一种仿刺参育种全基因组50k snp芯片及应用
技术领域:
1.涉及分子生物学、功能基因组学、生物信息学和分子育种领域,具体涉及一种与生长性状相关的snp位点筛选,与仿刺参的基因组snp芯片及制备方法,同时还涉及这种仿刺参snp芯片的用途。
背景技术:
2.分子标记(molecular markers),是以个体间遗传物质内核苷酸序列变异为基础的遗传标记,是dna水平遗传多态性的直接的反映。与其他几种遗传标记——形态学标记、生物化学标记、细胞学标记相比,dna分子标记具有的优越性有:大多数分子标记为共显性,对隐性的性状的选择十分便利;基因组变异极其丰富,分子标记的数量几乎是无限的;检测手段简单、迅速。随着分子生物学技术的发展,dna分子标记技术已有数十种,广泛应用于遗传育种、基因组作图、基因定位、物种亲缘关系鉴别、基因库构建、基因克隆等方面。遗传标记经历了4个发展阶段:形态学水平;细胞学和染色体水平;蛋白质和同工酶标记水平;dna分子水平(朱玉贤,李毅,郑晓峰,郭红卫.现代分子生物学(第5版)[j]. 生命世界,2019(07):2.)。dna分子标记相对之前的标记具有显著的优势:以dna的形式表现,不受发育阶段和外界环境因素的限制,在生物体的各组织均可进行检测;标记在全基因组中呈均匀分布,数目多,多态性高,且在自然界中普遍存在,不需对生物进行人为改造,不会影响物种的自然状态,dna分子标记的检测手段快速简便(李为民,李思锋,黎斌.dna分子标记在野生大豆遗传多样性研究中的应用进展[j].中国农学通报,2014,30(21):246
‑
250.);dna标记多为选择中性,不会对生物性状造成影响。
[0003]
以单核苷酸多态性(snp)标记为代表的dna分子标记技术,是分子标记技术的一种。与已有的标记相比较,snp标记技术的具有显著的优势(朱向博,张丽.单核苷酸多态性及其在畜牧兽医领域的研究进展[j].现代畜牧兽医,2020(07):48
‑
51.):由于在二倍体生物中snp位点多以二等位多态的形式存在,因而较容易估计出各位点的等位基因的频率;由于其单核苷酸突变的特性,因而其在全基因范围内大量存在,与应用较为广泛的dna分子标记
‑
微卫星标记相比,snp标记在基因组中的分布更为广泛,并且更为稳定;对位于编码区内的snp突变,可分为同义突变和非同义突变两类,非同义突变的snp由于改变了基因转录翻译产物的蛋白质序列,可能会造成蛋白质结构或基因表达水平的改变,因此,对于编码区snp的研究意义显得更为重大;snp标记的筛查过程可实现快速、规模化,后续的数据分析过程基本为自动化,对缩短研究周期十分重要(方宣钧,农业,吴为人,唐纪良,生物遗传学.作物dna标记辅助育种[m].科学出版社,2001.);snp标记在全基因组水平呈现不均匀分布的状态,自然界内的snp突变绝大多数发生在基因组的非编码区。随着高通量测序技术的发展,基于测序手段的snp筛查逐渐成为研究人员关注的热点。
[0004]
全基因组重测序,虽能获取最为全面的基因组变异信息,但若应用于上百乃至上千个体的大规模分析,其测序成本仍较高。简化基因组或低深度重测序技术的出现,虽较好地降低了测序成本,但由于其测序位点的随机性,难于实现对许多重要性状相关的已知基
因或基因通路的全面覆盖和分型(ruiqiang li,yingrui li,xiaodong fang,huanmingyang,jian wang,karsten kristiansen,jun wang.snp detection for massively parallelwhole
‑
genome resequencing[j].cold spring harbor laboratorypress,2009,19(6).xiangyang xu,guihua bai.whole
‑
genome resequencing:changingthe paradigms of snp detection,molecular mapping and gene discovery[j].molecularbreeding,2015,35(1).)。基因芯片技术是一种准确率高、重复性好的靶位点分型技术 ([1]jay shendure,robi d.mitra,chris varma,george m.church.advanced sequencingtechnologies:methods and goals[j].nature reviews genetics,2004,5(5).),在模式生物或农作物、畜禽的育种研究中应用广泛(andreas kranis,almas a gheyas,clarissaboschiero,frances turner,le yu,sarah smith,richard talbot,ali pirani,fionabrew,pete kaiser,paul m hocking,mark fife,nigel salmon,janet fulton,tim mstrom,georg haberer,steffen weigend,rudolf preisinger,mahmood gholami,saberqanbari,henner simianer,kellie a watson,john a woolliams,david w burt. development of a high density 600k snp genotyping array for chicken[j].biomedcentral,2013,14(1).),但对仿刺参等养殖品种仍然缺乏成熟的商业化芯片(shikailiu,luyang sun,yun li,fanyue sun,yanliang jiang,yu zhang,jiaren zhang,jianbinfeng,ludmilla kaltenboeck,huseyin kucuktas,zhanjiang liu.development of thecatfish 250k snp array for genome
‑
wide association studies[j].biomedcentral,2014,7(1).),定制费用较为昂贵,且固相芯片难以满足位点灵活选择的应用需求。hd
‑
marker技术是基于液相分子杂交的基因分型技术。该技术通过在单个pcr管内的高集成度探针杂交
‑
延伸
‑
连接反应,实现对多达上万个已知基因变异位点进行高通量筛查和分析相比固相芯片平台,由于hd
‑
marker是基于引物池的方式进行芯片的合成,可根据实际研究需求增减位点,具有更高的使用灵活性。该技术将液相杂交反应的位点选择灵活性和高通量测序平台通量高、成本低的优势有效地结合,突破了目前固相定制芯片平台费用高昂、灵活性差、难于大规模应用等技术瓶颈,为非模式生物提供了一种兼容不同通量级别、不同标记类型的高效灵活的靶向基因分型技术。(lv j,jiao w,guo h,et al. hd
‑
marker:a highly multiplexed and flexible approach for targeted genotyping ofmore than 10,000genes in a single
‑
tube assay.genome research,2018,28(12): 1919
‑
1930.zhu x,wang j,lv j,et al.sequencing
‑
based transcriptome
‑
wide targetedgenotyping for evolutionary and ecological studies.evolutionary bioinformatics, 2019,15(1176934319836074.)由于hd
‑
marker在靶向精准性、灵活性、和成本等具有明显的优势,在仿刺参分子育种研究中是一种具有应用潜力的高通量标记检测技术。
[0005]
仿刺参(apostichopus japonicus),又称刺参,隶属于棘皮动物门(echinodermata)、海参纲(holothuroideaia)、楯手目(aspidochirotidae)、仿刺参科(stichopodidae)、仿刺参属(apostichopus)(廖玉麟.中国动物志:棘皮动物门.海参纲[m].科学出版社,1997.)。仿刺参养殖是世界水产养殖业中一个非常重要的组成部分,自2003年以来刺参的养殖总产量和面积一直处于持续增长的态势,到2019年,增养殖面积达到24.67万公顷,年产量达到17.17万吨,直接经济产值逾300亿元,成为我国海水养殖单品种产值较高的种类之一,刺参养殖也成为继藻、对虾、贝、鱼之后第五次新的海水养殖浪潮
的主体部分,成为沿海渔业经济结构的支柱性产业。据不完全统计,目前刺参养殖业吸纳从业人员70万人,积聚了庞大的资金投入,同时也带动了加工、饲料、保健食品等相关产业的发展,为沿海经济结构调整和渔民就业、增收开辟了一条新的途径。([1]廖梅杰,王印庚, 李彬,荣小军,范瑞用,刘清兵.我国海参养殖产业现状、存在问题及对策探讨(上)[j].科学养鱼,2021(02):24
‑
25.),但伴随着这种爆炸性的养殖规模的扩增,导致由近亲繁殖造成的日渐严重的近交衰退(p a hohenlohe等,2010),仿刺参种质资源严重衰退,严重影响了仿刺参养殖行业的发展。另外,仿刺参对温度变化极其敏感,其生理活动随水温的变化而发生改变。通常在水温较高,即当所处水体温度达到18℃时,仿刺参的摄食及运动会开始有所降低;当水温达到20℃以上时,仿刺参的生活会发生改变,它们会选择移动到水深较深处的礁石中间,摄食量和运动量会降低到很低的水平,甚至停止进食和运动,进入夏眠的状态(f li等,1996)。夏眠是仿刺参的一个标志性的生物学特性,是个体为适应高温环境而产生的一种维持基本生存的自我保护性应激活动(f li等,1996;y liu等, 1996)。2018年夏季,辽宁多地持续高温,局部地区最高气温甚至突破了40摄氏度,使得海参养殖产业遭遇了前所未有的打击。辽宁省海洋渔业厅透露,全省海参养殖面积184.4 万亩,其中池塘养殖面积98.5万亩。目前初步统计,海参受灾损失面积95万亩,损失产量6.8万吨,直接经济损失68.7亿元人民币。
[0006]
针对上述,以改良种质资源为目的的分子生物学手段在棘皮动物中应用的需求显得尤为重要和急迫。然而目前仿刺参的分子育种研究中仍然缺乏稳定高效的液相芯片,本发明基于hd
‑
marker技术设计的液相芯片,能够为仿刺参经济性状研究、品种鉴定以及标记辅助育种提供重要的技术手段,可以满足仿刺参大规模商业化育种的需求。
技术实现要素:
[0007]
本发明的目的就在于snp芯片技术一直以来是经济养殖动物或农作物研究中靶位点分型便捷高效的工具,但是在仿刺参等非模式生物中尚未有可用的商业化snp芯片。为开展仿刺参经济性状选择等相关遗传育种工作提供可靠的技术平台,促进我国仿刺参分子育种的发展,同时也为其他水产生物的芯片的设计和开发提供初步的理论参考和方法指导。本研究对仿刺参不同地域的群体进行了高通量的snp标记的开发,进行了生长,抗高温性状的精细定位;初步设计和开发了包含50k位点数目的snp芯片,可以快捷地对仿刺参性状遗传参数准确评估,为指导研究人员开展仿刺参的分子育种工作提供高效可靠的技术手段,满足仿刺参优良品种培育工程的要求。
[0008]
本发明提供了一种仿刺参育种全基因组50k snp芯片,所述的snp芯片包括用于仿刺参育种的snp标记组合以及用于仿刺参育种的液相育种芯片,所述用于仿刺参抗高温性状育种的snp标记组合,由48755个snp位点组成,snp所在核苷酸序列分别为seq no.001
‑
seqid no.48755所示序列,长度为49bp,所述用于仿刺参育种的液相育种芯片,由48755对探针序列,每个snp位点对应两条探针序列,分别为forward探针和reverse探针。
[0009]
本发明同时提供了一种仿刺参育种全基因组50k snp芯片的制备方法以及该芯片相关的应用,具体包括仿刺参全基因组育种芯片在不同群体仿刺参的遗传背景分析中的作用,仿刺参全基因组育种芯片在仿刺参性状关联分析中的应用。
[0010]
一种仿刺参抗高温育种低密度12k snp芯片的制备方法,包括以下步骤:
[0011]
1、构建仿刺参样品群体
[0012]
在辽宁、山东不同海域,随机选取500只仿刺参样品,取组织放置于95%乙醇中带回实验室保存备用。
[0013]
2、仿刺参全基因组范围的snp分型
[0014]
2.1dna提取
[0015]
①
在1.5ml的管中加入500ul ste裂解缓冲液(100mm nacl;10mm tris
‑
cl,ph8.0; 1mm edta,ph8.0),50ul 10%sds,3.5ul蛋白酶k(20mg/ml),16ul rnase a(100mg/ml),取扇贝闭壳肌约0.1克,加入,剪碎,研磨棒研磨,研磨至絮状,56℃处理约2h,期间每隔30mins颠倒混匀一次,最终裂解液澄清状态。
[0016]
②
加入500ul的tris饱和酚,100ul氯仿/异戊醇(24:1),轻轻晃动20min,室温 12000rpm离心10分钟。
[0017]
③
抽取上清液至新的1.5ml的ep管中,加入300ultris饱和酚,300ul氯仿/异戊醇 (24:1),轻轻晃动20mins,室温12000rpm离心10分钟。
[0018]
④
重复步骤
③
两至三遍,直至无蛋白层为止。
[0019]
⑤
抽取上清,加入等体积氯仿/异戊醇,约500ul,轻轻晃动20min,室温8000rpm,常温离心10分钟。
[0020]
⑥
取上清加入1ml的冰无水乙醇,50ul醋酸钠(3m),
‑
20℃放置40min,12000rpm 离心10min,使核酸沉淀。
[0021]
⑦
弃上清,70%乙醇洗涤沉淀2次,每次8000rpm低温离心5min。
[0022]
⑧
干燥至乙醇全部挥发,加入30ul ddh2o溶解,再加0.75ul rnase于37℃消化rna 1.5h。
[0023]
⑨
利用qubit试剂盒进行对dna进行定量,1%琼脂糖凝胶电泳检测dna质量。提取的 dna放置在
‑
20℃保存备用。
[0024]
2.2建库及测序
[0025]
利用covaris破碎仪将提取的仿刺参基因组dna进行打断处理,打断范围设置在 350bp左右,利用基因组dna建库试剂盒进行dna片段的末端修复加a,然后两端连接上接头后进行扩增,利用带有barcode的引物进行文库扩增,完成文库的构建。利用qubit 2.0 进行文库定量。寄送至测序公司对文库的插入片段大小及文库的有效浓度进行定量,质检合格后在illumina hiseq x ten pe150平台进行测序。
[0026]
2.3重测序数据处理及比对分型
[0027]
①
参考基因组建立索引
[0028]
使用bwa软件的index命令、samtools的index命令、picard的 createsequencedictionary.jar构建参考序列的索引。
[0029]
②
序列比对
[0030]
利用bwa
‑
mem命令将双末端测序reads进行比对,生成bam文件,利用samtools的sort命令进行排序,生成排序的bam文件。
[0031]
③
去除pcr duplicate
[0032]
由于pcr过程中可能产生的偏好性,某些位置的片段会被过度扩增,从而使该位置上有大量冗余序列,造成分型的错误,因此要去除这些pcr duplicate,消除由于pcr实验过
程中产生的假阳性序列。利用picard markduplicates.jar命令,通过设置参数 remove_duplicates=true来丢弃duplicated序列。
[0033]
④
建立bam文件的索引
[0034]
利用samtools index对每个个体生成的bam文件建立索引,为后续gatk流程做好文件准备。
[0035]
⑤
gatk分型
[0036]
使用gatk软件中适用于群体变异检测的haplotypecaller模块对所有的样本进行变异检测,该方法首先使每样本生成一个gvcf文件,然后再进行群体的joint
‑
genotype,依据群体的变异信息校正个体的变异和基因型数据。
[0037]
3、仿刺参50k snp标记筛选
[0038]
3.1snp标记初步过滤
[0039]
按照下述步骤对生成的原始变异位点依次进行过滤,生成高质量的snp数据集。
[0040]
①
挑选出二态性snp位点
[0041]
②
过滤掉snp过于密集的区域,即10bp window内超过3的snp位点(bowen et al., 2011)
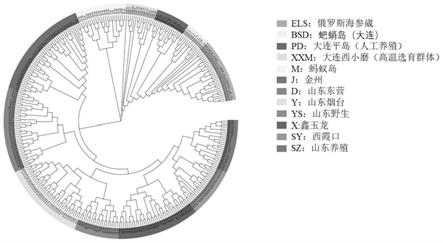
[0042]
③
根据官网推荐的hard filtering的过滤参数,过滤掉低质量snp位点,即qd<2.0, fs>20.0,mq<40.0,dp<6.0,dp>1000.0,mqranksum<
‑
12.5,readposranksum<
‑
8.0。
[0043]
④
过滤最小等位基因小于0.05的位点。
[0044]
⑤
最后得到高质量967万snp,进行下一步低密度snp选择。
[0045]
3.2snp标记优化选择
[0046]
估计动物个体分子育种可靠的遗传参数,需要进一步在初步过滤的snp中筛选一组适用的snp。一般需要两个条件:第一,筛选的snp是该物种不同地理群体共有的snp;第二,挑选的snp具有较高的信息含量,可以对个体snp基因效应及育种值进行准确评估。
[0047]
①
筛选出不同群体共有snp,然后在这些共有snp中减少或删除处于高连锁不平衡的 snp,我们采用ld的r2>0.35作为删除snp尺度,结果表明在保持snp育种参数估计准确性的前提下,使用这个尺度来筛选snp,可以明显降低所需snp标记的数目。
[0048]
②
筛选高信息量的snp可以依据不同的统计指标,根据wright的fst、snp基因频率的平均欧式距离、信息熵等综合指标,构建具有约束条件下snp选择最优化模型,利用r 软件优化求解包,获得snp基因频率含有高信息量snp,且snp能较均匀分布基因组上。
[0049]
③
根据已有文献报道的已克隆的仿刺参重要生长,抗高温性状相关基因的与功能相关的snp位点,利用gwas关联分析,通过gwas pval值得到与皮重、抗高温性状相关显著的snp位点。
[0050]
④
使用snpeff软件对高质量的snp进行注释,确定snp所在的基因元件,及对氨基酸的变化影响等。
[0051]
最后,根据第一至第四步,选择后snp总数控制到5万个标记左右。
[0052]
4、hd
‑
marker高密度芯片的设计和开发探针设计
[0053]
4.1靶向性探针的的设计和筛选
[0054]
根据hd
‑
marker探针池的设计思路,选择snp位点上游22bp的碱基序列及下游22bp 序列作为位点的特异探针。按照以下hd
‑
marker探针设计原则进行侧翼探针的设计和评估:
[0055]
·
侧翼探针序列需要满足gc含量在40%
‑
60%之间
[0056]
·
tm值在55~65℃之间,
[0057]
·
侧翼探针内不能有超过5个连续碱基的区域
[0058]
·
侧翼序列匹配度在80%以上的区域不能大于5处
[0059]
·
探针侧翼序列内的变异位点个数不超过3个。
[0060]
最终符合设计标准的位点数目为48755,对通过设计标准的位点信息进行整合,形成了一个包含位点信息、探针序列以及注释信息的hd
‑
marker液相芯片池。芯片上来源于基因区上的位点22778个,覆盖的基因数目为6955个,基因间区的位点为25977个。
[0061]
仿刺参液相芯片上的位点分布
[0062][0063]
4.2探针池的序列合成
[0064]
为使后续的pcr扩增中有引物结合的靶位点,将上游探针的5’端和下游探针的3’端都分别连接一段22bp的illumina平台测序通用的引物序列,形成forward探针和 reverse探针。
[0065]
以illumina测序平台为例,侧翼杂交探针forward的结构为:
[0066]
cctacacgacgctcttccgatctxxxxxxxxxxxxxxxxxxxxxx,侧翼杂交探针reverse的结构为: xxxxxxxxxxxxxxxxxxxxxxagatcggaagagcacacgtctga。其中x和y代表位点两侧的特异性序列。
[0067]
将所有位点的f探针集合形成一个f探针池,所有位点的r探针集合形成一个r探针池,合成f探针池和r探针池获得48k位点的液相芯片池。
[0068]
4.3仿刺身芯片的检测
[0069]
①
仿刺参dna提取:利用天根植物基因组提取试剂盒(rt405
‑
12)提取肌肉组织的基因组dna,
[0070]
②
dna样品质量检测:利用1%的琼脂糖凝胶电泳检测dna条带的完整性;用nanodrop 微量核酸定量仪检测浓度,将dna浓度调整到100ng/ul.
[0071]
③
hd
‑
marker芯片检测:参照hd
‑
marker标准实验流程,制备100个仿刺参dna样品的hd
‑
marker文库。利用qubit4分光光度计检测文库的浓度,在8.9
‑
10.6ng/ul之间,文库浓度均匀,质量符合测序要求。
[0072]
④
芯片性能检测分析:
[0073]
液相芯片效能分析:芯片的效能从位点的靶向性、捕获率、准确率及均一性等几个指标进行评价。结果显示,在所有样品中位点的捕获效率均能达到97%以上,分型位点比例
均在95%以上,位点测序深度上具有较高的一致性,重复样品深度一致性的皮尔逊系数均能达到在0.96以上。与标准wgs文库数据相比,位点分型准确性也在90%
‑
94%之间。结果表明仿刺参的50k液相芯片具有较好的分型效果。
[0074]
一种仿刺参抗高温育种低密度12k snp芯片的应用,包括下列步骤:
[0075]
1、不同群体仿刺参的遗传背景分析:筛选群体分型率大于90%,最小等位基因频率大于0.05的位点,得到46232个高质量位点的基因型信息46232个,利用分型数据对仿刺参的个体聚类分析。
[0076]
2、仿刺参性状关联分析中的应用:利用我们开发的仿刺参50ksnp芯片,对仿刺参生长、抗逆性状相关位点进行全基因组分析,进而证明了证明了50ksnp芯片覆盖了仿刺参生长、抗逆性状相关的突变位点和标记。
[0077]
本发明的有益效果:
[0078]
(1)本发明的液相芯片靶向性好,位点测序深度上具有较高的一致性,重复样品深度一致性的皮尔逊系数均能达到在0.96以上,位点分型准确性高,具有较好的分型效果。
[0079]
(2)本发明可通过筛选群体分型率大于90%,最小等位基因频率大于0.05的位点,得到46232个高质量位点的基因型信息46232个,并利用分型数据对仿刺参的个体聚类分析,其结果清楚的显示了仿刺参芯片覆盖的snp位点在仿刺参群体中具有较好的多态性,可以应用于仿刺参材料的遗传背景分析,是一种群体通用的snp芯片。
[0080]
(3)本发明可利用仿刺参50ksnp芯片,对仿刺参生长、抗逆性状相关位点进行全基因组分析,其p<1e
‑
07的显著位点有7个,定位于3号连锁群和11号连锁群,证明了50ksnp 芯片覆盖了仿刺参生长、抗逆性状相关的突变位点和标记。
[0081]
(4)本发明可为开展仿刺参抗高温性状选择等相关遗传育种工作提供可靠的技术平台,促进我国水产养殖业的发展,同时也为其他水产生物的芯片的设计和开发提供初步的理论参考和方法指导。
附图说明:
[0082]
为了易于说明,本发明由下述的具体实施及附图作以详细描述。
[0083]
图1为本发明的仿刺参样本系统进化树;
[0084]
图2为本发明的全基因组分析曼哈顿图;
具体实施方式:
[0085]
实施例1:仿刺参全基因组范围的50ksnp芯片的开发
[0086]
1、构建仿刺参样品群体
[0087]
在辽宁、山东不同海域,随机选取500只仿刺参样品,取组织放置于95%乙醇中带回实验室保存备用。
[0088]
2、仿刺参全基因组范围的snp分型
[0089]
2.1dna提取
[0090]
①
在1.5ml的管中加入500ul ste裂解缓冲液(100mm nacl;10mm tris
‑
cl,ph8.0; 1mm edta,ph8.0),50ul 10%sds,3.5ul蛋白酶k(20mg/ml),16ul rnase a(100mg/ml),取扇贝闭壳肌约0.1克,加入,剪碎,研磨棒研磨,研磨至絮状,56℃处理约2h,期间每隔30mins
颠倒混匀一次,最终裂解液澄清状态。
[0091]
②
加入500ul的tris饱和酚,100ul氯仿/异戊醇(24:1),轻轻晃动20min,室温 12000rpm离心10分钟。
[0092]
③
抽取上清液至新的1.5ml的ep管中,加入300ultris饱和酚,300ul氯仿/异戊醇 (24:1),轻轻晃动20mins,室温12000rpm离心10分钟。
[0093]
④
重复步骤
③
两至三遍,直至无蛋白层为止。
[0094]
⑤
抽取上清,加入等体积氯仿/异戊醇,约500ul,轻轻晃动20min,室温8000rpm,常温离心10分钟。
[0095]
⑥
取上清加入1ml的冰无水乙醇,50ul醋酸钠(3m),
‑
20℃放置40min,12000rpm 离心10min,使核酸沉淀。
[0096]
⑦
弃上清,70%乙醇洗涤沉淀2次,每次8000rpm低温离心5min。
[0097]
⑧
干燥至乙醇全部挥发,加入30ul ddh2o溶解,再加0.75ul rnase于37℃消化rna 1.5h。
[0098]
⑨
利用qubit试剂盒进行对dna进行定量,1%琼脂糖凝胶电泳检测dna质量。提取的 dna放置在
‑
20℃保存备用。
[0099]
2.2建库及测序
[0100]
利用covaris破碎仪将提取的仿刺参基因组dna进行打断处理,打断范围设置在 350bp左右,利用基因组dna建库试剂盒进行dna片段的末端修复加a,然后两端连接上接头后进行扩增,利用带有barcode的引物进行文库扩增,完成文库的构建。利用qubit 2.0 进行文库定量。寄送至测序公司对文库的插入片段大小及文库的有效浓度进行定量,质检合格后在illumina hiseq x ten pe150平台进行测序。
[0101]
2.3重测序数据处理及比对分型
[0102]
①
参考基因组建立索引
[0103]
使用bwa软件的index命令、samtools的index命令、picard的 createsequencedictionary.jar构建参考序列的索引。
[0104]
②
序列比对
[0105]
利用bwa
‑
mem命令将双末端测序reads进行比对,生成bam文件,利用samtools的 sort命令进行排序,生成排序的bam文件。
[0106]
③
去除pcr duplicate
[0107]
由于pcr过程中可能产生的偏好性,某些位置的片段会被过度扩增,从而使该位置上有大量冗余序列,造成分型的错误,因此要去除这些pcr duplicate,消除由于pcr实验过程中产生的假阳性序列。利用picard markduplicates.jar命令,通过设置参数 remove_duplicates=true来丢弃duplicated序列。
[0108]
④
建立bam文件的索引
[0109]
利用samtools index对每个个体生成的bam文件建立索引,为后续gatk流程做好文件准备。
[0110]
⑤
gatk分型
[0111]
使用gatk软件中适用于群体变异检测的haplotypecaller模块对所有的样本进行变异检测,该方法首先使每样本生成一个gvcf文件,然后再进行群体的joint
‑
genotype,依
据群体的变异信息校正个体的变异和基因型数据。
[0112]
3、仿刺参50k snp标记筛选
[0113]
3.1snp标记初步过滤
[0114]
按照下述步骤对生成的原始变异位点依次进行过滤,生成高质量的snp数据集。
[0115]
①
挑选出二态性snp位点
[0116]
②
过滤掉snp过于密集的区域,即10bp window内超过3的snp位点(bowen et al., 2011)
[0117]
③
根据官网推荐的hard filtering的过滤参数,过滤掉低质量snp位点,即qd<2.0, fs>20.0,mq<40.0,dp<6.0,dp>1000.0,mqranksum<
‑
12.5,readposranksum<
‑
8.0。
[0118]
④
过滤最小等位基因小于0.05的位点。
[0119]
⑤
最后得到高质量967万snp,进行下一步低密度snp选择。
[0120]
3.2snp标记优化选择
[0121]
估计动物个体分子育种可靠的遗传参数,需要进一步在初步过滤的snp中筛选一组适用的snp。一般需要两个条件:第一,筛选的snp是该物种不同地理群体共有的snp;第二,挑选的snp具有较高的信息含量,可以对个体snp基因效应及育种值进行准确评估。
[0122]
①
筛选出不同群体共有snp,然后在这些共有snp中减少或删除处于高连锁不平衡的 snp,我们采用ld的r2>0.35作为删除snp尺度,结果表明在保持snp育种参数估计准确性的前提下,使用这个尺度来筛选snp,可以明显降低所需snp标记的数目。
[0123]
②
筛选高信息量的snp可以依据不同的统计指标,根据wright的fst、snp基因频率的平均欧式距离、信息熵等综合指标,构建具有约束条件下snp选择最优化模型,利用r 软件优化求解包,获得snp基因频率含有高信息量snp,且snp能较均匀分布基因组上。
[0124]
③
根据已有文献报道的已克隆的仿刺参重要生长,抗高温性状相关基因的与功能相关的snp位点,利用gwas关联分析,通过gwas pval值得到与皮重、抗高温性状相关显著的snp位点。
[0125]
④
使用snpeff软件对高质量的snp进行注释,确定snp所在的基因元件,及对氨基酸的变化影响等。
[0126]
最后,根据第一至第四步,选择后snp总数控制到5万个标记左右。
[0127]
4、hd
‑
marker高密度芯片的设计和开发探针设计
[0128]
4.1靶向性探针的的设计和筛选
[0129]
根据hd
‑
marker探针池的设计思路,选择snp位点上游22bp的碱基序列及下游22bp 序列作为位点的特异探针。按照以下hd
‑
marker探针设计原则进行侧翼探针的设计和评估:
[0130]
·
侧翼探针序列需要满足gc含量在40%
‑
60%之间
[0131]
·
tm值在55~65℃之间,
[0132]
·
侧翼探针内不能有超过5个连续碱基的区域
[0133]
·
侧翼序列匹配度在80%以上的区域不能大于5处
[0134]
·
探针侧翼序列内的变异位点个数不超过3个。
[0135]
最终符合设计标准的位点数目为48755,对通过设计标准的位点信息进行整合,形成了一个包含位点信息、探针序列以及注释信息的hd
‑
marker液相芯片池。芯片上来源于基因区上的位点22778个,覆盖的基因数目为6955个,基因间区的位点为25977个。
[0136]
仿刺参液相芯片上的位点分布
[0137][0138]
4.2探针池的序列合成
[0139]
为使后续的pcr扩增中有引物结合的靶位点,将上游探针的5’端和下游探针的3’端都分别连接一段22bp的illumina平台测序通用的引物序列,形成forward探针和 reverse探针。
[0140]
以illumina测序平台为例,侧翼杂交探针forward的结构为:
[0141]
cctacacgacgctcttccgatctxxxxxxxxxxxxxxxxxxxxxx,侧翼杂交探针reverse的结构为: xxxxxxxxxxxxxxxxxxxxxxagatcggaagagcacacgtctga。其中x和y代表位点两侧的特异性序列。
[0142]
将所有位点的f探针集合形成一个f探针池,所有位点的r探针集合形成一个r探针池,合成f探针池和r探针池获得48k位点的液相芯片池。
[0143]
4.3仿刺身芯片的检测
[0144]
①
仿刺参dna提取:利用天根植物基因组提取试剂盒(rt405
‑
12)提取肌肉组织的基因组dna,
[0145]
②
dna样品质量检测:利用1%的琼脂糖凝胶电泳检测dna条带的完整性;用nanodrop 微量核酸定量仪检测浓度,将dna浓度调整到100ng/ul.
[0146]
③
hd
‑
marker芯片检测:参照hd
‑
marker标准实验流程,制备100个仿刺参dna样品的hd
‑
marker文库。利用qubit4分光光度计检测文库的浓度,在8.9
‑
10.6ng/ul之间,文库浓度均匀,质量符合测序要求。
[0147]
④
芯片性能检测分析:
[0148]
液相芯片效能分析:芯片的效能从位点的靶向性、捕获率、准确率及均一性等几个指标进行评价。结果显示,在所有样品中位点的捕获效率均能达到97%以上,分型位点比例均在95%以上,位点测序深度上具有较高的一致性,重复样品深度一致性的皮尔逊系数均能达到在0.96以上。与标准wgs文库数据相比,位点分型准确性也在90%
‑
94%之间。结果表明仿刺参的50k液相芯片具有较好的分型效果。
[0149]
实施例2:仿刺参50k snp芯片在分子育种中应用
[0150]
为了验证仿刺参芯片在仿刺参分子育种中应用效果,我们使用50k芯片对来自俄罗斯(30个)、大连(100个)、山东(100个)的样本进行检测,并进行分子育种中:
[0151]
(1)不同地理群体遗传背景分析;(2)与生长相关的仿刺参肉刺个数进行全基因组关联分析(gwas)
[0152]
1、仿刺参全基因组育种芯片在不同群体仿刺参的遗传背景分析中的作用:
[0153]
筛选群体分型率大于90%,最小等位基因频率大于0.05的位点,得到46232个高质量位点的基因型信息46232个,利用分型数据对仿刺参的个体聚类分析,如图1显示,样本中12个群体在系统进化树上的位置清晰,分类明确。该结果显示,仿刺参芯片覆盖的snp 位点在仿刺参群体中具有较好的多态性,可以应用于仿刺参材料的遗传背景分析,是一种群体通用的snp芯片。
[0154]
2、仿刺参全基因组育种芯片在仿刺参性状关联分析中的应用:
[0155]
利用本技术方案的仿刺参50ksnp芯片,对仿刺参生长、抗逆性状相关位点进行全基因组分析,结果如图2所示,p<1e
‑
07的显著位点有7个,定位于3号连锁群和11号连锁群,证明了50ksnp芯片覆盖了仿刺参生长、抗逆性状相关的突变位点和标记。
[0156]
以上显示和描述了本发明的基本原理和主要特征和本发明的优点,本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下,本发明还会有各种变化和改进,这些变化和改进都落入要求保护的本发明范围内,本发明要求保护范围由所附的权利要求书及其等效物界定。
[0157]
序列表
[0158]
[0159]
[0160]
[0161]
[0162]
[0163]
[0164]
[0165]
[0166]
[0167]
[0168]
[0169]
[0170]
[0171]
[0172]
[0173]
[0174]
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。