一种基于snv array技术的药物基因突变检测试剂盒及检测方法
技术领域
1.本发明属于基因检测领域,具体涉及一种基于snv array技术的药物基因突变检测试剂盒及检测方法。
背景技术:
2.研究表明,药物有效性在不同个体间反应差异很大,特别是在高血压、糖尿病以及神经系统等疾病的治疗中,药物的有效率低于50%,这是药物个体化差异造成的。而且很多药物存在药物不良反应(adverse drug reaction, adr),据国家食品药品监督管理局(nmpa)发布的国家药品不良反应监测报告显示我国每年因adr住院人数超过250万人,死亡人数超过20万。引起药物个体差异的因素有很多,除年龄、合并药物或者疾病外,遗传因素也证实是影响药物个体化反应的重要因素。
3.药物基因检测主要对患者药物代谢酶和药物作用靶点基因进行检测,如果这两类基因发生变异,可能导致药物的动力学和药效学改变,因此针对不同病人携带的药物基因突变选择合适的药物种类和剂量,就可以做到个性化的药物治疗。
4.检测药物基因突变的方法有很多,包括荧光原位杂交(fish)、sanger测序、荧光定量pcr、核酸质谱、二代测序和基因芯片(主要包括acgh和snp array)。其中,fish、sanger、pcr类方法为低中通量技术手段,一次只能检测少量样本的一个或几个突变位点,耗时耗力,而且检测的灵敏度和特异性均不高,不利于高效预测药物疗效及指导药物。
5.cn108728522a和cn108707658a采用二代测序可以进行高通量的药基因检测,一次检测多个基因、多个样本,且缩短了检测流程和检测时间,然而基于引物设计的捕获技术往往会出现扩增均一性差,扩增区域gc含量差异大以及高度同源区域导致引物特异性低,引物之间相互影响大难以实现一管操作等问题,从而导致检测准确率难以保证。此外,目前的测序成本仍然较高,测序结果的解读还存在挑战,还无法有效地结合药物基因组学实现对患者用药的临床指导。
6.传统点样法制成的基因芯片法具有高通量的优点,但易出现假阳性、假阴性等缺点。而基因芯片中的snp array技术虽然广泛应用于临床,具有高准确性、操作简便且成本较低但是仅能检测拷贝数变异。最近出现的snv array技术是snp array技术的创新性衍生,通过将snp探针替换为snv探针,结合改进的分型算法,可以实现对单基因点突变进行检测。因此,本发明基于snv array技术,在保证了临床应用价值的基础上,开发了一种精准、快速、高效、便捷、经济的检测药物基因突变的试剂盒,积极的推动精准用药在临床的应用和普及。
技术实现要素:
7.鉴于现有药物基因突变检测手段存在的问题,本发明的目的之一在于提供一种基于snv array技术的药物基因突变检测试剂盒,能精准、高效、快速、便捷、经济的检测常见
的药物基因相关突变。
8.本发明的目的之二在于提供一种snv array技术检测药物基因突变的方法,该方法延续传统snp array技术的的高便捷度、敏感性和特异性,可有效克服现有药物基因检测技术的不足,为临床精准用药提供新的技术思路和个性化用药指导依据。
9.具体的,本发明所述试剂盒包括seq id no.1~ seq id no.63所示的探针组(表1),用于杂交捕获药物基因突变位点;表1 覆盖的药物基因突变探针组
核苷酸序列编号覆盖基因位置探针序列突变seqidno.1abcb1chr7:87531302taatcaatcatatttagtttgactcaccttcccag[a/c]accttctagttctttcttatctttcagtgcttgtcc.2677t>gseqidno.2abcb1chr7:87531302taatcaatcatatttagtttgactcaccttcccag[a/t]accttctagttctttcttatctttcagtgcttgtcc.2677t>aseqidno.3abcb1chr7:87509329aaggcatgtatgttggcctcctttgctgccctcac[a/t]atctcttcctgtgacaccacccggctgttgtctccc.3435t>aseqidno.4abcb1chr7:87509329aaggcatgtatgttggcctcctttgctgccctcac[a/g]atctcttcctgtgacaccacccggctgttgtctccc.3435t>cseqidno.5cyp2c19chr10:94781859tgcaataattttcccactatcattgattatttccc[a/g]ggaacccataacaaattacttaaaaaccttgctttc.681g>aseqidno.6cyp2c19chr10:94781859tgcaataattttcccactatcattgattatttccc[g/c]ggaacccataacaaattacttaaaaaccttgctttc.681g>cseqidno.7cyp2c19chr10:94761900tttttttttcaaatttgtgtcttctgttctcaaag[c/a]atctctgatgtaagagataatgcgccacgatgggcc.
‑
806c>aseqidno.8cyp2c19chr10:94761900tttttttttcaaatttgtgtcttctgttctcaaag[c/t]atctctgatgtaagagataatgcgccacgatgggcc.
‑
806c>tseqidno.9cyp2c19chr10:94781859tgcaataattttcccactatcattgattatttccc[g/c]ggaacccataacaaattacttaaaaaccttgctttc.681g>cseqidno.10cyp2c19chr10:94761900tttttttttcaaatttgtgtcttctgttctcaaag[c/a]atctctgatgtaagagataatgcgccacgatgggcc.
‑
806c>aseqidno.11htr2cchrx:114903581cagcttcagcttatctacagtgactttgctaccct[c/g]tcttgctgtcccttgacaaggcctactttaggctcg.114903581c>gseqidno.12cyp3a5chr7:99648291tctggggcacagctttcttgaagaccaaagtagaa[a/g]tccttagaataactcattctccacttagggttccac.219
‑
237a>gseqidno.13slco2b1chr12:21176804cttacagttacaggtattctaaagaaactaatatc[a/g]attcatcagaaaattcaacatcgaccttatccactc.388a>gseqidno.14slco2b1chr12:21176804cttacagttacaggtattctaaagaaactaatatc[a/t]attcatcagaaaattcaacatcgaccttatccactc.388a>tseqidno.15slco2b1chr12:21176804cttacagttacaggtattctaaagaaactaatatc[a/c]attcatcagaaaattcaacatcgaccttatccactc.388a>cseqidno.16slco2b1chr12:21176804cttacagttacaggtattctaaagaaactaatatc[a/t]attcatcagaaaattcaacatcgaccttatccactc.388a>tseqidno.17ephx1chr1:225838705catccacgtgaagcccccccagctgcccgcaggcc[a/t]taccccgaagcccttgctgatggtgcacggctggcc.416a>tseqidno.18ephx1chr1:225838705catccacgtgaagcccccccagctgcccgcaggcc[a/g]taccccgaagcccttgctgatggtgcacggctggcc.416a>gseqidno.19ephx1chr1:225838705catccacgtgaagcccccccagctgcccgcaggcc[a/g]taccccgaagcccttgctgatggtgcacggctggcc.416a>gseqidno.20pla2g4achr1:186880054agaactttggttaaagtttagttaacatagtccag[a/g]atcacagagctgttatgttttggggctggaactcac.115 9538a>gseqidno.21ugt1achr2:233772770cagtaaagatatttgaatatgtatcgtgccccctc[c/t]ggtgtctttgatcaggatgacatgtgccatttttcc.*211t>cseqidno.22ugt1achr2:233772898ctgtttgggagtgcgggattcaaaggtggtcccac[c/g]gctgcccctactgcaaatggcagttttaatcttatc.*339g>cseqidno.23ugt1achr2:233772898ctgtttgggagtgcgggattcaaaggtggtcccac[g/t]gctgcccctactgcaaatggcagttttaatcttatc.*339g>tseqidno.24ugt1achr2:233772999tccttaaccaataatggtcagtcctcatctctgtc[c/g]tgcttcataggtgccaccttgtgtgtttaaagaagc.*440g>c
seqidno.25ugt1achr2:233760235aaacattaacttggtgtatcgattggtttttgcca[tatatatata/tatatatatatata]agtaggagagggcgaacctctggcaggagcaaaggg.233760235tatatatatatata>tatatatataseqidno.26ugt1achr2:233760235aaacattaacttggtgtatcgattggtttttgcca[tatatatatata/tatatatatatata]agtaggagagggcgaacctctggcaggagcaaaggg.233760235tatatatatatata>tatatatatataseqidno.27ugt1achr2:233760235aaacattaacttggtgtatcgattggtttttgcca[tatatatatatata/tatatatatatatata]agtaggagagggcgaacctctggcaggagcaaaggg.233760235tatatatatatata>tatatatatatatataseqidno.28ugt1achr2:233772898ctgtttgggagtgcgggattcaaaggtggtcccac[g/t]gctgcccctactgcaaatggcagttttaatcttatc.*339g>tseqidno.29ugt1achr2:233760235aaacattaacttggtgtatcgattggtttttgcca[tatatatata/tatatatatatata]agtaggagagggcgaacctctggcaggagcaaaggg.233760235tatatatatatata>tatatatataseqidno.30cyp2d6chr22:42129130gacttcttgcccaggcccaagttgcgcaaggtgga[c/g]acggagaagcgcctctgctcgcgccacgcgggcccc.408g>cseqidno.31acechr17:63488529cccatcctttctcccatttctctagacctgctgcc[t/tatacagtcactttttttttttttttgagacggagtctcgctctgtcgccc]atacagtcacttttatgtggtttcgccaattttatc.2306
‑
105_2306
‑
104insseqidno.32acechr17:63488529cccatcctttctcccatttctctagacctgctgcc[t/tg]atacagtcacttttatgtggtttcgccaattttatc.2306
‑
105_2306
‑
104insseqidno.33acechr17:63488529cccatcctttctcccatttctctagacctgctgcc[t/tatacagtcactttttttttttttttgagacggagtctcgctctgtcgccc]atacagtcacttttatgtggtttcgccaattttatc.2306
‑
105_2306
‑
104insseqidno.34drd2chr11:113475529ccaaaggagctgtacctcctcggcgatccccggcc[t/ta]gaacgggtaggaggggttgggggattccgccatccg.113475529t>taseqidno.35drd2chr11:113475529ccaaaggagctgtacctcctcggcgatccccggcc[t/tg]gaacgggtaggaggggttgggggattccgccatccg.113475529t>tgseqidno.36drd2chr11:113475629accgcccactgcgctcccacccacacccagagtaa[c/t]aagctgtgattgcaggctgggtcctcaccgtctgcg.113475629t>cseqidno.37drd2chr11:113475529ccaaaggagctgtacctcctcggcgatccccggcc[t/ta]gaacgggtaggaggggttgggggattccgccatccg.113475529t>taseqidno.38crhr1chr17:45834159tcctcccctcccagggctgcctctctccctccctg[c/t]tcctaggtccctaggggtatgctgctgggagccccc.668 111c>tseqidno.39fkbp5chr6:35702206aaccctaacctctctggactcctacattttcctct[a/c/]tcttggtccaataactcctcactatctcgttaggtg.35702206a>cseqidno.40fkbp5chr6:35702206aaccctaacctctctggactcctacattttcctct[a/g]tcttggtccaataactcctcactatctcgttaggtg.35702206a>gseqidno.41fkbp5chr6:35702206aaccctaacctctctggactcctacattttcctct[a/t]tcttggtccaataactcctcactatctcgttaggtg.35702206a>tseqidno.42fkbp5chr6:35702206aaccctaacctctctggactcctacattttcctct[a/c]tcttggtccaataactcctcactatctcgttaggtg.35702206a>cseqidno.43vkorc1chr16:31093557atcctctgttccccgacctcccatcctagtccaag[a/g]gtcgatgatctcctggcaccgggcacctttggccac.1173c>tseqidno.44vkorc1chr16:31093557atcctctgttccccgacctcccatcctagtccaag[c/g]gtcgatgatctcctggcaccgggcacctttggccac.1173c>gseqidno.45vkorc1chr16:31096368aaaatgctaggattataggcgtgagccaccgcacc[c/g]ggccaatggttgtttttcaggtcttctcttgcttgc.
‑
1639g>cseqidno.46vkorc1chr16:31096368aaaatgctaggattataggcgtgagccaccgcacc[c/a]ggccaatggttgtttttcaggtcttctcttgcttgc.
‑
1639g>tseqidno.47vkorc1chr16:31096368aaaatgctaggattataggcgtgagccaccgcacc[c/t]ggccaatggttgtttttcaggtcttctcttgcttgc.
‑
1639g>aseqidno.48vkorc1chr16:31093557atcctctgttccccgacctcccatcctagtccaag[c/g]gtcgatgatctcctggcaccgggcacctttggccac.1173c>gseqidno.49vkorc1chr16:31096368aaaatgctaggattataggcgtgagccaccgcacc[c/g]ggccaatggttgtttttcaggtcttctcttgcttgc.
‑
1639g>cseqidno.50cyp1a1chr15:74724835ggtatttccagctccctgcagttggcaatctgtca[a/c]cctgattgtctcccagcaaaggacaaagaagatggc.
‑
30 606g>tseqidno.51hla_bchr6:30731607ctctgatgagcccccgtttaatctatttttcccac[c/g]tgtgcccccttctagagtataaataccttgagggcg.30731607g>cseqidno.52hla_bchr6:30978371atgggtcaggatgggcttctctgaggtctccaagg[a/g]attggccatagagatggtcatgatcaaaatcaagag.30978371g>aseqidno.53ugt1a4chr2:233718962ctgatggcagcccctggctcagcatgcgggaggcc[a/t]tgcgggagctccatgccagaggccaccaggcggtgg.233718962t>a
seqidno.54ugt1a4chr2:233718962ctgatggcagcccctggctcagcatgcgggaggcc[g/t]tgcgggagctccatgccagaggccaccaggcggtgg.233718962t>gseqidno.55ugt1a4chr2:233718962ctgatggcagcccctggctcagcatgcgggaggcc[a/t]tgcgggagctccatgccagaggccaccaggcggtgg.233718962t>aseqidno.56mc4rchr18:60215554acaggtcatgtcttaattctgttgtcattagttcc[c/a]gtttgttaaatgtttacagcgtggcaggtataccag.60215554c>aseqidno.57mc4rchr18:60215554acaggtcatgtcttaattctgttgtcattagttcc[c/t]gtttgttaaatgtttacagcgtggcaggtataccag.60215554c>tseqidno.58mc4rchr18:60215554acaggtcatgtcttaattctgttgtcattagttcc[c/a]gtttgttaaatgtttacagcgtggcaggtataccag.60215554c>aseqidno.59tpmtchr6:18130687tttatctatgtctcatttacttttctgtaagtaga[c/g]ataacttttcaaaaagacagtcaattccccaacttc.719a>gseqidno.60tpmtchr6:18130687tttatctatgtctcatttacttttctgtaagtaga[t/g]ataacttttcaaaaagacagtcaattccccaacttc.719a>cseqidno.61tpmtchr6:18130687tttatctatgtctcatttacttttctgtaagtaga[t/g]ataacttttcaaaaagacagtcaattccccaacttc.719a>cseqidno.62mt
‑
rnr1chrmt:1494acagggccctgaagcgcgtacacaccgcccgtcac[c/t]ctcctcaagtatacttcaaaggacatttaactaaam.1494c>tseqidno.63mt
‑
rnr1chrmt:1555tttaactaaaacccctacgcatttatatagaggag[a/g]caagtcgtaacatggtaagtgtactggaaagtgcam.1555a>g
所述探针组包含药物相关基因abcb1、cyp2c19、htr2c、cyp3a5、slco2b1、ephx1、pla2g4a、ugt1a、cyp2d6、ace、drd2、crhr1、fkbp5、vkorc1、cyp1a1、hla_b、ugt1a4、mc4r、tpmt、mt
‑
rnr1。
[0010]
优选地,所述探针组均通过与含有相应药物基因突变位点的dna片段以碱基互补配对原则进行杂交发挥作用。结果的判读基于该部分探针的检测值来实现的,所用的方法为genotype_new和es
‑
cluster聚类算法。
[0011]
常规的snp array技术对探针单色荧光标记,snv array方法是通过对snp探针 进行改造,采用双色荧光标记实现基因分型,所述探针会得到两种荧光检测值a和b, 通过对a和b值进行计算,可得到三种基因分型结果,aa、ab和bb,其中aa和 bb代表纯和类型,野生型或纯合子,需根据实际情况判读属于野生型或纯合子,ab 代表杂合子。计算公式如下:f(z)=(a
‑
b)/(a b)当f(z)≈1
±
0.05时,分型结果为aa;当f(z)≈
‑1±
0.05时,分型结果为bb;当f(z)≈0
±
0.05时,分型结果为ab;进一步的,所述es
‑
cluster聚类算法对基因分型结果进行聚类,计算公式如下:f(x)=log2(a/b);f(y)=[log2(a) log2(b)]/2;所述es
‑
cluster聚类算法以f(x)为横轴,f(y)为纵轴进行聚类,根据样本分型结 果和聚类数目判断检测结果属于阳性还是阴性。一般来说,三种分型聚类结果均包含 大量样本,说明该位点人群频率较高,考虑为snp,一般无临床意义;三种分型聚类 结果中包含极少量样本,说明该位点人群频率较低,考虑为阳性的药物基因变异位点 (图2),全部聚类为aa或bb中的一种结果,考虑该位点为野生型(图3)。
[0012]
所述探针组包含的上述药物基因突变位点中,每个设计1~2个探针,正义链和反义链方向均可设计,重复的探针用于增加检测的准确度和结果的可信度。
[0013]
进一步地,所述试剂盒还包括变性混合液、扩增混合液、片段化混合液、沉淀混合液和杂交混合液。
[0014]
所述变性混合液由10
×
变性液用纯水稀释10倍而成。
[0015]
所述扩增混合液由1体积的扩增酶加45体积的扩增液混合制备而成。
[0016]
所述片段化混合液由1体积的片段化酶、10.3体积的片段化稀释液加45.7体积的10
×
片段化缓冲液混合制备所得。
[0017]
所述沉淀混合液由1体积的沉淀溶液2加119体积的沉淀溶液1混合制备所得。
[0018]
所述杂交混合液由1体积的杂交液1、18体积的杂交液2和140体积的杂交缓冲液混合制备所得。
[0019]
优选地,所述试剂盒在cnvplus
®
微阵列芯片扫描仪中进行检测使用。
[0020]
所述cnvplus
®
微阵列芯片扫描仪配套试剂包括染料混合物a、染料混合物b、稳定混合物、连接混合物a和连接混合物b。
[0021]
所述染料混合物a由1体积的染色液1
‑
a、1体积的染色液1
‑
b、2体积的染色缓冲液和96体积的洗液a混合制备所得。
[0022]
所述染料混合物b由1体积的染色液2
‑
a、1体积的染色液2
‑
b、2体积的染色缓冲液和96体积的洗液a混合制备所得。
[0023]
所述稳定混合物由1体积的稳定液、8体积的固定稀释液用纯水稀释7.9倍而成。
[0024]
所述连接混合物a由1体积的连接液1、0.4体积的连接液2和5体积的连接缓冲液混合制备所得。
[0025]
所述连接混合物b由1体积的连接酶、6.6体积的探针混合液1、6.6体积的探针混合液2和52体积的连接混合物a混合制备所得。
[0026]
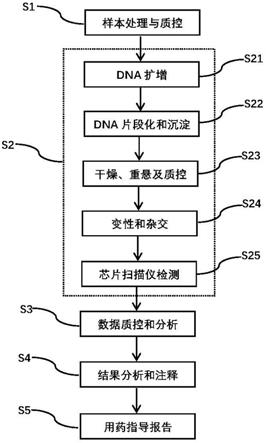
本发明的另一个目的是提供一种snv array技术检测药物基因突变的方法,所述方法包括以下步骤:s1:样品处理与质控:临床样品应先提取dna,然后通过凝胶电泳,nanodrop等方法进行质控,确保每个dna样品无降解,无杂质污染,高纯度。质控标准是光密度(optical density, od)260/280nm比值介于1.8~2.0,od 260/230nm比值介于1.5~2.0。其中胶上样染料为invitrogen
™
trackit cyan/orange loading buffer (invitrogen p/n 10482
‑
028);25bp invitrogen ladder (invitrogen p/n 10488
‑
022);凝胶电泳系统为invitrogen e
‑
gel
™ꢀ
48 agarose gels 4%,g8008
‑
04。
[0027]
s2:检测反应:s21:dna扩增:100ng dna样本(5 ng/
µ
l)中加入20μl变性混合液,室温孵育10min;加入130 μl中和液,震荡混匀后沿管壁缓慢加入230μl扩增混合液,震荡混匀1000rpm离心1min;在37℃孵育箱中孵育23
±
1h;65℃孵育箱中孵育20min;37℃孵育箱中继续孵育45min;s22:dna片段化和沉淀:对步骤s21孵育的样本加入57μl片段化混合液,混匀、简短离心后37℃孵育30min,而后在室温环境下加入19
µ
l 终止液,进行震荡混匀离心;室温环境下加入240
µ
l沉淀混合液和600
µ
l异丙醇,用移液器上下充分混匀;转移至
‑
20℃环境中放置16~24h;s23:干燥、重悬及质控:对步骤s22处理后的样本进行4℃ 3200rpm离心40min,去除废液,置于37℃杂交炉中20min晾干,加入35
µ
l重悬缓冲液进行振荡混匀,震荡仪中震荡10min,加入80
µ
l 杂交混合液,进行震荡混匀离心,产生115
µ
l变性杂交液;
质控使用10
µ
l的变性杂交液,包括用nanodrop测量吸光度和凝胶电泳,其中胶上样染料为invitrogen
™ꢀ
trackit cyan/orange loading buffer(invitrogen p/n 10482
‑
028);25bp invitrogen ladder(invitrogen p/n 10488
‑
022);凝胶电泳系统为invitrogen e
‑
gel
™ꢀ
48 agarose gels 4%,g8008
‑
04;s24:变性和杂交:将步骤s23产生的变性杂交液放于杂交板,将杂交板放置于pcr仪中进行变性程序:95℃变性10 min; 48℃变性3 min;冷却;使用移液器转移105
µ
l变性杂交液至杂交盘,载入cnvplus
®
微阵列芯片处理仪,持续杂交23.5h~24h;采用cnvplus
®
微阵列芯片扫描仪和自动分析软件,可以实现一次实验同时处理96个样本的检测通量,显著提高结果的可靠性。
[0028]
s25:芯片扫描仪检测:对步骤s24产生的混合物进行染料盘对应分装:其中染料混合物a的染料盘2个,每孔105
µ
l;染料混合物b的染料盘1个,每孔105
µ
l;稳定混合物的染料盘1个,每孔105
µ
l;连接混合物b的染料盘1个,每孔105
µ
l;将分装后的染料盘载入芯片扫描仪,同时向染料盘每孔加入150
µ
l固定缓冲液后避免接触扫描盘底部。将盖子的切口角对准扫描盘的切口边缘后盖住扫描,放置于工作台顶部进行检测;s3:数据质控和分析:成像完成后会得到每块杂交板的原始荧光信号数据,利用自动分析软件对其进行质控和分析,即可得到相应样品的变异位点信息结果文件。
[0029]
s4:结果分析与注释:结果包含每个样品检测到的药物基因突变位点信息,这些信息再经过进一步的注释分析,包括突变评级和医学解读,最终得到的明确的真实的位点信息。
[0030]
s5:用药指导报告:对于检测为阳性的样本发放用药指导报告,报告依据是临床药物基因组学实施联盟(cpic)公布的guideline。
[0031]
有益效果本发明提供的一种基于snv array技术检测药物基因突变的试剂盒,能够同时特异性杂交捕获常见药物基因相关变异类型,实现高效、精准、全面的药物基因突变检测。
[0032]
本发明提供的一种snv array技术检测药物基因突变的方法延续了snp array技术的的高便捷度、敏感性和特异性,又能够显著提升临床对药物基因突变检测的标准化程度,为临床精准用药提供新的技术思路和个性化用药指导依据。
附图说明
[0033]
图1为本发明的方法检测流程图;图2为药物基因突变阳性结果图;图3为药物基因突变阴性结果图。
具体实施方式
[0034]
下面结合具体实施例,进一步阐述本发明设计、阳性样本验证和结果分析。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。此外应理解,在阅读了本发明讲授的内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本技术所附权利要求书所限定的范围。
[0035]
实施例1
本实施例的一种基于snv array技术的药物基因突变检测的试剂盒,该试剂盒包括seq id no.1~ seq id no.63所示的探针组。
[0036]
上述试剂盒中,优选地,所述探针组的设计结合临床实验室的文献报道,针对药物基因相关的临床诊疗指南,结合clinvar、cpic和pharmagkb等数据库对这些基因相关位点进行人群频率的统计和筛选,确认了每个基因的中国人群遗传特征高发位点,并将这些位点合成相应探针并制成芯片,每个位点至少1个探针。部分位点是2个重复探针,这是为了在芯片不同位置放置相同的探针,从而增加对应基因位点的结果准确度和阳性检出率,筛选原则如下:(1)临床药物指南收录的中国人群高发位点;(2)clinvar数据库评级为drug response,证据强度为二星及以上;(3)cpic和pharmagkb数据库公布有guidelines且cpic等级为a的位点。
[0037]
试剂盒的组分还包括变性混合液、扩增混合液、片段化混合液、沉淀混合液和杂交混合液。
[0038]
变性混合液由10
×
变性液用纯水稀释10倍而成。
[0039]
扩增混合液由1体积的扩增酶加45体积的扩增液混合制备而成。
[0040]
片段化混合液由1体积的片段化酶、10.3体积的片段化稀释液加45.7体积的10
×
片段化缓冲液混合制备所得。
[0041]
沉淀混合液由1体积的沉淀溶液2加119体积的沉淀溶液1混合制备所得。
[0042]
杂交混合液由1体积的杂交液1、18体积的杂交液2和140体积的杂交缓冲液混合制备所得。
[0043]
试剂盒在cnvplus
®
微阵列芯片扫描仪中进行检测使用,扫描仪配套试剂包括染料混合物a、染料混合物b、稳定混合物、连接混合物a和连接混合物b。
[0044]
染料混合物a由1体积的染色液1
‑
a、1体积的染色液1
‑
b、2体积的染色缓冲液和96体积的洗液a混合制备所得。
[0045]
染料混合物b由1体积的染色液2
‑
a、1体积的染色液2
‑
b、2体积的染色缓冲液和96体积的洗液a混合制备所得。
[0046]
稳定混合物由1体积的稳定液、8体积的固定稀释液用纯水稀释7.9倍而成。
[0047]
连接混合物a由1体积的连接液1、0.4体积的连接液2和5体积的连接缓冲液混合制备所得。
[0048]
连接混合物b由1体积的连接酶、6.6体积的探针混合液1、6.6体积的探针混合液2和52体积的连接混合物a混合制备所得。
[0049]
试剂盒中所用试剂可以选自由江苏源隆医疗科技有限公司生产的试剂,如表2 所示:表2试剂盒中的试剂
实施例2本实施例中的一种基于snv array技术的药物基因突变检测方法包括以下步骤:s1:样品处理与质控:用临床获取的12例样本作为发明的阳性验证样品。这些 验证样品先进行dna抽提,通过凝胶电泳,nanodrop等进行质控,确保每个dna 样品无降解,无杂质污染,高纯度。光密度(optical density,od)260/280nm比值介 于1.8~2.0,od 260/230nm比值介于1.5~2.0,不满足任一条件的样品需进行纯化等处 理。其中胶上样染料为invitrogen
tm trackit cyan/orange loading buffer(invitrogenp/n 10482
‑
028);25bp invitrogen ladder(invitrogen p/n 10488
‑
022);凝胶电泳系统 为invitrogen e
‑
gel
tm 48agarose gels 4%,g8008
‑
04。
[0050]
s2:检测反应:s21:dna扩增:100ng dna样本(5 ng/
µ
l)中加入20μl变性混合液,室温孵育10min;加入130 μl中和液,震荡混匀后沿管壁缓慢加入230μl扩增混合液,震荡混匀1000rpm离心1min;在37℃孵育箱中孵育23
±
1h;65℃孵育箱中孵育20min;37℃孵育箱中继续孵育45min;s22:dna片段化和沉淀:对步骤s21孵育的样本加入57μl片段化混合液,混匀、简短离心后37℃孵育30min,而后在室温环境下加入19
µ
l 终止液,进行震荡混匀离心;室温环境下加入240
µ
l沉淀混合液和600
µ
l异丙醇,用移液器上下充分混匀;转移至
‑
20℃环境中放置16~24h;s23:干燥、重悬及质控:对步骤s22处理后的样本进行4℃ 3200rpm离心40min,去除废液,置于37℃杂交炉中20min晾干,加入35
µ
l重悬缓冲液进行振荡混匀,震荡仪中震荡10min,加入80
µ
l 杂交混合液,进行震荡混匀离心,产生115
µ
l变性杂交液;质控使用10
µ
l的变性杂交液,包括用nanodrop测量吸光度和凝胶电泳,其中胶上样染料为invitrogen
™
trackit cyan/orange loading buffer(invitrogen p/n 10482
‑
028);25bp invitrogen ladder(invitrogen p/n 10488
‑
022);凝胶电泳系统为invitrogen e
‑
gel
™ꢀ
48 agarose gels 4%,g8008
‑
04;s24:变性和杂交:将步骤s23产生的变性杂交液放于杂交板,将杂交板放置于pcr
仪中进行变性程序:95℃变性10 min; 48℃变性3 min;冷却;使用移液器转移105
µ
l变性杂交液至杂交盘,载入cnvplus
®
微阵列芯片处理仪,持续杂交23.5h~24h;采用cnvplus
®
微阵列芯片扫描仪和自动分析软件,可以实现一次实验同时处理96个样本的检测通量,显著提高结果的可靠性。
[0051]
s25:芯片扫描仪检测:对步骤s24产生的混合物进行染料盘对应分装:其中染料混合物a的染料盘2个,每孔105
µ
l;染料混合物b的染料盘1个,每孔105
µ
l;稳定混合物的染料盘1个,每孔105
µ
l;连接混合物b的染料盘1个,每孔105
µ
l;将分装后的染料盘载入芯片扫描仪,同时向染料盘每孔加入150
µ
l固定缓冲液后避免接触扫描盘底部。将盖子的切口角对准扫描盘的切口边缘后盖住扫描,放置于工作台顶部进行检测;s3:数据质控和分析:成像完成后会得到每块杂交板的原始荧光信号数据,利用自动分析软件对其进行质控和分析,即可得到相应样品的变异位点信息结果文件。
[0052]
s4:结果分析与注释:结果包含每个样品检测到的药物基因突变位点信息,这些信息再经过进一步的注释分析,包括突变评级和医学解读,最终得到的明确的真实的位点信息。
[0053]
s5:用药指导报告:对于检测为阳性的样本发放用药指导报告,报告依据是临床药物基因组学实施联盟(cpic)公布的guideline。
[0054]
本次验证样品及检测结果信息如下表3所示。
[0055]
表3验证样品及检测结果表3验证样品及检测结果注:na以及nocall表示无解读结果;failed表示qc未通过;affected表示检 测到突变,且有影响本发明对12个阳性样本进行检测,所有样本均检出成功,如附图2、图3所示, 检出率为100%。说明本发明产品所设计的探针具有以下特点:(1)精准高效:有效覆盖药物基因相关所有的高发变异热点,仅需一次实验, 即可对abcb1、cyp2c19、htr2c、cyp3a5、slco2b1、ephx1、pla2g4a、 ugt1a、cyp2d6、ace、drd2、crhr1、fkbp5、vkorc1、cyp1a1、hla_b、 ugt1a4、mc4r、tpmt、mt
‑
rnr1基因相关的变异热点进行检出;(2)快速便捷:通过精准高效检测和高样本检测通量,操作简单,诊断高效, 且仅需一次临床费用支出,适合临床药物基因突变检测使用;
(3)创新性:本发明提供一种snv array法检测药物基因突变的方法,采用 genotype_new和es
‑
cluster聚类算法,突破了传统snp array仅能检测拷贝数异常的 局限,实现了单基因突变位点的检测。
[0056]
以上对本发明优选的具体实施方式和实施例作了详细说明,但是本发明并不限于上述实施方式和实施例,在本领域技术人员所具备的知识范围内,还可以在不脱离本发明构思的前提下作出各种变化。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。