1.本发明属于生物学技术领域,具体涉及一种基于家系denovo突变的分析方法及其应用。
背景技术:
2.随着基因组高通量测序技术的发展,近年来,许多研究表明denovo突变在家系疾病中起着重要的作用,运用全外显子或者全基因组测序技术,能够筛查可能致病基因,但目前没有一套完整的流程来进行家系的denovo突变分析。因此,亟需发现一种基于denovo测序的家系突变分析方法。
技术实现要素:
3.基于上述问题,本发明的目的是提供基于denovo测序的家系突变分析方法,所述方法包括以下步骤:
4.s1、数据过滤:使用fastp对denovo测序下机的数据进行过滤;
5.s2、比对:经过滤以后的数据和人的参考基因组hg19进行比对并对比对结果进行质控,在去除重复序列后,对数据比对结果进行统计;
6.s3、家系denovo突变的检测:对家系中的父母、孩子进行snv、indel的家系分析;
7.s4、对snv、indel的结果进行过滤。
8.在本发明的一种实施方式中,所述s1的具体方法如下:
9.s11、去除含adaptor的reads,自动识别接头序列,进行过滤;
10.s12、去除n的比例大于10%的reads;
11.s13、去除低质量reads,所述低质量reads是指质量值q≤5的碱基数占整个read的50%以上的reads。
12.在本发明的一种实施方式中,所述s2的具体方法如下:
13.s21、使用bwa对过滤以后的数据和人的参考基因组hg19进行比对;
14.s22、使用samtools,对比对以后的bam进行排序;
15.s23、使用picard,对排序以后的bam标注并去除重复序列;
16.s24、对测序覆盖度、比对率等进行统计。
17.在本发明的一种实施方式中,所述s3的具体方法如下:
18.s31、使用gatk得到每个样本的gvcf,然后对家系中的父母、孩子的样本进行家系的snv、indel检测;
19.s32、对snv、indel的结果,使用统计学方法预测denovo突变位点;
20.s33、对家系snp、indel突变位点进行注释。
21.在本发明的一种实施方式中,s32所述预测denovo突变位点的方法为根据父、母、子的基因型及突变位点支持突变reads数信息、基因型质量值,进行过滤。
22.在本发明的一种实施方式中,s33所述注释包括:变异位点基本信息注释、基因及
区域信息注释、正常人数据库注释、保守性预测注释。
23.在本发明的一种实施方式中,所述s4的具体方法如下:
24.s41、选取功能缺失和预测为有害的错义突变位点、移码突变位点和非移码突变位点;
25.s42、过滤正常人群突变频率数据库,根据项目要求修改maf筛选阈值;
26.s43、过滤支持变异位点reads数≤4的variants;
27.s44、过滤基因型:如果变异位点的基因型为纯合,过滤该位点支持突变的reads与该位点覆盖的reads数的比例<0.8的variants;如果变异位点的基因型为杂合,过滤该位点支持突变的reads与该位点覆盖的reads数的比例<0.2或>0.8的variants。
28.s45、使用igv查看得到结果的覆盖,过滤假阳性位点。
29.在本发明的一种实施方式中,s42选取的数据库包括千人基因组数据库、esp6500siv2all数据库、exacall数据库及exac eas数据库。
30.本发明还提供了上述分析方法在疾病预测中的应用。
31.本发明的有益效果:
32.本发明提供的分析方法能够筛查可能致病基因,预测常见疾病、罕见疾病或遗传疾病的发生。
附图说明
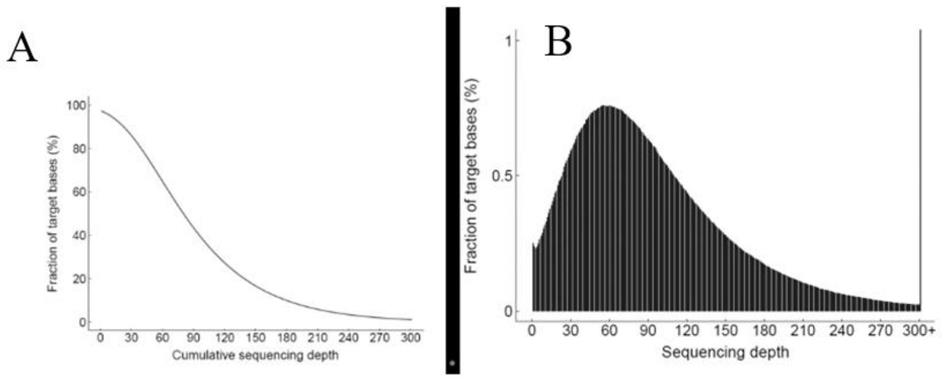
33.图1为覆盖度结果图;其中,图1中的a为不同测序深度的碱基比例,横坐标表示测序深度,纵坐标表示测序深度碱基在所有碱基中的比例;图1中的b为不同深度上的累积碱基比例;
34.图2为使用igv查看得到结果的覆盖图。
具体实施方式
35.下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
36.实施例一:基于家系denovo突变的分析方法
37.基于家系denovo突变的分析方法包括以下步骤:
38.s1、数据过滤:使用fastp对denovo测序下机的数据进行过滤,具体过滤步骤如下:
39.s11、去除含adaptor的reads,自动识别接头序列,进行过滤;
40.s12、去除n的比例大于10%的reads;
41.s13、去除低质量reads,所述低质量reads是指质量值q≤5的碱基数占整个read的50%以上的reads。
42.s2、比对:经过滤以后的数据和人的参考基因组hg19进行比对并对比对结果进行质控,在去除重复序列后,对数据比对结果进行统计,具体方法如下:
43.s21、使用bwa对过滤以后的数据和人的参考基因组hg19进行比对;
44.s22、使用samtools,对比对以后的bam进行排序;
45.s23、使用picard,对排序以后的bam标注并去除重复序列;
46.s24、对测序覆盖度、比对率等进行统计,覆盖度分布是衡量测序均一性的重要指标,覆盖度的统计结果如图1所示。
47.s3、家系denovo突变的检测:对家系中的父母、孩子进行snv、indel的家系分析,具体方法如下:
48.s31、使用gatk得到每个样本的gvcf,然后对家系中的父母、孩子的样本进行家系的snv、indel检测;
49.s32、对snv、indel的结果,使用统计学方法预测denovo突变位点(根据父、母、子的基因型及突变位点支持突变reads数信息、基因型质量值,进行过滤。);
50.s33、对家系snp、indel突变位点进行注释;所述注释包括:变异位点基本信息注释、基因及区域信息注释、正常人数据库(频率)注释、保守性(有害)预测注释。
51.变异位点基本信息注释:此部分信息为变异位点的详细信息,包括变异位点的覆盖深度,突变前后碱基型和纯杂合信息,变异质量值等。变异位点的信息能在家系分析或者筛选中起到重要的作用,同时也能评估结果的准确性。
52.基因及区域信息注释:此部分注释能详细了解变异位点所处的基因结构(对应的氨基酸)的具体位置和区域,有助于了解变异与疾病的相关性。
53.正常人数据库(频率)注释:人群中有很多变异位点是多态性的(高频的),而真正有害的变异位点一般是低频的。数据库主要包含千人、esp6500等有助于了解这个变异位点发生的频率的高低,找出致病突变位点。
54.保守(有害)性预测注释:一般个体突变往往都非常多,而真正有害的突变却是罕见的,此部分注释使用了多种国际惯用的变异有害性预测软件及数据库对变异位点进行有害性预测及评估,预测结果能协助找出真正有害的突变位点。
55.数据库注释:对突变位点所在基因进行疾病相关数据库注释,能了解到该突变位点是否已知与某类型疾病相关,也能了解到该突变位点所在基因存在于哪些通路中,对了解该基因的生物学功能有重要意义。
56.家系基因型信息:父、母、子的基因型及该位点支持突变reads数信息、基因型质量值。
57.s4、对snv、indel的结果进行过滤,具体方法如下:
58.s41、选取功能缺失和预测为有害的错义突变位点、移码突变位点和非移码突变位点;
59.s42、过滤正常人群突变频率数据库,根据项目要求修改maf筛选阈值,选取的数据库包括千人基因组数据库、esp6500siv2 all数据库、exac all数据库及exac eas数据库。若家系样本多来源于中国家庭,则将亚洲人突变频率的过滤加入到筛选致病性变异位点的分析流程中。
60.s43、过滤支持变异位点reads数≤4的variants;
61.s44、过滤基因型:如果变异位点的基因型为纯合,过滤该位点支持突变的reads与该位点覆盖的reads数的比例<0.8的variants;如果变异位点的基因型为杂合,过滤该位点支持突变的reads与该位点覆盖的reads数的比例<0.2或>0.8的variants。
62.s45、使用igv查看得到结果的覆盖(如图2),过滤假阳性位点。
63.以上所述实施例仅表达了本技术的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本技术专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本技术构思的前提下,还可以做出若干变形和改进,这些都属于本技术的保护范围。因此,本技术专利的保护范围应以所附权利要求为准。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。