超级起泡细菌
发明领域
1.本发明涉及新型遗传修饰的革兰氏阴性菌及其在制备和制造天然外膜囊泡(nomv)中的用途。
2.发明背景
3.革兰氏阴性菌自发地释放被称为天然外膜囊泡(nomv)的外细胞壁膜的泡状颗粒(kulp a.等人,annu rev mirobiol,2010,64:163-84),其可以用于多种生物技术应用(ellis tn和kuehn mj,microbiol mol biol rev.2010年3月;74(1):81-94),例如基于omv的疫苗领域(acevedo r等人,front.immunol,2014,5:121)。事实上,最近的证据显示了,基于omv的疫苗可以通过诱导体液免疫原性来保护免受百日咳博德特氏菌(bordetella pertussis)感染(gasperini g等人,mol cell proteomics,2018年2月;17(2):205-215),但是,尽管百日咳博德特氏菌在液体生长过程中天然地释放nomv,但所产生的量不足以用于大规模疫苗加工。外膜囊泡也可以人工产生,例如通过去污剂提取(被称为domv)或通过遗传改造细菌以显示出超级起泡(hyper blebbing)表型,其中,作为遗传修饰的后果,较大数量的外膜出芽从而提供了实际的膜材料来源。
4.对于商业疫苗生产,超级起泡表型通常通过缺失编码关键膜结构组分的基因例如gna33或tolr(berlanda scorza f等人,plos one,2012;7(6):e35616)来实现。然而,革兰氏阴性菌是高度遗传多样性的,并且在第一物种中诱导超级起泡的突变在第二物种中可以是不可能的(例如,因为该基因已经从野生型菌株中消失),或者可以在第二物种中并不诱导超级起泡(例如,由于控制膜结构的不同生物化学机制)。当用于新物种(即,其中并未应用超级起泡突变的物种)时,有必要通过例如与其它物种中的超级起泡相关联的基因的定向突变或随机突变和测试,在功能上筛选潜在的超级起泡突变。因此,鉴定在尽可能多的物种类型中以及理想地在所有革兰氏阴性菌中诱导超级起泡表型的遗传修饰将是有利的。
5.因为nomv特别适合于疫苗开发,所以本发明的一个目的是提供用于在多个革兰氏阴性物种和属中产生增加量的nomv的方法。
6.发明概述
7.通过修饰rpsa基因(编码30s核糖体蛋白s1)、rpsa操纵子和/或30s核糖体蛋白s1,而不是通过破坏编码膜结构组分的基因和/或操纵子和/或通过破坏膜结构组分,发明人已令人惊讶地开发了新的超级起泡革兰氏阴性菌细胞。
8.与已知的超级起泡突变相比,本文所述的遗传修饰提供了几个优点例如,与其操作涉及超级起泡的已知基因(其可能在细菌属和种上实质上不同)形成对比,rpsa(编码30s核糖体蛋白s1)在细菌属和种上广泛地表达且是保守的(machulin av等人,plos one,2019年8月22日;14(8):e0221370)。因此,本发明可以应用于编码30s核糖体蛋白s1的任何革兰氏阴性菌细胞。
9.值得注意的是,发现引起革兰氏阴性菌细胞的超级起泡的突变有利于在致死环境应激下的细菌存活(manning aj和kuehn mj,bmc microbiol,2011年12月1日;11:258)。特别地,nomv释放被描述为随着涉及调控细胞壁稳定性的错误折叠蛋白的累积而增加
(mcbroom aj和kuehn mj,mol miocrobiol,2007年1月;63(2):545-58)。令人惊讶的是,30s核糖体蛋白s1与膜稳定性无关。因此,本文所述的遗传修饰与nomv释放的调控机制相关联,推测是通过核糖体活性特化的转录调控(xue s和barna m,nat rev mol cell biol,2012年5月23日;13(6):355-69).
10.rpsa基因在细菌物种中是高度保守的,并且编码30s核糖体蛋白s1,其涉及细菌中的蛋白质翻译和核糖体蛋白合成的自身调节(aseev lv等人,rna,2008年9月;14(9):1882-94)。相应地,它牵涉调节属于其途径的那些蛋白质的浓度和折叠。起因于rpsa基因的3
′‑
区域内的突变的30s核糖体蛋白s1的c末端结构域的缺失,被描述为在大肠杆菌(e.coli)中不致死的(schnier j,j biol chem,1986年9月5日;261(25):11866-71;boni iv等人,j bacteriol 2000年10月;182(20):5872-9)。然而,不存在rpsa基因、rpsa操纵子或30s核糖体蛋白s1的修饰对nomv释放到培养基内具有影响的先前报道。
11.编码整合宿主因子(ihf)蛋白的β亚基的ihfb基因,定位紧邻rpsa下游。在一些细菌(例如百日咳博德特氏菌)中,在rpsa和ihfb之间存在非常短的基因间区域,其可以提示操纵子组构;而对于其它细菌(例如大肠杆菌),存在较长的基因间区域,其可能含有专用于ihfb表达的另一个启动子。因此,这两种基因很可能在一些革兰氏阴性菌如百日咳博德特氏菌中是共转录的。
12.附图描述
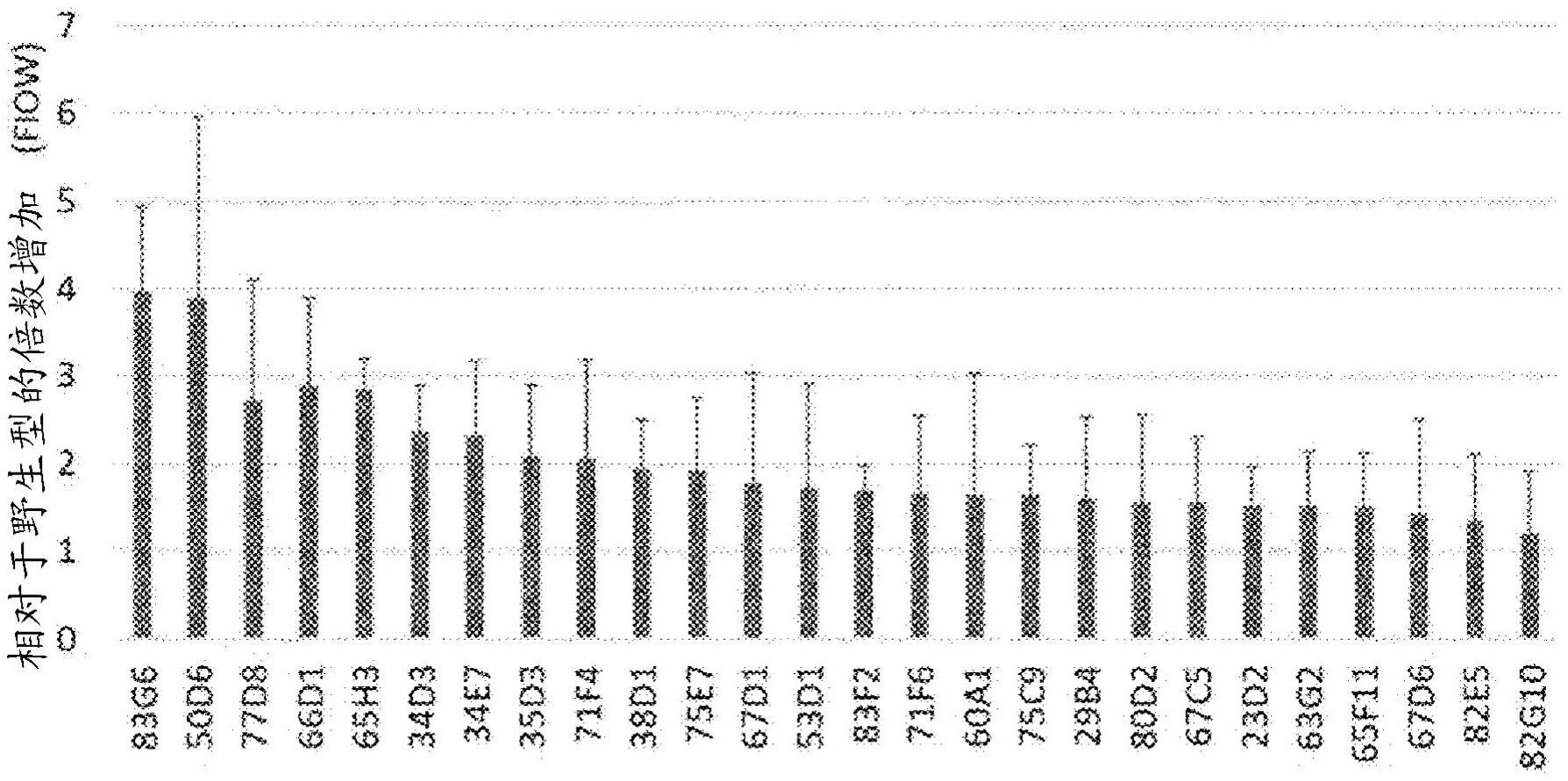
13.图1:通过荧光分析的随机突变体筛选结果。wt和随机突变株在37℃和350rpm下在深孔板中生长,直到晚期稳定生长期。对于每个克隆执行至少两次实验。在600nm处监测细胞培养物的光密度,同时通过fm4-64染料染色来测量上清液中的囊泡荧光。随机突变体的荧光强度针对每种培养物的光密度进行归一化(比产率),并且相对于wt菌株进行表示(相对于野生型的荧光强度-fiow)。
14.图2:微小规模生长中的gmma释放增加的确认。增加的nomv释放通过对于8个克隆的多重微小规模测试得到确认。通过使用fm4-64染料的荧光分析来揭示培养物上清液中的囊泡浓度。
15.图3:rpsa基因的示意性组构。结构域通过方框报告。50d6、83g6、77d8克隆中的tn5插入物通过箭头指示。
16.图4:来自小规模生长中的随机突变体的gmma释放。(a)wt和突变株在37℃和180rpm下,使用250ml具有通风杯的带挡板烧瓶,在补充的ss培养基(50m1)中生长。纯化的囊泡由细菌生长的晚期稳定期进行制备。(b)培养物上清液的fm4-64染料染色,将gmma释放的量表示为关于光密度归一化的fiow(比产率)。(c和d)小规模生长中的随机突变体gmma的释放分析。wt和突变株在37℃和350rpm下在250ml具有通风杯的带挡板烧瓶中生长。纯化的囊泡由晚期稳定期进行制备,并且通过nta分析进行定量。结果表示为绝对值(c)以及针对光密度(d)归一化的相对于wt(fiow)两者。
17.图5:通过大规模生长中的随机突变体的gmma释放。wt和两个随机克隆(50d6和66d1)在37℃和180rpm下,使用2l带挡板烧瓶,在补充的ss培养基(300ml)中生长。生长曲线以图形方式表示(a)。纯化的囊泡在细菌生长的大约72小时后进行制备,并且通过lowry测定进行定量(b):总蛋白产率针对每种培养物的光密度进行归一化,并且通过nta相对于wt菌株(fiow)表示:颗粒浓度表示为绝对值(d)或针对每种培养物的光密度进行归一化,并且
相对于wt菌株(fiow)表示(e);以及fm464(c):荧光强度针对每种培养物的光密度进行归一化,并且相对于wt菌株(fiow)表示。结果相对于光密度和wt值两者表示。
18.图6:来自50d6(rpsa突变体)和66d1克隆的上清液和纯化囊泡的蛋白谱。衍生自小规模培养物(左图)和分离的nomv(右图)的wt和突变体上清液的囊泡含量分别通过fm4-64和lowry测定进行定量。将样品加载到4-12%预制丙烯酰胺凝胶(加载5μg囊泡,荧光归一化的上清液)上,用于在室温和恒定电压下的电泳。蛋白质通过用于nomv的考马斯染色和用于上清液的银染色来显现。分子量梯在左侧上进行标记。
19.图7:培养物上清液的蛋白质印迹分析。为了确认上清液制剂含有gmma,来自wt和突变体大规模培养物的荧光强度(fi)归一化的上清液在sds-page上进行分离,并且印迹到硝酸纤维素膜上,用于用抗omv小鼠血清的免疫印迹分析。分子量梯在左侧上进行标记。
20.图8:tem图像。纯化的nomv从处于晚期稳定期的wt和突变体的培养物中进行分离,并且通过具有负染色的tem进行分析。比例尺,200nm。
21.图9:百日咳博德特氏菌清洁突变体验证。wt和突变株在37℃和350rpm下在深孔板中生长,直到晚期稳定生长期。在600nm处监测细胞培养物的光密度,同时通过fm4-64染料染色来测量上清液中的囊泡荧光。荧光强度针对每种培养物的光密度进行归一化,并且相对于wt菌株(fiow)进行表示。所报告的数据是16个独立培养物的平均值(对于两次实验中的每种突变体的8个单一菌落)
22.图10:s1比对。百日咳博德特氏菌(bp)和大肠杆菌(ec)30s核糖体蛋白s1序列使用clustal omega在线软件(hypertexttransferprotocolsecure://worldwideweb.ebi.ac.uk/tools/msa/clustalo/)进行比对。星号指示同一性,破折号指示空位,并且空格指示不匹配。在突变体中中断的第一个氨基酸在两个序列中以粗体且加下划线报告。
23.图11:在37℃下来自突变株的gmma产率(fiow)。荧光强度直接对无细胞的耗尽上清液进行测量,并且与对于培养物体积(体积产率)或光密度(比产率)归一化的野生型进行比较。所报告的数据是来自两次独立实验的平均值。
24.图12:来自突变株的gmma纯化(fiow)。gmma产率在纯化后进行评估,并且与对于培养物体积(体积产率)或光密度(比产率)归一化的野生型进行比较。所报告的数据是来自两次独立实验的平均值。
25.图13:gmma蛋白谱。由来自两次独立实验(i:泳道1-5;ii:泳道:6-10)的ec bl21(de3)野生型和突变体纯化的gmma在考马斯染色的sds-page中进行分析。由于极低的浓度,对于除δihfb#3外的所有样品都加载了5微克的总蛋白。
26.图14:ec突变体在30℃和25℃下的生长曲线。记录生长曲线,监测在600nm处的od。
27.图15:在30℃和25℃下来自突变株的gmma产率(fiow)。荧光强度直接对不同突变体的无细胞上清液进行测量,并且与对于培养物体积(体积产率)或光密度(比产率)归一化的野生型进行比较。所报告的数据是来自两次独立实验的平均值。
28.图16:来自粘膜炎莫拉氏菌(moraxella catarrhalis)中的rpsa和ihfb突变株的gmma产率。
29.图17:来自粘膜炎莫拉氏菌中的rpsa和ihfb突变株的gmma样品的负染色电子显微检查。
30.图18:rpsa过表达分析。在考马斯染色的sds-page上分析总蛋白谱和可溶性蛋白谱,以评估rpsa fl和tr过表达。预计的分子量分别为约61和48kda。
31.图19:来自rpsa过表达菌株的gmma产率(fiow)。荧光强度直接对不同突变体的无细胞的耗尽上清液进行测量,并且与对于培养物体积(体积产率)或最终生物质(比产率)归一化的用空载体转化的野生型进行比较。所报告的数据是来自三个技术重复的平均值。
32.图20:gmma蛋白谱。蛋白谱在考马斯染色的sds-page上进行分析,以比较蛋白质模式。
33.图21:来自rpsa过表达菌株的纯化的gmma产率(fiow)。来自不同菌株的纯化的gmma通过fm4-64染色进行定量,并且与用空载体转化的野生型进行比较,并且对于最终生物质(比产率)进行归一化。所报告的数据是来自三个技术重复的平均值。
34.发明详述
35.发明人已发现了,相对于未修饰的细菌细胞,rpsa基因、rpsa操纵子和/或30s核糖体蛋白s1的修饰可以增加由革兰氏阴性菌细胞释放的nomv。“未修饰的细菌细胞”意指或包括在其它方面等价的细菌细胞,其并不包含修饰的rpsa基因、修饰的rpsa操纵子和/或修饰的30s核糖体蛋白s1。
36.相应地,本发明的第一个方面提供了遗传修饰的革兰氏阴性菌细胞,其包含修饰的rpsa基因、修饰的rpsa操纵子和/或修饰的30s核糖体蛋白s1。“修饰的”(以及“修饰”)意指或包括以任何方式改变的任何生物材料,包括但不限于基因、操纵子、蛋白质和细胞。可以对生物材料进行化学或遗传修饰。修饰可以包括任何突变、基因产物表达的任何改变(例如上调和下调)或其组合(例如突变基因产物表达的改变)。
37.细菌细胞
38.遗传修饰的革兰氏阴性菌细胞可以属于选自以下的任何科(例如物种):肠杆菌科(enterobacteriaceae)、奈瑟氏球菌科(neisseriaceae)、螺杆菌科(helicobacteraceae)、弯曲菌科(campylobacteraceae)、耶尔森氏菌科(yersiniaceae)、弧菌科(vibrionaceae)、巴斯德氏菌科(pasteurellaceae)、产碱菌科(alcaligenaceae)、假单胞菌科(pseudomonadaceae)和莫拉氏菌科(moraxellaceae)。
39.可替代地或另外,遗传修饰的革兰氏阴性菌细胞是选自以下的属的物种:埃希氏菌属(escherichia)、志贺氏菌属(shigella)、奈瑟氏球菌属(neisseria)、莫拉氏菌属(moraxella)、博德特氏菌属(bordetella)、疏螺旋体属(borrelia)、布鲁氏菌属(brucella)、衣原体属(chlamydia)、嗜血杆菌属(haemophilus)、军团菌属(legionella)、假单胞菌属(pseudomonas)、耶尔森氏菌属(yersinia)、螺杆菌属(helicobacter)、沙门氏菌属(salmonella)、弧菌属(vibrio)等等。例如,革兰氏阴性菌细胞可以选自百日咳博德特氏菌、伯氏疏螺旋体(borrelia burgdorferi)、马耳他布鲁氏菌(brucella melitensis)、羊布鲁氏菌(brucella ovis)、鹦鹉热衣原体(chlamydia psittaci)、沙眼衣原体(chlamydia trachomatis)、粘膜炎莫拉氏菌、大肠杆菌、流感嗜血杆菌(haemophilus influenzae)(包括不可分型菌株)、嗜肺军团菌(legionella pneumophila)、淋病奈瑟氏球菌(neisseria gonorrhoeae)、脑膜炎奈瑟氏球菌(neisseria meningitidis)、乳糖奈瑟氏球菌(neisseria lactamica)、铜绿假单胞菌(pseudomonas aeruginosa)、小肠结肠炎耶尔森氏菌(yersinia enterocolitica)、幽门螺杆菌(helicobacter pylori)、肠道沙门氏菌
(salmonella enterica)(包括伤寒和鼠伤寒血清型、以及副伤寒和肠炎血清型)、志贺氏菌属(包括痢疾志贺氏菌(s.dysenteriae)、福氏志贺氏菌(s.flexneri)、鲍氏志贺氏菌(s.boydii)、宋内氏志贺氏菌(s.sonnei))、霍乱弧菌(vibrio cholerae)等。
40.可替代地或另外,革兰氏阴性菌细胞是百日咳博德特氏菌、奈瑟氏球菌属(例如脑膜炎奈瑟氏球菌或淋病奈瑟氏球菌)、沙门氏菌属(例如伤寒沙门氏菌(salmonella typhi)或鼠伤寒沙门氏菌(salmonella typhimurium))、志贺氏菌属(例如痢疾志贺氏菌、福氏志贺氏菌、鲍氏志贺氏菌或宋内氏志贺氏菌)、大肠杆菌(包括肠外致病性菌株)和流感嗜血杆菌(例如不可分型流感嗜血杆菌或nthi)。
41.可替代地或另外,遗传修饰的革兰氏阴性菌细胞是百日咳博德特氏菌菌株,例如tohama i菌株。可替代地或另外,遗传修饰的革兰氏阴性菌是表达遗传脱毒的百日咳类毒素,例如表达遗传脱毒的百日咳类毒素pt 9k/129g的百日咳博德特氏菌菌株。特别地,如其公开内容通过引用并入本文的wo/2020/094580中所述,可以使用这样的百日咳博德特氏菌菌株,所述百日咳博德特氏菌菌株包含已进行修饰以包括突变r9k和e129g的亚基1基因,并且表达遗传脱毒的百日咳类毒素pt-9k/129g。可替代地或另外,其中arnt基因已被敲除或缺失的百日咳博德特氏菌菌株的使用也是有利的。衍生自此类菌株的omv包含脂质a,其具有修饰的结构,不含在核心结构的远端磷酸基上的葡糖胺(glcn)取代。结果,当与衍生自具有功能性arnt基因的菌株的omv相比时,这些omv显示出降低水平的tlr4激活,如其公开内容通过引用并入本文的wo/2020/094580中所述的。
42.可替代地或另外,遗传修饰的革兰氏阴性菌细胞是脑膜炎奈瑟氏球菌菌株,例如血清群b菌株。可替代地或另外,脑膜炎奈瑟氏球菌菌株是除b外的血清群,例如a、c、w135、y或x之一。菌株可以具有任何血清型(例如,1、2a、2b、4、14、15、16等),任何血清亚型(例如,p.1.1、p.1.2、p.1.3、p1.4、p.1.5、p.1.6、p.1.7、p.1.9、p.1.10、p.1.12、p.1.13、p.1.14、p.1.15、p.1.16、p.1.9、p.1.22a)和任何免疫型(例如,l1;l2;l3;l3,7;l3,7,9;l10;等),如tondella ml等人,j clin microbiol.2000年9月;38(9):3323-8中所述。可替代地或另外,脑膜炎球菌可以来自任何合适的谱系,包括高侵袭性和高毒力谱系,包括选自以下的高毒力谱系:亚组i;亚组iii;亚组iv-1;et-5复合体;et-37复合体;a4簇;谱系3。可替代地或另外,脑膜炎奈瑟氏球菌菌株是nz98/254(b:4:p1.7-2,4)、或具有p1.4pora血清亚型的另一种菌株。
43.可替代地或另外,遗传修饰的革兰氏阴性菌细胞是淋病奈瑟氏球菌菌株,例如任何porbia或porbib菌株。可替代地或另外,淋病奈瑟氏球菌菌株是fa1090菌株或f62菌株。可替代地或另外,遗传修饰的革兰氏阴性菌细胞是大肠杆菌菌株,例如bl21、bl21(de3)、c43(de3)和dh5a菌株。可替代地或另外,遗传修饰的革兰氏阴性菌细胞不是大肠杆菌菌株,例如mb3001、csr603、jm105cag18478、iq646、ens0或ens0-xtir菌株。可替代地或另外,遗传修饰的革兰氏阴性菌细胞是铜绿假单胞菌菌株,例如pao1菌株。可替代地或另外,遗传修饰的革兰氏阴性菌细胞是流感嗜血杆菌菌株,例如hib atcc 10211、hib rd_kw20 hi1220、nthi r2866或nthi 86028np菌株。
44.可替代地或另外,遗传修饰的革兰氏阴性菌细胞是突变株,其已被操作以增强囊泡产生,表达一种或多种所需抗原例如一种或多种外源抗原,和/或敲除或修饰不需要的基因(例如,编码毒素或编码涉及生成有毒产物如内毒素的酶的基因)。例如,遗传修饰的革兰
氏阴性菌细胞可以通过选自下组的一种或多种方法进行进一步遗传改造:(a)下调免疫显性可变抗原或非保护性抗原的表达的方法,(b)上调保护性外膜蛋白(omp)抗原表达的方法,(c)下调涉及致使lps的脂质a部分有毒的基因的方法,(d)上调涉及致使lps的脂质a部分较少毒性的基因的方法,以及(e)对革兰氏阴性菌细胞进行遗传修饰以表达异源抗原的方法。
45.本发明的遗传修饰的革兰氏阴性菌细胞可能能够增殖。“能够增殖”意指或包括经历细胞分裂以生成后代细胞的能力。遗传修饰的革兰氏阴性菌细胞可能能够在关于细菌细胞的属和/或种的标准条件(参见例如,通过引用并入本文的peleg m和corradini mg,crit rev food sci nutr,2011年12月;51(10):917-45)或允许如此的其它条件下增殖,例如当在合适的温度(例如25℃、30℃、37℃、42℃)下、在有氧或厌氧环境中、在提供营养素的培养基中培养时。培养基可以是固体或液体,并且优选包含盐和营养素组分(例如,碳源、维生素、辅因子)。培养基可以含有血清。培养基优选提供合适的渗透性。培养基可以补充有对于每种菌株适当的抗生素,例如,它可以补充有链霉素、卡那霉素、萘啶酸、氯霉素和/或氨苄青霉素。例如,用于百日咳博德特氏菌tohama i菌株的合适生长条件是在37℃下三-五天的regan-lowe charcoal琼脂平板,或补充有0.4%(w/v)l-半胱氨酸单盐酸盐、0.1%(w/v)feso4、0.4%(w/v)抗坏血酸、0.04%(w/v)烟酸、1%(w/v)还原型谷胱甘肽的改良stainer-scholte培养基(ss),使用2ml 96孔深孔板用于微小规模培养(600μl),和使用在37℃下在定轨振荡器中的0.25-2l具有通风杯的带挡板烧瓶用于小规模和大规模培养(50-300ml),使微板以350rpm搅动且使烧瓶以180rpm搅动。例如,用于大肠杆菌bl21(de3)的合适生长条件是在25℃、30℃和37℃和180rpm下,在旋转振荡器中的luria bertani(lb)培养基或htmc培养基8小时、16小时或直到晚期稳定期。
46.遗传修饰的革兰氏阴性菌细胞的生长根据生物质、浊度和/或营养素摄取进行确定。评价细菌生长和增殖的方法是本领域已知的(peleg m和corradini mg,crit rev food sci nutr,2011年12月;51(10):917-45),例如,测量在600nm处的光密度(od),例如通过经由infinity 200pro分光光度计(tecan)测量在600nm处的od,用于评价液体培养物中的细菌生长,以及用于评价固体培养基中的细菌生长的菌落形成单位(cfu)。
47.可替代地或另外,遗传修饰的革兰氏阴性菌细胞能够生成的生物质是在相同培养条件下生长的未修饰细菌细胞的生物质的至少10%,例如在相同培养条件下生长的未修饰细胞的培养物的生物质的20%、30%、40%、50%、60%、70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%。例如,生物质在培养物达到稳定期时进行测量。可替代地或另外,遗传修饰的革兰氏阴性菌细胞能够实现的浊度是在相同培养条件下生长的未修饰细菌细胞的浊度的至少10%,例如在相同培养条件下生长的未修饰细菌细胞的培养物的浊度的20%、30%、40%、50%、60%、70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%。例如,浊度在培养物达到稳定期时进行测量。浊度可以通过本领域常见的方法,例如通过测量在600nm处的光密度(od)进行测量。例如,浊度通过以下进行测量:使5ml培养基(例如,对于每种菌株合适的培养基,例如lb)的预接种物在180rpm下在25℃或30℃下温育16-18小时或在37℃下温育8小时(预培养),在具有通风杯的带一次性带挡板的250ml烧瓶中的50ml培养基(例如,对于每种菌株合适的培养基,例如htmc)中1∶100稀释预培养物,分别在25℃、30℃和37℃下在180rpm振荡下温育直到
稳定期,并且用infinity 200pro分光光度计(tecan)测量在600nm处的od。可替代地或另外,革兰氏阴性菌细胞的培养物能够实现的营养素摄取是在相同培养条件下生长的未修饰细菌细胞的培养物的营养素摄取的至少10%,例如在相同培养条件下生长的未修饰细菌细胞的培养物的营养素摄取的20%、30%、40%、50%、60%、70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%。可替代地或另外,遗传修饰的革兰氏阴性菌细胞能够比在相同培养条件下生长的未修饰的细菌细胞更早地达到稳定期。可替代地或另外,遗传修饰的革兰氏阴性菌细胞能够与在相同培养条件下生长的未修饰的细菌细胞同时达到稳定期。可替代地或另外,遗传修饰的革兰氏阴性菌细胞能够在未修饰的细菌细胞在相同培养条件下生长后不迟于120小时,例如在未修饰的细菌细胞在相同培养条件下生长后不迟于1、2、3、4、5、6、7、8、9、10、12、15、20、25、30、40、50、60、70、80、90、100或110小时达到稳定期。例如,达到稳定期的能力通过浊度测量进行评价。例如,浊度通过以下进行测量:使5ml培养基(例如,对于每种菌株合适的培养基,例如lb)的预接种物在180rpm下在25℃或30℃下温育16-18小时或在37℃下温育8小时(预培养),在具有通风杯的带一次性带挡板的250ml烧瓶中的50ml培养基(例如,对于每种菌株合适的培养基,例如htmc)中1:100稀释预培养物,分别在25℃、30℃和37℃下在180rpm振荡下温育直到稳定期,并且用infinity 200pro分光光度计(tecan)测量在600nm处的od。
48.可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa基因,其中所述修饰的rpsa基因包含相对于野生型rpsa基因的一个或多个突变,其中所述一个或多个突变可能定位于rpsa基因的编码区内和/或rpsa基因的非编码区内。可替代地或另外,一个或多个突变可以借助于改变其表达和/或编码突变蛋白来改变rpsa基因产物。“突变”意指或包括多核苷酸的核苷酸序列中的改变和/或多肽的氨基酸序列中的改变。多核苷酸的突变可以选自缺失,例如一个或多个核苷酸的缺失;插入,例如一个或多个核苷酸的插入;插入的一个或多个核苷酸可以是外源的,例如源自野生型细菌细胞外部的核苷酸,或内源的,例如野生型细菌细胞的基因一部分的拷贝(例如重复);和/或取代,例如一个或多个核苷酸的取代。多核苷酸的缺失、插入和取代突变可以适当地引起由携带所述突变的多核苷酸编码的多肽中的错义或无义突变。多核苷酸的缺失和插入突变可以适当地引起由携带所述突变的多核苷酸编码的多肽中的移码突变。多肽的突变可以选自缺失,例如一个或多个氨基酸的缺失;插入,例如一个或多个氨基酸的插入和/或外源序列的插入;和/或取代,例如一个或多个氨基酸的取代。氨基酸取代可以是或可以不是保守的,即一个氨基酸由具有相关侧链的另一个氨基酸的替换。多肽的突变可以适当地修饰多肽的生物学活性。“翻译后修饰”意指或包括多肽在其生物合成之后的共价修饰,包括但不限于磷酸化、糖基化、酰化、乙酰化、脂化、二硫键形成和蛋白酶解切割。
49.可替代地或另外,一个或多个突变可以包含一个或多个核苷酸的插入或由一个或多个核苷酸的插入组成,其中所插入的一个或多个核苷酸选自转座子移动元件、选择标记物例如抗生素抗性盒、编码荧光蛋白的基因或编码抗毒素的基因、和/或rpsa基因的片段。“rpsa基因的片段”意指或包括由至少10个核苷酸,例如至少12、15、20、25、30、40、50、60、70、80、90、100、150、200个核苷酸组成的rpsa基因的核苷酸序列,其中所述rpsa基因与由未修饰的细菌细胞和/或野生型细菌细胞编码的rpsa基因相同或不同,例如其中所述rpsa基因是由相对于未修饰的细菌细胞不同物种的革兰氏阴性菌细胞编码的rpsa基因。“野生型
细菌细胞”意指或包括未以任何方式进行化学或遗传修饰的细菌细胞。
50.可替代地或另外,一个或多个突变使用选自以下的方法进行制备,或者通过选自以下的方法获得或可获得:定点诱变、重组酶介导的方法、λ-red重组酶介导的方法、基于原噬菌体的方法、移动ii组内含子敲除、转座子介导的基因组编辑和crispr-cas基因组编辑。可替代地或另外,转座子移动元件的插入使用转座子介导的基因组编辑进行制备,或者通过转座子介导的基因组编辑获得或可获得。可替代地或另外,转座子移动元件选自tn5转座子、tn10转座子、tn3转座子、tn7转座子、is5376转座子、细菌噬菌体mua转座子、is200/is605转座子和is91转座子。可替代地或另外,转座子移动元件是tn5转座子。
51.可替代地或另外,修饰的rpsa基因包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与野生型rpsa具有至少60%的序列同一性,例如与野生型rpsa至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。野生型rpsa可以选自seq id no:1、seq id no:2、seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:55和seq id no:60。
52.可替代地或另外,相对于野生型rpsa基因的一个或多个突变存在于指定的核苷酸序列中。可替代地或另外,相对于野生型rpsa基因的一个或多个突变并不存在于指定的核苷酸序列中,例如其中革兰氏阴性菌细胞是大肠杆菌。指定的核苷酸序列可以基于编码30s核糖体蛋白s1“r”结构域的核苷酸进行限定。“r”结构域意指或包括大约70个氨基酸的寡核苷酸/寡糖结合(ob)基序,其折叠成五链反向平行的β桶。多重“r”结构域可以以连续方式排列在30s核糖体蛋白s1中,所述30s核糖体蛋白s1可以包含1至6个结构域(machulin av等人,plos one,2019年8月22日;14(8):e0221370)。“r”结构域由10-15个氨基酸的链连接。“r”结构域可以使用合适的序列比对工具进行指定。指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的r3结构域的3
′
端上游的1至10个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端上游的11至20、21至50、或51至100个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的r3结构域的3’端上游的1至100个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端上游的10至50、或20至30个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因1%至10%的核苷酸,例如相对于野生型rpsa基因11%至20%、21%至30%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的5
′
端和相对于野生型rpsa基因的r3结构域的3
′
端之间。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因1%至30%的核苷酸,例如相对于野生型rpsa基因5%至25%、10%至20%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的5
′
端和相对于野生型rpsa基因的r3结构域的3
′
端之间。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的r3结构域的3’端下游的1至10个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端下游的11至20、21至50、或51至100个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的r3结构域的3’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端下游的10至50、或20至30个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的r4结构域的5’端下游的1至10个核苷酸,例如相对于野生型rpsa基因的r4结构域的5’端下游的11至20、21至50、或51至100个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组
成:相对于野生型rpsa基因的r4结构域的5’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r4结构域的5’端下游的10至50、或20至30个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的r4结构域的3’端下游的1至10个核苷酸,例如相对于野生型rpsa基因的r4结构域的3’端下游的11至20、21至50、或51至100个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的r4结构域的3’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r4结构域的3’端下游的10至50、或20至30个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因1%至10%的核苷酸,例如相对于野生型rpsa基因11%至20%、21%至30%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的r3结构域的3
′
端和相对于野生型rpsa基因的3
′
端之间。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因1%至30%的核苷酸,例如相对于野生型rpsa基因5%至25%、10%至20%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的r3结构域的3
′
端和相对于野生型rpsa基因的3
′
端之间。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的3
′
端上游的1至5个核苷酸,例如相对于野生型rpsa基因的3
′
端上游的6至10、11至20、21至30、31至40、41至50、51至100、101至150、151至200、201至250、251至300、301至350、351至400、401至450、451至500个核苷酸。可替代地或另外,指定的序列可以包含以下或由以下组成:相对于野生型rpsa基因的3
′
端上游的1至500个核苷酸,例如相对于野生型rpsa基因的3
′
端上游的50至450、100至400、150至380、200至378、或250至376个核苷酸。相对于指定的核苷酸序列(例如野生型rpsa基因或结构域,例如结构域1、结构域2、r1、r2、r3或r4)的“3端上游”,意指或包括直到并包括指定序列的最后一个核苷酸的核苷酸。相对于指定的核苷酸序列(例如野生型rpsa基因或结构域,例如结构域1、结构域2、r1、r2、r3或r4)的“3端下游”,意指或包括在指定序列的最后一个核苷酸之后的核苷酸。相对于指定的核苷酸序列(例如野生型rpsa基因或结构域,例如结构域1、结构域2、r1、r2、r3或r4)的“5端上游”,意指或包括直到并包括指定序列的第一个核苷酸的核苷酸。相对于指定的核苷酸序列(例如野生型rpsa基因或结构域,例如结构域1、结构域2、r1、r2、r3或r4)的“5端下游”,意指或包括在指定序列的第一个核苷酸之后的核苷酸。
53.可替代地或另外,相对于野生型rpsa基因的一个或多个突变包含在对应于编码百日咳博德特氏菌30s核糖体蛋白s1蛋白质的r4结构域的区域和/或编码百日咳博德特氏菌30s核糖体s1蛋白在r3和r4结构域之间的一部分的区域的区域中的一个或多个突变。
54.如果一个或多个突变在与特定结构域、部分或位置对齐的氨基酸序列或核苷酸序列中,则该一个或多个突变在“对应于”特定结构域、部分或位置的区域中。氨基酸序列或核苷酸序列是否与特定结构域、部分或位置对齐可以使用标准序列比对工具进行确定。任选地,百日咳博德特氏菌的r4结构域由seq id_no:24的核苷酸序列编码。任选地,百日咳博德特氏菌30s核糖体蛋白s1在r3和r4结构域之间的部分由seq id no:1的核苷酸1348至1383的核苷酸序列编码。
55.可替代地或另外,相对于野生型rpsa基因的一个或多个突变包含与未修饰的革兰氏阴性菌细胞相比,引起nomv释放的增加的一个或多个突变。如果当突变引入革兰氏阴性菌细胞中的基因内时,与除了突变之外相同的等价革兰氏阴性菌细胞相比,所述革兰氏阴性菌细胞释放更多的nomv,则突变引起nomv释放的增加。nomv释放水平可以通过选自以下
的参数进行评估:nomv的绝对量、上清液中的nomv浓度(在旋转后)、相对于细菌细胞数目的nomv绝对量、以及相对于细菌细胞数目的nomv浓度,如下文更详细地描述的。
56.可替代地或另外,与未修饰的革兰氏阴性菌细胞相比,一个或多个突变引起至少1.2倍、1.4倍、1.6倍、1.8倍、2.0倍或2.5倍的nomv释放增加。可替代地或另外,与未修饰的革兰氏阴性菌细胞相比,一个或多个突变引起至少2.0倍的nomv释放增加。可替代地或另外,一个或多个突变包含改变所编码的30s核糖体蛋白s1蛋白质的氨基酸序列的突变。可替代地或另外,一个或多个突变包含改变30s核糖体蛋白s1蛋白质的区域的编码氨基酸序列的突变,所述区域对应于百日咳博德特氏菌的r4结构域。可替代地或另外,一个或多个突变包含改变所编码的30s核糖体蛋白s1蛋白质的突变,以修饰和/或缺失至少5、至少10、至少25、至少50、至少75、至少100、至少110、至少125、5至150、25至130、100至130、或125至130个氨基酸。可替代地或另外,一个或多个突变包含改变所编码的30s核糖体蛋白s1蛋白质的突变,以缺失至少5、至少10、至少25、至少50、至少75、至少100、至少110、至少125、5至150、25至130、100至130、或125至130个氨基酸。可替代地或另外,修饰的rpsa基因编码如本文所述的修饰的30s核糖体蛋白s1蛋白质。可替代地或另外,一个或多个突变包含对应于百日咳博德特氏菌rpsa基因的核苷酸1655至1731的区域的至少15、至少30、至少45、至少60或至少75个核苷酸的突变或缺失。任选地,百日咳博德特氏菌rpsa基因是具有seq id no:1的核苷酸序列的基因。可替代地或另外,一个或多个突变包含对应于百日咳博德特氏菌rpsa基因的核苷酸1424至1731的区域的至少60、至少90、至少150、至少225或至少300个核苷酸的突变或缺失。可替代地或另外,一个或多个突变包含对应于百日咳博德特氏菌rpsa基因的核苷酸1355至1731的区域的至少60、至少90、至少150、至少225、至少300或至少360个核苷酸的突变或缺失。
57.可替代地或另外,一个或多个突变通过(a)改变其表达和/或(b)编码突变的30s核糖体蛋白s1来改变rpsa基因产物。
58.可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa操纵子。可替代地或另外,修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与野生型rpsa操纵子具有至少60%的序列同一性,例如与野生型rpsa操纵子至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。野生型rpsa操纵子可以选自seq id no:7、seq id no:8、seq id no:9、seq id no:10、seq id no:11和seq id no:12。
59.可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1。可替代地或另外,修饰的30s核糖体蛋白s1包含相对于野生型30s核糖体蛋白s1的一个或多个突变和/或包含相对于野生型30s核糖体蛋白s1的一种或多种翻译后修饰。
60.可替代地或另外,相对于野生型30s核糖体蛋白s1的一个或多个突变包含在对应于百日咳博德特氏菌30s核糖体蛋白s1的r4结构域的区域和/或对应于百日咳博德特氏菌30s核糖体蛋白s1在r3和r4结构域之间的部分的区域中的一个或多个突变。任选地,百日咳博德特氏菌的r4结构域由seq id no:24的核苷酸序列编码。任选地,百日咳博德特氏菌30s核糖体蛋白s1在r3和r4结构域之间的部分由seq id no:1的核苷酸1348至1383的核苷酸序列编码。
61.可替代地或另外,相对于野生型30s核糖体蛋白s1的一个或多个突变包含至少5、
至少10、至少25、至少50、至少75、至少100、至少110、至少125、5至150、25至130、100至130、或125至130个氨基酸的突变或缺失。可替代地或另外,相对于野生型30s核糖体蛋白s1的一个或多个突变包含对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸550至576的区域的至少5、至少10、至少15、至少20、或至少25个氨基酸的突变或缺失。可替代地或另外,相对于野生型30s核糖体蛋白s1的一个或多个突变包含对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸473至576的区域的至少20、至少30、至少50、至少75、或至少100个氨基酸的突变或缺失。可替代地或另外,相对于野生型30s核糖体蛋白s1的一个或多个突变包含对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸450至576的区域的至少20、至少30、至少50、至少100、或至少120个氨基酸的突变或缺失。可替代地或另外,相对于野生型30s核糖体蛋白s1的一个或多个突变包含来自30s核糖体蛋白s1的c末端端部的至少20、至少30、至少50、至少100、或至少120个连续氨基酸的截短。可替代地或另外,相对于野生型30s核糖体蛋白s1的一个或多个突变包含对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸560至576、552至576、500至576485至576、460至576、或453至576的氨基酸的突变或缺失。任选地,百目咳博德特氏菌30s核糖体蛋白s1具有seq id no:13的氨基酸序列。
62.可替代地或另外,修饰的30s核糖体蛋白s1相对于未修饰的细菌细胞的30s核糖体蛋白s1是下调的。“下调”或“下调的”意指或包括水平低于通过未修饰的细菌表达的基因产物表达。“较低水平”意指或包括相对于对照具有统计显著性的任何较低水平,例如相对于对照为至多1/2的水平。例如,下调可以通过遗传修饰来实现,所述遗传修饰包括改变或修饰与目的基因表达相关的调控序列或位点(例如,修饰启动子或调控序列),修饰目的基因的染色体定位,改变与目的基因相邻的核酸序列例如核糖体结合位点,修饰涉及目的基因转录和/或基因产物翻译的蛋白质(例如调控蛋白、阻抑制子、增强子、转录激活因子),或本领域已知的解除对目的基因表达的调控的任何其它常规手段(包括但不限于反义核酸分子的使用)。下调用jocelyn e.krebs,elliott s.goldstein,stephen t.kilpatrick,lewin’s genes xii,2017中公开的方法适当地进行评估,所述参考文献的公开内容通过引用并入本文。
63.可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1相对于野生型30s核糖体蛋白s1是截短的。可替代地或另外,修饰的30s核糖体蛋白s1相对于野生型30s核糖体蛋白s1并非截短的,例如,其中所述革兰氏阴性菌细胞是大肠杆菌。“截短的”意指或包括至少缺少野生型多肽的片段的野生型多肽的变体。此类片段可以定位于野生型多肽的n末端或c末端处,或者可以定位于多肽的n末端和c末端之间。此类片段可以是至少7个氨基酸长,例如,至少8、9、10、15、20、25、30、35、40、45、50、60、70、80、100、150、200个氨基酸长。
64.可替代地或另外,修饰的30s核糖体蛋白s1由修饰的rpsa基因编码。可替代地或另外,修饰的rpsa基因是染色体的或染色体外的,例如,修饰的rpsa基因由质粒或粘粒编码。
65.可替代地或另外,修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型s1具有至少60%的序列同一性,例如与野生型30s核糖体蛋白s1具有至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。野生型30s核糖体蛋白s1可以选自seq id no:13、seq id no:14、seq id no:15、seq id no:16、seq id no:17、seq id no:18和seq id no:57。可替代地或另外,
遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型30s核糖体蛋白s1的片段具有至少90%、至少95%、至少98%、至少99%或100%的同一性,所述片段对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸1至452。可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型30s核糖体蛋白s1的片段具有至少98%、至少99%或100%的同一性,所述片段对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸1至453。
66.可替代地或另外,修饰的30s核糖体蛋白s1包含选自以下的“r”结构域或由其组成:r1结构域或其片段、r2结构域或其片段、r3结构域或其片段和r4结构域或其片段;r1结构域或其片段、r2结构域或其片段和r3结构域或其片段;r2结构域或其片段、r3结构域或其片段和r4结构域或其片段;r2结构域或其片段和r3结构域或其片段;r3结构域或其片段和r4结构域或其片段;以及r3结构域或其片段。
67.可替代地或另外,30s核糖体蛋白s1可以由6个“r”结构域组成。此类结构域从n末端到c末端被指定为“结构域1”、“结构域2”、“r1”、“r2”、“r3”和“r4”。结构域1和结构域2涉及蛋白质-核糖体和蛋白质-蛋白质相互作用。“r”结构域可能具有rna结合性质,其对于单链区域可以是特异性的(bycroft m等人,cell,1997年1月24日;88(2):235-42;duval m等人,plos biol,2013年12月;11(12):e1001731)。另外,30s核糖体蛋白s1的c末端结构域可以结合rna结合区中具有保守残基的rna片段(fan y等人,biochem and biophys res commun,2017年5月27日;487(2):268-273),并且r4结构域的缺失被描述为在大肠杆菌中不致死的(schnier j等人,j biol chem,1986年9月5日;261(25):11866-71)。技术人员可以使用本领域已知的比对工具(machulin av等人,plos one,2019年8月22日;14(8):e0221370),来评价由1、2、3、4、5个或更多个“r”结构域组成的30s核糖体蛋白s1的哪些“r”结构域对应于六结构域30s核糖体蛋白s1的“结构域1”、“结构域2”、“r1”、“r2”、“r3”和“r4”。
68.可替代地或另外,结构域1由seq id no:19的核苷酸序列或其变体编码。可替代地或另外,结构域2由seq id no:20的核苷酸序列或其变体编码。可替代地或另外,r1由seq id no:21的核苷酸序列或其变体编码。可替代地或另外,r2由seq id no:22的核苷酸序列或其变体编码。可替代地或另外,r3由seq id no:23的核苷酸序列或其变体编码。可替代地或另外,r4由seq id no:24的核苷酸序列或其变体编码。“变体”意指或包括与给定序列具有至少60%序列同一性的序列,例如与给定序列至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。“r结构域的片段”意指包括至少9个核苷酸长,例如至少12、15、18、21、24、30、36、42、48、54、60、72、81、90、99、120、150、180、210个核苷酸长的核苷酸序列和/或至少3个氨基酸长,例如至少4、5、6、7、8、10、12、14、16、18、20、22、27、30、33、40、50、60、70个核苷酸长的多肽序列。
69.可替代地或另外,修饰的30s核糖体蛋白s1可以包含具有r1结构域的氨基酸序列或由其组成,当存在时,所述r1结构域与野生型s1具有至少60%的序列同一性,例如与野生型r1结构域具有至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。可替代地或另外,修饰的s1可以包含具有r2结构域的氨
基酸序列或由其组成,当存在时,所述r2结构域与野生型s1具有至少60%的序列同一性,例如与野生型r2结构域具有至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。可替代地或另外,修饰的s1可以包含具有r3结构域的氨基酸序列或由其组成,所述r3结构域与野生型s1具有至少60%的序列同一性,例如与野生型r3结构域具有至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。可替代地或另外,修饰的s1可以包含具有r4结构域的氨基酸序列或由其组成,当存在时,所述r4结构域与野生型s1具有至少40%的序列同一性,例如与野生型r4结构域具有至少50%、60%、70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。
70.可替代地或另外,修饰的30s核糖体蛋白s1可以包含r1、r2、r3和/或r4结构域的第一个和/或最后一个氨基酸上游的氨基酸序列,当存在时,其与野生型30s核糖体蛋白s1的r1、r2、r3和/或r4结构域的第一个和/或最后一个残基上游的氨基酸序列具有至少60%、70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。
71.可替代地或另外,修饰的30s核糖体蛋白s1使用以下进行修饰,或者通过以下获得或可获得:基因组编辑、基因沉默、片段化、rna转录后修饰和/或翻译后修饰。
72.可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含修饰的ihfb基因和/或修饰的整合宿主因子(ihf)蛋白。
73.可替代地或另外,修饰的ihfb基因包含相对于野生型ihfb基因的一个或多个突变,其中所述一个或多个突变定位于ihfb基因的编码区内和/或ihfb基因的非编码区内。可替代地或另外,一个或多个突变可以借助于改变其表达和/或编码突变蛋白来改变ihfb基因产物。可替代地或另外,修饰的ihfb基因相对于野生型ihfb基因被敲除。可替代地或另外,修饰的ihfb基因包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型ihfb基因具有至少60%的序列同一性,例如与野生型ihfb基因具有至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。野生型ihfb基因可以选自seq id no:25、seq id no:26、seq id no:27、seq id no:28、seq id no:29和seq id no:30。
74.可替代地或另外,相对于野生型ihf蛋白的一个或多个突变包含至少20、至少30、至少40、至少50、至少60、至少70、至少80、至少90、至少100、至少110、50至119、或80至119个氨基酸的缺失。可替代地或另外,相对于野生型ihf蛋白的一个或多个突变包含至少20、至少30、至少40、至少50、至少60、至少70、至少80、至少100、至少110、50至119、或80至119个邻接氨基酸的缺失。可替代地或另外,与未修饰的革兰氏阴性菌细胞相比,相对于野生型ihf蛋白的一个或多个突变引起nomv释放的增加。如果当突变引入革兰氏阴性菌细胞中的基因内时,与除了突变之外相同的等价革兰氏阴性菌细胞相比,所述革兰氏阴性菌细胞释放更多的nomv,则突变引起nomv释放的增加。nomv释放水平可以通过选自以下的参数进行评估:nomv的绝对量、上清液中的nomv浓度(在旋转后)、相对于细菌细胞数目的nomv绝对量、以及相对于细菌细胞数目的nomv浓度,如下文更详细地描述的。
75.可替代地或另外,与未修饰的革兰氏阴性菌细胞相比,相对于野生型ihf蛋白的一个或多个突变引起至少1.2倍、至少1.4倍、至少1.6倍、至少1.8倍、至少2.0倍或至少2.5倍
的nomv释放增加。可替代地或另外,革兰氏阴性菌细胞是天然共转录ihfb基因和rpsa基因的细菌物种。
76.可替代地或另外,修饰的ihf蛋白包含相对于野生型ihf蛋白的一个或多个突变。可替代地或另外,修饰的ihf蛋白包含相对于野生型ihf蛋白的一种或多种翻译后修饰。可替代地或另外,修饰的ihf蛋白包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型ihf蛋白具有至少60%的序列同一性,例如与野生型ihf蛋白至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。野生型ihf蛋白可以选自seq id no:31、seq id no:32、seq id no:33、seq id no:34、seq id no:35、seq id no:36和seq id no:62。
77.可替代地或另外,修饰的ihf蛋白相对于未修饰的细菌细胞的ihf蛋白是下调的。可替代地或另外,修饰的ihf蛋白由修饰的ihfb基因编码。
78.可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含野生型rpsa基因。可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含野生型rpsa基因,并且不包含修饰的rpsa基因,但表达修饰的30s核糖体蛋白s1(例如由于转录后修饰或由于rpsa操纵子的修饰)。可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含野生型rpsa基因和修饰的rpsa基因。可替代地或另外,遗传修饰的革兰氏阴性菌细胞包含野生型30s核糖体蛋白s1和修饰的30s核糖体蛋白s1。可替代地或另外,遗传修饰的革兰氏阴性菌细胞并不包含野生型rpsa基因。可替代地或另外,遗传修饰的革兰氏阴性菌细胞并不包含野生型30s核糖体蛋白s1。
79.可替代地或另外,修饰的rpsa基因包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:37或seq id no:56具有至少60%的序列同一性,例如与seq id no:37或seq id no:56至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
80.可替代地或另外,修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:38、seq id no:51或seq id no:59具有至少60%的序列同一性,例如与seq id no:38、seq id no:51或seq id no:59至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。
81.可替代地或另外,修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与选自seq id no:39、seq id no:40、seq id no:41和seq id no:53的多肽具有至少60%的序列同一性,例如与选自seq id no:39、seq id no:40、seq id no:41和seq id no:53的多肽至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。
82.可替代地或另外,修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:42或seq id no:43具有至少60%的序列同一性,例如与seq id no:42或seq id no:43至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。
83.可替代地或另外,修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与seq id no:44或seq id no:45具有至少60%的序列同一性,例如与seq id no:44或seq id no:45至少70%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。
(malvern ltd)来执行nta。纯化的nomv或细菌培养物的双重过滤上清液以在0.02μm过滤的d-pbs中1:200-1:5000的稀释度范围加载到测量室内。测量通过捕获60秒的5次测量以流动模式来执行,具有20(~2.1μl/分钟)的流速,产生60-90个颗粒/帧。所有测量都在室温下执行,并且捕获结果使用nanosight nta 3.2软件进行分析。在样品测量前,通过测量milliq稀释剂60秒,确认了milliq稀释剂含有少于1.0个颗粒/帧。囊泡浓度可以表示为“通过nta的总颗粒”(颗粒数目/ml)、“通过nta的特定颗粒”(颗粒数目/ml/od)或“相对于野生型的颗粒数目”(颗粒数目/ml/od)突变体/(颗粒数目/mi/od)wt。
90.可替代地或另外,革兰氏阴性菌细胞并不包含引起30s核糖体蛋白s1的c末端结构域缺失的修饰,其例如起因于rpsa基因的3
′‑
区域内的突变。可替代地或另外,革兰氏阴性菌细胞并不包含修饰的rpsa基因、rpsa操纵子和/或30s核糖体蛋白s1,如schnier j,j biol chem,1986年9月5日;261(25):11866-71中所述的,所述参考文献的公开内容通过引用并入本文。可替代地或另外,革兰氏阴性菌细胞并不包含修饰的rpsa基因、rpsa操纵子和/或30s核糖体蛋白s1,如boni iv等人,j bacteriol 2000年10月;182(20):5872-9中所述的,所述参考文献的公开内容通过引用并入本文。
91.在第二个方面,本发明提供了生成遗传修饰的革兰氏阴性菌细胞的方法,其包括修饰野生型rpsa基因、操纵子、rna和/或30s核糖体蛋白s1的步骤,使得修饰引起这样的遗传修饰的革兰氏阴性菌细胞,当在培养基中生长时,与未修饰的细菌细胞相比,其将更多数量的nomv释放到培养基内。在第三个方面,本发明提供了生成遗传修饰的革兰氏阴性菌细胞的方法,其包括提供突变型30s核糖体蛋白s1的步骤,使得修饰引起这样的遗传修饰的革兰氏阴性菌细胞,当在培养基中生长时,与未修饰的细菌细胞相比,其将更多数量的nomv释放到培养基内。
92.此外,提供了包含一种或多种本发明的遗传修饰的革兰氏阴性菌细胞的细胞培养物。
93.omv
94.术语“天然外膜囊泡”或“nomv”在本文中指示在培养基中通过革兰氏阴性菌自发地释放的外膜囊泡。nomv不被洗涤剂或变性剂提取。本发明的nomv呈现以其在天然膜环境中的天然构象和正确取向的外膜蛋白(omp)和/或脂多糖(lps),并且通常缺乏细胞质组分。相反地,术语“去污剂提取的omc”或“domv”涵盖了通过通常经由去污剂提取过程破坏革兰氏阴性菌的外膜以由其形成囊泡而获得的各种蛋白质脂质体囊泡。
95.术语“用于膜抗原的一般化模块”或“gmma”也可以用于指从突变型细菌获得的nomv。
96.nomv可以从本发明的遗传修饰的革兰氏阴性菌细胞的培养物中获得。它们可以从在肉汤或固体培养基培养物中生长的细菌获得,例如通过将细菌细胞与培养基分开(例如通过过滤或通过低速离心以使细胞形成团块),以及将外膜级分与细胞质分子分开(例如通过过滤,通过外膜和/或nomv的差异沉淀或聚集,通过使用特异性识别外膜分子的配体的亲和分离方法,或通过使外膜和/或nomv形成团块的高速离心)。
97.用于nomv制备的一种有用的方法涉及粗nomv的超滤,而不是高速离心。该方法可能涉及在超滤发生后的超速离心步骤。
98.用于本发明中的nomv制剂一般基本上不含完整细菌,无论是活的还是死的。囊泡
的大小意味着它们可以容易地通过例如如通常用于过滤器灭菌的过滤,从整个细菌中分离。
99.在第四个方面,本发明提供了用于制备nomv的方法,其包括以下步骤:接种含有适合于遗传修饰的革兰氏阴性菌细胞生长的营养培养基的培养容器,在允许nomv通过革兰氏阴性菌细胞释放到培养基内的条件下,培养遗传修饰的革兰氏阴性菌细胞,从培养基中回收nomv;并且将nomv与药学上可接受的稀释剂或载体混合。可替代地或另外,在回收nomv后,该方法可以包括无菌过滤nomv的制剂的进一步步骤。
100.本发明的第五个方面提供了nomv,其得自或可得自本发明的遗传修饰的细菌细胞、或者通过本发明的方法或通过本发明的过程获得或可获得的遗传修饰的革兰氏阴性菌细胞。
101.本发明的第六个方面提供了用于制备nomv的方法,其包括以下步骤:接种含有适合于遗传修饰的革兰氏阴性菌细胞生长的营养培养基的培养容器,在允许nomv通过细菌释放到培养基内的条件下,培养遗传修饰的革兰氏阴性菌细胞,其中所述条件包括添加本文所述的修饰的30s核糖体蛋白s1,从培养基中回收nomv,并且将nomv与药学上可接受的稀释剂或载体混合。
102.制药方法
103.在第六个方面,本发明提供了免疫原性组合物,其包含从本发明的革兰氏阴性菌细胞获得或可获得的nomv。可替代地或另外,组合物可以进一步包含来自相同或不同病原体的一种或多种另外的抗原。
104.本发明的免疫原性组合物可以进一步包含药学上可接受的载体。通常的药学上可接受的载体包括本身并不诱导对接受组合物的个体有害的抗体产生的任何载体。合适的载体通常是大的、缓慢代谢的大分子,例如蛋白质、多糖、聚乳酸、聚乙醇酸、聚合氨基酸、氨基酸共聚物、蔗糖、海藻糖、乳糖和脂质聚集体(例如油滴或脂质体)。此类载体是本领域普通技术人员众所周知的。疫苗还可以含有稀释剂,例如水、盐水、甘油等。
105.另外,可以存在辅助物质,例如润湿剂或乳化剂、ph缓冲物质等等。无菌无热原的磷酸盐缓冲生理盐水是通常的载体。
106.本发明的组合物可以以水性形式(即,溶液或悬浮液)或干燥形式(例如,冻干的)。如果使用干燥的疫苗,则它将在注射之前重构成液体介质。
107.本发明的组合物可以以各种形式进行制备。例如,可以将组合物制备为作为液体溶液或悬浮液的注射剂。组合物可以制备用于肺部施用,例如作为吸入剂,使用细粉或喷雾。组合物可以制备为栓剂或阴道栓剂。组合物可以制备用于鼻、耳或眼部施用,例如作为喷雾剂、滴剂、凝胶或粉末。本发明的组合物一般直接施用于患者。直接递送可以通过肠胃外注射(例如,皮下、腹膜内、静脉内、肌内或对于组织的间隙),或者通过直肠、口腔、阴道、局部、经皮、鼻内、眼、耳、肺部或其它粘膜施用来完成。对大腿或上臂的肌内施用是优选的。注射可以经由针(例如皮下注射针),但可替代地可以使用无针注射。
108.本发明的组合物一般包括缓冲液。磷酸盐缓冲液是通常的。
109.本发明的组合物可以与其它免疫调节剂结合进行施用。特别地,组合物可以包括一种或多种佐剂。此类佐剂包括但不限于含矿物质组合物。适合用作本发明中的佐剂的含矿物质组合物包括矿物盐,例如铝盐和钙盐(或其混合物)。钙盐包括磷酸钙。铝盐包括氢氧
化物、磷酸盐、硫酸盐等,其中盐采取任何合适的形式(例如凝胶、结晶、无定形等)。对这些盐的吸附是优选的。可以使用称为氢氧化铝和磷酸铝的佐剂。
110.医疗用途
111.当施用于哺乳动物时,本发明的nomv能够引发针对它们已由其获得的细菌细胞的免疫应答。由nomv引发的免疫应答可以指向或针对nomv中存在的一种或多种细菌蛋白质抗原。免疫应答可以是细胞或体液免疫应答。特别地,免疫应答是抗体应答。针对nomv的“抗体应答”,例如朝向或针对nomv中存在的一种或多种细菌蛋白质抗原的抗体应答,意指或包括在受试者中诱导免疫应答的能力,所述免疫应答生成(例如,刺激其释放)能够结合nomv或者nomv中存在的一种或多种细菌蛋白质抗原的抗体。优选的是结合部分能够在体内,即,在其中nomv或者nomv中存在的一种或多种细菌蛋白质抗原存在于受试者的身体上或内部的生理条件下结合。此类结合特异性可以通过本领域众所周知的方法来确定,所述方法例如使用细菌细胞的nomv的elisa、免疫组织化学、免疫沉淀、蛋白质印迹和流式细胞术。
112.可替代地或另外,免疫应答是可以中和细菌细胞的感染和/或毒力的t细胞免疫应答。可替代地或另外,免疫应答是免疫激活应答,例如保护性免疫应答。当施用于受试者时,nomv可能能够引发体外保护性免疫应答和/或体内保护性免疫应答。“免疫激活反应”意指和/或包括nomv在受试者中诱导或能够诱导免疫应答,其并不引起压制或终止炎症或炎症信号,并且优选引起炎症或炎症信号(m如细胞因子)的激活或增强。本发明的组合物是免疫原性的并且更优选是疫苗组合物。本发明的nomv、组合物和疫苗可以是预防性的(即,预防感染)或治疗性的(即,治疗感染),但通常是预防性的。
113.在第七个方面,本发明提供了用于在脊椎动物中,优选在哺乳动物中产生免疫应答的nomv、免疫原性组合物或疫苗,包括向脊椎动物施用本发明的nomv、免疫原性组合物或疫苗。nomv、免疫原性组合物和疫苗优选地能够在脊椎动物中,优选在哺乳动物中产生免疫应答。免疫应答优选是保护性的并且优选在宿主中诱导抗体应答,即保护性抗体应答。
114.在第八个方面,本发明还提供了用于在脊椎动物中,优选在哺乳动物中产生免疫应答的方法,其包括向哺乳动物施用本发明的nomv、免疫原性组合物或疫苗。免疫应答优选是保护性的并且优选在宿主中诱导抗体应答,即保护性抗体应答。
115.哺乳动物优选是人。当疫苗用于预防用途时,人优选是儿童(例如幼儿或婴儿,特别是新生儿)或青少年。预期用于儿童的疫苗也可以施用于成人,例如以评价安全性、剂量、免疫原性等。用于治疗的一类优选的人是育龄(例如,青少年及以上)女性。另一个优选类别是怀孕的女性。
116.本发明的第九个方面提供了用于药物中的本发明的组合物。本发明的第十个方面提供了本发明的nomv、组合物或疫苗在制造用于在脊椎动物中,优选在哺乳动物中产生免疫应答的药剂中的用途。这些用途和方法优选用于预防和/或治疗通过nomv由其获得的细菌细胞、或者与nomv由其获得的细菌细胞同一属或种的细菌细胞引起的疾病,所述细菌细胞例如百日咳博德特氏菌、粘膜炎莫拉氏菌、脑膜炎奈瑟氏球菌、淋病奈瑟氏球菌、大肠杆菌、铜绿假单胞菌、流感嗜血杆菌。
117.本发明可以用于引发全身免疫和/或粘膜免疫。本发明的免疫原性组合物可以以单剂量或多剂量进行施用。
118.一般内容
119.为了便于理解本发明,下文定义了许多术语和短语。下述术语和短语(包括过去时态、现在时态等)的本领域公认的同义词或替代词,即使没有具体描述,也是考虑的。
120.如本公开内容和权利要求中使用的,单数形式“一个”、“一种”和“该/所述”包括复数形式,除非上下文另有明确规定;即,除非另有说明,否则“一个/种”意指“一个/种或多个/种”。
121.术语“约”或“大约”意指大致、围绕或在区域中。术语“约”或“大约”进一步意指如通过本领域普通技术人员确定的,在关于特定值的可接受的上下文误差范围内,其将部分取决于该值如何进行测量或确定,即,测量系统的限制或特定目的所需的精确度,例如饲料制剂内的营养素的量。当术语“约”或“大约”与数值范围结合使用时,它通过将界限扩展到所示数值之上和之下来修改该范围。例如“在约0.2和5.0mg/ml之间”意指数值范围的界限扩展到低于0.2和高于5.0,使得所讨论的特定值实现了与范围内相同的功能结果。例如,按照本领域的实践,“约”和“大约”可以意指在1个标准差或多于1个标准差内。可替代地,“约”和“大约”可以意指给定值的至多20%,优选至多10%,更优选至多5%,并且更优选至多1%的范围。
122.如短语例如“a和/或b”中使用的术语“和/或”预期包括“a和b”、“a或b”、“a”和“b”。同样地,如短语例如“a、b和/或c”中使用的术语“和/或”预期涵盖下述实施方案中的每个:a、b和c;a、b或c;a或c;a或b;b或c;a和c;a和b;b和c;a(单独的);b(单独的);和c(单独的)。
123.除非另有说明,否则所有指定“a%-b%”、“a-b%”、“a%至b%”、“a至b%”、“a%-b”、“a%至bb都给予其普通和习惯的含义。在一些实施方案中,这些指定是同义词。
124.术语“基本上”或“基本的”意指所描述或请求保护的条件在所有重要方面都与所描述的标准一样发挥功能。因此,“基本上不含”意欲涵盖在所有重要方面作为不含条件发挥功能的条件,即使数值指示一些杂质或物质的存在。“基本的”一般意指大于90%,优选大于95%,最优选大于99%的值。在说明书和权利要求中使用特定值的情况下,除非另有说明,否则术语“基本上”意指对于特定值具有可接受的误差范围。
[0125]“有效量”意指足以引起所提及的效应或结果的量。“有效量”可以使用与所述目的有关的已知技术,以经验和常规方式进行确定。
[0126]
如本文使用的,提及查询核苷酸序列或氨基酸序列与核苷酸或主题氨基酸序列之间的“百分比序列同一性”应理解为指的是使用本领域已知的执行成对序列比对的合适算法或软件程序计算的同一性值。查询核苷酸序列或氨基酸序列可以通过本文的一项或多项权利要求中鉴定的核苷酸序列或氨基酸序列进行描述。查询序列可以与主题序列100%相同,或者与主题序列相比,它可以包括至多特定整数数目的核苷酸或氨基酸改变(例如点突变、取代、缺失、插入等),使得%同一性小于100%。例如,查询序列与主题序列至少60、70、80、85、90、91、92、93、94、95、96、97、98或99%等同。为了计算百分比同一性,查询序列和主题序列可以对于指定区域(例如,长度至少约40、45、50、55、60、65个或更多个核苷酸或氨基酸的区域,并且可以直到主题核苷酸序列或氨基酸序列的全长)上的最大对应性进行比较并比对。比对可以通过needleman&wunsch(1970)j.mol.biol.48,443-453中公开的needleman-wunsch全局比对算法来确定,并且在可公开获得的全局比对工具emboss needle(hypertexttransferprotocolsecure://worldwideweb.ebi.ac.uk/tools/psa/)中实施。比对可以用空位开放罚分10和空位延伸罚分0.5、blosum矩阵62来确定。实施
needleman-wunsch全局比对算法的替代或另外的全局比对工具是emboss stretcher。
[0127]
除非另有说明,否则术语“多肽”指能够充当所选抗原的任何长度的多肽。形成本发明的多肽的氨基酸聚合物可以是线性或分支的,它可以包含修饰的氨基酸,并且它可以被非氨基酸中断。该术语还涵盖了已天然地或通过干预进行修饰的氨基酸聚合物;例如,二硫键形成、糖基化、脂化、乙酰化、磷酸化、或者任何其它操作或修饰,例如与标记组分缀合。在该定义内还包括的是例如含有一种或多种氨基酸类似物(包括例如非天然氨基酸等)的多肽,以及本领域已知的其它修饰。多肽可以作为单链或结合链存在。
[0128]
术语“包含”涵盖了“包括”以及“由......组成”,例如,“包含”x的组合物可以专一地由x组成,或可以包括另外的事物例如x y。
实施例
[0129]
实施例1.百日咳博德特氏菌的遗传改造:材料和方法
[0130]
细菌菌株和生长条件
[0131]
携带遗传脱毒的百日咳毒素的百日咳博德特氏菌tohama ipt-9k/129g(ptg)用于本研究中。细菌在regan-lowe charcoal(becton dickinson)琼脂平板上在37℃下生长三-五天。bp的液体培养在改良的stainer-scholte培养基(ss)中执行,所述培养基补充有0.4%(w/v)l-半胱氨酸单盐酸盐(sigma)、0.1%(w/v)feso4(aros organics)、0.4%(w/v)抗坏血酸(sigma)、0.04%(w/v)烟酸(sigma)、1%(w/v)还原型谷胱甘肽(sigma),使用2ml 96孔深孔板(eppendorf)用于微小规模培养(600μl),并且使用0.25-2l具有通风杯的带挡板烧瓶(corning)用于小规模和大规模培养(50-300ml)。
[0132]
细菌培养物在37℃下在定轨振荡器中生长,使微板以350rpm搅动且使烧瓶以180rpm搅动。在整个生长过程中监测在600nm处的培养物吸光度。当达到稳定期时,通过在4℃下以4000xg离心30
′
(微小规模培养)或在4℃下以10000xg离心20
′
(烧瓶培养)来抽取细菌培养物,团块在-20℃下冷冻用于基因组dna纯化,并且上清液用0.22μm真空过滤瓶(millipore)进行双重过滤,并贮存于4℃下用于进一步加工。
[0133]
通过经由infinity 200pro分光光度计(tecan)对培养物等分试样的吸光度测量(在600nm处的od)来监测微小规模细菌生长。
[0134]
psortp1,prtp1质粒的衍生物,大肠杆菌s17-1λpir(biomedal)供体菌株和put-mini-tn5-kmr载体(biomedal)用于百日咳博德特氏菌接合。
[0135]
大肠杆菌细胞在luria bertani(lb)培养基(bectondickinson)中在旋转振荡器上在37℃和180rpm下常规生长16小时。培养基补充有对于每种菌株适当的sigma抗生素:链霉素400μg/ml、卡那霉素25-50μg/ml、萘啶酸20μg/ml、氨苄青霉素50-100μg/ml。
[0136]
百日咳博德特氏菌中的新型超级起泡突变的鉴定
[0137]
细菌基因组中的转座子插入位点通过转座子末端随机扩增(random amplification of transposon ends)(rate)(karlyshev av等人,biotechniques,2000年6月;28(6):1078、1080、1082)进行鉴定。rate是以rate pcr和rate产物的测序为代表的两步过程。简言之,将单个细菌cfu接种到50μl水内,并且在99℃下温育10分钟。使用high-fidelity dna polymerase(neb)和下述加热程序执行pcr:1个循环(94℃1分钟);30
个循环(94℃30秒;55℃30秒;72℃30秒);包括taq spyking的30个循环(94℃30秒;30℃30秒;72℃30秒);30个循环(94℃30秒;55℃30秒;72℃1分钟);1个循环(72℃5分钟)。rate pcr产物通过在1%琼脂糖凝胶上运行的电泳进行显现。阳性pcr产物通过qiaquick pcr纯化试剂盒(qiagen)遵循程序进行纯化,然后通过p3730xl dna analyzer (thermofisher)进行测序。
[0138]
基因组dna纯化
[0139]
基因组dna从来自携带转座子插入(tn5)的bp克隆的微小规模生长的细菌团块中进行纯化。根据制造商对于革兰氏阴性菌的说明,通过genelute bacterial genomic dna kit(sigma)分离基因组dna。在100μl预热的无核酸酶的水中执行dna洗脱。通过在nanodrop nd-1000分光光度计(euroclone)处的测量来确定纯化的dna浓度。所有样品都稀释至10ng/μl,并且通过在miseq(illumina)处的ngs测序进行分析。
[0140]
通过超速离心的gmma纯化
[0141]
来自百日咳博德特氏菌或大肠杆菌培养物的无细胞上清液经受在聚碳酸酯离心管(beckman)中以175,000xg和在4℃下的超速离心3小时,所得到的gmma团块用dulbecco氏磷酸盐缓冲盐水(d-pbs)进行洗涤,在4℃下以175,000xg进一步超速离心1小时。通过在4℃下的过夜振荡,将团块重悬浮到200μl d-pbs内。通过在室温下用100ubenzonase酶(roche)的最后2小时处理,从gmma样品中去除任何dna污染。最后使用0.22μm孔径过滤器(millipore)过滤纯化的gmma。
[0142]
通过切向流过滤(tff)的gmma纯化
[0143]
通过tff获得来自大量细胞培养物上清液(300ml)的细胞外囊泡的分离和浓缩。样品使用0.22μm孔径过滤器(millipore)进行过滤,在4℃下与100u benzonase一起温育48小时,然后使用具有300kda膜孔径的中空纤维过滤模块(sartorius)在tff系统(sartorius)中浓缩至200ml。在所有tff步骤中,跨膜压力设定为0.45psi。柱用500ml milli-q水进行引发,并且样品用4l过滤的pbs1
×
进行渗滤,以去除蛋白质污染物并同时进行缓冲液更换。囊泡在tff中进一步浓缩至50ml,用2l过滤的pbs1
×
进行渗滤,在tff中浓缩至大致15ml,并且最后使用0.22μm孔径过滤器(millipore)进行过滤。
[0144]
通过lowry测定的gmma蛋白质定量
[0145]
来自gmma和上清液样品的蛋白质含量用改良的lowry方法(markwell ma等人,anal biochem,1978年6月15日;87(1):206-10)进行确定。该测定在96孔平底透明聚苯乙烯板(costar)上进行,使用每个样品的一式三份等分试样并采用bsa(biorad)来执行标准曲线。将5μl每个未稀释的样品和pbs稀释的样品以及bsa加入每个孔中,并且与新鲜制备的dctm protein assay reagent(biorad)混合。lowry试剂s以2%(v/v)与lowry试剂a混合,并且混合物按25μl/孔进行等分。然后向每个样品中加入200μl lowry试剂b,并且使板在室温下温育15分钟。使用infinity 200pro分光光度计(tecan),针对pbs/试剂空白测量在750nm处的吸光度。
[0146]
通过fm4-64染料染色的gmma脂质定量
[0147]
荧光染料fm4-64(mcbroom aj等人,j bacteriol,2006年8月;188(15):5385-92)用作培养物上清液中的gmma的定量标记物。5μl fm4-64染料(molecular probes,100μg/ml)与96孔平底黑色聚苯乙烯板(costar)中的45μl 0.22μm过滤的上清液混合,并立即涡
旋。通过将板旋转几秒钟去除气泡,并且通过infinity 200pro板阅读器(tecan)伴随在515nm处的激发和在640nm处的发射来测量fm464荧光。与培养基混合的fm4-64探针用作基线并减去样品读数。在培养物稀释物中确定荧光强度(fi)值,并且通过将荧光值除以在收获时记录的培养物od
600nm
来计算囊泡的克隆特异性产生。相对囊泡形成通过每个实验组中克隆的特定荧光与野生型菌株的特定荧光之间的比率进行确定。
[0148]
纳米颗粒跟踪分析(nta)
[0149]
nanosight ns300(malvern ltd)用于确定膜颗粒大小和浓度,如先前所述的(olaya-abril a等人,j proteomics,2014年6月25日;106:46-60)。纯化的gmma或细菌培养物的双重过滤上清液以在0.02μm过滤的d-pbs中1:200-1:5000的稀释度范围加载到测量室内。测量通过捕获60秒的5次测量以流动模式来执行,具有20(~2.1μl/分钟)的流速,产生60-90个颗粒/帧。所有测量都在室温下执行,并且捕获结果使用nanosight nta 3.2软件进行分析。在样品测量前,通过测量milliq稀释剂60秒,确认了milliq稀释剂含有少于1.0个颗粒/帧。所有测量都在室温下执行。
[0150]
sds-page
[0151]
如先前所述的(laemmli uk,nature 1970年8月15日;227(5259):680-5),通过sds-page(4-12%分离凝胶,thermofisher)分析无细胞培养物上清液和纯化的gmma级分。在细菌噬菌体t4的头部组装过程中结构蛋白的切割。通过novex sharp pre-stained protein standard(thermofisher)执行样品的分子量估计。凝胶随后用于通过simplyblue safestain(thermofisher)的考马斯染色或通过银染色试剂盒(thermofisher)的银染色分析。染色根据制造商的说明来执行。
[0152]
免疫印迹分析
[0153]
上清液中包含的蛋白质在sds-聚丙烯酰胺凝胶上进行分离,通过i-blot迷你系统(thermofisher)转移到硝酸纤维素膜上,然后用补充有5%脱脂奶粉的pbs 0,1%v/v tween 20缓冲液(sigma)进行封闭。膜用作为一抗的抗全bp omv小鼠血清进行探测,并且用作为二抗的与hrp缀合的抗小鼠igg(sigma)进行处理。化学发光信号用supersignal west pico plus化学发光底物(thermofisher)进行显色。
[0154]
透射电子显微镜检查(tem)
[0155]
通过具有负染色的tem分析来显现nomv。将样品(5μl)加载到辉光放电的铜300正方形网状格栅(agar scientifc)上30秒,吸干过量样品,并且使用nanow(nanoprobes)对格栅进行负染色30秒。使用tecnai g2 spirit(fei thermofisher)获取显微照片,并且使用veleta ccd(emsis)获取囊泡图像。
[0156]
百日咳博德特氏菌突变体验证设计
[0157]
突变型构建体被设计为生成重组psortp1质粒,其包含定位在psortp1限制位点处的tn5突变的构建体序列。每个构建体都含有限制位点,在待突变的每个基因的上游和下游的tohama i基因组区域,以及用于转化接合子(transconjugants)选择的卡那霉素抗性盒(kanr)。
[0158]
重组质粒如通过生产商描述的通过ezna plasmid mini kit ii(omega)进行纯化,然后根据制造商的说明用于转化大肠杆菌s17-1λpir接合供体菌株。
[0159]
实施例2.百日咳博德特氏菌rpsa突变体的omv释放增加
[0160]
开发了实施以微小规模板形式筛选的转座子tn5文库的使用的随机诱变方法,以鉴定很可能引起nomv释放增加的突变。转化接合子通过新型接合方案生成,所述方案进行优化以产生4x106单个突变体/接合的tn5突变体文库。转化接合子通过单菌落挑取进行分离,并且在96孔深孔板内生长。确定为了具有相似的克隆生长曲线,所选菌落的大小必须是可比较的。通过使用能够防止液体蒸发的盖子进一步优化微小规模培养物的生长。
[0161]
培养物上清液中的nomv释放通过fm4-64荧光来评估,将每个荧光值针对相应克隆的光密度进行归一化,并且最终将其与在同一板中生长的wt克隆的光密度(fiow)进行比较。总共筛选了5,670个随机克隆。在其中,选择了fiow超过2倍的95个克隆,并且通过微小规模培养物中的至少两次进一步实验进行重新评估。如图1中报告的,所选的95个克隆中的26个在后续微小规模生长过程中确认了fiow》1。最后,在其中的八个克隆在以微小规模的多重测试过程中导致具有fiow》2(图2)。
[0162]
bp染色体中的转座子插入通过rate进行定位。有趣的是,3个得分最高的克隆中的插入与单个基因rpsa(bp0950,30s核糖体蛋白s1)相关。
[0163]
为了精确地定位这些克隆中的转座子插入,基因组dna进行纯化并且通过下一代测序(ngs)进行分析,揭示了在rpsa基因内的所有转座子插入(50d6、83g6、77d8克隆)都存在于r3结构域的下游(图3)。
[0164]
在50d6突变体克隆内的tn5转座子插入存在于77d8和83g6克隆中分别在最后376个rpsa核苷酸之前以及在最后308和77个rpsa核苷酸之前。在转座子插入的5
′
端处检测到重复序列(关于50d6的gctcga,关于77d8的catcaatg,关于83g6克隆的tgtccgagg)。
[0165]
为了进一步确认以可持续生长曲线过量生产gmma的能力,3个rpsa突变体克隆(50d6、83g6、77d8)和通过随机诱变鉴定但与rpsa无关的另外克隆(66d1)在小规模培养中生长。监测wt和突变株在37℃和180rpm下的细菌生长,直到生长的晚期稳定期。释放的囊泡在生长结束时从培养物上清液中进行纯化,并且通过荧光分析进行定量。为了评估纯化的释放囊泡的量,执行了nta。两次实验的结果在图4(a-b)中报告。50d6和66d1克隆在小规模培养中显示出与其亲本wt菌株相似的生长速率,并且如通过荧光结果确认的,相对于其亲本wt菌株,在上清液中释放两倍的gmma量。增加的囊泡形成也通过纯化囊泡的nta得到确认(图4c-d)。
[0166]
在大规模培养(300ml)中进一步调查了gmma释放。如图5中示意性地表示的,50d6和66d1克隆显示出其亲本wt菌株相似的生长速率。gmma通过tff从细胞培养物上清液中进行分离,然后通过lowry、nta和荧光分析进行定量。所有程序都确认了相对于wt菌株,随机突变体克隆中释放颗粒的数目增加。
[0167]
实施例3.来自rpsa突变体的囊泡的表征
[0168]
为了评估wt和随机突变体克隆两者的蛋白谱,从细菌生长的晚期稳定期收集上清液和纯化的囊泡。上清液的sds-page分析揭示了类似的条带模式,而在纯化的gmma中可以观察到差异,其潜在地通过在样品的超速离心过程中发生的部分沉淀引起(图6)。通过经由使用抗omv血清的免疫印迹分析蛋白谱,确认了培养物上清液中的膜囊泡的存在。在图7中报告的结果显示了通过血清识别的清楚的omv蛋白质梯。
[0169]
通过透射电子显微镜检查(tem)分析了来自wt和随机突变体的囊泡的形态。如图8中所示的,在所有样品中都可以显现圆形囊泡。囊泡的直径在50至100nm的范围内是可比较
的。
[0170]
实施例4.百日咳博德特氏菌中的tn5突变体的验证
[0171]
50d6克隆的干净突变体在bp tohama iptg菌株中生成。c7(rpsa 377-ins-kanr)突变体(seq id no:51和seq id no:53)包含在bp0950基因(rpsa)的最后376个核苷酸之前的kanr插入,即在50d6克隆中的tn5插入的相同位置处。c8突变体包含bp0950基因(rpsa)的最后376个核苷酸的缺失,即50d6克隆中的tn5插入位置下游的序列被缺失。缺失区域被kanr和spei限制位点替换。c9(ihfb::kanr)突变体(seq id no:52)包含整个bp0951基因(ihfb)的缺失。缺失区域被kanr和spei限制位点替换。
[0172]
突变体如实施例2中所述的进行分析。c7和c9(ihfb::kanr)两者均显示了与野生型(“wt”)相比显著更高的fiow,如图9中所示的。在c7和c9菌株之间并未观察到显著差异。
[0173]
实施例5:大肠杆菌的遗传改造:材料和方法
[0174]
构建体设计
[0175]
为了确认通过rpsa突变诱导的nomv释放增加,另一种革兰氏阴性菌,非致病性大肠杆菌,通过定点诱变进行突变。
[0176]
来自bp tohama i基因组的rpsa-ihfb基因座中的rpsa和ihfb基因的编码序列之间不存在基因间区域,提示了可能的多顺反子转录物。用bprom,softberry (solovyev v,&a salamov,metagenomics and its applications in agriculture,biomedicine and environmental studies,2011,61-78)并未预测到规范的启动子序列。与bp相比,大肠杆菌bl21(de3)rpsa-ihfb基因座的基因组序列展示了在两个基因之间更长的基因间。另外,在大肠杆菌中,预测在159个碱基对的基因间区域内的功能性启动子以及ihf转录因子(tf)键合位点(bprom,softberry)。
[0177]
使用clustal omega在线软件(hypertexttransferprotocolsecure://worldwideweb.ebi.ac.uk/tools/msa/clustalo/)比对bp(seq id no:13,不包含其5个n末端氨基酸)和ec s1(seq id no:14)的氨基酸序列,并且显示彼此具有63%的百分比同一性。bp 50d6克隆中的tn5插入点在图10中以粗体指示,并且落入谷氨酸编码gaa密码子的第二个碱基内。被ec中的相应突变中断的第一个氨基酸是丙氨酸而不是谷氨酸,但之前(以及将保留在最终菌株中)的序列是充分保守的(图10)。
[0178]
ihfb基因在两种生物之间是不同的。bp蛋白更长(119aa相对于94aa),并且同一性在94aa片段上为57%。
[0179]
在ec中设计了三种不同的构建体:(i)ins rpsa,重现50d6 bp克隆的tn5插入,具有在bp0950基因(rpsa)的最后376个核苷酸之前插入的kanr;(ii)δrpsa 3’,具有rpsa的最后一个r4结构域的缺失(从先前构建体的插入点到rpsa终止密码子),具有倒置的卡那霉素盒以重现80g6和77d8 bp克隆;(iii)δihfb,具有ihfb基因的缺失(rpsa的下游)。
[0180]
基因组突变也使用相同的方法在脑膜炎奈瑟氏球菌和淋病奈瑟氏球菌中获得。生成基因组突变体,并且通过直接检测至少在两个生物重复中的培养物上清液中释放的囊泡,在不同温度(25℃、30℃和37℃)下测试gmma产生。为了确认gmma产生并评价通过突变株产生的gmma中的蛋白谱,gmma进行纯化并且通过lowry和sds-page进行分析,如上文节段中所述的。
[0181]
突变体生成
[0182]
ec bl21(de3)的基因组突变体通过λred重组生成,如先前所述的(datsenko和wanner proc natl acad sci usa,2000年6月6日;97(12):6640-5)。用50ng携带λred重组酶基因的质粒dna pdk46转化ec bl21(de3)(化学感受态,neb)的等分试样,并且在30℃下在100mg/l氨苄青霉素的存在下进行选择。然后通过在100mg/l氨苄青霉素和1mm阿拉伯糖(以诱导λred重组酶表达)的存在下,在30℃下培养直至中期指数生长期(od
600nm
=0,4),使重组pkd46-ec bl21(de3)成为电感受态的。然后收获细菌并用无菌且冰冷的h2o洗涤3次,并且最后重悬浮于无菌冰冷的甘油10%v/v中。细菌在洗涤步骤过程中浓缩至50x。
[0183]
然后在2,5kv下对50ul电感受态细菌进行电穿孔,其中对于每种构建体使用300-400ng纯化的pcr。突变体选择在lb琼脂中的25mg/l卡那霉素的存在下在37℃下进行24小时。通过菌落pcr用外部引物筛选关于每种转化的三个克隆。
[0184]
小规模培养
[0185]
突变体在不同温度(25℃、30℃和37℃)下生长,并且每一种条件测试至少2次。简言之,使5ml lb培养基的预接种物对于25℃和30℃条件温育16-18小时,并且对于37℃条件在37℃和180rpm下温育8小时。预培养物在具有通风杯的一次性的带挡板的250ml烧瓶(corning)中的50ml htmc培养基中以1:100进行稀释,并且在不同温度和180rpm振荡下温育直至晚期稳定期。od
600nm
以分光光度法在时间过程中进行监测。通过在4℃下以4000rpm离心30’收获达到晚期稳定生长期时的培养物。所得到的上清液通过0.22μm stericup(millipore)进行过滤,并且贮存于4℃下用于进一步分析。
[0186]
gmma纯化
[0187]
使35ml上清液经受在4℃下以32000rpm的超速离心3小时(使用32ti摆动转子)。用35ml无菌pbs以32000rpm/4℃/2小时执行一个洗涤步骤。在振荡的同时,使含有囊泡的团块在4℃下重悬浮于0.15-0.6ml无菌pbs中16小时。
[0188]
gmma定量
[0189]
遵循来自biorad的方案“dc蛋白质测定”(cat n
°
500-0114),通过lowry测定对纯化的gmma进行定量。1mg/ml bsa的连续2步稀释物用于标准曲线。gmma也通过用fm4-64染料(thermofisher scientific,目录号:t13320)以最终稀释度1:100染色,直接从无细胞上清液中进行定量。荧光强度也对od
600nm
值进行归一化,以计算关于每种突变体的特定gmma产率。然后将获得的值除以野生型值,并且报告为相对于野生型的倍数增加(fiow)。
[0190]
实施例6:大肠杆菌的遗传改造:结果和讨论
[0191]
在37℃下来自突变株的gmma生产
[0192]
如材料和方法中所述的,在摇瓶中培养突变株和相应的野生型菌株,并且记录生长曲线(表1)。与野生型相比,所有三种突变体都显示了略微更低的生长速率和最终生物质产率。在不同的突变体之间观察到可比较的生长曲线。
[0193]
表1
[0194][0195]
上清液中释放的gmma直接从无细胞培养物上清液中进行测量,揭示了两种rpsa突变体(插入或3
′
缺失突变体)产生的gmma是野生型ecbl21(de3)菌株的3至5倍,而ihfb敲除突变体产生无法检测的nomv量(图11)。
[0196]
gmma通过超速离心从培养物上清液中进行纯化,并且基于总蛋白含量进行定量。两种rpsa突变体均显示了与野生型菌株相比,在体积产率方面3至4倍的gmma生产增加(图12)。这略微低于通过在上清液中的直接测量观察到的,很可能是由于来自不同培养物的不同纯化产率以及野生型菌株的部分裂解,所述部分裂解可以在上清液中释放细胞内蛋白,其可以影响总蛋白定量。确认了δihfb#3突变体的极低产率。
[0197]
在sds-page中分析纯化的gmma,以比较蛋白质模式(图13)。两种rpsa突变体和wt的蛋白谱是可叠加的,并且ihfb突变株的普遍条带对应于在其它菌株中观察到的蛋白谱。所有gmma制剂都显得干净,指示了细菌在收获时仍然存活。在第一个实验中,仅对于野生型制剂可检测到轻微的涂片,提示了培养物在它开始裂解时进行取样,提示了关于纯化的gmma计算的fiow可能被略微低估。
[0198]
在25℃和30℃下来自突变株的gmma生产
[0199]
调查了大肠杆菌突变体在各温度下释放gmma的能力,所述温度频繁用于重组蛋白表达-例如用于生产富含目的抗原的gmma。因此,如材料和方法中所述的,在25℃和30℃下在摇瓶中培养突变株和相应的野生型菌株。与野生型相比,突变体的生长曲线的特征在于较低的生长速率和略微更低的最终生物质产率(图14)。不同的突变体显示了可比较的生长曲线。
[0200]
上清液中释放的gmma直接从无细胞培养物上清液中进行测量,揭示了两种rpsa突变体(插入或3
′
缺失突变体)在30℃下产生的gmma是野生型ec bl21(de3)菌株的4至6倍,并且在25℃下是野生型ec bl21(de3)菌株的4至5倍。最高的比产率在30℃下获得。尽管ihfb敲除突变体在较低的温度下产生可检测量的gmma,但其gmma产率低于野生型之一(图15)。
[0201]
实施例7:粘膜炎莫拉氏菌的遗传改造
[0202]
在另一种革兰氏阴性菌粘膜炎莫拉氏菌中执行了进一步的实验,以确认通过rpsa和ihfb突变诱导的nomv释放增加。具体而言,评估了δrpsa-ct突变(rpsa的最后363bp的缺失)和δihfb突变(ihfb的完全缺失)。
[0203]
粘膜炎莫拉氏菌转化
[0204]
使用了粘膜炎莫拉氏菌bbh18(genbank程序集登录:gca_000092265.1)菌株,从来自在恶化期间的copd患者的痰分离物中分离的血清抗性谱系菌株。
[0205]
粘膜炎莫拉氏菌bbh18的定向基因缺失突变体通过靶基因与卡那霉素抗性盒(通过seq id no:56和58显示)的等位基因交换获得。取代基因的编码序列的卡那霉素抗性盒
与其5
′
和3
′
~600bp侧翼区融合,并且转化到天然感受态的粘膜炎莫拉氏菌bbh18细菌内。为此,将~1μg纯化的pcr产物与重悬浮于pbs中的25μl细菌混合,在37℃下温育5-6小时,并且转化体在bhi 30μg/ml卡那霉素平板上选择两次。转化体通过pcr验证在基因组中的正确重组。
[0206]
生长条件
[0207]
在37℃下,将菌株从甘油原种划线培养到bhi琼脂平板上on;将单个菌落以od600=0.1接种到150ml bhi内,并且在37℃、185rpm下温育on。培养物以4000rpm离心20分钟,并且上清液通过0.22μm过滤器进行过滤。
[0208]
gmma纯化
[0209]
为了纯化囊泡,使140ml上清液经受在4℃下以32000rpm的超速离心3小时(使用32ti摆动转子)。一个洗涤步骤在4℃下用140ml无菌pbs以32000rpm执行2小时。最后,在振荡的同时,使含有囊泡的团块在4℃下重悬浮于1.5-2.2ml无菌pbs中16小时。
[0210]
gmma定量
[0211]
遵循来自biorad的方案“dc蛋白质测定”(cat n
°
500-0114),通过lowry测定对纯化的gmma进行定量。2mg/ml bsa的连续2步稀释物用于标准曲线。
[0212]
负染色电子显微镜检查
[0213]
将5μl的每个样品(wt、δrpsa-ct和δihfb gmma)(2od/ml)加载到辉光放电的铜300正方形网状格栅上30秒。在吸干过量样品后,使用nanow对格栅进行负染色30秒。使用tecnai g2 spirit分析样品,并且使用tvips temcam-f216(em-menu软件)获取图像。
[0214]
实施例8:粘膜炎莫拉氏菌的遗传改造:结果
[0215]
rpsa和ihfb突变体显示了与野生型菌株相比,在体积产率方面7.5至14倍以及在比产率方面12至22倍的gmma生产增加(图16)。
[0216]
负染色电子显微镜检查(图17)也确认了gmma产生。它还显示了相应gmma的大小,相对于δrpsa-ct和δihfb gmma,对于wt gmma具有大的gmma,并且δihfb gmma略微大于δrpsa-ct gmma。δrpsa-ct和δihfb gmma也具有一致的大小。
[0217]
实施例9:确定rpsa截短是否负责超级起泡:材料和方法
[0218]
为了进一步调查rpsa超级起泡突变背后的机制,且特别是它是否是由于rpsa截短或下调,生成了在野生型和rpsa突变体背景下过表达全长(“fl”)或r4截短(“tr”)ecrpsa的大肠杆菌菌株,并且测试了gmma生产产率。
[0219]
引物设计和构建体生成
[0220]
表达载体pet15-tev-ccdb质粒(ampr)用于rpsa过表达,以允许pipe克隆并使用强诱导型t7启动子。并未添加标签,并且因此从最终构建体中排除his标签和tev位点。
scientific c862003)内
[0227]
根据制造商的说明,通过使用gotaq2x green master mix(promega)的菌落pcr,用外部引物筛选关于每种转化的三个克隆。反应在20μl中进行。
[0228]
循环:95℃5
′‑
(95℃30
″
;55℃30
″
;72℃1
′
30
″
)x30-72℃7
′
[0229]
最后,使所选的克隆经受测序,以确认在克隆过程中并未将突变引入编码序列内。
[0230]
菌株生成
[0231]
用下述质粒转化自制的化学感受态大肠杆菌bl21(de3)ins rpsa(构建体#7)突变体细胞和野生型bl21(de3)(one shot
tm bl21(de3)化学感受态大肠杆菌,thermo scientific目录#c600003):
[0232]
a.pet21b 空(pet-21b( )dna-novagen|69741-merck millipore,批号#m00067689):作为对照的空载体
[0233]
b.pet15-rpsafl:关于rpsa全长的过表达载体
[0234]
c.pet15-rpsatr:关于截短的rpsa的过表达载体
[0235]
小规模培养
[0236]
使5ml lb培养基 氨苄青霉素100mg/l的预接种物在37℃和180rpm下温育8小时。
[0237]
预培养物在具有通风杯的一次性的带挡板的250ml烧瓶(corning)中的50ml htmc培养基 氨苄青霉素100mg/l中进行1:100稀释,并且在30℃和200rpm下温育15小时。记录了od600nm(在htmc培养基中的1:40稀释度)。然后将培养物在4℃下以2000rpm离心10
′
,并且弃去上清液。将团块重悬浮于补充有100mg/l氨苄青霉素和iptg 1mm的50ml新鲜htmc培养基中,以诱导重组rpsa表达。由于死细菌与耗尽的上清液一起被弃去,因此在重悬浮后记录以od600nm表示的新细菌浓度。培养物在30℃和200rpm下再次温育,并且监测od600nm直至达到稳定生长期(在htmc培养基中的1:40稀释度)。
[0238]
最终通过在4℃下以4000rpm离心30’收获培养物。所得到的上清液通过0.22μm stericup(millipore)进行过滤,并且贮存于4℃下用于进一步分析。团块贮存于-20℃下直至进一步表征。
[0239]
rpsa重组表达和溶解度的评估
[0240]
为了验证rpsa(fl和tr)的表达,由收集的团块制备总裂解物和可溶性裂解物,以在sds-page中进行比较。简言之,来自50ml培养物的团块重悬浮于对应于最终od600nm值的体积中,以获得过滤灭菌的pbs中的50od/ml悬浮液,0.5ml的每种悬浮液在4℃下以13000rmp离心5
′
,并且弃去上清液。然后将每个团块重悬浮于先前溶解于pbs中的1ml cellytic express(sigma aldrich)中,并且在室温下在温和搅动下温育30’。将50μl的总裂解物转移到新管中(总裂解物),同时剩余的悬浮液在4℃下以13000rmp离心15’,以获得可溶性级分。总裂解物和可溶性裂解物贮存于-20℃下直至进一步操作。
[0241]
sds-page分析
[0242]
为了检查rpsa表达,如上文所述制备并预先在pbs中1:5稀释(20μl样品 80μl pbs)的13μl每种总裂解物和可溶性裂解物与5μl的4x上样缓冲液和2μl的10x还原剂在总共20μl中混合。
[0243]
对于gmma蛋白质模式分析,将5μg的每种gmma制剂与5μl的4x上样缓冲液和2μl的10x还原剂在总共20μl中混合。
[0244]
所有样品都在100℃下在热块(thermoblock)中处理5
′
用于变性。将样品加载到4-12%bis-tris聚丙烯酰胺凝胶(nupage,thermo scientific)中。作为分子量标记物,加载了10μl的novexsharp预染色的蛋白质梯(thermo scientific)。凝胶在mes缓冲液中在200v下运行40分钟。染色在室温下在搅动下用coomassie(giottobiotech)执行1小时。脱色在室温下在搅动下过夜完成。凝胶图像用geldoc xr(biorad)和imagelab软件来获取。
[0245]
gmma纯化
[0246]
为了纯化囊泡,使35ml上清液经受在4℃下以32000rpm的超速离心3小时,使35ml上清液在32000rpm/4℃/3小时(使用32ti摆动转子)下超速离心。用35ml无菌pbs以32000rpm/4℃/2小时执行一个洗涤步骤。最后,在振荡的同时,使含有囊泡的团块在4℃下重悬浮于0.15-0.6ml无菌pbs中16小时。
[0247]
gmma定量
[0248]
遵循来自biorad的方案“dc蛋白质测定”(cat n
°
500-0114),通过lowry测定对纯化的gmma进行定量。1mg/ml bsa的连续2步稀释物用于标准曲线。
[0249]
gmma也通过使用fm4-64染料(thermofisher scientific,目录号:t13320)的荧光测定,直接从上清液中进行定量。它用红色荧光(激发/发射515/640nm)染色外膜囊泡。染料以1:100的最终稀释度使用。
[0250]
实施例10:确定rpsa截短是否负责超级起泡:结果
[0251]
菌株培养和rpsa过表达
[0252]
下述菌株以小规模进行培养,以诱导rpsa过表达并评估gmma产生:
[0253]
1.bl21(de3)-pet21b 空(wt_空)
[0254]
2.bl21(de3)-pet15-rpsafl(wt_rpsa_fl)
[0255]
3.bl21(de3)-pet15-rpsatr(wt_rpsa_tr)
[0256]
4.bl21(de3)rpsains-pet21b 空(rpsains_空)
[0257]
5.bl21(de3)rpsains-pet15-rpsafl(rpsains_rpsa_fl)
[0258]
6.bl21(de3)rpsains-pet15-rpsatr(rpsains_rpsa_tr)
[0259]
在bl21(de3)野生型和bl21(de3)rpsains超级起泡突变体两者中,诱导了rpsa全长(fl)和截短(tr)两者的过表达。用空pet载体转化的菌株用作阴性对照,因为培养基中的氨苄青霉素可以影响在生长过程中的囊泡释放。
[0260]
为了评估测试菌株中的rpsa表达和溶解度,如图18中所示的运行sds-page。rpsa fl和tr两者的重组表达在两种菌株中均以高水平得到确认并且是完全可溶的。
[0261]
培养物上清液中的gmma生产产率的评估
[0262]
使用具有fm4-64染料的荧光测定,直接从培养物上清液中评估对于不同菌株获得的gmma产率。这种染料在它与双脂质层相互作用时变得发荧光,并且所产生的荧光与测试样品中的囊泡量成比例。
[0263]
由培养物上清液记录的荧光强度作为三个技术重复的平均值的绝对荧光进行比较(误差条计算为从空白值扣除得出),并且报告为体积产率。荧光强度也对od
600nm
值进行归一化,以便计算关于每种突变体的特定gmma产率。使用由空质粒转化的野生型菌株的平均值作为基线,来计算作为体积和特异性fiow两者的fiow(相对于野生型的倍数增加)(图19)。
[0264]
gmma纯化和表征
[0265]
为了确认在培养物上清液中观察到的,gmma通过超速离心进行纯化,并且通过lowry测定基于总蛋白含量进行定量。使用由空质粒转化的野生型菌株的平均值作为基线,来计算体积产率和比产率两者,并且报告为相对于野生型的倍数增加。所获得的值在下表中:
[0266][0267]
为了确认,上表中突出显示的框中的数字是9,739013。
[0268]
所获得的fiow值类似于通过fm4-64染色在培养物上清液中用直接gmma定量获得的数据,除了显示远高于预计的fiow的rpsains_空之外。
[0269]
为了验证纯化的gmma的纯度,运行了sds-page(图20)。
[0270]
gmma制剂都是高度纯的,除了其中可溶性蛋白质的明确污染在凝胶上显而易见的rpsains_空样品之外。这是预计的,因为定量结果与培养物上清液中检测到的荧光不一致。
[0271]
因为由于所获得样品的低纯度,rpsains_空产率被极大地高估,因此关于这种菌株基于总蛋白含量(通过lowry)计算的gmma产率必须进行归一化。
[0272]
为了尝试具有通过rpsa突变/互补获得的gmma产率的更准确估计,密度测定法用于评估30kda孔蛋白的纯度,其量使用imagelab软件在不同gmma制剂中是恒定的。
[0273]
然后fiow值对于不同菌株的纯度比进行归一化,如下表所报告的:
[0274][0275]
为了确认,上表中突出显示的框中的数字是9,739013。
[0276]
通过归一化,新数据与培养物上清液中用荧光强度观察到的更一致。
[0277]
为了进一步确认通过培养物上清液中的gmma的直接定量以及基于蛋白质含量定量且关于纯度归一化纯化的gmma所发现的,还使用荧光探针fm4-64对纯化的gmma进行定量。原理在于,如果低纯度是通过超速离心与gmma共纯化的培养物上清液中的可溶性细胞内蛋白的特定释放的问题,则基于脂质含量的定量将克服这个问题。由于细胞碎片的存在,
无论如何都可能存在低纯度样品的高估,但这与在培养物上清液中观察到的并未不同。
[0278]
基于记录的荧光强度来计算比产率(关于最终od值进行归一化)(图21)。使用基于脂质的定量,所获得的fiow概况与当直接分析培养物上清液时发现的一致,确认了rpsa截短对超级起泡表型的作用。
[0279]
序列描述
[0280]
seq id no:1
[0281]
》百日咳博德特氏菌tohama i rpsa基因[结构域加下划线]
[0282][0283]
seq id no:2
[0284]
》大肠杆菌bl21(de3)rpsa基因
[0285][0286]
seq id no:3
[0287]
>流感嗜血杆菌rd_kw20 hi1220rpsa基因
[0288][0289]
seq id no:4
[0290]
>淋病奈瑟氏球菌fa1090 rpsa基因
[0291]
seq id no:5
[0292]
》脑膜炎奈瑟氏球菌mc58rpsa基因
[0293][0294]
seq id no:6
[0295]
》铜绿假单胞菌pao1 rpsa基因
[0296][0297]
seq id no:7
[0298]
》百日咳博德特氏菌tohama i rpsa操纵子(rpsa-ihfb基因座)[50d6克隆插入位点为粗体,77d8插入位点用单线加下划线,83g6用双线加下划线]
[0299]
[0300][0301]
seq id no:8
[0302]
》大肠杆菌bl21(de3)rpsa操纵子(rpsa-ihfb基因座)[基因间区域加下划线]
[0303][0304]
seq id no:9
[0305]
》脑膜炎奈瑟氏球菌mc58rpsa操纵子(rpsa-ihfb基因座)[基因间区域加下划线;cmr的插入位点为粗体]
[0306][0307]
seq id no:10
[0308]
》淋病奈瑟氏球菌fa1090rpsa操纵子(rpsa-ihfb基因座)[基因间区域加下划线]
[0309][0310][0311]
seq id no:11
[0312]
>流感嗜血杆菌rd_kw20 hi1220 rpsa操纵子(rpsa-ihfb基因座)[基因间区域加下划线]
[0313][0314]
seq id no:12
[0315]
》铜绿假单胞菌pao1 rpsa操纵子(rpsa-ihfb基因座)[基因间区域加下划线]
[0316][0317]
[0318]
seq id no:13
[0319]
》百日咳博德特氏菌tohama i s1蛋白
[0320][0321]
seq id no:14
[0322]
》大肠杆菌bl21(de3)s1蛋白
[0323][0324]
seq id no:15
[0325]
》流感嗜血杆菌rd_kw20 h11220 s1蛋白
[0326][0327]
seq id no:16
[0328]
》淋病奈瑟氏球菌fa1090s1蛋白
[0329][0330]
seq id no:17
[0331]
》脑膜炎奈瑟氏球菌mc58 s1蛋白
[0332][0333]
seq id no:18
[0334]
》铜绿假单胞菌pao1 s1蛋白
[0335][0336]
seq id no:19
[0337]
》百日咳博德特氏菌tohama i rpsa基因,结构域1
[0338][0339]
seq id no:20
[0340]
》百日咳博德特氏菌tohama i rpsa基因,结构域2
[0341][0342]
seq id no:21
[0343]
>百日咳博德特氏菌tohama i rpsa基因,r1
[0344][0345]
seq id no:22
[0346]
>百日咳博德特氏菌tohama i rpsa基因,r2
[0347][0348]
seq id no:23
[0349]
>百日咳博德特氏菌tohama i rpsa基因,r3
[0350][0351]
seq id no:24
[0352]
>百日咳博德特氏菌tohama i rpsa基因,r4
[0353][0354]
seq id no:25
[0355]
>百日咳博德特氏菌tohama i ihfb基因
[0356][0357]
seq id no:26
[0358]
》大肠杆菌bl21(de3)ihfb基因
[0359]
[0360]
seq id no:27
[0361]
>流感嗜血杆菌rd_kw20 hi1220 ihfb基因
[0362][0363]
seq id no:28
[0364]
》淋病奈瑟氏球菌fa1090ihfb基因
[0365][0366]
seq id no:29
[0367]
》脑膜炎奈瑟氏球菌mc58ihfb基因
[0368][0369]
seq id no:30
[0370]
>铜绿假单胞菌pao 1ihfb基因
[0371][0372]
seq id no:31
[0373]
>百日咳博德特氏菌tohama i ihf蛋白
[0374][0375]
seq id no:32
[0376]
》大肠杆菌bl21(de3)ihf蛋白
[0377][0378]
seq id no:33
[0379]
》流感嗜血杆菌rd_kw20 hi1220 ihf蛋白
[0380][0381]
seq id no:34
[0382]
>淋病奈瑟氏球菌fa1090 ihf蛋白
[0383][0384]
seq id no:35
[0385]
>脑膜炎奈瑟氏球菌mc58ihf蛋白
[0386][0387]
seq id no:36
[0388]
》铜绿假单胞菌pao1 ihf蛋白
[0389][0390]
seq id no:37
[0391]
》百日咳博德特氏菌50d6克隆nt序列rpsa基因[tn5加下划线]
[0392][0393]
seq id no:38
[0394]
>百目咳博德特氏菌50d6克隆nt序列rpsa操纵子[tn5加下划线]
[0395][0396]
seq id no:39
[0397]
>百日咳博德特氏菌50d6克隆aa序列s1蛋白
[0398][0399]
seq id no:40
[0400]
》百日咳博德特氏菌77d8克隆aa序列s1蛋白
[0401]
[0402]
seq id no:41
[0403]
》百日咳博德特氏菌83g6克隆aa序列s1蛋白
[0404][0405]
seq id no:42
[0406]
》大肠杆菌突变体ins rpsa#1[定位50d6克隆的kanr插入加下划线]
[0407]
[0408][0409]
seq id no:43
[0410]
》大肠杆菌突变体rpsa 3’#2[kanr插入加下划线]
[0411][0412]
[0413]
seq id no:44
[0414]
》大肠杆菌突变体ins rpsa#1s 1蛋白
[0415][0416]
seq id no:45
[0417]
>大肠杆菌突变体δrpsa 3’#2s 1蛋白
[0418][0419]
seq id no:46
[0420]
>脑膜炎奈瑟氏球菌ng7构建体[氯霉素抗性盒cmr的插入加下划线]
[0421][0422]
[0423]
seq id no:47
[0424]
》脑膜炎奈瑟氏球菌ng8构建体[氯霉素抗性盒cmr的插入加下划线;缺失s1c末端]
[0425][0426]
seq id no:48
[0427]
》脑膜炎奈瑟氏球菌ng9构建体[氯霉素抗性盒cmr插入加下划线;下游基因缺失]
[0428][0429]
seq id no:49
[0430]
》脑膜炎奈瑟氏球菌ng7构建体中的s1蛋白序列
[0431][0432]
seq id no:50
[0433]
>脑膜炎奈瑟氏球菌ng8构建体中的s1蛋白序列
[0434][0435]
seq id no:51
[0436]
》百日咳博德特氏菌突变体c7[定位50d6克隆的kanr插入加下划线]
[0437][0438]
seq id no:52
[0439]
》百日咳博德特氏菌突变体c9[通过用kanr取代的ihfb缺失加下划线]
[0440][0441]
seq id no:53
[0442]
>百日咳博德特氏菌c7构建体中的s1蛋白序列
[0443][0444]
seq id no:54
[0445]
》百日咳博德特氏菌
[0446][0447]
seq id no:55
[0448]
》粘膜炎莫拉氏菌bbh18rpsa基因
[0449][0450]
seq id no:56
[0451]
》突变的粘膜炎莫拉氏菌bbh18rpsa基因(δrpsa-ct)
[0452][0453]
seq id no:57
[0454]
》粘膜炎莫拉氏菌bbh18 s1蛋白
[0455][0456]
seq id no:58
[0457]
》突变的粘膜炎莫拉氏菌bbh18s1蛋白(δrpsa-ct)
[0458][0459]
卡那霉素抗性盒
[0460]
seq id no:59
[0461]
》粘膜炎莫拉氏菌bbh18rpsa操纵子(rpsa-ihfb基因座)[基因间区域加下划线]
[0462][0463]
seq id no:60
[0464]
》粘膜炎莫拉氏菌bbh18rpsa基因
[0465][0466]
seq id no:61
[0467]
》粘膜炎莫拉氏菌bbh18ihfb基因
[0468][0469]
seq id no:62
[0470]
》粘膜炎莫拉氏菌bbh18ihf蛋白
[0471][0472]
seq id no:63
[0473]
>rpsafl(全长rpsa)(基因)
[0474][0475][0476]
seq id no:64
[0477]
>rpsafl(全长rpsa)(蛋白质)
[0478][0479]
seq id no:65
[0480]
》rpsatr(截短的rpsa)(基因)
[0481][0482]
seq id no:66
[0483]
>rpsatr(截短的rpsa)(蛋白质)
[0484][0485]
seq id no:67
[0486]
》v-pipe univ-f引物(pet15载体扩增)
[0487][0488]
seq id no:68
[0489]
>pet15notagrv(pet15载体扩增)
[0490][0491]
seq id no:69
[0492]
》rpsafw(用于rpsa fl和tr扩增的正向引物)
[0493][0494]
seq id no:70
[0495]
》rpsaflrv(用于rpsa fl扩增的反向引物)
[0496][0497]
seq id no:71
[0498]
》rpsatrrv(rpsa tr和tr扩增的反向引物)
[0499]
[0500]
seq id no:72
[0501]
》t7prom(菌落筛选)
[0502][0503]
seq id no:73
[0504]
》seq pet rv(菌落筛选)
[0505][0506]
实施方案
[0507]
1.一种遗传修饰的革兰氏阴性菌细胞,其包含修饰的rpsa基因、修饰的rpsa操纵子和/或修饰的30s核糖体蛋白s1蛋白质。
[0508]
2.实施方案1的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的细菌细胞能够分泌天然外膜囊泡(nomv)。
[0509]
3.实施方案2的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞能够分泌更多数量的nomv。
[0510]
4.实施方案3的遗传修饰的革兰氏阴性菌细胞,其中与野生型细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞能够分泌更多数量的nomv。
[0511]
5.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa基因,其中所述修饰的rpsa基因包含相对于野生型rpsa基因的一个或多个突变,其中所述一个或多个突变定位于rpsa基因的编码区内和/或rpsa基因的非编码区内。
[0512]
6.实施方案5的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含一个或多个核苷酸的插入或由一个或多个核苷酸的插入组成,其中所插入的一个或多个核苷酸选自以下:
[0513]
a.转座子移动元件;
[0514]
b.选择标记物,例如抗生素抗性盒;编码荧光蛋白的基因;或编码抗毒素的基因;和/或
[0515]
c.rpsa基因的片段。
[0516]
7.实施方案5和6中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变使用选自以下的方法进行制备,或者通过选自以下的方法获得或可获得:定点诱变、重组酶介导的方法、λ-red重组酶介导的方法、基于原噬菌体的方法、移动ii组内含子敲除、转座子介导的基因组编辑和crispr-cas基因组编辑。
[0517]
8.实施方案6(a)和7中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述转座子移动元件的插入使用转座子介导的基因组编辑进行制备,或者通过转座子介导的基因组编辑获得或可获得。
[0518]
9.实施方案6(a)至8中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述转座子移动元件选自tn5转座子、tn10转座子、tn3转座子、tn7转座子、is5376转座子、细菌噬菌体mua转座子、is200/is605转座子和is91转座子。
[0519]
10.实施方案9的遗传修饰的革兰氏阴性菌细胞,其中所述转座子移动元件是tn5转座子。
[0520]
11.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修
饰的细菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1:
[0521]
a.包含相对于野生型30s核糖体蛋白s1的一个或多个突变;和/或
[0522]
b.包含相对于野生型30s核糖体蛋白s1的一种或多种翻译后修饰。
[0523]
12.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的细菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1相对于未修饰的细菌细胞的30s核糖体蛋白s1是下调的。
[0524]
13.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1相对于野生型30s核糖体蛋白s1是截短的。
[0525]
14.实施方案11至13中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述修饰的30s核糖体蛋白s1由修饰的rpsa基因编码。
[0526]
15.实施方案5至10中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述修饰的rpsa基因是i)染色体的,或ii)染色体外的,例如,所述修饰的rpsa基因由质粒或粘粒编码。
[0527]
16.实施方案1至4和11至14中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型30s核糖体蛋白s1具有至少60%的序列同一性,例如与野生型30s核糖体蛋白s1至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0528]
17.实施方案1至4、11至14和16中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含选自以下的“r”结构域或由其组成:
[0529]
a.r1结构域或其片段、r2结构域或其片段、r3结构域或其片段和r4结构域或其片段;
[0530]
b.r1结构域或其片段、r2结构域或其片段和r3结构域或其片段;
[0531]
c.r2结构域或其片段、r3结构域或其片段和r4结构域或其片段;
[0532]
d.r2结构域或其片段和r3结构域或其片段;
[0533]
e.r3结构域或其片段和r4结构域或其片段;和
[0534]
f.r3结构域或其片段。
[0535]
18.实施方案1至4、11至14和16至17中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含具有以下的氨基酸序列或由其组成:
[0536]
a.r1结构域,当存在时,所述r1结构域与野生型s1具有至少60%的序列同一性,例如与野生型r1结构域至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性;
[0537]
b.r2结构域,当存在时,所述r2结构域与野生型s1具有至少60%的序列同一性,例如与野生型r2结构域至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性;
[0538]
c.r3结构域,其与野生型s 1具有至少60%的序列同一性,例如与野生型r3结构域至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性;和/
或
[0539]
d.r4结构域,当存在时,所述r4结构域与野生型s1具有至少40%的序列同一性,例如与野生型r4结构域至少50%、60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0540]
19.实施方案1至4、11至14和16至18中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含r1、r2、r3和/或r4结构域的第一个和/或最后一个氨基酸上游的氨基酸序列,当存在时,其与野生型30s核糖体蛋白s1的r1、r2、r3和/或r4结构域的第一个和/或最后一个残基上游的氨基酸序列具有至少60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0541]
20.实施方案1至4、11至14和16至19中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1使用以下进行修饰,或者通过以下获得或可获得:基因组编辑、基因沉默、片段化、rna转录后修饰和/或翻译后修饰。
[0542]
21.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述细菌细胞包含野生型rpsa基因。
[0543]
22.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述革兰氏阴性菌细胞包含野生型30s核糖体蛋白s1和修饰的30s核糖体蛋白s1蛋白质。
[0544]
23.实施方案1至20和22中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞并不包含野生型rpsa基因。
[0545]
24.实施方案1至21和23中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞并不包含野生型30s核糖体蛋白s1。
[0546]
25.实施方案3至24中任何一个的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞在液体培养物中生长时能够释放至少2.0倍的nomv,例如在液体培养物中生长时能够释放2.5倍、3.0倍、3.5倍、4.0倍、4.5倍、5.0倍、5.5倍、6.0倍、10倍、20倍、30倍、40倍、50倍、60倍、70倍、80倍、90倍或100倍的nomv。
[0547]
26.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其特征在于它能够增殖。
[0548]
27.实施方案26的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞的培养物能够生成的生物质是在相同培养条件下生长的未修饰细菌细胞的培养物的生物质的至少10%,例如在相同培养条件下生长的未修饰细菌细胞的培养物的生物质的20%、30%、40%、50%、60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%。
[0549]
28.实施方案26和27中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞的培养物能够实现的浊度是在相同培养条件下生长的未修饰细菌细胞的培养物的浊度的至少10%,例如在相同培养条件下生长的未修饰细菌细胞的培养物的浊度的20%、30%、40%、50%、60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%。
[0550]
29.实施方案28的遗传修饰的革兰氏阴性菌细胞,其中所述浊度作为在600nm处的
od进行测量。
[0551]
30.实施方案26的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞的培养物能够实现的营养素摄取是在相同培养条件下生长的未修饰细菌细胞的培养物的营养素摄取的至少10%,例如在相同培养条件下生长的未修饰细菌细胞的培养物的营养素摄取的20%、30%、40%、50%、60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%。
[0552]
31.实施方案26至30中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞能够在未修饰的细菌细胞在相同培养条件下生长后不迟于120小时,例如在未修饰的细菌细胞在相同培养条件下生长后不迟于1、2、3、4、5、6、7、8、9、10、12、15、20、25、30、40、50、60、70、80、90、100或110小时达到稳定期。
[0553]
32.实施方案26的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞的生长根据生物质、浊度和/或营养素摄取进行确定。
[0554]
33.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型rpsa基因的一个或多个突变存在于包含以下或由以下组成的指定的核苷酸序列中:
[0555]
a.相对于野生型rpsa基因的r3结构域的3
′
端上游的1至10个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端上游的11至20、21至50、或51至100个核苷酸;
[0556]
b.相对于野生型rpsa基因的r3结构域的3’端上游的1至100个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端上游的10至50、或20至30个核苷酸;
[0557]
c.相对于野生型rpsa基因1%至10%的核苷酸,例如相对于野生型rpsa基因11%至20%、21%至30%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的5
′
端和相对于野生型rpsa基因的r3结构域的3
′
端之间;
[0558]
d.相对于野生型rpsa基因1%至30%的核苷酸,例如相对于野生型rpsa基因5%至25%、10%至20%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的5
′
端和相对于野生型rpsa基因的r3结构域的3
′
端之间;
[0559]
e.相对于野生型rpsa基因的r3结构域的3’端下游的1至10个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端下游的11至20、21至50、或51至100个核苷酸;
[0560]
f.相对于野生型rpsa基因的r3结构域的3’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端下游的10至50、或20至30个核苷酸;
[0561]
g.相对于野生型rpsa基因的r4结构域的5’端下游的1至10个核苷酸,例如相对于野生型rpsa基因的r4结构域的5’端下游的11至20、21至50、或51至100个核苷酸;
[0562]
h.相对于野生型rpsa基因的r4结构域的5’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r4结构域的5’端下游的10至50、或20至30个核苷酸;
[0563]
i.相对于野生型rpsa基因的r4结构域的3’端下游的1至10个核苷酸,例如相对于野生型rpsa基因的r4结构域的3’端下游的11至20、21至50、或51至100个核苷酸;
[0564]
j.相对于野生型rpsa基因的r4结构域的3’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r4结构域的3’端下游的10至50、或20至30个核苷酸;
[0565]
k.相对于野生型rpsa基因1%至10%的核苷酸,例如相对于野生型rpsa基因11%至20%、21%至30%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的r3结构域的3
′
端和相对于野生型rpsa基因的3
′
端之间;
[0566]
1.相对于野生型rpsa基因1%至30%的核苷酸,例如相对于野生型rpsa基因5%至25%、10%至20%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的r3结构域的3
′
端和相对于野生型rpsa基因的3
′
端之间;
[0567]
m.相对于野生型rpsa基因的3
′
端上游的1至5个核苷酸,例如相对于野生型rpsa基因的3
′
端上游的6至10、11至20、21至30、31至40、41至50、51至100、101至150、151至200、201至250、251至300、301至350、351至400、401至450、451至500个核苷酸;和/或
[0568]
n.相对于野生型rpsa基因的3
′
端上游的1至500个核苷酸,例如相对于野生型rpsa基因的3
′
端上游的50至450、100至400、150至380、200至378、或250至376个核苷酸。
[0569]
34.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的ihfb基因和/或修饰的ihf蛋白。
[0570]
35.实施方案34的遗传修饰的革兰氏阴性菌细胞,其中:
[0571]
a.所述修饰的ihfb基因包含相对于野生型ihfb基因的一个或多个突变,其中所述一个或多个突变定位于ibfb基因的编码区内和/或ihfb基因的非编码区内;
[0572]
b.所述修饰的ihfb基因相对于野生型ihfb基因被敲除;
[0573]
c.所述修饰的ihf蛋白包含相对于野生型ihf蛋白的一个或多个突变;
[0574]
d.所述修饰的ihf蛋白包含相对于野生型ihf蛋白的一种或多种翻译后修饰;
[0575]
e.所述修饰的ihf蛋白相对于未修饰的细菌细胞的ihf蛋白是下调的;和/或
[0576]
f.所述修饰的ihf蛋白由修饰的ihfb基因编码。
[0577]
36.实施方案34和35中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述突变体ihf蛋白包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型ihf蛋白具有至少60%的序列同一性,例如与野生型ihf蛋白至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0578]
37.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述野生型rpsa基因选自以下:seq id no:1、seq id no:2、seq id no:3、seq id no:4、seq id no:5和seq id no:6。
[0579]
38.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述野生型s1选自以下:seq id no:13、seq id no:14、seq id no:15、seq id no:16、seq id no:17和seq id no:18。
[0580]
39.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述野生型ihf蛋白选自以下:seq id no:31、seq id no:32、seq id no:33、seq id no:34、seq id no:35和seq id no:36。
[0581]
40.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其选自肠杆菌科、奈瑟氏球菌科、螺杆菌科、弯曲菌科、耶尔森氏菌科、弧菌科、巴斯德氏菌科、产碱菌科、假单胞菌科和莫拉氏菌科。
[0582]
41.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其选自大肠杆菌、脑膜炎奈瑟氏球菌、乳糖奈瑟氏球菌、淋病奈瑟氏球菌、幽门螺杆菌、伤寒沙门氏菌、鼠伤寒沙门氏菌、霍乱弧菌、志贺氏菌属物种、流感嗜血杆菌、百日咳博德特氏菌、铜绿假单胞菌和粘膜炎莫拉氏菌。
[0583]
42.实施方案41的遗传修饰的革兰氏阴性菌细胞,其中所述革兰氏阴性菌细胞是
百日咳博德特氏菌。
[0584]
43.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa基因,其中所述修饰的rpsa基因包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与突变体seq id no:37具有至少60%的序列同一性,例如与seq id no:37至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0585]
44.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa操纵子,其中所述修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:38或seq id no:51具有至少60%的序列同一性,例如与seq id no:38或seq id no:51至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0586]
45.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与选自seq id no:39、seq id no:40、seq id no:41和seq id no:53的多肽具有至少60%的序列同一性,例如与选自seq id no:39、seq id no:40、seq id no:41和seq id no:53的多肽至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0587]
46.实施方案41的遗传修饰的革兰氏阴性菌细胞,其中所述细菌细胞是大肠杆菌。
[0588]
47.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa操纵子,其中所述修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:42或seq id no:43具有至少60%的序列同一性,例如与seq id no:42或seq id no:43至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0589]
48.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与seq id no:44或seq id no:45具有至少60%的序列同一性,例如与seq id no:44或seq id no:45至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0590]
49.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞表达一种或多种外源抗原。
[0591]
50.任何前述实施方案的遗传修饰的革兰氏阴性菌细胞,其已通过选自下组的一种或多种方法进行进一步遗传改造:(a)下调免疫显性可变抗原或非保护性抗原的表达的方法,(b)上调保护性omp抗原表达的方法,(c)下调涉及致使lps的脂质a部分有毒的基因的方法,(d)上调涉及致使lps的脂质a部分较少毒性的基因的方法,以及(e)对细菌细胞进行遗传修饰以表达异源抗原的方法。
[0592]
51.一种生成遗传修饰的革兰氏阴性菌细胞的方法,其包括修饰野生型rpsa基因、操纵子、rna和/或30s核糖体蛋白s1蛋白质的步骤,使得修饰产生这样的遗传修饰的革兰氏阴性菌细胞,当在培养基中生长时,与未修饰的细菌细胞相比,其将更多数量的nomv释放到培养基内。
[0593]
52.一种生成遗传修饰的革兰氏阴性菌细胞的方法,其包括提供修饰的30s核糖体蛋白s1的步骤,使得修饰产生这样的遗传修饰的革兰氏阴性菌细胞,当在培养基中生长时,与未修饰的细菌细胞相比,其将更多数量的nomv释放到培养基内。
[0594]
53.一种用于制备nomv的方法,其包括以下步骤:
[0595]
a.接种含有适合于实施方案1至50中任何一个的遗传修饰的革兰氏阴性菌细胞生长的营养培养基的培养容器;
[0596]
b.在允许nomv通过所述细菌释放到培养基内的条件下,培养遗传修饰的革兰氏阴性菌细胞;和
[0597]
c.从培养基中回收nomv;和
[0598]
d.将nomv与药学上可接受的稀释剂或载体混合。
[0599]
54.实施方案53的方法,其进一步包括步骤(c)之后的步骤,其包括无菌过滤nomv的制剂。
[0600]
55.一种nomv,其得自或可得自实施方案1至50中任何一个的遗传修饰的革兰氏阴性菌细胞、或者通过实施方案51和52的方法或通过实施方案53和54的方法获得或可获得的遗传修饰的革兰氏阴性菌细胞。
[0601]
56.一种免疫原性组合物,其包含实施方案55的nomv。
[0602]
57.实施方案55的免疫原性组合物,其进一步包含来自相同或不同病原体的一种或多种另外的抗原。
[0603]
58.一种疫苗,其包含实施方案55的nomv。
[0604]
59.实施方案55的nomv、实施方案56和57中任何一个的免疫原性组合物或实施方案58的疫苗,其用于药物中。
[0605]
60.实施方案55的nomv、实施方案56和57中任何一个的免疫原性组合物或实施方案58的疫苗,其用于在脊椎动物中,优选在哺乳动物中诱导免疫应答。
[0606]
61.实施方案55的nomv、实施方案56和57中任何一个的免疫原性组合物或实施方案58的疫苗,其用于治疗或预防通过nomv由其获得或可获得的遗传修饰的革兰氏阴性菌细胞同一属或种的细菌细胞引起的疾病。
[0607]
62.实施方案55的nomv、实施方案56和57中任何一个的免疫原性组合物或实施方案58的疫苗,其用于治疗或预防由博德特氏菌属,优选百日咳博德特氏菌引起的疾病。
[0608]
63.实施方案55的nomv、实施方案56和57中任何一个的免疫原性组合物或实施方案58的疫苗,其用于治疗或预防由大肠杆菌引起的疾病。
[0609]
64.一种针对细菌细胞免疫有此需要的受试者的方法,其通过施用实施方案55的nomv、实施方案56和57中任何一个的免疫原性组合物或实施方案58的疫苗。
[0610]
65.实施方案55的nomv、实施方案56和57中任何一个的免疫原性组合物或实施方案58的疫苗在制造用于针对细菌细胞免疫有此需要的受试者的药剂中的用途。
[0611]
进一步的实施方案
[0612]
1.一种遗传修饰的革兰氏阴性菌细胞,其包含修饰的rpsa基因、修饰的rpsa操纵子和/或修饰的30s核糖体蛋白s1蛋白质。
[0613]
2.实施方案1的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的细菌细胞能够分泌天然外膜囊泡(nomv)。
[0614]
3.实施方案2的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞能够分泌更多数量的nomv。
[0615]
4.实施方案3的遗传修饰的革兰氏阴性菌细胞,其中与野生型细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞能够分泌更多数量的nomv。
[0616]
5.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa基因,其中所述修饰的rpsa基因包含相对于野生型rpsa基因的一个或多个突变,其中所述一个或多个突变定位于rpsa基因的编码区内和/或rpsa基因的非编码区内。
[0617]
6.实施方案5的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含一个或多个核苷酸的插入或由一个或多个核苷酸的插入组成,其中所插入的一个或多个核苷酸选自以下:
[0618]
a.转座子移动元件;
[0619]
b.选择标记物,例如抗生素抗性盒;编码荧光蛋白的基因;或编码抗毒素的基因;和/或
[0620]
c.rpsa基因的片段。
[0621]
7.实施方案5或6的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变使用选自以下的方法进行制备,或者通过选自以下的方法获得或可获得:定点诱变、重组酶介导的方法、λ-red重组酶介导的方法、基于原噬菌体的方法、移动ii组内含子敲除、转座子介导的基因组编辑和crispr-cas基因组编辑。
[0622]
8.实施方案6(a)或7的遗传修饰的革兰氏阴性菌细胞,其中所述转座子移动元件的插入使用转座子介导的基因组编辑进行制备,或者通过转座子介导的基因组编辑获得或可获得。
[0623]
9.实施方案6(a)至8中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述转座子移动元件选自tn5转座子、tn10转座子、tn3转座子、tn7转座子、is5376转座子、细菌噬菌体mua转座子、is200/is605转座子和is91转座子。
[0624]
10.实施方案9的遗传修饰的革兰氏阴性菌细胞,其中所述转座子移动元件是tn5转座子。
[0625]
11.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1:
[0626]
a.包含相对于野生型30s核糖体蛋白s1的一个或多个突变;和/或
[0627]
b.包含相对于野生型30s核糖体蛋白s1的一种或多种翻译后修饰。
[0628]
12.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1相对于未修饰的细菌细胞的30s核糖体蛋白s1是下调的。
[0629]
13.实施方案11或12的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型30s核糖体蛋白s1的一个或多个突变包含在对应于百日咳博德特氏菌30s核糖体蛋白s1的r4结构域的区域和/或对应于百日咳博德特氏菌30s核糖体蛋白s1在r3和r4结构域之间的部分的区域中的一个或多个突变。
[0630]
14.实施方案11至13中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野
生型30s核糖体蛋白s1的一个或多个突变包含至少5、至少10、至少25、至少50、至少75、至少100、至少110、至少125、5至150、25至130、100至130、或125至130个氨基酸的突变或缺失。
[0631]
15.实施方案11至14中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型30s核糖体蛋白s1的一个或多个突变包含对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸550至576的区域的至少5、至少10、至少15、至少20、或至少25个氨基酸的突变或缺失。
[0632]
16.实施方案11至15中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型30s核糖体蛋白s1的一个或多个突变包含对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸473至576的区域的至少20、至少30、至少50、至少75、或至少100个氨基酸的突变或缺失。
[0633]
17.实施方案11至16中任何一个的遗传修饰的革兰氏阴性菌细胞,相对于野生型30s核糖体蛋白s1的一个或多个突变包含对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸450至576的区域的至少20、至少30、至少50、至少100、或至少120个氨基酸的突变或缺失。
[0634]
18.实施方案11至17中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型30s核糖体蛋白s1的一个或多个突变包含来自30s核糖体蛋白s1的c末端端部的至少20、至少30、至少50、至少100、或至少120个连续氨基酸的截短。
[0635]
19.实施方案11至18中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型30s核糖体蛋白s1的一个或多个突变包含对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸560至576、552至576、500至576485至576、460至576、或453至576的氨基酸的突变或缺失。
[0636]
20.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1相对于野生型30s核糖体蛋白s1是截短的。
[0637]
21.实施方案11至20中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述修饰的30s核糖体蛋白s1由修饰的rpsa基因编码。
[0638]
22.实施方案1至21中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述修饰的rpsa基因是i)染色体的,或ii)染色体外的,例如,所述修饰的rpsa基因由质粒或粘粒编码。
[0639]
23.实施方案1至22中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型30s核糖体蛋白s1具有至少60%的序列同一性,例如与野生型30s核糖体蛋白s1至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0640]
24.实施方案1至23中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型30s核糖体蛋白s1的片段具有至少90%、至少95%、至少98%、至少99%或100%的同一性,所述片段对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸1至452。
[0641]
25.实施方案1至24中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修
饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型30s核糖体蛋白s1的片段具有至少98%、至少99%或100%的同一性,所述片段对应于百日咳博德特氏菌30s核糖体蛋白s1的氨基酸1至453。
[0642]
26.实施方案1至25中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含选自以下的“r”结构域或由其组成:
[0643]
a.r1结构域或其片段、r2结构域或其片段、r3结构域或其片段和r4结构域或其片段;
[0644]
b.r1结构域或其片段、r2结构域或其片段和r3结构域或其片段;
[0645]
c.r2结构域或其片段、r3结构域或其片段和r4结构域或其片段;
[0646]
d.r2结构域或其片段和r3结构域或其片段;
[0647]
e.r3结构域或其片段和r4结构域或其片段;和
[0648]
f.r3结构域或其片段。
[0649]
27.实施方案1至26中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含具有以下的氨基酸序列或由其组成:
[0650]
a.r1结构域,当存在时,所述r1结构域与野生型s1具有至少60%的序列同一性,例如与野生型r1结构域至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性;
[0651]
b.r2结构域,当存在时,所述r2结构域与野生型s1具有至少60%的序列同一性,例如与野生型r2结构域至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性;
[0652]
c.r3结构域,其与野生型s1具有至少60%的序列同一性,例如与野生型r3结构域至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性;和/或
[0653]
d.r4结构域,当存在时,所述r4结构域与野生型s1具有至少40%的序列同一性,例如与野生型r4结构域至少50%、60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0654]
28.实施方案1至27中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s 1包含具有以下的氨基酸序列或由其组成:
[0655]
a.r1结构域,当存在时,所述r1结构域与野生型s1具有至少60%的序列同一性,例如与野生型r1结构域至少98%、99%或100%的序列同一性;
[0656]
b.r2结构域,当存在时,所述r2结构域与野生型s1具有至少60%的序列同一性,例如与野生型r2结构域至少98%、99%或100%的序列同一性;
[0657]
c.r3结构域,其与野生型s1具有至少60%的序列同一性,例如与野生型r3结构域至少98%、99%或100%的序列同一性;和/或
[0658]
d.r4结构域,当存在时,所述r4结构域与野生型s1具有至少40%的序列同一性,例
如与野生型r4结构域至少98%、99%或100%的序列同一性。
[0659]
29.实施方案1至28中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含r1、r2、r3和/或r4结构域的第一个和/或最后一个氨基酸上游的氨基酸序列,当存在时,其与野生型30s核糖体蛋白s 1的r1、r2、r3和/或r4结构域的第一个和/或最后一个残基上游的氨基酸序列具有至少60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0660]
30.实施方案1至29中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含r1、r2、r3和/或r4结构域的第一个和/或最后一个氨基酸上游的氨基酸序列,当存在时,其与野生型30s核糖体蛋白s1的r1、r2、r3和/或r4结构域的第一个和/或最后一个残基上游的氨基酸序列具有至少95%、98%、99%或100%的序列同一性。
[0661]
31.实施方案1至30中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1使用以下进行修饰,或者通过以下获得或可获得:基因组编辑、基因沉默、片段化、rna转录后修饰和/或翻译后修饰。
[0662]
32.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述革兰氏阴性菌细胞包含野生型rpsa基因。
[0663]
33.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述革兰氏阴性菌细胞包含野生型30s核糖体蛋白s1和修饰的30s核糖体蛋白s1蛋白质。
[0664]
34.实施方案1至31中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞并不包含野生型rpsa基因。
[0665]
35.实施方案1至31和34中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞并不包含野生型30s核糖体蛋白s1。
[0666]
36.实施方案1至35中任何一个的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞在液体培养物中生长时能够释放至少2.0倍的nomv,例如在液体培养物中生长时能够释放至少2.5倍、3.0倍、3.5倍、4.0倍、4.5倍、5.0倍、5.5倍、6.0倍、10倍、20倍、30倍、40倍、50倍、60倍、70倍、80倍、90倍或100倍的nomv。
[0667]
37.实施方案1至36中任何一个的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞在液体培养物中生长时能够释放至少2.0倍的nomv,例如在液体培养物中生长时能够释放至少2.5倍、3.0倍、3.5倍、4.0倍、4.5倍、5.0倍、5.5倍、6.0倍、10倍、20倍、30倍、40倍、50倍、60倍、70倍、80倍、90倍或100倍的nomv。
[0668]
38.实施方案1至37中任何一个的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞在液体培养物中生长时能够释放至少1.2倍、至少1.4倍、至少1.6倍、至少1.8倍或至少2.0倍的nomv。
[0669]
39.实施方案1至38中任何一个的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的细菌细胞相比,所述遗传修饰的革兰氏阴性菌细胞在液体培养物中生长时能够释放至少
1.2倍、至少1.4倍、至少1.6倍、至少1.8倍或至少2.0倍的nomv。
[0670]
40.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其特征在于它能够增殖。
[0671]
41.实施方案40的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞的培养物能够生成的生物质是在相同培养条件下生长的未修饰细菌细胞的培养物的生物质的至少10%,例如在相同培养条件下生长的未修饰细菌细胞的培养物的生物质的至少20%、30%、40%、50%、60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%。
[0672]
42.实施方案40至41中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞的培养物能够实现的浊度是在相同培养条件下生长的未修饰细菌细胞的培养物的浊度的至少10%,例如在相同培养条件下生长的未修饰细菌细胞的培养物的浊度的20%、30%、40%、50%、60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%。
[0673]
43.实施方案42的遗传修饰的革兰氏阴性菌细胞,其中所述浊度作为在600nm处的od进行测量。
[0674]
44.实施方案40的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞的培养物能够实现的营养素摄取是在相同培养条件下生长的未修饰细菌细胞的培养物的营养素摄取的至少10%,例如在相同培养条件下生长的未修饰细菌细胞的培养物的营养素摄取的20%、30%、40%、50%、60%、70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%。
[0675]
45.实施方案40至44中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞能够在未修饰的细菌细胞在相同培养条件下生长后不迟于120小时,例如在未修饰的细菌细胞在相同培养条件下生长后不迟于1、2、3、4、5、6、7、8、9、10、12、15、20、25、30、40、50、60、70、80、90、100或110小时达到稳定期。
[0676]
46.实施方案40至45中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞的生长根据生物质、浊度和/或营养素摄取进行确定。
[0677]
47.实施方案5至46中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型rpsa基因的一个或多个突变存在于包含以下或由以下组成的指定的核苷酸序列中:
[0678]
a.相对于野生型rpsa基因的r3结构域的3
′
端上游的1至10个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端上游的11至20、21至50或51至100个核苷酸;
[0679]
b.相对于野生型rpsa基因的r3结构域的3’端上游的1至100个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端上游的10至50、或20至30个核苷酸;
[0680]
c.相对于野生型rpsa基因1%至10%的核苷酸,例如相对于野生型rpsa基因11%至20%、21%至30%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的5
′
端和相对于野生型rpsa基因的r3结构域的3
′
端之间;
[0681]
d.相对于野生型rpsa基因1%至30%的核苷酸,例如相对于野生型rpsa基因5%至25%、10%至20%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的5
′
端和相对于野生型rpsa基因的r3结构域的3
′
端之间;
[0682]
e.相对于野生型rpsa基因的r3结构域的3’端下游的1至10个核苷酸,例如相对于
野生型rpsa基因的r3结构域的3’端下游的11至20、21至50、或51至100个核苷酸;
[0683]
f.相对于野生型rpsa基因的r3结构域的3’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r3结构域的3’端下游的10至50、或20至30个核苷酸;
[0684]
g.相对于野生型rpsa基因的r4结构域的5’端下游的1至10个核苷酸,例如相对于野生型rpsa基因的r4结构域的5’端下游的11至20、21至50、或51至100个核苷酸;
[0685]
h.相对于野生型rpsa基因的r4结构域的5’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r4结构域的5’端下游的10至50、或20至30个核苷酸;
[0686]
i.相对于野生型rpsa基因的r4结构域的3’端下游的1至10个核苷酸,例如相对于野生型rpsa基因的r4结构域的3’端下游的11至20、21至50、或51至100个核苷酸;
[0687]
j.相对于野生型rpsa基因的r4结构域的3’端下游的1至100个核苷酸,例如相对于野生型rpsa基因的r4结构域的3’端下游的10至50、或20至30个核苷酸;
[0688]
k.相对于野生型rpsa基因1%至10%的核苷酸,例如相对于野生型rpsa基因11%至20%、21%至30%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的r3结构域的3
′
端和相对于野生型rpsa基因的3
′
端之间;
[0689]
l.相对于野生型rpsa基因1%至30%的核苷酸,例如相对于野生型rpsa基因5%至25%、10%至20%的核苷酸,其中所述指定的区域定位于相对于野生型rpsa基因的r3结构域的3
′
端和相对于野生型rpsa基因的3
′
端之间;
[0690]
m.相对于野生型rpsa基因的3
′
端上游的1至5个核苷酸,例如相对于野生型rpsa基因的3
′
端上游的6至10、11至20、21至30、31至40、41至50、51至100、101至150、151至200、201至250、251至300、301至350、351至400、401至450、451至500个核苷酸;和/或
[0691]
n.相对于野生型rpsa基因的3
′
端上游的1至500个核苷酸,例如相对于野生型rpsa基因的3
′
端上游的50至450、100至400、150至380、200至378、或250至376个核苷酸。
[0692]
48.实施方案5至47中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型rpsa基因的一个或多个突变包含在对应于编码百日咳博德特氏菌30s核糖体蛋白s1蛋白质的r4结构域的区域和/或编码百日咳博德特氏菌30s核糖体s1蛋白在r3和r4结构域之间的部分的区域的区域中的一个或多个突变。
[0693]
49.实施方案5至48中任何一个的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型rpsa基因的一个或多个突变包含引起与未修饰的革兰氏阴性菌细胞相比的nomv释放增加的一个或多个突变。
[0694]
50.实施方案49的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的革兰氏阴性菌细胞相比,所述一个或多个突变引起至少1.2倍、1.4倍、1.6倍、1.8倍、2.0倍或2.5倍的nomv释放增加。
[0695]
51.实施方案50的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的革兰氏阴性菌细胞相比,所述一个或多个突变引起至少2.0倍的nomv释放增加。
[0696]
52.实施方案5至51中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含改变所编码的30s核糖体蛋白s1蛋白质的氨基酸序列的突变。
[0697]
53.实施方案52的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含改变30s核糖体蛋白s1蛋白质的区域的编码氨基酸序列的突变,所述区域对应于百日咳博德特氏菌的r4结构域。
[0698]
54.实施方案47至53中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含改变所编码的30s核糖体蛋白s1蛋白质的突变,以修饰和/或缺失至少5、至少10、至少25、至少50、至少75、至少100、至少110、至少125、5至150、25至130、100至130、或125至130个氨基酸。
[0699]
55.实施方案47至54中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含改变所编码的30s核糖体蛋白s1蛋白质的突变,以缺失至少5、至少10、至少25、至少50、至少75、至少100、至少110、至少125、5至150、25至130、100至130、或125至130个氨基酸。
[0700]
56.实施方案47至55中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述修饰的rpsa基因编码根据实施方案13至36中任何一个的修饰的30s核糖体蛋白s1蛋白质。
[0701]
57.实施方案47至56中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含对应于百日咳博德特氏菌rpsa基因的核苷酸1655至1731的区域的至少15、至少30、至少45、至少60或至少75个核苷酸的突变或缺失。
[0702]
58.实施方案47至57中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含对应于百日咳博德特氏菌rpsa基因的核苷酸1424至1731的区域的至少60、至少90、至少150、至少225或至少300个核苷酸的突变或缺失。
[0703]
59.实施方案47至58中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述一个或多个突变包含对应于百日咳博德特氏菌rpsa基因的核苷酸1355至1731的区域的至少60、至少90、至少150、至少225、至少300或至少360个核苷酸的突变或缺失。
[0704]
60.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的ihfb基因和/或修饰的ihf蛋白。
[0705]
61.一种遗传修饰的革兰氏阴性菌细胞,其包含修饰的ihfb基因和/或修饰的ihf蛋白。
[0706]
62.实施方案60或61的遗传修饰的革兰氏阴性菌细胞,其中:
[0707]
a.所述修饰的ihfb基因包含相对于野生型ihfb基因的一个或多个突变,其中所述一个或多个突变定位于ihfb基因的编码区内和/或ihfb基因的非编码区内;
[0708]
b.所述修饰的ihfb基因相对于野生型ihfb基因被敲除;
[0709]
c.所述修饰的ihf蛋白包含相对于野生型ihf蛋白的一个或多个突变;
[0710]
d.所述修饰的ihf蛋白包含相对于野生型ihf蛋白的一种或多种翻译后修饰;
[0711]
e.所述修饰的ihf蛋白相对于未修饰的细菌细胞的ihf蛋白是下调的;和/或
[0712]
f.所述修饰的ihf蛋白由修饰的ihfb基因编码。
[0713]
63.实施方案62的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型ihf蛋白的一个或多个突变包含至少20、至少30、至少40、至少50、至少60、至少70、至少80、至少90、至少100、至少110、50至119、或80至119个氨基酸的缺失。
[0714]
64.实施方案63的遗传修饰的革兰氏阴性菌细胞,其中相对于野生型ihf蛋白的一个或多个突变包含至少20、至少30、至少40、至少50、至少60、至少70、至少80、至少100、至少110、50至119、或80至119个邻接氨基酸的缺失。
[0715]
65.实施方案62至64中任何一个的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的革兰氏阴性菌细胞相比,相对于野生型ihf蛋白的一个或多个突变引起nomv释放的增加。
[0716]
66.实施方案62至65中任何一个的遗传修饰的革兰氏阴性菌细胞,其中与未修饰的革兰氏阴性菌细胞相比,相对于野生型ihf蛋白的一个或多个突变引起至少1.2倍、至少1.4倍、至少1.6倍、至少1.8倍、至少2.0倍或至少2.5倍的nomv释放增加。
[0717]
67.实施方案62至66中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述修饰的ihfb基因编码根据实施方案62至66中任何一个的修饰的ihf蛋白。
[0718]
68.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述革兰氏阴性菌细胞是天然共转录ihfb基因和rpsa基因的细菌物种。
[0719]
69.实施方案62至68中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述修饰的ihf蛋白包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与野生型ihf蛋白具有至少60%的序列同一性,例如与野生型ihf蛋白至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0720]
70.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述野生型rpsa基因选自以下:seq id no:1、seq id no:2、seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:55和seq id no:60。
[0721]
71.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述野生型s1选自以下:seq id no:13、seq id no:14、seq id no:15、seq id no:16、seq id no:17、seq id no:18和seq id no:57。
[0722]
72.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述野生型ihf蛋白选自以下:seq id no:31、seq id no:32、seq id no:33、seq id no:34、seq id no:35、seq id no:36和seq id no:62。
[0723]
73.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其选自肠杆菌科、奈瑟氏球菌科、螺杆菌科、弯曲菌科、耶尔森氏菌科、弧菌科、巴斯德氏菌科、产碱菌科、假单胞菌科和莫拉氏菌科。
[0724]
74.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其选自大肠杆菌、脑膜炎奈瑟氏球菌、乳糖奈瑟氏球菌、淋病奈瑟氏球菌、幽门螺杆菌、伤寒沙门氏菌、鼠伤寒沙门氏菌、霍乱弧菌、志贺氏菌属物种、流感嗜血杆菌、百日咳博德特氏菌、铜绿假单胞菌和粘膜炎莫拉氏菌。
[0725]
75.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其选自大肠杆菌、百日咳博德特氏菌和粘膜炎莫拉氏菌。
[0726]
76.实施方案74或75的遗传修饰的革兰氏阴性菌细胞,其中所述革兰氏阴性菌细胞是百日咳博德特氏菌。
[0727]
77.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa基因,其中所述修饰的rpsa基因包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与突变体seq id no:37或seq id no:56具有至少60%的序列同一性,例如与seq id no:37或seq id no:56至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0728]
78.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa基因,其中所述修饰的rpsa基因包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与突变体seq id no:37或seq id no:56具有至少95%
的序列同一性。
[0729]
79.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa操纵子,其中所述修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:38、seq id no:51或seq id no:59具有至少60%的序列同一性,例如与seq id no:38、seq id no:51或seq id no:59至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0730]
80.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa操纵子,其中所述修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:38、seq id no:51或seq id no:59具有至少95%的序列同一性。
[0731]
81.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与选自seq id no:39、seq id no:40、seq id no:41和seq id no:53的多肽具有至少60%的序列同一性,例如与选自seq id no:39、seq id no:40、seq id no:41和seq id no:53的多肽至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0732]
82.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与选自seq id no:39、seq id no:40、seq id no:41和seq id no:53的多肽具有至少98%的序列同一性。
[0733]
83.实施方案81或82的遗传修饰的革兰氏阴性菌细胞,其中所述细菌细胞是大肠杆菌或百日咳博德特氏菌。
[0734]
84.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa操纵子,其中所述修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:42或seq id no:43具有至少60%的序列同一性,例如与seq id no:42或seq id no:43至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0735]
85.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的rpsa操纵子,其中所述修饰的rpsa操纵子包含核苷酸序列或由核苷酸序列组成,所述核苷酸序列与seq id no:42或seq id no:43具有至少98%的序列同一性。
[0736]
86.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与seq id no:44或seq id no:45具有至少60%的序列同一性,例如与seq id no:44或seq id no:45至少70%、80%、85%、90%、92%、95%、96%、97%、98%、99%或100%的序列同一性。
[0737]
87.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞包含修饰的30s核糖体蛋白s1,其中所述修饰的30s核糖体蛋白s1包含氨基酸序列或由氨基酸序列组成,所述氨基酸序列与seq id no:44或seq id no:45具有
至少98%的序列同一性。
[0738]
88.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其中所述遗传修饰的革兰氏阴性菌细胞表达一种或多种外源抗原。
[0739]
89.前述实施方案中任何一个的遗传修饰的革兰氏阴性菌细胞,其已通过选自下组的一种或多种方法进行进一步遗传改造:(a)下调免疫显性可变抗原或非保护性抗原的表达的方法,(b)上调保护性omp抗原表达的方法,(c)下调涉及致使lps的脂质a部分有毒的基因的方法,(d)上调涉及致使lps的脂质a部分较少毒性的基因的方法,以及(e)对细菌细胞进行遗传修饰以表达异源抗原的方法。
[0740]
90.一种生成遗传修饰的革兰氏阴性菌细胞的方法,其包括修饰野生型rpsa基因、操纵子、rna和/或30s核糖体蛋白s1蛋白质的步骤,使得修饰产生这样的遗传修饰的革兰氏阴性菌细胞,当在培养基中生长时,与未修饰的细菌细胞相比,其将更多数量的nomv释放到培养基内。
[0741]
91.一种生成遗传修饰的革兰氏阴性菌细胞的方法,其包括提供修饰的30s核糖体蛋白s1的步骤,使得修饰产生这样的遗传修饰的革兰氏阴性菌细胞,当在培养基中生长时,与未修饰的细菌细胞相比,其将更多数量的nomv释放到培养基内。
[0742]
92.实施方案90或91的方法,其中所生成的遗传修饰的革兰氏阴性细胞是根据实施方案1至98中任何一个的革兰氏阴性菌细胞。
[0743]
93.一种用于制备nomv的方法,其包括以下步骤:
[0744]
a.接种含有适合于实施方案1至89中任何一个的遗传修饰的革兰氏阴性菌细胞生长的营养培养基的培养容器;
[0745]
b.在允许nomv通过所述细菌释放到培养基内的条件下,培养遗传修饰的革兰氏阴性菌细胞;和
[0746]
c.从培养基中回收nomv;和
[0747]
d.将nomv与药学上可接受的稀释剂或载体混合。
[0748]
94.一种用于制备nomv的方法,其包括以下步骤:
[0749]
a.接种含有适合于遗传修饰的革兰氏阴性菌细胞生长的营养培养基的培养容器;
[0750]
b.在允许nomv通过所述细菌释放到培养基内的条件下,培养遗传修饰的革兰氏阴性菌细胞,其中所述条件包括添加根据实施方案11至31中任何一个的修饰的30s核糖体蛋白s1;
[0751]
c.从培养基中回收nomv;和
[0752]
d.将nomv与药学上可接受的稀释剂或载体混合。
[0753]
95.实施方案94的方法,其中所述革兰氏阴性菌细胞是实施方案1至89中任何一个的革兰氏阴性菌细胞。
[0754]
96.实施方案93至95中任何一个的方法,其进一步包括步骤(c)之后的步骤,其包括无菌过滤nomv的制剂。
[0755]
97.一种nomv,其得自或可得自实施方案1至89中任何一个的遗传修饰的革兰氏阴性菌细胞、或者通过实施方案90至92中任何一个的方法或通过实施方案93至96中任何一个的方法获得或可获得的遗传修饰的革兰氏阴性菌细胞。
[0756]
98.一种免疫原性组合物,其包含实施方案97的nomv。
[0757]
99.实施方案98的免疫原性组合物,其进一步包含来自相同或不同病原体的一种或多种另外的抗原。
[0758]
100.一种疫苗,其包含实施方案97的nomv。
[0759]
101.实施方案97的nomv、实施方案98和99中任何一个的免疫原性组合物或实施方案100的疫苗,其用于药物中。
[0760]
102.实施方案97的nomv、实施方案98和99中任何一个的免疫原性组合物或实施方案100的疫苗,其用于在脊椎动物中,优选在哺乳动物中诱导免疫应答。
[0761]
103.实施方案97的nomv、实施方案98和99中任何一个的免疫原性组合物或实施方案100的疫苗,其用于治疗或预防由nomv从其获得或可获得的遗传修饰的革兰氏阴性菌细胞同一属或种的细菌细胞引起的疾病。
[0762]
104.实施方案97的nomv、实施方案98和99中任何一个的免疫原性组合物或实施方案100的疫苗,其用于治疗或预防由博德特氏菌属,优选百日咳博德特氏菌引起的疾病。
[0763]
105.实施方案97的nomv、实施方案98和99中任何一个的免疫原性组合物或实施方案100的疫苗,其用于治疗或预防由大肠杆菌引起的疾病。
[0764]
106.实施方案97的nomv、实施方案98和99中任何一个的免疫原性组合物或实施方案100的疫苗,其用于治疗或预防由粘膜炎莫拉氏菌引起的疾病。
[0765]
107.一种针对细菌细胞免疫有此需要的受试者的方法,其通过施用实施方案97的nomv、实施方案98和99中任何一个的免疫原性组合物或实施方案100的疫苗。
[0766]
108.实施方案97的nomv、实施方案98和99中任何一个的免疫原性组合物或实施方案100的疫苗在制造用于针对细菌细胞免疫有此需要的受试者的药剂中的用途。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。