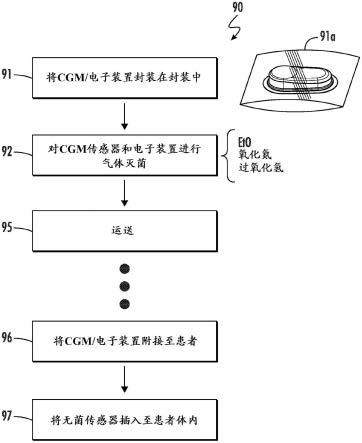
1.本发明涉及可用于预防或治疗冠状病毒科病毒感染的多肽。
背景技术:
::2.初步研究表明sars-cov-2与sars-cov相似,为此,以前存在关于保护性免疫应答的研究数据。有关sars-cov的各种报道提示体液和细胞介导二者的免疫应答的保护作用。针对sars-cov的刺突(s)和核壳(n)蛋白产生的抗体应答在sars-cov感染的患者中特别普遍。尽管有效,但发现抗体应答在恢复期sars-cov患者中是短暂的。相反,t细胞应答已证实在恢复的患者中提供感染后的长期记忆。3.生产有效疫苗的挑战之一是,在不同人类受试者的免疫系统与由感染性病毒表达的不同抗原的相互作用的方式中存在巨大的可变性。本发明人先前已经表明,个体受试者的免疫应答通过单个抗原t细胞表位被受试者的多个hla等位基因识别的能力来预测。被受试者的多个hla等位基因所限制的t细胞表位充当预测个体患者的肽特异性t细胞应答的遗传生物标志物。这些遗传生物标记物被称为“个人表位”或“pepi”。多hla等位基因结合性pepi以比被接种对象的单一hla等位基因所限制的t细胞表位显著更高的速率诱导t细胞应答。在疫苗组合物的多肽中t细胞抗原决定部位的鉴定,其为在人类模型群体中的个体的多hla等位基因结合性pepi,已显示预测在临床试验中报导的免疫反应率(wo2018/158456、wo2018/158457和wo2018/158455)。4.开发有效疫苗的第二个挑战是病毒通过突变的持续进化和感染病毒异质性的潜力。5.第三个挑战是需要快速开发、安全性试验和验证新出现的sars-cov-2冠状病毒的疫苗的效力,并随后大规模生产疫苗,以满足即时的群体需求。常规疫苗开发是复杂且具有挑战性的过程。与由死亡或减毒病原体、灭活毒素、和重组亚单位制成的常规疫苗相比,肽疫苗提供了若干优点。短多肽可以快速合成,并且肽疫苗生产相对便宜。另外,肽疫苗避免了包含对宿主具有高反应原性的不必要组分,例如脂多糖、脂质、和毒素。含有montanide佐剂的肽疫苗的安全性和免疫原性已经在涉及超过6,000名患者和超过200名健康志愿者的多个临床试验中得到证实。6.肽疫苗开发策略通常以选择寻求最大化覆盖全球群体的hla等位基因限制性表位的组合为目标。根据这种方法,选择具有不同hla结合特异性的多种肽,以提供由基于肽(表位)的疫苗靶向的患者群体的增加的覆盖,还考虑到不同的hla类型在不同的种族中以显著不同的频率表达。例如,sfahmed等人提出了一组筛选的t细胞表位,估计其提供了全球以及中国群体对sars-cov-2的广泛覆盖(ahmedetal.viruses,12(3).2020)。他们使用hla限制性sars-cov衍生表位和公众可获得的iedbpopulationcoveragetool(http://tools.iedb.org/population)指导针对sars-cov-2的疫苗的实验工作。7.这种方法试图考虑不同种族群体中hla多态性和频率。然而,实际上,最常见的hla限制性表位不诱导hla匹配个体中的免疫应答,并且临床试验导致比预期更低的免疫应答率(slingluffcl.cancerj2011;17(5):343–50)。此外,cd8t细胞识别的肽已证实既是选择性的且是极其敏感的;一个氨基酸变化可以将特定表位改变为非免疫原性肽。8.其它方法包含mrna或pdna载体中s蛋白的全序列。(smithtrfetal.underreview10.21203/rs.3.rs-16261/v1;safetyandimmunogenicitystudyof2019-ncovvaccine(mrna-1273)forprophylaxissarscov-2infection,nct04283461)。9.因此,迫切需要一种疫苗,这种疫苗对全球人口的高比例有效,对病毒抗原突变具有抵抗力,并能迅速通过临床验证和生产的必要步骤。技术实现要素:10.发明人集中精力开发针对sars-cov-2的全球性多肽疫苗,以解决不同个体的免疫反应的异质性和感染病毒的潜在异质性的双重挑战。本文详述的肽设计概念将共有的个人表位设计与b细胞表位序列的进一步选择合并,导致在个体内的重叠的多hla结合表位,旨在诱导cd4 、cd8 和产生抗体的b细胞应答。因此,发明人已经鉴定了目前已知的sars-cov-2病毒抗原的保守区的30聚体多肽片段,其包括(i)在具有代表全球群体的hla基因型的人类受试者的模型群体中的最大cd8 个人表位(pepi);(ii)全球群体中的最大cd4 个人表位(pepi);和(iii)线性b细胞表位。包括由发明人鉴定的多肽的肽疫苗,预计在人类群体中的惊人高比例的受试者中诱导细胞毒性t细胞、辅助t细胞和b细胞应答。通过组合多于一个的由发明人鉴定的抗原片段,优选地通过组合由发明人从不同的sars-cov-2结构蛋白鉴定的抗原片段,可以在人类群体中实现甚至更高的应答率,和针对进化的异源感染病毒的持续效力。11.因此,在第一方面,本公开提供了一种多肽、或一种长度至多50个氨基酸,或长度至多60个氨基酸的多肽组,其中所述多肽包括选自seqidno:1至17的氨基酸序列或由其组成。12.在另一个方面,本发明提供了一种长度多至50个氨基酸、或长度至多60个氨基酸的多肽组,其中每个多肽包括选自seqidno:1至17的不同氨基酸序列或由其组成。13.多肽的一或多个可以由冠状病毒科、β-冠状病毒科或sars-cov-2蛋白的片段组成。多肽中的每一个可包括选自seqidno:1至17中的序列的核酸序列,其是不同的冠状病毒科、β-冠状病毒科或sars-cov-2蛋白的片段。多肽组可以包含来自以下组中的至少二组、三组或全部四组的至少一个序列:(a)seqidno:1至11(sars-cov-2表面蛋白的片段);(b)seqidno:12至15(sars-cov-2核壳蛋白的片段);(c)seqidno:16(sars-cov-2膜蛋白的片段);和(d)seqidno:17(sars-cov-2包膜蛋白的片段)。在一些情况下,肽组可包括至少3、4、5、6、7、8、9、10、11、12、13、14、15、16或17个不同的多肽,其每个包括选自seqidno:1至17的不同的氨基酸序列。在一个实施方式中,肽组包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列。在一个实施方式中,多肽组包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列。在一个实施方式中,多肽组包括seqidno:6、和9至17的氨基酸序列。在一个实施方式中,多肽组包括十个多肽,其中十个多肽中的每一个包括选自seqidno:2、5、7、9、12、13、14、15、16、和17的不同的氨基酸序列或由其组成。在一个实施方式中,多肽组包括九个多肽,其中九个多肽中的每一个包括选自seqidno:2、5、9、12、13、14、15、16、和17的不同的氨基酸序列或由其组成。在一个实施方式中,多肽组包括十个多肽,其中十个多肽中的每一个包括选自seqidno:6、和9至17的不同的氨基酸序列或由其组成。14.在另一方面,本公开提供了具有上述多肽或多肽组作为活性成分的药物组合物或试剂盒。在一些情况下,药物组合物或试剂盒可包括编码每个多肽的核糖核酸。15.在另一方面,本公开提供了在受试者中接种疫苗、提供免疫疗法或诱导免疫应答的方法,所述方法包括向受试者施用多肽、多肽组或药物组合物。在一些情况下,所述方法是治疗受试者中冠状病毒科感染、β-冠状病毒科感染、sars-cov-2感染、或sars-cov感染,与冠状病毒科或β-冠状病毒科感染、covid-19或严重急性呼吸综合征(sars)相关的疾病或病况的方法。在一些情况下,所述方法是预防受试者中冠状病毒科感染、β-冠状病毒科感染、sars-cov-2或sars-cov感染,或预防个体中与冠状病毒科或β-冠状病毒科感染相关的、或与covid-19或sars感染相关的疾病或病况的发展,的方法。在一些情况下,多肽的至少一个,或至少二个、三个、四个、五个、六个或每一个包括冠状病毒科蛋白、β-冠状病毒科蛋白、sars-cov-2或sars-cov蛋白的片段,其是预测为被受试者的至少二个、或在一些情况下三个或至少三个hlai类等位基因所限制的cd8 t细胞表位。在一些情况下,多肽的至少一个,或至少二个、三个、四个、五个、六个或每一个包括冠状病毒科蛋白、β-冠状病毒科蛋白、sars-cov-2或sars-cov蛋白的片段,其是预测为被受试者的至少二个、或在一些情况下至少三个、或在一些情况下四个或至少四个hlaii类等位基因所限制的cd4 t细胞表位。在一些情况下,多肽的至少一个、或至少二个、三个、四个、五个、六个或每个包括线性b细胞表位。16.在另一方面,本公开提供了17.–如上所述的多肽、多肽组或药物组合物,其用于在受试者中接种疫苗、提供免疫疗法或诱导细胞毒性t细胞应答的方法中,或用于在受试者中治疗或预防冠状病毒科感染、β-冠状病毒科感染、sars-cov-2感染或sars-cov感染,或在受试者中治疗或预防与冠状病毒科或β-冠状病毒科感染或covid-19或sars相关的疾病或病况的发展的方法中,任选地其中受试者如上所述;以及18.–多肽、多肽组或编码如上所述的多肽或多肽组的一或多个多核苷酸或细胞在制备,用于在受试者中接种疫苗、提供免疫疗法或诱导细胞毒性t细胞应答的,或在治疗或预防冠状病毒科感染、β-冠状病毒科感染、sars-cov-2感染或sars-cov感染,在受试者中治疗或预防与冠状病毒科或β-冠状病毒科感染、或covid-19或sars相关的疾病或病况的发展的方法中的,药物中的用途,任选地其中受试者如上所述。19.在一些情况下,本文所述的方法可以包括确定受试者的hlai类和/或ii类基因型的步骤。20.现在将通过示例而非限制的方式并且通过参考附图来更详细地描述本公开。当给出本公开时,许多等同的修改和变化对于本领域技术人员将是显而易见的。因此,所阐述的本公开的示例性实施例被认为是说明性的而非限制性的。在不脱离本公开的范围的情况下,可以对所描述的实施例进行各种改变。本文引用的所有文献,无论是上文还是下文,均明确地通过引用整体并入。21.本公开包含所描述的方面和优选特征的组合,除非这样的组合明显是不允许的或被声明为明确避免。如本说明书和所附权利要求中所用,单数形式“一”、“一个”、和“所述”包含复数指代,除非内容明确另外指明。因此,例如,提及“肽”包含二或更多个这样的肽。22.本文所用的章节标题仅为了方便,而不应被解释为以任何方式进行限制。附图说明23.图1polypepi1018疫苗接种后用富集elispot分析测量的免疫应答。a)按患者绘制的具有免疫反应的疫苗抗原的数量。深灰色实心条:与接种前相比减少或无变化,深灰色条纹条:与接种前相比应答增强(至少增加二倍),浅灰条纹条:从头诱导的疫苗特异性免疫应答。b)在接种前(黑色)、一次接种后(深灰色)、二剂后(浅灰色)、和3剂后(白色)检测polypepi1018疫苗特异性cd4t细胞应答,c)对于八名具有所有三个节点数据的患者,免疫应答经过基线(接种前)和一次接种后第12周测量的时间过程。每条线代表单个患者(n=8)。24.图2疫苗特异性多功能cd8(a)和cd4(b)t细胞应答的平均分布(n=9,在单剂polypepi1018疫苗后测量)。25.图3在患者010007接种前(pre)和38周(post)样品中用ihc检测til。26.图4患者的临床反应。a)11名入选患者对一线治疗的反应和试验期间的recist评估结果的游泳者图,b)显示了oberto-101试验期间靶病变体积的变化(加总)的蜘蛛图。每个数据点被与基线(接种前)测量的病变大小比较,c)单剂和多剂组的无进展存活的kaplan-meier分析。27.图5应答者患者的靶病变大小变化。a)020004患者接受单剂,基线时有二个靶病变,b)010004患者接受多剂,基线时有三个靶病变,c)010007患者接受多剂,基线时有一个靶病变,和d)010002患者接受多剂,基线时有一个靶病变。28.图6预测的(a–b)和测量的(c–d)多抗原免疫应答(agp)倾向于与多剂组(n=5)中的肿瘤体积减少(a和c)和pfs(b和d)相关。29.图7在16个种族的~16,000名受试者中,sfahmed等人提出的随机选择的表位疫苗和polypepi-scov-2疫苗的10个肽的,预测的疫苗诱导的免疫应答率(cd8)的比较。30.图8在16个种族的~16,000名受试者中,sfahmed等人选择的所有59个肽或polypepi-scov-2疫苗的10个肽的,预测的疫苗诱导的免疫应答率(cd8)的比较。31.图9具有针对polypepi-scov-2疫苗的至少2个肽的cd4和cd8t细胞二者的受试者的比例。在16个种族的~16,000个受试者组群中进行预测。32.图10具有针对由sfahmed等人提出的随机选择的表位疫苗诱导和polypepi-scov-2疫苗的10个多肽诱导的≥1–10个疫苗表位的免疫应答的受试者(在~16,000的组群中)的比例。33.图11具有针对由sfahmed等人提出的表位疫苗的所有59个肽或polypepi-scov-2疫苗的10个肽诱导的≥1–10个疫苗表位的免疫应答的受试者(在~16,000的组群中)的比例。34.图12在种族多样的计算机模拟(insilico)人类组群中sars-cov-2spike-1蛋白的热点分析。通过预测每位受试者的≥3个hla等位基因结合个人表位(pepi)进行分析。左图:纵轴的每行代表模型群体中的一个受试者,而横轴代表sars-cov-2s-1蛋白亚基序列。垂直带代表最常见的表位,即,大多数受试者的优势免疫原性蛋白区域(热点)或pepi。ceu,中欧裔;chb,华裔;jpt,日裔;yri,非裔;mix,混合种族受试者。颜色代表被限制在一人上的表位数:浅灰色,3;镭灰色,4;黑色,》5。pepi定义为被限制在一人上的≥3个等位基因的表位。右图,不同种族的受试者的表位/pepi的平均数量。35.图13在二个动物模型中由polypepi-scov-2疫苗接种引发的ifn-γ t细胞应答。与仅接受载体的各对照组群相比,balb/c小鼠(a)和人源化小鼠(hu小鼠)(b)中polypepi-scov-2疫苗诱导的t细胞应答的倍数变化。在第28天在balb/c(c)和人源化(hu小鼠)中检测到,二剂后对sars-cov-2蛋白衍生的疫苗肽特异性的疫苗诱导的t细胞应答(d)。试验条件:s池含有来源于s蛋白的三个肽;n池含有衍生自n蛋白的四个肽;m和e分别是9聚体和30聚体池二者中衍生自m和e蛋白的肽。将结果与相同时间点的载体(用montanide乳化的dmso/水)对照组进行比较。通过用9聚体和30聚体肽刺激进行离体(exvivo)elispot测定。小鼠在第0天和第14天接受二剂疫苗或载体。在每个时间点每个组群包括六只动物。斑点形成单位(sfu)代表2×105个脾细胞的未受刺激的背景校正值。用t检验来计算显著性。36.图14polypepi-scov-2处理增加了小鼠中产生ifn-γ的t细胞。polypepi-scov-2接种的小鼠用深灰色的点显示,并与用浅灰色的点显示的载体(用montanide乳化的dmso/水)对照动物比较。在第14天(a,balb/c;和d,hu小鼠)、第21天(b,balb/c;和e,hu小鼠)、和第28天(c,balb/c;和f,hu小鼠)用肽再刺激后,通过离体elispot分析脾中ifn-γ的产生。条件1,s池;s2、s5、和s9肽的刺突特异性30聚体池。条件2,n池;n1、n2、n3、和n4肽的核蛋白特异性30聚体池。条件3,m1膜特异性30聚体肽。条件4,e1包膜特异性30聚体肽。条件5,s池;相应30聚体的s2、s5、和s9hlai类pepi热点片段的刺突特异性9聚体池。条件6,n池;相应30聚体的n1、n2、n3、和n4hlai类pepi热点片段的核蛋白特异性9聚体池。条件7,m1相应30聚体的膜特异性9聚体hlai类pepi热点片段。条件8,e1相应30聚体的包膜特异性9聚体hlai类pepi热点片段。条件9,未刺激对照。显示了单个斑点形成细胞(sfc)值和平均值,代表每2×105个脾细胞的斑点。分析了每组n=6只小鼠。通过mann-whitney检验进行统计学分析。*,p《0.05;**,p《0.01。37.图15在小鼠模型中th1优势免疫应答且polypepi-scov-2没有诱导显著的th2细胞因子。使用ics检测,balb/c(a)和hu小鼠模型(b)二者的免疫组和载体对照组产生il2、tnf-α、ifn-γ、il-5或il-10的平均cd4 和cd8 t细胞。显示了平均值 /-sem。分析了2×105个细胞,门控cd45 细胞、cd3 t细胞、cd4 或cd8 t细胞。通过汇集cd4 和cd8 t细胞的4个刺激条件(30聚体s池、n池、e1和m1肽)的背景减去的值,得到平均百分比。38.图16balb/c小鼠在第28天th1/th2平衡。使用ics检测,平均cd4 和cd8 t细胞为每个免疫小鼠(n=6)产生il2、tnf-α、ifn-γ(th1细胞因子)和il10(th2细胞因子)。分析了2×105个细胞,门控cd45 细胞、cd3 t细胞、cd4 或cd8 t细胞。通过汇集cd4 和cd8 t细胞的4个刺激条件(30聚体s池、n池、e1和m1肽)的背景减去的值,得到平均百分比。39.图17从balb/c小鼠(a)和hu小鼠(b)模型的血浆中测量的疫苗诱导的igg产生。在第0和14天,小鼠接受二剂polypepi-scov-2疫苗或载体。每个组群包括六只动物。使用t检验来计算显著性。*,p《0.0540.图18covid-19康复者的t细胞对polypepi-scov-2肽的反应性产生细胞因子,通过细胞内染色法测定从他们的pbmc离体确定。(a)通过用9聚体和30聚体肽刺激获得的cd4 和cd8 t细胞 的细胞因子分布(n=17);(b)在用30聚体肽刺激的疫苗特异性t细胞中th1优势。41.图19来自covid-19康复供体的polypepi-scov-2特异性t细胞应答。a.通过离体fluorospot测定法检测的高度特异性疫苗衍生的9聚体肽反应性cd8 t细胞应答和30聚体肽反应性cd4 t细胞应答。b.用30聚体肽池、9聚体肽池、和商业snmo肽池活化的t细胞的富集elispot结果。c.用由对应于具有相同名称的每个30聚体肽的单独9聚体肽活化的cd8 t细胞的富集elispot结果(表9粗体)。dsfu,δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。42.图20通过富集elispot测定法测量,检测到covid-19康复供体针对polypepi-scov-2疫苗的9聚体肽(pepi热点)的ifn-γ t细胞应答。s2、s5、和s9是衍生自刺突特异性疫苗30聚体的三个s特异性9聚体肽序列。n1–n4是衍生自n特异性疫苗30聚体的四个核蛋白特异性9聚体肽序列。e1和m1分别是衍生自e或m特异性疫苗30聚体的包膜和膜特异性9聚体肽序列(表9粗体)。dsfu,δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。呈现了每个受试者的平均和个体数据。pbmc,外周血单核细胞。43.图21covid-19康复供体的t细胞应答相对于从症状发作开始的时间的幅度和广度。a)polypepi-scov-2反应性t细胞应答的幅度。b)用富集elispot测定法检测的来自康复期供体的疫苗肽反应性cd8 t细胞应答的幅度。dsfu代表δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。统计分析:pearson相关分析。r——pearson相关系数。44.图22在covid-19康复期个体中sars-cov-2特异性抗体水平与polypepi-scov-2特异性产生ifn-γ的cd4 t细胞之间的相关性。a)将对polypepi-scov-2肽的30聚体池反应的t细胞应答,对igg-s1(euroimmun)作图。b)将对s-1蛋白衍生的30聚体肽(s2和s5)反应的平均t细胞应答,对igg-s1(euroimmun)作图。c)将对包括n1、n2、n3和n4的30聚体n肽池反应的t细胞应答,对用roche分析测量的总igg-n作图。d)将对polypepi-scov-2的30聚体池反应的t细胞应答,对用diaproigaelisa测定测量的iga抗体量作图。通过pearson检验进行相关性分析。r——相关系数。45.图23预测了具有不同种族的大群体的全球覆盖。a)具有针对九个polypepi-scov-2疫苗肽中的至少一个的hlai类pepi的受试者的比例。b)具有针对polypepi-scov-2疫苗中的至少二个肽的hlai类和ii类pepi二者的受试者的比例。c)ferretti等人(27)提出的基于所选择的六个hla等位基因(a*02:01、a*01:01、a*03:01、a*11:01、a*24:02、和b*07:02)的频率估计的理论全球覆盖率。46.图24代表全球等位基因频率的模型群体中的等位基因频率分布。在模型群体中的hla等位基因频率代表与ciwd数据库中》8百万hla基因分型受试者的等位基因频率相似的分布。ciwd3.0:常见(10,000中≥1)、中间(100,000中≥1)和文献充分记载(≥5次出现)的hla数据库3.0(2020年发布)。r——pearson相关系数。与图20相关。47.图25covid-19康复期内多个自体等位基因结合表位和cd8 t细胞应答之间的相关性。将预测的多个自体hla结合表位(n=9)与n=15个供体中的相同肽反应性cd8 t细胞应答(135个数据点)匹配。数字表示通过富集荧光斑点分析确定的dsfu。颜色代码是指结合特异性肽的自体hla等位基因的预测数目。dsfu,δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。48.图26具有不同种族的个体基于其完整hla基因型的sars-cov-2s1蛋白特异性表位产生能力。与图12相关。对于s2、s5、s9、n1、n2、n3、n4、m1和e1中的每一个,从左到右,这些条分别对应于“全部”、“ceu”、“chb”、“jpt”、“yri”和“mix”。49.图27从最初为sars-cov-2设计的17个30聚体肽为共享sars-cov表位预测在模型群体(n=433)(a)中和在n=16,000大群体(b)中的多肽应答率。50.图28从最初为sars-cov-2设计的17个30聚体肽为共享sars-cov表位预测在模型群体(n=433)(a)中和在n=16,000大群体(b)中的多抗原(多蛋白)应答率。51.图29covid-19康复期个体中多个自体hla等位基因结合表位与产生polypepi-scov-2特异性ifn-γ的cd8 t细胞应答之间的相关性。(a)多个自体hla等位基因结合表位与t细胞应答的量级之间的相关性。rs——spearman系数(也通过pearson相关分析证实)。(b)通过富集fluorospot分析检测的pepi(结合≥3个自体hlai类等位基因)和非pepi(结合≤3个自体hlai类等位基因)的cd8 t细胞应答的量级,(p=0.008,t检验)。给出了每个受试者的中值和个体数据,n=15。(c)变量依赖性分析,使用2×2偶然性表和fisher确切概率检验。(d)对于每个受试者,通过产生ifn-γ的cd8 t细胞证实个人表位(pepi)[阳性预测值=真阳性/预测的总量=37/44(84%)]。dsfu,δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。pbmc,外周血单核细胞。[0052]序列表说明[0053]seqidno:1至17中列出了描述于表6a中的30聚体t细胞表位。[0054]seqidno:18–233列出了本文公开的各种序列。[0055]seqidno:234至267列出了如表15所示的编码seqidno:1至17的氨基酸序列的相应rna或dna序列。具体实施方式[0056]hla基因型[0057]hlas由人类基因组的最多态的基因编码。每个人具有三个hlai类分子(hla-a*、hla-b*、hla-c*)和四个hlaii类分子(hla-dp*、hla-dq*、hla-drb1*、hla-drb3*/4*/5*)的母本和父本等位基因。实际上,每个人表达呈递来自相同蛋白抗原的不同表位的6个i类hla和8个ii类hla分子的不同组合。[0058]用于命名hla分子的氨基酸序列的命名法如下:基因名称*等位基因:蛋白质编号,其例如可以看起来像:hla-a*02:25。在这个例子中,“02”指等位基因。在大多数情况下,等位基因通过血清型定义——意味着给定等位基因的蛋白质在血清学测定中不会彼此反应。当发现蛋白质时,连续分配蛋白质编号(在上述例子中为“25”)。为具有不同氨基酸序列的任何蛋白质分配新的蛋白质编号(例如,即使序列中一个氨基酸的变化也被认为是不同的蛋白质编号)。关于给定基因座的核酸序列的进一步信息可以附加到hla命名中。[0059]个体的i类hla基因型或ii类hla基因型可以指个体的每个i类或ii类hla的实际氨基酸序列,或可以指如上所述的命名,其最低限度指定每个hla基因的等位基因和蛋白质编号。hla基因型可以使用任何合适的方法来确定。例如,序列可以通过使用本领域已知的方法和方案对hla基因座测序来确定。或者,个人的hla集可存储在数据库中并使用本领域已知的方法访问。[0060]一些受试者可能具有编码相同hla分子的二个hla等位基因(例如,在纯合性的情况下,二个拷贝的hla-a*02:25)。由这些等位基因编码的hla分子结合所有相同的t细胞表位。出于本公开的目的,如本文所用的“结合至受试者的至少二个hla分子”包含结合至由单个受试者中的二个相同hla等位基因编码的hla分子。换句话说,“结合至受试者的至少二个hla分子”等可以另外表达为“结合至受试者的至少二个hla等位基因编码的hla分子”。[0061]多肽[0062]本公开涉及衍生自sars-cov-2抗原的多肽,其对大部分人类群体具有免疫原性。[0063]如本文所用,术语“多肽”是指特征为一串氨基酸的全长蛋白质、蛋白质的一部分、或肽。如本文所用,术语“肽”是指包括2、或3、或4、或5、或6、或7、或8、或9、或10、或11、或12、或13、或14、或15和10、或11、或12、或13、或14、或15、或20、或25、或30、或35、或40、或45、或50或55或60个氨基酸的短多肽。[0064]如本文所用,术语“片段”或“多肽片段”是指相对于参考多肽通常具有减小的长度并且在共同部分上包括与参考多肽相同的氨基酸序列的一串氨基酸或氨基酸序列。根据本公开的此类片段可以在适当的情况下被包含在其作为组成部分的较大多肽中。在一些情况下,片段可以包括多肽的全长,例如,其中整个多肽,例如一个9个氨基酸的肽,是单个t细胞表位。在一些情况下,本文提及的片段可以是8或9至20,或25,或30,或35,或40,或45,或50个氨基酸。[0065]如本文所用,术语“表位”或“t细胞表位”是指包含在蛋白抗原内的连续氨基酸序列,其具有对一或多个hla的结合亲和性(能够结合到一或多个hla)。表位是hla和抗原特异性的(hla–表位对,用已知方法预测),但不是受试者特异性的。如果能够在特定的人类受试者中诱导t细胞应答(细胞毒性t细胞应答或辅助t细胞应答)的话,表位、t细胞表位、多肽、多肽片段或包括多肽或其片段的组合物对于该受试者是“免疫原性的”。在一些情况下,辅助t细胞应答是th1型辅助t细胞应答。术语“t细胞应答”和“免疫应答”在此可互换使用,并且是指在识别一或多个hla–表位结合对之后t细胞的活化和/或一或多个效应子功能的诱导。在一些情况下,“免疫应答”包含抗体应答,因为ii类hla分子刺激参与诱导持久的ctl应答和抗体应答二者的辅助应答。效应子功能包含细胞毒性、细胞因子产生和增殖。根据本公开内容,表位、t细胞表位或多肽片段如果能够结合特定受试者的至少二个、或在一些情况下至少三个,i类或至少二个、或在一些情况下至少三个或至少四个ii类hla,则对于受试者是免疫原性的。[0066]“个人表位”(或“pepi”)是由多肽的连续氨基酸序列组成的多肽片段,该多肽是能够结合特定人类受试者的一或多个hlai类分子的t细胞表位。在其它情况下,“pepi”是由多肽的连续氨基酸序列组成的多肽片段,该多肽是能够结合特定人类受试者的一或多个hlaii类分子的t细胞表位。换句话说,“pepi”是被特定个体的hla组识别的t细胞表位,因此除了hla和抗原之外,还对受试者具有特异性。与仅对hla和抗原特异的“表位”相反,pepi对个体是特异的,因为不同的个体具有各自结合不同t细胞表位的不同hla分子。[0067]如本文所用的“pepi1”是指可结合个体的一个hlai类分子(或在特定情况下hlaii类分子)的肽或多肽片段。“pepi1 ”是指能与个体的一或多个hlai类分子结合的多肽、或多肽片段。“pepi2”是指能与个体的二个hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi2 ”是指能与个体的二或更多个的hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi3”是指能与个体的三个hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi3 ”是指能与个体的三或更多个hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi4”是指能与个体的三个hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi4 ”是指能与个体的三或更多个hlai类(或ii类)分子结合的多肽、或多肽片段。[0068]一般而言,由hlai类分子呈递的表位的长度为约九个氨基酸,而由hlaii类分子呈递的表位的长度为约十五个氨基酸。然而,为了本公开的目的,表位可以多于或少于九个(对于i类hla)或十五个(对于ii类hla)氨基酸长度,只要该表位能够结合hla即可。例如,能够结合i类hla的表位长度可以在7、或8或9和9或10或11个氨基酸之间。能够结合ii类hla的表位的长度可以在13、或14或15和15或16或17个氨基酸之间。[0069]受试者的给定hla仅向t细胞呈递通过在抗原呈递细胞(apc)中加工蛋白抗原产生的有限数量的不同肽。如本文所用,“展示”或“呈递”,当与hla相关使用时,是指肽(表位)和hla之间的结合。在这方面,“展示”或“呈递”肽与“结合”肽同义。[0070]使用本领域已知的技术,有可能确定将结合已知hla的表位。可以使用任何合适的方法,只要使用相同的方法来确定被直接比较的多个hla–表位结合对。例如,可以使用生化分析。也可以使用已知与给定hla结合的表位列表。也可以使用预测或建模软件来确定哪些表位可以被给定的hla结合。表1提供了示例。在一些情况下,如果t细胞表位具有小于5000nm、小于2000nm、小于1000nm、或小于500nm的ic50或预测ic50,则该t细胞表位能够结合给定的hla。[0071]表1用于确定表位–hla结合的示例软件[0072][0073]本公开的肽可以包括一或多个冠状病毒科、β-冠状病毒科或sars-cov-2的选自表面糖蛋白(别名刺突蛋白)、核壳磷蛋白、包膜蛋白和膜糖蛋白的抗原,的一或多个片段,或由其组成。本文提供了参考序列。[0074]在一些情况下,氨基酸序列在n和/或c末端侧翼为不是冠状病毒科、β-冠状病毒科或sars-cov-2抗原序列的一部分的额外氨基酸,换句话说,不是与靶多肽抗原中所选片段相邻的连续氨基酸的相同序列。在一些情况下,序列在n和/或c末端侧翼有至多20、19、18、17、16、15、14、13、12、11、10、9、8、7、6、5、4、3、2或1个额外的氨基酸。[0075]发明人先前发现癌症疫苗中存在至少二个可结合个体的至少三个hlai类(≥2pepi3 )的多肽片段(表位)预示了临床反应。换句话说,如果在疫苗的活性成分多肽中鉴定出≥2pepi3 ,则个体可能是临床应答者。[0076]不希望被理论所束缚,发明人相信,从包括至少二个多hla结合性pepi的疫苗/免疫疗法中获得临床益处的可能性增加的一个原因,是患病的细胞群体,例如癌症或肿瘤细胞或被病毒或病原体(例如hiv)感染的细胞,在受影响的受试者内和受试者间二者都通常是异质的。此外,当更多的hla结合性pepi被包含或被疫苗靶向时,发展抗性的可能性被降低,因为患者不太可能通过靶pepi的突变发展对组合物的抗性。[0077]同样,在病毒感染的疫苗的情况下,其中病毒感染可以是异源的,向受试者施用疫苗肽是有利的,该疫苗肽被预测为包括多个受试者特异性多hla等位基因结合性pepi(用于治疗具有已知hla基因型的受试者)或多个群体最佳epi(bestepi),即在高比例的靶标群体中是或包括多hla等位基因结合性pepi的氨基酸序列。包含更多的最佳epi序列也增加了对治疗有反应的人类受试者的总比例。因此,在一些情况下,多肽组包括至少2、3、4、5、6、7、8、9、10、11、12、13、14、15、16或17个多肽,其各自包括选自seqidno:1至17的一个不同的氨基酸序列。在一些情况下,多肽的组合排除以下组合中的一或多个:seqidno:1和2;seqidno:3和4;seqidno:7和8;和/或seqidno:9和10;和/或排除以下组合中的一或多个:seqidno:2和3;和/或seqidno:13和14。[0078]多肽可以是或包括相同或不同病毒抗原的片段。不同的病毒结构蛋白可能倾向于以不同的速率突变。因此,在一些情况下,每个多肽包括选自seqidno:1至17的氨基酸序列,该序列是不同的冠状病毒科、β-冠状病毒科或sars-cov-2蛋白的片段。在一些情况下,多肽组包含来自以下组中的至少二组、三组或所有四组的至少一个序列:(a)seqidno:1至11(sars-cov-2表面蛋白的片段),任选地排除seqidno:1和2、seqidno:2和3、seqidno:3和4、seqidno:7和8、和/或seqidno:9和10;(b)seqidno:12至15(sars-cov-2核壳蛋白的片段),任选地排除seqidno:13和14;(c)seqidno:16(sars-cov-2膜蛋白的片段);和(d)seqidno:17(sars-cov-2包膜蛋白的片段)。在一些情况下,多肽的组合包括十个多肽或由其组成,多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列或由其组成。在另一个实施方式中,多肽组包括九个多肽,肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列或由其组成。在另一个实施方式中,多肽组包括十个多肽,多肽包括seqidno:6、和9至17的氨基酸序列或由其组成。[0079]多肽和患者的选择[0080]本文所述的肽可用于在有需要的受试者中诱导t细胞应答或提供疫苗接种或免疫治疗。所述的肽可用于治疗或预防受试者的冠状病毒科感染、β-冠状病毒科感染sars-cov-2感染、sars-cov感染、与冠状病毒科或β-冠状病毒科感染、covid-19或sars相关的疾病或病况。通常选择多于一个肽用于治疗受试者。在一些情况下,用于治疗的肽可以基于(i)受试者中待治疗的疾病或病况;(ii)受试者的hla基因型;和/或(iii)受试者(例如国籍或族群)的遗传背景。[0081]根据本发明可以治疗的冠状病毒科感染(和相关疾病)包含任何其中的病毒表达至少一个包括seqidno:1至17中任何一个的氨基酸序列(或表6a中所示seqidno:1至17中的最佳epi序列)的抗原。通常,病毒表达一起包括至少二个、或更通常至少3、4、5、6、7、8、9或10个不同的选自seqidno:1至17的序列(或最佳epi序列)的一或多个抗原。更典型地,病毒表达二或更多个不同的抗原,其中每一个抗原包括选自seqidno:1至17的序列(或最佳epi序列)。合适的如本文所述用于治疗特定冠状病毒科的本发明的多肽或本发明的药物组合物或试剂盒,是与特定病毒表达的抗原片段序列匹配的那些多肽或药物组合物或试剂盒。技术人员可以基于本文提供的公开内容和实施例容易地鉴定和选择这样的多肽。[0082]通常用于疫苗接种和免疫疗法的多肽抗原、特别是衍生自多肽抗原的短肽,仅在一部分人类受试者中诱导免疫应答。本公开的肽被特别选择以在全球群体的大部分中诱导免疫应答。然而,由于hla基因型异质性,它们可能不是在所有个体中都有效。[0083]发明人发现,由个体表达的多种hla通常需要呈递相同的肽以触发t细胞应答。因此,预计对特定个体(pepi)具有免疫原性的多肽抗原(表位)的片段是那些可以结合由该个体表达的多种i类(激活的细胞毒性t细胞)或ii类(激活的辅助t细胞)hla的片段。一般而言,受试者中对特定疫苗肽的细胞毒性t细胞应答通过疫苗肽中存在≥1pepi3 (结合受试者的三个或更多个i类hla等位基因的表位)最佳地预测。辅助t细胞应答通常由≥1pepi3 或≥1pepi4 (结合受试者的三或更多个或四或更多个ii类hla等位基因的表位)最佳地预测。[0084]因此,本公开提供了预测人类受试者对施用如本文所述的多肽组或药物组合物将具有t细胞应答(细胞毒性和/或辅助)的方法。所述方法可以包括(a)(i)确定多肽组或药物组合物的活性成分多肽包括被受试者的至少三个hlai类分子所限制的t细胞表位;和(ii)预测受试者将对多肽组或药物组合物的施用具有细胞毒性(cd8 )t细胞应答;和/或(b)(i)确定多肽组或药物组合物的活性成分多肽包括被受试者的至少三个、或在一些情况下至少四个hlaii类分子所限制的t细胞表位;和(ii)预测受试者将对多肽组或药物组合物的施用具有辅助(cd4 )t细胞应答。[0085]本公开还提供了确定特定人类受试者对施用本文所述的多肽组或药物组合物将具有t细胞应答(细胞毒性/cd8 或辅助/cd4 )的可能性的方法,其中所述方法包括鉴定多肽或活性成分多肽中被受试者的至少三个i类hla或至少三个或至少四个ii类hla所限制的t细胞表位,并且其中(a)(a)被受试者的至少三个i类hla所限制的较高数量的t细胞表位;和/或[0086](b)既(i)被受试者的至少三个hlai类所限制;且(ii)为不同sars-cov-2结构蛋白的片段,的较高数量的t细胞表位,对应于受试者中细胞毒性/cd8 t细胞应答的较高概率;且/或(b)(a)被受试者的至少三个或至少四个ii类hla所限制的更高数量的t细胞表位;和/或(b)既(i)被受试者的至少三个或至少四个ii类hla所限制;且为(ii)不同sars-cov-2结构蛋白的片段,的更高数量的t细胞表位,对应于受试者中辅助t细胞/cd4 t细胞应答的较高概率。[0087]在一些情况下,可以预测受试者具有细胞毒性t细胞应答,或高于对肽组或药物组合物的施用具有细胞毒性t细胞应答的预定阈值概率,并且所述方法还包括选择或推荐施用药物组合物作为治疗受试者的方法,并且任选地还包括通过向受试者施用多肽组或药物组合物来治疗受试者。[0088]本公开还提供了如本文所述的治疗方法,其中已经预测接受治疗的受试者对使用本文所述的方法施用多肽组或药物组合物具有细胞毒性或辅助t细胞应答,或对使用本文所述的方法施用多肽组或药物组合物具有细胞毒性t或辅助细胞应答的预定阈值概率高。所述方法可包括使用本文所述的方法选择预测对特定受试者具有免疫原性的肽。包括为受试者选择的肽作为活性成分的试剂盒的药物组合物可被认为是为受试者个性化的(即个性化药物)。所述方法可进一步包括向受试者施用。[0089]发明人进一步发现,在疫苗或免疫治疗组合物中存在(i)对应于一或多个靶多肽抗原的片段,且(ii)可以结合个体的至少三个i类hla等位基因的至少二个t细胞表位,是临床反应的前兆。本文所用的“临床反应”或“临床益处”可以是预防或延迟疾病或病况的发作、改善一或多个症状、诱导或延长缓解、或延迟复发或再发或恶化、或受试者疾病状态的任何其它改善或稳定。临床反应还可以是预防由冠状病毒科病毒的不同突变变体引起的感染。[0090]因此,本公开的一些方面涉及预测特定人类受试者将对如本文所述的治疗方法或对施用如本文所述的肽组或药物组合物具有临床反应的方法,或确定临床反应的概率的方法。所述方法类似于本文所述的用于预测t细胞应答的方法,但是通过确定多肽组或药物组合物的活性成分多肽包括各自被受试者的至少三个hlai类分子所限制的二个不同的t细胞表位来预测临床应答。[0091]药物组合物、治疗方法和给药方式[0092]在一些方面,本公开涉及包括如本文所述的肽、多核酸、载体或细胞中的一或多个的药物组合物或试剂盒。这样的药物组合物或试剂盒可以用于诱导免疫应答、治疗、接种或向受试者提供免疫治疗的方法中。药物组合物或试剂盒可以是疫苗或免疫治疗组合物或试剂盒。本文所述的治疗方法可包括向受试者施用药物组合物。[0093]如本文所用的术语“活性成分”是指旨在诱导免疫应答的多肽,并且可以包含在施用给受试者后在体内产生的疫苗或免疫治疗组合物的多肽产物。对于dna或rna免疫治疗组合物,多肽可以由施用组合物的受试者的细胞在体内产生。对于基于细胞的组合物,多肽可以由组合物的细胞加工和/或呈递,例如自体树突细胞或用多肽脉冲的或包括编码多肽的表达构建体的抗原呈递细胞。药物组合物或试剂盒可以包括编码一或多个活性成分多肽的多核苷酸或细胞。[0094]除了一或多个肽、核酸、载体或细胞之外,本文所述的药物组合物或试剂盒可以包括药学上可接受的赋形剂、载体、稀释剂、缓冲剂、稳定剂、防腐剂、佐剂或本领域技术人员熟知的其它材料。这样的物质优选是无毒的,并且优选不干扰活性成分的药物活性。药物载体或稀释剂可以是,例如,含水溶液和水/油乳液。载体或其它材料的确切性质可取决于施用途径,例如口服、静脉内、皮肤或皮下、鼻、肌内、皮内、和腹膜内途径。[0095]本公开的药物组合物可以包括一或多个“药学上可接受的载体”。这些通常是大的、代谢缓慢的大分子,例如蛋白质、糖、聚乳酸、聚乙醇酸、聚合氨基酸、氨基酸共聚物、蔗糖(paoletti,2001,vaccine,19:2118–2126)、海藻糖(wo00/56365)、乳糖和脂质聚集体(例如油滴或脂质体)。这些载体是本领域普通技术人员公知的。药物组合物还可以含有稀释剂,例如水、盐水、甘油等。另外,可以存在辅助物质,例如润湿剂或乳化剂、ph缓冲物质等。无菌无热原的磷酸盐缓冲生理盐水是典型的载体(gennaro,2000,remington:scienceandpracticeofpharmacy,第20版,isbn:0683306472)。[0096]本公开的药物组合物可以是冻干的或水性形式,即,溶液或悬浮液。这种类型的液体制剂允许组合物直接以其包装形式给药,而不需要在水性介质中重构,因此对于注射是理想的。药物组合物可以存在于小瓶中,或者它们可以存在于已填充的注射器中。注射器可以具有或不具有针头。注射器包含单剂,而小瓶可包含单剂或多剂。[0097]本公开的液体制剂也适于从冻干形式重构其它药物。当药物组合物用于这种临时重构时,本公开提供了试剂盒,其可以包括二个小瓶,或可以包括一个预填充注射器和一个小瓶,其中注射器的内容物用于在注射前重构小瓶的内容物。[0098]本公开的药物组合物可以包含抗微生物剂,特别是当以多剂形式包装时。可以使用抗微生物剂,例如2-苯氧基乙醇或对羟基苯甲酸酯(对羟基苯甲酸甲酯、对羟基苯甲酸乙酯、对羟基苯甲酸丙酯)。任何防腐剂优选以低水平存在。防腐剂可以外源加入且/或可以是混合形成组合物的大量抗原的组分(例如,作为百日咳抗原中的防腐剂存在)。[0099]本公开的药物组合物可包括去污剂,例如吐温(聚山梨醇酯)、dmso(二甲亚砜)、dmf(二甲基甲酰胺)。去污剂通常以低含量存在,例如《0.01%,但也可以较高含量使用,例如0.01–50%。[0100]本公开的药物组合物可以包含钠盐(例如氯化钠)和溶液中的游离磷酸根离子(例如通过使用磷酸盐缓冲液)。[0101]在某些实施方式中,药物组合物可以被包封在合适的载体中,以将肽递送到抗原呈递细胞中或增加稳定性。如本领域技术人员将理解的,多种载体适于递送本公开的药物组合物。合适的结构化流体递送系统的非限制性实例可包含纳米颗粒、脂质体、微乳液、胶束、树枝状聚合物和其它含磷脂的系统。将药物组合物掺入递送载体的方法是本领域已知的。[0102]为了增加组合物的免疫原性,药理学组合物可以包括一或多个佐剂和/或细胞因子。[0103]合适的佐剂包含铝盐,例如氢氧化铝或磷酸铝,但也可以是钙盐、铁盐或锌盐,或者可以是酰化酪氨酸、或酰化糖的不溶性悬浮液,或者可以是阳离子或阴离子衍生的糖、聚磷腈、生物可降解微球、单磷酰脂质a(mpl)、脂质a衍生物(例如毒性降低的)、3-o-脱酰mpl(3d-mpl)、quila、皂苷、qs21、弗氏不完全佐剂(密歇根州底特律的difcolaboratories)、merck佐剂65(新泽西州rahway的merckandcompany,inc.)、as-2(宾夕法尼亚州费城的smith-klinebeecham)、cpg寡核苷酸、生物粘合剂和粘膜粘合剂、微粒、脂质体、聚氧乙烯醚制剂、聚氧乙烯酯制剂、胞壁酰肽或咪唑并喹诺酮化合物(例如咪喹莫特及其同系物)。在本发明公开内容中适合用作佐剂的人免疫调节剂包含细胞因子,例如白介素(例如il-1、il-2、il-4、il-5、il-6、il-7、il-12等)、巨噬细胞集落刺激因子(m-csf)、肿瘤坏死因子(tnf)、粒细胞、巨噬细胞集落刺激因子(gm-csf)也可用作佐剂。[0104]在一些实施方式中,组合物包括佐剂,其选自由montanideisa-51(美国新泽西州fairfield的seppic,inc.)、qs-21(美国马萨诸塞州lexington的aqualiabiopharmaceuticals,inc.)、gm-csf、环磷酰胺、卡介苗(bcg)、短小棒状杆菌、左旋咪唑、azimezone、异丙肾上腺素、二硝基氯苯(dncb)、匙孔血蓝蛋白(klh)、弗氏佐剂(完全和不完全)、矿物凝胶、氢氧化铝(明矾)、溶血卵磷脂、普朗尼克多元醇、聚阴离子、油乳剂、二硝基苯酚、白喉毒素(dt),组成的组。在一个具体实施方式中,佐剂是montanide佐剂。[0105]例如,细胞因子可以选自由转化生长因子(tgf)例如但不限于tgf-α和tgf-β;胰岛素样生长因子i和/或胰岛素样生长因子ii;红细胞生成素(epo);骨诱导因子;干扰素例如但不限于干扰素α、干扰素β、和干扰素γ;集落刺激因子(csf)例如但不限于巨噬细胞-csf(m-csf)、粒细胞-巨噬细胞-csf(gm-csf)、和粒细胞-csf(g-csf),组成的组。在一些实施方式中,细胞因子选自由神经生长因子例如ngf-β;血小板生长因子;转化生长因子(tgf)例如但不限于tgf-α和tgf-β;胰岛素样生长因子i和胰岛素样生长因子ii;红细胞生成素(epo);骨诱导因子;干扰素(ifn)例如但不限于ifn-α、ifn-β、和ifn-γ;集落刺激因子(csf)例如巨噬细胞-csf(m-csf)、粒细胞-巨噬细胞-csf(gm-csf)、和粒细胞-csf(g-csf);白细胞介素(il)例如但不限于il-1、il-1α、il-2、il-3、il-4、il-5、il-6、il-7、il-8、il-9、il-10、il-11、il-12、il-13、il-14、il-15、il-16、il-17、il-18;lif;kit-配体或flt-3;制管张素;血小板反应蛋白;内皮抑素;肿瘤坏死因子(tnf);和lt,组成的组。[0106]预期佐剂或细胞因子可以以每剂约0.01mg至约10mg的量加入,优选以每剂约0.2mg至约5mg的量加入。或者,佐剂或细胞因子的浓度可以是约0.01至50%,优选约2%至30%。[0107]在某些方面,本公开的药物组合物是根据已知技术通过在适当的无菌条件下将佐剂和/或细胞因子与本文所述的肽物理混合以产生最终产物来制备的。[0108]多肽片段的合适组合物和施用方法的实例在esseku和adeye(2011)和vandenmooterg.(2006)中提供。疫苗和免疫疗法组合物的制备一般在vaccinedesign(“thesubunitandadjuvantapproach”(edspowellm.f.&newmanm.j.(1995)plenumpressnewyork)中描述。fullerton在美国专利4,235,877中描述了在脂质体内的封装,这也是设想中的封装。[0109]在一些实施方式中,本文公开的组合物被制备为(核糖)核酸疫苗。在一些实施方式中,核酸疫苗是dna疫苗。在一些实施方式中,dna疫苗或基因疫苗包括具有启动子和适当转录和翻译控制元件的质粒以及编码一或多个本公开内容的多肽的核酸序列。在一些实施方式中,质粒还包含增强例如表达水平、细胞内靶向或蛋白酶体加工的序列。在一些实施方式中,dna疫苗包括含有编码一或多个本公开内容的多肽的核酸序列的病毒载体。在另外的方面,本文公开的组合物包括一或多个编码被确定与生物样品具有免疫反应性的肽的核酸。例如,在一些实施方式中,组合物包括编码1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、或更多个肽的一或多个核苷酸序列,所述肽包括作为能够结合患者的至少三个hlai类分子和/或至少三个或四个hlaii类分子的t细胞表位的片段。在一些实施方式中,dna或基因疫苗还编码免疫调节分子以操纵所产生的免疫应答,例如增强疫苗的效力、刺激免疫系统或降低免疫抑制。增强dna或基因疫苗免疫原性的策略包含编码异种形式的抗原,将抗原与激活t细胞或触发联合识别的分子融合,用dna载体引发,然后用病毒载体加强,以及利用免疫调节分子。在一些实施方式中,通过针、基因枪、气溶胶注射器、贴片、微针、研磨等形式引入dna疫苗。在一些形式中,dna疫苗被掺入脂质体或其它形式的纳米体中。在一些实施方式中,dna疫苗包含选自由转染剂;鱼精蛋白;鱼精蛋白脂质体;多糖颗粒;阳离子纳米乳液;阳离子聚合物;阳离子聚合物脂质体;阳离子纳米颗粒;阳离子脂质和胆固醇纳米颗粒;阳离子脂质、胆固醇、和peg纳米颗粒;树枝状聚合物纳米颗粒,组成的组。在一些实施方式中,dna疫苗通过吸入或摄取施用。在一些实施方式中,将dna疫苗引入血液、胸腺、胰腺、皮肤、肌肉、肿瘤或其它部位。[0110]在一些实施方式中,本文公开的组合物被制备为rna疫苗。在一些实施方式中,rna是非复制性mrna或病毒衍生的自扩增rna。在一些实施方式中,非复制mrna编码本文公开的肽,并含有5′和3′非翻译区(utr)。在一些实施方式中,病毒衍生的自扩增rna不仅编码本文公开的肽,而且编码能够使细胞内rna扩增和大量蛋白表达的病毒复制机制。在一些实施方式中,rna被直接引入个体。在一些实施方式中,rna是化学合成的或体外(invitro)转录的。在一些实施方式中,使用t7、t3或sp6噬菌体rna聚合酶从线性dna模板产生mrna,所得产物含有编码本文公开的肽的开放阅读框、侧翼utr、5′帽和聚腺苷酸尾。在一些实施方式中,在使用痘苗病毒加帽酶或通过掺入合成的帽或抗反向帽类似物的转录反应期间或之后加入各种形式的5′帽。在一些实施方式中,将最佳长度的poly(a)尾直接从编码dna模板或通过使用poly(a)聚合酶加到mrna上。rna可以编码一或多个,包括能够结合患者的至少三个i类hla和/或至少三个或四个ii类hla分子的t细胞表位的片段,的肽。所述片段来源于在冠状病毒科中表达的抗原。在一些实施方式中,rna包含增强稳定性和翻译的信号。在一些实施方式中,rna还包含非天然核苷酸以增加半衰期,或修饰的核苷以改变免疫刺激特征。在一些实施方式中,通过针、基因枪、气溶胶注射器、贴片、微针、研磨等形式引入rna。在一些形式中,rna疫苗被掺入脂质体或其它形式的纳米抗体中,该纳米抗体促进rna的细胞摄取并保护其免于降解。在一些实施方式中,rna疫苗包含递送系统,其选自由转染剂;鱼精蛋白;鱼精蛋白脂质体;多糖颗粒;阳离子纳米乳液;阳离子聚合物;阳离子聚合物脂质体;阳离子纳米颗粒;阳离子脂质和胆固醇纳米颗粒;阳离子脂质、胆固醇、和peg纳米颗粒;树枝状聚合物纳米颗粒;和/或裸mrna;体内电穿孔的裸mrna;鱼精蛋白复合的mrna;与带正电荷的水包油阳离子纳米乳液缔合的mrna;与化学修饰的树枝状聚合物相关并与聚乙二醇(peg)–脂质复合的mrna;peg–脂质纳米颗粒中的鱼精蛋白复合的mrna;与阳离子聚合物如聚乙烯亚胺(pei)结合的mrna;与阳离子聚合物如pei和脂质组分结合的mrna;与多糖(例如壳聚糖)颗粒或凝胶结合的mrna;阳离子脂质纳米颗粒(例如,1,2-二油酰氧基3-三甲基铵丙基丙酮(dotap)或二油酰磷脂酰乙醇胺(dope)脂质)中的mrna;与阳离子脂质和胆固醇复合的mrna;或与阳离子脂质、胆固醇和peg–脂质复合的mrna,组成的组。在一些实施方式中,rna疫苗通过吸入或摄取施用。在一些实施方式中,rna被引入血液、胸腺、胰腺、皮肤、肌肉、肿瘤、或其他部位,和/或通过皮内、肌内、皮下、鼻内、结节内、静脉内、脾内、肿瘤内或其他递送途径引入。[0111]多核苷酸或寡核苷酸组分可以是裸露的核苷酸序列或与阳离子脂质、聚合物或靶向系统组合。它们可以通过任何可用的技术来递送。例如,多核苷酸或寡核苷酸可以通过针注射导入,优选皮内、皮下或肌内注射。或者,多核苷酸或寡核苷酸可以使用递送装置,例如颗粒介导的基因递送,直接递送穿过皮肤。多核苷酸或寡核苷酸可局部施用于皮肤或粘膜表面,例如通过鼻内、口服、或直肠内施用。[0112]核酸构建体的摄取可通过几种已知的转染技术来增强,例如那些包含使用转染剂的技术。这些试剂的实施例包含阳离子试剂,例如,磷酸钙和deae-dextran和脂质转染试剂,例如lipofectam和transfectam。可以改变待施用的多核苷酸或寡核苷酸的剂量。[0113]因此,本发明提供了疫苗或药物组合物或试剂盒,其包括编码一或多个、或至少一个(或至少2、3、4、5、6、7、8、9或10个)选自seqidno:1至17的多肽序列的,一或多个多核苷酸(多核酸)或多核糖核苷酸(核糖多核酸)。所述的多核苷酸或核糖多核苷酸可编码一或多个冠状病毒科蛋白、或β-冠状病毒科科蛋白、sars-cov-2蛋白、或sars-cov蛋白的一或多个片段,或任何冠状病毒科的表达一或多个蛋白的包括一或多个(或2、或3、4、5、6、7、8、9、或10或更多个)选自seqidno:1至17的氨基酸序列的蛋白。所述片段包括选自seqidno:1至17的氨基酸序列,并且通常长度至多50个氨基酸。多核苷酸或多核糖核苷酸可以包括至少一个(或至少2、3、4、5、6、7、8、9或10个)选自seqidno:234至267的序列。通常,多核苷酸或多核糖核苷酸一起包括选自seqidno:234至250或251至267的序列,所述序列编码选自seqidno:1–17的氨基酸序列。更典型地,所述编码选自编码seqidno:1–17的不同氨基酸序列的多核苷酸或核糖多核苷酸,是由冠状病毒科表达的不同蛋白质的片段。例如,多核苷酸或核糖多核苷酸可一起包括至少一个选自以下组中的至少二组、至少三组或所有四组中的每一组的序列:(a)seqidno:234至244或seqidno:251至261;(b)seqidno:245至248或seqidno:262至265;(c)seqidno:249或seqidno:266;和(d)seqidno:250或seqidno:267。多核苷酸或核糖多核苷酸可一起包括以下列表之一中的至少一个(或至少2、3、4、5、6、7、8、9个或全部)序列:seqidno:236、238、242、245、246、247、248、249和250;seqidno:236、238、240、242、245、246、247、248、249和250;seqidno:239、242、243、244、245、246、247、248、249和250;seqidno:252、255、259、262、263、264、265、266和267;seqidno:252、255、257、259、262、263、264、265、266和267;和seqidno:256、259、260、261、262、263、264、265、266和267。多核苷酸或核糖多核苷酸可以编码本文所述的本发明的任何多肽组。多核苷酸可以是dna。多核糖核苷酸可以是rna。例如,多核糖核苷酸可以是mrna。[0114]本发明还包括基于细胞的组合物。一或多个多肽或多肽组存在于细胞表面,特别是在施用后存在于患者体内。在一些情况下,细胞可以是(自体)树突细胞或抗原呈递细胞。细胞可以用多肽冲击,或者包括编码多肽的一或多个表达构建体/盒。表达构建体/表达盒可包括/表达上文所述的任何多核苷酸或核糖多核苷酸。[0115]如本文所用,术语“治疗”包含治疗性和预防性治疗。给药通常是“预防有效量”或“治疗有效量”(视情况而定,尽管预防可以被认为是治疗),这足以导致临床反应或对个体显示临床益处,例如,有效量预防或延迟疾病或病况的发作,改善一或多个症状,诱导或延长缓解,或延迟复发或再发。[0116]剂可以根据各种参数确定,尤其是根据所用的物质;待治疗个体的年龄、体重和状况;给药途径;以及所需的方案。每剂中的抗原量选择为诱导免疫应答的量。医师将能够确定任何特定个体所需的给药途径和剂量。剂可以作为单剂提供,或者可以作为多剂提供,例如以规则间隔服用,例如每小时施用2、3或4剂。典型地,肽、多核苷酸或寡核苷酸的给药范围典型地为1pg至1mg、更典型地为1pg至10μg,用于颗粒介导的递送,以及1μg至1mg、更典型地为1–100μg、更典型地为5–50μg,用于其它途径。通常,预期每剂将包括0.01–3mg抗原。特定疫苗的最佳量可以通过涉及观察受试者中的免疫应答的研究来确定。[0117]上述技术和方案的实例可以在remington'spharmaceuticalsciences,20thedition,2000,pub.lippincott,williams&wilkins中找到。[0118]在一些情况下,治疗方法可包括向受试者施用多于一个的肽、多核酸或载体。这些可以一起/同时和/或在不同时间或顺序施用。使用任选靶向不同抗原的不同肽的组合,对于克服个体的病毒异质性和hla异质性的挑战可能是重要的。组合使用本公开的肽扩大了可经历来自疫苗接种的临床益处的个体的组。为在一个方案中使用而制造的多种药物组合物可以定义药物产品。在一些情况下,可以在例如1年、或6个月、或3个月、或60或50或40或30天的时间段内向受试者施用单次治疗的不同的肽、多核酸或载体。[0119]给药途径包含但不限于鼻内、口服、皮下、皮内、和肌内。特别优选皮下给药。皮下给药可以例如通过注射到腹部、上臂或大腿的侧面和前面、背部的肩胛区域、或上腹侧臀部区域。[0120]本公开的组合物还可以以一、或多剂,以及,通过其他施用途径施用。例如,这些其它途径包含皮内、静脉内、血管内、动脉内、腹膜内、鞘内、气管内、心内、叶内、髓内、肺内、和阴道内。根据所需的治疗持续时间,根据本公开的组合物可以一次或多次施用,也可以间歇施用,例如每月施用数月或数年,并且以不同的剂量施用。[0121]用于口服给药的固体剂型包含胶囊、片剂、囊片、丸剂、粉剂、丸剂、和颗粒剂。在这样的固体剂型中,活性成分通常与一或多个药学上可接受的赋形剂组合,其例子如上详述。口服制剂也可以作为水性悬浮液、酏剂或糖浆施用。为此,活性成分可以与各种甜味剂或调味剂、着色剂、以及如果需要的话,乳化剂和/或悬浮剂、以及稀释剂如水、乙醇、甘油及其组合,来结合。[0122]本公开的一或多个组合物的施用、或根据本公开的用于治疗的方法和用途的进行,可以单独,或与其他药理学组合物或治疗,例如其他免疫疗法、疫苗或抗病毒剂组合。其它治疗性组合物或治疗可以与本公开的组合物或治疗同时或相继(之前或之后)施用。[0123]在一些情况下,治疗方法是疫苗接种方法或提供免疫治疗的方法。如本文所用,“免疫疗法”是通过诱导或增强个体中的免疫应答来治疗疾病或病况。在某些实施方式中,免疫治疗是指包括向个体施用一或多个药物以引发t细胞应答的治疗。在一个具体的实施方式中,免疫治疗是指一种治疗,其包括向个体施用或表达含有一或多个pepi的多肽,以引发t细胞应答,从而识别并杀死在其细胞表面展示与i类hla结合的一或多个pepi的细胞。在另一个实施方式中,免疫治疗是指一种治疗,其包括向个体施用或表达含有由ii类hla呈递的一或多个pepi的多肽,以引发辅助t细胞应答,从而向识别并杀死在其细胞表面展示与i类hla结合的一或多个pepi的患病细胞的细胞毒性t细胞提供共刺激。在更另一个具体实施方式中,免疫治疗是指包括向个体施用一或多个药物的治疗,所述药物再活化存在的t细胞以杀死靶细胞和/或病毒。[0124]本发明包括治疗或预防受试者中的冠状病毒科感染或与冠状病毒科感染相关的疾病或病况的方法。与冠状病毒科感染相关的疾病或状况包含由感染引起的,例如直接由感染引起的任何疾病或病况、症状或其他疾病属性。[0125]在一些情况下,冠状病毒科是β-冠状病毒科,例如sars-cov-2或其变体或突变菌株。冠状病毒科可以是sars-cov。具体地,冠状病毒科表达一或多个抗原/多肽,所述抗原/多肽包括选自seqidno:1至17所示的一或多个氨基酸序列,或一或多个在表6a中以粗体和/或下划线显示的最佳epi序列。更具体地,特定的冠状病毒科可使用组合物或试剂盒来治疗,其中活性成分多肽包括选自seqidno:1至17(或最佳epi序列)的一或多个(通常是2或更多个,或3、4、5、6、7、8、或9或更多个)序列,这些序列在特定病毒表达的抗原中发现。本文描述了特别适合于或优化了的用于治疗或预防由sars-cov-2或sars-cov感染引起的疾病的特定组合物。然而,技术人员能够使用本公开内容来确定其他组合物或试剂盒,这些组合物或试剂盒具有包括seqidno:1至17的氨基酸序列的不同组合的多肽作为活性成分,用于预防或治疗其他冠状病毒科。可以如上文和下文实施例中所述选择用于特定冠状病毒科和/或患者的合适治疗。[0126]公开内容的进一步实施方式——(1)[0127]1.一种多肽疫苗,其包括包括选自由seqidno:1至17组成的组的氨基酸序列的多肽,以及药学上可接受的佐剂、稀释剂、载体、防腐剂、赋形剂、缓冲剂、稳定剂或其组合。[0128]2.项1所述的多肽疫苗,其包括二或更多个多肽,每个多肽包括选自由seqidno:1至17组成的组的不同氨基酸序列。[0129]3.项1所述的多肽疫苗,其包括来自以下组中的至少二组的至少一个多肽:[0130](a)seqidno:1至11;[0131](b)seqidno:12至15;[0132](c)seqidno:16;和[0133](d)seqidno:17。[0134]4.项1所述的多肽疫苗,其包括至少二个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0135]5.项1所述的多肽疫苗,其包括至少四个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0136]6.项1所述的多肽疫苗,其包括至少六个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0137]7.项1所述的多肽疫苗,其包括至少八个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0138]8.项1所述的多肽疫苗,其包括至少十个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0139]9.项1所述的多肽疫苗,其中所述多肽中的一或多个包括,是被所述个体的至少二个hlai类等位基因所限制的cd8 t细胞表位的,冠状病毒科蛋白的片段。[0140]10.项1所述的多肽,其中所述多肽中的一或多个包括,是被所述个体的至少二个hlaii类等位基因所限制的cd4 t细胞表位的,冠状病毒科蛋白的片段。[0141]11.项1所述的多肽,其中所述多肽中的一或多个包括线性b细胞表位。[0142]12.一种在有需要的个体中治疗或预防冠状病毒科感染的方法,所述方法包括向所述个体施用项1的多肽疫苗。[0143]13.项12所述的方法,其中所述冠状病毒科感染是sars-cov-2感染。[0144]公开内容的进一步实施方式——(2)[0145]1.一种免疫原性组合物,其包括(a)至少二个不同的多肽,每个多肽由至少30个氨基酸且不超过60个氨基酸组成,并且包括选自由seqidno:1至17组成的组的氨基酸序列,和(b)增加所述多肽的免疫原性的药学上可接受的化合物。[0146]2.项1的免疫原性组合物,其中所述组合物包括:[0147](a)至少一个不同的多肽,其由至少30个氨基酸且不超过60个氨基酸残基组成,且包括选自seqidno:1至11的氨基酸序列;[0148](b)至少一个不同的多肽,其由至少30个氨基酸且不超过60个氨基酸组成,且包括选自seqidno:12至15的氨基酸序列;[0149](c)至少一个不同的多肽,其由至少30个氨基酸且不超过60个氨基酸组成,且包括选自seqidno:16的氨基酸序列;和[0150](d)至少一个不同的多肽,其由至少30个氨基酸且不超过60个氨基酸组成,且包括选自seqidno:17的氨基酸序列。[0151]3.项1的免疫原性组合物,其中所述不同的氨基酸序列选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组。[0152]4.项3的免疫原性组合物,其中所述氨基酸序列选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组。[0153]5.项1的免疫原性组合物,其中所述组合物包括六个不同的多肽,每个多肽由至少30个氨基酸且不超过60个氨基酸组成,且包括选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组的不同的氨基酸。[0154]6.项1的免疫原性组合物,其中所述组合物包括八个不同的多肽,每个多肽由至少30个氨基酸且不超过60个氨基酸组成,且包括选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组的不同的氨基酸。[0155]7.项1的免疫原性组合物,其中所述组合物包括十个不同的多肽,每个多肽由至少30个氨基酸且不超过60个氨基酸组成,且包括选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组的不同的氨基酸。[0156]8.项1的免疫原性组合物,其中所述至少一个多肽包括,是被个体的至少二个hlai类等位基因所限制的cd8 t细胞表位的,冠状病毒科蛋白质的片段。[0157]9.项1的免疫原性组合物,其中所述至少一个多肽包括,是被个体的至少二个hlaii类等位基因所限制的cd4 t细胞表位的,冠状病毒科蛋白质的片段。[0158]10.项1的免疫原性组合物,其中所述至少一个多肽包括线性b细胞表位。[0159]11.一种在有需要的个体中刺激针对sars-cov-2感染的免疫应答的方法,包括向所述个体施用项1的免疫原性组合物。[0160]12.项1的免疫原性组合物,其中所述组合物包括由根据seqidno:2的序列组成的多肽、由根据seqidno:5的序列组成的多肽、由根据seqidno:7的序列组成的多肽、由根据seqidno:9的序列组成的多肽、由根据seqidno:12的序列组成的多肽、由根据seqidno:13的序列组成的多肽、由根据seqidno:14的序列组成的多肽、由根据seqidno:15的序列组成的多肽、由根据seqidno:16的序列组成的多肽、和由根据seqidno:17的序列组成的多肽。[0161]实施例[0162]实施例1——polypepi1018肽疫苗在结肠直肠癌患者中的安全性[0163]如美国专利10,213,497中所述,已经设计了结肠直肠癌疫苗polypepi1018以优化pepi3 群体。用polypepi1018接种是安全的和耐受良好的。如所预期的,与接种疫苗相关的大多数不良事件是注射部位反应和轻度流感样症状。只有一个三级严重不良事件被记录为可能与治疗有关(非感染性脑炎),然而,安全审查小组和医疗监督员都将该事件归为非相关事件。表2收集了在试验中记录为与接种疫苗相关的不良事件。[0164]表2在11名患者中记录的与接种疫苗相关或可能相关的不良事件。[0165][0166]*凸起的红斑、皮下结节、皮下结节后臂和大腿[0167]实施例2——polypepi1018疫苗在结肠直肠癌患者中的免疫原性[0168]11名患者中的十名有足够的pmbc样本,可以用免疫分析法进行分析。十名患者中的七名预先存在针对至少一个polypepi1018疫苗抗原的免疫应答,并且十名患者中的七名针对至少一个疫苗抗原加强了免疫应答。在十名患者中的八名中,诱导针对至少一个疫苗抗原的重新免疫应答(图1a)。发现了针对所有七个靶抗原的预先存在的免疫应答,这证实了疫苗设计策略和靶抗原选择。所有十名患者都具有疫苗特异性cd4t细胞应答,其中九名在接种后加强应答(图1b)。[0169]八名患者具有从接种前至第12周获得的样品以分析免疫应答的动力学。患者在整个时间过程中表现出不同的动力学模式,其中大多数人在第3周 第6周的合并时间点上出现了一个峰值(图1c),这与病毒再攻击后免疫应答动力学的观察结果相一致。nascimbeni等人在黑猩猩身上发现,针对大多数hcv特异性试验肽的cd8t细胞反应的第一个峰值是在静脉内再攻击后5–6周,以及hcv肝内再攻击后3–4周左右测量到的。(nascimbenietal.,jvirol,77,4781–93.2003)。[0170]除了富集elispot测定之外,我们还进行了直接离体elispot和定量细胞内细胞因子染色(ics)测定,使用流式细胞术作为检测方法来研究效应型t细胞应答。在接种前样品中,离体elispot在九位分析患者中的三个中发现阳性疫苗特异性cd8应答、一个中发现cd4应答。由于接种疫苗,五名患者的cd8反应和七名患者的cd4反应得到了增强或新诱导(表3)。在cd8应答被加强或从头诱导的所有五位患者中,接种也诱导了cd4应答。如通过离体cs所测量的,疫苗特异性cd8和cd4t细胞应答是多功能的,并且接种后功能性cd8和cd4细胞的频率增加(表3)。在cd8t细胞级分中,tnf-α阳性细胞占优势,而在cd4池中检测到的最常见细胞因子是il-2(图2)。[0171]表3离体检测的疫苗特异性效应t细胞应答和肿瘤浸润淋巴细胞。“ ”代表加强:预先存在的响应≥2x。ics百分比显示为疫苗特异性ifn-γ、il-2、tnf-α单或双阳性t细胞频率的总和。nt——未测试[0172][0173][0174]11名患者中有四名有足够的肿瘤样本来分析肿瘤浸润性淋巴细胞(til)。对于四个试验患者中的三个,接种诱导til募集到侵入性边缘和核心肿瘤区域。对于接种后经历临床益处的二个患者,cd8t细胞在核心肿瘤中积累(表3),表明疫苗能够将冷肿瘤转变为热肿瘤。在图3中显示了患者010007活检接种前和接种后(38周)的ihc照片,其中cd8 til增加超过200%。[0175]实施例3——polypepi1018疫苗在结肠直肠癌患者中的效力[0176]在11个患者中,根据recist,三个患者具有客观的肿瘤应答。在只接受单剂的患者中(n=6),到第12周时有1人获得部分反应,2人病情稳定,导致17%的客观应答率(orr)和50%的疾病控制率(dcr)(图4a和4b)。在接受多剂治疗的亚组中,二名患者获得了部分应答,三名患者病情稳定,也就是40%的orr和80%的dcr。单剂和多剂组的中值pfs(包含试验后随访数据)分别为4.5个月和12.2个月(p=0.03)(图4c)。相比之下,在modul试验中5-fu加上贝伐单抗维持疗法与相当的患者群体(n=148)的mpfs为7.39个月。(grotheyetal.,annalsofoncology29,2018)[0177]患者020004在一线化疗期间是部分应答者。在接受单剂疫苗后,他继续缓解,并且接种后6周,他的靶损伤大小减少超过30%(图5a)。这个结果表明,所实现的部分应答可能部分地对诱导阶段有贡献。010004号患者在诱导期病性稳定,在第6周(第一次接种后6周)已经获得部分应答。在进一步接种的整个过程中肿瘤缩小继续,三个靶病变中的二个在第24周完全消失,在第26周进行治愈性手术以除去第三个剩余的病变(图5b)。患者010007在一线治疗中病情稳定后,在接种疫苗期间具有非常缓慢的缓解,具有轻微的斜率,并且仅在其第38周的最后访问时获得部分应答(图5c)。与患者010004类似,靶损伤的缩小也允许对该患者进行根治性手术,并且病理分析未在原发性肿瘤中发现癌细胞并且在肝脏中未发现残余转移。除了后三位患者出现客观应答外,患者010002也必须强调。他取得了稳定的病情(19%的靶病变缩小)作为最佳反应,反应时间为53周(~12个月)。对该患者和具有客观应答的患者的延长的肿瘤应答提示polypepi1018疫苗的抗肿瘤活性。[0178]实施例4pepi测验的回顾性和前瞻性验证[0179]开发pepi测验以预测受试者的t细胞应答。(tokeetal.,journalofclinicaloncology37,2019)pepi试验鉴定了受试者的抗原特异性个人表位(pepi),其结合受试者的至少三个hlai类等位基因。pepi试验的输入是受试者的6个hlai类等位基因和所讨论的抗原的氨基酸序列。用重叠的9聚体肽扫描抗原以鉴定结合受试者hlai类等位基因的肽。pepi试验从表位数据库(epdb)中获得hla–肽对,该数据库是通过纳入结合截止值≤5的肽(iedbpercentilerank)来组装的。根据6项临床试验的免疫原性数据对pepi试验的回顾性验证,确定的pepi引起免疫反应的概率为84%。(表4)。通过对71例癌症和9例hiv感染患者进行的六次临床试验的回顾性分析进行了性能评价研究(验证)。(bagarazzietal.,scitranslmed4,2012,bioleyetal.,clincancerres15,2009,gudmundsdotteretal.,vaccine29,2011,kakimietal.,intjcancer129,2011,valmorietal.,procnatlacadsciusa104,2007,wadaetal.,jimmunother37,2014,yuanetal.,procnatlacadsciusa108,2011)我们通过随机化可获得的患者数据创建了研究组群,并且由于除了数据可获得性之外的原因不排除任何患者。我们没有考虑从原始临床试验中排除,因为我们的研究不是旨在回顾性地分析这些临床试验。我们没有获得任何患者的个人数据。相反,我们使用患者标识符,如同行综述出版物中公开的,以及它们的hla基因型。抗原序列获自公开可获得的蛋白质序列数据库或同等的综述出版物。用标准随机数发生器将源自涉及80名患者的6个临床试验的可用的157个数据集随机化,以创建用于训练和验证研究的二个独立组群。训练和验证组群涉及相同患者群体的不同数据集。48名患者的76个数据集包含在训练组群中,51名患者的81个数据集包含在验证组群中。使用训练数据组,我们确定pepi计数≥1作为预测免疫应答的截止值。[0180]表4pepi试验的回顾性验证(n=81)。通过将pepi试验结果(如果pepi计数≥1为阳性)与在临床试验中通过生物测定法测量的抗原特异性ctl应答比较,获得诊断性能特征:真阳性(a):46,真阴性(d):11,假阳性(b):9,假阴性(c):15。[0181][0182]用于设计polypepi1018疫苗的pepi试验的计算机模拟工具使用符合免疫分析条件的10名患者的免疫原性数据进行了前瞻性验证。70个数据集(7个目标抗原×10个病人)被用来评估pepi试验预测抗原特异性ctl反应的能力。对于每个数据集,确定pepi试验是否能够预测免疫应答。总一致性百分比为64%,阳性预测值为79%,表示具有预测的pepi的患者将产生针对被分析的抗原的cd8t细胞特异性免疫应答的概率(表5)。临床试验数据与回顾性试验结果显著相关(p=0.01)。[0183]表5pepi试验的前瞻性验证。预测的和elispot测量的匹配结果用灰色突出显示。tp:真阳性,tn:真阴性,fp:假阳性,fn:假阴性,opa:整体一致性,ppv:阳性预测值,npv:阴性预测值。[0184][0185]实施例5可能的伴随诊断的可行性的证明(cdx)[0186]oberto-101试验的一个目的是确定一种生物标记,该生物标记除了pepi试验预测的免疫原性之外,还预期预测临床功效。从文献中已经清楚,所测量的免疫应答不能与recist所测量的临床应答直接相关,然而已经发现多抗原免疫应答率和癌症疫苗临床试验的客观应答率之间的相关性(klebanoffetal.,immunologicalreviews239,2011,lorinczetal.,annalsofoncology30,2019)。候选生物标记物可以是agp(具有pepi的抗原),其不仅考虑疫苗中包含的靶抗原的基数,而且考虑每种疫苗抗原的表达概率。受试者的agp计数指示疫苗能够用pepi“命中”的抗原的预期数目。在oberto-101研究的多剂组中,我们研究了agp作为潜在的生物标记物,并发现与肿瘤体积减小和pfs皆相关的趋势(图6a和b)。由于低样本数,不能确定显著性。我们也发现了与所测量的多抗原免疫应答类似的相关模式(图6c和d)。[0187]实施例6——polypepi-scov-2疫苗设计[0188]sars-cov基因组的大小为~30千碱基,像其它冠状病毒一样,编码多个结构和非结构蛋白。结构蛋白包含刺突蛋白(s)、包膜蛋白(e)、膜蛋白(m)、和核壳蛋白(n)。[0189]本文公开的polypepi-scov-2疫苗由一或多个30个氨基酸长的肽组成,该肽能够在全球群体的高比例的个体中诱导阳性的、期望的t细胞(cd8细胞毒性的和cd4辅助细胞二者)应答和b细胞介导的针对一或多个、并优选所有4种结构病毒抗原的抗体应答。[0190]2020年3月28日,从ncbi数据库(https://www.ncbi.nm.nih.gov/genome/genomes/86693)中共下载了19个covid-19的全基因组序列。登录号id如下:nc_045512.2、mn938384.1、mn975262.1、mn985325.1、mn988713.1、mn994467.1、mn994468.1、mn997409.1、mn988668.1、mn988669.1、mn996527.1、mn996528.1、mn996529.1、mn996530.1、mn996531.1、mt135041.1、mt135043.1、mt027063.1、mt027062.1。[0191]第一个id代表genbank参考序列。将翻译的编码序列的四个结构蛋白序列(表面糖蛋白、包膜蛋白、膜糖蛋白、核衣壳磷蛋白)进行比对,并与多序列比对进行比较。在19个序列中,15个是完全相同的。然而,我们在4个核壳蛋白中获得了单个氨基酸变化。这些替换如下:mn988713.1:核衣壳194s→x,mt135043.1:核衣壳343d→v,mt027063.1:核衣壳194s→l,mt027062.1:核衣壳194s→l。这些变化都不影响本疫苗多肽中被选为目标的表位。[0192]从目前已知的sars-cov-2结构蛋白的病毒抗原序列的保守区中选择十七个肽片段。选择片段以使模型群体中的多hlai类结合pepi3 和多hlaii类结合pepi4 ,即共享的个人表位,最大化。这些多肽还被设计成包含线性b细胞表位。具体地,选择在模型群体中最高比例受试者中的四个靶抗原pepi2 的保守区中的9聚体序列。这些9聚体被扩展到包含靶抗原的保守序列中的附近线性b细胞表位。然后选择同时包含9聚体“最佳epi”和线性b细胞表位二者的靶抗原的30聚体片段,以使模型群体中30聚体片段中具有hlaii类结合的pepi4 的受试者比例最大化。该模型群体包括从骨髓移植生物库获得的~16,000个hla基因分型的受试者,具有来自16个不同族群每个的约1,000个受试者。在模型群体中比例最高的受试者中,选定的30聚体肽片段和hlai类结合表位是pepi3 ,hlaii类结合表位是pepi4 ,其序列见表6a。[0193]表6apolypepi-scov-2肽序列列表。粗体/斜体:9聚体hlai最佳epi序列,下划线:15聚体最佳epi序列。[0194][0195]*含有肽的b细胞表位,b细胞表位列于表6b[0196]表6b线性b细胞表位[0197][0198][0199]参考文献:preliminaryidentificationofpotentialvaccinetargetsforthecovid-19coronavirus(sars-cov-2)basedonsars-covimmunologicalstudies。表4。来自s的sars-cov衍生的线性b细胞表位(23;其中20个位于亚基s2中)和n(22)蛋白在sars-cov-2中相同(总共45个表位)。[0200]表6c最佳hlai和hlaiipepi的辅助表:[0201][0202]实施例7——polypepi-scov-2与现有技术疫苗的比较[0203]如文章“preliminaryidentificationofpotentialvaccinetargetsforthecovid-19coronavirus(sars-cov-2)basedonsars-covimmunologicalstudies”(ahmed等人)中所提示的,我们基于其中鉴定的靶标建立了疫苗的可能效力(免疫原性)的模型。将结果与本文所述的选择的polypepi-scov-2疫苗肽进行比较。[0204]sfahmed等人鉴定了与19个hlai等位基因相关的61个t细胞表位,以提供基于全局等位基因频率的96.29%的估计的累积群体覆盖率。(ahmedetal.viruses,12(3).2020)表7所示的下列t细胞表位被认为是疫苗的潜在靶(该文章的表3;61个中的2个仅是8聚体表位,我们从模拟中排除)。[0205]表7采用sfahmed等人:sars-cov衍生的刺突(s)和核壳(n)蛋白t细胞表位的集合(从阳性mhc结合测定获得),其在sars-cov-2中是相同的,并且最大化估计的总体群体覆盖。[0206][0207][0208][0209][0210]ahmed等人提出,通过为每个列出的hla等位基因(即19个序列)选择至少一个表位,可以获得估计的最大群体覆盖率。因此,我们从该t细胞表位组中进行随机选择,为每个hla等位基因选择一个表位(完全如作者所提示的)。因为这些是混杂的hla结合表位,因此有时我们为多于一个hla等位基因选择相同的表位。将此重复30次,并将所选表位与针对polypepi-scov-2(seqidno:2、5、7、9、12、13、14、15、16、17)所选的10种肽进行比较。对从骨髓移植生物库获得的我们~16,000个hla基因分型受试者数据库进行计算机模拟比较。我们的数据库含有来自16个族群的数据(每族群约1,000个受试者)。我们计算了具有针对至少一个表位的cd8 免疫应答的受试者的比例。polypepi-scov-2的全球覆盖率为99.8%,相比之下,模拟疫苗(随机表位选择)的平均覆盖率为61%(±9.9%),一些族群(如高加索人)获得的保护低于其他群体(如日裔)(图7)。[0211]建立了另一种特殊(不实际)的情况,其中ahmed等人(n=59)中列出的所有t细胞表位都被选入疫苗。在这种情况下,全球覆盖率增加至88%,但是仍然没有达到polypepi-scov-2的水平(图8)。这表明在族群之间也均匀覆盖表位疫苗。[0212]我们还模拟了polypepi-scov-2疫苗(选择相同的10种肽)除了诱导cd8应答之外还诱导hlaii类限制性cd4应答(hlaii类pepi)的能力(图9)。在每个种族组群中,至少97%的受试者引发针对polypepi-scov-2疫苗的至少2种肽的cd8和cd4t细胞皆应答。[0213]实施例8——polypepi-scov-2和现有技术肽疫苗的免疫原性表位的数目的比较[0214]基于先前来自ahmed等人的数据集,我们计算了模型群体中每个受试者的免疫原性表位的数目。该数目的分布显示了疫苗对潜在突变的效力。[0215]图10显示61%(±9.9%)的受试者具有针对至少一个疫苗表位的免疫应答,但仅25%(±10.4%)的受试者具有针对至少2个来自19的表位的应答。这意味着,如果病毒在一个特定表位上突变,则另一个表位仍可以产生免疫应答(对于一部分受试者)。相反地,99%的用polypepi-scov-2处理的模型群体具有针对至少2个表位的应答。对于至少3个目标表位,差距甚至更大(polypepi-scov-2为96%,相对epitopevaccine为6%)。[0216]对于含有所有59个表位的疫苗,情况将稍微更好:69%的受试者可具有针对2个或更多个表位的免疫应答(图11),但与polypepi-scov-2疫苗(10个肽)相比,这仍然是群体中较小的比例。[0217]实施例9[0218]covid-19感染和预测的建模警告快速进化,这可能破坏接种疫苗对抗和治疗感染的尝试。迫切需要设计在未来几年中如何开展新型β冠状病毒sars-cov-2的传播。这些动力学将取决于季节性、免疫的持续时间、和针对/来自其它人冠状病毒的交叉免疫的强度。使用来自美国的数据,发明人测量了这些因素如何影响人冠状病毒hcov-oc43和hcov-hku1的传播。(kissler等人,2020,https://doi.org/10.1101/2020.03.04.20031112)。本文所述的疫苗肽和组合物的设计对于快速病毒进化是稳健的,并且通过选择多个免疫原性但保守的序列,优选地衍生自多个结构蛋白,覆盖全球群体。[0219]可以预料,随着病毒继续进化和收集更多的数据,将观察到另外的突变。这样的突变将不影响本文所述的多肽和多肽疫苗的整体覆盖,条件是突变发生在鉴定的表位区域之外。即使突变确实发生在任何选定的表位区域内,剩余的免疫原性表位仍然提供了针对病毒的强大保护,因为大多数受试者将保留病毒特异性记忆t细胞克隆的广泛所有组成成分,例如,对于包括seqidno:2,5,7,9,12,13,14,15,16、和17的多肽的十肽疫苗,94%的患者具有针对至少3个疫苗肽的免疫应答,并且分别针对4和5个肽具有85%和71%的免疫应答。[0220]实施例10[0221]概述[0222]本实施例描述了针对sars-cov-2的全肽疫苗的开发,其解决了不同个体的免疫应答中的异质性和感染病毒的潜在异质性的双重挑战。在本实施例中,“polypepi-scov-2”是含有九个30聚体肽的多肽疫苗,其衍生自下文所述的sars-cov-2病毒的所有四个主要结构蛋白。疫苗肽的选择是基于它们作为hlai类和ii类个人表位(pepi)的频率,这些表位限制在433名不同种族的受试者的计算机模拟组群中的多个自体hla等位基因。与montanideisa51vg佐剂一起施用的polypepi-scov-2疫苗在皮下注射到balb/c和cd34转基因小鼠中后,产生针对病毒的所有四个结构蛋白以及结合抗体的强cd8 和cd4 t细胞应答。此外,在症状发作后1–5个月,在研究的17个无症状/轻度covid-19康复期血液的每一个中离体检测到polypepi-scov-2特异性、多功能cd8 和cd4 t细胞。用于从covid-19回收的polypepi-scov-2特异性t细胞库极其多样化:供体具有针对sars-cov-2蛋白的平均七个不同的肽特异性t细胞;87%的供体具有针对至少三个sars-cov-2蛋白的多个靶而53%的供体具有针对所有四个蛋白的多个靶。此外,基于康复供体的完整i类hla基因型确定的pepi被验证,准确率为84%,以预测对个体测量的pepi特异性cd8 t细胞应答。将上述发现外推至具有16个不同族群(每个族群n=1,000)的16,000个hla基因分型个体的美国骨髓供体组群,表明在一般群体中的polypepi-scov-2疫苗接种将可能在98%的,与种族无关的,包含黑人、亚裔、和少数种族(bame)组群的个体中,引发广泛的多抗原cd8 和cd4 t细胞应答。与montanideisa51vg一起施用的polypepi-scov-2在皮下注射到模拟人免疫系统的balb/c和hcd34 转基因小鼠中后产生针对所有代表性蛋白质以及结合抗体的稳健、th1偏态的cd8 和cd4 t细胞应答。[0223]介绍[0224]由新型冠状病毒sars-cov-2引起的流行在2019年12月爆发后仍在发展。根据世界卫生组织(who),至少三分之二的处于临床开发中的候选疫苗被设计为主要产生针对病毒刺突(s)蛋白的中和抗体(1),但是从sars-cov和mers流行学以及covid-19康复期学到的教训表明了这种疫苗设计策略的潜在挑战。(2,3)潜在的问题是双重的:降低抗体水平和仅针对刺突蛋白的无效t细胞应答产生。[0225]感染了2003年流行的sars-cov病毒和2012年流行的mers病毒的患者通常具有短暂的(检测仅至多3–6年)体液免疫。甚至,由感冒冠状病毒的低风险实验感染产生的抗体在1年内下降,并且不能保护免受再攻击。(4,5)类似地,对于sars-cov-2,与sars-cov-2病毒感染的自然过程相关的免疫应答表明抗刺突igg抗体应答通常是弱的(除了幸运地较不频繁的严重病例),并且它们的持久性在大多数情况下持续高达3个月,或者在这段时间内下降高达70%。(6)此外,2–9%的个体甚至在感染sars-cov-2后2个月没有血清转化,(7)表明个体使用另一分支的适应性免疫系统t细胞达到免疫。事实上,可以得出结论,事实上所有有sars-cov-2感染史的对象都产生了针对病毒的t细胞应答,包含血清阴性和患有严重疾病的对象。(2,8–10)t细胞应答是多样的,即针对病毒的整个抗原库,而不太受刺突蛋白支配。具体地,几项研究报道了尽管是最大的结构蛋白,但刺突特异性t细胞应答仅占天然感染引起的cd4 和cd8 t细胞总数的25–27%。此外,这种多样性与无症状/轻度疾病有关,因为恢复的患者具有更多的针对膜(m)和核蛋白(n)蛋白而不是s蛋白的cd8 t细胞,而t细胞强度和多样性不随疾病严重程度而增加,如mers、sars-cov-1和sars-cov-2所证实的。(6,9,11,12)实际上,在covid-19患者中,低cd8 t细胞计数是死亡风险较高的预示,而患有严重疾病的患者或死亡的患者具有耗竭的t细胞。(2,13)认为感染后较早时间可检测的病毒特异性cd8 t细胞应答导致较低的病毒负荷和因此较低的抗体水平,解释了为什么这些患者具有更有利的结果。(12)为了支持这一点,最近报道了在病毒暴露后不久和在任何抗体检测之前对sars-cov-2特异性t细胞受体进行作图是可能的。[0226]这些观察结果还表明,在感染(通过接种)之前获得数量增加的多种病毒特异性记忆t细胞,可能有助于sars-cov-2感染早期的病毒和病毒储库的消除。(9,15)这些预期得到了动物攻击研究的支持,这些研究表明,重新激活的t细胞为sars-cov-1的致死剂量感染提供了保护。值得注意的是,在sars-cov-1病毒康复17年后,23名接受试验的患者中有23名报告了针对sars-cov-1病毒的n蛋白的记忆t细胞。(9)其它报道也支持由冠状病毒感染引起的记忆t细胞的持久性。(18–23)[0227]因此,在临床开发中旨在产生针对病毒s蛋白的t细胞应答的候选疫苗将可能仅产生恢复期免疫应答的一部分,因此不太可能诱导稳健的记忆t细胞应答。疫苗技术使用完整病毒,多个大蛋白理论上可以解决与缺乏多样性相关的问题。然而,这些具有着包括不必要的抗原负载的限制,这不必要的抗原负载不仅对保护性免疫应答贡献很小、而且通过诱导变应原性和/或反应原性应答而使情况复杂化。类似地,编码靶抗原的复制缺陷型病毒构建体可以引发针对病毒载体的非特异性免疫应答,尤其是在重复剂量下。[0228]肽疫苗是一种替代的亚单位疫苗策略,其依赖于短肽片段、表位的使用,能够诱导阳性的、期望的t细胞和b细胞介导的免疫应答。[0229]然而,困扰肽疫苗设计的核心问题是每个人具有独特的免疫应答谱。实际上,对于sars-cov-2,疾病过程根据由不同种族代表的遗传多样性而变化,尤其是黑人、亚裔和少数民族(bame)族群;然而,其原因还未被很好地理解。(25,26)遗传多样性可通过人类白细胞抗原(hla)等位基因的遗传变异来捕获,这等位基因是病毒抗原(表位)呈递途径的关键组分,其引发能够识别并杀死体内癌症或感染细胞的细胞毒性t细胞(ctl)。为了在设计针对sars-cov-2的整体疫苗中捕获这种异质性,已经广泛使用基于常见人hla等位基因的病毒抗原表位预测。(27)然而,实际上,这些表位作图研究在验证的t细胞应答方面具有低产量。例如,在一项研究中,测试了100个预测为10个最普遍hlai类等位基因的sars-cov-2衍生表位,仅有12个被证实为显性表位,即被》50%的covid-19供体cd8 t细胞识别。这与在本领域中对于若干感染性和癌症疫苗临床试验以及对于个性化突变的基于新抗原的表位的相对低的确认水平观察到的免疫应答率一致。[0230]为了克服肽疫苗设计的这些限制,我们数字化地开发了polypepi-scov-2,其使用具有完整hla基因型的个体的种族差异的计算机模拟人类组群,而不是使用单一hla等位基因。选择了多个所谓的个人表位(pepi),不仅被每个个体的一个而是被多个自身hla等位基因所限制,而且还在种族多样性群体中的大部分受试者之间共享。值得注意的是,这个计算机模拟人类组群与pepi概念一起先前回顾性地预测79个疫苗临床试验的免疫应答率,以及在转移性结肠直肠癌患者中进行的临床试验中我们的polypepi1018癌症疫苗的显著免疫原性(80%cd8 t细胞应答针对六个抗原中的至少三个)。(31–33)由为乳腺癌患者制备的个性化多肽混合物中的pepi产生的cd8 t细胞应答证明是长期持续的,因为在最后一次针对四个肿瘤抗原接种后14个月(436天)检测到它们。[0231]与sars-cov-2在自然感染过程中明显的长期记忆t细胞形成能力一致,本发明的多肽疫苗被设计为(1)通过靶向sars-cov-2的所有四个结构蛋白,在每个对象中诱导稳健的和广泛的免疫应答;(2)通过确保每个患者中的多个免疫原性靶来解决和克服潜在的病毒进化效应;且(3)通过肽的个人表位覆盖解决人类种族的不同敏感性。本文描述了针对covid-19的候选疫苗的设计和临床前表征。在二个小鼠模型中证实了免疫原性和耐受性,导致诱导了由第二剂加强的强cd4 和cd8 t细胞应答,以及体液应答。在康复期的covid-19血样中,在所有受试者中检测到抗所有肽的疫苗特异性免疫细胞,代表引起感染恢复的sars-cov-2诱导的免疫组库的重要组分。与由死亡或减毒病毒和重组蛋白制成的传统疫苗相比,肽疫苗是安全和经济的技术。以多公斤规模生产合成肽相对便宜,并且比mrna生产肯定更成熟。该技术不仅能够鉴定特定疾病/病原体的抗原靶标,而且更重要的是,能够计算确定大组群中个体的免疫系统能够应答的抗原。[0232]材料和方法[0233]患者/供体[0234]根据其sars-cov-1或sars-cov-2感染的临床史征募供体。血样在尼德兰独立医学研究中心以批准的方案(nl57912.075.16)从康复个体(n=15)收集,或由peptc疫苗有限公司收集(n=2)。来自未暴露个体(n=5)的血清和pbmc样品在2018年前收集并由nexelis-imxp(比利时)提供。所有供体提供书面知情同意书。该研究是按照赫尔辛基宣言进行的。症状发作后17–148天获得covid-19康复病人(n=17;16名无症状/轻微疾病和一名严重疾病)的血样。令人惊奇的是,在健康供体中发现了一个阳性igm抗体应答,其被排除在进一步分析之外。受试者的人口统计学和基线信息提供于表8中。[0235]来自尼德兰的康复期供体患者的hla基因分型是由imgm实验室有限公司(德国martinsried)使用下一代测序技术完成的。二个peptc供体的hla基因分型是由实验室公司(美国弗吉尼亚州burlington的labcorp)的颊拭子使用下一代测序(illumina)进行的,而hla等位基因解释是基于imgt/hla数据库版本3.38.0。来自尼德兰的康复期供体患者的hla基因分型是由imgm实验室有限公司(德国martinsried)使用下一代测序技术完成的。该组群使用总共46个不同的hlai类等位基因(15个hla-a*、18个hla-b*和13个hla-c*)和35个不同的hlaii类等位基因(14个drb1、12个dqb1和9个dpb1)。受试者的hla基因型数据提供于表8b中。[0236]动物[0237]cd34 转基因人源化小鼠(hu小鼠)。使用从人脐带血分离的造血干细胞(cd34 )对雌性nod/shi-scid/il-2rγ无效免疫缺陷小鼠(charlesriverlaboratories,法国)进行人源化。在研究期间仅使用人源化率(hcd45/总cd45)》50%的小鼠。用20–23周龄的雌性小鼠进行实验。[0238]balb/c小鼠。用6–8周龄的雌性balb/c小鼠(法国janvier)进行实验。[0239]表8供体基线和人口统计信息。所有供体都是高加索人种,具有轻度/无症状疾病且未住院治疗(除了一个用*标记的)。s/co,样品/对照比率;根据制造商的说明书确定值,并且试验结果解释为s/co《0.9为阴性、若s/co=0.9–1.1则不确定、且若s/co》1.1则为阳性。coi,截止指数;根据制造商的说明书确定数值,并且试验结果解释为coi《0.9为阴性、coi为0.9–1.1为不确定、且若coi》1.1为阳性。na,数据不可用。斜体,阴性或不确定的数值。**主诉:a,咳嗽;b,喉咙痛;c,发烧;d,气短;e,胃/肠道不适;f,胸痛;g,眼睛痛;h,嗅觉或味觉丧失;i,头痛;j,疲劳;k,其他主诉(imxp00759为肺栓塞和心脏停搏;腿痛、手臂痛、肌肉痛、眼睛痛)。[0240][0241]表8b恢复期供者的完全hla基因型[0242][0243][0244]疫苗设计[0245]筛选sars-cov-2结构蛋白(s、n、m、e),并在多步骤过程中选择九个不同的30聚体肽。首先,进行序列多样性分析(如ncbi数据库中截至2020年3月28日)。(35)登录号id如下:nc_045512.2、mn938384.1、mn975262.1、mn985325.1、mn988713.1、mn994467.1、mn994468.1、mn997409.1、mn988668.1、mn988669.1、mn996527.1、mn996528.1、mn996529.1、mn996530.1、mn996531.1、mt135041.1、mt135043.1、mt027063.1、和mt027062.1。第一个(粗体)id代表genbank参考序列。然后,使用多序列比对(英国embl-ebi的clustalomega)比对和比较翻译的四个结构蛋白序列的编码序列。(36)在19个序列中,15个是相同的;然而,在四个n蛋白序列中发生单个氨基酸变化:mn988713.1,n194s→x;mt135043.1,n343d→v;mt027063.1,n194s→l;mt027062.1,n194s→l。所得氨基酸取代仅影响n蛋白序列的二个位置(aa194和343),都不是发生在已被选作疫苗开发靶标的表位中(seqidno:2、5、9、和12–17)。在polypepi-scov-2疫苗中,仅涉及十三个报道的病毒s蛋白中的单字母变化(d614g,s943p,l5f,l8v,v367f,g476s,v483a,h49y,y145h/del,q239k,a831v,d839y/n/e,p1263l)中的一个(h49y),但该变体的流行在后来的病毒分离物中降低。(37)关于肽选择的进一步细节在结果部分提供,并且九个选择的30聚体肽的所得组成在表9中显示。[0246]与人冠状病毒株的交叉反应性[0247]将polypepi-scov-2疫苗的序列与sars-cov、mers-cov和普通(季节性)人冠状病毒株的序列进行比较,以揭示可能的交叉反应区域。根据疾病控制和预防中心(cdc),人类群体中常见的冠状病毒感染由四个冠状病毒组引起:α冠状病毒229e和nl63、以及β冠状病毒oc43和hku1。(38)结构蛋白的成对比对还使用uniprot(39)进行,具有以下参考序列id:229e:p15423(s)、p15130(n)、p19741(e)、p15422(m);nl63:q6q1s2(s)、q6q1r8(n)、q6q1s0(e)、q6q1r9(m);oc43:p36334(s)、p33469(n)、q04854(e)、q01455(m);hku1(分离株n1):q5mqd0(s)、q5mqc6(n)、q5mqc8(e)、q5mqc7(m)。此外,冠状病毒菌株与构成polypepi-scov-2疫苗的9个30聚体肽进行了比对。对于表位的最低要求,筛选八个氨基酸长的区域(滑动窗)作为负责潜在交叉反应性的区域。此外,在分析期间记录和报告较短(和较长)长度匹配的连续肽片段。[0248]计算机模拟人类组群[0249]模型群体是433个个体的组群,代表世界范围内的几个族群,对于它们可获得完整的hlai类基因型(2×hla-a,2×hla-b,2×hla-c)。模型群体由90名约鲁巴非裔(yri)、90名欧裔(ceu)、45名华裔(chb)、45名日裔(jpt)、67名混合种族(美国、加拿大、澳大利亚、新西兰)、和96名来自hiv数据库(mix)的对象组成。(40–43)使用pcr技术、affymetrix6.0和illumina1.0百万snpmassarray,以及通过参照链介导的构象分析(rsca)或基于测序的分型(sbt)对六个hla基因进行高分辨hla分型,确定hla基因型。(44–46)以前已经报道了模型群体的表征。(31)该组群使用总共152个不同的hlai类等位基因(49个hla-a*、71个hla-b*和32个hla-c*),代表97.4%的当前全球常见、中间和有据可查的(ciwd)等位基因,并充分代表主要种族(发行于2020的数据库3.0)(表8a)(hurley等人,2020)。模型群体的a*、b*、c*等位基因的频率与ciwd数据库中大于8百万个hla分型对象的记录频率相关(分别是r=0.943、0.869、0.942,p《0.00001)(图24)。[0250]表8a在模型群体中呈现的等位基因的hla覆盖度。非裔/非裔美国人(afa)、亚裔/太平洋岛(api)、欧裔/欧洲血统(euro)、中东/非洲北海岸(mena)、南美或中美洲/西语裔/拉丁裔(his)、美国原住民群体(nam)、未知/未询问/多祖先/其他(unk)。ciwd3.0:常见(10,000中≥1)、中间(100,000中≥1)和文献充分记载(≥5次出现)的hla数据库3.0(2020年发布)。与图24相关。[0251][0252]从dbmhc数据库(47)获得了356个个体的第二模型组群,这些个体具有四位等位基因分辨率的已表征ii类hla基因型(2×hla-drb、2×hla-dp、和2×hla-dq),dbmhc数据库是由国家生物技术信息中心(ncbi)操作的在线可用储存库,开发用于评价cdna或基因组序列的等位基因组成。在全世界许多国家中对广泛的种族进行采样。总之,该数据库中的356名受试者具有足够分辨率的hlaii类基因型数据(2×hla-drb、2×hla-dp、和2×hla-dq,具有至少四位分辨率)。hla基因分型通过sbt进行。[0253]大,美国组群(n=16,000)[0254]通过从来自国家骨髓供体计划(nmdp)的16个族群(500名男性和500名女性)中的每一个获得1,000个供体,创建包括来自16,000个个体的数据库。(48)16个族群是:非裔、非裔美国人、亚洲太平洋岛裔、菲律宾裔、黑加勒比海人、高加索人、华裔、西语裔、日裔、韩裔、美国印地安原住民、南亚裔、越南裔、美国、中东/非洲北海岸、夏威夷裔、和其它太平洋岛裔。hla基因分型通过nmdp募集实验室使用序列特异性寡核苷酸(sso)和序列特异性引物(ssp)方法进行,平均“分型分辨率得分”》0.7。(49)[0255]肽和polypepi-scov-2疫苗制剂[0256]9聚体(s2、s5、s9、n1、n2、n3、n4、e1、m1)和30聚体(s2、s5、s7、n1、n2、n3、n4、e1、m1)肽由intavispeptideservicesgmbh&co.kg(德国tübingen)和pepscan(尼德兰lelystad)使用固相肽合成来生产。表9中提供了9聚体试验肽(表9,粗体)和30聚体疫苗肽二者的氨基酸序列。通过将等质量的九个30聚体肽溶解于dmso中以达到1mg/ml的浓度,然后用纯水稀释至0.2mg/ml的终浓度并冷冻储存直至使用,来制备用于动物研究的肽疫苗。按照制造商提供的标准双注射器方案,通过乳化等体积的解冻的肽混合溶液和montanideisa51vg佐剂(法国巴黎seppic)制备准备注射的疫苗制剂。[0257]表位预测和分析[0258]使用基于免疫表位数据库(iedb)的表位预测方法预测每个个体中的≥3个hlai类等位基因结合表位(pepi)。用重叠的9聚体和15聚体肽扫描抗原以鉴定结合受试者hlai类等位基因的肽。用已经通过使用直接结合测定法进行实验表征的427个hla–肽对的内部集合(327个结合和100个未结合hla–表位对)验证选择参数。对于真实hla等位基因–表位对的预测,特异性和灵敏度结果皆为93%。ii类hla表位的预测是通过netmhcpan(2.4)预测算法进行的,每个个体≥4个ii类hla结合表位被定义为hlaii类pepi。[0259]临床前小鼠研究设计[0260]36只hu小鼠和36只balb/c小鼠接受在第0天和第14天皮下施用的polypepi-scov-2疫苗(0.66mg/kg/肽的200μl溶液;n=18)或乳化在montanideisa51vg佐剂中的20%dmso/水(200μl载体;n=18);随访期在第28天结束,分析第14、21、和28天的样品(每组群n=6)。研究在transcurebioservicefacility(法国archiamps)进行。每天监测小鼠的意外痛苦迹象。每周通过监测被毛(0–2分)、运动(0–3分)、活动(0–3分)、苍白(0–2分)、和体重(0–3分)进行完全临床评分;正常状况评分为0。[0261]本研究中描述的所有程序都经过当地伦理委员会(celeag)的审查和批准,并由法国研究部验证。通过小鼠脾细胞的离体elispot和细胞内细胞因子染色试验(ics)评估接种诱导的t细胞应答(下文详述)。通过测量血浆样品中的总igg研究抗体应答(详述如下)。[0262]elispot/fluorospot分析[0263]用于动物研究的离体elispot测定如下进行。使用2×105个被刺激了20小时/肽(10μg/ml,终浓度)的脾细胞鉴定产生ifn-γ的t细胞。用9聚体肽(四个n特异性肽的池:n池(n1、n2、n3、n4),三个s特异性肽的池:s池(s2、s5、s9),e蛋白衍生肽:e1,或m蛋白衍生肽:mm1)),或用与9聚体相同方式聚类的30聚体肽(包括肽n1、n2、n3、和n4的n池,包括肽s2、s5和s9的s池,以及单独的肽e1和m1),处理脾细胞。按照生产商的说明书,使用mabtech的人ifn-γelispotpro试剂盒(alp;ref3321-4apt-2)对hu小鼠组群进行、并使用mabtech的小鼠ifn-γelispotpro试剂盒(alp;ref3321-4apt-10)对balb/c小鼠组群进行elispot测定。从每个数据点中减去未刺激(dmso)测定对照背景斑点形成单位(sfu),然后计算δsfu(dsfu)。pma/离子霉素(invitrogen)用作阳性对照。[0264]用于康复供体试验的离体fluorospot测定通过nexelis-imxp(比利时)如下进行:用rpmi-10%fbs封闭ifn-γ/il-2fluorospot板,然后将肽(5μg/ml终浓度)或肽池(5μg/ml每肽终浓度)加入到相关孔中。从低温储存中回收pbmc并在培养基中解冻。然后,将200,000个pbmc细胞/孔三重复(刺激条件)或六重复(参考条件)铺板,并在培养前于37℃,5%co2温育过夜。根据制造商的建议进行fluorospot板的显影。除去细胞后,将稀释在含0.1%bsa的pbs中的检测抗体加入孔中,并将fluorospot板在室温下温育2小时。在使用mabtechiristm自动化fluorospot读数器读出之前,将fluorospot板倒空并在室温下避光干燥24小时。所有数据用mabtechiristm读数器获得,并用mabtechapextm软件分析。未刺激(dmso)阴性对照、cef阳性对照(来自cmv、ebv和流感病毒的t细胞表位,瑞典mabtech)、和商业sars-cov-2肽库(sars-cov-2snmo限定的肽池(3622-1)——瑞典mabtech),被包含作为测定对照。在减去非刺激对照(dsfu)后,当试验结果高于dmso阴性对照时,离体fluorospot结果被认为是阳性的。[0265]用于康复期供体检测的富集elispot检测由nexelis-imxp(比利时)进行,具体方法如下:从低温储存中回收pbmc并在培养基中解冻。在肽池存在下(每肽5μg/ml)以4,000,000个细胞/24孔接种pbmc,并在37℃、5%co2下温育7天。在培养的第1天和第4天,更新培养基,并分别补充5ng/ml的il-7或5ng/ml的il-7和4ng/ml的il-2(r&dsystems)。培养7天后,收获pbmc并静置16小时,然后使用trypanblue溶液0.4%(vwr)和cellometerk2荧光生存细胞计数器(nexcelom)对静置的pbmc计数,并以200,000细胞/孔在ifn-γ/粒酶–b/tnf-αfluorospot板(mabtech)上接种在含有10%人血清hi、2mml-谷氨酰胺、50μg/ml庆大霉素、和100μmβ-me的rpmi1640上,至含有肽(5μg/ml)或肽池(每肽5μg/ml)的相关fluorospot孔中。在培养前,将fluorospot板在37℃,5%co2下温育过夜。所有数据用mabtechiristm读数器获得,并用mabtechapextm软件分析。dmso、仅培养基、商业的covid肽库(sars-cov-2snmo定义的肽池[3622-1]——mabtech)、和cef作为测定对照,以1μg/ml的浓度包含在内。在减去背景(dsfu)后,阳性标准为》1.5倍的未刺激对照。[0266]细胞内细胞因子染色(ics)测定[0267]临床前小鼠研究的离体ics测定如下进行:刺激20小时后,从elispot板上取下脾细胞,转移到常规的96孔平底板上,根据制造商的建议,用bdgolgistoptm培养4小时。使用bdcytofix/cytopermplus试剂盒与bdgolgistoptm蛋白转运抑制剂(含有莫能星;目录号554715)按照制造商的说明进行流式细胞术。流式细胞术分析和细胞因子分布测定在attunenxt流式细胞仪(lifetechnologies)上进行。分析了2×105个细胞,门控cd45 、cd3 、cd4 或cd8 t细胞。低于25的计数被评价为0。斑点计数≥25是通过减去未刺激的(dmso)对照而校正的背景。pma/离子霉素(invitrogen)用作阳性对照。作为测定对照,使用mann-whitney检验比较每个细胞因子的阴性对照(未刺激)和阳性对照(pma/伊屋诺霉素)。当确定对照之间的统计学差异时,分析对应于其它刺激条件的值。下列染色剂用于hu小鼠组群:mab11502932(biolegend),mp4-25d2500836(biolegend),4s.b3502536(biolegend),hi30304044(biolegend),sk7344842(biolegend),jes6-5h4503806(biolegend),vit4130-113-218(miltenyi),jes1-39d10500904(biolegend),sk1344744(biolegend),jes10-5a2501914(biolegend),jes3-19f1554707(bd),以及na564997(bd)。下列染色用于balb/c小鼠组群:11b11562915(bd)、mp6-xt22506339(biomension)、xmg1.2505840(biomension)、30-f11103151(biomension)、145-2c11100355(biomension)、jes6-5h4503806(biomension)、gk1.5100762(biomension)、jes1-39d10500904(biomension)、53-6.7100762(biomension)、ebio13a25-7133-82(thermoscientific)、jess-16e3505010(biomension)、和na564997(bd)。[0268]用nexelis-imxp(比利时)进行恢复期供体检测的离体ics分析。简言之,在解冻200,000个pbmc细胞/孔后,在肽(5μg/ml)或肽池(每肽5μg/ml)的存在下,将pbmc接种在无菌圆底96孔板中,该无菌圆底96孔板中的rpmi总量为10%人血清hi、2mml-谷氨酰胺、50μg/ml庆大霉素、和100μm2-me。温育2小时后,将bdgolgiplugtm(bdbiosciences)以培养基中1μl/ml的浓度加入96孔板中。温育10小时后,将板离心(800g,3分钟,8℃)并在37℃温育10分钟,并将zombienir生存力染料(biolegne)加入到每个孔中。将板在避光的条件下于室温温育15分钟。温育后,每孔加入pbs/0.1%bsa,并将板离心(800g,3分钟,8℃)。此后,细胞在fcr封闭试剂中于4℃温育5分钟,然后将染色混合物(含有抗cd3、biolegen、抗cd4和抗cd8抗体;bdbiosciences)加入每个孔中。在4℃下孵育30分钟后,洗涤并离心(800g,3分钟,8℃),根据制造商的建议(bdbiosciences)将细胞透化并固定。固定后,将细胞因子染色混合物(含有抗ifn-γ、抗il-2、抗il-4、抗il-10和抗tnf-α抗体,biolegen)加入每个孔中。将板在4℃下温育30分钟,然后在获得之前洗涤二次。所有流式细胞术数据用lsrfortessatmx-20获得,并使用flowjov10软件分析。从使用试验肽或池获得的每个数据点中减去dmso阴性对照。[0269]抗体elisa[0270]根据制造商的说明书,对balc/c组群使用igg(总)小鼠未包被elisa试剂盒(invitrogen,#88-50400-22),并对用于hu小鼠组群使用igg(总)人未包被elisa试剂盒(invitrogen,#88-50550-22),进行用于小鼠研究的elisa,以定量测量血浆样品中总小鼠igg的产生。使用在第14、21、和28天收获的样品进行分析(每组每时间点n=6)。在epoch微板读数器(biotech)上读取吸光度,并使用gen5软件分析。[0271]康复供体试验的elisas通过mikromikomedkft(匈牙利布达佩斯)进行,根据生产商的说明书(意大利diaprodiagnosticbiobesr.l),使用diaprocovid-19igm酶免疫测定来确定人血清和血浆中的抗covid-19的igm抗体,使用diaprocovid-19igg酶免疫测定来确定人血清和血浆中的抗covid-19的igg抗体,并使用diaprocovid-19iga酶免疫测定来确定人血清和血浆中的抗covid-19的iga抗体。为了确定总的n特异性ig抗体,根据制造商的说明书,使用用于定性检测抗sars-cov-2的抗体(包含igg)的roche抗sars-cov-2免疫测定法,用cobase411分析仪(瑞士罗氏的disksystem)。进行euroimmunelisa测定以通过独立的医学研究中心确定康复期供体中的s1特异性igg水平。抗sars-cov-2elisa板用sars-cov-2的抗体结合的sars-cov-2的重组s1结构蛋白包被。选择与其它冠状病毒,特别是sars-cov-1同源性相对较低的抗原。按照生产商的说明书进行免疫测定。[0272]假颗粒中和试验[0273]根据剂量范围发现,使用基于细胞的假颗粒中和测定法(pna)评价小鼠血清中的中和抗体:sars-cov-2假颗粒中和试验。将表达ace-2受体的veroe6细胞(veroc1008(美国,atcc编号crl-1586)),以20000个细胞/孔的速度播种,使细胞汇合度达到80%。将血清样本和对照组(内部生产的人类康复血清池)在细胞生长培养基中二重复稀释,起始稀释度为1/25或1/250(样本)或1/100(对照组),然后进行连续稀释(2倍稀释,5次)。同时,sars-cov-2假病毒(由nexelis公司使用kerafast系统制备,使用武汉株的刺突(减去19个c端氨基酸))被稀释,以达到所需的浓度(根据预先确定的tu/ml)。然后将假病毒加入稀释的血清样本中,在37℃下用co2预温育1小时。然后将该混合物加入到预先接种的veroe6细胞层中,在37℃和5%的co2条件下将板培养18-24小时。孵化并去除培养基后,将one-gloex荧光素酶检测底物(promega,目录号e8110)添加到细胞中,并在室温下摇晃孵化3分钟。使用spectramaxi3x微板阅读器和softmaxprov6.5.1(moleculardevices)测量发光。每个稀释度的发光结果被用来生成一个滴定曲线,使用微软excel(适用于微软office365)的4参数逻辑回归(4pl)。效价定义为发光等于预定的50截点(对应于50%中和)的样品稀释度的倒数。使用50%的侧翼点,使用线性回归建立该截止点。[0274]统计分析[0275]使用两尾t检验或mann-whitney检验比较组间和组间的显著性,适当时,p《0.05被认为是显著的。进行pearson检验以评价相关性。若r》0.7则认为相关性强,若0.5《r≤0.7则认为相关性中等,若0.3《r≤0.5则认为相关性弱。[0276]结果[0277]将polypepi-scov-2定制至个体[0278]在设计polypepi-scov-2期间,我们使用计算机模拟人类组群中受试者的hla基因型数据来确定四个选择的sars-cov-2结构蛋白的旨在诱导cd8 t细胞应答的最免疫原性肽(即hlai类pepi热点,9聚体)。然后基于病毒蛋白序列将鉴定的9聚体pepi热点的序列延伸至30聚体,以最大化旨在诱导cd4 t细胞应答的hlaii类结合pepi(15聚体)的数目。[0279]首先,我们分别使用9聚体(i类hla)和15聚体(ii类hla)框架,对其i类hla和ii类hla基因型(六个i类hla和八个ii类hla等位基因)的19个已知sars-cov-2病毒蛋白的保守区的氨基酸序列,在计算机模拟人类组群中对每个受试者进行表位预测(图1)。然后,我们选择能够结合多个(≥3)自体hla等位基因的表位(pepi)。我们为每个人鉴定了几个hla限制性表位(平均,仅s-1蛋白有166个表位)。相反,pepi在所有种族中以低得多的水平呈现(平均,14个多hla结合表位,图12)。[0280]从所得的pepi列表中,我们鉴定了九个30聚体多肽片段,其包括重叠的、在模型群体中的高百分比个体之间共享(频繁)的i类和ii类pepi,与种族无关(表9)。为了在个体水平和群体水平上皆最大化多抗原免疫应答,我们从大刺突(s)和核蛋白(n)蛋白中选择多余一个的肽,并且从较短的膜(m)和包膜(e)蛋白中仅选择一个肽。从sars-cov-2病毒的四个结构性病毒抗原中,共选择了九个30聚体的多肽用于疫苗,同时考虑了多肽的化学和可制造性:来自s的三个多肽、来自n的四个多肽、来自m和e的各一个多肽。没有包含来自s-1蛋白的受体结合域的多肽。总之,模型群体的每个成员具有九个肽中至少二个的pepi,97%具有至少三个(表9)。[0281]表9在计算机模拟人类组群内的包括pepi频率的polypepi-scov-2肽。粗体:包括30聚体肽的polypepi-scov-2内的9聚体hlai类pepi序列;下划线:15聚体ii类hlapepi序列。百分比显示了来自模型群体的具有至少一个i类hla(cd8 t细胞特异性)pepi或至少一个ii类hla(cd4 t细胞特异性)pepi的个体的比例。标记的肽含有实验证实的b细胞表位,具有在sars康复期患者中发现的抗体(ig)应答。n/a:数据不可用。[0282][0283][0284]我们通过实验鉴定了来源于sars-cov-1的线性b细胞表位,其与相关sars-cov-2抗原具有100%的序列同一性,以说明长肽的潜在b细胞生产能力。(54)polypepi-scov-2疫苗的位于n蛋白衍生肽中的三个重叠表位和位于s蛋白衍生肽中的一个表位与患严重急性呼吸综合征(sars)的恢复期患者的血清反应。这表明上述s和n蛋白上的抗原位点在引发人体内针对sars-cov-1和可能针对sars-cov-2的体液免疫应答中是重要的。[0285]如所预期的,polypepi-scov-2含有几个(九个中的八个)肽,由于sars-cov-2和sars-cov之间的高度序列同源性,而与sars-cov交叉反应。polypepi-scov-2肽与属于α冠状病毒(229e和nl63)、β冠状病毒(oc43,hku1)和mers的普通(季节性)冠状病毒株之间的序列相似性低。因此,疫苗和先前冠状病毒感染个体之间的交叉反应性仍然较低,可能仅涉及疫苗的m1肽(参见方法和表10)。[0286]表10polypepi-scov-2和其它冠状病毒株之间的序列比对结果。序列比较是用比对的蛋白序列对之间的8聚体长的肽匹配进行的,定义为cd8 t细胞表位的最小长度要求。最大氨基酸匹配:最长相同的氨基酸序列长度。突出显示的灰色值表示至少八个氨基酸的相同序列。[0287][0288]在小鼠中polypepi-scov-2疫苗诱导的广泛t细胞应答[0289]在非人源化balb/c模型中和在cd34 hu-ncg(hu小鼠)小鼠的人源化免疫环境中,在相隔二周(第0天和第14天)施用一和二个疫苗剂后,进行polypepi-scov-2疫苗的临床前免疫原性试验以测量诱导的免疫应答。免疫后,没有小鼠呈现任何临床评分(评分0,表示与正常无偏差),表明不存在任何副作用或免疫厌恶。此外,在每个时间点通过肉眼观察进行的尸检没有揭示脾、肝、肾、胃和肠中的任何可见的器官变化。重复的疫苗施用也是良好耐受的,并且没有检测到免疫毒性或其他全身性不良事件的迹象。总之,这些数据强烈提示本研究中使用的制剂在小鼠中是安全的。[0290]在第14天(d14)第一剂后和在第21天(d21)和第28天(d28)第二剂后测量产生ifn-γ的疫苗诱导的t细胞。在第14天,polypepi-scov-2处理没有比载体(用montanide乳化的dmso/水)诱导更多的cd8 t细胞的ifn-γ产生,后者导致异常高的应答,可能是由于montanide介导的非特异性应答。然而,在第21天和第28天,对于用30聚体和9聚体肽池刺激的脾细胞,第二剂的polypepi-scov-2与媒介物对照组相比将ifn-γ产生提高六倍和3.5倍(图13a)。在第21天和第28天,来自polypepi-scov-2处理的小鼠的t细胞的ifn-γ产生增加通过斑点形成细胞的数目增加而证实,其对于以下条件达到统计学显著性:分别是第21天的30聚体s池、肽e1、9聚体n池和肽e1,以及第28天的肽e1和9聚体s池(图14a–c)。[0291]在免疫缺陷的hu小鼠中,在第14天,用polypepi-scov-2处理使ifn-γ产生增加二倍,其中脾细胞用9聚体肽池刺激,但用30聚体刺激的脾细胞未观察到增加。在第21和28天,第二剂的polypepi-scov-2分别对用9聚体和30聚体肽池刺激的脾细胞增强ifn-γ的产生二倍和四倍(图13b)。重要的是,在二种动物模型中,9聚体检测的cd8 t细胞和30聚体检测的cd4 和cd8 t细胞应答皆同等地针对疫苗靶向的所有四个病毒蛋白(图13c、d和图14d–f中的详细结果)。[0292]进行细胞内染色(ics)分析以研究由接种引起的t细胞应答的极化。由于t细胞的频率低,与elispot相比,ics更难以观察单个肽特异性t细胞,但与仅接受载体(用montanide乳化的dmso/水)的动物相比,在balb/c和hu小鼠模型中皆可检测到产生tnf-α和il-2的th1型细胞因子的cd4 和cd8 t细胞的清晰群体(图15)。对于th2型细胞因子il-4和il-13,分析在任何时间点都没有显示任何特异性应答。在这二个模型中皆检测到低水平的il-5和/或il-10细胞因子产生cd4 t细胞,但在第28天仅balb/c小鼠与载体对照显著不同。即使对于该组群,6只小鼠中有5只(一个异常值)th1/th2平衡保持向th1偏移,证实了由疫苗引起的总体th1偏态t细胞应答(图16)。[0293]polypepi-scov-2接种也诱导体液应答,如通过balb/c的总小鼠igg和hu小鼠的人igg所测量的。在balb/c小鼠中,与仅接受载体的对照动物相比,接种在第一剂(第14天)后导致疫苗诱导的igg产生。在第二剂后的稍后时间点,对于balb/c和hu小鼠模型皆观察到igg升高(图17)。如所预期的,假定polypepi-scov-2肽不含构象表位,则疫苗接种不产生中和抗体,如使用用假颗粒的中和测定法从hu小鼠血清所评估的(数据未显示)。[0294]polypepi-scov-2疫苗在二种动物模型中诱导广泛的t细胞应答[0295]在balb/c和hu小鼠模型中,进行polypepi-scov-2疫苗的临床前免疫原性试验,以测量在相隔二周(第0天和第14天)施用一和二个疫苗剂后诱导的免疫应答。免疫后,在第14、21或28天没有小鼠出现任何临床评分(评分0,表示与正常没有偏差),表明没有任何副作用或免疫厌恶(表10a和b)。此外,在每个时间点通过肉眼观察进行的尸检没有揭示脾、肝、肾、胃和肠中的任何可见的器官变化(表10c)。重复的疫苗施用也是良好耐受的,并且没有检测到免疫毒性或其他全身性不良事件的迹象。总之,这些数据强烈提示polypepi-scov-2在小鼠中是安全的。[0296]在第14天第一剂后和在第21天和第28天第二剂后测量疫苗诱导的产生ifn-γ的t细胞。根据它们的来源蛋白质:s、n、m、和e,使用分组在四个池中的九个30聚体疫苗肽检测疫苗诱导的t细胞,以评估cd4 和cd8 t细胞应答。cd8 t细胞应答也使用短的9聚体试验肽特异性测量,该试验肽对应于上文对四个池(s、n、m和、e肽;表9粗体)中的九个疫苗肽中的每一个定义的共享hlai类pepi。[0297]在balb/c小鼠中,在第14天,polypepi-scov-2疫苗接种没有比载体(用montanide乳化的dmso/水)诱导更多的ifn-γ产生,后者导致异常高的应答,可能是由于montanide介导的非特异性应答。然而,在第21天和第28天,分别地,对于用30聚体和9聚体肽检测的脾细胞,第二剂的polypepi-scov-2与媒介物对照组相比使ifn-γ产生增加六倍和3.5倍(图13a)。[0298]在免疫缺陷的hu小鼠中,在第14天,用对9聚体肽池特异的脾细胞进行polypepi-scov-2接种使ifn-γ产生增加二倍,但用30聚体刺激的脾细胞未观察到增加。在第21和28天,分别地,对于用30聚体和9聚体肽检测的脾细胞,第二剂的polypepi-scov-2使ifn-γ产生增加四倍和二倍(图13b)。重要的是,在二种动物模型中,9聚体检测的cd8 t细胞和30聚体检测的cd4 和cd8 t细胞应答皆针对疫苗靶向的所有四个病毒蛋白(图13c–d和图14d–f)。由于hu小鼠模型是通过移植人cd34 造血干细胞产生人抗原呈递细胞和t-和b-淋巴细胞进入nod/shi-scid/il-2rγ无效免疫缺陷小鼠而开发的,该模型提供了真实的人免疫系统模型(brehm等人,2013)。因此,在该模型中获得的稳健的多抗原cd4 和cd8 t细胞应答表明,疫苗接种导致hu小鼠的人免疫细胞的适当加工的和hla呈递的表位和随后的抗原特异性t细胞应答。[0299]进行ics分析以研究由接种引起的t细胞应答的极化。由于t细胞的频率低,与elispot相比,ics更难以观察到单个肽特异性t细胞,但是与接受载体的动物相比,在balb/c和hu小鼠模型中皆可检测到产生tnf-α和il-2的th1型细胞因子的cd4 和cd8 t细胞的清晰群体(图15)。对于il-4和il-13th2型细胞因子,分析在任何时间点都没有显示任何特异性应答。在这二个模型中检测到低水平的il-5和/或il-10细胞因子产生cd4 t细胞,但在第28天仅balb/c小鼠与载体对照显著不同。即使对于该组群,6只小鼠中有5只(一个异常值)th1/th2平衡保持向th1偏移,证实了由疫苗引起的总体th1偏态t细胞应答(图16)。[0300]polypepi-scov-2接种也诱导体液应答,如通过balb/c的总小鼠igg和hu小鼠模型的人igg所测量的。在balb/c小鼠中,与载体对照组相比,第一剂(第14天)后接种导致疫苗诱导的igg产生。在第二剂后的稍后时间点,对于balb/c和hu小鼠模型皆观察到igg升高(图17)。在第28天时,从hu小鼠血浆测量的igg水平(平均115ng/ml,图17b)低于balb/c(平均529ng/ml,图17a)。这与已知的nod/shi-scid/il-2rγ无效免疫缺陷小鼠的局限性相一致,即它难以产生导致类别转换和igg产生的人类体液反应。(brehm等人,2013)。hu小鼠模型中人源化率为~50%进一步降低了理论上预期的igg水平。尽管有这些限制,但剂量依赖性的人igg产生表明疫苗产生的人体液应答。如所预期的,假定polypepi-scov-2肽不含构象b细胞表位,接种不产生可测量的中和抗体,如使用pna测定从hu小鼠的血清所评估的。在1∶25稀释度的检测极限下,每个试验样本的50%中和抗体滴度(nt50)是无法检测的(数据未显示)。[0301]表10abalb/c小鼠的安全性分析、临床评分数据表。临床安全评分是通过对五个不同的临床症状(被毛、运动、活动、苍白、体重)的描述来确定的,具体如下。被毛:0分——正常;1分——缺乏梳理、部分脱毛;2分——大量脱毛、伤口、出血、炎症。运动:0分——正常;2分——运动缓慢,一只动物瘫痪;3分——吃喝困难,多于一只动物瘫痪。活动:0分——正常;1分——激动、反应过度、反应不足;3分——瘫痪。苍白:0分——正常;1分——轻微(看不到耳部血管);2分——严重(耳朵和脚受到影响)。体重:0分——正常;2分——椎体分节明显,骨盆骨可触及;3分——骨骼结构突出。允许的最大累积临床评分:6。n.a.:不适用。[0302][0303]表10bhu小鼠的安全性分析、临床评分数据表。临床安全评分是通过对五个不同的临床症状(被毛、运动、活动、苍白、体重)的描述来确定的,具体如下。被毛:0分——正常;1分——缺乏梳理、部分脱毛;2分——大量脱毛、伤口、出血、炎症。运动:0分——正常;2分——运动缓慢,一只动物瘫痪;3分——吃喝困难,多于一只动物瘫痪。活动:0分——正常;1分——激动、反应过度、反应不足;3分——瘫痪。苍白:0分——正常;1分——轻微(看不到耳部血管);2分——严重(耳朵和脚受到影响)。体重:0分——正常;2分——椎体分节明显,骨盆骨可触及;3分——骨骼结构突出。允许的最大累积临床评分:6。n.a.:不适用。[0304][0305]表10c安全性分析、尸检数据表。通过肉眼观察脾、肝、肾、胃和肠进行尸检。[0306][0307][0308]covid-19康复供体的polypepi-scov-2肽特异性t细胞应答[0309]接下来,我们旨在通过研究在covid-19康复供体的血液中循环的疫苗特异性t细胞,证明在接种疫苗的动物中检测到的强和广泛的t细胞应答与人类相关(基线数据报道在表8中)。[0310]在17个康复期和四个健康供体中研究疫苗肽与康复期免疫组分的反应性。根据来源蛋白,使用分为四个池的九个30聚体疫苗肽检测疫苗反应性cd4 t细胞:s,n,m、和e肽。cd8 t细胞应答使用对应于为根据其源蛋白(s、n、m、和e肽;表8粗体)分组为四个池的九个疫苗肽中的每一个所定义的显性和共有hlai类pepi的9聚体试验肽测量,如动物实验中所用。[0311]使用可以检测快速活化的效应期t细胞应答的离体fluorospot分析,与健康受试者相比,用30聚体(平均dsfu:48.1,p=0.014)和9聚体肽(平均dsfu:16.5,p=0.011)皆检测到显着数量的疫苗反应性的表达ifn-γ的t细胞(图19a和b)。对四个蛋白质特异性肽池的详细分析,揭示了17个供体中的三个用30聚体疫苗肽与所有四个结构抗原反应;82%的供体与二个抗原反应,59%的供体与三个抗原反应。值得注意的是,也可以鉴定出对所有17个供体中的四个抗原中的至少一个和对53%中的至少二个抗原的,短的9聚体特异性cd8 t细胞应答(表11)。[0312]表11通过离体fluorospot分析测定,covid-19恢复期供体患者对polypepi-scov-2疫苗靶向的一个、二个、三个或所有四个病毒抗原的应答率。九聚体是hlai类热点pepi,其嵌入在与以下四个结构蛋白相应的每个30聚体疫苗肽中:s,刺突;n,核蛋白;m,隔膜;包膜蛋白。[0313][0314]如通过ics分析所确定的,用9聚体肽刺激导致平均而言t细胞组成83%的cd8 t细胞、和17%的cd4 t细胞(图18a)。有趣的是,30聚体试验肽与cd4 和cd8 t细胞皆以50∶50的平均比例反应(图18a)。t细胞的功能性试验揭示cd8 t细胞主要产生ifn-γ、tnf-α、和il-2(具有少量il-4和il-10),而cd4 t细胞主要对il-2和ifn-γ呈阳性。回忆应答显示了清楚的th1细胞因子特征;th2应答不存在于用30聚体疫苗肽的回忆应答中(图18b)。[0315]接着,我们确定离体检测的、可用的、快速活化的t细胞是否也可以在疫苗肽存在下体外扩增。使用富集的elispot,与健康受试者相比,用30聚体(平均dsfu=3746,p=0.025)和9聚体(平均dsfu=2088,p=0.028)肽池皆检测到显着数量的疫苗反应性的表达ifn-γ的t细胞(图19a)。还相对于用含有47个长的肽的商业化的、大的sars-cov-2肽集合(smno)检测的应答,评价了polypepi-scov-2衍生的t细胞应答(30聚体池)的强度,所述肽衍生自结构(s、m、n)和非结构(开放阅读框orf-3a和7a)蛋白质二者。从二个库中获得的相对强度有利于covid-19供体中的疫苗池,而更健康的供体与商业肽库反应,证实了polypepi-scov-2疫苗的特异性提高(图19b)。[0316]为了证实和进一步描述在covid-19回收的个体中离体检测到的polypepi-scov-2特异性t细胞应答的多特异性,我们定义了由它们的t细胞靶向的独特肽。我们首先去卷积肽池,并使用体外扩增试验对与每个疫苗肽对应的每个9聚体hlai类pepi特异性的cd8 t细胞应答(图20)。分析揭示,每个9聚体肽被若干受试者识别;对于n4(93%)、s9(87%)、s2、n1、m1(80%)、e1(60%)、s5、n2(40%)观察到covid-19康复期供体中的最高识别率(图19c)。[0317]九个肽特异性cd8 t细胞应答的详细分析揭示100%的covid-19回收的受试者具有用至少一个肽再活化的polypepi-scov-2特异性t细胞,93%用二个以上肽再活化、87%用五个以上肽再活化、而27%具有对所有九个疫苗肽特异性的t细胞池。在蛋白质水平上,87%的受试者具有针对多(三)种蛋白质的t细胞,并且15个测量的供体中的八个(53%)与所有四个靶向病毒蛋白质反应(图19c)。这些数据证实了polypepi-scov-2肽对于个体是显性的并且在covid-19受试者之间是共享的,并且在康复群体中获得的多肽特异性t细胞频率与计算机模拟组群的共享pepi的预测频率良好比对(表9)。此外,ferretti等人在涉及康复的系统、费力的t细胞表位筛选研究中独立地证实了我们的疫苗肽的一些片段(表位)是共享的免疫显性表位。(27)[0318]对于我们的恢复期对象组群,疫苗特异性t细胞应答的宽度和强度与从症状发作的时间不相关,表明这些t细胞是持久的(长达5个月)(图21)。[0319]为了证明pepi(多hla结合表位)可以在个体水平产生t细胞应答,我们首先确定每个受试者的完整i类hla基因型,然后从elispot测定法中使用的九个9聚体肽的列表中预测可以结合人的至少三个hla等位基因的肽。对于每个受试者,九个肽中的二个和七个之间被证明是pepi。在预测的肽(pepi)中,84%通过elispot证实产生高度特异性t细胞应答(表12)。[0320]表11pepi预测与通过富集elispot测定法测量的covid-19康复期的免疫应答之间的一致性。个人表位(pepi)预测为≥3个自体hlai类等位基因结合9聚体表位,并与用相同的9聚体刺激测量的产生ifn-γ的cd8 t细胞应答比较。真阳性值以粗体字母突出显示。[0321][0322]多个自体等位基因结合表位与cd8 t细胞应答之间的相关性[0323]研究了对每个受试者检测的免疫原性肽的hla结合能力。首先,我们确定了每个受试者的完整hlai类基因型,然后预测了可以结合至fluorospot测定中所使用的九个共享9聚体肽中的每一个的自体hla等位基因的数目。然后我们将预测的hla结合表位与每个患者中对每个肽测量的cd8 t细胞应答相匹配(总共15×9=135个数据点,图25)。cd8 t细胞应答的强度倾向于与被多个自体hla等位基因所限制的表位相关(rs=0.188,p=0.028,图29b)。此外,我们观察到pepi产生的cd8 t细胞应答的强度(hla≥3)(dsfu中值=458)显著高于非pepi产生的(hla《3)(dsfu中值=110)(p=0.008)(图29b)。[0324]在135个数据点上,记录了98个阳性反应和37个阴性反应。在98个阳性反应中有37个是由pepi产生,而在37个阴性中仅有7个是pepi,其它的是限制于≤3个自体hla等位基因的表位(图25)。总之,2×2偶然性表揭示了t细胞应答与pepi关联(p=0.041,fisher确切概率),但不与hla限制性表位关联(p=1.000,fisher确切概率)(图25)。对于每个受试者,九个肽中有一到七个被证明是pepi。在预测的pepi中,44个中的37个(84%)通过ifn-γ荧光斑点分析证实其在给定的受试者中产生特异性t细胞应答(图29d和图25)。[0325]这些数据表明,受试者的完整hla基因型影响他们的cd8 t细胞应答,并且多个自体等位基因结合能力是免疫原性表位的关键特征。pepi通常低估了受试者的全部t细胞库,然而,它们精确地预测了受试者的pepi特异性cd8 t细胞应答。[0326]polypepi-scov-2反应性t细胞和sars-cov-2特异性抗体应答之间的相关性[0327]抗体产生需要t细胞依赖性b细胞活化。对于每个受试者,使用不同的商业试剂盒确定针对sars-cov-2的s和n抗原二者的不同水平的抗体应答(表8)。所有受试者均用euroimmuneelisa(igg)针对病毒s-1和roche试剂盒检测n相关抗体,检测为阳性。所有受试者测试为diaproigg和igm阳性(除了2个供体),17个中有7个检测为diaproiga检测混合的s-1和n蛋白特异性抗体应答(表8)。[0328]我们接着评价了polypepi-scov-2特异性cd4 t细胞反应性和抗体应答之间的相关性(图22)。通过elisa测量的与s-1蛋白特异性igg量相关的polypepi-scov-2反应性cd4 t细胞的总数(r=0.59,p=0.02,图22a)。接下来,分析了polypepi-scov-2疫苗(s2和s5)的特异性s-1蛋白衍生肽,而相关性是相似的(r=0.585,p=0.02,图22b)。类似地,用n蛋白衍生的polypepi-scov-2肽(n1、n2、n3和n4)检测的t细胞应答与用roche试剂盒检测的n特异性抗体呈现弱但不显著的相关性(图22c)。这些数据表明covid-19康复供体的polypepi-scov-2特异性cd4 t细胞应答和随后igg产生之间的联系。[0329]有趣的是,iga的产生与polypepi-scov-2特异性记忆cd4 t细胞应答相关(r=0.63,p=0.006,图6d)。对smno肽池反应的t细胞应答与任何抗体亚群都没有表现出相关性。这表明不是所有cd4 t细胞都有助于b细胞应答,因此有助于igg产生(数据未显示)。[0330]不同种族中预测的免疫原性[0331]我们在分布于来自美国骨髓供体数据库的16个不同族群的16,000个hla分型受试者的大组群中进行了我们的polypepi-scov-2疫苗的计算机模拟试验。(49)对于该大组群中的每个受试者,我们基于其完整的hlai类和ii类基因型预测了polypepi-scov-2特异性pepi。大多数受试者具有广泛的pepi库,其可能被转化至病毒特异性记忆cd8 t细胞克隆:预测98%的受试者具有抗至少二个疫苗肽的pepi,而95%、86%和70%分别抗三个、四个和五个肽(图23a)。[0332]计算机模拟试验显示≥98%的每个族群中的受试者将可能产生强细胞应答,具有针对疫苗中至少二个肽的cd8 和cd4 t细胞应答(图23b)。这种预测的高应答率对于covid-19报告的临床结果较差的种族(黑人、亚裔)也是如此。(25)基于这些数据,我们预计该疫苗将提供全球覆盖,不受种族和地理位置的影响。[0333]我们还使用16,000个群体(并且包括族群)来评估由其他人提出的理论总体覆盖率,即,筛选具有六个普遍hlai类等位基因中的至少一个的亚群,其被认为覆盖了总体群体的95%。(27,55,56)使用这种方法,我们观察到种族水平的显著异质性。尽管我们证实了挑选出的六个hla等位基因在高加索人和北美组群中是普遍的(91–93%),但是这些等位基因在所有其它族群中的频率较低,尤其是在非裔群体中(48–54%)(图23c)。我们推断,所提出的普遍hla等位基因集合可能涵盖种族加权的全球群体中的hla频率,但仅基于这些等位基因的用于疫苗接种目的表位选择将区分一些种族。因此,我们建议使用一种代表性的模型群体,该模型群体对人类种族中的异质性敏感,并且允许选择在种族之间的个体之间共享的pepi。值得注意的是,虽然我们没有观察到不同种族的个体的sars-cov-2蛋白衍生的表位生成能力基于它们的完整hla基因型的任何差异(图12)(这似乎没有解释在bame中观察到的较高的感染和死亡率),但是可以仔细选择用于亚单位疫苗的表位以解决bame组的hla基因型特征。观察到在不同族群中共有的pepi频率的异质性,特别是对于蛋白n,对整体疫苗的设计具有很大的影响(图26)。通过在大量真实受试者群体中进行“计算机模拟临床试验”,将族群内和族群间不同频率的靶组合为具有高整体覆盖率的候选疫苗是可行的。[0334]因此,为了在个体和群体/种族水平上皆最大化多抗原免疫应答,并且还考虑到肽的化学和可制造性特性,从sars-cov-2的四个结构蛋白中总共选择九个30聚体肽:来自刺突的三个肽(s)、来自核蛋白的四个肽(n)、和来自基质和包膜的各一个肽(m)和(e)。没有包含来自s蛋白受体结合结构域(rbd)的肽。总之,模型群体中的每个成员在九个肽中至少有二个hlai类pepi,97%的成员至少有三个(表9)。每个受试者具有多个用于疫苗肽的ii类pepi(表9)。每个受试者具有多个用于疫苗肽的ii类pepi(表9)。[0335]讨论[0336]我们证明了,当与montanideisa51vg佐剂一起施用时,polypepi-scov-2,一种包括九个合成的衍生自sars-cov-2病毒(s、n、m、e)的四个结构蛋白的长(30聚体)肽的多肽疫苗,在balb/c小鼠和人源化cd34 小鼠中是安全的和高度免疫原性的。此外,通过成功地再活化polypepi-scov-2特异性t细胞,在covid-19康复供体中证实了疫苗的免疫原性潜力,这与sars-cov-2感染产生的t细胞免疫广泛重叠。[0337]本发明的疫苗设计概念,在个体和群体水平上皆靶向多抗原免疫应答,代表了一种新的靶鉴定方法,其已经成功地用于癌症疫苗开发中以实现与临床环境中的初始功效相关的前所未有的免疫应答率。(32)对于covid-19,我们集中于选择sars-cov-2蛋白的片段,其含有在种族差异的hla基因分型个体之间共有的i类和ii类hlat细胞表位的重叠,并且其还产生针对整个病毒结构的差异和广泛的免疫应答。因此,我们选择相对长的30聚体片段以利于产生多重效应应答(b细胞和细胞毒性t细胞)和辅助t细胞应答。[0338]polypepi-scov-2疫苗在接种的balb/c和人源化cd34 小鼠中引发针对所有四个sars-cov-2蛋白的疫苗特异性cd8 和cd4 t细胞二者的期望的多抗原ifn-γ产生性t细胞应答,并且这些应答在加强剂量后更显著。covid-19康复期的回忆应答包括针对所有九个肽的快速激活效应型(离体测得)和扩展(体外测得)记忆型cd8 和cd4 t细胞应答,在100%供体中检测到polypepi-scov-2特异性t细胞。在个体水平上,用于从covid-19回收的polypepi-scov-2特异性t细胞池极其多样化:在其患病后1–5个月,每个供体平均具有七个不同的肽特异性t细胞集合,具有针对sars-cov-2蛋白的多个靶;87%的供体具有针对至少三个sars-cov-2蛋白的、而53%的供体具有针对所有四个的。此外,87%的受试者具有抗s蛋白的cd8 t细胞,这与i/ii期临床试验中报道的前沿covid-19疫苗候选物的免疫应答率相似)。(57)然而,我们发现s特异性(记忆)t细胞仅代表用疫苗肽检测的恢复期总t细胞所有组成成分的36%;剩余的64%几乎相等地分布在n、m、和e蛋白中。这些数据证实了越来越多的关注,即基于s蛋白的候选疫苗不能发挥人抗sars-cov-2免疫的全部潜力,特别是因为多样性t细胞应答与温和/无症状的covid-19有关。(4)[0339]t细胞和b细胞之间的相互作用是已知的产生抗体的浆细胞和产生记忆b细胞二者的机制。(59)在康复期抗体亚群的分析期间,我们发现抗原特异性igg水平和相应的肽特异性cd4 t细胞应答之间的相关性。这种相关性可能代表cd4 t细胞和抗体产生之间的联系,这一概念也得到动物模型中总igg产生的支持。结合igg抗体可以在随后的sars-cov-2暴露于接种者时与疫苗诱导的cd8 杀伤t细胞协同作用。这种相互作用可能导致有效的cd8 t细胞介导的直接杀伤感染细胞和igg介导的杀伤病毒感染细胞和病毒颗粒,也抑制th2依赖的免疫病理过程。以这种方式,预期二个细胞内端的细胞外病毒储库都受到攻击,以帮助快速清除病毒,即使在不存在中和抗体的情况下。(59,60)[0340]本数据证明个体对polypepi-scov-2肽组反应的抗sars-cov-2t细胞应答是hla基因型依赖性的。具体地,多个自体hla结合表位(pepi)以84%的准确度确定抗原特异性cd8 t细胞应答。尽管pepi通常低估了受试者的全部t细胞库,但它们是精确的靶标鉴定“工具”和pepi特异性免疫应答的预测物,克服了在仅使用表位结合亲和力作为t细胞应答预测物的领域中通常观察到的高假阳性率。(34,61)因此,通过基于(仅有)高加索人个体的完整hla基因型的t细胞应答的经验证的pepi预测,以及在对人免疫应答建模的动物中对polypepi-scov-2诱导的免疫应答的仔细解释,我们的发现可以外推至16,000个hla基因分型的个体和16个人种族的大组群。在该大美国组群中代表的族群涵盖全球人口的组成,但是它们没有就其全球代表性进行加权(对于每个种族有意使用n=1,000个受试者)。(62)基于此,polypepi-scov-2将可能在》98%的全球群体中产生有意义的免疫应答(针对至少二个疫苗肽的cd8 和cd4 t细胞应答),而与种族无关。相比之下,基于全球频繁出现的hla等位基因的t细胞表位的疫苗设计方法会漏掉对黑加勒比海人、非裔、非裔美国人、和越南裔中约50%的人产生的免疫反应。通过接种疫苗在高百分比的接种疫苗的受试者中均匀地诱导有意义的免疫应答(广泛和多功能记忆应答,模拟由sars-cov-2诱导的免疫的异质性)对于实现期望的“群体免疫”是必要的。据认为,约25–50%的人群必须对病毒免疫以实现对群体传播的抑制。(3)然而,第一代covid-19疫苗正被测试显示疾病风险降低至少50%,但它们不能预期将病毒传播降低到相当的程度。这个事实与这些疫苗也不提供长期免疫力的可能性一起暗示需要第二代工具来对抗大流行。(3)[0341]我们相信,集中于每个受试者中的几个靶将更好地重演由病毒诱导的天然t细胞免疫,导致有效的、长期的记忆应答。为此目的,基于合成多肽的平台技术被认为是具有若干关键优点的安全和免疫原性疫苗接种策略。具有montanide佐剂的相同类型的肽疫苗在》6,000名患者和231名健康志愿者中是安全的和良好耐受的。(63–69)对sars的研究表明,限制包含完整病毒蛋白的候选冠状病毒疫苗具有更有益的安全性。此外,有效的th1偏态t细胞应答和非构象(线性)b细胞表位减轻了理论抗体介导的疾病增强和基于th2的免疫病理过程的风险。[0342]基于肽的疫苗迄今为止仅取得有限的成功,但这可归因于缺乏关于使用哪些肽的知识。通过理解个体的遗传背景如何能够响应特定的肽,降低了这种不确定性,如我们以上所证明的。[0343]总之,本文所述的基于肽的多表位编码疫苗设计证明了安全性和异常广泛的临床前免疫原性,并且在仔细的临床试验后,预期提供针对sars-cov-2的有效的第二代疫苗。[0344]实施例11——针对sars的交叉保护polypepi-scov-2特异性多肽的分析。[0345]通过预测模型群体(mp,n=433)内的自体的≥3hla结合表位的高频率选择疫苗组合物的多肽,使得能够选择在不同病原体物种之间共享的序列。例如,在sars病毒和sars-cov-2的蛋白质组中有几个保守的和100%相同的肽片段(seqidno:6和9–17)。通过分析这些片段,发明人在高百分比的mp个体中鉴定出了适当的≥3个hla结合9聚体pepi。99%具有至少一个来自所分析的seqidno:1–17(表13和图27a)的组合物的预测共有表位,94%的mp具有至少二个hlai类特异性(cd8 t细胞)pepi,因此预测该组合物在94%的mp中诱导多肽免疫应答。平均而言,3或4个sars衍生的共享肽被预测为与17个30聚体的肽发生反应。如果一个肽代表一个t细胞克隆(预测的和elispot测定的免疫应答的相关性在实施例5中得到证实,例如ppv=0.79),如果这17个肽用于免疫,这些计算机模拟结果可解释为诱导多肽特异性免疫原性t细胞应答。[0346]表13在模型群体(n=433)中预测的来自最初为sars-cov-2设计的17个30聚体肽的共享sars-cov表位的多抗原(多蛋白)应答率。[0347]多重肽(n=433)polypepi-scov-2共享sars-cov》=1100%99%》=2100%94%》=399%88%》=499%61%》=597%31%》=693%7%》=790%0%》=883%0%》=972%0%》=1064%0%》=1153%0%》=1242%0%》=1331%0%》=1420%0%》=1511%0%》=162%0%》=171%0%肽平均:10.573.79[0348]针对至少二个抗原的多蛋白或多抗原应答在91%的个体中共享,如果用于sars组群,则代表对该组合物的良好覆盖(图28a)。这些数据表明,这17个肽具有诱导抗sars的多肽和多蛋白特异性t细胞应答的潜力。[0349]结果也在16,000人的人类群体中得到证实。95%的个体具有针对至少2个共有肽的多hla限定的个人表位。[0350]表14从最初为sars-cov-2设计的17个30聚体多肽中预测出的人类大群体(n=16,000)对共享sars-cov表位的多抗原(多蛋白)反应率。[0351]多重肽(n=16,000)polypepi-scov-2共享sars-cov》=1100%98%》=2100%95%》=399%87%》=498%62%》=596%29%》=692%8%》=788%0%》=882%0%》=974%0%》=1065%0%》=1156%0%》=1245%0%》=1333%0%》=1421%0%》=1511%0%》=165%0%》=171%0%肽平均:10.653.79[0352]这些数据代表了鉴定具有针对不同病原体,例如针对sars的共有特异性的丰富的、多hla结合表位的机会(图27b和图28b)。[0353]实施例12——编码和表达seqidno:1–17的核酸制剂[0354]seqidno:1–17多肽可以不同的制剂制备,以水溶性或含佐剂的肽溶解或乳化用于注射。另外,如果3个字母的氨基酸编码被翻译成与seqidno:1–17相同的氨基酸序列,该多肽可以被编码成mrna、rna或dna制剂并在质粒dna或病毒载体中表达。用于递送和/或表达所编码的多肽的合适载体包含但不限于腺病毒载体、腺相关载体、慢病毒或逆转录病毒载体、痘病毒衍生载体、新城疫病毒载体、植物病毒载体如镶嵌病毒载体、和杂交载体。[0355]抗原呈递细胞中表达的蛋白质通过与疫苗皮下或皮内递送时apc摄取的多肽类似的hlai类和ii类抗原呈递途径加工和呈递。[0356]表15seqidno:1–17的氨基酸序列的对应的rna和dna序列。iupac核苷酸代码缩写:a:腺嘌呤;c:胞嘧啶,g:鸟嘌呤,t:胸腺嘧啶,u:尿嘧啶,r:a或g,y:c或t/u,s:g或c,w:a或t/u,k:g或t/u,m:a或c,b:c或g或t/u,d:a或g或t/u,h:a或c或t/u,v:a或c或g,n:a或c或t/u或g。t/u代表等同的核苷酸:t表示dna序列,u表示rna序列。[0357][0358][0359]实施例13——多个自体等位基因结合表位和cd8 t细胞应答之间的相关性[0360]发明人研究了从分析的covid-19康复供体对每个受试者(n=15)检测的免疫原性肽seqidno:2、5、9、12–17的hla结合能力。[0361]首先,发明人确定了每个受试者的完整hlai类基因型,然后预测了可以结合在用于检测肽特异性的产生ifn-γ的t细胞的fluorospot测定中所使用的九个共享的9聚体肽中的每一个的自体hla等位基因的数目。然后将预测的hla结合表位与每个患者中对每个肽测量的cd8 t细胞应答相匹配(总共15×9=135个数据点,图25)。cd8 t细胞应答的强度倾向于与被多个自体hla等位基因所限制的表位相关(rs=0.189,p=0.029,图29a)。此外,pepi产生的cd8 t细胞应答的强度(hla≥3)(dsfu中值=458)显着高于非pepi产生的(hla《3)(dsfu中值=110)(p=0.008)(图29b)。[0362]在135个数据点上,记录了98个阳性反应和37个阴性反应。在98个阳性反应中有37个是由pepi产生,而在37个阴性中仅有7个是pepi,其它的是限制于≤3个自体hla等位基因的表位(图25)。总之,2×2偶然性表揭示了t细胞应答与pepi关联(p=0.041,fisher确切概率),但不与hla限制性表位关联(p=1.000,fisher确切概率)(图29c)。对于每个受试者,九个肽中有一到七个被证明是pepi。在预测的pepi中,44个中的37个(84%)通过ifn-γ荧光斑点分析证实其在给定的受试者中产生特异性t细胞应答(图29d和图25)。[0363]这些数据表明,受试者的完整hla基因型影响其cd8 t细胞应答,并且多个自体等immuneresponsetoexperimentalcoronavirusinfectionofman.epidemiolinfect105,435-446(1990).[0373]5.q.x.long,x.j.tang,q.l.shi,q.li,h.j.deng,j.yuan,j.l.hu,w.xu,y.zhang,f.j.lv,k.su,f.zhang,j.gong,b.wu,x.m.liu,j.j.li,j.f.qiu,j.chen,a.l.huang,clinicalandimmunologicalassessmentofasymptomaticsars-cov-2infections.natmed26,1200-1204(2020).[0374]6.t.sekine,a.perez-potti,o.rivera-ballesteros,k.j.-b.gorin,a.olsson,s.llewellyn-lacey,h.kamal,g.bogdanovic,s.muschiol,d.j.wullimann,t.kammann,j.t.parrot,e.folkesson,o.rooyackers,l.i.eriksson,a.t.allander,j.albert,m.nielsen,j.s.gredmark-russ,n.k.j.k.sandberg,d.a.price,h.-g.ljunggren,s.aleman,m.buggert,robusttcellimmunityinconvalescentindividualswithasymptomaticormildcovid-19.(2020).[0375]7.h.m.staines,d.e.kirwan,d.j.clark,e.r.adams,y.augustin,r.l.byrne,m.cocozza,a.i.cubas-atienza,l.e.cuevas,m.cusinato,b.m.o.davies,m.davis,p.davis,a.duvoix,n.m.eckersley,d.forton,a.fraser,g.garrod,l.hadcocks,q.hu,m.johnson,g.a.kay,k.klekotko,z.lewis,j.mensah-kane,s.menzies,i.monahan,c.moore,g.nebe-von-caron,s.i.owen,c.sainter,a.a.sall,j.schouten,c.williams,j.wilkins,k.woolston,j.r.a.fitchett,s.krishna,t.planche,dynamicsofiggseroconversionandpathophysiologyofcovid-19infections.(2020).[0376]8.a.grifoni,d.weiskopf,s.i.ramirez,j.mateus,j.m.dan,c.r.moderbacher,s.a.rawlings,a.sutherland,l.premkumar,r.s.jadi,d.marrama,a.m.desilva,a.frazier,a.f.carlin,j.a.greenbaum,b.peters,f.krammer,d.m.smith,s.crotty,a.sette,targetsoftcellresponsestosars-cov-2coronavirusinhumanswithcovid-19diseaseandunexposedindividuals.cell181,1–13(2020).[0377]9.n.l.bert,a.t.tan,k.kunasegaran,c.y.l.tham,m.hafezi,a.chia,m.chng,m.lin,n.tan,m.linster,w.n.chia,m.i.c.chen,l.-f.wang,e.e.ooi,s.kalimuddin,p.a.tambyah,j.g.-h.low,y.-j.tan,a.bertoletti,differentpatternofpre-existingsars-cov-2specifictcellimmunityinsars-recoveredanduninfectedindividuals.(2020).[0378]10.y.peng,a.j.mentzer,g.liu,x.yao,z.yin,d.dong,w.dejnirattisai,t.rostron,p.supasa,c.liu,c.lopez-camacho,j.slon-campos,y.zhao,d.i.stuart,g.c.paesen,j.m.grimes,a.a.antson,o.w.bayfield,d.hawkins,d.s.ker,b.wang,l.turtle,k.subramaniam,p.thomson,p.zhang,c.dold,j.ratcliff,p.simmonds,t.desilva,p.sopp,d.wellington,u.rajapaksa,y.l.chen,m.salio,g.napolitani,w.paes,p.borrow,b.m.kessler,j.w.fry,n.f.schwabe,m.g.semple,j.k.baillie,s.c.moore,p.j.m.openshaw,m.a.ansari,s.dunachie,e.barnes,j.frater,g.kerr,p.goulder,t.lockett,r.levin,y.zhang,r.jing,l.p.ho,t.c.c.oxfordimmunologynetworkcovid-19response,i.c.investigators,r.j.cornall,c.p.conlon,p.klenerman,g.r.screaton,j.mongkolsapaya,a.mcmichael,j.c.knight,g.ogg,t.dong,broadandstrongmemorycd4( )andcd8( )tcellsinducedbysars-cov-2inukconvalescentindividualsfollowingcovid-19.natimmunol,(2020).[0379]11.a.nelde,t.bilich,j.s.heitmann,y.maringer,h.r.salih,m.roerden,m.lübke,j.bauer,j.rieth,m.wacker,a.peter,s.b.traenkle,p.d.kaiser,u.rothbauer,m.becker,d.junker,g.krause,m.strengert,n.schneiderhan-marra,m.f.templin,t.o.joos,d.j.kowalewski,v.stos-zweifel,m.fehr,m.graf,l.-c.gruber,d.rachfalski,b.preuβ,i.hagelstein,m.t.bakchoul,c.gouttefangeas,o.kohlbacher,r.klein,s.h.-g.rammensee,j.s.walz,sars-cov-2t-cellepitopesdefineheterologousandcovid-19-inducedt-cellrecognition.(2020).[0380]12.j.zhao,a.n.alshukairi,s.a.baharoon,w.a.ahmed,a.a.bokhari,a.m.nehdi,l.a.layqah,m.g.alghamdi,m.m.algethamy,a.m.dada,i.khalid,m.boujelal,s.m.aljohani,l.vogel,k.subbarao,a.mangalam,c.wu,p.teneyck,s.perlman,j.zhao,recoveryfromthemiddleeastrespiratorysyndromeisassociatedwithantibodyandt-cellresponses.sciimmunol2,(2017).[0381]13.b.diao,c.wang,y.tan,x.chen,y.liu,l.ning,l.chen,m.li,y.liu,g.wang,z.yuan,z.feng,y.zhang,y.wu,y.chen,reductionandfunctionalexhaustionoftcellsinpatientswithcoronavirusdisease2019(covid-19).frontimmunol11,827(2020).[0382]14.t.m.snyder,r.m.gittelman,m.klinger,d.h.may,e.j.osborne,r.taniguchi,h.j.zahid,i.m.kaplan,j.n.dines,m.n.noakes,r.pandya,x.chen,s.elasady,e.svejnoha,p.ebert,m.w.pesesky,p.dealmeida,h.o'donnell,q.degottardi,g.keitany,j.lu,a.vong,r.elyanow,p.fields,j.greissl,l.baldo,s.semprini,c.cerchione,m.mazza,o.m.delmonte,k.dobbs,g.carreno-tarragona,s.barrio,l.imberti,a.sottini,e.quiros-roldan,c.rossi,a.biondi,l.r.bettini,m.d'angio,p.bonfanti,m.f.tompkins,c.alba,c.dalgard,v.sambri,g.martinelli,j.d.goldman,j.r.heath,h.c.su,l.d.notarangelo,j.martinez-lopez,j.m.carlson,h.s.robins,magnitudeanddynamicsofthet-cellresponsetosars-cov-2infectionatbothindividualandpopulationlevels.medrxiv,(2020).[0383]15.j.zhao,j.zhao,s.perlman,tcellresponsesarerequiredforprotectionfromclinicaldiseaseandforvirusclearanceinsevereacuterespiratorysyndromecoronavirus-infectedmice.jvirol84,9318-9325(2010).[0384]16.r.channappanavar,c.fett,j.zhao,d.k.meyerholz,s.perlman,virus-specificmemorycd8tcellsprovidesubstantialprotectionfromlethalsevereacuterespiratorysyndromecoronavirusinfection.jvirol88,11034-11044(2014).[0385]17.r.channappanavar,j.zhao,s.perlman,tcell-mediatedimmuneresponsetorespiratorycoronaviruses.immunolres59,118-128(2014).[0386]18.w.li,m.d.joshi,s.singhania,k.h.ramsey,a.k.murthy,peptidevaccine:progressandchallenges.vaccines(basel)2,515-536(2014).[0387]19.m.black,a.trent,m.tirrell,c.olive,advancesinthedesignanddeliveryofpeptidesubunitvaccineswithafocusontoll-likereceptoragonists.expertreviewofvaccines9,157-173(2010).[0388]20.a.l.thompson,h.f.staats,cytokines:thefutureofintranasalvaccineadjuvants.clindevimmunol2011,289597(2011).[0389]21.n.petrovsky,j.c.aguilar,vaccineadjuvants:currentstateandfuturetrends.immunolcellbiol82,488-496(2004).[0390]22.d.sesardic,syntheticpeptidevaccines.jmedmicrobiol39,241-242(1993).[0391]23.m.s.bijker,c.j.melief,r.offringa,s.h.vanderburg,designanddevelopmentofsyntheticpeptidevaccines:past,presentandfuture.expertrevvaccines6,591-603(2007).[0392]24.s.nayak,r.w.herzog,progressandprospects:immuneresponsestoviralvectors.genether17,295-304(2010).[0393]25.d.pan,s.sze,j.s.minhas,m.n.bangash,n.pareek,p.divall,c.m.williams,m.r.oggioni,i.b.squire,l.b.nellums,w.hanif,k.khunti,m.pareek,theimpactofethnicityonclinicaloutcomesincovid-19:asystematicreview.eclinicalmedicine23,100404(2020).[0394]26.r.w.aldridge,d.lewer,s.v.katikireddi,r.mathur,n.pathak,r.burns,e.b.fragaszy,a.m.johnson,d.devakumar,i.abubakar,a.hayward,black,asianandminorityethnicgroupsinenglandareatincreasedriskofdeathfromcovid-19:indirectstandardisationofnhsmortalitydata.wellcomeopenres5,88(2020).[0395]27.a.p.ferretti,t.kula,y.wang,d.m.nguyen,a.weinheimer,g.s.dunlap,q.xu,n.nabilsi,c.r.perullo,a.w.cristofaro,h.j.whitton,a.virbasius,k.j.olivier,l.b.baiamonte,a.t.alistar,e.d.whitman,s.a.bertino,s.chattopadhyay,g.macbeath,covid-19patientsformmemorycd8 tcellsthatrecognizeasmallsetofsharedimmunodominantepitopesinsars-cov-2.medrxiv,2020.2007.2024.20161653(2020).[0396]28.u.sahin,e.derhovanessian,m.miller,b.p.kloke,p.simon,m.lower,v.bukur,a.d.tadmor,u.luxemburger,b.schrors,t.omokoko,m.vormehr,c.albrecht,a.paruzynski,a.n.kuhn,j.buck,s.heesch,k.h.schreeb,f.muller,i.ortseifer,i.vogler,e.godehardt,s.attig,r.rae,a.breitkreuz,c.tolliver,m.suchan,g.martic,a.hohberger,p.sorn,j.diekmann,j.ciesla,o.waksmann,a.k.bruck,m.witt,m.zillgen,a.rothermel,b.kasemann,d.langer,s.bolte,m.diken,s.kreiter,r.nemecek,c.gebhardt,s.grabbe,c.holler,j.utikal,c.huber,c.loquai,o.tureci,personalizedrnamutanomevaccinesmobilizepoly-specifictherapeuticimmunityagainstcancer.nature547,222-226(2017).[0397]29.p.a.ott,z.hu,d.b.keskin,s.a.shukla,j.sun,d.j.bozym,w.zhang,a.luoma,a.giobbie-hurder,l.peter,c.chen,o.olive,t.a.carter,s.li,d.j.lieb,t.eisenhaure,e.gjini,j.stevens,w.j.lane,i.javeri,k.nellaiappan,a.m.salazar,h.daley,m.seaman,e.i.buchbinder,c.h.yoon,m.harden,n.lennon,s.gabriel,s.j.rodig,d.h.barouch,j.c.aster,g.getz,k.wucherpfennig,d.neuberg,j.ritz,e.s.lander,e.f.fritsch,n.hacohen,c.j.wu,animmunogenicpersonalneoantigenvaccineforpatientswithmelanoma.nature547,217-221(2017).[0398]30.a.w.purcell,j.mccluskey,j.rossjohn,morethanonereasontorethinktheuseofpeptidesinvaccinedesign.natrevdrugdiscov6,404-414(2007).[0399]31.o.lorincz,j.toth,m.megyesi,k.pántya,i.miklos,e.somogyi,z.csiszovszki,p.pales,l.molnar,e.r.1935pcomputationalmodeltopredictresponserateofclinicaltrials.annalsofoncology30,(2019).[0400]32.j.hubbard,c.cremolini,r.graham,r.moretto,j.mitchell,j.wessling,e.toke,z.csiszovszki,o.l.molnár,e.somogyi,m.megyesi,k.pántya,j.tóth,p.páles,i.miklós,a.falcone,p329polypepi1018off-theshelfvaccineasadd-ontomaintenancetherapyachieveddurabletreatmentresponsesinpatientswithmicrosatellite-stablemetastaticcolorectalcancerpatients(mssmcrc).journalforimmunotherapyofcancer7(34thannualmeeting&pre-conferenceprogramsofthesocietyforimmunotherapyofcancer(sitc2019):part1),282(2019).[0401]33.j.hubbard,c.cremolini,r.p.graham,r.moretto,j.mitchell,j.wessling,e.r.z.csiszovszki,o.lorincz,l.molnar,e.somogyi,m.megyesi,k.pántya,j.toth,p.pales,i.miklos,a.falcone,606pevaluationofsafety,immunogenicityandpreliminaryefficacyofpolypepi1018vaccineinsubjectswithmetastaticcolorectalcancer(mcrc)withapredictivebiomarker.annalsofoncology30,(2019).[0402]34.e.somogyi,z.csiszovszki,o.lorincz,j.toth,l.molnar,w.s.urban,t.k.pántya,p.pales,m.megyesi,e.r.1181pdpersonalantigenselectioncalculator(pascal)forthedesignofpersonalcancervaccines.annalsofoncology30,(2019).[0403]35.u.s.nationallibraryofmedicine.severeacuterespiratorysyndromecoronavirus2https://www.ncbi.nlm.nih.gov/genome/browse#!/viruses/86693/.[0404]36.f.madeira,y.m.park,j.lee,n.buso,t.gur,n.madhusoodanan,p.basutkar,a.r.n.tivey,s.c.potter,r.d.finn,r.lopez,theembl-ebisearchandsequenceanalysistoolsapisin2019.nucleicacidsres47,w636-w641(2019).[0405]37.b.korber,w.m.fischer,s.gnanakaran,h.yoon,j.theiler,w.abfalterer,b.foley,e.e.giorgi,t.bhattacharya,m.d.parker,d.g.partridge,c.m.evans,t.m.freeman,t.i.desilva,c.c.labranche,d.c.montefiori,spikemutationpipelinerevealstheemergenceofamoretransmissibleformofsars-cov-2.(2020).[0406]38.centersfordiseasecontrolandprevention.coronavirushttps://www.cdc.gov/coronavirus/types.html.[0407]39.t.u.consortium,uniprot:aworldwidehubofproteinknowledge.nucleicacidsresearch47,d506-d515(2018).[0408]40.c.internationalhapmap,d.m.altshuler,r.a.gibbs,l.peltonen,d.m.altshuler,r.a.gibbs,l.peltonen,e.dermitzakis,s.f.schaffner,f.yu,l.peltonen,e.dermitzakis,p.e.bonnen,d.m.altshuler,r.a.gibbs,p.i.debakker,p.deloukas,s.b.gabriel,r.gwilliam,s.hunt,m.inouye,x.jia,a.palotie,m.parkin,p.whittaker,f.yu,k.chang,a.hawes,l.r.lewis,y.ren,d.wheeler,r.a.gibbs,d.m.muzny,c.barnes,k.darvishi,m.hurles,j.m.korn,k.kristiansson,c.lee,s.a.mccarrol,j.nemesh,e.dermitzakis,a.keinan,s.b.montgomery,s.pollack,a.l.price,n.soranzo,p.e.bonnen,r.a.gibbs,c.gonzaga-jauregui,a.keinan,a.l.price,f.yu,v.anttila,w.brodeur,m.j.daly,s.leslie,g.mcvean,l.moutsianas,h.nguyen,s.f.schaffner,q.zhang,m.j.ghori,r.mcginnis,w.mclaren,s.pollack,a.l.price,s.f.schaffner,f.takeuchi,s.r.grossman,i.shlyakhter,e.b.hostetter,p.c.sabeti,c.a.adebamowo,m.w.foster,d.r.gordon,j.licinio,m.c.manca,p.a.marshall,i.matsuda,d.ngare,v.o.wang,d.reddy,c.n.rotimi,c.d.royal,r.r.sharp,c.zeng,l.d.brooks,j.e.mcewen,integratingcommonandraregeneticvariationindiversehumanpopulations.nature467,52-58(2010).[0409]41.j.robinson,j.a.halliwell,j.d.hayhurst,p.flicek,p.parham,s.g.marsh,theipdandimgt/hladatabase:allelevariantdatabases.nucleicacidsres43,d423-431(2015).[0410]42.j.robinson,j.a.halliwell,h.mcwilliam,r.lopez,s.g.marsh,ipd‑‑theimmunopolymorphismdatabase.nucleicacidsres41,d1234-1240(2013).[0411]43.k.yusim,b.t.m.korber,c.brander,d.barouch,r.deboer,b.f.haynes,r.koup,j.p.moore,b.d.walker,d.i.watkins,inhivmolecularimmunology2014.(losalamosnationallaboratory,theoreticalbiologyandbiophysics,https://www.hiv.lanl.gov/content/immunology/compendium.html,2014),vol.la-ur15-20754.[0412]44.g.pawelec,s.g.marsh,estdab:acollectionofimmunologicallycharacterisedmelanomacelllinesandsearchabledatabank.cancerimmunolimmunother55,623-627(2006).[0413]45.j.robinson,c.h.roberts,i.a.dodi,j.a.madrigal,g.pawelec,l.wedel,s.g.marsh,theeuropeansearchabletumourlinedatabase.cancerimmunolimmunother58,1501-1506(2009).[0414]46.j.r.arguello,a.m.little,e.bohan,j.m.goldman,s.g.marsh,j.a.madrigal,highresolutionhlaclassitypingbyreferencestrandmediatedconformationanalysis(rsca).tissueantigens52,57-66(1998).[0415]47.w.helmberg,r.dunivin,m.feolo,thesequencing-basedtypingtoolofdbmhc:typinghighlypolymorphicgenesequences.nucleicacidsres32,w173-175(2004).10,10895(2020).[0429]61.e.r.toke,m.megyesi,l.molnar,j.tóth,o.s.h.vanderburg,m.welters,c.j.melief,w.s.urban,t.roehnisch,u.heine,e.somogyi,z.csiszovszki,k.pántya,p.páles,i.miklós,f.lori,j.lisziewicz,predictiontheclinicaloutcomesofcancerpatientsafterpeptidevaccination.journalofclinicaloncology37,e14295(2019).[0430]62.demographicsoftheworld.https://www.quora.com/what-are-all-the-races-and-their-world-population-demographics-the-entire-world.[0431]63.s.ascarateil,a.puget,m.-e.koziol,safetydataofmontanideisa51vgandmontanideisa720vg,twoadjuvantsdedicatedtohumantherapeuticvaccines.journalforimmunotherapyofcancer3,(2015).[0432]64.j.atsmon,e.kate-ilovitz,d.shaikevich,y.singer,i.volokhov,k.y.haim,t.ben-yedidia,safetyandimmunogenicityofmultimeric-001‑‑anoveluniversalinfluenzavaccine.jclinimmunol32,595-603(2012).[0433]65.b.s.graham,m.j.mcelrath,m.c.keefer,k.rybczyk,d.berger,k.j.weinhold,j.ottinger,g.ferarri,d.c.montefiori,d.stablein,c.smith,r.ginsberg,j.eldridge,a.duerr,p.fast,b.f.haynes,immunizationwithcocktailofhiv-derivedpeptidesinmontanideisa-51isimmunogenic,butcausessterileabscessesandunacceptablereactogenicity.plosone5,e11995(2010).[0434]66.s.herrera,o.l.fernández,o.vera,w.cárdenas,o.ramírez,r.palacios,m.chen-mok,g.corradin,m.arévalo-herrera,phaseisafetyandimmunogenicitytrialofplasmodiumvivaxcsderivedlongsyntheticpeptidesadjuvantedwithmontanideisa720ormontanideisa51.theamericanjournaloftropicalmedicineandhygiene84,12-20(2011).[0435]67.g.pialoux,j.l.excler,y.rivière,g.gonzalez-canali,v.feuillie,p.coulaud,j.c.gluckman,t.j.matthews,b.meignier,m.p.kieny,etal.,aprime-boostapproachtohivpreventivevaccineusingarecombinantcanarypoxvirusexpressingglycoprotein160(mn)followedbyarecombinantglycoprotein160(mn/lai).theagisgroup,andl'agencenationalederecherchesurlesida.aidsreshumretroviruses11,373-381(1995).[0436]68.o.pleguezuelos,s.robinson,g.a.stoloff,w.caparrós-wanderley,syntheticinfluenzavaccine(flu-v)stimulatescellmediatedimmunityinadouble-blind,randomised,placebo-controlledphaseitrial.vaccine30,4655-4660(2012).[0437]69.y.wu,r.d.ellis,d.shaffer,e.fontes,e.m.malkin,s.mahanty,m.p.fay,d.narum,k.rausch,a.p.miles,j.aebig,a.orcutt,o.muratova,g.song,l.lambert,d.zhu,k.miura,c.long,a.saul,l.h.miller,a.p.durbin,phase1trialofmalariatransmissionblockingvaccinecandidatespfs25andpvs25formulatedwithmontanideisa51.plosone3,e2636(2008).[0438]其他参考资料[0439]ahmed,s.f.,quadeer,a.a.,&mckay,m.r.(2020).preliminaryidentificationofpotentialvaccinetargetsforthecovid-19coronavirus(sars-cov-2)basedonsars-covimmunologicalstudies.viruses,12(3).doi:10.3390/v12030254[0440]bagarazzi,m.l.,yan,j.,morrow,m.p.,shen,x.,parker,r.l.,lee,j.c.,giffear,m.,pankhong,p.,khan,a.s.,broderick,k.e.,knott,c.,lin,f.,boyer,j.d.,draghia-akli,r.,white,c.j.,kim,j.j.,weiner,d.b.&sardesai,n.y.,2012.immunotherapyagainsthpv16/18generatespotentth1andcytotoxiccellularimmuneresponses.scitranslmed,4,155ra138.[0441]bioley,g.,guillaume,p.,luescher,i.,yeh,a.,dupont,b.,bhardwaj,n.,mears,g.,old,l.j.,valmori,d.&ayyoub,m.,2009.hlaclassi-associatedimmunodominanceaffectsctlresponsivenesstoanesorecombinantproteintumorantigenvaccine.clincancerres,15,299-306.[0442]essekuf,adeyeyemc.bacteriaandph-sensitivepolysaccharide-polymerfilmsforcolontargeteddelivery.critrevtherdrugcarriersyst2011;28:395–445.[0443]gennaro,2000,remington:thescienceandpracticeofpharmacy,20thedition,isbn:0683306472[0444]grothey,a.,tabernero,j.,arnold,d.,degramont,a.,ducreux,m.p.,o'dwyer,p.j.,vancutsem,e.,bosanac,i.,srock,s.,mancao,c.,gilberg,f.,winter,j.&schmoll,h.-j.,2018.lba19fluoropyrimidine(fp) bevacizumab(bev) atezolizumabvsfp/bevinbrafwtmetastaticcolorectalcancer(mcrc):findingsfromcohort2ofmodul–amulticentre,randomizedtrialofbiomarker-drivenmaintenancetreatmentfollowingfirst-lineinductiontherapy.annalsofoncology,29.[0445]gudmundsdotter,l.,wahren,b.,haller,b.k.,boberg,a.,edback,u.,bernasconi,d.,butto,s.,gaines,h.,imami,n.,gotch,f.,lori,f.,lisziewicz,j.,sandstrom,e.&hejdeman,b.,2011.amplifiedantigen-specificimmuneresponsesinhiv-1infectedindividualsinadoubleblinddnaimmunizationandtherapyinterruptiontrial.vaccine,29,5558-66.[0446]kakimi,k.,isobe,m.,uenaka,a.,wada,h.,sato,e.,doki,y.,nakajima,j.,seto,y.,yamatsuji,t.,naomoto,y.,shiraishi,k.,takigawa,n.,kiura,k.,tsuji,k.,iwatsuki,k.,oka,m.,pan,l.,hoffman,e.w.,old,l.j.&nakayama,e.,2011.aphaseistudyofvaccinationwithny-eso-1fpeptidemixedwithpicibanilok-432andmontanideisa-51inpatientswithcancersexpressingtheny-eso-1antigen.intjcancer,129,2836-46.[0447]kisslersm,tedijantoc,goldsteine,gradyh,lipsitchm.thecopyrightholderforthispreprint.https://doi.org/10.1101/2020.03.04.20031112[0448]klebanoff,c.a.,acquavella,n.,yu,z.&restifo,n.p.,2011.therapeuticcancervaccines:arewethereyet?immunologicalreviews,239,27-44.antigen.jimmunother,37,84-92.[0457]vandenmooterg,weutsi,deriddert,blatonn.evaluationofinutecsp1asanewcarrierintheformulationofsoliddispersionsforpoorlysolubledrugs.intjpharm.2006jun19;316(1-2):1-6.[0458]yuan,j.,adamow,m.,ginsberg,b.a.,rasalan,t.s.,ritter,e.,gallardo,h.f.,xu,y.,pogoriler,e.,terzulli,s.l.,kuk,d.,panageas,k.s.,ritter,g.,sznol,m.,halaban,r.,jungbluth,a.a.,allison,j.p.,old,l.j.,wolchok,j.d.&gnjatic,s.,2011.integratedny-eso-1antibodyandcd8 t-cellresponsescorrelatewithclinicalbenefitinadvancedmelanomapatientstreatedwithipilimumab.procnatlacadsciusa,108,16723-8.当前第1页12当前第1页12
背景技术:
::2.初步研究表明sars-cov-2与sars-cov相似,为此,以前存在关于保护性免疫应答的研究数据。有关sars-cov的各种报道提示体液和细胞介导二者的免疫应答的保护作用。针对sars-cov的刺突(s)和核壳(n)蛋白产生的抗体应答在sars-cov感染的患者中特别普遍。尽管有效,但发现抗体应答在恢复期sars-cov患者中是短暂的。相反,t细胞应答已证实在恢复的患者中提供感染后的长期记忆。3.生产有效疫苗的挑战之一是,在不同人类受试者的免疫系统与由感染性病毒表达的不同抗原的相互作用的方式中存在巨大的可变性。本发明人先前已经表明,个体受试者的免疫应答通过单个抗原t细胞表位被受试者的多个hla等位基因识别的能力来预测。被受试者的多个hla等位基因所限制的t细胞表位充当预测个体患者的肽特异性t细胞应答的遗传生物标志物。这些遗传生物标记物被称为“个人表位”或“pepi”。多hla等位基因结合性pepi以比被接种对象的单一hla等位基因所限制的t细胞表位显著更高的速率诱导t细胞应答。在疫苗组合物的多肽中t细胞抗原决定部位的鉴定,其为在人类模型群体中的个体的多hla等位基因结合性pepi,已显示预测在临床试验中报导的免疫反应率(wo2018/158456、wo2018/158457和wo2018/158455)。4.开发有效疫苗的第二个挑战是病毒通过突变的持续进化和感染病毒异质性的潜力。5.第三个挑战是需要快速开发、安全性试验和验证新出现的sars-cov-2冠状病毒的疫苗的效力,并随后大规模生产疫苗,以满足即时的群体需求。常规疫苗开发是复杂且具有挑战性的过程。与由死亡或减毒病原体、灭活毒素、和重组亚单位制成的常规疫苗相比,肽疫苗提供了若干优点。短多肽可以快速合成,并且肽疫苗生产相对便宜。另外,肽疫苗避免了包含对宿主具有高反应原性的不必要组分,例如脂多糖、脂质、和毒素。含有montanide佐剂的肽疫苗的安全性和免疫原性已经在涉及超过6,000名患者和超过200名健康志愿者的多个临床试验中得到证实。6.肽疫苗开发策略通常以选择寻求最大化覆盖全球群体的hla等位基因限制性表位的组合为目标。根据这种方法,选择具有不同hla结合特异性的多种肽,以提供由基于肽(表位)的疫苗靶向的患者群体的增加的覆盖,还考虑到不同的hla类型在不同的种族中以显著不同的频率表达。例如,sfahmed等人提出了一组筛选的t细胞表位,估计其提供了全球以及中国群体对sars-cov-2的广泛覆盖(ahmedetal.viruses,12(3).2020)。他们使用hla限制性sars-cov衍生表位和公众可获得的iedbpopulationcoveragetool(http://tools.iedb.org/population)指导针对sars-cov-2的疫苗的实验工作。7.这种方法试图考虑不同种族群体中hla多态性和频率。然而,实际上,最常见的hla限制性表位不诱导hla匹配个体中的免疫应答,并且临床试验导致比预期更低的免疫应答率(slingluffcl.cancerj2011;17(5):343–50)。此外,cd8t细胞识别的肽已证实既是选择性的且是极其敏感的;一个氨基酸变化可以将特定表位改变为非免疫原性肽。8.其它方法包含mrna或pdna载体中s蛋白的全序列。(smithtrfetal.underreview10.21203/rs.3.rs-16261/v1;safetyandimmunogenicitystudyof2019-ncovvaccine(mrna-1273)forprophylaxissarscov-2infection,nct04283461)。9.因此,迫切需要一种疫苗,这种疫苗对全球人口的高比例有效,对病毒抗原突变具有抵抗力,并能迅速通过临床验证和生产的必要步骤。技术实现要素:10.发明人集中精力开发针对sars-cov-2的全球性多肽疫苗,以解决不同个体的免疫反应的异质性和感染病毒的潜在异质性的双重挑战。本文详述的肽设计概念将共有的个人表位设计与b细胞表位序列的进一步选择合并,导致在个体内的重叠的多hla结合表位,旨在诱导cd4 、cd8 和产生抗体的b细胞应答。因此,发明人已经鉴定了目前已知的sars-cov-2病毒抗原的保守区的30聚体多肽片段,其包括(i)在具有代表全球群体的hla基因型的人类受试者的模型群体中的最大cd8 个人表位(pepi);(ii)全球群体中的最大cd4 个人表位(pepi);和(iii)线性b细胞表位。包括由发明人鉴定的多肽的肽疫苗,预计在人类群体中的惊人高比例的受试者中诱导细胞毒性t细胞、辅助t细胞和b细胞应答。通过组合多于一个的由发明人鉴定的抗原片段,优选地通过组合由发明人从不同的sars-cov-2结构蛋白鉴定的抗原片段,可以在人类群体中实现甚至更高的应答率,和针对进化的异源感染病毒的持续效力。11.因此,在第一方面,本公开提供了一种多肽、或一种长度至多50个氨基酸,或长度至多60个氨基酸的多肽组,其中所述多肽包括选自seqidno:1至17的氨基酸序列或由其组成。12.在另一个方面,本发明提供了一种长度多至50个氨基酸、或长度至多60个氨基酸的多肽组,其中每个多肽包括选自seqidno:1至17的不同氨基酸序列或由其组成。13.多肽的一或多个可以由冠状病毒科、β-冠状病毒科或sars-cov-2蛋白的片段组成。多肽中的每一个可包括选自seqidno:1至17中的序列的核酸序列,其是不同的冠状病毒科、β-冠状病毒科或sars-cov-2蛋白的片段。多肽组可以包含来自以下组中的至少二组、三组或全部四组的至少一个序列:(a)seqidno:1至11(sars-cov-2表面蛋白的片段);(b)seqidno:12至15(sars-cov-2核壳蛋白的片段);(c)seqidno:16(sars-cov-2膜蛋白的片段);和(d)seqidno:17(sars-cov-2包膜蛋白的片段)。在一些情况下,肽组可包括至少3、4、5、6、7、8、9、10、11、12、13、14、15、16或17个不同的多肽,其每个包括选自seqidno:1至17的不同的氨基酸序列。在一个实施方式中,肽组包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列。在一个实施方式中,多肽组包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列。在一个实施方式中,多肽组包括seqidno:6、和9至17的氨基酸序列。在一个实施方式中,多肽组包括十个多肽,其中十个多肽中的每一个包括选自seqidno:2、5、7、9、12、13、14、15、16、和17的不同的氨基酸序列或由其组成。在一个实施方式中,多肽组包括九个多肽,其中九个多肽中的每一个包括选自seqidno:2、5、9、12、13、14、15、16、和17的不同的氨基酸序列或由其组成。在一个实施方式中,多肽组包括十个多肽,其中十个多肽中的每一个包括选自seqidno:6、和9至17的不同的氨基酸序列或由其组成。14.在另一方面,本公开提供了具有上述多肽或多肽组作为活性成分的药物组合物或试剂盒。在一些情况下,药物组合物或试剂盒可包括编码每个多肽的核糖核酸。15.在另一方面,本公开提供了在受试者中接种疫苗、提供免疫疗法或诱导免疫应答的方法,所述方法包括向受试者施用多肽、多肽组或药物组合物。在一些情况下,所述方法是治疗受试者中冠状病毒科感染、β-冠状病毒科感染、sars-cov-2感染、或sars-cov感染,与冠状病毒科或β-冠状病毒科感染、covid-19或严重急性呼吸综合征(sars)相关的疾病或病况的方法。在一些情况下,所述方法是预防受试者中冠状病毒科感染、β-冠状病毒科感染、sars-cov-2或sars-cov感染,或预防个体中与冠状病毒科或β-冠状病毒科感染相关的、或与covid-19或sars感染相关的疾病或病况的发展,的方法。在一些情况下,多肽的至少一个,或至少二个、三个、四个、五个、六个或每一个包括冠状病毒科蛋白、β-冠状病毒科蛋白、sars-cov-2或sars-cov蛋白的片段,其是预测为被受试者的至少二个、或在一些情况下三个或至少三个hlai类等位基因所限制的cd8 t细胞表位。在一些情况下,多肽的至少一个,或至少二个、三个、四个、五个、六个或每一个包括冠状病毒科蛋白、β-冠状病毒科蛋白、sars-cov-2或sars-cov蛋白的片段,其是预测为被受试者的至少二个、或在一些情况下至少三个、或在一些情况下四个或至少四个hlaii类等位基因所限制的cd4 t细胞表位。在一些情况下,多肽的至少一个、或至少二个、三个、四个、五个、六个或每个包括线性b细胞表位。16.在另一方面,本公开提供了17.–如上所述的多肽、多肽组或药物组合物,其用于在受试者中接种疫苗、提供免疫疗法或诱导细胞毒性t细胞应答的方法中,或用于在受试者中治疗或预防冠状病毒科感染、β-冠状病毒科感染、sars-cov-2感染或sars-cov感染,或在受试者中治疗或预防与冠状病毒科或β-冠状病毒科感染或covid-19或sars相关的疾病或病况的发展的方法中,任选地其中受试者如上所述;以及18.–多肽、多肽组或编码如上所述的多肽或多肽组的一或多个多核苷酸或细胞在制备,用于在受试者中接种疫苗、提供免疫疗法或诱导细胞毒性t细胞应答的,或在治疗或预防冠状病毒科感染、β-冠状病毒科感染、sars-cov-2感染或sars-cov感染,在受试者中治疗或预防与冠状病毒科或β-冠状病毒科感染、或covid-19或sars相关的疾病或病况的发展的方法中的,药物中的用途,任选地其中受试者如上所述。19.在一些情况下,本文所述的方法可以包括确定受试者的hlai类和/或ii类基因型的步骤。20.现在将通过示例而非限制的方式并且通过参考附图来更详细地描述本公开。当给出本公开时,许多等同的修改和变化对于本领域技术人员将是显而易见的。因此,所阐述的本公开的示例性实施例被认为是说明性的而非限制性的。在不脱离本公开的范围的情况下,可以对所描述的实施例进行各种改变。本文引用的所有文献,无论是上文还是下文,均明确地通过引用整体并入。21.本公开包含所描述的方面和优选特征的组合,除非这样的组合明显是不允许的或被声明为明确避免。如本说明书和所附权利要求中所用,单数形式“一”、“一个”、和“所述”包含复数指代,除非内容明确另外指明。因此,例如,提及“肽”包含二或更多个这样的肽。22.本文所用的章节标题仅为了方便,而不应被解释为以任何方式进行限制。附图说明23.图1polypepi1018疫苗接种后用富集elispot分析测量的免疫应答。a)按患者绘制的具有免疫反应的疫苗抗原的数量。深灰色实心条:与接种前相比减少或无变化,深灰色条纹条:与接种前相比应答增强(至少增加二倍),浅灰条纹条:从头诱导的疫苗特异性免疫应答。b)在接种前(黑色)、一次接种后(深灰色)、二剂后(浅灰色)、和3剂后(白色)检测polypepi1018疫苗特异性cd4t细胞应答,c)对于八名具有所有三个节点数据的患者,免疫应答经过基线(接种前)和一次接种后第12周测量的时间过程。每条线代表单个患者(n=8)。24.图2疫苗特异性多功能cd8(a)和cd4(b)t细胞应答的平均分布(n=9,在单剂polypepi1018疫苗后测量)。25.图3在患者010007接种前(pre)和38周(post)样品中用ihc检测til。26.图4患者的临床反应。a)11名入选患者对一线治疗的反应和试验期间的recist评估结果的游泳者图,b)显示了oberto-101试验期间靶病变体积的变化(加总)的蜘蛛图。每个数据点被与基线(接种前)测量的病变大小比较,c)单剂和多剂组的无进展存活的kaplan-meier分析。27.图5应答者患者的靶病变大小变化。a)020004患者接受单剂,基线时有二个靶病变,b)010004患者接受多剂,基线时有三个靶病变,c)010007患者接受多剂,基线时有一个靶病变,和d)010002患者接受多剂,基线时有一个靶病变。28.图6预测的(a–b)和测量的(c–d)多抗原免疫应答(agp)倾向于与多剂组(n=5)中的肿瘤体积减少(a和c)和pfs(b和d)相关。29.图7在16个种族的~16,000名受试者中,sfahmed等人提出的随机选择的表位疫苗和polypepi-scov-2疫苗的10个肽的,预测的疫苗诱导的免疫应答率(cd8)的比较。30.图8在16个种族的~16,000名受试者中,sfahmed等人选择的所有59个肽或polypepi-scov-2疫苗的10个肽的,预测的疫苗诱导的免疫应答率(cd8)的比较。31.图9具有针对polypepi-scov-2疫苗的至少2个肽的cd4和cd8t细胞二者的受试者的比例。在16个种族的~16,000个受试者组群中进行预测。32.图10具有针对由sfahmed等人提出的随机选择的表位疫苗诱导和polypepi-scov-2疫苗的10个多肽诱导的≥1–10个疫苗表位的免疫应答的受试者(在~16,000的组群中)的比例。33.图11具有针对由sfahmed等人提出的表位疫苗的所有59个肽或polypepi-scov-2疫苗的10个肽诱导的≥1–10个疫苗表位的免疫应答的受试者(在~16,000的组群中)的比例。34.图12在种族多样的计算机模拟(insilico)人类组群中sars-cov-2spike-1蛋白的热点分析。通过预测每位受试者的≥3个hla等位基因结合个人表位(pepi)进行分析。左图:纵轴的每行代表模型群体中的一个受试者,而横轴代表sars-cov-2s-1蛋白亚基序列。垂直带代表最常见的表位,即,大多数受试者的优势免疫原性蛋白区域(热点)或pepi。ceu,中欧裔;chb,华裔;jpt,日裔;yri,非裔;mix,混合种族受试者。颜色代表被限制在一人上的表位数:浅灰色,3;镭灰色,4;黑色,》5。pepi定义为被限制在一人上的≥3个等位基因的表位。右图,不同种族的受试者的表位/pepi的平均数量。35.图13在二个动物模型中由polypepi-scov-2疫苗接种引发的ifn-γ t细胞应答。与仅接受载体的各对照组群相比,balb/c小鼠(a)和人源化小鼠(hu小鼠)(b)中polypepi-scov-2疫苗诱导的t细胞应答的倍数变化。在第28天在balb/c(c)和人源化(hu小鼠)中检测到,二剂后对sars-cov-2蛋白衍生的疫苗肽特异性的疫苗诱导的t细胞应答(d)。试验条件:s池含有来源于s蛋白的三个肽;n池含有衍生自n蛋白的四个肽;m和e分别是9聚体和30聚体池二者中衍生自m和e蛋白的肽。将结果与相同时间点的载体(用montanide乳化的dmso/水)对照组进行比较。通过用9聚体和30聚体肽刺激进行离体(exvivo)elispot测定。小鼠在第0天和第14天接受二剂疫苗或载体。在每个时间点每个组群包括六只动物。斑点形成单位(sfu)代表2×105个脾细胞的未受刺激的背景校正值。用t检验来计算显著性。36.图14polypepi-scov-2处理增加了小鼠中产生ifn-γ的t细胞。polypepi-scov-2接种的小鼠用深灰色的点显示,并与用浅灰色的点显示的载体(用montanide乳化的dmso/水)对照动物比较。在第14天(a,balb/c;和d,hu小鼠)、第21天(b,balb/c;和e,hu小鼠)、和第28天(c,balb/c;和f,hu小鼠)用肽再刺激后,通过离体elispot分析脾中ifn-γ的产生。条件1,s池;s2、s5、和s9肽的刺突特异性30聚体池。条件2,n池;n1、n2、n3、和n4肽的核蛋白特异性30聚体池。条件3,m1膜特异性30聚体肽。条件4,e1包膜特异性30聚体肽。条件5,s池;相应30聚体的s2、s5、和s9hlai类pepi热点片段的刺突特异性9聚体池。条件6,n池;相应30聚体的n1、n2、n3、和n4hlai类pepi热点片段的核蛋白特异性9聚体池。条件7,m1相应30聚体的膜特异性9聚体hlai类pepi热点片段。条件8,e1相应30聚体的包膜特异性9聚体hlai类pepi热点片段。条件9,未刺激对照。显示了单个斑点形成细胞(sfc)值和平均值,代表每2×105个脾细胞的斑点。分析了每组n=6只小鼠。通过mann-whitney检验进行统计学分析。*,p《0.05;**,p《0.01。37.图15在小鼠模型中th1优势免疫应答且polypepi-scov-2没有诱导显著的th2细胞因子。使用ics检测,balb/c(a)和hu小鼠模型(b)二者的免疫组和载体对照组产生il2、tnf-α、ifn-γ、il-5或il-10的平均cd4 和cd8 t细胞。显示了平均值 /-sem。分析了2×105个细胞,门控cd45 细胞、cd3 t细胞、cd4 或cd8 t细胞。通过汇集cd4 和cd8 t细胞的4个刺激条件(30聚体s池、n池、e1和m1肽)的背景减去的值,得到平均百分比。38.图16balb/c小鼠在第28天th1/th2平衡。使用ics检测,平均cd4 和cd8 t细胞为每个免疫小鼠(n=6)产生il2、tnf-α、ifn-γ(th1细胞因子)和il10(th2细胞因子)。分析了2×105个细胞,门控cd45 细胞、cd3 t细胞、cd4 或cd8 t细胞。通过汇集cd4 和cd8 t细胞的4个刺激条件(30聚体s池、n池、e1和m1肽)的背景减去的值,得到平均百分比。39.图17从balb/c小鼠(a)和hu小鼠(b)模型的血浆中测量的疫苗诱导的igg产生。在第0和14天,小鼠接受二剂polypepi-scov-2疫苗或载体。每个组群包括六只动物。使用t检验来计算显著性。*,p《0.0540.图18covid-19康复者的t细胞对polypepi-scov-2肽的反应性产生细胞因子,通过细胞内染色法测定从他们的pbmc离体确定。(a)通过用9聚体和30聚体肽刺激获得的cd4 和cd8 t细胞 的细胞因子分布(n=17);(b)在用30聚体肽刺激的疫苗特异性t细胞中th1优势。41.图19来自covid-19康复供体的polypepi-scov-2特异性t细胞应答。a.通过离体fluorospot测定法检测的高度特异性疫苗衍生的9聚体肽反应性cd8 t细胞应答和30聚体肽反应性cd4 t细胞应答。b.用30聚体肽池、9聚体肽池、和商业snmo肽池活化的t细胞的富集elispot结果。c.用由对应于具有相同名称的每个30聚体肽的单独9聚体肽活化的cd8 t细胞的富集elispot结果(表9粗体)。dsfu,δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。42.图20通过富集elispot测定法测量,检测到covid-19康复供体针对polypepi-scov-2疫苗的9聚体肽(pepi热点)的ifn-γ t细胞应答。s2、s5、和s9是衍生自刺突特异性疫苗30聚体的三个s特异性9聚体肽序列。n1–n4是衍生自n特异性疫苗30聚体的四个核蛋白特异性9聚体肽序列。e1和m1分别是衍生自e或m特异性疫苗30聚体的包膜和膜特异性9聚体肽序列(表9粗体)。dsfu,δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。呈现了每个受试者的平均和个体数据。pbmc,外周血单核细胞。43.图21covid-19康复供体的t细胞应答相对于从症状发作开始的时间的幅度和广度。a)polypepi-scov-2反应性t细胞应答的幅度。b)用富集elispot测定法检测的来自康复期供体的疫苗肽反应性cd8 t细胞应答的幅度。dsfu代表δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。统计分析:pearson相关分析。r——pearson相关系数。44.图22在covid-19康复期个体中sars-cov-2特异性抗体水平与polypepi-scov-2特异性产生ifn-γ的cd4 t细胞之间的相关性。a)将对polypepi-scov-2肽的30聚体池反应的t细胞应答,对igg-s1(euroimmun)作图。b)将对s-1蛋白衍生的30聚体肽(s2和s5)反应的平均t细胞应答,对igg-s1(euroimmun)作图。c)将对包括n1、n2、n3和n4的30聚体n肽池反应的t细胞应答,对用roche分析测量的总igg-n作图。d)将对polypepi-scov-2的30聚体池反应的t细胞应答,对用diaproigaelisa测定测量的iga抗体量作图。通过pearson检验进行相关性分析。r——相关系数。45.图23预测了具有不同种族的大群体的全球覆盖。a)具有针对九个polypepi-scov-2疫苗肽中的至少一个的hlai类pepi的受试者的比例。b)具有针对polypepi-scov-2疫苗中的至少二个肽的hlai类和ii类pepi二者的受试者的比例。c)ferretti等人(27)提出的基于所选择的六个hla等位基因(a*02:01、a*01:01、a*03:01、a*11:01、a*24:02、和b*07:02)的频率估计的理论全球覆盖率。46.图24代表全球等位基因频率的模型群体中的等位基因频率分布。在模型群体中的hla等位基因频率代表与ciwd数据库中》8百万hla基因分型受试者的等位基因频率相似的分布。ciwd3.0:常见(10,000中≥1)、中间(100,000中≥1)和文献充分记载(≥5次出现)的hla数据库3.0(2020年发布)。r——pearson相关系数。与图20相关。47.图25covid-19康复期内多个自体等位基因结合表位和cd8 t细胞应答之间的相关性。将预测的多个自体hla结合表位(n=9)与n=15个供体中的相同肽反应性cd8 t细胞应答(135个数据点)匹配。数字表示通过富集荧光斑点分析确定的dsfu。颜色代码是指结合特异性肽的自体hla等位基因的预测数目。dsfu,δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。48.图26具有不同种族的个体基于其完整hla基因型的sars-cov-2s1蛋白特异性表位产生能力。与图12相关。对于s2、s5、s9、n1、n2、n3、n4、m1和e1中的每一个,从左到右,这些条分别对应于“全部”、“ceu”、“chb”、“jpt”、“yri”和“mix”。49.图27从最初为sars-cov-2设计的17个30聚体肽为共享sars-cov表位预测在模型群体(n=433)(a)中和在n=16,000大群体(b)中的多肽应答率。50.图28从最初为sars-cov-2设计的17个30聚体肽为共享sars-cov表位预测在模型群体(n=433)(a)中和在n=16,000大群体(b)中的多抗原(多蛋白)应答率。51.图29covid-19康复期个体中多个自体hla等位基因结合表位与产生polypepi-scov-2特异性ifn-γ的cd8 t细胞应答之间的相关性。(a)多个自体hla等位基因结合表位与t细胞应答的量级之间的相关性。rs——spearman系数(也通过pearson相关分析证实)。(b)通过富集fluorospot分析检测的pepi(结合≥3个自体hlai类等位基因)和非pepi(结合≤3个自体hlai类等位基因)的cd8 t细胞应答的量级,(p=0.008,t检验)。给出了每个受试者的中值和个体数据,n=15。(c)变量依赖性分析,使用2×2偶然性表和fisher确切概率检验。(d)对于每个受试者,通过产生ifn-γ的cd8 t细胞证实个人表位(pepi)[阳性预测值=真阳性/预测的总量=37/44(84%)]。dsfu,δ斑点形成单位,计算为每106pbmc的未刺激背景校正的斑点计数。pbmc,外周血单核细胞。[0052]序列表说明[0053]seqidno:1至17中列出了描述于表6a中的30聚体t细胞表位。[0054]seqidno:18–233列出了本文公开的各种序列。[0055]seqidno:234至267列出了如表15所示的编码seqidno:1至17的氨基酸序列的相应rna或dna序列。具体实施方式[0056]hla基因型[0057]hlas由人类基因组的最多态的基因编码。每个人具有三个hlai类分子(hla-a*、hla-b*、hla-c*)和四个hlaii类分子(hla-dp*、hla-dq*、hla-drb1*、hla-drb3*/4*/5*)的母本和父本等位基因。实际上,每个人表达呈递来自相同蛋白抗原的不同表位的6个i类hla和8个ii类hla分子的不同组合。[0058]用于命名hla分子的氨基酸序列的命名法如下:基因名称*等位基因:蛋白质编号,其例如可以看起来像:hla-a*02:25。在这个例子中,“02”指等位基因。在大多数情况下,等位基因通过血清型定义——意味着给定等位基因的蛋白质在血清学测定中不会彼此反应。当发现蛋白质时,连续分配蛋白质编号(在上述例子中为“25”)。为具有不同氨基酸序列的任何蛋白质分配新的蛋白质编号(例如,即使序列中一个氨基酸的变化也被认为是不同的蛋白质编号)。关于给定基因座的核酸序列的进一步信息可以附加到hla命名中。[0059]个体的i类hla基因型或ii类hla基因型可以指个体的每个i类或ii类hla的实际氨基酸序列,或可以指如上所述的命名,其最低限度指定每个hla基因的等位基因和蛋白质编号。hla基因型可以使用任何合适的方法来确定。例如,序列可以通过使用本领域已知的方法和方案对hla基因座测序来确定。或者,个人的hla集可存储在数据库中并使用本领域已知的方法访问。[0060]一些受试者可能具有编码相同hla分子的二个hla等位基因(例如,在纯合性的情况下,二个拷贝的hla-a*02:25)。由这些等位基因编码的hla分子结合所有相同的t细胞表位。出于本公开的目的,如本文所用的“结合至受试者的至少二个hla分子”包含结合至由单个受试者中的二个相同hla等位基因编码的hla分子。换句话说,“结合至受试者的至少二个hla分子”等可以另外表达为“结合至受试者的至少二个hla等位基因编码的hla分子”。[0061]多肽[0062]本公开涉及衍生自sars-cov-2抗原的多肽,其对大部分人类群体具有免疫原性。[0063]如本文所用,术语“多肽”是指特征为一串氨基酸的全长蛋白质、蛋白质的一部分、或肽。如本文所用,术语“肽”是指包括2、或3、或4、或5、或6、或7、或8、或9、或10、或11、或12、或13、或14、或15和10、或11、或12、或13、或14、或15、或20、或25、或30、或35、或40、或45、或50或55或60个氨基酸的短多肽。[0064]如本文所用,术语“片段”或“多肽片段”是指相对于参考多肽通常具有减小的长度并且在共同部分上包括与参考多肽相同的氨基酸序列的一串氨基酸或氨基酸序列。根据本公开的此类片段可以在适当的情况下被包含在其作为组成部分的较大多肽中。在一些情况下,片段可以包括多肽的全长,例如,其中整个多肽,例如一个9个氨基酸的肽,是单个t细胞表位。在一些情况下,本文提及的片段可以是8或9至20,或25,或30,或35,或40,或45,或50个氨基酸。[0065]如本文所用,术语“表位”或“t细胞表位”是指包含在蛋白抗原内的连续氨基酸序列,其具有对一或多个hla的结合亲和性(能够结合到一或多个hla)。表位是hla和抗原特异性的(hla–表位对,用已知方法预测),但不是受试者特异性的。如果能够在特定的人类受试者中诱导t细胞应答(细胞毒性t细胞应答或辅助t细胞应答)的话,表位、t细胞表位、多肽、多肽片段或包括多肽或其片段的组合物对于该受试者是“免疫原性的”。在一些情况下,辅助t细胞应答是th1型辅助t细胞应答。术语“t细胞应答”和“免疫应答”在此可互换使用,并且是指在识别一或多个hla–表位结合对之后t细胞的活化和/或一或多个效应子功能的诱导。在一些情况下,“免疫应答”包含抗体应答,因为ii类hla分子刺激参与诱导持久的ctl应答和抗体应答二者的辅助应答。效应子功能包含细胞毒性、细胞因子产生和增殖。根据本公开内容,表位、t细胞表位或多肽片段如果能够结合特定受试者的至少二个、或在一些情况下至少三个,i类或至少二个、或在一些情况下至少三个或至少四个ii类hla,则对于受试者是免疫原性的。[0066]“个人表位”(或“pepi”)是由多肽的连续氨基酸序列组成的多肽片段,该多肽是能够结合特定人类受试者的一或多个hlai类分子的t细胞表位。在其它情况下,“pepi”是由多肽的连续氨基酸序列组成的多肽片段,该多肽是能够结合特定人类受试者的一或多个hlaii类分子的t细胞表位。换句话说,“pepi”是被特定个体的hla组识别的t细胞表位,因此除了hla和抗原之外,还对受试者具有特异性。与仅对hla和抗原特异的“表位”相反,pepi对个体是特异的,因为不同的个体具有各自结合不同t细胞表位的不同hla分子。[0067]如本文所用的“pepi1”是指可结合个体的一个hlai类分子(或在特定情况下hlaii类分子)的肽或多肽片段。“pepi1 ”是指能与个体的一或多个hlai类分子结合的多肽、或多肽片段。“pepi2”是指能与个体的二个hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi2 ”是指能与个体的二或更多个的hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi3”是指能与个体的三个hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi3 ”是指能与个体的三或更多个hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi4”是指能与个体的三个hlai类(或ii类)分子结合的多肽、或多肽片段。“pepi4 ”是指能与个体的三或更多个hlai类(或ii类)分子结合的多肽、或多肽片段。[0068]一般而言,由hlai类分子呈递的表位的长度为约九个氨基酸,而由hlaii类分子呈递的表位的长度为约十五个氨基酸。然而,为了本公开的目的,表位可以多于或少于九个(对于i类hla)或十五个(对于ii类hla)氨基酸长度,只要该表位能够结合hla即可。例如,能够结合i类hla的表位长度可以在7、或8或9和9或10或11个氨基酸之间。能够结合ii类hla的表位的长度可以在13、或14或15和15或16或17个氨基酸之间。[0069]受试者的给定hla仅向t细胞呈递通过在抗原呈递细胞(apc)中加工蛋白抗原产生的有限数量的不同肽。如本文所用,“展示”或“呈递”,当与hla相关使用时,是指肽(表位)和hla之间的结合。在这方面,“展示”或“呈递”肽与“结合”肽同义。[0070]使用本领域已知的技术,有可能确定将结合已知hla的表位。可以使用任何合适的方法,只要使用相同的方法来确定被直接比较的多个hla–表位结合对。例如,可以使用生化分析。也可以使用已知与给定hla结合的表位列表。也可以使用预测或建模软件来确定哪些表位可以被给定的hla结合。表1提供了示例。在一些情况下,如果t细胞表位具有小于5000nm、小于2000nm、小于1000nm、或小于500nm的ic50或预测ic50,则该t细胞表位能够结合给定的hla。[0071]表1用于确定表位–hla结合的示例软件[0072][0073]本公开的肽可以包括一或多个冠状病毒科、β-冠状病毒科或sars-cov-2的选自表面糖蛋白(别名刺突蛋白)、核壳磷蛋白、包膜蛋白和膜糖蛋白的抗原,的一或多个片段,或由其组成。本文提供了参考序列。[0074]在一些情况下,氨基酸序列在n和/或c末端侧翼为不是冠状病毒科、β-冠状病毒科或sars-cov-2抗原序列的一部分的额外氨基酸,换句话说,不是与靶多肽抗原中所选片段相邻的连续氨基酸的相同序列。在一些情况下,序列在n和/或c末端侧翼有至多20、19、18、17、16、15、14、13、12、11、10、9、8、7、6、5、4、3、2或1个额外的氨基酸。[0075]发明人先前发现癌症疫苗中存在至少二个可结合个体的至少三个hlai类(≥2pepi3 )的多肽片段(表位)预示了临床反应。换句话说,如果在疫苗的活性成分多肽中鉴定出≥2pepi3 ,则个体可能是临床应答者。[0076]不希望被理论所束缚,发明人相信,从包括至少二个多hla结合性pepi的疫苗/免疫疗法中获得临床益处的可能性增加的一个原因,是患病的细胞群体,例如癌症或肿瘤细胞或被病毒或病原体(例如hiv)感染的细胞,在受影响的受试者内和受试者间二者都通常是异质的。此外,当更多的hla结合性pepi被包含或被疫苗靶向时,发展抗性的可能性被降低,因为患者不太可能通过靶pepi的突变发展对组合物的抗性。[0077]同样,在病毒感染的疫苗的情况下,其中病毒感染可以是异源的,向受试者施用疫苗肽是有利的,该疫苗肽被预测为包括多个受试者特异性多hla等位基因结合性pepi(用于治疗具有已知hla基因型的受试者)或多个群体最佳epi(bestepi),即在高比例的靶标群体中是或包括多hla等位基因结合性pepi的氨基酸序列。包含更多的最佳epi序列也增加了对治疗有反应的人类受试者的总比例。因此,在一些情况下,多肽组包括至少2、3、4、5、6、7、8、9、10、11、12、13、14、15、16或17个多肽,其各自包括选自seqidno:1至17的一个不同的氨基酸序列。在一些情况下,多肽的组合排除以下组合中的一或多个:seqidno:1和2;seqidno:3和4;seqidno:7和8;和/或seqidno:9和10;和/或排除以下组合中的一或多个:seqidno:2和3;和/或seqidno:13和14。[0078]多肽可以是或包括相同或不同病毒抗原的片段。不同的病毒结构蛋白可能倾向于以不同的速率突变。因此,在一些情况下,每个多肽包括选自seqidno:1至17的氨基酸序列,该序列是不同的冠状病毒科、β-冠状病毒科或sars-cov-2蛋白的片段。在一些情况下,多肽组包含来自以下组中的至少二组、三组或所有四组的至少一个序列:(a)seqidno:1至11(sars-cov-2表面蛋白的片段),任选地排除seqidno:1和2、seqidno:2和3、seqidno:3和4、seqidno:7和8、和/或seqidno:9和10;(b)seqidno:12至15(sars-cov-2核壳蛋白的片段),任选地排除seqidno:13和14;(c)seqidno:16(sars-cov-2膜蛋白的片段);和(d)seqidno:17(sars-cov-2包膜蛋白的片段)。在一些情况下,多肽的组合包括十个多肽或由其组成,多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列或由其组成。在另一个实施方式中,多肽组包括九个多肽,肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列或由其组成。在另一个实施方式中,多肽组包括十个多肽,多肽包括seqidno:6、和9至17的氨基酸序列或由其组成。[0079]多肽和患者的选择[0080]本文所述的肽可用于在有需要的受试者中诱导t细胞应答或提供疫苗接种或免疫治疗。所述的肽可用于治疗或预防受试者的冠状病毒科感染、β-冠状病毒科感染sars-cov-2感染、sars-cov感染、与冠状病毒科或β-冠状病毒科感染、covid-19或sars相关的疾病或病况。通常选择多于一个肽用于治疗受试者。在一些情况下,用于治疗的肽可以基于(i)受试者中待治疗的疾病或病况;(ii)受试者的hla基因型;和/或(iii)受试者(例如国籍或族群)的遗传背景。[0081]根据本发明可以治疗的冠状病毒科感染(和相关疾病)包含任何其中的病毒表达至少一个包括seqidno:1至17中任何一个的氨基酸序列(或表6a中所示seqidno:1至17中的最佳epi序列)的抗原。通常,病毒表达一起包括至少二个、或更通常至少3、4、5、6、7、8、9或10个不同的选自seqidno:1至17的序列(或最佳epi序列)的一或多个抗原。更典型地,病毒表达二或更多个不同的抗原,其中每一个抗原包括选自seqidno:1至17的序列(或最佳epi序列)。合适的如本文所述用于治疗特定冠状病毒科的本发明的多肽或本发明的药物组合物或试剂盒,是与特定病毒表达的抗原片段序列匹配的那些多肽或药物组合物或试剂盒。技术人员可以基于本文提供的公开内容和实施例容易地鉴定和选择这样的多肽。[0082]通常用于疫苗接种和免疫疗法的多肽抗原、特别是衍生自多肽抗原的短肽,仅在一部分人类受试者中诱导免疫应答。本公开的肽被特别选择以在全球群体的大部分中诱导免疫应答。然而,由于hla基因型异质性,它们可能不是在所有个体中都有效。[0083]发明人发现,由个体表达的多种hla通常需要呈递相同的肽以触发t细胞应答。因此,预计对特定个体(pepi)具有免疫原性的多肽抗原(表位)的片段是那些可以结合由该个体表达的多种i类(激活的细胞毒性t细胞)或ii类(激活的辅助t细胞)hla的片段。一般而言,受试者中对特定疫苗肽的细胞毒性t细胞应答通过疫苗肽中存在≥1pepi3 (结合受试者的三个或更多个i类hla等位基因的表位)最佳地预测。辅助t细胞应答通常由≥1pepi3 或≥1pepi4 (结合受试者的三或更多个或四或更多个ii类hla等位基因的表位)最佳地预测。[0084]因此,本公开提供了预测人类受试者对施用如本文所述的多肽组或药物组合物将具有t细胞应答(细胞毒性和/或辅助)的方法。所述方法可以包括(a)(i)确定多肽组或药物组合物的活性成分多肽包括被受试者的至少三个hlai类分子所限制的t细胞表位;和(ii)预测受试者将对多肽组或药物组合物的施用具有细胞毒性(cd8 )t细胞应答;和/或(b)(i)确定多肽组或药物组合物的活性成分多肽包括被受试者的至少三个、或在一些情况下至少四个hlaii类分子所限制的t细胞表位;和(ii)预测受试者将对多肽组或药物组合物的施用具有辅助(cd4 )t细胞应答。[0085]本公开还提供了确定特定人类受试者对施用本文所述的多肽组或药物组合物将具有t细胞应答(细胞毒性/cd8 或辅助/cd4 )的可能性的方法,其中所述方法包括鉴定多肽或活性成分多肽中被受试者的至少三个i类hla或至少三个或至少四个ii类hla所限制的t细胞表位,并且其中(a)(a)被受试者的至少三个i类hla所限制的较高数量的t细胞表位;和/或[0086](b)既(i)被受试者的至少三个hlai类所限制;且(ii)为不同sars-cov-2结构蛋白的片段,的较高数量的t细胞表位,对应于受试者中细胞毒性/cd8 t细胞应答的较高概率;且/或(b)(a)被受试者的至少三个或至少四个ii类hla所限制的更高数量的t细胞表位;和/或(b)既(i)被受试者的至少三个或至少四个ii类hla所限制;且为(ii)不同sars-cov-2结构蛋白的片段,的更高数量的t细胞表位,对应于受试者中辅助t细胞/cd4 t细胞应答的较高概率。[0087]在一些情况下,可以预测受试者具有细胞毒性t细胞应答,或高于对肽组或药物组合物的施用具有细胞毒性t细胞应答的预定阈值概率,并且所述方法还包括选择或推荐施用药物组合物作为治疗受试者的方法,并且任选地还包括通过向受试者施用多肽组或药物组合物来治疗受试者。[0088]本公开还提供了如本文所述的治疗方法,其中已经预测接受治疗的受试者对使用本文所述的方法施用多肽组或药物组合物具有细胞毒性或辅助t细胞应答,或对使用本文所述的方法施用多肽组或药物组合物具有细胞毒性t或辅助细胞应答的预定阈值概率高。所述方法可包括使用本文所述的方法选择预测对特定受试者具有免疫原性的肽。包括为受试者选择的肽作为活性成分的试剂盒的药物组合物可被认为是为受试者个性化的(即个性化药物)。所述方法可进一步包括向受试者施用。[0089]发明人进一步发现,在疫苗或免疫治疗组合物中存在(i)对应于一或多个靶多肽抗原的片段,且(ii)可以结合个体的至少三个i类hla等位基因的至少二个t细胞表位,是临床反应的前兆。本文所用的“临床反应”或“临床益处”可以是预防或延迟疾病或病况的发作、改善一或多个症状、诱导或延长缓解、或延迟复发或再发或恶化、或受试者疾病状态的任何其它改善或稳定。临床反应还可以是预防由冠状病毒科病毒的不同突变变体引起的感染。[0090]因此,本公开的一些方面涉及预测特定人类受试者将对如本文所述的治疗方法或对施用如本文所述的肽组或药物组合物具有临床反应的方法,或确定临床反应的概率的方法。所述方法类似于本文所述的用于预测t细胞应答的方法,但是通过确定多肽组或药物组合物的活性成分多肽包括各自被受试者的至少三个hlai类分子所限制的二个不同的t细胞表位来预测临床应答。[0091]药物组合物、治疗方法和给药方式[0092]在一些方面,本公开涉及包括如本文所述的肽、多核酸、载体或细胞中的一或多个的药物组合物或试剂盒。这样的药物组合物或试剂盒可以用于诱导免疫应答、治疗、接种或向受试者提供免疫治疗的方法中。药物组合物或试剂盒可以是疫苗或免疫治疗组合物或试剂盒。本文所述的治疗方法可包括向受试者施用药物组合物。[0093]如本文所用的术语“活性成分”是指旨在诱导免疫应答的多肽,并且可以包含在施用给受试者后在体内产生的疫苗或免疫治疗组合物的多肽产物。对于dna或rna免疫治疗组合物,多肽可以由施用组合物的受试者的细胞在体内产生。对于基于细胞的组合物,多肽可以由组合物的细胞加工和/或呈递,例如自体树突细胞或用多肽脉冲的或包括编码多肽的表达构建体的抗原呈递细胞。药物组合物或试剂盒可以包括编码一或多个活性成分多肽的多核苷酸或细胞。[0094]除了一或多个肽、核酸、载体或细胞之外,本文所述的药物组合物或试剂盒可以包括药学上可接受的赋形剂、载体、稀释剂、缓冲剂、稳定剂、防腐剂、佐剂或本领域技术人员熟知的其它材料。这样的物质优选是无毒的,并且优选不干扰活性成分的药物活性。药物载体或稀释剂可以是,例如,含水溶液和水/油乳液。载体或其它材料的确切性质可取决于施用途径,例如口服、静脉内、皮肤或皮下、鼻、肌内、皮内、和腹膜内途径。[0095]本公开的药物组合物可以包括一或多个“药学上可接受的载体”。这些通常是大的、代谢缓慢的大分子,例如蛋白质、糖、聚乳酸、聚乙醇酸、聚合氨基酸、氨基酸共聚物、蔗糖(paoletti,2001,vaccine,19:2118–2126)、海藻糖(wo00/56365)、乳糖和脂质聚集体(例如油滴或脂质体)。这些载体是本领域普通技术人员公知的。药物组合物还可以含有稀释剂,例如水、盐水、甘油等。另外,可以存在辅助物质,例如润湿剂或乳化剂、ph缓冲物质等。无菌无热原的磷酸盐缓冲生理盐水是典型的载体(gennaro,2000,remington:scienceandpracticeofpharmacy,第20版,isbn:0683306472)。[0096]本公开的药物组合物可以是冻干的或水性形式,即,溶液或悬浮液。这种类型的液体制剂允许组合物直接以其包装形式给药,而不需要在水性介质中重构,因此对于注射是理想的。药物组合物可以存在于小瓶中,或者它们可以存在于已填充的注射器中。注射器可以具有或不具有针头。注射器包含单剂,而小瓶可包含单剂或多剂。[0097]本公开的液体制剂也适于从冻干形式重构其它药物。当药物组合物用于这种临时重构时,本公开提供了试剂盒,其可以包括二个小瓶,或可以包括一个预填充注射器和一个小瓶,其中注射器的内容物用于在注射前重构小瓶的内容物。[0098]本公开的药物组合物可以包含抗微生物剂,特别是当以多剂形式包装时。可以使用抗微生物剂,例如2-苯氧基乙醇或对羟基苯甲酸酯(对羟基苯甲酸甲酯、对羟基苯甲酸乙酯、对羟基苯甲酸丙酯)。任何防腐剂优选以低水平存在。防腐剂可以外源加入且/或可以是混合形成组合物的大量抗原的组分(例如,作为百日咳抗原中的防腐剂存在)。[0099]本公开的药物组合物可包括去污剂,例如吐温(聚山梨醇酯)、dmso(二甲亚砜)、dmf(二甲基甲酰胺)。去污剂通常以低含量存在,例如《0.01%,但也可以较高含量使用,例如0.01–50%。[0100]本公开的药物组合物可以包含钠盐(例如氯化钠)和溶液中的游离磷酸根离子(例如通过使用磷酸盐缓冲液)。[0101]在某些实施方式中,药物组合物可以被包封在合适的载体中,以将肽递送到抗原呈递细胞中或增加稳定性。如本领域技术人员将理解的,多种载体适于递送本公开的药物组合物。合适的结构化流体递送系统的非限制性实例可包含纳米颗粒、脂质体、微乳液、胶束、树枝状聚合物和其它含磷脂的系统。将药物组合物掺入递送载体的方法是本领域已知的。[0102]为了增加组合物的免疫原性,药理学组合物可以包括一或多个佐剂和/或细胞因子。[0103]合适的佐剂包含铝盐,例如氢氧化铝或磷酸铝,但也可以是钙盐、铁盐或锌盐,或者可以是酰化酪氨酸、或酰化糖的不溶性悬浮液,或者可以是阳离子或阴离子衍生的糖、聚磷腈、生物可降解微球、单磷酰脂质a(mpl)、脂质a衍生物(例如毒性降低的)、3-o-脱酰mpl(3d-mpl)、quila、皂苷、qs21、弗氏不完全佐剂(密歇根州底特律的difcolaboratories)、merck佐剂65(新泽西州rahway的merckandcompany,inc.)、as-2(宾夕法尼亚州费城的smith-klinebeecham)、cpg寡核苷酸、生物粘合剂和粘膜粘合剂、微粒、脂质体、聚氧乙烯醚制剂、聚氧乙烯酯制剂、胞壁酰肽或咪唑并喹诺酮化合物(例如咪喹莫特及其同系物)。在本发明公开内容中适合用作佐剂的人免疫调节剂包含细胞因子,例如白介素(例如il-1、il-2、il-4、il-5、il-6、il-7、il-12等)、巨噬细胞集落刺激因子(m-csf)、肿瘤坏死因子(tnf)、粒细胞、巨噬细胞集落刺激因子(gm-csf)也可用作佐剂。[0104]在一些实施方式中,组合物包括佐剂,其选自由montanideisa-51(美国新泽西州fairfield的seppic,inc.)、qs-21(美国马萨诸塞州lexington的aqualiabiopharmaceuticals,inc.)、gm-csf、环磷酰胺、卡介苗(bcg)、短小棒状杆菌、左旋咪唑、azimezone、异丙肾上腺素、二硝基氯苯(dncb)、匙孔血蓝蛋白(klh)、弗氏佐剂(完全和不完全)、矿物凝胶、氢氧化铝(明矾)、溶血卵磷脂、普朗尼克多元醇、聚阴离子、油乳剂、二硝基苯酚、白喉毒素(dt),组成的组。在一个具体实施方式中,佐剂是montanide佐剂。[0105]例如,细胞因子可以选自由转化生长因子(tgf)例如但不限于tgf-α和tgf-β;胰岛素样生长因子i和/或胰岛素样生长因子ii;红细胞生成素(epo);骨诱导因子;干扰素例如但不限于干扰素α、干扰素β、和干扰素γ;集落刺激因子(csf)例如但不限于巨噬细胞-csf(m-csf)、粒细胞-巨噬细胞-csf(gm-csf)、和粒细胞-csf(g-csf),组成的组。在一些实施方式中,细胞因子选自由神经生长因子例如ngf-β;血小板生长因子;转化生长因子(tgf)例如但不限于tgf-α和tgf-β;胰岛素样生长因子i和胰岛素样生长因子ii;红细胞生成素(epo);骨诱导因子;干扰素(ifn)例如但不限于ifn-α、ifn-β、和ifn-γ;集落刺激因子(csf)例如巨噬细胞-csf(m-csf)、粒细胞-巨噬细胞-csf(gm-csf)、和粒细胞-csf(g-csf);白细胞介素(il)例如但不限于il-1、il-1α、il-2、il-3、il-4、il-5、il-6、il-7、il-8、il-9、il-10、il-11、il-12、il-13、il-14、il-15、il-16、il-17、il-18;lif;kit-配体或flt-3;制管张素;血小板反应蛋白;内皮抑素;肿瘤坏死因子(tnf);和lt,组成的组。[0106]预期佐剂或细胞因子可以以每剂约0.01mg至约10mg的量加入,优选以每剂约0.2mg至约5mg的量加入。或者,佐剂或细胞因子的浓度可以是约0.01至50%,优选约2%至30%。[0107]在某些方面,本公开的药物组合物是根据已知技术通过在适当的无菌条件下将佐剂和/或细胞因子与本文所述的肽物理混合以产生最终产物来制备的。[0108]多肽片段的合适组合物和施用方法的实例在esseku和adeye(2011)和vandenmooterg.(2006)中提供。疫苗和免疫疗法组合物的制备一般在vaccinedesign(“thesubunitandadjuvantapproach”(edspowellm.f.&newmanm.j.(1995)plenumpressnewyork)中描述。fullerton在美国专利4,235,877中描述了在脂质体内的封装,这也是设想中的封装。[0109]在一些实施方式中,本文公开的组合物被制备为(核糖)核酸疫苗。在一些实施方式中,核酸疫苗是dna疫苗。在一些实施方式中,dna疫苗或基因疫苗包括具有启动子和适当转录和翻译控制元件的质粒以及编码一或多个本公开内容的多肽的核酸序列。在一些实施方式中,质粒还包含增强例如表达水平、细胞内靶向或蛋白酶体加工的序列。在一些实施方式中,dna疫苗包括含有编码一或多个本公开内容的多肽的核酸序列的病毒载体。在另外的方面,本文公开的组合物包括一或多个编码被确定与生物样品具有免疫反应性的肽的核酸。例如,在一些实施方式中,组合物包括编码1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、或更多个肽的一或多个核苷酸序列,所述肽包括作为能够结合患者的至少三个hlai类分子和/或至少三个或四个hlaii类分子的t细胞表位的片段。在一些实施方式中,dna或基因疫苗还编码免疫调节分子以操纵所产生的免疫应答,例如增强疫苗的效力、刺激免疫系统或降低免疫抑制。增强dna或基因疫苗免疫原性的策略包含编码异种形式的抗原,将抗原与激活t细胞或触发联合识别的分子融合,用dna载体引发,然后用病毒载体加强,以及利用免疫调节分子。在一些实施方式中,通过针、基因枪、气溶胶注射器、贴片、微针、研磨等形式引入dna疫苗。在一些形式中,dna疫苗被掺入脂质体或其它形式的纳米体中。在一些实施方式中,dna疫苗包含选自由转染剂;鱼精蛋白;鱼精蛋白脂质体;多糖颗粒;阳离子纳米乳液;阳离子聚合物;阳离子聚合物脂质体;阳离子纳米颗粒;阳离子脂质和胆固醇纳米颗粒;阳离子脂质、胆固醇、和peg纳米颗粒;树枝状聚合物纳米颗粒,组成的组。在一些实施方式中,dna疫苗通过吸入或摄取施用。在一些实施方式中,将dna疫苗引入血液、胸腺、胰腺、皮肤、肌肉、肿瘤或其它部位。[0110]在一些实施方式中,本文公开的组合物被制备为rna疫苗。在一些实施方式中,rna是非复制性mrna或病毒衍生的自扩增rna。在一些实施方式中,非复制mrna编码本文公开的肽,并含有5′和3′非翻译区(utr)。在一些实施方式中,病毒衍生的自扩增rna不仅编码本文公开的肽,而且编码能够使细胞内rna扩增和大量蛋白表达的病毒复制机制。在一些实施方式中,rna被直接引入个体。在一些实施方式中,rna是化学合成的或体外(invitro)转录的。在一些实施方式中,使用t7、t3或sp6噬菌体rna聚合酶从线性dna模板产生mrna,所得产物含有编码本文公开的肽的开放阅读框、侧翼utr、5′帽和聚腺苷酸尾。在一些实施方式中,在使用痘苗病毒加帽酶或通过掺入合成的帽或抗反向帽类似物的转录反应期间或之后加入各种形式的5′帽。在一些实施方式中,将最佳长度的poly(a)尾直接从编码dna模板或通过使用poly(a)聚合酶加到mrna上。rna可以编码一或多个,包括能够结合患者的至少三个i类hla和/或至少三个或四个ii类hla分子的t细胞表位的片段,的肽。所述片段来源于在冠状病毒科中表达的抗原。在一些实施方式中,rna包含增强稳定性和翻译的信号。在一些实施方式中,rna还包含非天然核苷酸以增加半衰期,或修饰的核苷以改变免疫刺激特征。在一些实施方式中,通过针、基因枪、气溶胶注射器、贴片、微针、研磨等形式引入rna。在一些形式中,rna疫苗被掺入脂质体或其它形式的纳米抗体中,该纳米抗体促进rna的细胞摄取并保护其免于降解。在一些实施方式中,rna疫苗包含递送系统,其选自由转染剂;鱼精蛋白;鱼精蛋白脂质体;多糖颗粒;阳离子纳米乳液;阳离子聚合物;阳离子聚合物脂质体;阳离子纳米颗粒;阳离子脂质和胆固醇纳米颗粒;阳离子脂质、胆固醇、和peg纳米颗粒;树枝状聚合物纳米颗粒;和/或裸mrna;体内电穿孔的裸mrna;鱼精蛋白复合的mrna;与带正电荷的水包油阳离子纳米乳液缔合的mrna;与化学修饰的树枝状聚合物相关并与聚乙二醇(peg)–脂质复合的mrna;peg–脂质纳米颗粒中的鱼精蛋白复合的mrna;与阳离子聚合物如聚乙烯亚胺(pei)结合的mrna;与阳离子聚合物如pei和脂质组分结合的mrna;与多糖(例如壳聚糖)颗粒或凝胶结合的mrna;阳离子脂质纳米颗粒(例如,1,2-二油酰氧基3-三甲基铵丙基丙酮(dotap)或二油酰磷脂酰乙醇胺(dope)脂质)中的mrna;与阳离子脂质和胆固醇复合的mrna;或与阳离子脂质、胆固醇和peg–脂质复合的mrna,组成的组。在一些实施方式中,rna疫苗通过吸入或摄取施用。在一些实施方式中,rna被引入血液、胸腺、胰腺、皮肤、肌肉、肿瘤、或其他部位,和/或通过皮内、肌内、皮下、鼻内、结节内、静脉内、脾内、肿瘤内或其他递送途径引入。[0111]多核苷酸或寡核苷酸组分可以是裸露的核苷酸序列或与阳离子脂质、聚合物或靶向系统组合。它们可以通过任何可用的技术来递送。例如,多核苷酸或寡核苷酸可以通过针注射导入,优选皮内、皮下或肌内注射。或者,多核苷酸或寡核苷酸可以使用递送装置,例如颗粒介导的基因递送,直接递送穿过皮肤。多核苷酸或寡核苷酸可局部施用于皮肤或粘膜表面,例如通过鼻内、口服、或直肠内施用。[0112]核酸构建体的摄取可通过几种已知的转染技术来增强,例如那些包含使用转染剂的技术。这些试剂的实施例包含阳离子试剂,例如,磷酸钙和deae-dextran和脂质转染试剂,例如lipofectam和transfectam。可以改变待施用的多核苷酸或寡核苷酸的剂量。[0113]因此,本发明提供了疫苗或药物组合物或试剂盒,其包括编码一或多个、或至少一个(或至少2、3、4、5、6、7、8、9或10个)选自seqidno:1至17的多肽序列的,一或多个多核苷酸(多核酸)或多核糖核苷酸(核糖多核酸)。所述的多核苷酸或核糖多核苷酸可编码一或多个冠状病毒科蛋白、或β-冠状病毒科科蛋白、sars-cov-2蛋白、或sars-cov蛋白的一或多个片段,或任何冠状病毒科的表达一或多个蛋白的包括一或多个(或2、或3、4、5、6、7、8、9、或10或更多个)选自seqidno:1至17的氨基酸序列的蛋白。所述片段包括选自seqidno:1至17的氨基酸序列,并且通常长度至多50个氨基酸。多核苷酸或多核糖核苷酸可以包括至少一个(或至少2、3、4、5、6、7、8、9或10个)选自seqidno:234至267的序列。通常,多核苷酸或多核糖核苷酸一起包括选自seqidno:234至250或251至267的序列,所述序列编码选自seqidno:1–17的氨基酸序列。更典型地,所述编码选自编码seqidno:1–17的不同氨基酸序列的多核苷酸或核糖多核苷酸,是由冠状病毒科表达的不同蛋白质的片段。例如,多核苷酸或核糖多核苷酸可一起包括至少一个选自以下组中的至少二组、至少三组或所有四组中的每一组的序列:(a)seqidno:234至244或seqidno:251至261;(b)seqidno:245至248或seqidno:262至265;(c)seqidno:249或seqidno:266;和(d)seqidno:250或seqidno:267。多核苷酸或核糖多核苷酸可一起包括以下列表之一中的至少一个(或至少2、3、4、5、6、7、8、9个或全部)序列:seqidno:236、238、242、245、246、247、248、249和250;seqidno:236、238、240、242、245、246、247、248、249和250;seqidno:239、242、243、244、245、246、247、248、249和250;seqidno:252、255、259、262、263、264、265、266和267;seqidno:252、255、257、259、262、263、264、265、266和267;和seqidno:256、259、260、261、262、263、264、265、266和267。多核苷酸或核糖多核苷酸可以编码本文所述的本发明的任何多肽组。多核苷酸可以是dna。多核糖核苷酸可以是rna。例如,多核糖核苷酸可以是mrna。[0114]本发明还包括基于细胞的组合物。一或多个多肽或多肽组存在于细胞表面,特别是在施用后存在于患者体内。在一些情况下,细胞可以是(自体)树突细胞或抗原呈递细胞。细胞可以用多肽冲击,或者包括编码多肽的一或多个表达构建体/盒。表达构建体/表达盒可包括/表达上文所述的任何多核苷酸或核糖多核苷酸。[0115]如本文所用,术语“治疗”包含治疗性和预防性治疗。给药通常是“预防有效量”或“治疗有效量”(视情况而定,尽管预防可以被认为是治疗),这足以导致临床反应或对个体显示临床益处,例如,有效量预防或延迟疾病或病况的发作,改善一或多个症状,诱导或延长缓解,或延迟复发或再发。[0116]剂可以根据各种参数确定,尤其是根据所用的物质;待治疗个体的年龄、体重和状况;给药途径;以及所需的方案。每剂中的抗原量选择为诱导免疫应答的量。医师将能够确定任何特定个体所需的给药途径和剂量。剂可以作为单剂提供,或者可以作为多剂提供,例如以规则间隔服用,例如每小时施用2、3或4剂。典型地,肽、多核苷酸或寡核苷酸的给药范围典型地为1pg至1mg、更典型地为1pg至10μg,用于颗粒介导的递送,以及1μg至1mg、更典型地为1–100μg、更典型地为5–50μg,用于其它途径。通常,预期每剂将包括0.01–3mg抗原。特定疫苗的最佳量可以通过涉及观察受试者中的免疫应答的研究来确定。[0117]上述技术和方案的实例可以在remington'spharmaceuticalsciences,20thedition,2000,pub.lippincott,williams&wilkins中找到。[0118]在一些情况下,治疗方法可包括向受试者施用多于一个的肽、多核酸或载体。这些可以一起/同时和/或在不同时间或顺序施用。使用任选靶向不同抗原的不同肽的组合,对于克服个体的病毒异质性和hla异质性的挑战可能是重要的。组合使用本公开的肽扩大了可经历来自疫苗接种的临床益处的个体的组。为在一个方案中使用而制造的多种药物组合物可以定义药物产品。在一些情况下,可以在例如1年、或6个月、或3个月、或60或50或40或30天的时间段内向受试者施用单次治疗的不同的肽、多核酸或载体。[0119]给药途径包含但不限于鼻内、口服、皮下、皮内、和肌内。特别优选皮下给药。皮下给药可以例如通过注射到腹部、上臂或大腿的侧面和前面、背部的肩胛区域、或上腹侧臀部区域。[0120]本公开的组合物还可以以一、或多剂,以及,通过其他施用途径施用。例如,这些其它途径包含皮内、静脉内、血管内、动脉内、腹膜内、鞘内、气管内、心内、叶内、髓内、肺内、和阴道内。根据所需的治疗持续时间,根据本公开的组合物可以一次或多次施用,也可以间歇施用,例如每月施用数月或数年,并且以不同的剂量施用。[0121]用于口服给药的固体剂型包含胶囊、片剂、囊片、丸剂、粉剂、丸剂、和颗粒剂。在这样的固体剂型中,活性成分通常与一或多个药学上可接受的赋形剂组合,其例子如上详述。口服制剂也可以作为水性悬浮液、酏剂或糖浆施用。为此,活性成分可以与各种甜味剂或调味剂、着色剂、以及如果需要的话,乳化剂和/或悬浮剂、以及稀释剂如水、乙醇、甘油及其组合,来结合。[0122]本公开的一或多个组合物的施用、或根据本公开的用于治疗的方法和用途的进行,可以单独,或与其他药理学组合物或治疗,例如其他免疫疗法、疫苗或抗病毒剂组合。其它治疗性组合物或治疗可以与本公开的组合物或治疗同时或相继(之前或之后)施用。[0123]在一些情况下,治疗方法是疫苗接种方法或提供免疫治疗的方法。如本文所用,“免疫疗法”是通过诱导或增强个体中的免疫应答来治疗疾病或病况。在某些实施方式中,免疫治疗是指包括向个体施用一或多个药物以引发t细胞应答的治疗。在一个具体的实施方式中,免疫治疗是指一种治疗,其包括向个体施用或表达含有一或多个pepi的多肽,以引发t细胞应答,从而识别并杀死在其细胞表面展示与i类hla结合的一或多个pepi的细胞。在另一个实施方式中,免疫治疗是指一种治疗,其包括向个体施用或表达含有由ii类hla呈递的一或多个pepi的多肽,以引发辅助t细胞应答,从而向识别并杀死在其细胞表面展示与i类hla结合的一或多个pepi的患病细胞的细胞毒性t细胞提供共刺激。在更另一个具体实施方式中,免疫治疗是指包括向个体施用一或多个药物的治疗,所述药物再活化存在的t细胞以杀死靶细胞和/或病毒。[0124]本发明包括治疗或预防受试者中的冠状病毒科感染或与冠状病毒科感染相关的疾病或病况的方法。与冠状病毒科感染相关的疾病或状况包含由感染引起的,例如直接由感染引起的任何疾病或病况、症状或其他疾病属性。[0125]在一些情况下,冠状病毒科是β-冠状病毒科,例如sars-cov-2或其变体或突变菌株。冠状病毒科可以是sars-cov。具体地,冠状病毒科表达一或多个抗原/多肽,所述抗原/多肽包括选自seqidno:1至17所示的一或多个氨基酸序列,或一或多个在表6a中以粗体和/或下划线显示的最佳epi序列。更具体地,特定的冠状病毒科可使用组合物或试剂盒来治疗,其中活性成分多肽包括选自seqidno:1至17(或最佳epi序列)的一或多个(通常是2或更多个,或3、4、5、6、7、8、或9或更多个)序列,这些序列在特定病毒表达的抗原中发现。本文描述了特别适合于或优化了的用于治疗或预防由sars-cov-2或sars-cov感染引起的疾病的特定组合物。然而,技术人员能够使用本公开内容来确定其他组合物或试剂盒,这些组合物或试剂盒具有包括seqidno:1至17的氨基酸序列的不同组合的多肽作为活性成分,用于预防或治疗其他冠状病毒科。可以如上文和下文实施例中所述选择用于特定冠状病毒科和/或患者的合适治疗。[0126]公开内容的进一步实施方式——(1)[0127]1.一种多肽疫苗,其包括包括选自由seqidno:1至17组成的组的氨基酸序列的多肽,以及药学上可接受的佐剂、稀释剂、载体、防腐剂、赋形剂、缓冲剂、稳定剂或其组合。[0128]2.项1所述的多肽疫苗,其包括二或更多个多肽,每个多肽包括选自由seqidno:1至17组成的组的不同氨基酸序列。[0129]3.项1所述的多肽疫苗,其包括来自以下组中的至少二组的至少一个多肽:[0130](a)seqidno:1至11;[0131](b)seqidno:12至15;[0132](c)seqidno:16;和[0133](d)seqidno:17。[0134]4.项1所述的多肽疫苗,其包括至少二个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0135]5.项1所述的多肽疫苗,其包括至少四个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0136]6.项1所述的多肽疫苗,其包括至少六个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0137]7.项1所述的多肽疫苗,其包括至少八个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0138]8.项1所述的多肽疫苗,其包括至少十个多肽,其中每个多肽包括seqidno:2、5、7、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:2、5、9、12、13、14、15、16、和17的氨基酸序列中的不同的一个,或其中每个多肽包括seqidno:6和9至17的氨基酸序列中的不同的一个。[0139]9.项1所述的多肽疫苗,其中所述多肽中的一或多个包括,是被所述个体的至少二个hlai类等位基因所限制的cd8 t细胞表位的,冠状病毒科蛋白的片段。[0140]10.项1所述的多肽,其中所述多肽中的一或多个包括,是被所述个体的至少二个hlaii类等位基因所限制的cd4 t细胞表位的,冠状病毒科蛋白的片段。[0141]11.项1所述的多肽,其中所述多肽中的一或多个包括线性b细胞表位。[0142]12.一种在有需要的个体中治疗或预防冠状病毒科感染的方法,所述方法包括向所述个体施用项1的多肽疫苗。[0143]13.项12所述的方法,其中所述冠状病毒科感染是sars-cov-2感染。[0144]公开内容的进一步实施方式——(2)[0145]1.一种免疫原性组合物,其包括(a)至少二个不同的多肽,每个多肽由至少30个氨基酸且不超过60个氨基酸组成,并且包括选自由seqidno:1至17组成的组的氨基酸序列,和(b)增加所述多肽的免疫原性的药学上可接受的化合物。[0146]2.项1的免疫原性组合物,其中所述组合物包括:[0147](a)至少一个不同的多肽,其由至少30个氨基酸且不超过60个氨基酸残基组成,且包括选自seqidno:1至11的氨基酸序列;[0148](b)至少一个不同的多肽,其由至少30个氨基酸且不超过60个氨基酸组成,且包括选自seqidno:12至15的氨基酸序列;[0149](c)至少一个不同的多肽,其由至少30个氨基酸且不超过60个氨基酸组成,且包括选自seqidno:16的氨基酸序列;和[0150](d)至少一个不同的多肽,其由至少30个氨基酸且不超过60个氨基酸组成,且包括选自seqidno:17的氨基酸序列。[0151]3.项1的免疫原性组合物,其中所述不同的氨基酸序列选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组。[0152]4.项3的免疫原性组合物,其中所述氨基酸序列选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组。[0153]5.项1的免疫原性组合物,其中所述组合物包括六个不同的多肽,每个多肽由至少30个氨基酸且不超过60个氨基酸组成,且包括选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组的不同的氨基酸。[0154]6.项1的免疫原性组合物,其中所述组合物包括八个不同的多肽,每个多肽由至少30个氨基酸且不超过60个氨基酸组成,且包括选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组的不同的氨基酸。[0155]7.项1的免疫原性组合物,其中所述组合物包括十个不同的多肽,每个多肽由至少30个氨基酸且不超过60个氨基酸组成,且包括选自由seqidno:2、5、7、9、12、13、14、15、16、和17组成的组的不同的氨基酸。[0156]8.项1的免疫原性组合物,其中所述至少一个多肽包括,是被个体的至少二个hlai类等位基因所限制的cd8 t细胞表位的,冠状病毒科蛋白质的片段。[0157]9.项1的免疫原性组合物,其中所述至少一个多肽包括,是被个体的至少二个hlaii类等位基因所限制的cd4 t细胞表位的,冠状病毒科蛋白质的片段。[0158]10.项1的免疫原性组合物,其中所述至少一个多肽包括线性b细胞表位。[0159]11.一种在有需要的个体中刺激针对sars-cov-2感染的免疫应答的方法,包括向所述个体施用项1的免疫原性组合物。[0160]12.项1的免疫原性组合物,其中所述组合物包括由根据seqidno:2的序列组成的多肽、由根据seqidno:5的序列组成的多肽、由根据seqidno:7的序列组成的多肽、由根据seqidno:9的序列组成的多肽、由根据seqidno:12的序列组成的多肽、由根据seqidno:13的序列组成的多肽、由根据seqidno:14的序列组成的多肽、由根据seqidno:15的序列组成的多肽、由根据seqidno:16的序列组成的多肽、和由根据seqidno:17的序列组成的多肽。[0161]实施例[0162]实施例1——polypepi1018肽疫苗在结肠直肠癌患者中的安全性[0163]如美国专利10,213,497中所述,已经设计了结肠直肠癌疫苗polypepi1018以优化pepi3 群体。用polypepi1018接种是安全的和耐受良好的。如所预期的,与接种疫苗相关的大多数不良事件是注射部位反应和轻度流感样症状。只有一个三级严重不良事件被记录为可能与治疗有关(非感染性脑炎),然而,安全审查小组和医疗监督员都将该事件归为非相关事件。表2收集了在试验中记录为与接种疫苗相关的不良事件。[0164]表2在11名患者中记录的与接种疫苗相关或可能相关的不良事件。[0165][0166]*凸起的红斑、皮下结节、皮下结节后臂和大腿[0167]实施例2——polypepi1018疫苗在结肠直肠癌患者中的免疫原性[0168]11名患者中的十名有足够的pmbc样本,可以用免疫分析法进行分析。十名患者中的七名预先存在针对至少一个polypepi1018疫苗抗原的免疫应答,并且十名患者中的七名针对至少一个疫苗抗原加强了免疫应答。在十名患者中的八名中,诱导针对至少一个疫苗抗原的重新免疫应答(图1a)。发现了针对所有七个靶抗原的预先存在的免疫应答,这证实了疫苗设计策略和靶抗原选择。所有十名患者都具有疫苗特异性cd4t细胞应答,其中九名在接种后加强应答(图1b)。[0169]八名患者具有从接种前至第12周获得的样品以分析免疫应答的动力学。患者在整个时间过程中表现出不同的动力学模式,其中大多数人在第3周 第6周的合并时间点上出现了一个峰值(图1c),这与病毒再攻击后免疫应答动力学的观察结果相一致。nascimbeni等人在黑猩猩身上发现,针对大多数hcv特异性试验肽的cd8t细胞反应的第一个峰值是在静脉内再攻击后5–6周,以及hcv肝内再攻击后3–4周左右测量到的。(nascimbenietal.,jvirol,77,4781–93.2003)。[0170]除了富集elispot测定之外,我们还进行了直接离体elispot和定量细胞内细胞因子染色(ics)测定,使用流式细胞术作为检测方法来研究效应型t细胞应答。在接种前样品中,离体elispot在九位分析患者中的三个中发现阳性疫苗特异性cd8应答、一个中发现cd4应答。由于接种疫苗,五名患者的cd8反应和七名患者的cd4反应得到了增强或新诱导(表3)。在cd8应答被加强或从头诱导的所有五位患者中,接种也诱导了cd4应答。如通过离体cs所测量的,疫苗特异性cd8和cd4t细胞应答是多功能的,并且接种后功能性cd8和cd4细胞的频率增加(表3)。在cd8t细胞级分中,tnf-α阳性细胞占优势,而在cd4池中检测到的最常见细胞因子是il-2(图2)。[0171]表3离体检测的疫苗特异性效应t细胞应答和肿瘤浸润淋巴细胞。“ ”代表加强:预先存在的响应≥2x。ics百分比显示为疫苗特异性ifn-γ、il-2、tnf-α单或双阳性t细胞频率的总和。nt——未测试[0172][0173][0174]11名患者中有四名有足够的肿瘤样本来分析肿瘤浸润性淋巴细胞(til)。对于四个试验患者中的三个,接种诱导til募集到侵入性边缘和核心肿瘤区域。对于接种后经历临床益处的二个患者,cd8t细胞在核心肿瘤中积累(表3),表明疫苗能够将冷肿瘤转变为热肿瘤。在图3中显示了患者010007活检接种前和接种后(38周)的ihc照片,其中cd8 til增加超过200%。[0175]实施例3——polypepi1018疫苗在结肠直肠癌患者中的效力[0176]在11个患者中,根据recist,三个患者具有客观的肿瘤应答。在只接受单剂的患者中(n=6),到第12周时有1人获得部分反应,2人病情稳定,导致17%的客观应答率(orr)和50%的疾病控制率(dcr)(图4a和4b)。在接受多剂治疗的亚组中,二名患者获得了部分应答,三名患者病情稳定,也就是40%的orr和80%的dcr。单剂和多剂组的中值pfs(包含试验后随访数据)分别为4.5个月和12.2个月(p=0.03)(图4c)。相比之下,在modul试验中5-fu加上贝伐单抗维持疗法与相当的患者群体(n=148)的mpfs为7.39个月。(grotheyetal.,annalsofoncology29,2018)[0177]患者020004在一线化疗期间是部分应答者。在接受单剂疫苗后,他继续缓解,并且接种后6周,他的靶损伤大小减少超过30%(图5a)。这个结果表明,所实现的部分应答可能部分地对诱导阶段有贡献。010004号患者在诱导期病性稳定,在第6周(第一次接种后6周)已经获得部分应答。在进一步接种的整个过程中肿瘤缩小继续,三个靶病变中的二个在第24周完全消失,在第26周进行治愈性手术以除去第三个剩余的病变(图5b)。患者010007在一线治疗中病情稳定后,在接种疫苗期间具有非常缓慢的缓解,具有轻微的斜率,并且仅在其第38周的最后访问时获得部分应答(图5c)。与患者010004类似,靶损伤的缩小也允许对该患者进行根治性手术,并且病理分析未在原发性肿瘤中发现癌细胞并且在肝脏中未发现残余转移。除了后三位患者出现客观应答外,患者010002也必须强调。他取得了稳定的病情(19%的靶病变缩小)作为最佳反应,反应时间为53周(~12个月)。对该患者和具有客观应答的患者的延长的肿瘤应答提示polypepi1018疫苗的抗肿瘤活性。[0178]实施例4pepi测验的回顾性和前瞻性验证[0179]开发pepi测验以预测受试者的t细胞应答。(tokeetal.,journalofclinicaloncology37,2019)pepi试验鉴定了受试者的抗原特异性个人表位(pepi),其结合受试者的至少三个hlai类等位基因。pepi试验的输入是受试者的6个hlai类等位基因和所讨论的抗原的氨基酸序列。用重叠的9聚体肽扫描抗原以鉴定结合受试者hlai类等位基因的肽。pepi试验从表位数据库(epdb)中获得hla–肽对,该数据库是通过纳入结合截止值≤5的肽(iedbpercentilerank)来组装的。根据6项临床试验的免疫原性数据对pepi试验的回顾性验证,确定的pepi引起免疫反应的概率为84%。(表4)。通过对71例癌症和9例hiv感染患者进行的六次临床试验的回顾性分析进行了性能评价研究(验证)。(bagarazzietal.,scitranslmed4,2012,bioleyetal.,clincancerres15,2009,gudmundsdotteretal.,vaccine29,2011,kakimietal.,intjcancer129,2011,valmorietal.,procnatlacadsciusa104,2007,wadaetal.,jimmunother37,2014,yuanetal.,procnatlacadsciusa108,2011)我们通过随机化可获得的患者数据创建了研究组群,并且由于除了数据可获得性之外的原因不排除任何患者。我们没有考虑从原始临床试验中排除,因为我们的研究不是旨在回顾性地分析这些临床试验。我们没有获得任何患者的个人数据。相反,我们使用患者标识符,如同行综述出版物中公开的,以及它们的hla基因型。抗原序列获自公开可获得的蛋白质序列数据库或同等的综述出版物。用标准随机数发生器将源自涉及80名患者的6个临床试验的可用的157个数据集随机化,以创建用于训练和验证研究的二个独立组群。训练和验证组群涉及相同患者群体的不同数据集。48名患者的76个数据集包含在训练组群中,51名患者的81个数据集包含在验证组群中。使用训练数据组,我们确定pepi计数≥1作为预测免疫应答的截止值。[0180]表4pepi试验的回顾性验证(n=81)。通过将pepi试验结果(如果pepi计数≥1为阳性)与在临床试验中通过生物测定法测量的抗原特异性ctl应答比较,获得诊断性能特征:真阳性(a):46,真阴性(d):11,假阳性(b):9,假阴性(c):15。[0181][0182]用于设计polypepi1018疫苗的pepi试验的计算机模拟工具使用符合免疫分析条件的10名患者的免疫原性数据进行了前瞻性验证。70个数据集(7个目标抗原×10个病人)被用来评估pepi试验预测抗原特异性ctl反应的能力。对于每个数据集,确定pepi试验是否能够预测免疫应答。总一致性百分比为64%,阳性预测值为79%,表示具有预测的pepi的患者将产生针对被分析的抗原的cd8t细胞特异性免疫应答的概率(表5)。临床试验数据与回顾性试验结果显著相关(p=0.01)。[0183]表5pepi试验的前瞻性验证。预测的和elispot测量的匹配结果用灰色突出显示。tp:真阳性,tn:真阴性,fp:假阳性,fn:假阴性,opa:整体一致性,ppv:阳性预测值,npv:阴性预测值。[0184][0185]实施例5可能的伴随诊断的可行性的证明(cdx)[0186]oberto-101试验的一个目的是确定一种生物标记,该生物标记除了pepi试验预测的免疫原性之外,还预期预测临床功效。从文献中已经清楚,所测量的免疫应答不能与recist所测量的临床应答直接相关,然而已经发现多抗原免疫应答率和癌症疫苗临床试验的客观应答率之间的相关性(klebanoffetal.,immunologicalreviews239,2011,lorinczetal.,annalsofoncology30,2019)。候选生物标记物可以是agp(具有pepi的抗原),其不仅考虑疫苗中包含的靶抗原的基数,而且考虑每种疫苗抗原的表达概率。受试者的agp计数指示疫苗能够用pepi“命中”的抗原的预期数目。在oberto-101研究的多剂组中,我们研究了agp作为潜在的生物标记物,并发现与肿瘤体积减小和pfs皆相关的趋势(图6a和b)。由于低样本数,不能确定显著性。我们也发现了与所测量的多抗原免疫应答类似的相关模式(图6c和d)。[0187]实施例6——polypepi-scov-2疫苗设计[0188]sars-cov基因组的大小为~30千碱基,像其它冠状病毒一样,编码多个结构和非结构蛋白。结构蛋白包含刺突蛋白(s)、包膜蛋白(e)、膜蛋白(m)、和核壳蛋白(n)。[0189]本文公开的polypepi-scov-2疫苗由一或多个30个氨基酸长的肽组成,该肽能够在全球群体的高比例的个体中诱导阳性的、期望的t细胞(cd8细胞毒性的和cd4辅助细胞二者)应答和b细胞介导的针对一或多个、并优选所有4种结构病毒抗原的抗体应答。[0190]2020年3月28日,从ncbi数据库(https://www.ncbi.nm.nih.gov/genome/genomes/86693)中共下载了19个covid-19的全基因组序列。登录号id如下:nc_045512.2、mn938384.1、mn975262.1、mn985325.1、mn988713.1、mn994467.1、mn994468.1、mn997409.1、mn988668.1、mn988669.1、mn996527.1、mn996528.1、mn996529.1、mn996530.1、mn996531.1、mt135041.1、mt135043.1、mt027063.1、mt027062.1。[0191]第一个id代表genbank参考序列。将翻译的编码序列的四个结构蛋白序列(表面糖蛋白、包膜蛋白、膜糖蛋白、核衣壳磷蛋白)进行比对,并与多序列比对进行比较。在19个序列中,15个是完全相同的。然而,我们在4个核壳蛋白中获得了单个氨基酸变化。这些替换如下:mn988713.1:核衣壳194s→x,mt135043.1:核衣壳343d→v,mt027063.1:核衣壳194s→l,mt027062.1:核衣壳194s→l。这些变化都不影响本疫苗多肽中被选为目标的表位。[0192]从目前已知的sars-cov-2结构蛋白的病毒抗原序列的保守区中选择十七个肽片段。选择片段以使模型群体中的多hlai类结合pepi3 和多hlaii类结合pepi4 ,即共享的个人表位,最大化。这些多肽还被设计成包含线性b细胞表位。具体地,选择在模型群体中最高比例受试者中的四个靶抗原pepi2 的保守区中的9聚体序列。这些9聚体被扩展到包含靶抗原的保守序列中的附近线性b细胞表位。然后选择同时包含9聚体“最佳epi”和线性b细胞表位二者的靶抗原的30聚体片段,以使模型群体中30聚体片段中具有hlaii类结合的pepi4 的受试者比例最大化。该模型群体包括从骨髓移植生物库获得的~16,000个hla基因分型的受试者,具有来自16个不同族群每个的约1,000个受试者。在模型群体中比例最高的受试者中,选定的30聚体肽片段和hlai类结合表位是pepi3 ,hlaii类结合表位是pepi4 ,其序列见表6a。[0193]表6apolypepi-scov-2肽序列列表。粗体/斜体:9聚体hlai最佳epi序列,下划线:15聚体最佳epi序列。[0194][0195]*含有肽的b细胞表位,b细胞表位列于表6b[0196]表6b线性b细胞表位[0197][0198][0199]参考文献:preliminaryidentificationofpotentialvaccinetargetsforthecovid-19coronavirus(sars-cov-2)basedonsars-covimmunologicalstudies。表4。来自s的sars-cov衍生的线性b细胞表位(23;其中20个位于亚基s2中)和n(22)蛋白在sars-cov-2中相同(总共45个表位)。[0200]表6c最佳hlai和hlaiipepi的辅助表:[0201][0202]实施例7——polypepi-scov-2与现有技术疫苗的比较[0203]如文章“preliminaryidentificationofpotentialvaccinetargetsforthecovid-19coronavirus(sars-cov-2)basedonsars-covimmunologicalstudies”(ahmed等人)中所提示的,我们基于其中鉴定的靶标建立了疫苗的可能效力(免疫原性)的模型。将结果与本文所述的选择的polypepi-scov-2疫苗肽进行比较。[0204]sfahmed等人鉴定了与19个hlai等位基因相关的61个t细胞表位,以提供基于全局等位基因频率的96.29%的估计的累积群体覆盖率。(ahmedetal.viruses,12(3).2020)表7所示的下列t细胞表位被认为是疫苗的潜在靶(该文章的表3;61个中的2个仅是8聚体表位,我们从模拟中排除)。[0205]表7采用sfahmed等人:sars-cov衍生的刺突(s)和核壳(n)蛋白t细胞表位的集合(从阳性mhc结合测定获得),其在sars-cov-2中是相同的,并且最大化估计的总体群体覆盖。[0206][0207][0208][0209][0210]ahmed等人提出,通过为每个列出的hla等位基因(即19个序列)选择至少一个表位,可以获得估计的最大群体覆盖率。因此,我们从该t细胞表位组中进行随机选择,为每个hla等位基因选择一个表位(完全如作者所提示的)。因为这些是混杂的hla结合表位,因此有时我们为多于一个hla等位基因选择相同的表位。将此重复30次,并将所选表位与针对polypepi-scov-2(seqidno:2、5、7、9、12、13、14、15、16、17)所选的10种肽进行比较。对从骨髓移植生物库获得的我们~16,000个hla基因分型受试者数据库进行计算机模拟比较。我们的数据库含有来自16个族群的数据(每族群约1,000个受试者)。我们计算了具有针对至少一个表位的cd8 免疫应答的受试者的比例。polypepi-scov-2的全球覆盖率为99.8%,相比之下,模拟疫苗(随机表位选择)的平均覆盖率为61%(±9.9%),一些族群(如高加索人)获得的保护低于其他群体(如日裔)(图7)。[0211]建立了另一种特殊(不实际)的情况,其中ahmed等人(n=59)中列出的所有t细胞表位都被选入疫苗。在这种情况下,全球覆盖率增加至88%,但是仍然没有达到polypepi-scov-2的水平(图8)。这表明在族群之间也均匀覆盖表位疫苗。[0212]我们还模拟了polypepi-scov-2疫苗(选择相同的10种肽)除了诱导cd8应答之外还诱导hlaii类限制性cd4应答(hlaii类pepi)的能力(图9)。在每个种族组群中,至少97%的受试者引发针对polypepi-scov-2疫苗的至少2种肽的cd8和cd4t细胞皆应答。[0213]实施例8——polypepi-scov-2和现有技术肽疫苗的免疫原性表位的数目的比较[0214]基于先前来自ahmed等人的数据集,我们计算了模型群体中每个受试者的免疫原性表位的数目。该数目的分布显示了疫苗对潜在突变的效力。[0215]图10显示61%(±9.9%)的受试者具有针对至少一个疫苗表位的免疫应答,但仅25%(±10.4%)的受试者具有针对至少2个来自19的表位的应答。这意味着,如果病毒在一个特定表位上突变,则另一个表位仍可以产生免疫应答(对于一部分受试者)。相反地,99%的用polypepi-scov-2处理的模型群体具有针对至少2个表位的应答。对于至少3个目标表位,差距甚至更大(polypepi-scov-2为96%,相对epitopevaccine为6%)。[0216]对于含有所有59个表位的疫苗,情况将稍微更好:69%的受试者可具有针对2个或更多个表位的免疫应答(图11),但与polypepi-scov-2疫苗(10个肽)相比,这仍然是群体中较小的比例。[0217]实施例9[0218]covid-19感染和预测的建模警告快速进化,这可能破坏接种疫苗对抗和治疗感染的尝试。迫切需要设计在未来几年中如何开展新型β冠状病毒sars-cov-2的传播。这些动力学将取决于季节性、免疫的持续时间、和针对/来自其它人冠状病毒的交叉免疫的强度。使用来自美国的数据,发明人测量了这些因素如何影响人冠状病毒hcov-oc43和hcov-hku1的传播。(kissler等人,2020,https://doi.org/10.1101/2020.03.04.20031112)。本文所述的疫苗肽和组合物的设计对于快速病毒进化是稳健的,并且通过选择多个免疫原性但保守的序列,优选地衍生自多个结构蛋白,覆盖全球群体。[0219]可以预料,随着病毒继续进化和收集更多的数据,将观察到另外的突变。这样的突变将不影响本文所述的多肽和多肽疫苗的整体覆盖,条件是突变发生在鉴定的表位区域之外。即使突变确实发生在任何选定的表位区域内,剩余的免疫原性表位仍然提供了针对病毒的强大保护,因为大多数受试者将保留病毒特异性记忆t细胞克隆的广泛所有组成成分,例如,对于包括seqidno:2,5,7,9,12,13,14,15,16、和17的多肽的十肽疫苗,94%的患者具有针对至少3个疫苗肽的免疫应答,并且分别针对4和5个肽具有85%和71%的免疫应答。[0220]实施例10[0221]概述[0222]本实施例描述了针对sars-cov-2的全肽疫苗的开发,其解决了不同个体的免疫应答中的异质性和感染病毒的潜在异质性的双重挑战。在本实施例中,“polypepi-scov-2”是含有九个30聚体肽的多肽疫苗,其衍生自下文所述的sars-cov-2病毒的所有四个主要结构蛋白。疫苗肽的选择是基于它们作为hlai类和ii类个人表位(pepi)的频率,这些表位限制在433名不同种族的受试者的计算机模拟组群中的多个自体hla等位基因。与montanideisa51vg佐剂一起施用的polypepi-scov-2疫苗在皮下注射到balb/c和cd34转基因小鼠中后,产生针对病毒的所有四个结构蛋白以及结合抗体的强cd8 和cd4 t细胞应答。此外,在症状发作后1–5个月,在研究的17个无症状/轻度covid-19康复期血液的每一个中离体检测到polypepi-scov-2特异性、多功能cd8 和cd4 t细胞。用于从covid-19回收的polypepi-scov-2特异性t细胞库极其多样化:供体具有针对sars-cov-2蛋白的平均七个不同的肽特异性t细胞;87%的供体具有针对至少三个sars-cov-2蛋白的多个靶而53%的供体具有针对所有四个蛋白的多个靶。此外,基于康复供体的完整i类hla基因型确定的pepi被验证,准确率为84%,以预测对个体测量的pepi特异性cd8 t细胞应答。将上述发现外推至具有16个不同族群(每个族群n=1,000)的16,000个hla基因分型个体的美国骨髓供体组群,表明在一般群体中的polypepi-scov-2疫苗接种将可能在98%的,与种族无关的,包含黑人、亚裔、和少数种族(bame)组群的个体中,引发广泛的多抗原cd8 和cd4 t细胞应答。与montanideisa51vg一起施用的polypepi-scov-2在皮下注射到模拟人免疫系统的balb/c和hcd34 转基因小鼠中后产生针对所有代表性蛋白质以及结合抗体的稳健、th1偏态的cd8 和cd4 t细胞应答。[0223]介绍[0224]由新型冠状病毒sars-cov-2引起的流行在2019年12月爆发后仍在发展。根据世界卫生组织(who),至少三分之二的处于临床开发中的候选疫苗被设计为主要产生针对病毒刺突(s)蛋白的中和抗体(1),但是从sars-cov和mers流行学以及covid-19康复期学到的教训表明了这种疫苗设计策略的潜在挑战。(2,3)潜在的问题是双重的:降低抗体水平和仅针对刺突蛋白的无效t细胞应答产生。[0225]感染了2003年流行的sars-cov病毒和2012年流行的mers病毒的患者通常具有短暂的(检测仅至多3–6年)体液免疫。甚至,由感冒冠状病毒的低风险实验感染产生的抗体在1年内下降,并且不能保护免受再攻击。(4,5)类似地,对于sars-cov-2,与sars-cov-2病毒感染的自然过程相关的免疫应答表明抗刺突igg抗体应答通常是弱的(除了幸运地较不频繁的严重病例),并且它们的持久性在大多数情况下持续高达3个月,或者在这段时间内下降高达70%。(6)此外,2–9%的个体甚至在感染sars-cov-2后2个月没有血清转化,(7)表明个体使用另一分支的适应性免疫系统t细胞达到免疫。事实上,可以得出结论,事实上所有有sars-cov-2感染史的对象都产生了针对病毒的t细胞应答,包含血清阴性和患有严重疾病的对象。(2,8–10)t细胞应答是多样的,即针对病毒的整个抗原库,而不太受刺突蛋白支配。具体地,几项研究报道了尽管是最大的结构蛋白,但刺突特异性t细胞应答仅占天然感染引起的cd4 和cd8 t细胞总数的25–27%。此外,这种多样性与无症状/轻度疾病有关,因为恢复的患者具有更多的针对膜(m)和核蛋白(n)蛋白而不是s蛋白的cd8 t细胞,而t细胞强度和多样性不随疾病严重程度而增加,如mers、sars-cov-1和sars-cov-2所证实的。(6,9,11,12)实际上,在covid-19患者中,低cd8 t细胞计数是死亡风险较高的预示,而患有严重疾病的患者或死亡的患者具有耗竭的t细胞。(2,13)认为感染后较早时间可检测的病毒特异性cd8 t细胞应答导致较低的病毒负荷和因此较低的抗体水平,解释了为什么这些患者具有更有利的结果。(12)为了支持这一点,最近报道了在病毒暴露后不久和在任何抗体检测之前对sars-cov-2特异性t细胞受体进行作图是可能的。[0226]这些观察结果还表明,在感染(通过接种)之前获得数量增加的多种病毒特异性记忆t细胞,可能有助于sars-cov-2感染早期的病毒和病毒储库的消除。(9,15)这些预期得到了动物攻击研究的支持,这些研究表明,重新激活的t细胞为sars-cov-1的致死剂量感染提供了保护。值得注意的是,在sars-cov-1病毒康复17年后,23名接受试验的患者中有23名报告了针对sars-cov-1病毒的n蛋白的记忆t细胞。(9)其它报道也支持由冠状病毒感染引起的记忆t细胞的持久性。(18–23)[0227]因此,在临床开发中旨在产生针对病毒s蛋白的t细胞应答的候选疫苗将可能仅产生恢复期免疫应答的一部分,因此不太可能诱导稳健的记忆t细胞应答。疫苗技术使用完整病毒,多个大蛋白理论上可以解决与缺乏多样性相关的问题。然而,这些具有着包括不必要的抗原负载的限制,这不必要的抗原负载不仅对保护性免疫应答贡献很小、而且通过诱导变应原性和/或反应原性应答而使情况复杂化。类似地,编码靶抗原的复制缺陷型病毒构建体可以引发针对病毒载体的非特异性免疫应答,尤其是在重复剂量下。[0228]肽疫苗是一种替代的亚单位疫苗策略,其依赖于短肽片段、表位的使用,能够诱导阳性的、期望的t细胞和b细胞介导的免疫应答。[0229]然而,困扰肽疫苗设计的核心问题是每个人具有独特的免疫应答谱。实际上,对于sars-cov-2,疾病过程根据由不同种族代表的遗传多样性而变化,尤其是黑人、亚裔和少数民族(bame)族群;然而,其原因还未被很好地理解。(25,26)遗传多样性可通过人类白细胞抗原(hla)等位基因的遗传变异来捕获,这等位基因是病毒抗原(表位)呈递途径的关键组分,其引发能够识别并杀死体内癌症或感染细胞的细胞毒性t细胞(ctl)。为了在设计针对sars-cov-2的整体疫苗中捕获这种异质性,已经广泛使用基于常见人hla等位基因的病毒抗原表位预测。(27)然而,实际上,这些表位作图研究在验证的t细胞应答方面具有低产量。例如,在一项研究中,测试了100个预测为10个最普遍hlai类等位基因的sars-cov-2衍生表位,仅有12个被证实为显性表位,即被》50%的covid-19供体cd8 t细胞识别。这与在本领域中对于若干感染性和癌症疫苗临床试验以及对于个性化突变的基于新抗原的表位的相对低的确认水平观察到的免疫应答率一致。[0230]为了克服肽疫苗设计的这些限制,我们数字化地开发了polypepi-scov-2,其使用具有完整hla基因型的个体的种族差异的计算机模拟人类组群,而不是使用单一hla等位基因。选择了多个所谓的个人表位(pepi),不仅被每个个体的一个而是被多个自身hla等位基因所限制,而且还在种族多样性群体中的大部分受试者之间共享。值得注意的是,这个计算机模拟人类组群与pepi概念一起先前回顾性地预测79个疫苗临床试验的免疫应答率,以及在转移性结肠直肠癌患者中进行的临床试验中我们的polypepi1018癌症疫苗的显著免疫原性(80%cd8 t细胞应答针对六个抗原中的至少三个)。(31–33)由为乳腺癌患者制备的个性化多肽混合物中的pepi产生的cd8 t细胞应答证明是长期持续的,因为在最后一次针对四个肿瘤抗原接种后14个月(436天)检测到它们。[0231]与sars-cov-2在自然感染过程中明显的长期记忆t细胞形成能力一致,本发明的多肽疫苗被设计为(1)通过靶向sars-cov-2的所有四个结构蛋白,在每个对象中诱导稳健的和广泛的免疫应答;(2)通过确保每个患者中的多个免疫原性靶来解决和克服潜在的病毒进化效应;且(3)通过肽的个人表位覆盖解决人类种族的不同敏感性。本文描述了针对covid-19的候选疫苗的设计和临床前表征。在二个小鼠模型中证实了免疫原性和耐受性,导致诱导了由第二剂加强的强cd4 和cd8 t细胞应答,以及体液应答。在康复期的covid-19血样中,在所有受试者中检测到抗所有肽的疫苗特异性免疫细胞,代表引起感染恢复的sars-cov-2诱导的免疫组库的重要组分。与由死亡或减毒病毒和重组蛋白制成的传统疫苗相比,肽疫苗是安全和经济的技术。以多公斤规模生产合成肽相对便宜,并且比mrna生产肯定更成熟。该技术不仅能够鉴定特定疾病/病原体的抗原靶标,而且更重要的是,能够计算确定大组群中个体的免疫系统能够应答的抗原。[0232]材料和方法[0233]患者/供体[0234]根据其sars-cov-1或sars-cov-2感染的临床史征募供体。血样在尼德兰独立医学研究中心以批准的方案(nl57912.075.16)从康复个体(n=15)收集,或由peptc疫苗有限公司收集(n=2)。来自未暴露个体(n=5)的血清和pbmc样品在2018年前收集并由nexelis-imxp(比利时)提供。所有供体提供书面知情同意书。该研究是按照赫尔辛基宣言进行的。症状发作后17–148天获得covid-19康复病人(n=17;16名无症状/轻微疾病和一名严重疾病)的血样。令人惊奇的是,在健康供体中发现了一个阳性igm抗体应答,其被排除在进一步分析之外。受试者的人口统计学和基线信息提供于表8中。[0235]来自尼德兰的康复期供体患者的hla基因分型是由imgm实验室有限公司(德国martinsried)使用下一代测序技术完成的。二个peptc供体的hla基因分型是由实验室公司(美国弗吉尼亚州burlington的labcorp)的颊拭子使用下一代测序(illumina)进行的,而hla等位基因解释是基于imgt/hla数据库版本3.38.0。来自尼德兰的康复期供体患者的hla基因分型是由imgm实验室有限公司(德国martinsried)使用下一代测序技术完成的。该组群使用总共46个不同的hlai类等位基因(15个hla-a*、18个hla-b*和13个hla-c*)和35个不同的hlaii类等位基因(14个drb1、12个dqb1和9个dpb1)。受试者的hla基因型数据提供于表8b中。[0236]动物[0237]cd34 转基因人源化小鼠(hu小鼠)。使用从人脐带血分离的造血干细胞(cd34 )对雌性nod/shi-scid/il-2rγ无效免疫缺陷小鼠(charlesriverlaboratories,法国)进行人源化。在研究期间仅使用人源化率(hcd45/总cd45)》50%的小鼠。用20–23周龄的雌性小鼠进行实验。[0238]balb/c小鼠。用6–8周龄的雌性balb/c小鼠(法国janvier)进行实验。[0239]表8供体基线和人口统计信息。所有供体都是高加索人种,具有轻度/无症状疾病且未住院治疗(除了一个用*标记的)。s/co,样品/对照比率;根据制造商的说明书确定值,并且试验结果解释为s/co《0.9为阴性、若s/co=0.9–1.1则不确定、且若s/co》1.1则为阳性。coi,截止指数;根据制造商的说明书确定数值,并且试验结果解释为coi《0.9为阴性、coi为0.9–1.1为不确定、且若coi》1.1为阳性。na,数据不可用。斜体,阴性或不确定的数值。**主诉:a,咳嗽;b,喉咙痛;c,发烧;d,气短;e,胃/肠道不适;f,胸痛;g,眼睛痛;h,嗅觉或味觉丧失;i,头痛;j,疲劳;k,其他主诉(imxp00759为肺栓塞和心脏停搏;腿痛、手臂痛、肌肉痛、眼睛痛)。[0240][0241]表8b恢复期供者的完全hla基因型[0242][0243][0244]疫苗设计[0245]筛选sars-cov-2结构蛋白(s、n、m、e),并在多步骤过程中选择九个不同的30聚体肽。首先,进行序列多样性分析(如ncbi数据库中截至2020年3月28日)。(35)登录号id如下:nc_045512.2、mn938384.1、mn975262.1、mn985325.1、mn988713.1、mn994467.1、mn994468.1、mn997409.1、mn988668.1、mn988669.1、mn996527.1、mn996528.1、mn996529.1、mn996530.1、mn996531.1、mt135041.1、mt135043.1、mt027063.1、和mt027062.1。第一个(粗体)id代表genbank参考序列。然后,使用多序列比对(英国embl-ebi的clustalomega)比对和比较翻译的四个结构蛋白序列的编码序列。(36)在19个序列中,15个是相同的;然而,在四个n蛋白序列中发生单个氨基酸变化:mn988713.1,n194s→x;mt135043.1,n343d→v;mt027063.1,n194s→l;mt027062.1,n194s→l。所得氨基酸取代仅影响n蛋白序列的二个位置(aa194和343),都不是发生在已被选作疫苗开发靶标的表位中(seqidno:2、5、9、和12–17)。在polypepi-scov-2疫苗中,仅涉及十三个报道的病毒s蛋白中的单字母变化(d614g,s943p,l5f,l8v,v367f,g476s,v483a,h49y,y145h/del,q239k,a831v,d839y/n/e,p1263l)中的一个(h49y),但该变体的流行在后来的病毒分离物中降低。(37)关于肽选择的进一步细节在结果部分提供,并且九个选择的30聚体肽的所得组成在表9中显示。[0246]与人冠状病毒株的交叉反应性[0247]将polypepi-scov-2疫苗的序列与sars-cov、mers-cov和普通(季节性)人冠状病毒株的序列进行比较,以揭示可能的交叉反应区域。根据疾病控制和预防中心(cdc),人类群体中常见的冠状病毒感染由四个冠状病毒组引起:α冠状病毒229e和nl63、以及β冠状病毒oc43和hku1。(38)结构蛋白的成对比对还使用uniprot(39)进行,具有以下参考序列id:229e:p15423(s)、p15130(n)、p19741(e)、p15422(m);nl63:q6q1s2(s)、q6q1r8(n)、q6q1s0(e)、q6q1r9(m);oc43:p36334(s)、p33469(n)、q04854(e)、q01455(m);hku1(分离株n1):q5mqd0(s)、q5mqc6(n)、q5mqc8(e)、q5mqc7(m)。此外,冠状病毒菌株与构成polypepi-scov-2疫苗的9个30聚体肽进行了比对。对于表位的最低要求,筛选八个氨基酸长的区域(滑动窗)作为负责潜在交叉反应性的区域。此外,在分析期间记录和报告较短(和较长)长度匹配的连续肽片段。[0248]计算机模拟人类组群[0249]模型群体是433个个体的组群,代表世界范围内的几个族群,对于它们可获得完整的hlai类基因型(2×hla-a,2×hla-b,2×hla-c)。模型群体由90名约鲁巴非裔(yri)、90名欧裔(ceu)、45名华裔(chb)、45名日裔(jpt)、67名混合种族(美国、加拿大、澳大利亚、新西兰)、和96名来自hiv数据库(mix)的对象组成。(40–43)使用pcr技术、affymetrix6.0和illumina1.0百万snpmassarray,以及通过参照链介导的构象分析(rsca)或基于测序的分型(sbt)对六个hla基因进行高分辨hla分型,确定hla基因型。(44–46)以前已经报道了模型群体的表征。(31)该组群使用总共152个不同的hlai类等位基因(49个hla-a*、71个hla-b*和32个hla-c*),代表97.4%的当前全球常见、中间和有据可查的(ciwd)等位基因,并充分代表主要种族(发行于2020的数据库3.0)(表8a)(hurley等人,2020)。模型群体的a*、b*、c*等位基因的频率与ciwd数据库中大于8百万个hla分型对象的记录频率相关(分别是r=0.943、0.869、0.942,p《0.00001)(图24)。[0250]表8a在模型群体中呈现的等位基因的hla覆盖度。非裔/非裔美国人(afa)、亚裔/太平洋岛(api)、欧裔/欧洲血统(euro)、中东/非洲北海岸(mena)、南美或中美洲/西语裔/拉丁裔(his)、美国原住民群体(nam)、未知/未询问/多祖先/其他(unk)。ciwd3.0:常见(10,000中≥1)、中间(100,000中≥1)和文献充分记载(≥5次出现)的hla数据库3.0(2020年发布)。与图24相关。[0251][0252]从dbmhc数据库(47)获得了356个个体的第二模型组群,这些个体具有四位等位基因分辨率的已表征ii类hla基因型(2×hla-drb、2×hla-dp、和2×hla-dq),dbmhc数据库是由国家生物技术信息中心(ncbi)操作的在线可用储存库,开发用于评价cdna或基因组序列的等位基因组成。在全世界许多国家中对广泛的种族进行采样。总之,该数据库中的356名受试者具有足够分辨率的hlaii类基因型数据(2×hla-drb、2×hla-dp、和2×hla-dq,具有至少四位分辨率)。hla基因分型通过sbt进行。[0253]大,美国组群(n=16,000)[0254]通过从来自国家骨髓供体计划(nmdp)的16个族群(500名男性和500名女性)中的每一个获得1,000个供体,创建包括来自16,000个个体的数据库。(48)16个族群是:非裔、非裔美国人、亚洲太平洋岛裔、菲律宾裔、黑加勒比海人、高加索人、华裔、西语裔、日裔、韩裔、美国印地安原住民、南亚裔、越南裔、美国、中东/非洲北海岸、夏威夷裔、和其它太平洋岛裔。hla基因分型通过nmdp募集实验室使用序列特异性寡核苷酸(sso)和序列特异性引物(ssp)方法进行,平均“分型分辨率得分”》0.7。(49)[0255]肽和polypepi-scov-2疫苗制剂[0256]9聚体(s2、s5、s9、n1、n2、n3、n4、e1、m1)和30聚体(s2、s5、s7、n1、n2、n3、n4、e1、m1)肽由intavispeptideservicesgmbh&co.kg(德国tübingen)和pepscan(尼德兰lelystad)使用固相肽合成来生产。表9中提供了9聚体试验肽(表9,粗体)和30聚体疫苗肽二者的氨基酸序列。通过将等质量的九个30聚体肽溶解于dmso中以达到1mg/ml的浓度,然后用纯水稀释至0.2mg/ml的终浓度并冷冻储存直至使用,来制备用于动物研究的肽疫苗。按照制造商提供的标准双注射器方案,通过乳化等体积的解冻的肽混合溶液和montanideisa51vg佐剂(法国巴黎seppic)制备准备注射的疫苗制剂。[0257]表位预测和分析[0258]使用基于免疫表位数据库(iedb)的表位预测方法预测每个个体中的≥3个hlai类等位基因结合表位(pepi)。用重叠的9聚体和15聚体肽扫描抗原以鉴定结合受试者hlai类等位基因的肽。用已经通过使用直接结合测定法进行实验表征的427个hla–肽对的内部集合(327个结合和100个未结合hla–表位对)验证选择参数。对于真实hla等位基因–表位对的预测,特异性和灵敏度结果皆为93%。ii类hla表位的预测是通过netmhcpan(2.4)预测算法进行的,每个个体≥4个ii类hla结合表位被定义为hlaii类pepi。[0259]临床前小鼠研究设计[0260]36只hu小鼠和36只balb/c小鼠接受在第0天和第14天皮下施用的polypepi-scov-2疫苗(0.66mg/kg/肽的200μl溶液;n=18)或乳化在montanideisa51vg佐剂中的20%dmso/水(200μl载体;n=18);随访期在第28天结束,分析第14、21、和28天的样品(每组群n=6)。研究在transcurebioservicefacility(法国archiamps)进行。每天监测小鼠的意外痛苦迹象。每周通过监测被毛(0–2分)、运动(0–3分)、活动(0–3分)、苍白(0–2分)、和体重(0–3分)进行完全临床评分;正常状况评分为0。[0261]本研究中描述的所有程序都经过当地伦理委员会(celeag)的审查和批准,并由法国研究部验证。通过小鼠脾细胞的离体elispot和细胞内细胞因子染色试验(ics)评估接种诱导的t细胞应答(下文详述)。通过测量血浆样品中的总igg研究抗体应答(详述如下)。[0262]elispot/fluorospot分析[0263]用于动物研究的离体elispot测定如下进行。使用2×105个被刺激了20小时/肽(10μg/ml,终浓度)的脾细胞鉴定产生ifn-γ的t细胞。用9聚体肽(四个n特异性肽的池:n池(n1、n2、n3、n4),三个s特异性肽的池:s池(s2、s5、s9),e蛋白衍生肽:e1,或m蛋白衍生肽:mm1)),或用与9聚体相同方式聚类的30聚体肽(包括肽n1、n2、n3、和n4的n池,包括肽s2、s5和s9的s池,以及单独的肽e1和m1),处理脾细胞。按照生产商的说明书,使用mabtech的人ifn-γelispotpro试剂盒(alp;ref3321-4apt-2)对hu小鼠组群进行、并使用mabtech的小鼠ifn-γelispotpro试剂盒(alp;ref3321-4apt-10)对balb/c小鼠组群进行elispot测定。从每个数据点中减去未刺激(dmso)测定对照背景斑点形成单位(sfu),然后计算δsfu(dsfu)。pma/离子霉素(invitrogen)用作阳性对照。[0264]用于康复供体试验的离体fluorospot测定通过nexelis-imxp(比利时)如下进行:用rpmi-10%fbs封闭ifn-γ/il-2fluorospot板,然后将肽(5μg/ml终浓度)或肽池(5μg/ml每肽终浓度)加入到相关孔中。从低温储存中回收pbmc并在培养基中解冻。然后,将200,000个pbmc细胞/孔三重复(刺激条件)或六重复(参考条件)铺板,并在培养前于37℃,5%co2温育过夜。根据制造商的建议进行fluorospot板的显影。除去细胞后,将稀释在含0.1%bsa的pbs中的检测抗体加入孔中,并将fluorospot板在室温下温育2小时。在使用mabtechiristm自动化fluorospot读数器读出之前,将fluorospot板倒空并在室温下避光干燥24小时。所有数据用mabtechiristm读数器获得,并用mabtechapextm软件分析。未刺激(dmso)阴性对照、cef阳性对照(来自cmv、ebv和流感病毒的t细胞表位,瑞典mabtech)、和商业sars-cov-2肽库(sars-cov-2snmo限定的肽池(3622-1)——瑞典mabtech),被包含作为测定对照。在减去非刺激对照(dsfu)后,当试验结果高于dmso阴性对照时,离体fluorospot结果被认为是阳性的。[0265]用于康复期供体检测的富集elispot检测由nexelis-imxp(比利时)进行,具体方法如下:从低温储存中回收pbmc并在培养基中解冻。在肽池存在下(每肽5μg/ml)以4,000,000个细胞/24孔接种pbmc,并在37℃、5%co2下温育7天。在培养的第1天和第4天,更新培养基,并分别补充5ng/ml的il-7或5ng/ml的il-7和4ng/ml的il-2(r&dsystems)。培养7天后,收获pbmc并静置16小时,然后使用trypanblue溶液0.4%(vwr)和cellometerk2荧光生存细胞计数器(nexcelom)对静置的pbmc计数,并以200,000细胞/孔在ifn-γ/粒酶–b/tnf-αfluorospot板(mabtech)上接种在含有10%人血清hi、2mml-谷氨酰胺、50μg/ml庆大霉素、和100μmβ-me的rpmi1640上,至含有肽(5μg/ml)或肽池(每肽5μg/ml)的相关fluorospot孔中。在培养前,将fluorospot板在37℃,5%co2下温育过夜。所有数据用mabtechiristm读数器获得,并用mabtechapextm软件分析。dmso、仅培养基、商业的covid肽库(sars-cov-2snmo定义的肽池[3622-1]——mabtech)、和cef作为测定对照,以1μg/ml的浓度包含在内。在减去背景(dsfu)后,阳性标准为》1.5倍的未刺激对照。[0266]细胞内细胞因子染色(ics)测定[0267]临床前小鼠研究的离体ics测定如下进行:刺激20小时后,从elispot板上取下脾细胞,转移到常规的96孔平底板上,根据制造商的建议,用bdgolgistoptm培养4小时。使用bdcytofix/cytopermplus试剂盒与bdgolgistoptm蛋白转运抑制剂(含有莫能星;目录号554715)按照制造商的说明进行流式细胞术。流式细胞术分析和细胞因子分布测定在attunenxt流式细胞仪(lifetechnologies)上进行。分析了2×105个细胞,门控cd45 、cd3 、cd4 或cd8 t细胞。低于25的计数被评价为0。斑点计数≥25是通过减去未刺激的(dmso)对照而校正的背景。pma/离子霉素(invitrogen)用作阳性对照。作为测定对照,使用mann-whitney检验比较每个细胞因子的阴性对照(未刺激)和阳性对照(pma/伊屋诺霉素)。当确定对照之间的统计学差异时,分析对应于其它刺激条件的值。下列染色剂用于hu小鼠组群:mab11502932(biolegend),mp4-25d2500836(biolegend),4s.b3502536(biolegend),hi30304044(biolegend),sk7344842(biolegend),jes6-5h4503806(biolegend),vit4130-113-218(miltenyi),jes1-39d10500904(biolegend),sk1344744(biolegend),jes10-5a2501914(biolegend),jes3-19f1554707(bd),以及na564997(bd)。下列染色用于balb/c小鼠组群:11b11562915(bd)、mp6-xt22506339(biomension)、xmg1.2505840(biomension)、30-f11103151(biomension)、145-2c11100355(biomension)、jes6-5h4503806(biomension)、gk1.5100762(biomension)、jes1-39d10500904(biomension)、53-6.7100762(biomension)、ebio13a25-7133-82(thermoscientific)、jess-16e3505010(biomension)、和na564997(bd)。[0268]用nexelis-imxp(比利时)进行恢复期供体检测的离体ics分析。简言之,在解冻200,000个pbmc细胞/孔后,在肽(5μg/ml)或肽池(每肽5μg/ml)的存在下,将pbmc接种在无菌圆底96孔板中,该无菌圆底96孔板中的rpmi总量为10%人血清hi、2mml-谷氨酰胺、50μg/ml庆大霉素、和100μm2-me。温育2小时后,将bdgolgiplugtm(bdbiosciences)以培养基中1μl/ml的浓度加入96孔板中。温育10小时后,将板离心(800g,3分钟,8℃)并在37℃温育10分钟,并将zombienir生存力染料(biolegne)加入到每个孔中。将板在避光的条件下于室温温育15分钟。温育后,每孔加入pbs/0.1%bsa,并将板离心(800g,3分钟,8℃)。此后,细胞在fcr封闭试剂中于4℃温育5分钟,然后将染色混合物(含有抗cd3、biolegen、抗cd4和抗cd8抗体;bdbiosciences)加入每个孔中。在4℃下孵育30分钟后,洗涤并离心(800g,3分钟,8℃),根据制造商的建议(bdbiosciences)将细胞透化并固定。固定后,将细胞因子染色混合物(含有抗ifn-γ、抗il-2、抗il-4、抗il-10和抗tnf-α抗体,biolegen)加入每个孔中。将板在4℃下温育30分钟,然后在获得之前洗涤二次。所有流式细胞术数据用lsrfortessatmx-20获得,并使用flowjov10软件分析。从使用试验肽或池获得的每个数据点中减去dmso阴性对照。[0269]抗体elisa[0270]根据制造商的说明书,对balc/c组群使用igg(总)小鼠未包被elisa试剂盒(invitrogen,#88-50400-22),并对用于hu小鼠组群使用igg(总)人未包被elisa试剂盒(invitrogen,#88-50550-22),进行用于小鼠研究的elisa,以定量测量血浆样品中总小鼠igg的产生。使用在第14、21、和28天收获的样品进行分析(每组每时间点n=6)。在epoch微板读数器(biotech)上读取吸光度,并使用gen5软件分析。[0271]康复供体试验的elisas通过mikromikomedkft(匈牙利布达佩斯)进行,根据生产商的说明书(意大利diaprodiagnosticbiobesr.l),使用diaprocovid-19igm酶免疫测定来确定人血清和血浆中的抗covid-19的igm抗体,使用diaprocovid-19igg酶免疫测定来确定人血清和血浆中的抗covid-19的igg抗体,并使用diaprocovid-19iga酶免疫测定来确定人血清和血浆中的抗covid-19的iga抗体。为了确定总的n特异性ig抗体,根据制造商的说明书,使用用于定性检测抗sars-cov-2的抗体(包含igg)的roche抗sars-cov-2免疫测定法,用cobase411分析仪(瑞士罗氏的disksystem)。进行euroimmunelisa测定以通过独立的医学研究中心确定康复期供体中的s1特异性igg水平。抗sars-cov-2elisa板用sars-cov-2的抗体结合的sars-cov-2的重组s1结构蛋白包被。选择与其它冠状病毒,特别是sars-cov-1同源性相对较低的抗原。按照生产商的说明书进行免疫测定。[0272]假颗粒中和试验[0273]根据剂量范围发现,使用基于细胞的假颗粒中和测定法(pna)评价小鼠血清中的中和抗体:sars-cov-2假颗粒中和试验。将表达ace-2受体的veroe6细胞(veroc1008(美国,atcc编号crl-1586)),以20000个细胞/孔的速度播种,使细胞汇合度达到80%。将血清样本和对照组(内部生产的人类康复血清池)在细胞生长培养基中二重复稀释,起始稀释度为1/25或1/250(样本)或1/100(对照组),然后进行连续稀释(2倍稀释,5次)。同时,sars-cov-2假病毒(由nexelis公司使用kerafast系统制备,使用武汉株的刺突(减去19个c端氨基酸))被稀释,以达到所需的浓度(根据预先确定的tu/ml)。然后将假病毒加入稀释的血清样本中,在37℃下用co2预温育1小时。然后将该混合物加入到预先接种的veroe6细胞层中,在37℃和5%的co2条件下将板培养18-24小时。孵化并去除培养基后,将one-gloex荧光素酶检测底物(promega,目录号e8110)添加到细胞中,并在室温下摇晃孵化3分钟。使用spectramaxi3x微板阅读器和softmaxprov6.5.1(moleculardevices)测量发光。每个稀释度的发光结果被用来生成一个滴定曲线,使用微软excel(适用于微软office365)的4参数逻辑回归(4pl)。效价定义为发光等于预定的50截点(对应于50%中和)的样品稀释度的倒数。使用50%的侧翼点,使用线性回归建立该截止点。[0274]统计分析[0275]使用两尾t检验或mann-whitney检验比较组间和组间的显著性,适当时,p《0.05被认为是显著的。进行pearson检验以评价相关性。若r》0.7则认为相关性强,若0.5《r≤0.7则认为相关性中等,若0.3《r≤0.5则认为相关性弱。[0276]结果[0277]将polypepi-scov-2定制至个体[0278]在设计polypepi-scov-2期间,我们使用计算机模拟人类组群中受试者的hla基因型数据来确定四个选择的sars-cov-2结构蛋白的旨在诱导cd8 t细胞应答的最免疫原性肽(即hlai类pepi热点,9聚体)。然后基于病毒蛋白序列将鉴定的9聚体pepi热点的序列延伸至30聚体,以最大化旨在诱导cd4 t细胞应答的hlaii类结合pepi(15聚体)的数目。[0279]首先,我们分别使用9聚体(i类hla)和15聚体(ii类hla)框架,对其i类hla和ii类hla基因型(六个i类hla和八个ii类hla等位基因)的19个已知sars-cov-2病毒蛋白的保守区的氨基酸序列,在计算机模拟人类组群中对每个受试者进行表位预测(图1)。然后,我们选择能够结合多个(≥3)自体hla等位基因的表位(pepi)。我们为每个人鉴定了几个hla限制性表位(平均,仅s-1蛋白有166个表位)。相反,pepi在所有种族中以低得多的水平呈现(平均,14个多hla结合表位,图12)。[0280]从所得的pepi列表中,我们鉴定了九个30聚体多肽片段,其包括重叠的、在模型群体中的高百分比个体之间共享(频繁)的i类和ii类pepi,与种族无关(表9)。为了在个体水平和群体水平上皆最大化多抗原免疫应答,我们从大刺突(s)和核蛋白(n)蛋白中选择多余一个的肽,并且从较短的膜(m)和包膜(e)蛋白中仅选择一个肽。从sars-cov-2病毒的四个结构性病毒抗原中,共选择了九个30聚体的多肽用于疫苗,同时考虑了多肽的化学和可制造性:来自s的三个多肽、来自n的四个多肽、来自m和e的各一个多肽。没有包含来自s-1蛋白的受体结合域的多肽。总之,模型群体的每个成员具有九个肽中至少二个的pepi,97%具有至少三个(表9)。[0281]表9在计算机模拟人类组群内的包括pepi频率的polypepi-scov-2肽。粗体:包括30聚体肽的polypepi-scov-2内的9聚体hlai类pepi序列;下划线:15聚体ii类hlapepi序列。百分比显示了来自模型群体的具有至少一个i类hla(cd8 t细胞特异性)pepi或至少一个ii类hla(cd4 t细胞特异性)pepi的个体的比例。标记的肽含有实验证实的b细胞表位,具有在sars康复期患者中发现的抗体(ig)应答。n/a:数据不可用。[0282][0283][0284]我们通过实验鉴定了来源于sars-cov-1的线性b细胞表位,其与相关sars-cov-2抗原具有100%的序列同一性,以说明长肽的潜在b细胞生产能力。(54)polypepi-scov-2疫苗的位于n蛋白衍生肽中的三个重叠表位和位于s蛋白衍生肽中的一个表位与患严重急性呼吸综合征(sars)的恢复期患者的血清反应。这表明上述s和n蛋白上的抗原位点在引发人体内针对sars-cov-1和可能针对sars-cov-2的体液免疫应答中是重要的。[0285]如所预期的,polypepi-scov-2含有几个(九个中的八个)肽,由于sars-cov-2和sars-cov之间的高度序列同源性,而与sars-cov交叉反应。polypepi-scov-2肽与属于α冠状病毒(229e和nl63)、β冠状病毒(oc43,hku1)和mers的普通(季节性)冠状病毒株之间的序列相似性低。因此,疫苗和先前冠状病毒感染个体之间的交叉反应性仍然较低,可能仅涉及疫苗的m1肽(参见方法和表10)。[0286]表10polypepi-scov-2和其它冠状病毒株之间的序列比对结果。序列比较是用比对的蛋白序列对之间的8聚体长的肽匹配进行的,定义为cd8 t细胞表位的最小长度要求。最大氨基酸匹配:最长相同的氨基酸序列长度。突出显示的灰色值表示至少八个氨基酸的相同序列。[0287][0288]在小鼠中polypepi-scov-2疫苗诱导的广泛t细胞应答[0289]在非人源化balb/c模型中和在cd34 hu-ncg(hu小鼠)小鼠的人源化免疫环境中,在相隔二周(第0天和第14天)施用一和二个疫苗剂后,进行polypepi-scov-2疫苗的临床前免疫原性试验以测量诱导的免疫应答。免疫后,没有小鼠呈现任何临床评分(评分0,表示与正常无偏差),表明不存在任何副作用或免疫厌恶。此外,在每个时间点通过肉眼观察进行的尸检没有揭示脾、肝、肾、胃和肠中的任何可见的器官变化。重复的疫苗施用也是良好耐受的,并且没有检测到免疫毒性或其他全身性不良事件的迹象。总之,这些数据强烈提示本研究中使用的制剂在小鼠中是安全的。[0290]在第14天(d14)第一剂后和在第21天(d21)和第28天(d28)第二剂后测量产生ifn-γ的疫苗诱导的t细胞。在第14天,polypepi-scov-2处理没有比载体(用montanide乳化的dmso/水)诱导更多的cd8 t细胞的ifn-γ产生,后者导致异常高的应答,可能是由于montanide介导的非特异性应答。然而,在第21天和第28天,对于用30聚体和9聚体肽池刺激的脾细胞,第二剂的polypepi-scov-2与媒介物对照组相比将ifn-γ产生提高六倍和3.5倍(图13a)。在第21天和第28天,来自polypepi-scov-2处理的小鼠的t细胞的ifn-γ产生增加通过斑点形成细胞的数目增加而证实,其对于以下条件达到统计学显著性:分别是第21天的30聚体s池、肽e1、9聚体n池和肽e1,以及第28天的肽e1和9聚体s池(图14a–c)。[0291]在免疫缺陷的hu小鼠中,在第14天,用polypepi-scov-2处理使ifn-γ产生增加二倍,其中脾细胞用9聚体肽池刺激,但用30聚体刺激的脾细胞未观察到增加。在第21和28天,第二剂的polypepi-scov-2分别对用9聚体和30聚体肽池刺激的脾细胞增强ifn-γ的产生二倍和四倍(图13b)。重要的是,在二种动物模型中,9聚体检测的cd8 t细胞和30聚体检测的cd4 和cd8 t细胞应答皆同等地针对疫苗靶向的所有四个病毒蛋白(图13c、d和图14d–f中的详细结果)。[0292]进行细胞内染色(ics)分析以研究由接种引起的t细胞应答的极化。由于t细胞的频率低,与elispot相比,ics更难以观察单个肽特异性t细胞,但与仅接受载体(用montanide乳化的dmso/水)的动物相比,在balb/c和hu小鼠模型中皆可检测到产生tnf-α和il-2的th1型细胞因子的cd4 和cd8 t细胞的清晰群体(图15)。对于th2型细胞因子il-4和il-13,分析在任何时间点都没有显示任何特异性应答。在这二个模型中皆检测到低水平的il-5和/或il-10细胞因子产生cd4 t细胞,但在第28天仅balb/c小鼠与载体对照显著不同。即使对于该组群,6只小鼠中有5只(一个异常值)th1/th2平衡保持向th1偏移,证实了由疫苗引起的总体th1偏态t细胞应答(图16)。[0293]polypepi-scov-2接种也诱导体液应答,如通过balb/c的总小鼠igg和hu小鼠的人igg所测量的。在balb/c小鼠中,与仅接受载体的对照动物相比,接种在第一剂(第14天)后导致疫苗诱导的igg产生。在第二剂后的稍后时间点,对于balb/c和hu小鼠模型皆观察到igg升高(图17)。如所预期的,假定polypepi-scov-2肽不含构象表位,则疫苗接种不产生中和抗体,如使用用假颗粒的中和测定法从hu小鼠血清所评估的(数据未显示)。[0294]polypepi-scov-2疫苗在二种动物模型中诱导广泛的t细胞应答[0295]在balb/c和hu小鼠模型中,进行polypepi-scov-2疫苗的临床前免疫原性试验,以测量在相隔二周(第0天和第14天)施用一和二个疫苗剂后诱导的免疫应答。免疫后,在第14、21或28天没有小鼠出现任何临床评分(评分0,表示与正常没有偏差),表明没有任何副作用或免疫厌恶(表10a和b)。此外,在每个时间点通过肉眼观察进行的尸检没有揭示脾、肝、肾、胃和肠中的任何可见的器官变化(表10c)。重复的疫苗施用也是良好耐受的,并且没有检测到免疫毒性或其他全身性不良事件的迹象。总之,这些数据强烈提示polypepi-scov-2在小鼠中是安全的。[0296]在第14天第一剂后和在第21天和第28天第二剂后测量疫苗诱导的产生ifn-γ的t细胞。根据它们的来源蛋白质:s、n、m、和e,使用分组在四个池中的九个30聚体疫苗肽检测疫苗诱导的t细胞,以评估cd4 和cd8 t细胞应答。cd8 t细胞应答也使用短的9聚体试验肽特异性测量,该试验肽对应于上文对四个池(s、n、m和、e肽;表9粗体)中的九个疫苗肽中的每一个定义的共享hlai类pepi。[0297]在balb/c小鼠中,在第14天,polypepi-scov-2疫苗接种没有比载体(用montanide乳化的dmso/水)诱导更多的ifn-γ产生,后者导致异常高的应答,可能是由于montanide介导的非特异性应答。然而,在第21天和第28天,分别地,对于用30聚体和9聚体肽检测的脾细胞,第二剂的polypepi-scov-2与媒介物对照组相比使ifn-γ产生增加六倍和3.5倍(图13a)。[0298]在免疫缺陷的hu小鼠中,在第14天,用对9聚体肽池特异的脾细胞进行polypepi-scov-2接种使ifn-γ产生增加二倍,但用30聚体刺激的脾细胞未观察到增加。在第21和28天,分别地,对于用30聚体和9聚体肽检测的脾细胞,第二剂的polypepi-scov-2使ifn-γ产生增加四倍和二倍(图13b)。重要的是,在二种动物模型中,9聚体检测的cd8 t细胞和30聚体检测的cd4 和cd8 t细胞应答皆针对疫苗靶向的所有四个病毒蛋白(图13c–d和图14d–f)。由于hu小鼠模型是通过移植人cd34 造血干细胞产生人抗原呈递细胞和t-和b-淋巴细胞进入nod/shi-scid/il-2rγ无效免疫缺陷小鼠而开发的,该模型提供了真实的人免疫系统模型(brehm等人,2013)。因此,在该模型中获得的稳健的多抗原cd4 和cd8 t细胞应答表明,疫苗接种导致hu小鼠的人免疫细胞的适当加工的和hla呈递的表位和随后的抗原特异性t细胞应答。[0299]进行ics分析以研究由接种引起的t细胞应答的极化。由于t细胞的频率低,与elispot相比,ics更难以观察到单个肽特异性t细胞,但是与接受载体的动物相比,在balb/c和hu小鼠模型中皆可检测到产生tnf-α和il-2的th1型细胞因子的cd4 和cd8 t细胞的清晰群体(图15)。对于il-4和il-13th2型细胞因子,分析在任何时间点都没有显示任何特异性应答。在这二个模型中检测到低水平的il-5和/或il-10细胞因子产生cd4 t细胞,但在第28天仅balb/c小鼠与载体对照显著不同。即使对于该组群,6只小鼠中有5只(一个异常值)th1/th2平衡保持向th1偏移,证实了由疫苗引起的总体th1偏态t细胞应答(图16)。[0300]polypepi-scov-2接种也诱导体液应答,如通过balb/c的总小鼠igg和hu小鼠模型的人igg所测量的。在balb/c小鼠中,与载体对照组相比,第一剂(第14天)后接种导致疫苗诱导的igg产生。在第二剂后的稍后时间点,对于balb/c和hu小鼠模型皆观察到igg升高(图17)。在第28天时,从hu小鼠血浆测量的igg水平(平均115ng/ml,图17b)低于balb/c(平均529ng/ml,图17a)。这与已知的nod/shi-scid/il-2rγ无效免疫缺陷小鼠的局限性相一致,即它难以产生导致类别转换和igg产生的人类体液反应。(brehm等人,2013)。hu小鼠模型中人源化率为~50%进一步降低了理论上预期的igg水平。尽管有这些限制,但剂量依赖性的人igg产生表明疫苗产生的人体液应答。如所预期的,假定polypepi-scov-2肽不含构象b细胞表位,接种不产生可测量的中和抗体,如使用pna测定从hu小鼠的血清所评估的。在1∶25稀释度的检测极限下,每个试验样本的50%中和抗体滴度(nt50)是无法检测的(数据未显示)。[0301]表10abalb/c小鼠的安全性分析、临床评分数据表。临床安全评分是通过对五个不同的临床症状(被毛、运动、活动、苍白、体重)的描述来确定的,具体如下。被毛:0分——正常;1分——缺乏梳理、部分脱毛;2分——大量脱毛、伤口、出血、炎症。运动:0分——正常;2分——运动缓慢,一只动物瘫痪;3分——吃喝困难,多于一只动物瘫痪。活动:0分——正常;1分——激动、反应过度、反应不足;3分——瘫痪。苍白:0分——正常;1分——轻微(看不到耳部血管);2分——严重(耳朵和脚受到影响)。体重:0分——正常;2分——椎体分节明显,骨盆骨可触及;3分——骨骼结构突出。允许的最大累积临床评分:6。n.a.:不适用。[0302][0303]表10bhu小鼠的安全性分析、临床评分数据表。临床安全评分是通过对五个不同的临床症状(被毛、运动、活动、苍白、体重)的描述来确定的,具体如下。被毛:0分——正常;1分——缺乏梳理、部分脱毛;2分——大量脱毛、伤口、出血、炎症。运动:0分——正常;2分——运动缓慢,一只动物瘫痪;3分——吃喝困难,多于一只动物瘫痪。活动:0分——正常;1分——激动、反应过度、反应不足;3分——瘫痪。苍白:0分——正常;1分——轻微(看不到耳部血管);2分——严重(耳朵和脚受到影响)。体重:0分——正常;2分——椎体分节明显,骨盆骨可触及;3分——骨骼结构突出。允许的最大累积临床评分:6。n.a.:不适用。[0304][0305]表10c安全性分析、尸检数据表。通过肉眼观察脾、肝、肾、胃和肠进行尸检。[0306][0307][0308]covid-19康复供体的polypepi-scov-2肽特异性t细胞应答[0309]接下来,我们旨在通过研究在covid-19康复供体的血液中循环的疫苗特异性t细胞,证明在接种疫苗的动物中检测到的强和广泛的t细胞应答与人类相关(基线数据报道在表8中)。[0310]在17个康复期和四个健康供体中研究疫苗肽与康复期免疫组分的反应性。根据来源蛋白,使用分为四个池的九个30聚体疫苗肽检测疫苗反应性cd4 t细胞:s,n,m、和e肽。cd8 t细胞应答使用对应于为根据其源蛋白(s、n、m、和e肽;表8粗体)分组为四个池的九个疫苗肽中的每一个所定义的显性和共有hlai类pepi的9聚体试验肽测量,如动物实验中所用。[0311]使用可以检测快速活化的效应期t细胞应答的离体fluorospot分析,与健康受试者相比,用30聚体(平均dsfu:48.1,p=0.014)和9聚体肽(平均dsfu:16.5,p=0.011)皆检测到显着数量的疫苗反应性的表达ifn-γ的t细胞(图19a和b)。对四个蛋白质特异性肽池的详细分析,揭示了17个供体中的三个用30聚体疫苗肽与所有四个结构抗原反应;82%的供体与二个抗原反应,59%的供体与三个抗原反应。值得注意的是,也可以鉴定出对所有17个供体中的四个抗原中的至少一个和对53%中的至少二个抗原的,短的9聚体特异性cd8 t细胞应答(表11)。[0312]表11通过离体fluorospot分析测定,covid-19恢复期供体患者对polypepi-scov-2疫苗靶向的一个、二个、三个或所有四个病毒抗原的应答率。九聚体是hlai类热点pepi,其嵌入在与以下四个结构蛋白相应的每个30聚体疫苗肽中:s,刺突;n,核蛋白;m,隔膜;包膜蛋白。[0313][0314]如通过ics分析所确定的,用9聚体肽刺激导致平均而言t细胞组成83%的cd8 t细胞、和17%的cd4 t细胞(图18a)。有趣的是,30聚体试验肽与cd4 和cd8 t细胞皆以50∶50的平均比例反应(图18a)。t细胞的功能性试验揭示cd8 t细胞主要产生ifn-γ、tnf-α、和il-2(具有少量il-4和il-10),而cd4 t细胞主要对il-2和ifn-γ呈阳性。回忆应答显示了清楚的th1细胞因子特征;th2应答不存在于用30聚体疫苗肽的回忆应答中(图18b)。[0315]接着,我们确定离体检测的、可用的、快速活化的t细胞是否也可以在疫苗肽存在下体外扩增。使用富集的elispot,与健康受试者相比,用30聚体(平均dsfu=3746,p=0.025)和9聚体(平均dsfu=2088,p=0.028)肽池皆检测到显着数量的疫苗反应性的表达ifn-γ的t细胞(图19a)。还相对于用含有47个长的肽的商业化的、大的sars-cov-2肽集合(smno)检测的应答,评价了polypepi-scov-2衍生的t细胞应答(30聚体池)的强度,所述肽衍生自结构(s、m、n)和非结构(开放阅读框orf-3a和7a)蛋白质二者。从二个库中获得的相对强度有利于covid-19供体中的疫苗池,而更健康的供体与商业肽库反应,证实了polypepi-scov-2疫苗的特异性提高(图19b)。[0316]为了证实和进一步描述在covid-19回收的个体中离体检测到的polypepi-scov-2特异性t细胞应答的多特异性,我们定义了由它们的t细胞靶向的独特肽。我们首先去卷积肽池,并使用体外扩增试验对与每个疫苗肽对应的每个9聚体hlai类pepi特异性的cd8 t细胞应答(图20)。分析揭示,每个9聚体肽被若干受试者识别;对于n4(93%)、s9(87%)、s2、n1、m1(80%)、e1(60%)、s5、n2(40%)观察到covid-19康复期供体中的最高识别率(图19c)。[0317]九个肽特异性cd8 t细胞应答的详细分析揭示100%的covid-19回收的受试者具有用至少一个肽再活化的polypepi-scov-2特异性t细胞,93%用二个以上肽再活化、87%用五个以上肽再活化、而27%具有对所有九个疫苗肽特异性的t细胞池。在蛋白质水平上,87%的受试者具有针对多(三)种蛋白质的t细胞,并且15个测量的供体中的八个(53%)与所有四个靶向病毒蛋白质反应(图19c)。这些数据证实了polypepi-scov-2肽对于个体是显性的并且在covid-19受试者之间是共享的,并且在康复群体中获得的多肽特异性t细胞频率与计算机模拟组群的共享pepi的预测频率良好比对(表9)。此外,ferretti等人在涉及康复的系统、费力的t细胞表位筛选研究中独立地证实了我们的疫苗肽的一些片段(表位)是共享的免疫显性表位。(27)[0318]对于我们的恢复期对象组群,疫苗特异性t细胞应答的宽度和强度与从症状发作的时间不相关,表明这些t细胞是持久的(长达5个月)(图21)。[0319]为了证明pepi(多hla结合表位)可以在个体水平产生t细胞应答,我们首先确定每个受试者的完整i类hla基因型,然后从elispot测定法中使用的九个9聚体肽的列表中预测可以结合人的至少三个hla等位基因的肽。对于每个受试者,九个肽中的二个和七个之间被证明是pepi。在预测的肽(pepi)中,84%通过elispot证实产生高度特异性t细胞应答(表12)。[0320]表11pepi预测与通过富集elispot测定法测量的covid-19康复期的免疫应答之间的一致性。个人表位(pepi)预测为≥3个自体hlai类等位基因结合9聚体表位,并与用相同的9聚体刺激测量的产生ifn-γ的cd8 t细胞应答比较。真阳性值以粗体字母突出显示。[0321][0322]多个自体等位基因结合表位与cd8 t细胞应答之间的相关性[0323]研究了对每个受试者检测的免疫原性肽的hla结合能力。首先,我们确定了每个受试者的完整hlai类基因型,然后预测了可以结合至fluorospot测定中所使用的九个共享9聚体肽中的每一个的自体hla等位基因的数目。然后我们将预测的hla结合表位与每个患者中对每个肽测量的cd8 t细胞应答相匹配(总共15×9=135个数据点,图25)。cd8 t细胞应答的强度倾向于与被多个自体hla等位基因所限制的表位相关(rs=0.188,p=0.028,图29b)。此外,我们观察到pepi产生的cd8 t细胞应答的强度(hla≥3)(dsfu中值=458)显著高于非pepi产生的(hla《3)(dsfu中值=110)(p=0.008)(图29b)。[0324]在135个数据点上,记录了98个阳性反应和37个阴性反应。在98个阳性反应中有37个是由pepi产生,而在37个阴性中仅有7个是pepi,其它的是限制于≤3个自体hla等位基因的表位(图25)。总之,2×2偶然性表揭示了t细胞应答与pepi关联(p=0.041,fisher确切概率),但不与hla限制性表位关联(p=1.000,fisher确切概率)(图25)。对于每个受试者,九个肽中有一到七个被证明是pepi。在预测的pepi中,44个中的37个(84%)通过ifn-γ荧光斑点分析证实其在给定的受试者中产生特异性t细胞应答(图29d和图25)。[0325]这些数据表明,受试者的完整hla基因型影响他们的cd8 t细胞应答,并且多个自体等位基因结合能力是免疫原性表位的关键特征。pepi通常低估了受试者的全部t细胞库,然而,它们精确地预测了受试者的pepi特异性cd8 t细胞应答。[0326]polypepi-scov-2反应性t细胞和sars-cov-2特异性抗体应答之间的相关性[0327]抗体产生需要t细胞依赖性b细胞活化。对于每个受试者,使用不同的商业试剂盒确定针对sars-cov-2的s和n抗原二者的不同水平的抗体应答(表8)。所有受试者均用euroimmuneelisa(igg)针对病毒s-1和roche试剂盒检测n相关抗体,检测为阳性。所有受试者测试为diaproigg和igm阳性(除了2个供体),17个中有7个检测为diaproiga检测混合的s-1和n蛋白特异性抗体应答(表8)。[0328]我们接着评价了polypepi-scov-2特异性cd4 t细胞反应性和抗体应答之间的相关性(图22)。通过elisa测量的与s-1蛋白特异性igg量相关的polypepi-scov-2反应性cd4 t细胞的总数(r=0.59,p=0.02,图22a)。接下来,分析了polypepi-scov-2疫苗(s2和s5)的特异性s-1蛋白衍生肽,而相关性是相似的(r=0.585,p=0.02,图22b)。类似地,用n蛋白衍生的polypepi-scov-2肽(n1、n2、n3和n4)检测的t细胞应答与用roche试剂盒检测的n特异性抗体呈现弱但不显著的相关性(图22c)。这些数据表明covid-19康复供体的polypepi-scov-2特异性cd4 t细胞应答和随后igg产生之间的联系。[0329]有趣的是,iga的产生与polypepi-scov-2特异性记忆cd4 t细胞应答相关(r=0.63,p=0.006,图6d)。对smno肽池反应的t细胞应答与任何抗体亚群都没有表现出相关性。这表明不是所有cd4 t细胞都有助于b细胞应答,因此有助于igg产生(数据未显示)。[0330]不同种族中预测的免疫原性[0331]我们在分布于来自美国骨髓供体数据库的16个不同族群的16,000个hla分型受试者的大组群中进行了我们的polypepi-scov-2疫苗的计算机模拟试验。(49)对于该大组群中的每个受试者,我们基于其完整的hlai类和ii类基因型预测了polypepi-scov-2特异性pepi。大多数受试者具有广泛的pepi库,其可能被转化至病毒特异性记忆cd8 t细胞克隆:预测98%的受试者具有抗至少二个疫苗肽的pepi,而95%、86%和70%分别抗三个、四个和五个肽(图23a)。[0332]计算机模拟试验显示≥98%的每个族群中的受试者将可能产生强细胞应答,具有针对疫苗中至少二个肽的cd8 和cd4 t细胞应答(图23b)。这种预测的高应答率对于covid-19报告的临床结果较差的种族(黑人、亚裔)也是如此。(25)基于这些数据,我们预计该疫苗将提供全球覆盖,不受种族和地理位置的影响。[0333]我们还使用16,000个群体(并且包括族群)来评估由其他人提出的理论总体覆盖率,即,筛选具有六个普遍hlai类等位基因中的至少一个的亚群,其被认为覆盖了总体群体的95%。(27,55,56)使用这种方法,我们观察到种族水平的显著异质性。尽管我们证实了挑选出的六个hla等位基因在高加索人和北美组群中是普遍的(91–93%),但是这些等位基因在所有其它族群中的频率较低,尤其是在非裔群体中(48–54%)(图23c)。我们推断,所提出的普遍hla等位基因集合可能涵盖种族加权的全球群体中的hla频率,但仅基于这些等位基因的用于疫苗接种目的表位选择将区分一些种族。因此,我们建议使用一种代表性的模型群体,该模型群体对人类种族中的异质性敏感,并且允许选择在种族之间的个体之间共享的pepi。值得注意的是,虽然我们没有观察到不同种族的个体的sars-cov-2蛋白衍生的表位生成能力基于它们的完整hla基因型的任何差异(图12)(这似乎没有解释在bame中观察到的较高的感染和死亡率),但是可以仔细选择用于亚单位疫苗的表位以解决bame组的hla基因型特征。观察到在不同族群中共有的pepi频率的异质性,特别是对于蛋白n,对整体疫苗的设计具有很大的影响(图26)。通过在大量真实受试者群体中进行“计算机模拟临床试验”,将族群内和族群间不同频率的靶组合为具有高整体覆盖率的候选疫苗是可行的。[0334]因此,为了在个体和群体/种族水平上皆最大化多抗原免疫应答,并且还考虑到肽的化学和可制造性特性,从sars-cov-2的四个结构蛋白中总共选择九个30聚体肽:来自刺突的三个肽(s)、来自核蛋白的四个肽(n)、和来自基质和包膜的各一个肽(m)和(e)。没有包含来自s蛋白受体结合结构域(rbd)的肽。总之,模型群体中的每个成员在九个肽中至少有二个hlai类pepi,97%的成员至少有三个(表9)。每个受试者具有多个用于疫苗肽的ii类pepi(表9)。每个受试者具有多个用于疫苗肽的ii类pepi(表9)。[0335]讨论[0336]我们证明了,当与montanideisa51vg佐剂一起施用时,polypepi-scov-2,一种包括九个合成的衍生自sars-cov-2病毒(s、n、m、e)的四个结构蛋白的长(30聚体)肽的多肽疫苗,在balb/c小鼠和人源化cd34 小鼠中是安全的和高度免疫原性的。此外,通过成功地再活化polypepi-scov-2特异性t细胞,在covid-19康复供体中证实了疫苗的免疫原性潜力,这与sars-cov-2感染产生的t细胞免疫广泛重叠。[0337]本发明的疫苗设计概念,在个体和群体水平上皆靶向多抗原免疫应答,代表了一种新的靶鉴定方法,其已经成功地用于癌症疫苗开发中以实现与临床环境中的初始功效相关的前所未有的免疫应答率。(32)对于covid-19,我们集中于选择sars-cov-2蛋白的片段,其含有在种族差异的hla基因分型个体之间共有的i类和ii类hlat细胞表位的重叠,并且其还产生针对整个病毒结构的差异和广泛的免疫应答。因此,我们选择相对长的30聚体片段以利于产生多重效应应答(b细胞和细胞毒性t细胞)和辅助t细胞应答。[0338]polypepi-scov-2疫苗在接种的balb/c和人源化cd34 小鼠中引发针对所有四个sars-cov-2蛋白的疫苗特异性cd8 和cd4 t细胞二者的期望的多抗原ifn-γ产生性t细胞应答,并且这些应答在加强剂量后更显著。covid-19康复期的回忆应答包括针对所有九个肽的快速激活效应型(离体测得)和扩展(体外测得)记忆型cd8 和cd4 t细胞应答,在100%供体中检测到polypepi-scov-2特异性t细胞。在个体水平上,用于从covid-19回收的polypepi-scov-2特异性t细胞池极其多样化:在其患病后1–5个月,每个供体平均具有七个不同的肽特异性t细胞集合,具有针对sars-cov-2蛋白的多个靶;87%的供体具有针对至少三个sars-cov-2蛋白的、而53%的供体具有针对所有四个的。此外,87%的受试者具有抗s蛋白的cd8 t细胞,这与i/ii期临床试验中报道的前沿covid-19疫苗候选物的免疫应答率相似)。(57)然而,我们发现s特异性(记忆)t细胞仅代表用疫苗肽检测的恢复期总t细胞所有组成成分的36%;剩余的64%几乎相等地分布在n、m、和e蛋白中。这些数据证实了越来越多的关注,即基于s蛋白的候选疫苗不能发挥人抗sars-cov-2免疫的全部潜力,特别是因为多样性t细胞应答与温和/无症状的covid-19有关。(4)[0339]t细胞和b细胞之间的相互作用是已知的产生抗体的浆细胞和产生记忆b细胞二者的机制。(59)在康复期抗体亚群的分析期间,我们发现抗原特异性igg水平和相应的肽特异性cd4 t细胞应答之间的相关性。这种相关性可能代表cd4 t细胞和抗体产生之间的联系,这一概念也得到动物模型中总igg产生的支持。结合igg抗体可以在随后的sars-cov-2暴露于接种者时与疫苗诱导的cd8 杀伤t细胞协同作用。这种相互作用可能导致有效的cd8 t细胞介导的直接杀伤感染细胞和igg介导的杀伤病毒感染细胞和病毒颗粒,也抑制th2依赖的免疫病理过程。以这种方式,预期二个细胞内端的细胞外病毒储库都受到攻击,以帮助快速清除病毒,即使在不存在中和抗体的情况下。(59,60)[0340]本数据证明个体对polypepi-scov-2肽组反应的抗sars-cov-2t细胞应答是hla基因型依赖性的。具体地,多个自体hla结合表位(pepi)以84%的准确度确定抗原特异性cd8 t细胞应答。尽管pepi通常低估了受试者的全部t细胞库,但它们是精确的靶标鉴定“工具”和pepi特异性免疫应答的预测物,克服了在仅使用表位结合亲和力作为t细胞应答预测物的领域中通常观察到的高假阳性率。(34,61)因此,通过基于(仅有)高加索人个体的完整hla基因型的t细胞应答的经验证的pepi预测,以及在对人免疫应答建模的动物中对polypepi-scov-2诱导的免疫应答的仔细解释,我们的发现可以外推至16,000个hla基因分型的个体和16个人种族的大组群。在该大美国组群中代表的族群涵盖全球人口的组成,但是它们没有就其全球代表性进行加权(对于每个种族有意使用n=1,000个受试者)。(62)基于此,polypepi-scov-2将可能在》98%的全球群体中产生有意义的免疫应答(针对至少二个疫苗肽的cd8 和cd4 t细胞应答),而与种族无关。相比之下,基于全球频繁出现的hla等位基因的t细胞表位的疫苗设计方法会漏掉对黑加勒比海人、非裔、非裔美国人、和越南裔中约50%的人产生的免疫反应。通过接种疫苗在高百分比的接种疫苗的受试者中均匀地诱导有意义的免疫应答(广泛和多功能记忆应答,模拟由sars-cov-2诱导的免疫的异质性)对于实现期望的“群体免疫”是必要的。据认为,约25–50%的人群必须对病毒免疫以实现对群体传播的抑制。(3)然而,第一代covid-19疫苗正被测试显示疾病风险降低至少50%,但它们不能预期将病毒传播降低到相当的程度。这个事实与这些疫苗也不提供长期免疫力的可能性一起暗示需要第二代工具来对抗大流行。(3)[0341]我们相信,集中于每个受试者中的几个靶将更好地重演由病毒诱导的天然t细胞免疫,导致有效的、长期的记忆应答。为此目的,基于合成多肽的平台技术被认为是具有若干关键优点的安全和免疫原性疫苗接种策略。具有montanide佐剂的相同类型的肽疫苗在》6,000名患者和231名健康志愿者中是安全的和良好耐受的。(63–69)对sars的研究表明,限制包含完整病毒蛋白的候选冠状病毒疫苗具有更有益的安全性。此外,有效的th1偏态t细胞应答和非构象(线性)b细胞表位减轻了理论抗体介导的疾病增强和基于th2的免疫病理过程的风险。[0342]基于肽的疫苗迄今为止仅取得有限的成功,但这可归因于缺乏关于使用哪些肽的知识。通过理解个体的遗传背景如何能够响应特定的肽,降低了这种不确定性,如我们以上所证明的。[0343]总之,本文所述的基于肽的多表位编码疫苗设计证明了安全性和异常广泛的临床前免疫原性,并且在仔细的临床试验后,预期提供针对sars-cov-2的有效的第二代疫苗。[0344]实施例11——针对sars的交叉保护polypepi-scov-2特异性多肽的分析。[0345]通过预测模型群体(mp,n=433)内的自体的≥3hla结合表位的高频率选择疫苗组合物的多肽,使得能够选择在不同病原体物种之间共享的序列。例如,在sars病毒和sars-cov-2的蛋白质组中有几个保守的和100%相同的肽片段(seqidno:6和9–17)。通过分析这些片段,发明人在高百分比的mp个体中鉴定出了适当的≥3个hla结合9聚体pepi。99%具有至少一个来自所分析的seqidno:1–17(表13和图27a)的组合物的预测共有表位,94%的mp具有至少二个hlai类特异性(cd8 t细胞)pepi,因此预测该组合物在94%的mp中诱导多肽免疫应答。平均而言,3或4个sars衍生的共享肽被预测为与17个30聚体的肽发生反应。如果一个肽代表一个t细胞克隆(预测的和elispot测定的免疫应答的相关性在实施例5中得到证实,例如ppv=0.79),如果这17个肽用于免疫,这些计算机模拟结果可解释为诱导多肽特异性免疫原性t细胞应答。[0346]表13在模型群体(n=433)中预测的来自最初为sars-cov-2设计的17个30聚体肽的共享sars-cov表位的多抗原(多蛋白)应答率。[0347]多重肽(n=433)polypepi-scov-2共享sars-cov》=1100%99%》=2100%94%》=399%88%》=499%61%》=597%31%》=693%7%》=790%0%》=883%0%》=972%0%》=1064%0%》=1153%0%》=1242%0%》=1331%0%》=1420%0%》=1511%0%》=162%0%》=171%0%肽平均:10.573.79[0348]针对至少二个抗原的多蛋白或多抗原应答在91%的个体中共享,如果用于sars组群,则代表对该组合物的良好覆盖(图28a)。这些数据表明,这17个肽具有诱导抗sars的多肽和多蛋白特异性t细胞应答的潜力。[0349]结果也在16,000人的人类群体中得到证实。95%的个体具有针对至少2个共有肽的多hla限定的个人表位。[0350]表14从最初为sars-cov-2设计的17个30聚体多肽中预测出的人类大群体(n=16,000)对共享sars-cov表位的多抗原(多蛋白)反应率。[0351]多重肽(n=16,000)polypepi-scov-2共享sars-cov》=1100%98%》=2100%95%》=399%87%》=498%62%》=596%29%》=692%8%》=788%0%》=882%0%》=974%0%》=1065%0%》=1156%0%》=1245%0%》=1333%0%》=1421%0%》=1511%0%》=165%0%》=171%0%肽平均:10.653.79[0352]这些数据代表了鉴定具有针对不同病原体,例如针对sars的共有特异性的丰富的、多hla结合表位的机会(图27b和图28b)。[0353]实施例12——编码和表达seqidno:1–17的核酸制剂[0354]seqidno:1–17多肽可以不同的制剂制备,以水溶性或含佐剂的肽溶解或乳化用于注射。另外,如果3个字母的氨基酸编码被翻译成与seqidno:1–17相同的氨基酸序列,该多肽可以被编码成mrna、rna或dna制剂并在质粒dna或病毒载体中表达。用于递送和/或表达所编码的多肽的合适载体包含但不限于腺病毒载体、腺相关载体、慢病毒或逆转录病毒载体、痘病毒衍生载体、新城疫病毒载体、植物病毒载体如镶嵌病毒载体、和杂交载体。[0355]抗原呈递细胞中表达的蛋白质通过与疫苗皮下或皮内递送时apc摄取的多肽类似的hlai类和ii类抗原呈递途径加工和呈递。[0356]表15seqidno:1–17的氨基酸序列的对应的rna和dna序列。iupac核苷酸代码缩写:a:腺嘌呤;c:胞嘧啶,g:鸟嘌呤,t:胸腺嘧啶,u:尿嘧啶,r:a或g,y:c或t/u,s:g或c,w:a或t/u,k:g或t/u,m:a或c,b:c或g或t/u,d:a或g或t/u,h:a或c或t/u,v:a或c或g,n:a或c或t/u或g。t/u代表等同的核苷酸:t表示dna序列,u表示rna序列。[0357][0358][0359]实施例13——多个自体等位基因结合表位和cd8 t细胞应答之间的相关性[0360]发明人研究了从分析的covid-19康复供体对每个受试者(n=15)检测的免疫原性肽seqidno:2、5、9、12–17的hla结合能力。[0361]首先,发明人确定了每个受试者的完整hlai类基因型,然后预测了可以结合在用于检测肽特异性的产生ifn-γ的t细胞的fluorospot测定中所使用的九个共享的9聚体肽中的每一个的自体hla等位基因的数目。然后将预测的hla结合表位与每个患者中对每个肽测量的cd8 t细胞应答相匹配(总共15×9=135个数据点,图25)。cd8 t细胞应答的强度倾向于与被多个自体hla等位基因所限制的表位相关(rs=0.189,p=0.029,图29a)。此外,pepi产生的cd8 t细胞应答的强度(hla≥3)(dsfu中值=458)显着高于非pepi产生的(hla《3)(dsfu中值=110)(p=0.008)(图29b)。[0362]在135个数据点上,记录了98个阳性反应和37个阴性反应。在98个阳性反应中有37个是由pepi产生,而在37个阴性中仅有7个是pepi,其它的是限制于≤3个自体hla等位基因的表位(图25)。总之,2×2偶然性表揭示了t细胞应答与pepi关联(p=0.041,fisher确切概率),但不与hla限制性表位关联(p=1.000,fisher确切概率)(图29c)。对于每个受试者,九个肽中有一到七个被证明是pepi。在预测的pepi中,44个中的37个(84%)通过ifn-γ荧光斑点分析证实其在给定的受试者中产生特异性t细胞应答(图29d和图25)。[0363]这些数据表明,受试者的完整hla基因型影响其cd8 t细胞应答,并且多个自体等immuneresponsetoexperimentalcoronavirusinfectionofman.epidemiolinfect105,435-446(1990).[0373]5.q.x.long,x.j.tang,q.l.shi,q.li,h.j.deng,j.yuan,j.l.hu,w.xu,y.zhang,f.j.lv,k.su,f.zhang,j.gong,b.wu,x.m.liu,j.j.li,j.f.qiu,j.chen,a.l.huang,clinicalandimmunologicalassessmentofasymptomaticsars-cov-2infections.natmed26,1200-1204(2020).[0374]6.t.sekine,a.perez-potti,o.rivera-ballesteros,k.j.-b.gorin,a.olsson,s.llewellyn-lacey,h.kamal,g.bogdanovic,s.muschiol,d.j.wullimann,t.kammann,j.t.parrot,e.folkesson,o.rooyackers,l.i.eriksson,a.t.allander,j.albert,m.nielsen,j.s.gredmark-russ,n.k.j.k.sandberg,d.a.price,h.-g.ljunggren,s.aleman,m.buggert,robusttcellimmunityinconvalescentindividualswithasymptomaticormildcovid-19.(2020).[0375]7.h.m.staines,d.e.kirwan,d.j.clark,e.r.adams,y.augustin,r.l.byrne,m.cocozza,a.i.cubas-atienza,l.e.cuevas,m.cusinato,b.m.o.davies,m.davis,p.davis,a.duvoix,n.m.eckersley,d.forton,a.fraser,g.garrod,l.hadcocks,q.hu,m.johnson,g.a.kay,k.klekotko,z.lewis,j.mensah-kane,s.menzies,i.monahan,c.moore,g.nebe-von-caron,s.i.owen,c.sainter,a.a.sall,j.schouten,c.williams,j.wilkins,k.woolston,j.r.a.fitchett,s.krishna,t.planche,dynamicsofiggseroconversionandpathophysiologyofcovid-19infections.(2020).[0376]8.a.grifoni,d.weiskopf,s.i.ramirez,j.mateus,j.m.dan,c.r.moderbacher,s.a.rawlings,a.sutherland,l.premkumar,r.s.jadi,d.marrama,a.m.desilva,a.frazier,a.f.carlin,j.a.greenbaum,b.peters,f.krammer,d.m.smith,s.crotty,a.sette,targetsoftcellresponsestosars-cov-2coronavirusinhumanswithcovid-19diseaseandunexposedindividuals.cell181,1–13(2020).[0377]9.n.l.bert,a.t.tan,k.kunasegaran,c.y.l.tham,m.hafezi,a.chia,m.chng,m.lin,n.tan,m.linster,w.n.chia,m.i.c.chen,l.-f.wang,e.e.ooi,s.kalimuddin,p.a.tambyah,j.g.-h.low,y.-j.tan,a.bertoletti,differentpatternofpre-existingsars-cov-2specifictcellimmunityinsars-recoveredanduninfectedindividuals.(2020).[0378]10.y.peng,a.j.mentzer,g.liu,x.yao,z.yin,d.dong,w.dejnirattisai,t.rostron,p.supasa,c.liu,c.lopez-camacho,j.slon-campos,y.zhao,d.i.stuart,g.c.paesen,j.m.grimes,a.a.antson,o.w.bayfield,d.hawkins,d.s.ker,b.wang,l.turtle,k.subramaniam,p.thomson,p.zhang,c.dold,j.ratcliff,p.simmonds,t.desilva,p.sopp,d.wellington,u.rajapaksa,y.l.chen,m.salio,g.napolitani,w.paes,p.borrow,b.m.kessler,j.w.fry,n.f.schwabe,m.g.semple,j.k.baillie,s.c.moore,p.j.m.openshaw,m.a.ansari,s.dunachie,e.barnes,j.frater,g.kerr,p.goulder,t.lockett,r.levin,y.zhang,r.jing,l.p.ho,t.c.c.oxfordimmunologynetworkcovid-19response,i.c.investigators,r.j.cornall,c.p.conlon,p.klenerman,g.r.screaton,j.mongkolsapaya,a.mcmichael,j.c.knight,g.ogg,t.dong,broadandstrongmemorycd4( )andcd8( )tcellsinducedbysars-cov-2inukconvalescentindividualsfollowingcovid-19.natimmunol,(2020).[0379]11.a.nelde,t.bilich,j.s.heitmann,y.maringer,h.r.salih,m.roerden,m.lübke,j.bauer,j.rieth,m.wacker,a.peter,s.b.traenkle,p.d.kaiser,u.rothbauer,m.becker,d.junker,g.krause,m.strengert,n.schneiderhan-marra,m.f.templin,t.o.joos,d.j.kowalewski,v.stos-zweifel,m.fehr,m.graf,l.-c.gruber,d.rachfalski,b.preuβ,i.hagelstein,m.t.bakchoul,c.gouttefangeas,o.kohlbacher,r.klein,s.h.-g.rammensee,j.s.walz,sars-cov-2t-cellepitopesdefineheterologousandcovid-19-inducedt-cellrecognition.(2020).[0380]12.j.zhao,a.n.alshukairi,s.a.baharoon,w.a.ahmed,a.a.bokhari,a.m.nehdi,l.a.layqah,m.g.alghamdi,m.m.algethamy,a.m.dada,i.khalid,m.boujelal,s.m.aljohani,l.vogel,k.subbarao,a.mangalam,c.wu,p.teneyck,s.perlman,j.zhao,recoveryfromthemiddleeastrespiratorysyndromeisassociatedwithantibodyandt-cellresponses.sciimmunol2,(2017).[0381]13.b.diao,c.wang,y.tan,x.chen,y.liu,l.ning,l.chen,m.li,y.liu,g.wang,z.yuan,z.feng,y.zhang,y.wu,y.chen,reductionandfunctionalexhaustionoftcellsinpatientswithcoronavirusdisease2019(covid-19).frontimmunol11,827(2020).[0382]14.t.m.snyder,r.m.gittelman,m.klinger,d.h.may,e.j.osborne,r.taniguchi,h.j.zahid,i.m.kaplan,j.n.dines,m.n.noakes,r.pandya,x.chen,s.elasady,e.svejnoha,p.ebert,m.w.pesesky,p.dealmeida,h.o'donnell,q.degottardi,g.keitany,j.lu,a.vong,r.elyanow,p.fields,j.greissl,l.baldo,s.semprini,c.cerchione,m.mazza,o.m.delmonte,k.dobbs,g.carreno-tarragona,s.barrio,l.imberti,a.sottini,e.quiros-roldan,c.rossi,a.biondi,l.r.bettini,m.d'angio,p.bonfanti,m.f.tompkins,c.alba,c.dalgard,v.sambri,g.martinelli,j.d.goldman,j.r.heath,h.c.su,l.d.notarangelo,j.martinez-lopez,j.m.carlson,h.s.robins,magnitudeanddynamicsofthet-cellresponsetosars-cov-2infectionatbothindividualandpopulationlevels.medrxiv,(2020).[0383]15.j.zhao,j.zhao,s.perlman,tcellresponsesarerequiredforprotectionfromclinicaldiseaseandforvirusclearanceinsevereacuterespiratorysyndromecoronavirus-infectedmice.jvirol84,9318-9325(2010).[0384]16.r.channappanavar,c.fett,j.zhao,d.k.meyerholz,s.perlman,virus-specificmemorycd8tcellsprovidesubstantialprotectionfromlethalsevereacuterespiratorysyndromecoronavirusinfection.jvirol88,11034-11044(2014).[0385]17.r.channappanavar,j.zhao,s.perlman,tcell-mediatedimmuneresponsetorespiratorycoronaviruses.immunolres59,118-128(2014).[0386]18.w.li,m.d.joshi,s.singhania,k.h.ramsey,a.k.murthy,peptidevaccine:progressandchallenges.vaccines(basel)2,515-536(2014).[0387]19.m.black,a.trent,m.tirrell,c.olive,advancesinthedesignanddeliveryofpeptidesubunitvaccineswithafocusontoll-likereceptoragonists.expertreviewofvaccines9,157-173(2010).[0388]20.a.l.thompson,h.f.staats,cytokines:thefutureofintranasalvaccineadjuvants.clindevimmunol2011,289597(2011).[0389]21.n.petrovsky,j.c.aguilar,vaccineadjuvants:currentstateandfuturetrends.immunolcellbiol82,488-496(2004).[0390]22.d.sesardic,syntheticpeptidevaccines.jmedmicrobiol39,241-242(1993).[0391]23.m.s.bijker,c.j.melief,r.offringa,s.h.vanderburg,designanddevelopmentofsyntheticpeptidevaccines:past,presentandfuture.expertrevvaccines6,591-603(2007).[0392]24.s.nayak,r.w.herzog,progressandprospects:immuneresponsestoviralvectors.genether17,295-304(2010).[0393]25.d.pan,s.sze,j.s.minhas,m.n.bangash,n.pareek,p.divall,c.m.williams,m.r.oggioni,i.b.squire,l.b.nellums,w.hanif,k.khunti,m.pareek,theimpactofethnicityonclinicaloutcomesincovid-19:asystematicreview.eclinicalmedicine23,100404(2020).[0394]26.r.w.aldridge,d.lewer,s.v.katikireddi,r.mathur,n.pathak,r.burns,e.b.fragaszy,a.m.johnson,d.devakumar,i.abubakar,a.hayward,black,asianandminorityethnicgroupsinenglandareatincreasedriskofdeathfromcovid-19:indirectstandardisationofnhsmortalitydata.wellcomeopenres5,88(2020).[0395]27.a.p.ferretti,t.kula,y.wang,d.m.nguyen,a.weinheimer,g.s.dunlap,q.xu,n.nabilsi,c.r.perullo,a.w.cristofaro,h.j.whitton,a.virbasius,k.j.olivier,l.b.baiamonte,a.t.alistar,e.d.whitman,s.a.bertino,s.chattopadhyay,g.macbeath,covid-19patientsformmemorycd8 tcellsthatrecognizeasmallsetofsharedimmunodominantepitopesinsars-cov-2.medrxiv,2020.2007.2024.20161653(2020).[0396]28.u.sahin,e.derhovanessian,m.miller,b.p.kloke,p.simon,m.lower,v.bukur,a.d.tadmor,u.luxemburger,b.schrors,t.omokoko,m.vormehr,c.albrecht,a.paruzynski,a.n.kuhn,j.buck,s.heesch,k.h.schreeb,f.muller,i.ortseifer,i.vogler,e.godehardt,s.attig,r.rae,a.breitkreuz,c.tolliver,m.suchan,g.martic,a.hohberger,p.sorn,j.diekmann,j.ciesla,o.waksmann,a.k.bruck,m.witt,m.zillgen,a.rothermel,b.kasemann,d.langer,s.bolte,m.diken,s.kreiter,r.nemecek,c.gebhardt,s.grabbe,c.holler,j.utikal,c.huber,c.loquai,o.tureci,personalizedrnamutanomevaccinesmobilizepoly-specifictherapeuticimmunityagainstcancer.nature547,222-226(2017).[0397]29.p.a.ott,z.hu,d.b.keskin,s.a.shukla,j.sun,d.j.bozym,w.zhang,a.luoma,a.giobbie-hurder,l.peter,c.chen,o.olive,t.a.carter,s.li,d.j.lieb,t.eisenhaure,e.gjini,j.stevens,w.j.lane,i.javeri,k.nellaiappan,a.m.salazar,h.daley,m.seaman,e.i.buchbinder,c.h.yoon,m.harden,n.lennon,s.gabriel,s.j.rodig,d.h.barouch,j.c.aster,g.getz,k.wucherpfennig,d.neuberg,j.ritz,e.s.lander,e.f.fritsch,n.hacohen,c.j.wu,animmunogenicpersonalneoantigenvaccineforpatientswithmelanoma.nature547,217-221(2017).[0398]30.a.w.purcell,j.mccluskey,j.rossjohn,morethanonereasontorethinktheuseofpeptidesinvaccinedesign.natrevdrugdiscov6,404-414(2007).[0399]31.o.lorincz,j.toth,m.megyesi,k.pántya,i.miklos,e.somogyi,z.csiszovszki,p.pales,l.molnar,e.r.1935pcomputationalmodeltopredictresponserateofclinicaltrials.annalsofoncology30,(2019).[0400]32.j.hubbard,c.cremolini,r.graham,r.moretto,j.mitchell,j.wessling,e.toke,z.csiszovszki,o.l.molnár,e.somogyi,m.megyesi,k.pántya,j.tóth,p.páles,i.miklós,a.falcone,p329polypepi1018off-theshelfvaccineasadd-ontomaintenancetherapyachieveddurabletreatmentresponsesinpatientswithmicrosatellite-stablemetastaticcolorectalcancerpatients(mssmcrc).journalforimmunotherapyofcancer7(34thannualmeeting&pre-conferenceprogramsofthesocietyforimmunotherapyofcancer(sitc2019):part1),282(2019).[0401]33.j.hubbard,c.cremolini,r.p.graham,r.moretto,j.mitchell,j.wessling,e.r.z.csiszovszki,o.lorincz,l.molnar,e.somogyi,m.megyesi,k.pántya,j.toth,p.pales,i.miklos,a.falcone,606pevaluationofsafety,immunogenicityandpreliminaryefficacyofpolypepi1018vaccineinsubjectswithmetastaticcolorectalcancer(mcrc)withapredictivebiomarker.annalsofoncology30,(2019).[0402]34.e.somogyi,z.csiszovszki,o.lorincz,j.toth,l.molnar,w.s.urban,t.k.pántya,p.pales,m.megyesi,e.r.1181pdpersonalantigenselectioncalculator(pascal)forthedesignofpersonalcancervaccines.annalsofoncology30,(2019).[0403]35.u.s.nationallibraryofmedicine.severeacuterespiratorysyndromecoronavirus2https://www.ncbi.nlm.nih.gov/genome/browse#!/viruses/86693/.[0404]36.f.madeira,y.m.park,j.lee,n.buso,t.gur,n.madhusoodanan,p.basutkar,a.r.n.tivey,s.c.potter,r.d.finn,r.lopez,theembl-ebisearchandsequenceanalysistoolsapisin2019.nucleicacidsres47,w636-w641(2019).[0405]37.b.korber,w.m.fischer,s.gnanakaran,h.yoon,j.theiler,w.abfalterer,b.foley,e.e.giorgi,t.bhattacharya,m.d.parker,d.g.partridge,c.m.evans,t.m.freeman,t.i.desilva,c.c.labranche,d.c.montefiori,spikemutationpipelinerevealstheemergenceofamoretransmissibleformofsars-cov-2.(2020).[0406]38.centersfordiseasecontrolandprevention.coronavirushttps://www.cdc.gov/coronavirus/types.html.[0407]39.t.u.consortium,uniprot:aworldwidehubofproteinknowledge.nucleicacidsresearch47,d506-d515(2018).[0408]40.c.internationalhapmap,d.m.altshuler,r.a.gibbs,l.peltonen,d.m.altshuler,r.a.gibbs,l.peltonen,e.dermitzakis,s.f.schaffner,f.yu,l.peltonen,e.dermitzakis,p.e.bonnen,d.m.altshuler,r.a.gibbs,p.i.debakker,p.deloukas,s.b.gabriel,r.gwilliam,s.hunt,m.inouye,x.jia,a.palotie,m.parkin,p.whittaker,f.yu,k.chang,a.hawes,l.r.lewis,y.ren,d.wheeler,r.a.gibbs,d.m.muzny,c.barnes,k.darvishi,m.hurles,j.m.korn,k.kristiansson,c.lee,s.a.mccarrol,j.nemesh,e.dermitzakis,a.keinan,s.b.montgomery,s.pollack,a.l.price,n.soranzo,p.e.bonnen,r.a.gibbs,c.gonzaga-jauregui,a.keinan,a.l.price,f.yu,v.anttila,w.brodeur,m.j.daly,s.leslie,g.mcvean,l.moutsianas,h.nguyen,s.f.schaffner,q.zhang,m.j.ghori,r.mcginnis,w.mclaren,s.pollack,a.l.price,s.f.schaffner,f.takeuchi,s.r.grossman,i.shlyakhter,e.b.hostetter,p.c.sabeti,c.a.adebamowo,m.w.foster,d.r.gordon,j.licinio,m.c.manca,p.a.marshall,i.matsuda,d.ngare,v.o.wang,d.reddy,c.n.rotimi,c.d.royal,r.r.sharp,c.zeng,l.d.brooks,j.e.mcewen,integratingcommonandraregeneticvariationindiversehumanpopulations.nature467,52-58(2010).[0409]41.j.robinson,j.a.halliwell,j.d.hayhurst,p.flicek,p.parham,s.g.marsh,theipdandimgt/hladatabase:allelevariantdatabases.nucleicacidsres43,d423-431(2015).[0410]42.j.robinson,j.a.halliwell,h.mcwilliam,r.lopez,s.g.marsh,ipd‑‑theimmunopolymorphismdatabase.nucleicacidsres41,d1234-1240(2013).[0411]43.k.yusim,b.t.m.korber,c.brander,d.barouch,r.deboer,b.f.haynes,r.koup,j.p.moore,b.d.walker,d.i.watkins,inhivmolecularimmunology2014.(losalamosnationallaboratory,theoreticalbiologyandbiophysics,https://www.hiv.lanl.gov/content/immunology/compendium.html,2014),vol.la-ur15-20754.[0412]44.g.pawelec,s.g.marsh,estdab:acollectionofimmunologicallycharacterisedmelanomacelllinesandsearchabledatabank.cancerimmunolimmunother55,623-627(2006).[0413]45.j.robinson,c.h.roberts,i.a.dodi,j.a.madrigal,g.pawelec,l.wedel,s.g.marsh,theeuropeansearchabletumourlinedatabase.cancerimmunolimmunother58,1501-1506(2009).[0414]46.j.r.arguello,a.m.little,e.bohan,j.m.goldman,s.g.marsh,j.a.madrigal,highresolutionhlaclassitypingbyreferencestrandmediatedconformationanalysis(rsca).tissueantigens52,57-66(1998).[0415]47.w.helmberg,r.dunivin,m.feolo,thesequencing-basedtypingtoolofdbmhc:typinghighlypolymorphicgenesequences.nucleicacidsres32,w173-175(2004).10,10895(2020).[0429]61.e.r.toke,m.megyesi,l.molnar,j.tóth,o.s.h.vanderburg,m.welters,c.j.melief,w.s.urban,t.roehnisch,u.heine,e.somogyi,z.csiszovszki,k.pántya,p.páles,i.miklós,f.lori,j.lisziewicz,predictiontheclinicaloutcomesofcancerpatientsafterpeptidevaccination.journalofclinicaloncology37,e14295(2019).[0430]62.demographicsoftheworld.https://www.quora.com/what-are-all-the-races-and-their-world-population-demographics-the-entire-world.[0431]63.s.ascarateil,a.puget,m.-e.koziol,safetydataofmontanideisa51vgandmontanideisa720vg,twoadjuvantsdedicatedtohumantherapeuticvaccines.journalforimmunotherapyofcancer3,(2015).[0432]64.j.atsmon,e.kate-ilovitz,d.shaikevich,y.singer,i.volokhov,k.y.haim,t.ben-yedidia,safetyandimmunogenicityofmultimeric-001‑‑anoveluniversalinfluenzavaccine.jclinimmunol32,595-603(2012).[0433]65.b.s.graham,m.j.mcelrath,m.c.keefer,k.rybczyk,d.berger,k.j.weinhold,j.ottinger,g.ferarri,d.c.montefiori,d.stablein,c.smith,r.ginsberg,j.eldridge,a.duerr,p.fast,b.f.haynes,immunizationwithcocktailofhiv-derivedpeptidesinmontanideisa-51isimmunogenic,butcausessterileabscessesandunacceptablereactogenicity.plosone5,e11995(2010).[0434]66.s.herrera,o.l.fernández,o.vera,w.cárdenas,o.ramírez,r.palacios,m.chen-mok,g.corradin,m.arévalo-herrera,phaseisafetyandimmunogenicitytrialofplasmodiumvivaxcsderivedlongsyntheticpeptidesadjuvantedwithmontanideisa720ormontanideisa51.theamericanjournaloftropicalmedicineandhygiene84,12-20(2011).[0435]67.g.pialoux,j.l.excler,y.rivière,g.gonzalez-canali,v.feuillie,p.coulaud,j.c.gluckman,t.j.matthews,b.meignier,m.p.kieny,etal.,aprime-boostapproachtohivpreventivevaccineusingarecombinantcanarypoxvirusexpressingglycoprotein160(mn)followedbyarecombinantglycoprotein160(mn/lai).theagisgroup,andl'agencenationalederecherchesurlesida.aidsreshumretroviruses11,373-381(1995).[0436]68.o.pleguezuelos,s.robinson,g.a.stoloff,w.caparrós-wanderley,syntheticinfluenzavaccine(flu-v)stimulatescellmediatedimmunityinadouble-blind,randomised,placebo-controlledphaseitrial.vaccine30,4655-4660(2012).[0437]69.y.wu,r.d.ellis,d.shaffer,e.fontes,e.m.malkin,s.mahanty,m.p.fay,d.narum,k.rausch,a.p.miles,j.aebig,a.orcutt,o.muratova,g.song,l.lambert,d.zhu,k.miura,c.long,a.saul,l.h.miller,a.p.durbin,phase1trialofmalariatransmissionblockingvaccinecandidatespfs25andpvs25formulatedwithmontanideisa51.plosone3,e2636(2008).[0438]其他参考资料[0439]ahmed,s.f.,quadeer,a.a.,&mckay,m.r.(2020).preliminaryidentificationofpotentialvaccinetargetsforthecovid-19coronavirus(sars-cov-2)basedonsars-covimmunologicalstudies.viruses,12(3).doi:10.3390/v12030254[0440]bagarazzi,m.l.,yan,j.,morrow,m.p.,shen,x.,parker,r.l.,lee,j.c.,giffear,m.,pankhong,p.,khan,a.s.,broderick,k.e.,knott,c.,lin,f.,boyer,j.d.,draghia-akli,r.,white,c.j.,kim,j.j.,weiner,d.b.&sardesai,n.y.,2012.immunotherapyagainsthpv16/18generatespotentth1andcytotoxiccellularimmuneresponses.scitranslmed,4,155ra138.[0441]bioley,g.,guillaume,p.,luescher,i.,yeh,a.,dupont,b.,bhardwaj,n.,mears,g.,old,l.j.,valmori,d.&ayyoub,m.,2009.hlaclassi-associatedimmunodominanceaffectsctlresponsivenesstoanesorecombinantproteintumorantigenvaccine.clincancerres,15,299-306.[0442]essekuf,adeyeyemc.bacteriaandph-sensitivepolysaccharide-polymerfilmsforcolontargeteddelivery.critrevtherdrugcarriersyst2011;28:395–445.[0443]gennaro,2000,remington:thescienceandpracticeofpharmacy,20thedition,isbn:0683306472[0444]grothey,a.,tabernero,j.,arnold,d.,degramont,a.,ducreux,m.p.,o'dwyer,p.j.,vancutsem,e.,bosanac,i.,srock,s.,mancao,c.,gilberg,f.,winter,j.&schmoll,h.-j.,2018.lba19fluoropyrimidine(fp) bevacizumab(bev) atezolizumabvsfp/bevinbrafwtmetastaticcolorectalcancer(mcrc):findingsfromcohort2ofmodul–amulticentre,randomizedtrialofbiomarker-drivenmaintenancetreatmentfollowingfirst-lineinductiontherapy.annalsofoncology,29.[0445]gudmundsdotter,l.,wahren,b.,haller,b.k.,boberg,a.,edback,u.,bernasconi,d.,butto,s.,gaines,h.,imami,n.,gotch,f.,lori,f.,lisziewicz,j.,sandstrom,e.&hejdeman,b.,2011.amplifiedantigen-specificimmuneresponsesinhiv-1infectedindividualsinadoubleblinddnaimmunizationandtherapyinterruptiontrial.vaccine,29,5558-66.[0446]kakimi,k.,isobe,m.,uenaka,a.,wada,h.,sato,e.,doki,y.,nakajima,j.,seto,y.,yamatsuji,t.,naomoto,y.,shiraishi,k.,takigawa,n.,kiura,k.,tsuji,k.,iwatsuki,k.,oka,m.,pan,l.,hoffman,e.w.,old,l.j.&nakayama,e.,2011.aphaseistudyofvaccinationwithny-eso-1fpeptidemixedwithpicibanilok-432andmontanideisa-51inpatientswithcancersexpressingtheny-eso-1antigen.intjcancer,129,2836-46.[0447]kisslersm,tedijantoc,goldsteine,gradyh,lipsitchm.thecopyrightholderforthispreprint.https://doi.org/10.1101/2020.03.04.20031112[0448]klebanoff,c.a.,acquavella,n.,yu,z.&restifo,n.p.,2011.therapeuticcancervaccines:arewethereyet?immunologicalreviews,239,27-44.antigen.jimmunother,37,84-92.[0457]vandenmooterg,weutsi,deriddert,blatonn.evaluationofinutecsp1asanewcarrierintheformulationofsoliddispersionsforpoorlysolubledrugs.intjpharm.2006jun19;316(1-2):1-6.[0458]yuan,j.,adamow,m.,ginsberg,b.a.,rasalan,t.s.,ritter,e.,gallardo,h.f.,xu,y.,pogoriler,e.,terzulli,s.l.,kuk,d.,panageas,k.s.,ritter,g.,sznol,m.,halaban,r.,jungbluth,a.a.,allison,j.p.,old,l.j.,wolchok,j.d.&gnjatic,s.,2011.integratedny-eso-1antibodyandcd8 t-cellresponsescorrelatewithclinicalbenefitinadvancedmelanomapatientstreatedwithipilimumab.procnatlacadsciusa,108,16723-8.当前第1页12当前第1页12
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。