用于血红蛋白a(hba)检测和基因分型的测定
优先权
1.本技术要求于2020年3月6日提交的美国临时申请号62/986,152的利益和优先权,其出于所有目的通过引用整体并入本文。
技术领域
2.本公开内容涉及血红蛋白a(hba)临床试验,特别涉及用于实施到临床试验中的hba测定和hba基因分型决策树矩阵的技术。
背景技术:
3.血红蛋白是红细胞中富含铁的蛋白质,其将氧气输送到全身的细胞中。该蛋白由两个α-珠蛋白和两个β-珠蛋白亚基组成,前者由hba1和hba2基因编码,后者由hbb基因编码。当这些基因发生改变(变化)或缺失时,发生地中海贫血。在alpha地中海贫血(α-地中海贫血)中受影响的血红蛋白蛋白亚基是α珠蛋白。从父母一方遗传缺陷型α-地中海贫血基因但从另一方遗传正常α-地中海贫血基因的人是α-地中海贫血的携带者。α-地中海贫血的携带者通常没有体征或症状。然而,他们可以将错误的基因传递给他们的孩子。患有中度至重度α-地中海贫血的人从父母双方遗传了多个错误的α-地中海贫血基因。这些都是以常染色体隐性模式遗传的。α-地中海贫血是世界上最常见的血红蛋白合成遗传性病症,在疟疾流行的热带和亚热带,突变等位基因频率在1%至98%之间。α-地中海贫血可发生于所有种族群体中,但在东南亚后裔中更常见。α-地中海贫血在特定群体中的高患病率可能是由于hba基因的1-2个缺陷拷贝提供的疟疾寄生虫(疟原虫的多个种)的感染保护作用。美国妇产科医师学会建议对非洲、东南亚、地中海、中东或西印度裔进行血红蛋白病筛查,尽管血红蛋白病在混合人群中越来越普遍。
4.α珠蛋白由16号染色体上的两个基因(α-珠蛋白基因hba1和hba2)编码。每个人需要四个功能性hba基因(每个亲本两个)来产生足够的α-珠蛋白,使身体的血红蛋白正常工作。如果这些基因中的一个或多个基因有缺陷,则会发生不同形式的α-地中海贫血。如果一个基因有缺陷,那么该人就是α-地中海贫血特征的“沉默”携带者,并且通常没有体征或症状。如果两个基因有缺陷,那么该人就有α-地中海贫血的特征(也称为轻度α地中海贫血),并且可能有轻度贫血。如果三个基因有缺陷,那么该人患有血红蛋白h病。这可能导致中度至重度贫血。如果所有四个基因都缺失,那么该人患有严重的α-地中海贫血(也称为血红蛋白bart’s或胎儿水肿)。这是最严重的α-地中海贫血类型。患有这种病症的胎儿通常会在子宫内死亡,或者婴儿出生后不久就会死亡,因为婴儿无法制造正常的血红蛋白来将氧气携带到全身。
5.超过90%的α-地中海贫血是由于16号染色体上两个或更多个拷贝的α-珠蛋白基因(hba1和hba2)的缺失所致。hba1和hba2基因位于16号染色体上的约30kbα-珠蛋白基因簇内,其中从端粒到着丝粒以此顺序包括以下α珠蛋白基因和(假基因):hbz、(hbzp1).hbm、(hbap1)、hba2、hba1、hbq1(参见例如图1)。hba1和hba2的编码序列与位于内含子和5'-和
3'-非翻译区中的分歧序列相同。此外,位于启动子区中hbz基因上游40kb处的hs-40主要超敏位点的缺失影响hba1和hba2的rna表达,从而在杂合子中引起α-地中海贫血特征。hba2中终止密码子的第一碱基处的hb constant spring点突变影响hba2的rna表达,并引起比hba2缺失等位基因更严重的表型。最后,基因转换在hba1和hba2之间由于其紧密靠近和高度同源性而常见,但无临床意义。鉴于这些因素,可能需要开发能够对α-珠蛋白区域的多个不同位点进行基因分型的用于hba检测的测定,以容易地筛查α-地中海贫血。
技术实现要素:
6.在各种实施方案中,提供了一种计算机实现的方法,其包括:从对多个样品进行的血红蛋白a(hba)测定中获得原始数据,其中所述hba测定使用能够检测所述多个样品中的每个样品的α-珠蛋白基因簇区域中的拷贝数丢失或增加的多个探针来进行,所述原始数据包括对于多个样品中的每个样品通过毛细管电泳解析的多个探针的hba拷贝数数据;从所述多个样品中选择参考样品;基于来自所述hba测定的原始数据和所述参考样品计算所述多个样品中的每个样品的第一组探针比;基于所述第一组探针比鉴定待组合为用于所述多个样品的合成参考样品的预定数量的参考样品;基于所述预定数量的参考样品生成所述合成参考样品;基于来自所述hba测定的原始数据和所述合成参考样品计算所述多个样品中的每个样品的第二组探针比;将每个样品的第二组探针比迭代地输入决策树矩阵;通过所述决策树矩阵,基于每个样品的第二组探针比和与所述多个探针中的每个探针相关联的样品探针/参考探针比的拷贝数调用阈值来确定每个样品的hba基因型;和提供每个样品的hba基因型。
7.在一些实施方案中,其中计算第一组探针比包括:(i)将所述多个样品中的每个样品中的对照探针峰高或信号与所述参考样品中的相应的对照探针峰高或信号进行比较,(ii)将每个样品中的对照探针峰高或信号与参考样品中的相应的对照探针峰高或信号之间的信号的变异性计算为对照探针标准偏差,(iii)当任何变异性度量大于预定阈值时,确定所述多个样品中的样品失败,(iv)当变异性度量中没有一个大于预定阈值时,确定所述多个样品中的样品不失败,和(v)对于不失败的每个样品,将样品中的测试探针峰高或信号与参考样品中的相应的测试探针峰高进行比较,并计算样品中的测试探针峰高或信号与参考样品中相应的测试探针峰高或信号之间的探针比。
8.在一些实施方案中,计算第二组探针比包括:(i)将所述多个样品中的每个样品中的对照探针峰高或信号与所述合成参考样品中的相应的对照探针峰高或信号进行比较,(ii)将每个样品中的对照探针峰高或信号与所述合成参考样品中的相应的对照探针峰高或信号之间的信号的变异性计算为对照探针标准偏差,(iii)当任何变异性度量大于预定阈值时,确定所述多个样品中的样品失败,(iv)当变异性度量中没有一个大于预定阈值时,确定所述多个样品中的样品不失败,和(v)对于不失败的每个样品,将样品中的测试探针峰高或信号与合成参考样品中的相应的测试探针峰高进行比较,并计算样品中的测试探针峰高或信号与合成参考样品中相应的测试探针峰高或信号之间的探针比。
9.在一些实施方案中,确定每个样品的hba基因型包括:(i)基于每个样品的第二组探针比和与所述多个探针中的每个探针相关联的样品探针/参考探针比的拷贝数调用阈值来确定每个样品的异常探针比模式,和(ii)基于异常探针比模式鉴定每个样品的hba基因
型。
10.在一些实施方案中,确定每个样品的异常探针比模式和鉴定hba基因型包括:基于异常探针比模式将每个样品分类为正常、具有拷贝数变异(cnv)或多态;以及基于异常探针比模式,将被分类为具有cnv的任何样品亚分类为大的靶向缺失、重复或“其他”。
11.在一些实施方案中,确定每个样品的异常探针比模式和鉴定hba基因型还包括:将被分类为具有大的靶向缺失的任何样品亚分类为大杂合缺失或纯合缺失;基于异常探针比模式,将被分类为大杂合缺失或纯合缺失的任何样品针对以下缺失中的一种或多种进行亚分类:sea,fil/thai、med或α
20.5
;和基于异常探针比模式,将被分类为具有大杂合缺失的任何样品针对以下中的一种或多种进行亚分类:α
3.7
缺失、α
4.2
缺失和/或α
3.7
重复。
12.在一些实施方案中,确定每个样品的异常探针比模式和鉴定hba基因型还包括:基于异常探针比模式,将被分类为“其他”的任何样品进行亚分类;将被分类为“其他”的任何样品亚分类为具有α
3.7
缺失、α
4.2
缺失和/或α
3.7
重复。
13.在一些实施方案中,所述方法还包括触发对具有不正常或不需要手动审查的hba基因型的多个样品中的每个样品的确认测试的性能。
14.在一些实施方案中,提供了一种系统,其包括一个或多个数据处理器和包含指令的非暂时性计算机可读存储介质,当在一个或多个数据处理器上执行时,所述指令导致一个或多个数据处理器执行本文公开的一种或多种方法或过程的一部分或全部。
15.在一些实施方案中,提供了一种计算机程序产品,其有形地体现在非暂时性机器可读存储介质中,并且包括被配置成导致一个或多个数据处理器执行本文公开的一种或多种方法的一部分或全部的指令。
16.本公开内容的一些实施方案包括包含一个或多个数据处理器的系统。在一些实施方案中,系统包括包含指令的非暂时性计算机可读存储介质,当在一个或多个数据处理器上执行时,所述指令导致一个或多个数据处理器执行本文公开的一种或多种方法的一部分或全部和/或一个或多个过程的一部分或全部。本公开内容的一些实施方案包括有形地体现在非暂时性机器可读存储介质中的计算机程序产品,其包括被配置成导致一个或多个数据处理器执行本文公开的一种或多种方法的一部分或全部和/或一个或多个过程的一部分或全部的指令。
17.所采用的术语和表达用作描述性而非限制性术语,并且在使用这些术语和表达时无意排除所示和描述的特征或其部分的任何等同物,但认可的是,在所要求保护的本发明的范围内可以进行各种修改。因此,应当理解,尽管本发明已经通过实施方案和任选特征具体地公开,但本领域技术人员可以求助于本文所公开的概念的修改和变型,并且这种修改和变型被认为在由所附权利要求限定的本发明的范围内。附图简述
18.鉴于以下非限制性附图,将更好地理解本发明,其中:
19.图1示出了根据各种实施方案的16号染色体的基因数据;
20.图2示出了根据各种实施方案的hba测定平台的框图;
21.图3a示出了根据各种实施方案的mlpa测定化学的概述;
22.图3b示出了根据各种实施方案的34个α-珠蛋白基因簇区域探针和常见的hba缺失的位置;
23.图4示出了根据各种实施方案的使用hba测定平台和基因分型技术的hba测定和hba基因分型的示例性流程;
24.图5示出了根据各种实施方案的使用hba测定平台和基因分型技术的hba基因分型的示例性流程;
25.图6示出了根据各种实施方案的示例性计算设备;和
26.图7示出了根据各种实施方案的α
3.7
杂合缺失(样品b09)的相对于参考的倍数变化的挨个探针图。
27.在所附图中,相似的组分和/或特征可以具有相同的参考标签。此外,同一类型的各种组分可通过在参考标签后面接上划线和区分相似组分的第二标签来区分。如果在说明书中仅使用了第一参考标签,则该描述适用于具有相同第一参考标签的任何一个相似组分,而与第二参考标签无关。发明详述
28.随后的描述仅提供优选的示例性实施方案,并且不旨在限制本公开内容的范围、适用性或配置。相反,优选示例性实施方案的后续描述将为本领域技术人员提供用于实施各种实施方案的启用性描述。应当理解,可以在不脱离所附权利要求中所述的精神和范围的情况下对元件的功能和布置进行各种变化。
29.具体细节在以下描述中给出,以提供对实施方案的透彻理解。然而,应当理解,可以在没有这些具体细节的情况下实施实施方案。例如,回路、系统、网络、过程和其他组件可以以框图形式示出为组件,以避免在不必要的细节中掩盖实施方案。在其他情况下,可以在没有不必要的细节的情况下示出众所周知的回路、过程、算法、结构和技术,以避免掩盖实施方案。
30.此外,值得注意的是,单个实施方案可以被描述为一个过程,其被描绘为流程图、作业图、数据流程图、结构图或框图。虽然流程图或作业图可以将操作描述为顺序过程,但许多操作可以并行或同时进行。此外,可以重新安排操作顺序。过程在其操作完成时终止,但可能有不包括在图中的其他步骤。过程可以对应于方法、函数、程序、次例程、子程序等。当过程对应于函数时,它的终止可能对应于函数返回到调用函数或主函数。i.引言
31.α-珠蛋白区域的dna分析可通过使用多重连接依赖性探针扩增(mlpa)靶向多个不同的位点进行。该方法由mrc holland(产品描述salsamlpaprobemix p140-c1 hba)开发,检测涉及该位点的基因组缺失和重复,包括7种最常见的α-地中海贫血缺失类型(α
3.7
、α
4.2
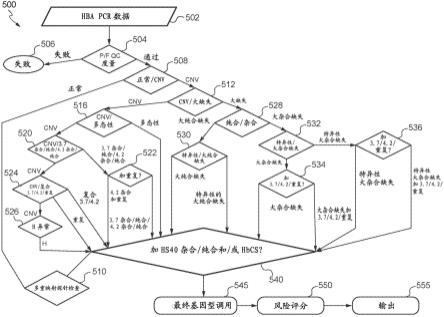
、sea、med、thai、fil和α
20.5
),以及constant spring点突变和hbax hs-40启动子缺失。由于α-珠蛋白区域的复杂性和通过mlpa测定分析的位点数量,通常手动审查hba mlpa拷贝数数据以确定基因型。确认测试可通过多重pcr和凝胶电泳或sanger测序进行。与传统hba mlpa测定和手动基因分型相关的一个问题是,它们从α-珠蛋白区域生成大量mlpa拷贝数数据,并使用复杂的表格指导mlpa拷贝数数据的手动评价,这需要大量的培训经验和时间。此外,传统的hba mlpa测定通常证明手动基因分型期间采取的评价步骤的错误率较高。该错误率会产生扩散效应,并不期望地影响筛选测试的质量(例如,假阳性或过度诊断),并可能错误地触发昂贵的确认测试。
32.为了解决这些局限性和问题,本文描述的各种实施方案涉及hba测定和基因分型
技术,其能够减少决策制定中的模糊性并提供对每个可能的决策的后果的综合分析,同时在有限的处理、存储器和功率资源下实现最小的错误率。在某些情况下,开发了这样的过程,其包括基于决策树是否得出样品包括指示α-地中海贫血的hba基因型的结论来对是否应对样品进行确认测试进行门控。例如,本公开内容的各种实施方案包括系统,其包含一个或多个处理器和耦合到一个或多个处理器的存储器。存储器编码有一组指令,所述指令被配置为执行过程,所述过程包括:从对多个样品进行的hba测定中获得原始数据,其中所述hba测定使用能够检测所述多个样品中的每个样品的α-珠蛋白基因簇区域中的拷贝数丢失或增加的多个探针来进行,所述原始数据包括对于多个样品中的每个样品通过毛细管电泳解析的多个探针的hba拷贝数数据(例如,mlpa拷贝数数据);从所述多个样品中选择参考样品;基于来自所述hba测定的原始数据和所述参考样品计算所述多个样品中的每个样品的第一组探针比;基于所述第一组探针比鉴定待组合为用于所述多个样品的合成参考样品的预定数量的参考样品;基于预定数量的参考样品生成合成参考样品;基于来自所述hba测定的原始数据和所述合成参考样品计算所述多个样品中的每个样品的第二组探针比;将每个样品的第二组探针比迭代地输入决策树矩阵中;通过所述决策树矩阵,基于每个样品的第二组探针比和与所述多个探针中的每个探针相关联的样品探针/参考探针比的拷贝数调用阈值来确定每个样品的hba基因型;和提供每个样品的hba基因型。在一些情况下,该技术还包括触发对具有不正常或不需要手动审查的hba基因型的多个样品中的每个样品的确认测试的性能。
33.有利地,这些方法提供了能够在有限的处理、存储器和功率资源下在诸如测序系统的环境中实现最小错误率的hba测定和基因分型技术。例如,决策树可以在低功率预算(例如,简单的条件树)下实现,并且提供多维搜索空间的强大的非线性分类能力。此外,非线性分类可用于对是否应对样品进行确认测试进行门控,从而节省错误执行确认测试的成本,并增加总体筛选hba测定的稳健性。
34.如本文所用,术语“基本上”、“大致”和“约”被定义为主要但不一定完全是本领域普通技术人员所理解的指定内容(并且包括完全的所指定的内容)。在任何公开的实施方案中,术语“基本上”、“大致”或“约”可被在指定内容的“[一定百分比]内”替换,其中所述百分比包括0.1、1、5和10%。如本文所用,当动作是“基于”某物时,这意味着该动作至少部分地基于某物的至少一部分。
[0035]
应当理解,与本文具体描述的mlpa拷贝数数据相比,本文公开的hba基因分型技术可应用于评估其他类型的测序原始数据。还应理解,预期使用其他测定方法和类型的聚合酶链反应(pcr)或多重(pcr)来鉴定给定染色体或基因区域(例如,16号染色体或α-珠蛋白区域)内的一个或多个位点。例如,替代地或另外地,反向斑点印迹杂交(rdb)、southern印迹(sb)或gap-pcr可用于鉴定16号染色体或α-珠蛋白区域内的一个或多个位点。ii.hba测定技术
[0036]
本文描述的一个或多个实施方案可以使用编程模块、引擎或组件来实现。编程模块、引擎或组件可以包括能够进行一个或多个所述任务或功能的程序、子程序、程序的一部分、或软件组件或硬件组件。如本文所用,模块或组件可以独立于其他模块或组件存在于硬件组件上。可替代地,模块或组件可以是其他模块、程序或机器的共享元件或过程。图2示出了用于检测位于16号染色体上的α-珠蛋白区域中的缺失和/或重复作为α-地中海贫血的潜
在原因、筛查工具和/或临床诊断的hba测定平台100的框图,并且示出了可由一个或多个处理器执行的模块、引擎或组件(例如,程序、代码、或指令),所述处理器可用于实现根据各种实施方案的分析仪系统205的各种子系统。模块、引擎或组件可以存储在非暂时性计算机介质上。根据需要,模块、引擎或组件中的一个或多个可以被加载到系统存储器(例如,ram)中,并且由分析仪系统205的一个或多个处理器执行。在图2所示的示例中,示出了实现基因分析仪子系统210和hba基因分型子系统215的模块、引擎或组件。
[0037]
图2还示出了包括实验室的湿实验室子系统220,在其中测试和分析需要水、直接通风和专用管道公用设施的化学品、药物或其他材料或生物物质。hba测定平台200包括在湿实验室子系统220内在框230处获得一个或多个样品225。在一些情况下,样品225包括从人细胞系中提取的核酸。在一些情况下,样品225包括从男性或女性患者获得的核酸。在一些情况下,样品225是从男性或女性患者获得的全血、羊水、羊水细胞培养物、绒毛膜绒毛取样或绒毛膜绒毛样品细胞培养物中提取的核酸。在某些情况下,样品或样品225在16号染色体的α-珠蛋白基因簇区域内具有一个或多个基因组缺失、点突变和/或重复。
[0038]
在湿实验室子系统220内的框235处,进行hba测定,包括通过使用多重测定方法250(例如,多重pcr方法)靶向多个不同位点来进行α-珠蛋白基因簇区域(hba1/hba2、omim 141800/141850、16pter-16p13.3)的dna分析。多重测定方法250检测涉及该位点的基因组缺失和重复,包括7种最常见的α-地中海贫血缺失类型(α
3.7
、α
4.2
、sea、med、thai、fil和α
20.5
),以及constant spring点突变和hbax hs-40启动子缺失。在某些情况下,hba测定用于:(i)筛查α
3.7
、α
4.2
、sea、med、thai、fil、α
20.5
和hs-40缺失的携带者,(ii)通过筛查携带者的伴侣的任何hba突变来鉴定高危夫妇,和/或(iii)针对hba缺失的靶向家族性检测和产前检查,其中父母一方或双方已被确定携带如本文所述的致病性突变。确认测试可通过多重测定方法和凝胶电泳或sanger测序进行。
[0039]
在各种实施方案中,多重测定方法250是mpla技术(例如,由mrc holland-product description salsamlpaprobemix p140-c1 hba开发的mpla方法),其是一种半定量、连接依赖性多重pcr方法,其可以通过测量样品或样品225和合成参考样品240之间的相对信号强度来确定目标区域中的拷贝数差异。hba测定使用mlpa寡核苷酸245的库,使得当在靶序列处紧邻彼此杂交的两个寡核苷酸连接在一起时形成探针。如图3a所示,每个连接的探针300可由两个寡核苷酸305(例如,来自寡核苷酸245的库)组成,其中一端包括对靶序列特异性的杂交序列310,另一端包括用于多重复用的pcr引物315(例如,标记的通用pcr引物)序列。在某些情况下,不同长度的填充序列320可用于允许不同的探针尺寸。在样品和参考变性后,将寡核苷酸305与靶序列杂交预定量的时间(例如,16-20小时)。杂交后,将与紧邻位点杂交的两个寡核苷酸305连接在一起,以形成独特大小的连接探针300。该反应具有高度特异性,并且在两个寡核苷酸305之间没有大于缺口的间隙时发生。使用pcr引物315对连接的探针300进行pcr扩增。由于snp或缺乏靶序列而与其靶序列结合不良或根本不结合的连接探针300将不会扩增。
[0040]
在一些实施方案中,hba测定包括使用45个靶特异性探针(扩增大小131-481个碱基),这些探针包括33个探针用于检测α-珠蛋白基因簇区域和周围序列中的缺失和重复,以及1个探针用于检测hb constant spring点突变的存在。在33个探针中,有5个对hba1或hba2具有特异性的单个探针,3个靶向两个基因中存在的序列的探针;即,在正常样品中,分
别通过映射到独特序列的探针或映射到hba1和hba2中存在的序列的探针来检测2或4个拷贝。由于这两个基因的高度同源性和接近性,也有5个探针对与hba1和hba2基因的相同位置中的不同序列结合,使得一对中的一个探针对hba1具有特异性,另一个探针对hba2具有特异性。这些探针对用于检测无临床意义但可能混淆基因分型调用的基因转换或多态性。当存在多态性时,该对中一个探针的倍数变化将指示重复,而另一个探针指示缺失。45个靶特异性探针还包括11个对照探针,这些对照探针靶向16号染色体以外的其他常染色体上的参考序列,并用于33个探针的标准化,以检测hba区域的拷贝数变化。在某些情况下,可包括质量控制(q-片段和d-片段)和性别特异性片段(其是单寡核苷酸),以确保mlpa化学如预期地工作。检测α-珠蛋白基因簇区域和周围序列中变异的探针的位置以及本测试中靶向的缺失的位置示意性地示出在图3b中。所显示的是配对的探针(p)(该探针可检测由hba1和hba2之间的基因转换引起的多态性)、映射到hba1和hba2的探针(粗体箭头)以及hb constant spring(cs)探针的位置。fil和thai缺失通过相同的探针检测,并且需要多重pcr以进行区分。在某些情况下,α
3.7
、α
4.2
和med的缺失边界可能不同,并且其检测可能涉及不同的探针。
[0041]
在任选的框255处,可以对pcr产物(例如,扩增的连接探针)进行pcr后清洁。在一些情况下,pcr后清洁包括将pcr产物与磁珠混合、用诸如70%乙醇的洗涤液洗涤、风干和洗脱纯化的pcr产物260以提高信噪比。在扩增和pcr后清洁后,纯化的pcr产物260可上样到分析仪系统205(例如,基于荧光的分离仪器系统)上用于下游处理。
[0042]
在框265处,使用毛细管电泳解析多重测定产物或任选纯化pcr产物260,使用标记物(例如荧光染料)检测,并输出为包含hba拷贝数数据的原始数据270。在一些情况下,原始数据270可以生成、收集和存储在存储器设备中的分析仪系统205上。在框275处,分析仪子系统210从分析仪系统205获得每个样品的原始数据270,确定扩增子的大小,并自动选择峰高比变异性最小且未检测到缺失/重复的标准化样品作为参考样品。分析仪子系统210将每个测试探针(正常探针)的原始峰信号标准化至对照探针的原始峰信号,以最小化较小片段的任何优先扩增。例如,可以将测试样品中的对照探针峰高或信号与参考样品中的相应对照探针峰高进行比较。两者之间信号比的变异性计算为对照探针标准偏差,并且大于预定阈值(例如,0.125阈值)的对照探针标准偏差的值可能导致样品失败。分析仪子系统210还可以检查质量控制d-片段和q-片段,以确保pcr反应符合预定的质量标准,并且超出预定质量标准的任何东西可能导致样品失败。
[0043]
然后将未失败样品的测试探针的标准化探针峰信号与参考样品中相应测试探针的峰信号进行比较。然后,通过分析仪子系统210计算样品和参考探针信号之间的相对探针比或倍数变化,并且样品探针/参考探针比的超出正常范围(例如,拷贝数调用阈值)的任何变化可以被分析仪子系统210鉴定为缺失或重复。因此,相对于参考样品,无缺失或重复的样品的相对探针比或倍数变化为约1,超过拷贝数调用阈值的探针比的降低或增加可分别解释为靶序列的缺失或重复。分析仪子系统210可以生成和导出包括每个样品225的相对探针比或倍数变化的初始结果文件。
[0044]
在框280处,通过hba基因分型子系统215基于缺失和/或重复分析在分析仪子系统210上计算的相对探针比数据的异常探针比模式,以鉴定被靶向用于测试的基因型。更具体地,hba基因分型子系统215被配置成用于以下几个目的:a)检查数据质量;b)使用阴性选择器工具选择用于创建合成参考的样品;和c)通过用于基因型调用的决策树矩阵依次解析由
分析仪子系统210计算的相对探针比数据。为了最大限度地增加可在一个批次中运行的测试的数量,并且由于约85-90%的用于携带者筛选的样品对于hba缺失或重复将是阴性的,开发了hba基因分型子系统215的阴性选择器工具,以提供技术优势,以鉴定具有最佳质量度量且对于任何拷贝数变化(无缺失/重复)为阴性的3至6个最佳质量样品以用于分析仪子系统210中的对照样品选择应用。然后由分析仪子系统210中的对照样品选择应用通信和/或选择3至6个最佳质量样品,并且分析仪子系统210将这3至6个最佳质量样品组合以创建合成参考样品。然后,分析仪子系统210使用该合成参考样品重新计算每个样品225的相对探针比或倍数变化。该重新计算考虑了平板的所有部分的样品,因此,除了最大限度地增加每个平板可运行的样品数量外,合成参考样品标准化了整个平板上的倍数变化比,使样品失败的可能性降低。分析仪子系统210可以生成和导出包括每个样品225的新计算的相对探针比或倍数变化的新结果文件。hba基因分型子系统215使用这些新计算的相对探针比或倍数变化和决策树矩阵对每个样品225中的hba基因型进行分类和/或鉴定。
[0045]
在各种实施方案中,在检查新计算的相对探针比或数据质量的倍数变化之后,hba基因分型子系统215使用拷贝数调用阈值来鉴定涉及多个异常基因型的探针的拷贝数丢失或增加的区域,并将每个样品分类为正常、多态性或拷贝数变异(cnv)类别。然后可以将cnv样品分类为大的靶向缺失、重复或其他。确定大的靶向缺失组中的样品的合子性(zygosity),并进一步分类为sea、fil/thai、med或α-20.5。进一步检查杂合的样品的α
3.7
和α
4.2
缺失。也可以检查“其他”类别中的那些的α
3.7
和α
4.2
缺失,如果检测到其中之一,则检查合子性。此时,所有样品应分类为靶向的基因型或“其他”。然后hba基因分型子系统215将探针的组合与具有每种可能情景的表进行比较,以针对靶向缺失和重复鉴定复合杂合子。还可通过hba基因分型子系统215分析所有样品的hs-40缺失和hb constant spring点突变,并且任何阳性结果均串联到先前确定的基因型上。仍被分类为“其他”的样品被标记以供医疗保健专业人员(例如临床管理者)审查,从而确保hba基因分型子系统215不会错过任何具有临床意义的结果,无论是否具有靶向性。
[0046]
每个样品的hba基因型和每个样品的任选风险结果由分析仪系统205输出作为最终结果285。在一些情况下,分析仪子系统205和hba基因分型子系统215使用的所有阈值和qc参数以及决策树矩阵均维持在一个或多个单独的配置文件中,并且可用于任何数量的hba pcr测定。iii.hba基因分型技术
[0047]
图4示出了使用hba测定平台和基因分型技术(例如,参照图1描述的hba测定平台100)进行hba基因分型的过程400。过程400从框405开始,其中从对多个样品进行的hba测定中获得原始数据。在一些情况下,使用能够检测多个样品中的每个样品的α-珠蛋白基因簇区域中的拷贝数丢失或增加(例如缺失、重复和hb constant spring点突变)的多个探针(例如,连接的探针)进行hba测定。多个探针可包括一个或多个对照探针和一个或多个测试探针。在一些情况下,原始数据包括对于多个样品中的每个样品通过毛细管电泳解析的多个探针的hba拷贝数数据。在框410处,从所述多个样品中选择参考样品。可以基于所述多个探针的原始数据选择参考样品以获得在所述多个探针中具有最小变异性且没有拷贝数变化(例如,没有缺失和/或重复)的参考样品。
[0048]
在一些情况下,在选择参考探针之前,检查原始数据的质量。原始数据的质量检查
可以包括使用表1中列出的一个或多个参数确认原始数据的质量。在一些情况下,进行以下质量检查中的一个或多个,并且基于一个或多个质量检查的结果评估原始数据的质量:(i)总探针数等于总探针的预定数量,例如45,(ii)测试(正常)探针数等于测试探针的预定数量,例如33,(iii)对照探针数等于对照探针的预定数量,例如11,(iv)峰信号大于或等于预定峰高阈值(例如,标准化后大于或等于200rfu),(v)q-片段(当dna量过低或连接失败时将优先扩增的单寡核苷酸(未连接的探针))代表≤预定阈值,例如,92nt基准片段的信号的33%,和(vi)d-片段(当变性反应不完全时将优先扩增的单寡核苷酸(未连接的探针))代表≤预定阈值,例如92nt基准片段的信号的50%。当原始数据质量不符合要求时,则过程可停止,并任选地要求再次进行hba测定,以获得新的原始数据。当原始数据质量符合要求时,过程可继续进行。表1:本研究中使用的质量度量和阈值
[0049]
在框415处,针对多个样品中的每个样品计算第一组探针比(在一些情况下,未针对参考样品计算第一组探针比)。在一些情况下,计算第一组探针比包括:(i)将每个样品中的对照探针峰高或信号与所选参考样品中的相应对照探针峰高进行比较,(ii)将每个样品中的对照探针峰高或信号与参考样品的对照探针之间的信号的变异性计算为对照探针标准偏差,(iii)当对照探针标准偏差大于或等于预定阈值(例如,》0.125阈值)时,确定多个样品中的样品失败,(iv)当对照探针标准偏差小于预定阈值(例如,《0.125阈值)时,确定多个样品中的样品不失败,和(v)对于不失败的每个样品,将样品中的测试探针峰高或信号与所选参考样品中的相应的测试探针峰高进行比较,并计算样品中的测试探针峰高或信号与所选参考样品中相应的测试探针峰高之间的探针比。
[0050]
在框420处,生成并输出初始结果文件,该初始结果文件包括为每个样品计算的第一组探针比。第一组探针比包括步骤415中计算的未失败的每个样品(和任选地不是参考样品)的相对探针比或倍数变化。在框425处,访问初始结果文件,并且分析样品的第一组探针比以鉴定预定数量(例如,在三个和六个之间)的参考样品,以组合为用于多个样品的合成参考样品。在某些情况下,基于质量度量和拷贝数变化鉴定参考样品。例如,所鉴定的参考样品应对于任何拷贝数变化是阴性的(例如,基于拷贝数调用阈值,没有超出正常范围的探针,因此没有缺失或重复),通过所有探针数度量,并且在样品的平板上具有最低的对照探针标准偏差度量。可以生成包括所鉴定的参考样品的列表,并且生成并输出包括所鉴定的样品的列表的合成参考样品文件。在框430处,将合成参考样品文件访问或上传至计算系统,并且基于列表中鉴定的参考样品生成合成参考样品。在一些情况下,合成参考样品被创建为列表中每个所鉴定的样品的峰高或信号的功能性串联。例如,对于列表中的每个鉴定的样品,合成参考样品可以是峰高或信号的平均值、中位数或模式。
[0051]
在框435处,针对所述多个样品中的每个样品计算第二组探针比。在一些情况下,计算第二组探针比包括:(i)将每个样品中的对照探针峰高或信号与合成参考样品中的相应对照探针峰高进行比较,(ii)将每个样品中的对照探针峰高或信号与合成参考样品的对照探针之间的信号的变异性计算为对照探针标准偏差,(iii)当任何变异性度量大于或等于预定阈值(例如,≥0.125阈值)时,确定所述多个样品中的样品失败,(iv)当变异性度量中没有一个大于或等于预定阈值(例如,≥0.125阈值)时,确定所述多个样品中的样品不失败,和(v)对于每个不失败的样品,将样品中的测试探针峰高或信号与合成参考样品中的相应的测试探针峰高进行比较,并计算样品中的测试探针峰高或信号与合成参考样品中相应的测试探针峰高之间的探针比。
[0052]
在框440处,生成并输出新的结果文件,其包括每个样品的新的相对探针比数据。
新的相对探针比数据包括步骤435中计算的未失败的每个样品的相对探针比或倍数变化。在框445处,访问新的结果文件,并将每个样品的新的相对探针比数据迭代输入决策树矩阵,以:(i)基于缺失和/或重复确定每个样品的异常探针比模式,和(ii)基于异常探针比模式确定被靶向用于测试的基因型。异常探针比模式的确定和被靶向用于测试的基因型的鉴定可以包括基于多个探针的新的相对探针比数据和与每个探针相关联的样品探针/参考探针比的正常范围或拷贝数调用阈值通过决策树矩阵鉴定拷贝数丢失或增加(例如,缺失和/或重复)的一个或多个区域。例如,超出正常范围(例如,拷贝数调用阈值)的样品探针/参考探针比的任何变化被鉴定为缺失或重复,并且可以用于鉴定拷贝数丢失或增加的一个或多个区域。异常探针比模式的确定还可包括基于所鉴定的拷贝数丢失或增加的一个或多个区域将样品分类为正常、具有拷贝数变异(cnv)或多态。例如,如果样品没有超出正常范围的探针,则可以将样品分类为正常。如果样品有一个或多个探针超出正常范围,则可以将样品分类为cnv。对于有一个或多个探针超出正常范围的样品,如果所有超出正常范围的探针均不在指示缺失的探针子集(例如,被鉴定为对调用cnv很重要的缺失探针的子集)内,则该样品可被分类为多态性。
[0053]
异常探针比模式的确定和被靶向用于测试的基因型的鉴定还可以包括基于所鉴定的拷贝数丢失或增加的一个或多个区域将被分类为具有cnv的样品亚分类为大的靶向缺失、重复或“其他”。例如,如果被分类为具有cnv的样品具有指示超出正常范围的大的靶向缺失的一个或多个探针,则该样品可以进一步分类为具有大的靶向缺失。如果被分类为具有cnv的样品具有指示超出正常范围的重复的一个或多个探针,则该样品可以进一步分类为具有重复。如果被分类为具有cnv的样品没有指示超出正常范围的大的靶向缺失或重复的一个或多个探针,则该样品可以进一步分类为“其他”。
[0054]
异常探针比模式的确定和被靶向用于测试的基因型的鉴定还可以包括基于指示合子性的一个或多个探针来确定cnv组中样品的缺失状态和合子性和重复状态(例如,纯合性或杂合缺失),并且基于指示超出正常范围的缺失α
3.7
和α
4.2
的一个或多个探针进一步将样品分类为以下缺失中的一个,或重复:α
3.7
和α
4.2
。大缺失定义为包含hba1和hba2基因两者的缺失。可以基于一个或多个指示杂合性的探针确定大的靶向缺失组中的样品的合子性(例如纯合或杂合的大缺失),并基于指示超出正常范围的缺失sea、fil/thai、med、α
20.5
、α
3.7a
/α
4.2c
的一个或多个探针进一步分类为以下缺失之一:sea、fil/thai、med或α
20.5
,或不确定的定相的α
3.7a
/α
4.2c
缺失。基于指示超出正常范围的缺失α
3.7
和α
4.2
的一个或多个探针,可进一步将被分类为杂合性大缺失的样品针对较小的α
3.7
和α
4.2
缺失进行分类。此时,样品应被分类为靶向基因型或“其他”。靶向基因型的该过程的结果、结果含义和可能的临床解读的示例见表2。表2:测试中靶向的突变的可能的mlpa结果及其含义
*注:mlpa技术无法检测到拷贝数中性倒位或易位。本表仅列出了测试的靶向突变的情况,并且不包括可能触发医疗保健提供者(例如临床管理者)审查的情况。het,杂合;hom,纯合;cs snv,constant spring单核苷酸变异
[0055]
异常探针比模式的确定和被靶向用于测试的基因型的鉴定可进一步包括将新的相对探针比数据与具有鉴定非靶基因型(例如用于靶向缺失和重复的复合杂合子)的各种可能情况的表进行比较。在表比较后仍分类为“其他”的样品可被标记用于医疗保健提供者(例如临床管理者)审查,从而确保基因分型技术不会错过任何具有临床意义的结果,无论
是否被靶向。此外,也可基于指示超出正常范围的hs-40缺失和hb constant spring点突变的一个或多个探针分析所有样品(分类为正常、cnv和多态性的样品)的hs-40缺失和hb constant spring点突变,并且任何阳性结果均可连接到先前确定的靶向或非靶向基因型上。
[0056]
任选地,在框450处,在框445中确定的每个样品的基因型可用于确定与样品相关联的受试者的风险评分。风险评分可鉴定:(i)受试者是α
3.7
、α
4.2
、sea、med、thai、fil、α
20.5
和hs-40缺失以及hb constant spring点突变的携带者的风险,(ii)夫妇被鉴定为α
3.7
、α
4.2
、sea、med、thai、fil、α
20.5
和hs-40缺失和hb constant spring点突变的携带者的风险,和/或(iii)胎儿遗传α
3.7
、α
4.2
、sea、med、thai、fil、α
20.5
和hs-40缺失以及hb constant spring点突变的风险。在框455处,可以输出在框445中确定的每个样品的基因型和在框450中确定的任选风险评分。每个样品的基因型和任选的风险评分的输出可以包括向最终用户提供输出和/或将输出记录在存储设备中(例如,在用户界面上显示输出和/或将输出存储在数据库的结果文件中)。在框460处,当在框445中确定的样品基因型表明α
3.7
、α
4.2
、sea、med、thai、fil、α
20.5
和hs-40缺失和hb constant spring点突变中的一个或多个被分配给样品时,可对样品进行确认测试。确认测试可通过多重测定方法和凝胶电泳或sanger测序进行。当在框445中样品的基因型未确定时,可对样品进行手动审查。在框465处,当在框445中确定的样品的基因型指示对样品分配正常分类时,停止样品的处理。因此,决策树矩阵的非线性分类可用于对是否应对样品进行确认测试进行门控,从而节省错误进行确认测试的成本,并增加总体筛选hba测定的稳健性。
[0057]
图5示出了决策树矩阵500,其示出了可实现以进行一个或多个样品的hba基因分型的基因分型技术。在框502处,访问新的结果文件,并且将包括多个样品中的每个样品的hba拷贝数数据的原始数据迭代输入决策树矩阵中,以:(i)基于缺失和/或重复确定每个样品的异常探针比模式,和(ii)基于异常探针比模式鉴定靶向用于测试的基因型。原始数据包括基于合成参考样品(如关于图4中的步骤425和430所讨论的)获得的每个样品的新的相对探针比数据(如关于图4中的步骤435、440和445所讨论的)。新的相对探针比数据包括用于分析多个样品中的每个样品的α-珠蛋白基因簇区域的多个探针中的每个探针的相对探针比或倍数变化。在一些情况下,新的相对探针比数据是从使用多个探针(例如,连接的探针)进行的hba测定中获得的,所述多个探针能够检测多个样品中的每个样品的α-珠蛋白基因簇区域中的缺失、重复和hb constant spring点突变,如关于图1和图4所讨论的。表3提供了可用于进行hba测定和基因分型的多个探针的示例性列表。多个探针包括一个或多个对照探针(例如,ctrl_5q31)和一个或多个测试探针(例如,hba_hba1)。表3:探针列表-共45个探针,包括11个对照探针和34个测试探针
[0058]
在框504处,可通过决策树矩阵进行质量检查,以确认原始数据对基因分型分析有效。原始数据的质量检查可包括使用表1中列出的一个或多个参数确认原始数据质量的决策树矩阵。在一些情况下,进行以下质量检查中的一个或多个,并且基于一个或多个质量检查的结果评估原始数据的质量:(i)总探针数等于总探针的预定数量,例如45,(ii)测试(正常)探针数等于测试探针的预定数量,例如33,(iii)对照探针数等于对照探针的预定数量,例如11,(iv)峰信号大于或等于预定峰高阈值(例如,标准化后大于或等于200rfu),(v)q-片段(当dna量太低或连接失败时优先扩增的单寡核苷酸(未连接的探针))代表≤预定阈值,例如,92nt基准片段的信号的33%,(vi)d-片段(当变性反应不完全时将优先扩增的单寡核苷酸(未连接的探针))代表≤预定阈值,例如92nt基准片段的信号的50%,和(vii)对照探针标准偏差中的任何偏差大于预定阈值(例如0.125阈值)。当原始数据质量不合格时,过程在框506处停止,并任选地发出要求以再次进行hba测定,以获得新的原始数据。当原始数据质量符合要求时,过程在框508处继续进行。
[0059]
在框508处,基于指示与cnv相关联的异常探针比模式的样品探针/参考探针比确定正常样品或具有cnv的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)来鉴定与cnv相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)表3中列出的所有探针的相对探针比数据,(ii)用于预测样品为正常的拷贝数调用阈值≥0.75且≤1.3,和(iii)用于预测样品具有cnv的拷贝数调用阈值《0.75或≥1.3。如果分析的结果是预测样品为正常,则将样品分类为正常。如果分析的结果是预测样品具有cnv,则将样品分类为具有cnv。当样品被分类为正常时,过程在框510处继续进行。当样品被分类为具有cnv时,过程在框512处继续进行。
[0060]
在框510处,对于具有“正常”或“多态性”分类的任何样品,检查13《0.85、14《0.85、17《0.85。如果是真的,则检测到异常探针,并将样品重新分类为h异常,其中h=3。这基本上是对重复探针13、14、17的检查,以确定重复探针13、14、17是否低于预期。这可能表明由于性能不佳,探针11、21或22未检测到缺失。如果任何探针的探针比》0.85,则将样品分类为正
常。当样品被分类为正常或分类为h=3为基因型时,该过程在框540处继续进行。
[0061]
在框512处,基于指示与大缺失相关联的异常探针比模式的样品探针/参考探针比确定具有大缺失的样品的分类。大缺失包含hba1和hba2基因两者,而小缺失仅包含hba1或hba2。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)来鉴定与大缺失相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)包括表3中列出的探针8、10、11、13、14和17的第一组探针的相对探针比数据,(ii)用于预测样品为具有大缺失的探针10的拷贝数调用阈值《0.75,和(iii)用于预测样品为具有大缺失的探针8和11的拷贝数调用阈值《0.75,和探针13、14和17的拷贝数调用阈值《0.63。如果分析结果预测样品具有大缺失,则将样品分类为具有大缺失。如果分析结果未能预测样品具有大缺失,则将样品分类为具有cnv而无大缺失。当样品被分类为具有大缺失时,该过程在框528处继续进行。当样品被分类为具有cnv而没有大缺失时,该过程在框516处继续进行。
[0062]
在框516处,基于指示与多态性相关联的异常探针比模式的样品探针/参考探针比确定具有多态性的样品的分类。分类的确定可以包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)来鉴定与多态性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)包括表3中列出的探针4、8、11、21和22的第二组探针的相对探针比数据,和(ii)用于预测样品的多态性的探针4、8、11、21和22的拷贝数调用阈值≥0.75且≤1.3。如果分析结果预测样品具有多态性,则将样品分类为具有多态性。如果分析结果未能预测样品具有多态性,则将样品分类为具有cnv而无多态性。当样品被分类为多态时,过程在框540处继续进行。当样品被分类为具有cnv而无多态性时,过程在框520处继续进行。
[0063]
在框520处,基于指示与α
3.7
缺失和合子性相关联的异常探针比模式的样品探针/参考探针比确定具有α
3.7
缺失和合子性的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与α
3.7
缺失和合子性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于α
3.7
纯合子的包括表3中列出的探针21、22和8的第三组探针的相对探针比数据,(ii)拷贝数调用阈值检查是否21《0.1、22《0.1、8》0.75以用于预测样品具有α
3.7
纯合缺失,(iii)对于α
3.7
杂合子的包括表3中列出的探针21、22和8的第四组探针的相对探针比数据,和(iv)拷贝数调用阈值检查是否21《0.75、22《0.75、8》0.75以用于预测样品具有α
3.7
杂合缺失。如果分析结果预测样品具有α
3.7
纯合缺失,则将样品分类为具有α
3.7
纯合缺失。如果分析结果预测样品具有α
3.7
杂合缺失,则将样品分类为具有α
3.7
杂合缺失。
[0064]
另外,在框520处,结合确定具有α
3.7
缺失和合子性的样品的分类、在确定具有α
3.7
缺失和合子性的样品的分类之前或之后,基于指示与α
4.2
缺失和合子性相关联的异常探针比模式的样品探针/参考探针比确定具有α
4.2
缺失和合子性的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与α
3.7
缺失和合子性相关联的拷贝数丢失或增加
(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于α
4.2
杂合子和纯合子的包括表3中列出的探针11、21和22的第四组探针的相对探针比数据,(ii)拷贝数调用阈值检查是否11《0.1、21》0.75、22》0.75以用于预测样品具有α
4.2
纯合缺失,和(iii)拷贝数调用阈值检查是否11《0.75、21》0.75、22》0.75以用于预测样品具有α
4.2
杂合缺失。如果分析结果预测样品具有α
4.2
纯合缺失则将样品分类为具有α
4.2
纯合缺失。如果分析结果预测样品具有α
4.2
杂合缺失则将样品分类为具有α
4.2
杂合缺失。如果分析结果未能预测样品具有α
4.2
缺失;然而,α
3.7
缺失分析的结果推断样品确实具有α
3.7
杂合或纯合缺失,则将样品分类为具有α
3.7
杂合或纯合缺失。如果分析结果未能预测样品具有α
4.2
缺失或α
3.7
缺失,则将样品分类为具有cnv而不具有α
4.2
杂合或纯合缺失或α
3.7
杂合或纯合缺失。当样品被分类为具有α
3.7
杂合或纯合缺失或α
4.2
杂合或纯合缺失时,过程在框522处继续进行。当样品被分类为具有cnv而没有α
3.7
杂合或纯合缺失或α
4.2
杂合或纯合缺失时,过程在框524处继续进行。
[0065]
在框522处,基于指示与α
3.7
重复相关联的异常探针比模式的样品探针/参考探针比确定具有α
4.2
杂合或纯合缺失和/或具有α
3.7
重复的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与α
3.7
重复相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)当存在α
4.2
杂合或纯合缺失时对于α
3.7
重复的包括表3中列出的探针8、11、21和22的第五组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8《1.3、》0.75、11《0.75、21》1.3、22》1.3以用于预测样品具有α
3.7
重复。如果分析结果预测分类为具有α
4.2
杂合或纯合缺失的样品还具有α
3.7
重复,则样品被分类为具有α
4.2
杂合或纯合缺失和α
3.7
重复。如果分析结果预测分类为具有α
4.2
杂合或纯合缺失的样品不具有α
3.7
重复,则样品的分类为具有α
4.2
杂合或纯合缺失。当样品被分类为具有α
4.2
杂合或纯合缺失和α
3.7
重复时,过程在框540处继续进行。当样品被分类为具有α
3.7
杂合或纯合缺失或α
4.2
杂合或纯合缺失时,过程在框540处继续进行。
[0066]
在框524处,基于指示与α
3.7
缺失和α
4.2
缺失相关联的异常探针比模式的样品探针/参考探针比确定具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与α
3.7
缺失和合子性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于α
3.7
缺失和α
4.2
缺失的包括表3中列出的探针8、29、1111、21、22、16和19的第六组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8《1.3、》0.75、29《1.3、》0.75、11《0.75、21《0.75、22《0.75、16《0.1或19《0.1以用于预测样品具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子。如果分析结果预测样品具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子,则将样品分类为具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子。当样品被分类为具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子时,过程在框540处继续进行。
[0067]
另外,在框524处,结合确定具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子的样品的分类、在确定具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子的样品的分类之前或之后,基于指示与重复相关联的异常探针比模式的样品探针/参考探针比确定具有重复的样品的分类。分
类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与重复相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于重复的包括表3中列出的探针8、21和22的第七组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8》0.75、21》1.3、22》1.3以用于预测样品具有重复。如果分析结果预测样品具有重复,则将样品分类为具有重复。当样品被分类为具有重复时,过程在框540处继续进行。如果分析结果未能预测样品具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子和/或重复,则将样品分类为具有cnv而不具有α
4.2
杂合或纯合缺失、α
3.7
杂合或纯合缺失和/或重复。当样品被分类为具有cnv而不具有α
4.2
杂合或纯合缺失、α
3.7
杂合或纯合缺失和重复时,过程在框526处继续进行。
[0068]
在框526处,基于指示与h异常的cnv相关联的异常探针比模式的样品探针/参考探针比确定h异常的cnv的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与h异常的cnv相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于h异常的cnv的包括表3中列出的探针1、4-6、8、11、21、22、29-37的第八组探针的相对探针比数据,和(ii)对于探针1、4-6、8、11、21、22、29-37,拷贝数调用阈值检查是否8《0.75或》1.3(确定有多少重要的缺失探针是异常的,并提供异常的那些探针的h计数)以用于预测样品具有h异常的cnv。如果分析结果预测样品具有h异常的cnv,则将样品分类为h异常的cnv(其中h代表有多少个重要的缺失探针是异常的)。当样品被分类为具有h异常的cnv时,过程在框540处继续进行。
[0069]
在框528处,基于指示与大缺失合子性相关联的异常探针比模式的样品探针/参考探针比确定具有大纯合缺失或大杂合缺失的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与大缺失合子性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于大纯合缺失的包括表3中列出的探针8、10、13、14和17的第九组探针的相对探针比数据,(ii)拷贝数调用阈值检查是否探针8、10或13、14、17《0.1以用于预测样品具有大纯合缺失,(iii)对于大杂合缺失的包括表3中列出的探针8、11、13、14和17的第十组探针的相对探针比数据,和(iv)拷贝数调用阈值检查是否探针8和11《0.75或13、14、17《0.63以用于预测样品具有大杂合缺失。如果分析结果预测样品具有大纯合缺失,则将样品分类为具有大纯合缺失。如果分析结果预测样品具有大杂合缺失,则将样品分类为具有大杂合缺失。当样品被分类为具有大纯合缺失时,过程在框530处继续进行。当样品被分类为具有大杂合缺失时,过程在框532处继续进行。
[0070]
在框530处,基于指示与规定缺失(包括sea、med1、med2、thai、fil和α
20.5
)相关联的异常探针比模式的样品探针/参考探针比确定具有特定的大纯合缺失的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与规定缺失相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探
针/参考探针比和拷贝数调用阈值包括:(i)对于大sea纯合缺失的包括表3中列出的探针8、31、32、5、6、33和34的第十一组探针的相对探针比数据,(ii)拷贝数调用阈值检查是否8《0.1、31《0.1、32《0.1、5》0.75、6》0.75、33》0.75、34》0.75以用于预测样品具有大sea纯合缺失,(iii)对于大med1纯合缺失的包括表3中列出的探针8、30、31、5、6、32和33的第十二组探针的相对探针比数据,(iv)拷贝数调用阈值检查是否8《0.1、30《0.1、31《0.1、5》0.75、6》0.75、32》0.75、33》0.75以用于预测样品具有大med1纯合缺失,(v)对于大med2纯合缺失的包括表3中列出的探针5、6、30、4、31和32的第十三组探针的相对探针比数据,(vi)拷贝数调用阈值检查是否5《0.1、6《0.1、30《0.1、4》0.75、31》0.75、32》0.75以用于预测样品具有大med2纯合缺失,(vii)对于大α
20.5
纯合缺失的包括表3中列出的探针8、6、29和30的第十四组探针的相对探针比数据,(viii)拷贝数调用阈值检查是否8《0.1、6》0.75、29》0.75、30》0.75以用于预测样品具有大α
20.5
纯合缺失,(ix)对于大fil/thai纯合缺失的包括表3中列出的探针6、8、31、32、5、33和34的第十五组探针的相对探针比数据,(x)拷贝数调用阈值检查是否6《0.1、8《0.1、31《0.1、32《0.1、5》0.75、33》0.75、34》0.75以用于预测样品具有大fil/thai纯合缺失,(xi)对于大α
3.7a
/α
4.2c
纯合缺失的包括表3中列出的探针9、22、6、8、29和30的第十六组探针的相对探针比数据,(xii)拷贝数调用阈值检查是否9《0.1、22《0.1、6》0.75、8》0.75、29》0.75、30》0.75以用于预测样品具有大α
3.7a
/α
4.2c
纯合缺失,(xiii)对于α
4.2
纯合缺失的包括表3中列出的探针11、21和22的第十七组探针的相对探针比数据,(xiv)拷贝数调用阈值检查是否11《0.1、21》0.75、22》0.75以用于预测样品具有α
4.2
纯合缺失,(xv)对于大α
4.2
杂合缺失的包括表3中列出的探针8、21、22和11的第十八组探针的相对探针比数据,和(xvi)拷贝数调用阈值检查是否8《0.75、》0.25、21《0.75、》0.25、22《0.75、》0.25、11《0.1以用于预测样品具有大α
4.2
杂合缺失。
[0071]
如果分析结果预测样品具有大sea纯合缺失,则将样品分类为具有大sea纯合缺失。如果分析结果预测样品具有大med1纯合缺失,则将样品分类为具有大med1纯合缺失。如果分析结果预测样品具有大med2纯合缺失,则将样品分类为具有大med2纯合缺失。如果分析结果预测样品具有大α
20.5
纯合缺失,则将样品分类为具有大α
20.5
纯合缺失。如果分析结果预测样品具有大fil/thai纯合缺失,则将样品分类为具有大fil/thai纯合缺失。如果分析结果预测样品具有大α
3.7a
/α
4.2c
纯合缺失,则将样品分类为具有大α
3.7a
/α
4.2c
纯合缺失。如果分析结果预测样品具有α
4.2
纯合缺失,则将样品分类为具有α
4.2
纯合缺失。如果分析结果预测样品具有大α
4.2
杂合缺失,则将样品分类为具有大α
4.2
杂合缺失。当样品被分类为具有大纯合缺失时,过程在框530处继续进行。当样品被分类为具有大sea、med1,med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
缺失,或α
4.2
纯合缺失,或大的α
4.2
杂合缺失时,过程在框540处继续进行。
[0072]
此时,如果样品仍为非特异性分类的纯合缺失(非大sea、med1、med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
缺失,或α
4.2
纯合缺失,或大α
4.2
杂合缺失),过程在框530处继续进行,其中基于指示与规定缺失(包括α
3.7
、α
4.2
、sea、med1、med2、thai、fil和α
20.5
)相关的其他异常探针比模式的样品探针/参考探针比确定具有特定大的纯合缺失的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与规定缺失相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于大sea/med1纯合缺失的包括表3中列出的探针8、13、14、
17、32、6和33的第十九组探针的相对探针比数据,(ii)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、17《0.1、32《0.75、》0.25、6》0.75、33》0.75以用于预测样品具有大sea/med1纯合缺失,(iii)对于大sea/(med2/dutch)纯合缺失的包括表3中列出的探针8、13、14、17、30、31、5、6和33的第二十组探针的相对探针比数据,(iv)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、17《0.1、30《0.1、31《0.75、》0.25、5《0.75、》0.25、6》0.75、33》0.75以用于预测样品具有大sea/(med2/dutch)纯合缺失,(v)对于大sea/α
20.5
纯合缺失的包括表3中列出的探针8、13、14、29、30、31、32、8和33的第二十一组探针的相对探针比数据,(vi)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、29《0.75、》0.25、30《0.75、》0.25、31《0.75、》0.25、32《0.75、》0.25、6》0.75、33》0.75以用于预测样品具有大sea/α
20.5
纯合缺失,(vii)对于大sea/(fil/thai)纯合缺失的包括表3中列出的探针8、13、14、17、32、8、6和33的第二十二组探针的相对探针比数据,(viii)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、17《0.1、32《0.1、8《0.75、》0.25、6》0.75、33》0.75以用于预测样品具有大sea/(fil/thai)纯合缺失,(ix)对于大med1/(med2/dutch)纯合缺失的包括表3中列出的探针8、13、14、17、30、31、5、6、4和32的第二十三组探针的相对探针比数据,(x)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、17《0.1、30《0.1、31《0.75、》0.25、5《0.75、》0.25、6《0.75、》0.25、4》0.75、32》0.75以用于预测样品具有大med1/(med2/dutch)纯合缺失,(xi)对于大med1/α
20.5
纯合缺失的包括表3中列出的探针8、13、14、17、31、6和32的第二十四组探针的相对探针比数据,(xii)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、17《0.1、31《0.75、》0.25、6》0.75、32》0.75以用于预测样品具有大med1/α
20.5
纯合缺失,(xiii)对于大med1/(fil/thai)纯合缺失的包括表3中列出的探针8、13、14、17、31、32、6、5和33的第二十五组探针的相对探针比数据,(xiv)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、17《0.1、31《0.1、32《0.75、》0.25、6《0.75、》0.25、5》0.75、33》0.75以用于预测样品具有大med1/(fil/thai)纯合缺失,(xiii)对于大(med2/dutch)/α
20.5
纯合缺失的包括表3中列出的探针13、14、17、5、6、29、30、31和4的第二十六组探针的相对探针比数据,(xiv)拷贝数调用阈值检查是否13《0.1、14《0.1、17《0.1、5《0.75、》0.25、6《0.75、》0.25、29《0.75、》0.25、30《0.75、》0.25、31》0.75、4》0.75以用于预测样品具有大(med2/dutch)/α
20.5
纯合缺失,(xv)对于大(med2/dutch)/(fil/thai)纯合缺失的包括表3中列出的探针8、13、14、17、6、31、5和33的第二十七组探针的相对探针比数据,(xvi)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、17《0.1、6《0.75、》0.25、31《0.75、》0.25、5》0.75、33》0.75以用于预测样品具有大(med2/dutch)/(fil/thai)纯合缺失,(xvii)对于大α
20.5
/(fil/thai)纯合缺失的包括表3中列出的探针8、13、14、17、6、29、5和33的二十八组探针的相对探针比数据,和(xviii)拷贝数调用阈值检查是否8《0.1、13《0.1、14《0.1、17《0.1、6《0.75、》0.25、29《0.75、》0.25、5》0.75、33》0.75以用于预测样品具有大α
20.5
/(fil/thai)纯合缺失。
[0073]
如果分析结果预测样品具有大sea/med1纯合缺失,则将样品分类为具有大sea/med1纯合缺失。如果分析结果预测样品具有大sea/(med2/dutch)纯合缺失,则将样品分类为具有大sea/(med2/dutch)纯合缺失。如果分析结果预测样品具有大sea/(med2/dutch)纯合缺失,则将样品分类为具有大sea/(med2/dutch)纯合缺失。如果分析结果预测样品具有大sea/α
20.5
纯合缺失,则将样品分类为具有大sea/α
20.5
纯合缺失。如果分析结果预测样品具有大sea/(fil/thai)纯合缺失,则将样品分类为具有大sea/(fil/thai)纯合缺失。如果分
析结果预测样品具有大med1/(med2/dutch)纯合缺失,则将样品分类为具有大med1/(med2/dutch)纯合缺失。如果分析结果预测样品具有大med1/α
20.5
纯合缺失,则将样品分类为具有大med1/α
20.5
纯合缺失。如果分析结果预测样品具有大med1/(fil/thai)纯合缺失,则将样品分类为具有大med1/(fil/thai)纯合缺失。如果分析结果预测样品具有大(med2/dutch)/α
20.5
纯合缺失,则将样品分类为具有大(med2/dutch)/α
20.5
纯合缺失。如果分析结果预测样品具有大(med2/dutch)/(fil/thai)纯合缺失,则将样品分类为具有大(med2/dutch)/(fil/thai)纯合缺失。如果分析结果预测样品具有大α
20.5
/(fil/thai)纯合缺失,则将样品分类为具有大α
20.5
/(fil/thai)纯合缺失。此时,当样品仍为非特异性分类的纯合缺失(非大sea、med1、med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
缺失,或α
4.2
纯合缺失,或大α
4.2
杂合缺失)时,过程在框540处继续进行。当样品被分类为具有大sea、med1、med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
缺失,或α
4.2
纯合缺失,或大α
4.2
杂合缺失时,过程在框540处继续进行。
[0074]
在框532处,基于指示与规定缺失(包括sea、med1、med2、thai、fil和α
20.5
)相关的异常探针比模式的样品探针/参考探针比确定具有特定大杂合缺失的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与规定缺失相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于大sea杂合缺失的包括表3中列出的探针8、31、32、5、6、33和34的第二十九组探针的相对探针比数据,(ii)拷贝数调用阈值检查是否8《0.75,31《0.75,32《0.75,5》0.75,6》0.75,33》0.75,345》0.75以用于预测样品具有大sea杂合缺失,(iii)对于大med1杂合缺失的包括表3中列出的探针8、30、31、5、6、32和33的第三十组探针的相对探针比数据,(iv)拷贝数调用阈值检查是否8《0.75、30《0.75、31《0.75、5》0.75、6》0.75、32》0.75、33》0.75以用于预测样品具有大med1杂合缺失,(v)对于大med2杂合缺失的包括表3中列出的探针5、6、30、4、31和32的第三十一组探针的相对探针比数据,(vi)拷贝数调用阈值检查是否5《0.75、6《0.75、30《0.75、4》0.75、31》0.75、32》0.75以用于预测样品具有大med2杂合缺失,(vii)对于大α
20.5
杂合缺失的包括表3中列出的探针8、6、29和30的第三十二组探针的相对探针比数据,(viii)拷贝数调用阈值检查是否8《0.75、6》0.75、29》0.75、30》0.75以用于预测样品具有大α
20.5
杂合缺失,(ix)对于大fil/thai杂合缺失的包括表3中列出的探针6、8、31、32、5、33和34的第三十三组探针的相对探针比数据,(x)拷贝数调用阈值检查是否6《0.75、85《0.75、31《0.75、32《0.75、5》0.75、33》0.75、34》0.75以用于预测样品具有大fil/thai杂合缺失,(xi)对于大α
3.7a
/α
4.2c
杂合缺失的包括表3中列出的探针9、22、6、8、29和30的第三十四组探针的相对探针比数据,(xii)拷贝数调用阈值检查是否9《0.75、22《0.75、6》0.75、8》0.75、29》0.75、30》0.75以用于预测样品具有大α
3.7a
/α
4.2c
杂合缺失,(xiii)对于α
4.2
纯合缺失的包括表3中列出的探针11、21和22的第三十五组探针的相对探针比数据,(xiv)拷贝数调用阈值检查是否11《0.1、21》0.75、22》0.75以用于预测样品具有α
4.2
纯合缺失,(xv)对于大α
3.7
杂合缺失的包括表3中列出的探针21、22和11的第三十六组探针的相对探针比数据,(xvi)拷贝数调用阈值检查是否21《0.15、22《0.15、11《0.75、》0.25以用于预测样品具有大α
3.7
杂合缺失,(xvii)对于α
3.7
纯合缺失的包括表3中列出的探针21、22和11的第三十七组探针的相对探针比数据,(xviii)拷贝数调用阈值检查是否21《0.15、22《0.15、11《0.75、》0.25以用于预测样品具有α
3.7
纯合缺失,(xix)对于大α
4.2
杂合缺失的包
括表3中列出的探针8、21、22和11的第三十八组探针的相对探针比数据,和(xviii)拷贝数调用阈值检查是否8《0.75、》0.25、21《0.75、》0.25、22《0.75、》0.25、11《0.1以用于预测样品具有大α
4.2
杂合缺失。
[0075]
如果分析结果预测样品具有大sea杂合缺失,则将样品分类为具有大sea杂合缺失。如果分析结果预测样品具有大med1杂合缺失,则将样品分类为具有大med1杂合缺失。如果分析结果预测样品具有大med2杂合缺失,则将样品分类为具有大med2杂合缺失。如果分析结果预测样品具有大α
20.5
杂合缺失,则将样品分类为具有大α
20.5
杂合缺失。如果分析结果预测样品具有大fil/thai杂合缺失,则将样品分类为具有大fil/thai杂合缺失。如果分析结果预测样品具有大α
3.7a
/α
4.2c
杂合缺失,则将样品分类为具有大α
3.7a
/α
4.2c
杂合缺失。如果分析结果预测样品具有α
4.2
纯合缺失,则将样品分类为具有α
4.2
纯合缺失。如果分析结果预测样品具有大α
3.7
杂合缺失,则将样品分类为具有大α
3.7
杂合缺失。如果分析结果预测样品具有α
3.7
纯合缺失,则将样品分类为具有α
3.7
纯合缺失。如果分析结果预测样品具有大α
4.2
杂合缺失,则将样品分类为具有大α
4.2
杂合缺失。当样品被分类为具有大杂合缺失时,过程在框534处继续进行。当样品被分类为具有大sea、med1、med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
或α
4.2
纯合缺失,或大α
3.7
杂合缺失,或α
3.7
纯合缺失,或大α
4.2
杂合缺失时,过程在框536处继续进行。
[0076]
在框534处,基于指示与大缺失和α
3.7
缺失两者相关的异常探针比模式的样品探针/参考探针比确定具有包括未指明的大缺失和α
3.7
缺失的复合杂合基因型的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与α
3.7
缺失和合子性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于α
3.7
缺失的包括表3中列出的探针8、29、11、21、22、16和19的第三十九组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8《0.75、29《0.75、11《0.75、21《0.1、22《0.1、16《0.1或19《0.1以用于预测样品具有包括未指明的大缺失和α
3.7
缺失的复合杂合子。如果分析结果预测样品具有包括α
3.7
缺失的复合杂合子,则将样品分类为具有包括α
3.7
缺失的复合杂合子的大杂合缺失。如果分析结果预测样品不具有包括α
3.7
缺失的复合杂合子,则将样品分类为大杂合缺失。
[0077]
另外,在框534处,基于指示与大缺失和α
4.2
缺失两者相关的异常探针比模式的样品探针/参考探针比确定具有包括未指明的大缺失和α
4.2
缺失的复合杂合基因型的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与α
4.2
缺失和合子性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于α
4.2
缺失的包括表3中列出的探针8、29、11、21、22、16和19的第四十组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8《0.75、29《0.75、11《0.1、21《0.75、22《0.75以用于预测样品具有包括未指明的大缺失和α
4.2
缺失的复合杂合子。如果分析结果预测样品具有包括α
4.2
缺失的复合杂合子,则将样品分类为具有包括α
4.2
缺失的复合杂合子的大杂合缺失。如果分析结果预测样品不具有包括α
4.2
缺失的复合杂合子,则将样品分类为大杂合缺失。
[0078]
另外,在框534处,结合确定具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子的样品的分
类、在确定具有包括α
3.7
缺失和α
4.2
缺失的复合杂合子的样品的分类之前或之后,基于指示与重复相关联的异常探针比模式的样品探针/参考探针比确定具有重复的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与重复相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于重复的包括表3中列出的探针8、21和22的第四十一组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8《0.75、21》0.75、22》0.75以用于预测样品具有重复。
[0079]
如果分析结果预测样品具有与α
3.7
缺失结合的未指明的大杂合缺失或然后将样品分类为与α
3.7
缺失结合的未指明的大杂合缺失。当样品被分类为具有包括α
4.2
缺失的复合杂合子的未指明的大杂合缺失时,过程在框540处继续进行。当样品被分类为与重复结合的未指明的大杂合缺失时,将样品分类为具有未指明的大缺失和重复,并且过程在框540处继续进行。如果分析结果预测样品具有大杂合缺失而不具有重复时,则将样品分类为大杂合缺失。当样品被分类为大杂合缺失时,过程在框540处继续进行。
[0080]
在框536处,基于指示与特异性靶向的大缺失和α
3.7
缺失两者相关的异常探针比模式的样品探针/参考探针比确定具有包括特异性靶向的大缺失和α
3.7
缺失的复合杂合子的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与α
3.7
缺失和合子性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于α
3.7
缺失的包括表3中列出的探针8、29、11、21、22、16和19的第四十二组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8《0.75、29《0.75、11《0.75、21《0.1、22《0.1、16《0.1或19《0.1以用于预测样品具有包括α
3.7
缺失的复合杂合子。如果分析结果预测样品具有包括α
3.7
缺失的复合杂合子,则将样品分类为具有包括α
3.7
缺失的复合杂合子的特定大杂合缺失(例如,大sea、med1、med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
)。如果分析结果预测样品不包含α
3.7
缺失,则将样品分类为特定大杂合缺失(例如,大sea、med1、med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
。
[0081]
另外,在框536处,基于指示与特异性靶向的大缺失和α
4.2
缺失两者相关的异常探针比模式的样品探针/参考探针比确定具有包括特异性靶向的大缺失和α
4.2
缺失的复合杂合子的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与α
4.2
缺失和合子性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于α
3.7
缺失的包括表3中列出的探针8、29、11、21、22、16和19的第四十三组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8《0.75、29《0.75、11《0.1、21《0.75、22《0.75以用于预测样品具有包括α
4.2
缺失的复合杂合子。如果分析结果预测样品具有包括α
4.2
缺失的复合杂合子,则将样品分类为具有包括α
4.2
缺失的复合杂合子的特定大杂合缺失(例如,大sea、med1、med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
)。如果分析结果预测样品不包含α
3.7
缺失,则将样品分类为特定大杂合缺失(例如,大sea、med1、med2、α
20.5
、fil/thai或α
3.7a
/α
4.2c
。
[0082]
另外,在框536处,结合确定具有包括特定大缺失和α
3.7
缺失或α
4.2
缺失的复合杂合
缺失的样品的分类、在确定具有包括特定大缺失和α
3.7
缺失或α
4.2
缺失的复合杂合缺失的样品的分类之前或之后,基于指示与重复相关联的异常探针比模式的样品探针/参考探针比确定具有特定大缺失和重复的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与重复相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于重复的包括表3中列出的探针8、21和22的第四十四组探针的相对探针比数据,和(ii)拷贝数调用阈值检查是否8》0.1、21》0.75、22》0.75以用于预测样品具有特定大缺失和重复。
[0083]
如果分析结果预测样品具有特定大缺失和包括α
3.7
缺失的复合杂合子,则将样品分类为具有包括α
3.7
缺失的复合杂合子的特定大杂合缺失。如果分析结果预测样品具有特定大缺失和包括重复的复合杂合子,则将样品分类为具有包括重复的复合杂合子的特定大杂合缺失。如果分析结果预测样品具有特定大缺失和包括α
4.2
重复的复合杂合子,则将样品分类为具有包括重复的复合杂合子的特定大杂合缺失。当样品被分类为具有包括α
3.7
缺失、α
4.2
缺失或重复的复合杂合子的特定大杂合缺失时,过程在框540处继续进行。如果分析结果预测具有特定大杂合缺失的样品没有α
3.7
缺失、α
4.2
缺失或重复,则将样品分类为特定大杂合缺失。当样品被分类为特定大杂合缺失时,过程在框540处继续进行。
[0084]
在框540处,基于指示与hs40缺失合子性相关的异常探针比模式的样品探针/参考探针比确定具有hs40纯合缺失或hs40杂合缺失的样品的分类。分类的确定可包括通过决策树矩阵基于新的相对探针比数据和与一个或多个探针相关联的样品探针/参考探针比的正常范围(拷贝数调用阈值)鉴定与hs40缺失合子性相关联的拷贝数丢失或增加(例如缺失、重复或点突变)的一个或多个区域。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于hs40纯合缺失的包括表3中列出的探针1、4、2和3的第四十五组探针的相对探针比数据,(ii)拷贝数调用阈值检查是否1》0.75、《1.3、4》0.75、《1.3、2《0.1、3《0.1以用于预测样品具有hs40纯合缺失,(iii)对于hs40杂合缺失的包括表3中列出的探针1、4、2和3的第四十五组探针的相对探针比数据,和(iv)拷贝数调用阈值检查是否探针8和1》0.75、《1.3、4》0.75、《1.3、2《0.75、3《0.75以用于预测样品具有大杂合缺失。此外,在样品被分类为具有h异常的cnv的情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于额外的异常探针的包括表3中列出的探针2和3的第四十二组探针的相对探针比数据,(ii)拷贝数调用阈值检查是否2》0.75、3《0.75或2》0.75、3《0.75以用于预测样品具有额外的异常探针并更新异常的探针的数量h(如果任一为真,则向任何数量h(1-17)添加1)。
[0085]
此外,在框540处,对于具有“正常”或“多态性”分类的任何样品,检查13《0.85、14《0.85、17《0.85。如果是真的,则检测到异常探针,并将样品重新分类为h异常,其中h=3。对于具有“h异常的cnv”分类的任何样品,该算法检查是否探针13《0.85、14《0.85、17《0.85。如果为真,则检测到额外的异常探针,并更新异常的探针的数量h(向任何数量h(1-17)添加3)。
[0086]
此外,在框540处,针对具有hb constant spring单核苷酸变异(snv)的样品的分类检查所有样品。snv的存在或不存在由constant spring snv探针的存在或不存在来确定。在某些情况下,用于分析的样品探针/参考探针比和拷贝数调用阈值包括:(i)对于hs40
纯合缺失的表3中列出的探针18的阳性探针信号数据,(ii)探针信号》0。
[0087]
如果分析结果预测样品具有hs40纯合缺失,则将样品的先验分类与hs40纯合缺失连接。如果分析结果预测样品具有hs40杂合缺失,则将样品的先验分类与hs40杂合缺失连接。如果分析结果预测样品不具有hs40纯合或杂合缺失,则样品的先验分类不变。如果分析结果预测样品具有额外的异常探针,则更新异常探针的数量h,并且分类保持为h异常的cnv。如果分析结果预测样品具有hb constant spring突变,则样品的先验分类与hb constant spring连接。如果分析结果预测样品不具有hb constant spring,则样品的先验分类不变。
[0088]
该步骤540的结果为最终分类。此时,所有样品应分类为正常、仅包含多态性、具有一个或多个可确认的突变(例如,α
3.7
、α
4.2
、sea、med1、med2、thai、fil和/或α
20.5
)、具有h异常探针的cnv或不可分类的大缺失。如果样品包含具有h异常探针的cnv或不可分类的大缺失,则应手动审查样品。
[0089]
在框545处,基于每个样品的最终分类确定每个样品的hba基因型。例如,具有杂合α
3.7
或α
4.2
缺失和杂合sea、fil、med、thai或α
20.5
分类的样品将被基因分型为
‑‑
/-α(参见例如表2)。在任选的框550处,可以基于为每个样品确定的hba基因型和结果含义计算风险评分,如表2所示。在一些情况下,风险评分可以鉴定:(i)受试者是α
3.7
、α
4.2
、sea、med、thai、fil、α
20.5
和hs-40缺失以及hb constant spring点突变的携带者的风险,(ii)夫妇被确定为α
3.7
、α
4.2
、sea、med、thai、fil、α
20.5
和hs-40缺失和hb constant spring点突变的携带者的风险,和/或(iii)胎儿遗传α
3.7
、α
4.2
、sea、med、thai、fil、α
20.5
和hs-40缺失以及hb constant spring点突变的风险。
[0090]
在框555处,可以输出针对每个等位基因确定的hba基因型和任选的风险评分。针对每个等位基因确定的hba基因型和任选的风险评分的输出可包括向最终用户提供输出和/或将输出记录在存储设备中(例如,在用户界面上显示输出和/或将输出存储在数据库的结果文件中)。
[0091]
图6示出了适用于与根据本公开内容的使用hba测定平台和基因分型技术的hba基因分型的系统和方法一起使用的示例性计算设备600。示例性计算设备600包括处理器605,处理器605使用一个或多个通信总线615与计算设备600的存储器610和其他组件通信。处理器605被配置成执行存储在存储器610中的处理器可执行的指令,以执行用于搜索和鉴定存在于原始数据中的hba峰、确定样品的hba基因型和/或根据不同实例确定患者的风险评分(例如上文关于图4和5描述的示例性过程400或500的部分或全部)的一种或多种方法。在该实例中,存储器610存储提供hba峰分析620和hba基因型确定625的处理器可执行指令,如上文关于图1、图2、图4和图5所讨论的。
[0092]
在该实例中,计算设备600还包括一个或多个用户输入设备630,例如键盘、鼠标、触摸屏、麦克风等,以接受用户输入。计算设备600还包括显示器635,以向用户诸如用户界面提供视觉输出。计算设备600还包括通信接口640。在一些实例中,通信接口640可以使用一个或多个网络(包括局域网(“lan”);广域网(“wan”),诸如因特网;城域网(“man”);点对点或端对端连接等)实现通信。可以使用任何合适的网络协议来实现与其他设备的通信。例如,一种合适的网络协议可以包括因特网协议(“ip”)、传输控制协议(“tcp”)、用户数据报协议(“udp”)或其组合,例如tcp/ip或udp/ip。
iv.实施例
[0093]
可以通过参考以下实施例更好地理解在各种实施方案中实施的系统和方法。实施例1:hba测定和hba基因分型决策树矩阵样品、数据采集和hba测定分析
[0094]
在本示例性研究中使用从224份血液、41份产前样品和2种细胞系(coriell na03433,na10797)中提取的基因组dna。在血液样品中,49份为新鲜或存档样品。在去鉴定前,保留每个样品的任何α-地中海贫血基因型信息(如果存在)。所有样品在其用于本示例性研究之前均匿名。使用abi foundation data collection软件v3.0在abi 3730xl基因分析仪上收集原始数据,并上传至genemarker软件v2.7.0中用于分析样品质量、生成合成参考并将样品信号标准化至合成参考的信号以检测缺失和重复。用于分析的质量度量列于表1中。本研究使用了当不存在缺失或重复时相对探针比的生产商推荐的相阈值(相对于参考的倍数变化)。根据经验确定对照探针标准偏差的阈值。分析灵敏度和特异性
[0095]
为了建立分析的灵敏度和特异性,如关于图1-3所述的在hba mlpa测定中测试了已知基因型的69份样品和细胞系(39个阳性和30个阴性;表4)。使用hba基因分型决策树矩阵(关于图4和图5讨论的)和通过手动审查进行基因型调用。使用spectramax m2荧光计对所有样品进行定量,输入范围为12.5ng至100ng。基因型在使用前对操作者设盲。表4.缺失和重复基因型的列表
质量度量
[0096]
总体平均对照探针标准偏差为0.048
±
0.030。在测试的69个样品中,6个最初未通过样品质量度量(6/69;8.7%;表5),并使用相同的dna等分试样重复测试。这些样品中的3个样品的输入为15ng、16ng和29ng,其低于生产商建议的下限(50-100ng)。其他3个样品冷冻存档至少15年,并且未获得关于所用提取方法的信息。重新测试后,所有6个样品均通过质量度量,并被认为对于基因型调用是可接受的(表5)。表5.用于灵敏度/特异性样品集的质量度量
基因型调用
[0097]
使用表1中的质量检查阈值分析所有样品。使用hba基因分型决策树矩阵(关于图4和图5讨论的)或通过手动分析,均未检测到假阴性,并且所有30个阴性样品均正确调用,导致特异性为100%。在39个阳性样品中,分别使用hba基因分型决策树矩阵和手动分析调用35个和37个样品的靶向基因型(表6)。对于其中检测到变异但基因型未特异性调用的样品,2个为重复并且不是靶向的变异体。
[0098]
对于两个重复样品,手动分析正确鉴定出一个为杂合α
3.7
重复(样品zz-59),另一个为杂合α
4.2
重复(样品zz-48)。hba基因分型决策树矩阵也检测到重复,但不能将靶向的基因型中的一个分配给它们,而是标记这些样品用于进行手动审查。也就是说,对于zz-59,针对α
3.7
区域,除一个探针外,所有探针均高于1.30阈值以调用重复,并且hba基因分型决策树矩阵将其标记为手动审查,而不是将其调用为正常。对于具有α
4.2
重复的zz-48,未对hba基因分型决策树矩阵进行编程以调用该特定基因型。重要的是,对于任一样品,hba基因分型决策树矩阵均未错过调用,而是默认为手动审查。
[0099]
尽管不是本测定的预期靶向突变之一,但本研究纳入了对于brit缺失杂合的两个样品,以测试hba基因分型决策树矩阵。使用hba基因分型决策树矩阵和手动分析两者,因为相同的mlpa探针将检测sea或brit缺失,因此样品被称为杂合sea缺失。多重pcr未证实任一样品中的sea缺失。对于这些样品的通过hba mlpa的sea缺失调用不应被视为假阳性,因为brit缺失不是测定中的靶向突变,并且两种调用方法均检测到与brit缺失大小大致相同的大缺失。由于hba mlpa测定可检测到除此测试靶向的那些外可能具有临床意义的任何数量的大缺失,因此设计hba基因分型决策树矩阵以确保通过多个探针检测到的任何拷贝数变化均未遗漏,而是进行手动审查。因此,在阳性样品中鉴定出所有靶向突变,导致使用hba基因分型决策树矩阵或手动方法的100%的测定灵敏度。表6.灵敏度/特异性基因型调用的总结
*brit缺失重现性
[0100]
对于测定内重现性,在同一测定运行中以一式三份测试3个样品。这些相同的样品还用于使用第二批试剂、不同操作人员和在不同日期进行的测定间重现性。通过手动分析和hba基因分型决策树矩阵进行数据分析。对于测定内和测定间重现性,除1个样品(g05-intra1)外,所有样品均通过了针对对照探针标准偏差的qc度量(表7),尽管该样品的重复样品通过表明样品质量不是问题。mlpa泳道评分或质量控制片段未检测到mlpa反应的问题,并且未目视检测到该样品的蒸发。对于通过的样品,每个探针的相对于参考的倍数变化是可重现的(参见例如图7-单个探针(#15、16、19-23)以约0.5倍变化一致地检测到缺失,并且对于重复探针(#13、14、17)以约0.75倍变化一致地检测到缺失。多态性探针#12还以约0.75倍变化检测到缺失,表明该特定α
3.7
缺失的可能跨度),使用该算法的基因型调用与手动进行的调用100%一致。基于该数据,该测定在运行间是可重现的。表7.测定内和测定间重现性结果
*正常探针总数=正常探针数量 缺失/重复探针数量hba基因分型决策树矩阵性能
[0101]
用267个盲样品进一步评价hba基因分型决策树矩阵的性能。将基因型调用与通过手动审查的结果和/或来自另一个实验室的先前测试的结果进行比较。总体而言,在首次通过hba mlpa测定时,hba基因分型决策树矩阵和手动方法/先前的测试结果对于261个样品是一致的(261/267;97.8%;表8)。具有最少基因分型信息的两个样品(一个标记为“hydrops”,另一个标记为“hemo.h”),通过hba基因分型决策树矩阵分别基因分型为纯合sea缺失和具有α
3.7
缺失的反式sea缺失,并通过多重pcr确认。此外,如前所述,在多重pcr测定中,通过hba mlpa测定与sea缺失难以区分的具有brit缺失的两个样品未被确认为sea或任何其他靶向缺失。这些样品将在mlpa测定中重复进行以在临床实验室进行确认测试。表8.与手动分析相比较的基因分型算法的性能
*通过hba基因分型决策树矩阵对于预期的基因型未特别调用而是指示用于管理者审查的样品**通过hba mlpa测定与sea缺失难以区分的具有brit缺失的2个样品
[0102]
对hba基因分型决策树矩阵进行训练以检测标准化样品探针(无论其是否可以分配靶向基因型之一)的倍数变化模式,如表9所示,算法标记用于临床管理者审查的所有6个样品都是由于一个区域的相对探针比的配置不一致。此外,hba基因分型决策树矩阵被设计为在灵敏度方面出错(即假阳性),而不是错过任何调用(即假阴性)。因此,尽管hba基因分型决策树矩阵没有特异性地调用α
3.7
重复,但它确实用一个探针检测到异常,而不是错过调用,并将样品标记为用于手动审查。同样,对于三个正常样品,hba基因分型决策树矩阵鉴定了多个探针的相对探针比的不规则性,并指定这些用于手动审查,而不是错误调用基因型。表9.基因分型算法与手动方法之间的调用的差异
其他注意事项
[0103]
在上述描述中给出了具体细节以提供对实施方案的透彻理解。然而,应当理解,实施方案可以在没有这些具体细节的情况下实施。例如,回路可以在框图中示出,以避免在不必要的细节中掩盖实施方案。在其他情况下,可以在没有不必要的细节的情况下示出众所周知的回路、过程、算法、结构和技术,以避免掩盖实施方案。
[0104]
上述技术、模块、步骤和手段的实施可以以各种方式进行。例如,这些技术、模块、步骤和手段可在硬件、软件或其组合中实现。对于硬件实现,处理单元可在一个或多个专用集成电路(asic)、数字信号处理器(dsp)、数字信号处理设备(dspd)、可编程逻辑设备(pld)、现场可编程门阵列(fpga)、处理器、控制器、微控制器、微处理器、设计用于进行上述功能的其他电子单元和/或其组合内实现。
[0105]
此外,值得注意的是,实施方案可以被描述为一个过程,其被描绘为流程图、作业图、数据流程图、结构图或框图。虽然流程图可以将操作描述为顺序过程,但许多操作可以并行或同时进行。此外,可以重新安排操作顺序。过程在其操作完成时终止,但可能有不包括在图中的其他步骤。过程可以对应于方法、函数、程序、次例程、子程序等。当过程对应于函数时,它的终止对应于函数返回到调用函数或主函数。
[0106]
此外,实施方案可通过硬件、软件、脚本语言、固件、中间件、微码、硬件描述语言和/或其任何组合来实现。当在软件、固件、中间件、脚本语言和/或微码中实现时,进行必要任务的程序代码或代码段可以存储在机器可读介质(例如存储介质)中。代码段或机器可执行指令可以表示过程、函数、子程序、程序、例程、次例程、模块、软件包、脚本、类或指令、数据结构和/或程序语句的任何组合。代码段可以通过传递和/或接收信息、数据、命令行参数、参数和/或存储内容而耦合到另一个代码段或硬件电路。信息、命令行参数、参数、数据等可以通过任何合适的方式(包括内存共享、消息传递、票据传递、网络传输等)传递、转发或传输。
[0107]
对于固件和/或软件实现,方法学可以用执行本文所述功能的模块(例如,程序、功能等)来实现。任何有形地体现指令的机器可读介质可用于实现本文所述的方法学。例如,软件代码可以存储在存储器中。存储器可以在处理器内或处理器外部实现。如本文所用,术语“存储器”是指任何类型的长期、短期、易失性、非易失性或其他存储介质,并且不限于任何特定类型的存储器或存储器的数量,或存储器存储在其上的介质的类型。
[0108]
此外,如本文所公开的,术语“存储介质”、“存储”或“存储器”可以表示用于存储数据的一个或多个存储器,包括只读存储器(rom)、随机存取存储器(ram)、磁性ram、核心存储器、磁盘存储介质、光存储介质、闪存装置和/或用于存储信息的其他机器可读介质。术语“机器可读介质”包括但不限于便携式或固定存储设备、光存储设备、无线通道和/或能够存储包含或携带指令和/或数据的各种其他存储介质。
[0109]
虽然本公开内容的原理已经结合具体的设备和方法在上文中描述,但应当清楚地理解,该描述仅作为示例给出而不是作为对本公开内容范围的限制。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。