1.本发明涉及状态预测领域,尤其涉及一种基于特征基因提取的癌症患者生存状态预测方法。
背景技术:
2.恶性肿瘤是一种高度影响人类健康的疾病,其在医学领域受到广泛的重视。不同组织来源的恶性肿瘤在形态学与生物行为上各有其特点,但是不同恶性肿瘤细胞均与肿瘤微环境(tumor microenvironment,tme)之间存在错综复杂的联系,当正常细胞转变为肿瘤细胞时,其基因表达发生了一定的变化,获得了一系列的生物标志物,与此同时,tme的组成细胞针对肿瘤细胞也会发生一系列成分与功能的改变。这些改变在基因的表达与修饰之中可以体现。状态预测方法皆是基于特征基因提取的方法上进行预测,而现有的特征基因提取方法在样本量少的情况下无法使用。
技术实现要素:
3.本发明的目的是提供一种基于特征基因提取的癌症患者生存状态预测方法,解决了在样本量少、特征变量多的情况下无法提取到有意义特征的问题,从而提高预测准确率。
4.本发明所采用的第一技术方案是:一种基于特征基因提取的癌症患者生存状态预测方法,包括以下步骤:
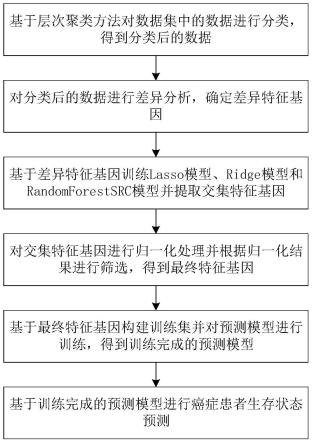
5.基于层次聚类方法对数据集中的数据进行分类,得到分类后的数据;
6.对分类后的数据进行差异分析,确定差异特征基因;
7.基于特征基因训练lasso模型、ridge模型和randomforestsrc模型并提取交集特征基因;
8.对交集特征基因进行归一化处理并计算对应的系数评分,得到最终特征基因;
9.基于最终特征基因和患者常规特征构建训练集并对预测模型进行训练,得到训练完成的预测模型;
10.基于训练完成的预测模型进行癌症患者生存状态预测。
11.进一步,还包括:
12.根据最终特征基因计算风险评估系数;
13.基于km曲线和hr值验证风险评估系数的准确性。
14.进一步,所述基于层次聚类方法对数据集中的数据进行分类,得到分类后的数据这一步骤,其具体包括:
15.获取数据集并对数据集中的数据进行预处理,得到预处理后的数据;
16.对预处理后的数据进行层次聚类分析,得到层次聚类结果;
17.根据层次聚类结果将数据集中的数据分类,得到分类后的数据。
18.进一步,所述对分类后的数据进行差异分析,确定差异特征基因这一步骤,其具体包括:
19.基于分类后的数据,两两之间进行差异性分析,得到差异性分析结果;
20.通过火山图显示差异性分析结果,并选择两两类别之间大于预设阈值的差异特征;
21.通过韦恩图显示各类别挑选出的差异特征,获得所有类别之间差异特征的交集,得到差异特征基因。
22.进一步,所述基于差异特征基因训练lasso模型、ridge模型和randomforestsrc模型并提取交集特征基因这一步骤,其具体包括:
23.基于特征基因训练lasso模型,提取非零特征系数绝对值前预设数量的特征基因;
24.基于特征基因训练ridge模型,提取非零特征系数绝对值前预设数量的特征基因;
25.基于特征基因训练randomforestsrc模型,提取非零特征系数绝对值前预设数量的特征基因;
26.对三种模型提取到的基因取交集,得到交集特征基因。
27.进一步,归一化处理的计算公式如下:
[0028][0029]
上式中,x表示特征基因的系数,x’表示归一化后的特征基因的系数。
[0030]
进一步,风险评估系数的计算公式如下:
[0031][0032]
上式中,表示lasso模型得到基因系数的归一化结果,表示ridge模型得到基因系数的归一化结果,表示randomforestsrc模型得到基因系数的归一化结果,coefficient(xi)表示系数评分。
[0033]
本发明方法的有益效果是:本发明先通过层次聚类方法和差异分析方法对数据集的大量基因进行初步筛选,最后基于lasso、ridge、randomforestsrc三种模型和归一化方法提取最有价值的基因,这种方法在针对特点为样本量不多,但是特征变量特别多、且个别变量间存在高度的线性相关性的数据集尤其有效。
附图说明
[0034]
图1是本发明一种基于特征基因提取的癌症患者生存状态预测方法的步骤流程图。
具体实施方式
[0035]
下面结合附图和具体实施例对本发明做进一步的详细说明。对于以下实施例中的步骤编号,其仅为了便于阐述说明而设置,对步骤之间的顺序不做任何限定,实施例中的各步骤的执行顺序均可根据本领域技术人员的理解来进行适应性调整。
[0036]
如图1所示,本发明提供了一种基于特征基因提取的癌症患者生存状态预测方法,
该方法包括以下步骤:
[0037]
s1、基于层次聚类方法对数据集中的数据进行分类,得到分类后的数据;
[0038]
s1.1、获取数据集并对数据集中的数据进行预处理,得到预处理后的数据;
[0039]
具体地,在tcga官网上下载宫颈癌的基因测序表达数据集,对数据进行预处理,预处理步骤包括转id和处理缺失值等步骤。
[0040]
s1.2、对预处理后的数据进行层次聚类分析,得到层次聚类结果;
[0041]
s1.3、根据层次聚类结果将数据集中的数据分类,得到分类后的数据。
[0042]
具体地,先对数据进行gsva分析后,使用层次聚类对gsva分析结果进行聚类分析。用热图显示聚类分析的结果,根据热图确定将样本划分为3类。
[0043]
gsva是一种非参数,无监督算法。全名gene set variation analysis(基因集变异分析)。gsva无需预先对样本进行分组,就可以计算每个样本中特定基因集的富集分数。可以理解成gsva转化了基因表达数据,把单个基因作为特征的表达矩阵,转化为特定基因集作为特征的表达矩阵。
[0044]
层次聚类是聚类分析的一中。它是通过计算数据点之间的相似度来创建一棵有层次的嵌套聚类树。可以采用“自底向上”的聚合策略,也可以采用“自顶向下”的分拆策略。
[0045]
差异性分析是常见的数据分析方法,用于检测科学实验中实验组和对照组之间是否有差异以及差异是否显著,又称为差异性显著检验。
[0046]
s2、对分类后的数据进行差异分析,确定差异特征基因;
[0047]
s2.1、基于分类后的数据,两两之间进行差异性分析,得到差异性分析结果;
[0048]
s2.2、通过火山图显示差异性分析结果,并选择两两类别之间大于预设阈值的差异特征;
[0049]
s2.3、通过韦恩图显示各类别挑选出的差异特征,获得所有类别之间差异特征的交集,得到差异特征基因。
[0050]
具体地,对分成3类的样本,两两之间进行差异性分析,使用火山图显示差异分析结果。根据火山图确定阈值p_value=0.01。挑选出差异分析结果中p-value《0.01的特征基因。使用韦恩图显示3类别两两类别之间差异基因的交集。通过上述的数据分析与对结果的筛选,得到481个基因,这些基因有免疫基因也有非免疫基因。因为是研究免疫相关基因,所以只提取481中的免疫基因。最终提取出309个免疫基因。
[0051]
s3、基于特征基因训练lasso模型、ridge模型和randomforestsrc模型并提取交集特征基因;
[0052]
s3.1、基于特征基因训练lasso模型,提取非零特征系数绝对值前预设数量的特征基因;
[0053]
s3.2、基于特征基因训练ridge模型,提取非零特征系数绝对值前预设数量的特征基因;
[0054]
s3.3、基于特征基因训练randomforestsrc模型,提取非零特征系数绝对值前预设数量的特征基因;
[0055]
s3.4、对三种模型提取到的基因取交集,得到交集特征基因。
[0056]
具体地,提取非零特征系数绝对值前top_n的特征基因,使用步骤s2提取的309个基因组成的数据集作为训练集。训练lasso、ridge、randomforestsrc模型。提取模型中重要
性系数绝对值的前100基因。取三种模型前100特征基因的交集特征基因。
[0057]
s4、对交集特征基因进行归一化处理并计算对应的系数评分,得到最终特征基因;
[0058]
具体地,将三种模型交集特征基因系数归一化后相加得到基因的重要性系数。根据得到的基因系数情况,取系数大于1的前5个基因作为后续分析的基因。到这里就已经从6万多的基因中提取5个基因。
[0059]
归一化处理的计算公式如下:
[0060][0061]
上式中,x表示特征基因的系数,x’表示归一化后的特征基因的系数。
[0062]
计算特征基因的系数评分,公式如下:
[0063][0064]
上式中,表示lasso模型得到基因系数的归一化结果,表示ridge模型得到基因系数的归一化结果,表示randomforestsrc模型得到基因系数的归一化结果,coefficient(xi)表示系数评分。
[0065]
s5、基于最终特征基因构建训练集并对预测模型进行训练,得到训练完成的预测模型;
[0066]
s5.1、使用步骤s4选择的top_n特征数据集,将数据3/7分成测试集和训练集
[0067]
s5.2、使用训练集训练svm、randomforest、neural net、adaboost模型。
[0068]
s5.3、使用测试集测试svm、randomforest、neural net、adaboost模型的预测准确率。
[0069]
具体地,选择步骤s4中得到的风险系数与患者年龄、患者种族、患者癌症分期、患者是否吸烟作为特征组成的数据集。将数据3/7划分成测试集和训练集,使用训练集训练randomforest、neural net、adaboost模型。三种模型在测试集上有很好的准确率。三种模型在测试集上预测结果可知,在测试集上的预测结果的准确率达到了80%以上。auc的面积值也达到了90%。
[0070]
s6、基于训练完成的预测模型进行癌症患者生存状态预测。
[0071]
进一步作为本方法的优选实施例,还包括:
[0072]
s7、根据最终特征基因计算风险评估系数;
[0073]
具体地,将特征基因系数评分乘以表达值的均值得到每个样本的风险评估系数,计算公式如下:
[0074][0075]
上式中,expression(mrna
ij
)表示样本i的第j个基因的表达值,coefficient(mrna
ij
)表示样本i的第j个基因的系数评分,risk_coefi表示样本i的风险评估系数。
[0076]
s8、基于km曲线和hr值验证风险评估系数的准确性。
[0077]
s8.1、按照风险评估系数把样本分成高低两组。大于mean(risk_coef)的样本为高组,不低于mean(risk_coef)的样本为低组;
[0078]
s8.2、画出高低两组的km曲线,通过p值检验风险评估系数的准确性;
[0079]
s8.3、计算hr值,检验风险评估系数的准确性。
[0080]
具体地,使用前5个基因系数乘以该基因的表达值的均值把样本分成高低两组,作km曲线图。p值是0.00003,说明high组比low组有更高的生存率。hr值是3.2,说明生存率高出了3.2倍多。
[0081]
km生存分析是指根据试验或调查得到的数据对生物或是人的生存时间进行分析和推断,研究生存时间和结局与众多影响因素间关系及其程度大小的方法。kaplan-meier曲线估计生存率是非参数的方法。kaplan-meier生存曲线,它纵轴表示生存概率,横轴表示生存事件。它呈现为一条下降的曲线,下降的坡度越陡,表示生存率越低或生存时间越短,其斜率表示死亡速率。
[0082]
hr(风险比率)的英文名称是hazard ratio。风险比率是两个风险率的比值。在医学和公共卫生研究中,人们常常使用风险比率来表示实验组和对照组之间的风险差别。km生存曲线能够直观表示风险比率。曲线上的点表示此时存活人数占全组人数的比值,即生存率。生存率与风险率之和为图中在任意一个时间点上,两个组的风险率之比,就是风险比率
[0083]
本方法提供一种基于lasso、ridge、randomforestsrc三种模型归一化特征基因提取方法。先使用gsva分析、聚类分析、差异分析方法从6万多基因中筛选出481多基因,最后使用基于lasso、ridge、randomforestsrc三种模型归一化方法提取top_n基因。并且用km曲线对top_n基因是否有意义做验证。这种方法在针对特点为样本量不多,但是特征变量特别多、且个别变量间存在高度的线性相关性的数据集。tcga上的30多种癌症数据集都属于这种类型的数据集,所以这种方法在tcga数据库上的多种癌症数据集都能提取非常有意义的基因。这种方法解决了在样本量少,但是特征变量特别多的情况下,很多现有的机器学习特征提取方法无法使用。因为任何一种有效的机器学习必有其归纳偏好,所以本方法采用三种机器学习算法对数据建模分析。最后使用归一化中和三种模型的学习结果,提取最终的目标特征基因。这也是为什么本方法在多种数据集都有好的结果。
[0084]
一种基于特征基因提取的癌症患者生存状态预测系统,包括:
[0085]
分类模块,基于层次聚类方法对数据集中的数据进行分类,得到分类后的数据;
[0086]
差异分析模块,对分类后的数据进行差异分析,确定差异特征基因;
[0087]
提取模型训练模块,基于特征基因训练lasso模型、ridge模型和randomforestsrc模型并提取交集特征基因;
[0088]
归一化模块,对交集特征基因进行归一化处理并根据归一化结果进行筛选,得到最终特征基因;
[0089]
预测模型训练模块,基于最终特征基因构建训练集并对预测模型进行训练,得到训练完成的预测模型;
[0090]
预测模块,基于训练完成的预测模型进行癌症患者生存状态预测。
[0091]
具体地,还包括:
[0092]
计算模块,根据最终特征基因计算风险评估系数;
[0093]
验证模块,基于km曲线和hr值验证风险评估系数的准确性。
[0094]
上述方法实施例中的内容均适用于本系统实施例中,本系统实施例所具体实现的功能与上述方法实施例相同,并且达到的有益效果与上述方法实施例所达到的有益效果也相同。
[0095]
一种基于特征基因提取的癌症患者生存状态预测装置:
[0096]
至少一个处理器;
[0097]
至少一个存储器,用于存储至少一个程序;
[0098]
当所述至少一个程序被所述至少一个处理器执行,使得所述至少一个处理器实现如上所述一种基于特征基因提取的癌症患者生存状态预测方法。
[0099]
上述方法实施例中的内容均适用于本装置实施例中,本装置实施例所具体实现的功能与上述方法实施例相同,并且达到的有益效果与上述方法实施例所达到的有益效果也相同。
[0100]
一种存储介质,其中存储有处理器可执行的指令,其特征在于:所述处理器可执行的指令在由处理器执行时用于实现如上所述一种基于特征基因提取的癌症患者生存状态预测方法。
[0101]
上述方法实施例中的内容均适用于本存储介质实施例中,本存储介质实施例所具体实现的功能与上述方法实施例相同,并且达到的有益效果与上述方法实施例所达到的有益效果也相同。
[0102]
以上是对本发明的较佳实施进行了具体说明,但本发明创造并不限于所述实施例,熟悉本领域的技术人员在不违背本发明精神的前提下还可做作出种种的等同变形或替换,这些等同的变形或替换均包含在本技术权利要求所限定的范围内。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。