1.本发明涉及基因工程技术领域,特别涉及一种拷贝数变异检测方法、装置、设备和计算机可读介质。
背景技术:
2.拷贝数变异(cnvs)对人类基因组的多样性和许多复杂疾病的发生有重大影响。拷贝数变异的检测和鉴定在生物学和生物医学领域都具有重要意义。新一代测序(ngs)技术为拷贝数变异的检测提供了丰富的数据,并且已经提出了很多基于ngs数据的拷贝数变异检测方法。然而,这些方法在检测低振幅拷贝数变异时,尤其是当拷贝数变异的长度很小时,灵敏度并不可靠。
技术实现要素:
3.为了解决上述现有技术中存在的至少一个技术问题,本发明实施例提供了一种拷贝数变异检测方法、装置、设备和计算机可读介质。所述技术方案如下:
4.第一方面,提供了一种拷贝数变异检测方法,所述方法包括:
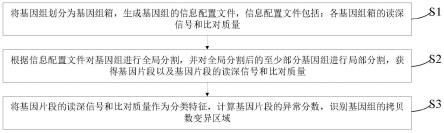
5.将基因组划分为基因组箱,生成所述基因组的信息配置文件,所述信息配置文件包括:各所述基因组箱的读深信号和比对质量;
6.根据所述信息配置文件对所述基因组进行全局分割,并对所述全局分割后的至少部分所述基因组进行局部分割,获得基因片段以及所述基因片段的读深信号和比对质量;
7.将所述基因片段的读深信号和比对质量作为分类特征,计算所述基因片段的异常分数,识别所述基因组的拷贝数变异区域。
8.进一步地,所述将基因组划分为基因组箱,生成所述基因组的信息配置文件,包括:
9.获取所述基因组的测试样本和参考样本;
10.将所述测试样本和所述参考样本进行比对,获得比对结果;
11.根据所述比对结果将所述基因组划分为所述基因组箱;
12.计算所述基因组箱中的读深信号和比对质量,生成所述信息配置文件。
13.进一步地,所述计算所述基因组箱中的读深信号和比对质量,生成所述信息配置文件,包括:
14.计算所述基因组箱中的原始读深信号;
15.对所述原始读深信号进行归一化处理校正。
16.进一步地,所述根据所述信息配置文件对所述基因组进行全局分割,包括:
17.确定一组读深信号连续的所述基因组箱;
18.根据最大统计量,将所述基因组箱的读深信号的平均值与剩余所述基因组箱的读深信号的平均值比较;
19.若比较结果符合变化阈值条件,则确定连续的所述基因组箱中存在所述基因组箱
对应有变化的读深信号,将连续的所述基因组箱划分为一个基因段。
20.进一步地,所述对所述全局分割后至少部分所述基因组进行局部分割,包括:
21.获取预设的分割长度;
22.根据所述分割长度将部分所述基因段划分为多个连续且不重叠的基因片段;
23.计算所述基因片段的读深信号和比对质量。
24.进一步地,在获得所述基因片段后,所述方法还包括:
25.对所述基因片段中的读深信号进行去噪处理。
26.进一步地,所述将所述基因片段的读深信号和比对质量作为分类特征,计算所述基因片段的异常值分数,识别所述基因组的拷贝数变异区域,包括:
27.将所有所述基因片段的读深信号和比对质量表示为一个标准化矩阵,所述标准化矩阵中一个所述基因片段的读深信号和比对质量作为一个数据样本;
28.根据所述标准化矩阵计算协方差矩阵;
29.计算所述协方差矩阵的特征值和特征向量;
30.计算各所述数据样本在所述特征向量上的投影距离作为异常分数;
31.根据所述异常分数以及设定的阈值判定所述数据样本中的异常样本;
32.根据所述异常样本对应的所述基因片段的读深信号确定基线,并声明所述拷贝数变异区域。
33.第二方面,提供了一种拷贝数变异检测装置,所述装置包括:
34.文件生成模块,用于将基因组划分为基因组箱,生成所述基因组的信息配置文件,所述信息配置文件包括:各所述基因组箱的读深信号和比对质量;
35.分段模块,用于根据所述信息配置文件对所述基因组进行全局分割,并对所述全局分割后的至少部分所述基因组进行局部分割,获得基因片段以及所述基因片段的读深信号和比对质量;
36.检测模块,用于将所述基因片段的读深信号和比对质量作为分类特征,计算所述基因片段的异常分数,识别所述基因组的拷贝数变异区域。
37.第三方面,提供了一种电子设备,包括:
38.一个或多个处理器;以及
39.与所述一个或多个处理器关联的存储器,所述存储器用于存储程序指令,所述程序指令在被所述一个或多个处理器读取执行时,执行如第一方面任一所述的方法。
40.第四方面,提供了一种计算机可读介质,其上存储有计算机程序,其中,该程序被处理器执行时实现如第一方面任一所述的方法。
41.本发明实施例提供的技术方案带来的有益效果是:
42.(1)本发明实施例公开的检测方法可提高拷贝数变异检测的敏感性,在检测低幅度拷贝数变异方面有效、可靠;
43.(2)发明实施例公开的检测方法采用了全局和局部分割,有效避免了低幅度拷贝数变异和长度小的拷贝数变异被平滑的问题;
44.(3)发明实施例公开的检测方法可使低的比对质量信号呈现较高的异常值分数,从而可以减少映射错误的干扰。
附图说明
45.为了更清楚地说明本发明实施例中的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
46.图1是本发明实施例提供的拷贝数变异检测方法流程图;
47.图2是本发明实施例提供的拷贝数变异检测装置结构示意图;
48.图3是评估实验中每种方法的精度、灵敏度、f1分数的评估结果图;
49.图4是评估实验中每种方法的wls直方图;
50.图5是方法有效性实验中五种方法检测到的拷贝数变异的分布弦图;
51.图6是方法有效性实验中五种方法检测到的拷贝数变异的数量;
52.图7是本发明实施例提供的电子设备结构示意图。
具体实施方式
53.为使本发明的目的、技术方案和优点更加清楚,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
54.过去,拷贝数变异的检测在很大程度上依赖于微阵列技术。但微阵列技术局限于探针的数量,且它只能检测到设计探针的参考组件中存在的拷贝数变异。近年来,下一代测序(ngs)技术发展迅速并成为主流测序方法。随后,基于序列的结构变异检测方法策略也应运而生。其中,基于读取深度的策略被广泛用于拷贝数变异的检测。其基本思想是,与正常区域相比,拷贝数扩增的区域将获得更高的读取深度,而缺失的区域将具有更低的读取深度。有许多方法是基于这一策略实现的,例如cnvnator、freec、readdepth、grom-rd和最近发布的icopydav、cnv-lof和cnv_iftv。
55.基于读取深度的方法的第一步是对齐基因组坐标中的读取,然后通过计算基因组箱中的平均读取计数获得读深信号。但是读深信号在gc含量较高或较低的区域存在偏差(gc-bias),所以需要根据基因组箱中的gc含量进行归一化处理,这在单样本情况下应用比较广泛。基于读取深度的方法的基本假设是读深信号与区域中的副本数成正比。受序列覆盖率限制和映射误差的干扰,低幅度拷贝数变异(获得不大于2个拷贝或丢失1个拷贝)的信号强度变化较小。同时,较低的肿瘤纯度进一步削弱了信号强度,导致低幅度的拷贝数变异在检测过程中很容易被忽略。
56.去除gc偏差之后进行分割,目标是将具有相似读深信号的相邻基因组箱聚类到同一段中。分割决定了拷贝数变异的长度和位置,流行的分割算法包括圆形二进制分割(cbs)、均值偏移、隐马尔可夫模型(hmm)和逻辑回归。例如,cnvnator使用均值偏移算法进行分割。它根据相邻基因组箱中的读深信号计算每个箱中的均值偏移向量,并根据向量的方向确定分段断点。该方法具有较高的灵敏度和定位精度。freec的分割是通过逻辑回归完成的。之后,通过选择与最大对数似然相对应的等位基因含量来预测基因组的扩增和缺失。它可以估计测序样本的肿瘤纯度,并可以估计预测的拷贝数变异的绝对拷贝数(cn)。
icopydav结合了cbs和总变异最小化(tvm)算法进行分割,弥补了cbs在分割低覆盖序列方面的不足,使其能够以高灵敏度和精度检测更大范围的拷贝数变异。但是,上述分割过程是在整个基因组上进行的(全局分割),并没有考虑局部读取计数的可变性。这导致一些具有微弱信号的局部拷贝数变异被忽略,特别是对于小的拷贝数变异(《6kb)。为了避免这个问题,cnv-lof从局部角度进行分割。它首先将目标基因组划分为多个连续且不重叠的长度相同的区域,然后使用cbs算法对每个子区域进行分割。最后,将异常因子分配给每个基因组片段以识别拷贝数变异区域。该方法对低振幅拷贝数变异显示出高灵敏度,并在低肿瘤纯度数据上表现良好。然而只关注局部区域可能会限制其在具有高肿瘤纯度的测序数据上的性能。
57.本发明实施例为了解决现有技术中存在的问题,提供了一种拷贝数变异检测方法、装置、设备和计算机可读介质,具体技术方案如下:
58.如图1所示,一种拷贝数变异检测方法,包括:
59.s1、将基因组划分为基因组箱,生成基因组的信息配置文件,信息配置文件包括:各基因组箱的读深信号和比对质量。
60.上述,划分基因箱组可根据预设的划分规则划分,例如先对基因组计数,按照计数排序划分基因组箱。读深和比对质量是在适当大小的非重叠基因组箱中计算的,可以减少由噪声信号引起的读取深度的随机波动。读深信号反映了基因组箱中的读取计数。比对质量反映了基因组箱中包含的读数的平均映射质量水平。
61.在一个实施例中,步骤s1包括:
62.获取基因组的测试样本和参考样本;
63.将测试样本和参考样本进行比对,获得比对结果;
64.根据比对结果将基因组划分为基因组箱;
65.计算基因组箱中的读深信号和比对质量,生成信息配置文件。
66.上述,测序样本(fastq格式)和参考序列(例如hg38)的比对生成可生成一个对比文件,例如可通过bwa-mem方法完成对齐,然后由samtools软件排序。
67.将基因组划分为基因组箱可按照固定大小划分,例如,使用bi(i=1,2,3,...,m)来表示第i个基因组箱,m表示基因组箱的总数。每个基因组箱的读深信号可以通过式(1)计算。
[0068][0069]
其中,rdi表示bi的读深信号的值,rcj表示该基因组箱中第j个位置的读取计数,size_bi表示bi的大小,设置为1kb。
[0070]
映射错误的区域呈现较低的值。特别是,当读取不能唯一映射到某个位置时,相关的映射质量为零。因此,较高的映射质量值表示更可靠的对齐。一个基因组箱的映射质量又称比对质量,可以通过公式(2)计算:
[0071][0072]
其中,mqi表示bi的映射质量值,mapgj表示该基因组箱中第j个位置的映射质量。
[0073]
在一个实施例中,计算基因组箱中的读深信号和比对质量,生成信息配置文件,包括:
[0074]
计算基因组箱中的原始读深信号;
[0075]
对原始读深信号进行归一化处理校正。
[0076]
上述,gc偏差(气相色谱偏差)是导致读深信号和序列覆盖率不一致的主要原因之一。读深信号值在gc含量低或高的区域会有偏差。为了得到具有代表性和准确的读深信号,使用一种常用的方法进行校正,公式如下。
[0077][0078]
其中和rdi分别表示第i个基因组箱b的校正读深信号值和原始读深信号值;r
av
g表示所有基因组箱的平均读深信号值,r
gc
表示与bi具有相似gc含量的所有基因组箱的平均读深信号值。
[0079]
s2、根据信息配置文件对基因组进行全局分割,并对全局分割后的至少部分基因组进行局部分割,获得基因片段以及基因片段的读深信号和比对质量。
[0080]
在一个实施例中,全局分割包括:
[0081]
确定一组读深信号连续的基因组箱;
[0082]
根据最大统计量,将基因组箱的读深信号的平均值与剩余基因组箱的读深信号的平均值比较;
[0083]
若比较结果符合变化阈值条件,则确定连续的基因组箱中存在基因组箱对应有变化的读深信号,将连续的基因组箱划分为一个基因段。
[0084]
上述,全局分割以cbs算法为例,在整个基因组上进行分割,将b1,
…
,bm分成许多片段。在每一步中,它确定一组连续的基因组箱bi,b
i 1
,...,bj(1≤i<j≤m)。然后利用最大t统计量,将从bi到bj的读深信号值的平均值与剩余基因组箱的平均值进行比较。如果p值小于阈值(通常为0.01),则bi和bj(如果j《m)可以最大化检验统计量,并被视为变化点的位置。换言之,即将bi到bj的区域划分为一个段。该过程递归地应用于整个基因组并将其分成多个片段。
[0085]
在一个实施例中,局部分割包括:
[0086]
获取预设的分割长度;
[0087]
根据分割长度将部分基因段划分为多个连续且不重叠的基因片段;
[0088]
计算基因片段的读深信号和比对质量。
[0089]
上述,全局分割完成后,对产生的基因片段进一步进行局部分割。在一种情况下,可以预设最终获得的基因片段长度,在全局分割的过程中,若全局分割后获得的基因片段大于预设的基因片段长度,则需要对相应的基因片段进行局部分割。这个过程可以有效地
识别出在大段中被平滑的拷贝数变异,例如低幅度和小的拷贝数变异。首先,指定子段的长度(lrs),它是基因组箱的大小(1kb)的整数倍。然后将长度大于lrs的段划分为多个连续且不重叠的子段。每个子段具有相同的长度(lrs),最后一个可能小于lrs。lrs的大小与拷贝数变异的分辨率相关。通常,较小的lrs将提供更高的检测分辨率和灵敏度,但会导致大量误报事件。较大的lrs将提供更高的精度,但假阴性很难控制。用户可以根据实际需要设置lrs的大小。在我们的研究中,lrs的大小设置为3kb。局部分割完成后,所有片段(局部分割生成的基因片段和未局部分割的片段)按顺序排列并由方程式(4)表示。
[0090]
rs={rs1,rs2,rs3,...,rsn}
ꢀꢀ
(4)
[0091]
其中,rsi表示第i个片段,n表示片段的总数。
[0092]
在一个实施例中,步骤s2还包括:
[0093]
对基因片段中的读深信号进行去噪处理。
[0094]
上述,分割完成后,需要对片段中的读深信号进行平滑以去除噪声。这是因为排序和分割过程中产生的噪声数据可能会导致新的错误。tv算法实现了平滑过程,包含噪声的读深信号表现出出较高的总方差。tv算法通过减少相邻段之间的总方差来恢复原始信号,同时又能很好地保留边缘信息。读深信号的平滑公式如下:
[0095][0096]
其中和分别表示第i个片段的原始读深信号值和平滑后的读深信号值;n表示片段的数量;公式的前一项表示原始rd值与平滑后读深信号值的拟合误差,后一项为总方差的l1范数。λ是该项的惩罚参数,用于调整总方差的约束大小。λ的值越大,惩罚越强。当它趋于无穷大时,所有读深信号值都收敛为相同的值。当λ为0时,则保留原始信号。用户可以指定λ的值。
[0097]
s3、将基因片段的读深信号和比对质量作为分类特征,计算基因片段的异常分数,识别基因组的拷贝数变异区域。
[0098]
上述,计算各基因片段的机场分数可采用主成分分类器算法(pcc),读深信号和比对质量作为分类算法的两个特征。
[0099]
在一个实施例中,步骤s3包括:
[0100]
s31、将所述基因片段的读深信号和比对质量表示为一个标准化矩阵,所述标准化矩阵中一个所述基因片段的读深信号和比对质量作为一个数据样本;
[0101]
s32、根据所述标准化矩阵计算协方差矩阵;
[0102]
s33、计算所述协方差矩阵的特征值和特征向量;
[0103]
s34、计算各所述数据样本在所述特征向量上的投影距离作为异常分数;
[0104]
s35、根据所述异常分数以及设定的阈值判定所述数据样本中的异常样本;
[0105]
s36、根据所述异常样本对应的所述基因片段的读深信号确定基线,并声明所述拷贝数变异区域。
[0106]
上述,举例说明:
[0107]
步骤s31中,读深信号和比对质量可以表示为向量r和m,即r=[r1,r2,...,rn]和m=[m1,m2,...,mn],其中n表示片段数量,ri和mi分别表示第i个段的读深信号值和比对质量
值,它们是该段中相应信号的平均值。这两个特征可以用矩阵n表示,每个列向量(ri,mi)
t
表示为一个样本。
[0108][0109]
上述,标准化矩阵n,用矩阵x表示。
[0110]
步骤s32和s33中,计算协方差矩阵:
[0111]
求解c的特征值-特征向量对:(λ1,e1)和(λ2,e2),λ1≥λ2;
[0112]
步骤s34中,计算每个数据样本xi在e1上的投影距离d作为异常分数:
[0113][0114]
步骤s35中,使用otsu算法设置阈值t,当score(xi)》t时判定为异常样本。
[0115]
步骤s36中,根据读深信号确定基线并声明拷贝数变异区域。
[0116]
上述,步骤s3采用的是主成分分类法,主成分分类器(pcc)建立在主成分分析(pca)之上,pca是一种常用于高维数据降维的算法。pca的主要原理是将原始高维数据通过线性变换投影到某个低维空间上,并使其方差尽可能大,从而最大限度地保留数据的有效信息。pca已作为一种数据校正技术应用于拷贝数变异检测问题之中,而不是作为确认拷贝数变异区域的主要方法。步骤s31-s36主要目标是将二维矩阵n投影到一维向量v上,并根据投影距离找到异常样本。
[0117]
在步骤s31中,将两个特征r和m归一化到相同的尺度。这是因为比对质量值一般大于读取信号的值,当投影到低维空间时,比对质量会在主成分中获得更大的权重。使用以下公式对这两特征进行标准化:
[0118][0119][0120]
其中r
′
表示标准化后的读深信号,和r
sd
分别表示读深信号的平均值和标准差。比对质量的归一化过程与读深信号相同,如方程式(8)所示。标准化后,每个特征的均值变为0,标准差变为1。这保证了两个特征对主成分变量的影响相同。
[0121]
在步骤s33中,协方差矩阵c可以分解为与特征值相关联的正交向量,称为特征向量。特征向量反映了样本数据方差变化的不同方向,特征值表示数据在相应方向上的方差大小。具有高特征值的特征向量e1捕获了数据中的大部分方差,并作为主成分向量。
[0122]
步骤s34中,读深信号是识别拷贝数变异的主要特征,因此只需要计算样本到e1的投影距离。离群值分数是每个样本到特征向量e1之间的加权欧几里得距离。具有较大异常值的样本表示潜在的拷贝数变异或映射错误区域。
[0123]
在步骤s35中,设置阈值以确定异常样本。对于不同序列覆盖的样本数据,投影到
e1上的距离差异很大。为了适应具有不同序列覆盖率的数据,我们使用otsu算法来计算阈值。otsu是一种全局二值分割算法,主要用于灰度图的分割。获得的最佳阈值可以使所得灰度级的可分离性最大化。它通过遍历一个区间内的所有分数来动态获取一个阈值,以最大化两个类之间的方差。在这一步中,我们首先将异常值分数转换为具有两位小数的浮点数。然后,我们遍历下35%分位数和上85%分位数之间的异常值分数,每次以0.01为增量找到最佳阈值t。分数高于t(score(xi)≥t)的样本被认为是异常样本。
[0124]
在步骤s36中,基线定义为去除异常样本后剩余样本的平均读深信号值(允许15%的误差)。读深信号值高于该基线的异常样本被识别为拷贝数增加,低于基线被认为是丢失。
[0125]
基于上述本发明实施例公开的拷贝数变异检测方法,如图2所示,本发明实施例还提供一种贝数变异检测装置,包括:
[0126]
文件生成模块201,用于将基因组划分为基因组箱,生成基因组的信息配置文件,信息配置文件包括:各基因组箱的读深信号和比对质量;
[0127]
分段模块202,用于根据信息配置文件对基因组进行全局分割,并对全局分割后的至少部分基因组进行局部分割,获得基因片段以及基因片段的读深信号和比对质量;
[0128]
检测模块203,用于将基因片段的读深信号和比对质量作为分类特征,计算基因片段的异常分数,识别基因组的拷贝数变异区域。
[0129]
进一步地,文件生成模块201,包括:
[0130]
输入模块,用于获取基因组的测试样本和参考样本;
[0131]
比对模块,用于将测试样本和参考样本进行比对,获得比对结果;
[0132]
分箱模块,用于根据比对结果将基因组划分为基因组箱;
[0133]
计算模块,用于计算基因组箱中的读深信号和比对质量,生成信息配置文件。
[0134]
进一步地,计算模块,具体用于:
[0135]
计算基因组箱中的原始读深信号;
[0136]
对原始读深信号进行归一化处理校正。
[0137]
进一步地,分段模块202,包括:
[0138]
全局分割模块,用于:
[0139]
确定一组读深信号连续的基因组箱;
[0140]
根据最大统计量,将基因组箱的读深信号的平均值与剩余基因组箱的读深信号的平均值比较;
[0141]
若比较结果符合变化阈值条件,则确定连续的基因组箱中存在基因组箱对应有变化的读深信号,将连续的基因组箱划分为一个基因段。
[0142]
进一步地,分段模块202,包括:
[0143]
局部分割模块,用于:
[0144]
获取预设的分割长度;
[0145]
根据分割长度将部分基因段划分为多个连续且不重叠的基因片段;
[0146]
计算基因片段的读深信号和比对质量。
[0147]
进一步地,分段模块202还包括:
[0148]
去噪模块,用于对基因片段中的读深信号进行去噪处理。
[0149]
进一步地,检测模块203,具体用于:
[0150]
将基因片段的读深信号和比对质量表示为一个标准化矩阵,标准化矩阵中一个基因片段的读深信号和比对质量作为一个数据样本;
[0151]
根据标准化矩阵计算协方差矩阵;
[0152]
计算协方差矩阵的特征值和特征向量;
[0153]
计算各数据样本在特征向量上的投影距离作为异常分数;
[0154]
根据异常分数以及设定的阈值判定数据样本中的异常样本;
[0155]
根据异常样本对应的基因片段的读深信号确定基线,并声明拷贝数变异区域。
[0156]
下面为了进一步说明本发明公开的技术方案的有益效果,对本发明公开的拷贝数变异检测的技术方案进行性能评估:
[0157]
首先在模拟数据上建立一个比较实验。模拟数据所具有的基本事实保证了评估的可靠性。将本发明公开的拷贝数变异检测方法(cnv-pcc)与五种流行的方法(cnvnator、freec、grom-rd、cnv-lof和cnv_iftv)在精度、灵敏度和f1分数方面进行了比较。为了确保实验的公平性,调整了某些方法的基因组箱的大小,以便它们可以检测到小的拷贝数变异。例如,cnvnator的基因组箱大小设置为推荐值(30倍覆盖度的设置为250bp,20倍覆盖度的为130bp,10倍覆盖度的为90bp),freec的基因组箱的大小设置为1kb。其余方法使用它们的默认参数。随后,真实样本用于验证cnv-pcc的有效性,它包括来自千人基因组计划的三个样本。
[0158]
利用样本数据对上述检测方法进行模拟。综合仿真软件sinc以及序列处理工具seqtk用于生成仿真数据集。所有模拟数据均基于参考基因组hg38中的21号染色体生成。覆盖度设置为10x、20x和30x,肿瘤纯度设置为0.4、0.5和0.6,每种配置模拟30个重复样本。拷贝数变异的长度限制在1kb到6kb,因为大多数方法在检测大型拷贝数变异时表现良好。同时,不考虑高振幅拷贝数变异(纯合缺失和拷贝增益》4),因为它们也很容易检测到。每次模拟复制共产生24个拷贝数变异,包括15个扩增和9个缺失。扩增的拷贝数数量为3和4,所有缺失均为半合子缺失。将cnv-pcc与五种方法在生成的模拟数据集上进行比较。如果被声明的拷贝数变异覆盖了真实拷贝数变异的50%的区域,则将其视为一个真阳性事件。评估中使用精度、灵敏度和f1分数作为指标,结果如图3所示。图中,评估指标的每个值是每个配置下30次模拟复制的平均值。图3显示cnv-pcc、cnv_iftv、freec和cnv-lof获得了更好的检测结果,而cnvnator和grom-rd在大多数模拟数据中性能较差。cnv-pcc在每个数据集中获得最高的灵敏度,这表明它检测到的拷贝数变异数量最多。除了第一个数据集外,它还具有最高的f1分数。当肿瘤纯度低于0.6时,cnv-lof在10x数据上的灵敏度排名第二,并且在肿瘤纯度为0.4时具有最大的f1分数。这反映了它对低纯度数据的有效性。其余依次为cnv_iftv、freec、grom-rd和cnvnator。随着覆盖范围的增加,cnv_iftv的灵敏度和f1分数超过了cnv-lof,位居第二。它在高覆盖度数据上表现出色。cnvnator和grom-rd的灵敏度和f1分数始终较低,即使在高覆盖率的数据上也是如此。这表明这两种方法不适用于低纯度数据的检测。当肿瘤纯度上升到0.6时,freec的灵敏度和f1分数显着增加。然而,cnv-lof的灵敏度和f1分数在20x和30x数据上有所下降。这一事实表明,仅依靠局部分割可能会限制其在具有高肿瘤纯度的数据上的性能。在精度方面,freec在10x数据上具有最大的价值,它产生了可靠的结果。随着序列覆盖率的增加,cnv-lof、cnv_iftv、cnv-pcc在30x数据上的精度超
过freec。这是因为读深信号的强度随着序列覆盖的增加而增加,从而减少了假阳性结果的产生。cnvnator和grom-rd的精度一直较低,这两种方法都会产生大量的假阳性结果。
[0159]
综合来看,cnv-pcc在所有模拟数据上都表现良好,并且优于其他五种方法。cnv_iftv也表现出较好的性能。cnv-lof适用于肿瘤纯度较低的cnv检测,结果稳定,但肿瘤纯度较高时效果下降。相比之下,freec适用于具有高肿瘤纯度的数据。除了肿瘤纯度高的条件外,cnvnator还需要高覆盖度。grom-rd在所有模拟数据上表现不佳,表明它不适合检测此类拷贝数变异。
[0160]
为了进一步评估这六种方法的有效性,设计了方程式(9)计算每种方法的加权长度得分(wls)。每种方法的wls直方图如图4所示。
[0161]
wls=w
×
lv
ꢀꢀ
(9)
[0162]
其中w表示加权值,它是一种方法识别出的真实拷贝数变异的长度与该方法检测到的所有的拷贝数变异的长度之比。lv表示该方法检测到的真实拷贝数变异的长度。wls可以间接反映已识别拷贝数变异的准确度水平。例如,当肿瘤纯度为0.6时,freec和cnv_iftv在10x数据上的灵敏度几乎相同,但图4显示freec的wls高于cnv_iftv。这很可能是因为iftv的断点偏差大于freec。在肿瘤纯度为0.4的30x数据上比较pcc和iftv之间的wls也可以发现相同的情况。当覆盖率为30x时,cnvnator的wls在肿瘤纯度为0.6的数据上排名第二。cnvnator超过了敏度比它更高的cnv_iftv和freec。这表明cnvnator具有较小的断点偏差。cnv-pcc在所有模拟数据上保持最高的wls,表现出强大的性能。
[0163]
在真实数据方面,从千人基因组计划中选取约鲁巴家族三人组的测序样本来检验cnv-pcc的有效性。它包括na19238(母亲)、na19239(父亲)和na19240(女儿)。cnv-pcc应用于每个样本的全基因组,并与五种现有方法(cnvnator、freec、grom-rd、cnv-lof、cnv_iftv)进行比较。弦图(图5)显示了五种方法检测到的拷贝数变异的分布(以kb为单位)。圆圈的上半部分有22个扇区,表示从第1到第22个常染色体。每个扇区的宽度代表在该染色体上检测到的cnv的数量。圆圈的下半部分分为五个扇区,每个扇区表示一种方法:cnv-lof(绿色)、cnvnator(红色)、cnv-pcc(紫色)、freec(黑色)、cnv_iftv(蓝色)和grom-rd(粉色的)。扇区的宽度表示该方法检测到的拷贝数变异的总数。观察到cnv-lof检测到的拷贝数变异数量最多,它检测到许多染色体上的大规模拷贝数变异区域。其余依次为cnvnator、cnv-pcc、freec、cnv_iftv和grom-rd。为便于查看检测结果,本技术图5采用彩色显示,灰度处理会大大影响显示效果。
[0164]
为了进一步分析这六种方法在真实样本上的分布情况,在图6中,展示了每种方法在三个样本上检测到的拷贝数变异的数量,以及两种方法之间重叠的拷贝数变异的数量。发现cnv-pcc的平均重叠拷贝数变异数量最多,这表明cnv-pcc与其他方法具有很高的一致性。紧随其后的是cnvnator和freec,这两种方法之间重叠的拷贝数变异数量很高。cnv-lof在每个样本中检测到的拷贝数变异数量最多,但与其他方法重叠的拷贝数变异数量较少。grom-rd检测到的拷贝数变异最少。
[0165]
由于真实样本缺乏像模拟数据中那样的完整基本事实,为了进行合理的评估,进一步计算了每种方法的重叠密度得分(ods)作为可靠的衡量标准。ods的计算公式如下:
[0166][0167]
其中lm表示一种方法与其他方法的平均重叠长度,l
sum
表示该方法检测到的拷贝数变异的总长度。不同方法之间的重叠区域被认为是真正的阳性结果。公式的前一项可视为灵敏度,后一项可视为精度。具有较高ods的方法具有更好的性能。五种方法的ods值如表1所示。
[0168]
表1
[0169] cnvnatorfreecgrom-rdcnv-lofcnv_iftvcnv-pccna192381577215469914135381046816044na1923817291156491101825571095516391na192381265816878912535411399617919
[0170]
从表1可以看出cnv-pcc在na19238和na19240中ods最大,cnvnator在na19239中ods最大。对于三个样本的平均ods,cnv-pcc有着最大值,其余依次为freec、cnvnator、cnv_iftv、grom-rd、cnv-lof。因此,可以得出结论,提出的方法在实际数据的应用中是有效且可靠的。
[0171]
另外本发明实施例还提供了一种电子设备,包括:
[0172]
一个或多个处理器;以及
[0173]
与所述一个或多个处理器关联的存储器,所述存储器用于存储程序指令,所述程序指令在被所述一个或多个处理器读取执行时,执行上述实施例公开的光信息编码方法。
[0174]
其中,如图7所示,计算机设备12以通用计算设备的形式表现。计算机设备12的组件可以包括但不限于:一个或者多个处理器或者处理单元16,系统存储器28,连接不同系统组件(包括系统存储器28和处理单元16)的总线18。总线18表示几类总线结构中的一种或多种,包括存储器总线或者存储器控制器,外围总线,图形加速端口,处理器或者使用多种总线结构中的任意总线结构的局域总线。举例来说,这些体系结构包括但不限于工业标准体系结构(isa)总线,微通道体系结构(mac)总线,增强型isa总线、视频电子标准协会(vesa)局域总线以及外围组件互连(pci)总线。
[0175]
计算机设备12典型地包括多种计算机系统可读介质。这些介质可以是任何能够被计算机设备12访问的可用介质,包括易失性和非易失性介质,可移动的和不可移动的介质。
[0176]
系统存储器28可以包括易失性存储器形式的计算机系统可读介质,例如随机存取存储器(ram)30和/或高速缓存存储器32。计算机设备12可以进一步包括其它可移动/不可移动的、易失性/非易失性计算机系统存储介质。仅作为举例,存储系统34可以用于读写不可移动的、非易失性磁介质(图中未显示,通常称为“硬盘驱动器”)。尽管图中未示出,可以提供用于对可移动非易失性磁盘(例如“软盘”)读写的磁盘驱动器,以及对可移动非易失性光盘(例如cd-rom,dvd-rom或者其它光介质)读写的光盘驱动器。在这些情况下,每个驱动器可以通过一个或者多个数据介质接口与总线18相连。存储器28可以包括至少一个程序产品,该程序产品具有一组(例如至少一个)程序模块,这些程序模块被配置以执行本发明各实施例的功能。
[0177]
具有一组(至少一个)程序模块42的程序/实用工具40,可以存储在例如存储器28
中,这样的程序模块42包括—但不限于—操作系统、一个或者多个应用程序、其它程序模块以及程序数据,这些示例中的每一个或某种组合中可能包括网络环境的实现。程序模块42通常执行本发明所描述的实施例中的功能和/或方法。
[0178]
计算机设备12也可以与一个或多个外部设备14(例如键盘、指向设备、显示器24等)通信,还可与一个或者多个使得用户能与该计算机设备12交互的设备通信,和/或与使得该计算机设备12能与一个或多个其它计算设备进行通信的任何设备(例如网卡,调制解调器等等)通信。这种通信可以通过输入/输出(i/o)接口22进行。另外,本实施例中的计算机设备12,显示器24不是作为独立个体存在,而是嵌入镜面中,在显示器24的显示面不予显示时,显示器24的显示面与镜面从视觉上融为一体。并且,计算机设备12还可以通过网络适配器20与一个或者多个网络(例如局域网(lan),广域网(wan)和/或公共网络,例如因特网)通信。如图所示,网络适配器20通过总线18与计算机设备12的其它模块通信。应当明白,尽管图中未示出,可以结合计算机设备12使用其它硬件和/或软件模块,包括但不限于:微代码、设备驱动器、冗余处理单元、外部磁盘驱动阵列、raid系统、磁带驱动器以及数据备份存储系统等。
[0179]
处理单元16通过运行存储在系统存储器28中的程序,从而执行各种功能应用以及数据处理。
[0180]
本说明书中的各个实施例均采用递进的方式描述,各个实施例之间相同相似的部分互相参见即可,每个实施例重点说明的都是与其他实施例的不同之处。尤其,对于系统或系统实施例而言,由于其基本相似于方法实施例,所以描述得比较简单,相关之处参见方法实施例的部分说明即可。以上所描述的系统及系统实施例仅仅是示意性的,其中所述作为分离部件说明的单元可以是或者也可以不是物理上分开的,作为单元显示的部件可以是或者也可以不是物理单元,即可以位于一个地方,或者也可以分布到多个网络单元上。可以根据实际的需要选择其中的部分或者全部模块来实现本实施例方案的目的。本领域普通技术人员在不付出创造性劳动的情况下,即可以理解并实施。
[0181]
以上对本技术所提供的技术方案进行了详细介绍,本文中应用了具体个例对本技术的原理及实施方式进行了阐述,以上实施例的说明只是用于帮助理解本技术的方法及其核心思想;同时,对于本领域的一般技术人员,依据本技术的思想,在具体实施方式及应用范围上均会有改变之处。综上所述,本说明书内容不应理解为对本技术的限制。
[0182]
上述所有可选技术方案,可以采用任意结合形成本发明的可选实施例,在此不再一一赘述。
[0183]
以上所述仅为本发明的较佳实施例,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。
