一种基于图注意力的疾病相关环状rna识别方法
技术领域
1.本发明涉及生物信息工程领域,特别涉及一种基于图注意力的疾病相关环状rna识别方法。
背景技术:
2.在内源性非编码rna研究邻域中,一个新的被研究的明星是circrna,因其具有单链的环状封闭结构引起了许多研究者的兴趣。circrna早在40年前就在植物样病毒中被发现。由于环状rna分子的丰度较低,且生物学功能未知,因此它们通常被认为是rna异常剪接的副产物。随着高通量测序技术和生物信息研究的深入发现环状rna除了作为mirna(一种常见的非编码rna)海绵和影响rna的剪接和转录,还可以通过独立翻译蛋白质来调节生命活动。
3.新出现的研究发现,circrna可以促进许多主要疾病的发生,如癌症、眼部疾病和神经退行性疾病。因此,探索circrna与疾病之间的潜在关联,有助于生物学家深入研究人类疾病的复杂致病机制,进一步促进疾病预防、疾病诊断和疾病治疗药物的研发。然而,通过传统的生物实验来发现circrna-疾病的潜在关联是费时、费力和消耗大量金钱的。基于已经被生物实验验证的circrna与疾病关联信息,可以采用计算预测的方法探寻circrna与疾病的相互作用关系,这将有助于研究人员发现circrna与疾病潜在关联的效率。
4.现存的方法大都基于传统机器学习和网络信息传播的方法去预测潜在的circrna与疾病的关联,但是这些方法都有一个明显的缺点,当出现一个新的circrna或者疾病时,需要重新计算预测方法中的相似性矩阵再进行后续步骤,这就造成“冷启动”问题,无疑将降低预测的效率。
技术实现要素:
5.为了解决上述技术问题,本发明提供一种算法简单、可靠性高、准确性强的基于图注意力的疾病相关环状rna识别方法。
6.本发明解决上述技术问题的技术方案是:一种基于图注意力的疾病相关环状rna识别方法,包括以下步骤:
7.步骤一:采用整合的cirrna相似性矩阵构建circrna-circrna关系子图,采用整合的疾病相似性矩阵构建疾病与疾病关系子图;
8.步骤二:利用构建的circrna-circrna关系子图、疾病与疾病关系子图以及已知的circrna-疾病关系图构建circrna与疾病的异构图;
9.步骤三:利用多头动态注意力机制学习异构图上每个节点特征的不同聚合表达,在此基础上利用具有不同大小卷积核的单层卷积网络从节点特征的不同聚合表达中提取具有信息的高阶特征;
10.步骤四:利用广义矩阵分解交互节点的高阶特征以表达异构图上节点之间的复杂关系;最后利用多层感知机网络学习节点之间的特征交互以达到预测circrna节点与疾病
节点之间潜在关系的目的。
11.上述基于图注意力的疾病相关环状rna识别方法,所述步骤一具体包括如下步骤,
12.1-1)由circrna高斯核相似性、circrna序列相似性和circrna功能相似性整合得到circrna相似性矩阵,基于cirrna相似性矩阵构建circrna-circrna关系子图;
13.1-2)由疾病高斯核相似性和疾病语义相似性整合得到疾病相似性矩阵,基于疾病相似性矩阵构建疾病与疾病关系子图。
14.上述基于图注意力的疾病相关环状rna识别方法,所述步骤1-1)具体过程为:
15.1-1-1)将circrna的序列信息视为字符串,用levenshtein距离算法计算两个字符串之间相互转换的编辑距离,编辑距离越短,两个字符串越相似,其计算遵循以下公式:
[0016][0017]
dist表示编辑的距离,seqlen(
·
)表示circrna的序列信息长度,ci表示第i个circrna,sc表示circrna与circrna之间的序列相似性矩阵,sc(ci,cj)表示ci与cj之间的序列相似性;
[0018]
1-1-2)基于circrna与相同疾病关联越多就越相似的原则,计算circrna-circrna之间的功能相似性,计算的公式如下:
[0019][0020]gi
与gj分别表示与ci和cj相关的疾病集合,||表示构成集合的circrna或者疾病的个数,sd(d,gn)表示疾病d与gn集合内疾病的相似性分数,fs表示circrna与circrna之间的功能相似性矩阵,fs(ci,cj)表示ci与cj之间的功能相似性;
[0021]
1-1-3)考虑circrna与疾病的已知相互作用关系,计算表示circrna与circrna之间线性关系的高斯核相似性,具体计算公式如下:
[0022]
kc(ci,cj)=exp(-γc||a
i.-a
j.
||2)(3)
[0023]
其中,kc表示circrna与circrna之间的高斯核相似性矩阵,kc(ci,cj)表示ci与cj之间的高斯核相似性,a
i.
与a
j.
分别表示circrna与疾病关联矩阵a的第i行和第j行,γc表示计算circrna高斯核相似性的宽度控制参数,用公式表示为:
[0024][0025]
nc表示circrna的个数;
[0026]
1-1-4)整合circrna的相似性矩阵构建circrna-circra关系子图,用如下公式整合相似矩阵:
[0027][0028]
rcs表示整合的circrna的相似性矩阵,rcs(ci,cj)表示ci与cj之间的相似性,假设circrna与circrna之间的相似性大于0.5,则认为两者之间具有强关联关系;构建circrna
子图的邻接矩阵mc,具体表示为:
[0029][0030]
上述基于图注意力的疾病相关环状rna识别方法,所述步骤1-2)具体过程为:
[0031]
1-2-1)基于疾病本体数据库diseaseontology中的doid信息计算疾病与疾病之间的语义相似性,计算的公式为:
[0032][0033]
其中sd表示疾病与疾病之间的语义相似性矩阵,sd(dm,dn)表示疾病dm与dn之间的语义相似性,表示疾病dm的祖先,且包括dm自身;d
′
表示dm和dn都存在关系的疾病,dv(dm)表示疾病dm的在祖先中的语义值,表示为:
[0034][0035]
其中d为疾病,dd(d
′
)表示疾病d
′
对疾病d的贡献值;公式(7)中表示疾病d
′
对疾病dm的贡献值,其具体计算为:
[0036][0037]
1-2-2)考虑到circrna与疾病的已知相互作用关系,计算表示疾病与疾病之间线性关系的高斯核相似性,具体计算公式如下:
[0038]
kd(dm,dn)=exp(-γc||a
.m-a
.n
||2)(10)
[0039]
其中,kd表示疾病与疾病之间的高斯核相似性矩阵,kd(dm,dn)表示疾病dm与dn之间的高斯核相似性,a
.m
与a
.n
分别表示circrna与疾病关联矩阵a的第m列和第n列,γd表示计算疾病高斯核相似性的宽度控制参数,用公式表示为:
[0040][0041]
nd表示疾病的个数;
[0042]
1-2-3)整合疾病的相似性矩阵,构建疾病关系子图,整合疾病相似性矩阵rds的具体公式如下:
[0043][0044]
构建疾病子图的邻接矩阵,同样假设疾病与疾病之间的相似性大于0.5,则认为两者之间具有强关联关系,疾病子图的邻接矩阵具体表示为:
[0045]
[0046]
上述基于图注意力的疾病相关环状rna识别方法,所述步骤二的具体过程为:
[0047]
利用已知的circrna-疾病关系图、circrna-circrna关系子图和疾病与疾病关系子图构建circrna与疾病的异构图;按照如下方法先构建异构图的邻接矩阵,再利用python中的dgl库构建异构图g;
[0048]
构建异构图邻接矩阵h的公式为:
[0049][0050]
异构图g中点的特征矩阵x为:
[0051][0052]
其中,a表示已知circrna与疾病构成的关联矩阵,a矩阵的行代表circrna,列代表疾病,如果第c
p
个circrna和第dq个疾病存在关联,p=1,2,
…
,nc;q=1,2,
…
,nd,则a中位于第c
p
行第dq列的值否则否则和为需要学习的特征投影矩阵,表示实数范围内取值,nc×
k表示wc的大小,nd×
k表示wd的大小,k为投影之后特征的维度。
[0053]
上述基于图注意力的疾病相关环状rna识别方法,所述步骤三的具体过程为:
[0054]
利用多头动态图注意力机制获取异构图上节点特征的不同聚合表示,动态注意力机制的核心思想是聚合节点不同邻居的特征时,给邻居分配不同的权重;
[0055]
异构图g中以circrna c
p
作为节点,以疾病dq作为c
p
的邻居节点,首先定义一个分数函数来计算异构图g上从节点c
p
到邻居节点dq的注意力权重分数
[0056][0057]
其中||表示向量的拼接,和分别表示异构图g中点的特征矩阵x中的第c
p
行和第dq行,a表示单层前馈神经网络,a
t
表示的a转置,leakyrelu()表示神经网络中的激活函数;使用softmax函数对邻居的注意权重分数进行归一化,得到关注邻居节点的注意力值
[0058][0059]
nc表示图上节点c
p
的邻居集合,则节点c
p
的邻居节点特征聚合表示为:
[0060][0061]
其中,σ()表示神经网络中的激活函数relu();另外,使用单一的注意机制来聚合节点的邻域特征并不足以完全表示节点之间的关系,因此,将多个注意力头输出的特征表示进行聚合,得到节点c更丰富的特征表示,表述如下:
[0062][0063]
k是注意力头的个数,表示第k注意头所聚合的节点特征表示,表示节点c
p
提取了多头注意力聚合邻居特征的高阶特征表示,f(
·
)表示一个特征提取的函数,计算的f(
·
)共分为三个步骤;
[0064]
第一步,通过多头注意力机制获得的节点c
p
特征的不同聚合表示被垂直地堆叠起来:
[0065][0066]
表示节点c
p
的特征堆叠矩阵;
[0067]
第二步,利用具有不同大小卷积核的单层卷积网络从特征堆叠矩阵中提取具有丰富信息的高阶特征,第l个卷积核的特征提取公式如下:
[0068][0069]
■
表示卷积操作,表示第l个卷积核参数矩阵,表示偏置项,表示节点c
p
的不同特征聚合表达堆叠矩阵经过第l个卷积核所提取出来的特征向量,ψ()表示神经网络中的激活函数relu();
[0070]
第三步,将由不同卷积核得到的特征向量拼接在一起,形成具有丰富信息的高阶特征表示,用公式表示如下:
[0071][0072]
表示节点c
p
提取了多头注意力聚合邻居特征的高阶特征表示,为公式(19)的输出表示;
[0073]
因此,异构图中点的特征矩阵x进一步用x
′
表示:
[0074][0075]
由公式(23)可知,表示x
′
的第(nc nd)行,x
′
[1:nc]和x
′
[nc 1:nc nd]分别表示异构图中circrna和疾病的高阶特征信息。
[0076]
上述基于图注意力的疾病相关环状rna识别方法,所述步骤四中,用广义矩阵分解来表示circrna与疾病之间复杂的高阶非线性特征交互;广义矩阵分解的定义如下:
[0077][0078]
其中,
⊙
表示哈达玛乘积,和分别表示x
′
中的第c
p
行和第dq行,p
pairwise
(c
p
,dq)表示circrna c
p
与疾病dq的高阶特征交互;
[0079]
利用多层感知器网络学习节点之间的特征交互被表示为:
[0080][0081]
公式(25)中的w,w2…wl
和ε1,ε2,
…
ε
l
均为需要训练的参数矩阵,表示circrna c
p
与疾病dq的关联预测得分,模型是通过用l2正则化来最小化二元交叉熵损失来训练的,通过下面的损失函数公式来计算:
[0082][0083]
其中,a已知的circrna与疾病的关联矩阵,表示a中位于第c
p
行第dq列的值,表示预测的circrna与疾病的关联矩阵,表示中位于第c
p
行第dq列的值,||θ||2表示l2正则化,l2正则化的参数矩阵为θ,s
为训练用的正样本,用来表示已知的circrna与疾病的关联;s-为训练用的负样本,用来表示未知的circrna与疾病的关联。
[0084]
本发明的有益效果在于:本发明利用图注意力网络中的多头动态注意力机制高效聚合异构图上邻居特征形成特征的不同聚合表示,再利用具有大小不同的单层卷积网络提取出高阶特征,最后通过多层感知机网络预测潜在circrna与疾病的关联。本发明具有可靠性高、准确性强等优点,在生物数据库、生物数据分析、生物数据挖掘、疾病治疗药物的靶向研发的应用及普及上有着广泛的市场前景。
附图说明
[0085]
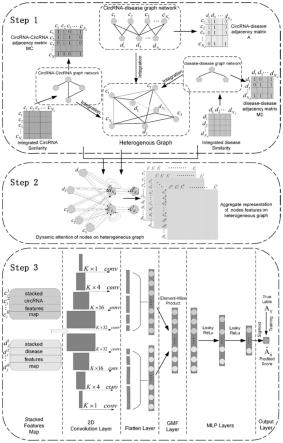
图1为本发明的流程图。
具体实施方式
[0086]
下面结合附图和实施例对本发明作进一步的说明。
[0087]
如图1所示,一种基于图注意力的疾病相关环状rna识别方法,包括以下步骤:
[0088]
步骤一:采用整合的cirrna相似性矩阵构建circrna-circrna关系子图,采用整合的疾病相似性矩阵构建疾病与疾病关系子图。
[0089]
步骤一具体包括如下步骤,
[0090]
1-1)由circrna高斯核相似性、circrna序列相似性和circrna功能相似性整合得到circrna相似性矩阵,基于cirrna相似性矩阵构建circrna-circrna关系子图。
[0091]
1-1)具体过程为:
[0092]
1-1-1)将circrna的序列信息视为字符串,用levenshtein距离算法计算两个字符串之间相互转换的编辑距离,编辑距离越短,两个字符串越相似,其计算遵循以下公式:
[0093][0094]
dist表示编辑的距离,seqlen(
·
)表示circrna的序列信息长度,ci表示第i个circrna,sc表示circrna与circrna之间的序列相似性矩阵,sc(ci,cj)表示ci与cj之间的序列相似性;
[0095]
1-1-2)基于circrna与相同疾病关联越多就越相似的原则,计算circrna-circrna之间的功能相似性,计算的公式如下:
[0096][0097]gi
与gj分别表示与ci和cj相关的疾病集合,||表示构成集合的circrna或者疾病的个数,sd(d,gn)表示疾病d与gn集合内疾病的相似性分数,fs表示circrna与circrna之间的功能相似性矩阵,fs(ci,cj)表示ci与cj之间的功能相似性;
[0098]
1-1-3)考虑circrna与疾病的已知相互作用关系,计算表示circrna与circrna之间线性关系的高斯核相似性,具体计算公式如下:
[0099]
kc(ci,cj)=exp(-γc||a
i.-a
j.
||2)(3)
[0100]
其中,kc表示circrna与circrna之间的高斯核相似性矩阵,kc(ci,cj)表示ci与cj之间的高斯核相似性,a
i.
与a
j.
分别表示circrna与疾病关联矩阵a的第i行和第j行,γc表示计算circrna高斯核相似性的宽度控制参数,用公式表示为:
[0101][0102]
nc表示circrna的个数;
[0103]
1-1-4)整合circrna的相似性矩阵构建circrna-circra关系子图,用如下公式整合相似矩阵:
[0104][0105]
rcs表示整合的circrna的相似性矩阵,rcs(ci,cj)表示ci与cj之间的相似性,假设circrna与circrna之间的相似性大于0.5,则认为两者之间具有强关联关系;构建circrna子图的邻接矩阵mc,具体表示为:
[0106][0107]
1-2)由疾病高斯核相似性和疾病语义相似性整合得到疾病相似性矩阵,基于疾病相似性矩阵构建疾病与疾病关系子图。
[0108]
1-2)具体步骤为:
[0109]
1-2-1)基于疾病本体数据库diseaseontology中的doid信息计算疾病与疾病之间的语义相似性,计算的公式为:
[0110][0111]
其中sd表示疾病与疾病之间的语义相似性矩阵,sd(dm,dn)表示疾病dm与dn之间的语义相似性,表示疾病dm的祖先,且包括dm自身;d
′
表示dm和dn都存在关系的疾病,dv(dm)表示疾病dm的在祖先中的语义值,表示为:
[0112][0113]
其中d为疾病,dd(d
′
)表示疾病d
′
对疾病d的贡献值;公式(7)中表示疾病d
′
对疾病dm的贡献值,其具体计算为:
[0114][0115]
1-2-2)考虑到circrna与疾病的已知相互作用关系,计算表示疾病与疾病之间线性关系的高斯核相似性,具体计算公式如下:
[0116]
kd(dm,dn)=exp(-γc||a
.m-a
.n
||2)(10)
[0117]
其中,kd表示疾病与疾病之间的高斯核相似性矩阵,kd(dm,dn)表示疾病dm与dn之间的高斯核相似性,a
.m
与a
.n
分别表示circrna与疾病关联矩阵a的第m列和第n列,γd表示计算疾病高斯核相似性的宽度控制参数,用公式表示为:
[0118][0119]
nd表示疾病的个数;
[0120]
1-2-3)整合疾病的相似性矩阵,构建疾病关系子图,整合疾病相似性矩阵rds的具体公式如下:
[0121][0122]
构建疾病子图的邻接矩阵,同样假设疾病与疾病之间的相似性大于0.5,则认为两者之间具有强关联关系,疾病子图的邻接矩阵具体表示为:
[0123][0124]
步骤二:利用构建的circrna-circrna关系子图、疾病与疾病关系子图以及已知的circrna-疾病关系图构建circrna与疾病的异构图。
[0125]
步骤二中,利用已知的circrna-疾病关系图、circrna-circrna关系子图和疾病与
疾病关系子图构建circrna与疾病的异构图;按照如下方法先构建异构图的邻接矩阵,再利用python中的dgl库构建异构图g;
[0126]
构建异构图邻接矩阵h的公式为:
[0127][0128]
异构图g中点的特征矩阵x为:
[0129][0130]
其中,a表示已知circrna与疾病构成的关联矩阵,a矩阵的行代表circrna,列代表疾病,如果第c
p
个circrna和第dq个疾病存在关联,p=1,2,
…
,nc;q=1,2,
…
,nd,则a中位于第c
p
行第dq列的值否则否则和为需要学习的特征投影矩阵,表示实数范围内取值,nc×
k表示wc的大小,nd×
k表示wd的大小,k为投影之后特征的维度。
[0131]
步骤三:利用多头动态注意力机制学习异构图上每个节点特征的不同聚合表达,在此基础上利用具有不同大小卷积核的单层卷积网络从节点特征的不同聚合表达中提取具有信息的高阶特征。
[0132]
步骤三的具体过程为:
[0133]
利用多头动态图注意力机制获取异构图上节点特征的不同聚合表示,动态注意力机制的核心思想是聚合节点不同邻居的特征时,给邻居分配不同的权重;
[0134]
异构图g中以circrna c
p
作为节点,以疾病dq作为c
p
的邻居节点,首先定义一个分数函数来计算异构图g上从节点c
p
到邻居节点dq的注意力权重分数
[0135][0136]
其中||表示向量的拼接,和分别表示异构图g中点的特征矩阵x中的第c
p
行和第dq行,a表示单层前馈神经网络,a
t
表示的a转置,leakyrelu()表示神经网络中的激活函数;使用softmax函数对邻居的注意权重分数进行归一化,得到关注邻居节点的注意力值
[0137][0138]
nc表示图上节点c
p
的邻居集合,则节点c
p
的邻居节点特征聚合表示为:
[0139][0140]
其中,σ()表示神经网络中的激活函数relu();另外,使用单一的注意机制来聚合节点的邻域特征并不足以完全表示节点之间的关系,因此,将多个注意力头输出的特征表
示进行聚合,得到节点c更丰富的特征表示,表述如下:
[0141][0142]
k是注意力头的个数,表示第k注意头所聚合的节点特征表示,表示节点c
p
提取了多头注意力聚合邻居特征的高阶特征表示,f(
·
)表示一个特征提取的函数,计算的f(
·
)共分为三个步骤;
[0143]
第一步,通过多头注意力机制获得的节点c
p
特征的不同聚合表示被垂直地堆叠起来:
[0144][0145]
表示节点c
p
的特征堆叠矩阵;
[0146]
第二步,利用具有不同大小卷积核的单层卷积网络从特征堆叠矩阵中提取具有丰富信息的高阶特征,第l个卷积核的特征提取公式如下:
[0147][0148]
■
表示卷积操作,表示第l个卷积核参数矩阵,表示偏置项,表示节点c
p
的不同特征聚合表达堆叠矩阵经过第l个卷积核所提取出来的特征向量,ψ()表示神经网络中的激活函数relu();
[0149]
第三步,将由不同卷积核得到的特征向量拼接在一起,形成具有丰富信息的高阶特征表示,用公式表示如下:
[0150][0151]
表示节点c
p
提取了多头注意力聚合邻居特征的高阶特征表示,为公式(19)的输出表示;
[0152]
因此,异构图中点的特征矩阵x进一步用x
′
表示:
[0153][0154]
由公式(23)可知,表示x
′
的第(nc nd)行,x
′
[1:nc]和x
′
[nc 1:nc nd]分别
表示异构图中circrna和疾病的高阶特征信息。
[0155]
步骤四:利用广义矩阵分解交互节点的高阶特征以表达异构图上节点之间的复杂关系;最后利用多层感知机网络学习节点之间的特征交互以达到预测circrna节点与疾病节点之间潜在关系的目的。
[0156]
步骤四中,用广义矩阵分解来表示circrna与疾病之间复杂的高阶非线性特征交互;广义矩阵分解的定义如下:
[0157][0158]
其中,
⊙
表示哈达玛乘积,和分别表示x
′
中的第c
p
行和第dq行,p
pairwise
(c
p
,dq)表示circrna c
p
与疾病dq的高阶特征交互;
[0159]
利用多层感知器网络学习节点之间的特征交互被表示为:
[0160][0161]
公式(25)中的w,w2…wl
和ε1,ε2,
…
ε
l
均为需要训练的参数矩阵,表示circrna
p
与疾病dq的关联预测得分,模型是通过用l2正则化来最小化二元交叉熵损失来训练的,通过下面的损失函数公式来计算:
[0162][0163]
其中,a已知的circrna与疾病的关联矩阵,表示a中位于第c
p
行第dq列的值,表示预测的circrna与疾病的关联矩阵,表示中位于第c
p
行第dq列的值,||θ||2表示l2正则化,l2正则化的参数矩阵为θ,s
为训练用的正样本,用来表示已知的circrna与疾病的关联;s-为训练用的负样本,用来表示未知的circrna与疾病的关联。
[0164]
实例
[0165]
相关预测circrna与疾病关联的方法介绍
[0166]
[1]lu c,zeng m,zhang f,et al.deep matrix factorization improves prediction of human circrna-disease associations[j].ieee journal of biomedical and health informatics,2020,25(3):891-899。
[0167]
文献[1]公开了一种深度矩阵分解的预测方法。它主要考虑已知circrna与疾病关联的显式和隐式的反馈,然后利用投影层自动学习circrna与疾病的表示来达到预测潜在circrna与疾病关联的目的。
[0168]
[2]zhang w,yu c,wang x,et al.predicting circrna-disease associations through linear neighborhood label propagation method[j].ieee access,2019,7:83474-83483。
[0169]
文献[2]公开了一种线性邻域标签传播方法来预测circrna与疾病的关联。首先,它使用基于已知circrna与疾病的关联来计算circrna-circrna相似性和疾病-疾病相似
性。接下来,分别基于circrna-circrna相似性的图和基于疾病-疾病相似性的图实现标签传播,以预测circrna-疾病关联。
[0170]
[3]ge e,yang y,gang m,et al.predicting human disease-associated circrnas based on locality-constrained linear coding[j].genomics,2020,112(2):1335-1342。
[0171]
文献[3]公开了一种基于局部约束线性编码和标签传播的预测方法。它首先在已知的关联矩阵上使用局部约束线性编码获得重构的circrna-circrna相似性矩阵和疾病-疾病相似性矩阵,然后利用标签传播方法在重构的相似性矩阵和原有的相似性矩阵上进行标签传播获得最终的circrna与疾病的预测关联得分。
[0172]
本发明方法与相关预测方法指标在性能上的比较:
[0173]
评价指标:准确率(acc.),精度(pre.),召回率(rec.),f1分数(f1-score):
[0174][0175][0176][0177][0178]
其中,从正样本和负样本中正确识别的样本数分别用tp和tn表示。从正样本和负样本中错误识别的样本数量分别用fp和fn表示。比较的结果如表1所示:
[0179]
表1
[0180][0181]
结论:跟其他预测circrna与疾病关联的方法相比,本发明所提出的预测方法在性能指标上都比其他方法要好,具有可靠性高,性能优越的特点。
[0182]
本发明方法在预测与疾病存在潜在关联的circrna上的结果,如表2、表3。
[0183]
检验预测的证据为生物医学文献免费检索系统(pubmed)中发布的证实某些环状rna与疾病存在医学关系的文章id(pmid)。
[0184]
表2预测与肝细胞癌有潜在关联的circrna(预测得分排名前15)
[0185][0186]
表3预测与非小细胞肺癌有潜在关联的circrna(预测得分排名前15)
[0187][0188]
结论:表2和表3中,通过预测与两种疾病存在潜在关联的circrna结果说明,本发明在实际使用过程中能达到可靠的效果。虽然有些预测的结果还没被证实,但是,这些未被证实的预测结果可以进一步帮助生物信息人员提供候选的检测目标,这将会极大地提高科研效率,有助于尽快找到疾病的病发位置,从而及时的对症下药。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。