基于二代测序数据检测mhc ii型肿瘤新生抗原的方法和装置
技术领域
1.本技术涉及肿瘤新生抗原检测技术领域,特别是涉及一种基于二代测序数据检测mhc ii型肿瘤新生抗原的方法和装置。
背景技术:
2.肿瘤是世界第一大病,并且近年肿瘤发病率有增无减。肿瘤免疫治疗就是通过重新启动并维持肿瘤-免疫循环,恢复机体正常的抗肿瘤免疫反应,从而控制与清除肿瘤的一种治疗方法。肿瘤免疫治疗药物包括单克隆抗体类免疫检查点抑制剂、治疗性抗体、癌症疫苗、细胞治疗和小分子抑制剂等。近几年,肿瘤免疫治疗的好消息不断,目前已在多种肿瘤,如黑色素瘤、非小细胞肺癌、肾癌和前列腺癌等实体瘤的治疗中展示出了强大的抗肿瘤活性,多个肿瘤免疫治疗药物已经获得美国fda(food and drug administration,fda)批准临床应用。肿瘤免疫治疗由于其卓越的疗效和创新性,在2013年被《科学》杂志评为年度最重要的科学突破。
3.细胞的癌变通常是体细胞中的基因突变长期积累的结果,但不是所有的体细胞突变都会导致细胞癌变。目前主流观点认为,只有在驱动基因上的特异突变才会赋予细胞癌变的特性,这种突变叫做驱动突变(driver mutation)。而驱动突变又会引发其他的基因突变,这导致癌细胞中的基因突变数量往往高于正常的体细胞。肿瘤突变负荷(tmb)是反映肿瘤细胞中总的基因突变程度的一个指标,通常以每百万碱基(mb)的肿瘤基因组区域中包含的肿瘤体细胞突变总数来表示。
4.肿瘤特异性抗原(tumor-specific antigens,缩写tsas)是指肿瘤细胞所特有的抗原,又称新生抗原(neoantigens)。肿瘤特异性抗原提出于上世纪前半叶,之后随着分子生物学发展及对主要组织相容性复合体(major histocompatibility complex,缩写mhc)的分子功能被深入认识。boon等人首先发现在肿瘤中,有肿瘤产生的特异性肽段与mhc分子复合物可以被cd8 或者是cd4 等t细胞识别。随后的研究认识到,这些能被t细胞识别的抗原来自于肿瘤的基因组变异表达成肿瘤特有的肽段(neo-epitopes),被定义为新生抗原(neoantigens)。所以新生抗原可以作为肿瘤治疗的理想靶点。
5.肿瘤特异性新生抗原分为mhc i型和mhc ii型,以往的检测装置多是针对mhc i型新生抗原;但是随着肿瘤免疫类基础研究的进展,发现被cd4 t细胞识别的mhc ii型新生抗原显示出越来越重要的作用。有研究表明,mhc ii型新生抗原可能才是引起肿瘤免疫反应的关键所在。然而,mhc ii型新生抗原一直是检测的难点,因为其亚型众多,多于mhc i型;所以,每种亚型可用数据较少;并且,mhc ii型新生抗原的抗原长度范围更广,与mhc分子的结合是非线性的,进一步增加了mhc ii型肿瘤新生抗原的检测和分析难度。
6.同时还有研究表明,一个突变同时产生能被mhc i型和mhc ii型分子识别的新生抗原肽,会提示出该变异位点的高免疫源性;所以种种缘由使得对mhc ii型新生抗原的检测显得尤为重要。
7.因此,如何更准确、有效的检测mhc ii型肿瘤新生抗原,是肿瘤免疫治疗技术领域
matrix,使用编码氨基酸序列的blosum50矩阵对mhc分子进行编码转换成blosum50matrix,将编码好的两个矩阵分别输入lstm模型中进行训练,训练至验证集的损失函数值不再明显改善,即获得mhc ii型肿瘤新生抗原预测模型。
21.其中,步骤(2)中,编码好的两个矩阵即分别使用blosum50矩阵对新生抗原肽段和mhc分子进行编码转换成的两个blosum50 matrix。步骤(2)中的新生抗原肽段,即步骤(1)的多肽,新生抗原肽段和多肽可以理解为同一对象,肿瘤特有的多肽就是新生抗原,用于评估软件的数据。步骤(2)中,mhc分子分为i型和ii型,针对ii型新生抗原的就是mhc ii型分子,人的mhc分子就是hla。
22.需要说明的是,本技术的关键在于实现了基于二代测序数据的mhc ii型肿瘤新生抗原检测,至于mhc ii型肿瘤新生抗原预测模型的训练步骤,可以理解,在已经存在mhc ii型肿瘤新生抗原预测模型的情况下,可以直接使用该模型,不必每次都进行该训练步骤。因此,本技术的mhc ii型肿瘤新生抗原检测方法,除了第一次需要进行预测模型训练以外,后续检测过程中,模型训练步骤并非必须进行。
23.本技术的一种实现方式中,mhc ii型肿瘤新生抗原预测步骤,采用mhc ii型肿瘤新生抗原预测模型进行mhc ii型肿瘤新生抗原预测,具体包括,(1)从人蛋白组数据集中随机截取长度为13-19长度的短肽,形成一个肽段数据集,使用mhc ii型肿瘤新生抗原预测模型对肽段数据集中的短肽进行预测,得到一个预测值数据集;(2)一个mhc ii型肿瘤新生抗原的预测结果值,用其在预测值数据集中处于前百分之多少来代表,即在预测值数据集中越靠前,则更有可能是高免疫源性的mhc ii型肿瘤新生抗原。其中,人蛋白组数据集就是一个人的蛋白数据,来自于uniprot。
24.本技术的一种实现方式中,将预测结果值小于5%判断为阳性,其对应的多肽即候选mhc ii型肿瘤新生抗原,从中筛选表达量值tpm大于tpm阈值的氨基酸序列,即获得mhc ii型肿瘤新生抗原。
25.本技术的一种实现方式中,tpm阈值为5。
26.本技术的第二方面公开了一种基于二代测序数据检测mhc ii型肿瘤新生抗原的装置,包括肿瘤样本dna数据获取和比对模块、肿瘤样本rna数据获取和比对模块、肿瘤样本hla分型鉴别模块、基因变异检测模块、候选新生抗原突变肽翻译模块、样本各基因的表达量值计算模块、mhc ii型肿瘤新生抗原预测模型训练模块、mhc ii型肿瘤新生抗原预测模块;
27.肿瘤样本dna数据获取和比对模块,包括用于获取肿瘤样本的dna测序数据,并将其比对到人全基因组参考序列,获得dna比对文件;
28.肿瘤样本rna数据获取和比对模块,包括用于获取肿瘤样本的rna测序数据,并将其比对到人全基因组参考序列,获得rna比对文件;
29.肿瘤样本hla分型鉴别模块,包括用于根据dna比对文件,分析鉴定肿瘤样本的hla分型;
30.基因变异检测模块,包括用于根据dna比对文件,检测dna数据中相较于人全基因组参考序列所发生的基因变异,包括点突变和插入缺失突变;
31.候选新生抗原突变肽翻译模块,包括用于根据基因变异检测模块的结果,将发生变异的基因翻译为氨基酸序列,将其作为候选肿瘤新生抗原集;
32.样本各基因的表达量值计算模块,包括用于根据rna比对文件分析肿瘤样本各基因的表达量值tpm;
33.mhc ii型肿瘤新生抗原预测模型训练模块,包括用于(1)获取蛋白质谱数据集,其含有分离mhc ii型分子与多肽的复合物,复合物中的多肽被洗脱下来,经由蛋白质谱测序即得到该多肽的氨基酸序列,由此获得细胞表达呈递的ii型抗原数据集,作为训练数据;(2)使用编码氨基酸序列的blosum50矩阵对新生抗原肽段进行编码转换成blosum50 matrix,使用编码氨基酸序列的blosum50矩阵对mhc分子进行编码转换成blosum50 matrix,将编码好的两个矩阵分别输入lstm模型中进行训练,训练至验证集的损失函数值不再明显改善,即获得mhc ii型肿瘤新生抗原预测模型;
34.mhc ii型肿瘤新生抗原预测模块,包括用于根据hla分型和mhc ii型肿瘤新生抗原预测模型,从候选肿瘤新生抗原集中筛选表达量值tpm大于tpm阈值的氨基酸序列,作为mhc ii型肿瘤新生抗原。
35.其中,mhc ii型肿瘤新生抗原预测模型训练模块除了在第一次进行mhc ii型肿瘤新生抗原预测模型训练需要运行以外,在已经获得mhc ii型肿瘤新生抗原预测模型的情况下,后续模块可以直接使用该预测模型即可,不需要每次检测都重复运行mhc ii型肿瘤新生抗原预测模型训练模块。mhc ii型肿瘤新生抗原预测模型训练模块的步骤(2)中,编码好的两个矩阵即分别使用blosum50矩阵对新生抗原肽段和mhc分子进行编码转换成的两个blosum50 matrix。新生抗原肽段,即步骤(1)的多肽,新生抗原肽段和多肽可以理解为同一对象,肿瘤特有的多肽就是新生抗原,用于评估软件的数据。mhc分子分为i型和ii型,针对ii型新生抗原的就是mhc ii型分子,人的mhc分子就是hla。
36.需要说明的是,本技术基于二代测序数据检测mhc ii型肿瘤新生抗原的装置,实际上就是通过各模块实现本技术基于二代测序数据检测mhc ii型肿瘤新生抗原的方法的各个步骤,因此,本技术装置中各模块的具体实现方式或参数条件可以参考本技术的方法,例如采用mhc ii型肿瘤新生抗原预测模型进行mhc ii型肿瘤新生抗原预测的具体步骤,阳性判断,tpm阈值等都可以参考本技术基于二代测序数据检测mhc ii型肿瘤新生抗原的方法,在此不累述。
37.本技术的第三方面公开了一种基于二代测序数据检测mhc ii型肿瘤新生抗原的装置,该装置包括存储器和处理器;存储器,用于存储程序;处理器,用于通过执行存储器存储的程序以实现本技术基于二代测序数据检测mhc ii型肿瘤新生抗原的方法。
38.本技术的第四方面公开了一种计算机可读存储介质,其包括程序,该程序能够被处理器执行以实现本技术基于二代测序数据检测mhc ii型肿瘤新生抗原的方法。
39.由于采用以上技术方案,本技术的有益效果在于:
40.本技术基于二代测序数据检测mhc ii型肿瘤新生抗原的方法和装置,直接通过二代测序数据预测mhc ii型肿瘤新生抗原,解决了肿瘤免疫治疗中对mhc ii型新生抗原预测和筛选的迫切需求,为肿瘤免疫的研究和针对新生抗原的免疫治疗提供了帮助。
附图说明
41.图1是本技术实施例中基于二代测序数据检测mhc ii型肿瘤新生抗原的方法的流程框图;
42.图2是本技术实施例中基于二代测序数据检测mhc ii型肿瘤新生抗原的装置的结构框图;
43.图3是本技术实施例中基于二代测序数据检测mhc ii型肿瘤新生抗原的方法预测的mhc ii型肿瘤新生抗原的roc曲线图;
44.图4是本技术实施例中基于二代测序数据检测mhc ii型肿瘤新生抗原的方法预测的另一mhc ii型肿瘤新生抗原的roc曲线图。
具体实施方式
45.下面通过具体实施方式结合附图对本技术作进一步详细说明。在以下的实施方式中,很多细节描述是为了使得本技术能被更好的理解。然而,本领域技术人员可以毫不费力的认识到,其中部分特征在不同情况下是可以省略的,或者可以由其他元件、材料、方法所替代。在某些情况下,本技术相关的一些操作并没有在说明书中显示或者描述,是为了避免本技术的核心部分被过多的描述所淹没,而对于本领域技术人员而言,详细描述这些相关操作并不是必要的,他们根据说明书中的描述以及本领域的一般技术知识即可完整了解相关操作。
46.mhc ii型肿瘤新生抗原检测一直是本领域的难点,由于mhc ii型肿瘤新生抗原亚型众多、抗原长度范围更广,且与mhc分子的结合是非线性的,本技术创造性的引入能处理复杂信息的非线性模型,来解决mhc ii型新生抗原预测的训练问题。另外,由于新生抗原的预测准确度一直不是很高,本技术旨在改善和提高基于肿瘤样本二代测序数据的mhc ii型肿瘤新生抗原的检测灵敏度和准确度。
47.因此,本技术创造性的研发了一种新的基于二代测序数据检测mhc ii型肿瘤新生抗原的方法,如图1所示,包括肿瘤样本dna数据获取和比对步骤11、肿瘤样本rna数据获取和比对步骤12、肿瘤样本hla分型鉴别步骤13、基因变异检测步骤14、候选新生抗原突变肽翻译步骤15、样本各基因的表达量值计算步骤16和mhc ii型肿瘤新生抗原预测步骤18。
48.其中,肿瘤样本dna数据获取和比对步骤11,包括获取肿瘤样本的dna测序数据,并将其比对到人全基因组参考序列,获得dna比对文件。例如,将dna样本二代测序fastq.gz文件作为输入,使用bwa mem(https://github.com/lh3/bwa)将序列比对到人全基因组参考序列,得到比对的dna.bam,即dna比对文件。
49.肿瘤样本rna数据获取和比对步骤12,包括获取肿瘤样本的rna测序数据,并将其比对到人全基因组参考序列,获得rna比对文件。例如,将rna样本二代测序fastq.gz文件作为输入,使用rna比对软件star(https://github.com/alexdobin/star)将序列比对到人全基因组参考序列,得到比对的rna.bam,即rna比对文件。
50.肿瘤样本hla分型鉴别步骤13,包括根据dna比对文件,分析鉴定肿瘤样本的hla分型。例如,将dna.bam作为输入,使用软件bwahla、polysolver做hla分型分析,并取其结果并集作为样本hla分型结果。
51.基因变异检测步骤14,包括根据dna比对文件,检测dna数据中相较于人全基因组参考序列所发生的基因变异,包括点突变和插入缺失突变。例如,以dna.bam作为输入,由vardict(https://github.com/astrazeneca-ngs/vardict)鉴定出其产生的非同义单核苷酸突变和插入缺失突变,输出mutation.vcf。
52.候选新生抗原突变肽翻译步骤15,包括根据基因变异检测步骤的结果,将发生变异的基因翻译为氨基酸序列,将其作为候选肿瘤新生抗原集。例如,将mutation.vcf作为输入,由vep(https://asia.ensembl.org/info/docs/tools/vep/index.html)注释突变所在的基因及其对应的氨基酸序列,并提取突变得到的氨基酸序列,得到mutation.vep.vcf。
53.样本各基因的表达量值计算步骤16,包括根据rna比对文件分析肿瘤样本各基因的表达量值tpm。例如,将rna.bam作为输入,由rsem(https://github.com/deweylab/rsem)分析出该样本各gene的表达量值tpm。
54.mhc ii型肿瘤新生抗原预测步骤18,包括根据hla分型和mhc ii型肿瘤新生抗原预测模型,从候选肿瘤新生抗原集中筛选表达量值tpm大于tpm阈值的氨基酸序列,作为mhc ii型肿瘤新生抗原;其中,mhc ii型肿瘤新生抗原预测模型为采用基于蛋白质谱数据集的细胞表面蛋白质谱检测出的肽段序列数据集作为训练数据,训练获得的根据hla分型预测mhc ii型肿瘤新生抗原的模型;蛋白质谱数据集含有分离mhc ii型分子与多肽的复合物,将多肽从mhc分型上洗脱下来,再经由蛋白质谱测序获得肽段序列数据集。
55.其中,mhc ii型肿瘤新生抗原预测模型,由mhc ii型肿瘤新生抗原预测模型训练步骤17获得,该步骤包括(1)获取蛋白质谱数据集,其含有分离mhc ii型分子与多肽的复合物,复合物中的多肽被洗脱下来,经由蛋白质谱测序即得到该多肽的氨基酸序列,由此获得细胞表达呈递的ii型抗原数据集,作为训练数据;(2)使用基于氨基酸之前关系的blosum50矩阵对新生抗原肽段进行编码转换成blosum50 matrix,使用基于氨基酸之前关系的blosum50矩阵对mhc分子(人的mhc分子就是hla)进行编码转换成blosum50 matrix,将编码好的两个矩阵分别输入lstm模型中进行训练,训练至验证集的损失函数值不再明显改善,即获得mhc ii型肿瘤新生抗原预测模型。
56.本技术的数据集引用自:
57.https://services.healthtech.dtu.dk/suppl/immunology/nar_netmhcpan_netmhciipan/netmhciipan_train.tar.gz
58.本领域技术人员可以理解,上述实施方式方法的全部或部分功能可以通过硬件的方式实现,也可以通过计算机程序的方式实现。当上述实施方式中全部或部分功能通过计算机程序的方式实现时,该程序可以存储于一个计算机可读存储介质中,存储介质可以包括:只读存储器、随机存储器、磁盘、光盘、硬盘等,通过计算机执行该程序以实现上述功能。例如,将程序存储在设备的存储器中,当通过处理器执行存储器中程序,即可实现上述全部或部分功能。另外,当上述实施方式中全部或部分功能通过计算机程序的方式实现时,该程序也可以存储在服务器、另一计算机、磁盘、光盘、闪存盘或移动硬盘等存储介质中,通过下载或复制保存到本地设备的存储器中,或对本地设备的系统进行版本更新,当通过处理器执行存储器中的程序时,即可实现上述实施方式中全部或部分功能。
59.因此,基于本技术的基于二代测序数据检测mhc ii型肿瘤新生抗原的方法,本技术提出了一种基于二代测序数据检测mhc ii型肿瘤新生抗原的装置,如图2所示,包括肿瘤样本dna数据获取和比对模块21、肿瘤样本rna数据获取和比对模块22、肿瘤样本hla分型鉴别模块23、基因变异检测模块24、候选新生抗原突变肽翻译模块25、样本各基因的表达量值计算模块26、mhc ii型肿瘤新生抗原预测模型训练模块27、mhc ii型肿瘤新生抗原预测模块28。
60.其中,肿瘤样本dna数据获取和比对模块21,包括用于获取肿瘤样本的dna测序数据,并将其比对到人全基因组参考序列,获得dna比对文件。例如,参考bwa mem将序列比对到人全基因组参考序列,得到dna比对文件。
61.肿瘤样本rna数据获取和比对模块22,包括用于获取肿瘤样本的rna测序数据,并将其比对到人全基因组参考序列,获得rna比对文件。例如,参考rna比对软件star将序列比对到人全基因组参考序列,得到rna比对文件。
62.肿瘤样本hla分型鉴别模块23,包括用于根据dna比对文件,分析鉴定肿瘤样本的hla分型。例如,参考软件bwahla、polysolver进行hla分型分析。
63.基因变异检测模块24,包括用于根据dna比对文件,检测dna数据中相较于人全基因组参考序列所发生的基因变异,包括点突变和插入缺失突变。例如,参考vardict进行基因变异鉴定。
64.候选新生抗原突变肽翻译模块25,包括用于根据基因变异检测步骤的结果,将发生变异的基因翻译为氨基酸序列,将其作为候选肿瘤新生抗原集。例如,参考vep注释突变所在的基因及其对应的氨基酸序列,并提取突变得到的氨基酸序列。
65.样本各基因的表达量值计算模块26,包括用于根据rna比对文件分析肿瘤样本各基因的表达量值tpm。例如,参考rsem分析各基因的表达量值tpm。
66.mhc ii型肿瘤新生抗原预测模型训练模块27,包括用于(1)获取蛋白质谱数据集,其含有分离mhc ii型分子与多肽的复合物,复合物中的多肽被洗脱下来,经由蛋白质谱测序即得到该多肽的氨基酸序列,由此获得细胞表达呈递的ii型抗原数据集,作为训练数据;其中,数据集引用自:https://services.healthtech.dtu.dk/suppl/immunology/nar_netmhcpan_netmhciipan/netmhciipan_train.tar.gz;(2)使用编码氨基酸序列的blosum50矩阵对新生抗原肽段进行编码转换成blosum50 matrix,使用编码氨基酸序列的blosum50矩阵对mhc分子(人的mhc分子就是hla)进行编码转换成blosum50 matrix,将编码好的两个矩阵分别输入lstm模型中进行训练,训练至验证集的损失函数值不再明显改善,即获得mhc ii型肿瘤新生抗原预测模型。lstm模型即常用时间序列深度学习模型。
67.mhc ii型肿瘤新生抗原预测模块28,包括用于根据hla分型和mhc ii型肿瘤新生抗原预测模型,从候选肿瘤新生抗原集中筛选表达量值tpm大于tpm阈值的氨基酸序列,作为mhc ii型肿瘤新生抗原。
68.本技术中,模型训练和mhc ii型肿瘤新生抗原预测具体方案如下:
69.1.训练数据,模型训练是基于一个蛋白质谱数据集,该数据集是有分离mhc ii型分子与多肽的复合物,然后将多肽从mhc分析上洗脱下来,再经由蛋白质谱测序得到该多肽的氨基酸序列,由于获得一个细胞表达呈递的ii型抗原数据集,数据集引用自:https://services.healthtech.dtu.dk/suppl/immunology/nar_netmhcpan_netmhciipan/netmhciipan_train.tar.gz
70.2.模型构建,(1)使用基于氨基酸之前关系的blosum50矩阵,即编码氨基酸序列的blosum50矩阵,对新生抗原肽段进行编码转换成blosum50matrix;(2)使用基于氨基酸之前关系的blosum50矩阵对mhc分子(人的mhc分子就是hla)进行编码转换成blosum50 matrix;(3)将编码好的2个矩阵分别输入一个由16个神经元组成的lstm(常用时间序列深度学习模型)模型中;(4)两个lstm的输出连接到一个60个神经元组成的全连接层,并最终输出预测
结果;(5)训练模型至验证集的损失函数值不再明显改善,即获得mhc ii型肿瘤新生抗原预测模型。
71.3.从人蛋白组数据集中随机截取长度为13-19长度的短肽,形成一个肽段数据集,使用“2.模型构建”中的模型对数据集中的短肽做预测,得到一个预测值数据集。其中,蛋白组数据集即来自于uniprot的蛋白数据。
72.4.一个新生抗原肽的预测结果值,用该值在前述步骤中的预测值数据集中处于前百分之多少来代表,即一个新生抗原肽在随机一个随机数据集中的预测值所处的位置,百分比越低代表更可能是一个高免疫源性的新生抗原,本技术中阈值为《5%则判定为阳性,否则为阴性。
73.mhc ii型肿瘤新生抗原预测:将肿瘤样本hla分型鉴别获得的样本hla分型结果,和候选新生抗原突变肽翻译获得的突变的氨基酸序列,即mutation.vep.vcf,输入mhc ii型肿瘤新生抗原预测模型,预测得到高免疫源性的ii型新生抗原:进一步的,根据样本各基因的表达量值计算获得的各基因的表达量值tpm,筛选tpm值》5的结果,作为mhc ii型肿瘤新生抗原。
74.本技术的另一实现方式中还提供了一种基于二代测序数据检测mhc ii型肿瘤新生抗原的装置,该装置包括存储器和处理器;存储器,包括用于存储程序;处理器,包括用于通过执行存储器存储的程序以实现以下方法:肿瘤样本dna数据获取和比对步骤,包括获取肿瘤样本的dna测序数据,并将其比对到人全基因组参考序列,获得dna比对文件;肿瘤样本rna数据获取和比对步骤,包括获取肿瘤样本的rna测序数据,并将其比对到人全基因组参考序列,获得rna比对文件;肿瘤样本hla分型鉴别步骤,包括根据dna比对文件,分析鉴定肿瘤样本的hla分型;基因变异检测步骤,包括根据dna比对文件,检测dna数据中相较于人全基因组参考序列所发生的基因变异,包括点突变和插入缺失突变;候选新生抗原突变肽翻译步骤,包括根据基因变异检测步骤的结果,将发生变异的基因翻译为氨基酸序列,将其作为候选肿瘤新生抗原集;样本各基因的表达量值计算步骤,包括根据rna比对文件分析肿瘤样本各基因的表达量值tpm;mhc ii型肿瘤新生抗原预测步骤,包括根据hla分型和mhc ii型肿瘤新生抗原预测模型,从候选肿瘤新生抗原集中筛选表达量值tpm大于tpm阈值的氨基酸序列,作为mhc ii型肿瘤新生抗原;其中,mhc ii型肿瘤新生抗原预测模型为采用基于蛋白质谱数据集的细胞表面蛋白质谱检测出的肽段序列数据集作为训练数据,训练获得的根据hla分型预测mhc ii型肿瘤新生抗原的模型;蛋白质谱数据集含有分离mhc ii型分子与多肽的复合物,将多肽从mhc分型上洗脱下来,再经由蛋白质谱测序获得肽段序列数据集。根据需求,还可以包括mhc ii型肿瘤新生抗原预测模型训练步骤,该步骤包括(1)获取蛋白质谱数据集,其含有分离mhc ii型分子与多肽的复合物,复合物中的多肽被洗脱下来,经由蛋白质谱测序即得到该多肽的氨基酸序列,由此获得细胞表达呈递的ii型抗原数据集,作为训练数据;(2)使用编码氨基酸序列的blosum50矩阵对新生抗原肽段进行编码转换成blosum50 matrix,使用编码氨基酸序列的blosum50矩阵对mhc分子(人的mhc分子就是hla)进行编码转换成blosum50 matrix,将编码好的两个矩阵分别输入lstm模型中进行训练,训练至验证集的损失函数值不再明显改善,即获得mhc ii型肿瘤新生抗原预测模型。
75.本技术另一种实现方式中还提供一种计算机可读存储介质,该存储介质中包括程序,该程序能够被处理器执行以实现如下方法:肿瘤样本dna数据获取和比对步骤,包括获
取肿瘤样本的dna测序数据,并将其比对到人全基因组参考序列,获得dna比对文件;肿瘤样本rna数据获取和比对步骤,包括获取肿瘤样本的rna测序数据,并将其比对到人全基因组参考序列,获得rna比对文件;肿瘤样本hla分型鉴别步骤,包括根据dna比对文件,分析鉴定肿瘤样本的hla分型;基因变异检测步骤,包括根据dna比对文件,检测dna数据中相较于人全基因组参考序列所发生的基因变异,包括点突变和插入缺失突变;候选新生抗原突变肽翻译步骤,包括根据基因变异检测步骤的结果,将发生变异的基因翻译为氨基酸序列,将其作为候选肿瘤新生抗原集;样本各基因的表达量值计算步骤,包括根据rna比对文件分析肿瘤样本各基因的表达量值tpm;mhc ii型肿瘤新生抗原预测步骤,包括根据hla分型和mhc ii型肿瘤新生抗原预测模型,从候选肿瘤新生抗原集中筛选表达量值tpm大于tpm阈值的氨基酸序列,作为mhc ii型肿瘤新生抗原;其中,mhc ii型肿瘤新生抗原预测模型为采用基于蛋白质谱数据集的细胞表面蛋白质谱检测出的肽段序列数据集作为训练数据,训练获得的根据hla分型预测mhc ii型肿瘤新生抗原的模型;蛋白质谱数据集含有分离mhc ii型分子与多肽的复合物,将多肽从mhc分型上洗脱下来,再经由蛋白质谱测序获得肽段序列数据集。根据需求,还可以包括mhc ii型肿瘤新生抗原预测模型训练步骤,该步骤包括(1)获取蛋白质谱数据集,其含有分离mhc ii型分子与多肽的复合物,复合物中的多肽被洗脱下来,经由蛋白质谱测序即得到该多肽的氨基酸序列,由此获得细胞表达呈递的ii型抗原数据集,作为训练数据;(2)使用编码氨基酸序列的blosum50矩阵对新生抗原肽段进行编码转换成blosum50 matrix,使用编码氨基酸序列的blosum50矩阵对mhc分子(人的mhc分子就是hla)进行编码转换成blosum50 matrix,将编码好的两个矩阵分别输入lstm模型中进行训练,训练至验证集的损失函数值不再明显改善,即获得mhc ii型肿瘤新生抗原预测模型。
76.下面通过具体试验对本技术作进一步详细说明。以下试验仅对本技术进行进一步说明,不应理解为对本技术的限制。
77.实施例1
78.按照以上基于二代测序数据检测mhc ii型肿瘤新生抗原的方法,本例使用公开数据库中两个细胞系的质谱检测数据集作为测试数据,验证本技术的mhc ii型肿瘤新生抗原检测方法在该数据集中的预测效果。
79.数据来源:
80.https://static-content.springer.com/esm/art%3a10.1038%2fs41587-019-0280-2/mediaobjects/41587_2019_280_moesm3_esm.xlsx
81.验证方法:
82.本例分别使用本技术的基于二代测序数据检测mhc ii型肿瘤新生抗原的方法,以及另外两种mhc ii型新生抗原预测方法,对数据集中的肽段进行预测,并将预测结果,与实际的阴性和阳性结果进行对比分析,比较三种方法在此数据集中的mhc ii型肿瘤新生抗原预测效果。
83.另外两种mhc ii型新生抗原预测方法分别为maria和mixmhciipred。
84.maria:predicting hla class ii antigen presentation through integrated deep learning|nature biotechnology
85.mixmhciipred:robust prediction of hla class ii epitopes by deep motif deconvolution of immunopeptidomes|nature biotechnology
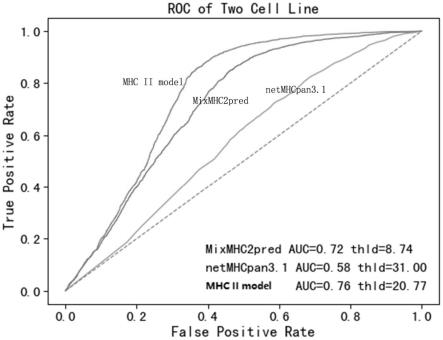
86.根据预测结果与实际的阴性和阳性结果的对比分析结果,绘制roc曲线,结果如图3所示。图3中,“mhc ii model”对应的曲线是本技术的mhc ii型肿瘤新生抗原检测方法的roc曲线,“mixmhc2pred”和“netmhcpan3.1”分别是另外两种mhc ii型新生抗原预测方法的roc曲线。
87.图3的结果显示,本技术基于二代测序数据检测mhc ii型肿瘤新生抗原的方法在roc曲线中得到最大的auc值0.76,优于另外两种方法,表明本技术方法在预测ii型新生抗原的呈递上的改善。
88.实施例2
89.按照以上基于二代测序数据检测mhc ii型肿瘤新生抗原的方法,本例使用公开数据库中一个实际引起cd4t细胞阳性反应的数据作为测试数据,用于验证本技术的mhc ii型肿瘤新生抗原检测方法在该数据集中的预测效果。
90.数据来源:
91.https://static-content.springer.com/esm/art%3a10.1038%2fs41587-019-0289-6/mediaobjects/41587_2019_289_moesm6_esm.xlsx
92.验证方法:
93.本例分别使用本技术的基于二代测序数据检测mhc ii型肿瘤新生抗原的方法,以及另外两种mhc ii型新生抗原预测方法,对数据集中的肽段进行预测,并将预测结果,与实际的阴性和阳性结果进行对比分析,比较三种方法在此数据集中的mhc ii型肿瘤新生抗原预测效果。另外两种mhc ii型新生抗原预测方法与实施例1相同。
94.根据预测结果与实际的阴性和阳性结果的对比分析结果,绘制roc曲线,结果如图4所示。图4中,“mhc ii model”对应的曲线是本技术的mhc ii型肿瘤新生抗原检测方法的roc曲线,“mixmhc2pred”和“netmhcpan3.1”分别是另外两种mhc ii型新生抗原预测方法的roc曲线。
95.图4的结果显示,本技术基于二代测序数据检测mhc ii型肿瘤新生抗原的方法在roc曲线中得到最大的auc值0.76,优于另外两种方法,表明本技术方法在筛选有免疫源性的ii型新生抗原上具有优势。
96.以上内容是结合具体的实施方式对本技术所作的进一步详细说明,不能认定本技术的具体实施只局限于这些说明。对于本技术所属技术领域的普通技术人员来说,在不脱离本技术构思的前提下,还可以做出若干简单推演或替换。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。