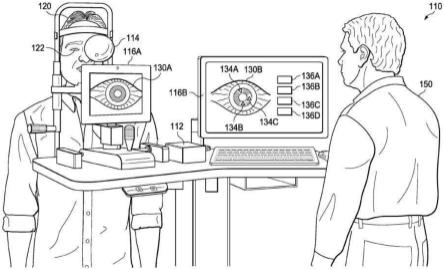
用于鉴定、分类和/或排序遗传序列的方法和系统1.相关申请的交叉引用2.本技术要求2020年3月23日提交的美国临时专利申请第62/993,567号和2019年11月12日提交的美国临时专利申请第62/934,323号的权益,所述申请中的每一者的公开内容以全文引用的方式并入本文中。3.序列表4.呈文本文件形式的序列表(标题为”2010794_2132_sl”,于2020年11月10日创建,并且大小为146,610字节)通过引用整体并入本文。
背景技术:
::5.近几十年来,基因组测序的速度和效率大大提高,从而能够收集到大量的基因组序列信息。可在可公开访问的数据库中获得超过100万个基因组序列,其中大部分是微生物基因组。例如,对于病原性冠状病毒sars-cov-2,大约160,000个基因组序列已寄存在可公开访问的数据库中。因此,有不断增长的不同基因组序列信息库。6.基因组序列信息的效用受到分析工具可用性的限制。分析所需的计算资源落后于序列数据的积累。例如,治疗和疫苗开发研究往往不能评估病原体群体的遗传多样性,从而导致临床试验失败。需要用于分析基因组序列信息的改进方法和系统,包括需要用于分析特定生物体、序列或基因的大量不同基因组序列的方法和系统。需要改进分析方法和系统,以为治疗开发提供信息并潜在地预测临床结果。另外,用于分析基因组序列信息的许多现有方法需要序列数据库的专用知识、序列分析软件的操作和/或数据输出的提炼。技术实现要素:7.本公开提供了用于分析基因组序列信息的方法和系统。近年来,基因组序列信息(包括微生物基因组序列信息)例如在可公开访问的数据库中激增。成本有效、高通量测序仪器和多重测序方案的开发已拓宽了基因组分析的吸引力,从而转变传染病的领域。然而,比较基因组分析通常由一组小的、有偏见的、经充分注释的库存基因组来指导,而没有考虑到公共数据库中可用的基因组多样性的广度。这些库存基因组通常被认为代表自然或相关多样性的广度,但实际上只代表自然群体的一微小部分。(例如)关于病原体的研究,这种鉴定、分析和/或代表自然多样性的问题尤其尖锐,其中所开发的治疗对不同病原体分离株的适用性是总体临床功效的重要组成部分。利用来自不同株系的可用序列在历史上需要计算技能和精心策划的最新基因组资源,其包括跨不同谱系(例如,跨病原体谱系)的基因组注释。至少部分由于大的可用基因组序列不是以这种方式完全组装,并且/或可用基因组序列(例如,病原体的不同株系的基因组序列)以不一致的方式注释,所以基因组分析(例如,种间或种内)在实践中是复杂的。随着测序的基因组数量的增加,对分析和计算工具的需要是确保优化利用这些资源的重要组成部分。8.本公开的方法和系统尤其提供了用于表征输入序列之中以及输入序列之间的序列保守性的方法和系统。如本文所讨论的,本公开的某些方法和系统包括在基于序列之间的比对的覆盖率百分比和序列之间的变异数量的多序列比较之后,将相似性或保守性评分分配给序列。9.在某些实施例中,本公开的方法和系统包括以下描述的一个或多个步骤。例如,在某些实施方案中,本文所述的方法和系统包括选择获取用于比较分析的基因组序列的生物体(例如病原体)的第一步骤。因此,在某些实施例中,用户在第一步骤中指示关于从中提取感兴趣序列的基因组的信息。第二步骤可包括例如通过从可公开访问的数据库获取序列数据,诸如通过从国家生物技术信息中心(nationalcenterforbiotechnologyinformationdatabase,ncbi)数据库下载,并且可选地从相同或不同来源获取序列注释和/或特征信息来提供序列。也可通过直接实验测量、例如从利用物理生物样品的高通量测序系统中读取来提供序列。因此,在某些实施例中,可通过直接测量、从ncbi数据库下载或两者来提供序列。序列和特征文件可从某些可公开访问的数据库(诸如ncbi数据库)自动下载。第三步骤可包括例如通过基本局部比对搜索工具(blast)成对比较分析序列。成对blast分析确立所有比较序列中每个分析的感兴趣序列的序列多样性水平。第四步骤可包括例如通过生成编译与序列保守性相关的信息的输出表编译与所有成对序列比较相关的信息。示例性表可包括关于特定序列的存在或不存在、特定序列基因座中的多样性水平、特定序列基因座中的变异性质和/或分析序列中特定特征的基因组坐标的信息。在各种实施方案中,可基于定义的评分系统为所分析的每个序列分配相似性评分,其中每个序列根据覆盖率百分比和序列变异数量进行归类。例如,在某些实施方案中,可根据表2对序列进行归类并分配相似性评分。在一些实施方案中,然后可从分析的序列中提取编码序列并翻译以产生核苷酸和氨基酸比对。可选的第五步骤可包括生成代表编译的序列保守性信息的视觉显示,例如,呈多样性图形、系统发育(例如,最大似然或简约系统发育)、热图和/或比对文件的形式。在某些实例中,基于基因组的系统发育和基因的系统发育是使用系统发育软件(诸如phyml或quicktree程序)生成,并保存到单独的文件中。10.在各种实施方案中,本文公开的方法和系统的步骤是通过使用计算机处理器和软件来实现。所述特定的专有软件在本文中称为“got_gene”,以r编程语言编写。got_gene使用blast算法和r包来鉴定、比较和表征一组序列的多样性,并可分析数千个序列间的多样性。11.在各种实施方案中,可用基因组序列的集合(主题序列,例如,参考序列)以成对方式与一个或多个用户选择的序列(查询序列)进行比较,以鉴定临床相关的序列特征。在各种实施方案中,本公开的方法和系统利用数据库(包括基因组序列信息的可公开访问的数据库)中可用的基因组序列信息的集合。在某些实施方案中,成对比较包括主题遗传序列和查询遗传序列(例如,主题编码遗传序列和查询编码遗传序列)的成对比较。在某些实施方案中,成对比较包括主题序列和查询序列编码的蛋白质的成对比较。12.在某些实施方案中,本公开的方法和系统可用于鉴定治疗效用的序列和序列特征。例如,本公开的方法和系统可用于鉴定候选抗原(例如,病原体抗原)以开发抗抗原治疗剂,诸如抗抗原治疗性抗体。在一些实施方案中,本公开的方法和系统可用于鉴定候选疫苗抗原。在一些实施方案中,本公开的方法和系统可用于确定一个或多个特定遗传序列(例如,实验室病原体株系的基因组)是否代表可比较的遗传序列的集合(例如,临床相关的病原体株系的基因组)。在一些实施方案中,本公开的方法和系统可用于鉴定抗生素抗性标记物。在一些实施方案中,本公开的方法和系统可用于生成肽发现资源,例如用于查询质谱法数据的预计肽和特征的列表。在一些实施方案中,本公开的方法和系统可用于鉴定序列内的多样性区域。在一些实施方案中,本公开的方法和系统可用于产生系统发育,例如,以增强对流行病(例如,病原体的传播)的临床理解。在一些实施方案中,本公开的方法和系统可用于鉴定物种之间或物种之中的直向同源序列。13.本公开的病原体可包括任何包括核酸或氨基酸序列或由核酸或氨基酸序列表征的病原体。本公开的病原体包括原核病原体和真核病原体。本发明的病原体的实例包括(但不限于)细菌、酵母、原生动物和病毒。在各种实施方案中,本公开的病原体选自鲍氏不动杆菌(acinetobacterbaumannii)、鲁氏不动杆菌(acinetobacterlwoffii)、不动杆菌属某些种(acinetobacterspp.)(例如,多药抗性不动杆菌(mdr-a))、放线菌(actinomycetes)、腺病毒(adenovirus)、气单胞菌属某些种(aeromonasspp.)、粪产碱菌(alcaligenesfaecalis)、产碱菌属某些种(alcaligenesspp.)/无色杆菌属某些种(achromobacterspp.)、木糖氧化产碱菌(alcaligenesxylosoxidans)(例如,广谱β-内酰胺酶(esbl)/多药抗性革兰氏阴性生物体(mrgn))、虫媒病毒(arbovirus)、人蛔虫(ascarislumbricoides)、曲菌属某些种(aspergillusspp.)、星状病毒(astrovirus)、炭疽芽孢杆菌(bacillusanthracis)、蜡状芽孢杆菌(bacilluscereus)、枯草杆菌(bacillussubtilis)、脆弱类杆菌(bacteriodesfragilis)、五日热巴尔通体(bartonellaquintana)、人芽囊原虫(blastocystishominis)、百日咳博德特氏菌(bordetellapertussis)、伯氏疏螺旋体(borreliaburgdorferi)、达氏疏螺旋体(borreliaduttoni)、回归热疏螺旋体(borreliarecurrentis)、缺陷短波单胞菌(brevundimonasdiminuta)、泡囊短波单胞菌(brevundimonasvesicularis)、布氏杆菌属某些种(brucellaspp.)、洋葱伯霍尔德杆菌(burkholderiacepacia)(例如,多药抗性(mdr))、鼻疽伯霍尔德杆菌(burkholderiamallei)、类鼻疽伯霍尔德杆菌(burkholderiapseudomallei)、空肠弯曲杆菌(campylobacterjejuni)/大肠弯曲杆菌(campylobactercoli)、白色念珠菌(candidaalbicans)、耳念珠菌(candidaauris)、克鲁斯念珠菌(candidakrusei)、近平滑念珠菌(candidaparapsilosis)、屈公病病毒(chikungunyavirus)(chikv)、肺炎衣原体(chlamydiapneumoniae)、鹦鹉衣原体(chlamydiapsittaci)、沙眼衣原体(chlamydiatrachomatis)、柠檬酸杆菌属某些种(citrobacterspp.)、肉毒梭菌(clostridiumbotulinum)、难养芽胞梭菌(clostridiumdifficile)、产气荚膜梭菌(clostridiumperfringens)、破伤风梭菌(clostridiumtetani)、冠状病毒(例如,严重急性呼吸综合征相关冠状病毒(sars-cov);严重急性呼吸综合征冠状病毒2(sars-cov2),其是引起冠状病毒疾病(covid-19)的病毒;以及中东呼吸综合征相关冠状病毒(mers-cov))、白喉棒状杆菌(corynebacteriumdiphtheriae)、假结核棒状杆菌(corynebacteriumpseudotuberculosis)、棒状杆菌属某些种(corynebacteriumspp.)、溃疡棒状杆菌(corynebacteriumulcerans)、伯内特考克斯体(coxiellaburnetii)、柯萨奇病毒(coxsackievirus)、克里米亚-刚果出血热病毒(crimean-congohaemorrhagicfevervirus)、新型隐球菌(cryptococcusneoformans)、人隐孢子虫(cryptosporidiumhominis)、小隐孢子虫(cryptosporidiumparvum)、卡耶塔环孢子虫(cyclosporacayetanensis)、巨细胞病毒(cytomegalovirus)、登革热病毒(denguevirus)、脆双核阿米species)、奇异变形杆菌(proteusmirabilis)(例如,esbl/mrgn)、普通变形杆菌(proteusvulgaris)、雷氏普罗威登斯菌(providenciarettgeri)、斯氏普罗威登斯菌(providenciastuartii)、绿脓假单胞菌(pseudomonasaeruginosa)、假单胞菌属某些种(pseudomonasspp.)、狂犬病病毒(rabiesvirus)、罗尔斯通氏菌属某些种(ralstoniaspp.)、呼吸道融合病毒、鼻病毒、普氏立克次体(rickettsiaprowazekii)、斑疹伤寒立克次体(rickettsiatyphi)、玫瑰单胞菌(roseomonasgilardii)、轮状病毒(rotavirus)、德国麻疹病毒(rubellavirus)、曼森氏住血吸虫(schistosomamansoni)、肠炎沙门氏菌(salmonellaenteritidis)、副伤寒沙门氏菌(salmonellaparatyphi)、沙门氏菌属某些种(salmonellaspp.)、伤寒沙门氏菌(salmonellatyphi)、鼠伤寒沙门氏菌(salmonellatyphimurium)、疥螨(sarcoptesscabiei,itchmite)、札如病毒(sapovirus)、粘质沙雷氏菌(serratiamarcescens)(例如,esbl/mrgn)、宋氏志贺氏菌(shigellasonnei)、假平胞菌属物种(sphingomonasspecies)、金黄色葡萄球菌(staphylococcusaureus)(例如,甲氧西林(methicillin)抗性金黄色葡萄球菌mrsa、万古霉素(vancomycin)抗性金黄色葡萄球菌(vrsa))、头葡萄球菌(staphylococcuscapitis)、表皮葡萄球菌(staphylococcusepidermidis)(例如,甲氧西林抗性表皮葡萄球菌(mrse))、溶血性葡萄球菌(staphylococcushaemolyticus)、人葡萄球菌(staphylococcushominis)、路邓葡萄球菌(staphylococcuslugdunensis)、巴斯德葡萄球菌(staphylococcuspasteuri)、腐生性葡萄球菌(staphylococcussaprophyticus)、嗜麦芽窄食单胞菌(stenotrophomonasmaltophilia)、肺炎链球菌(streptococcuspneumoniae)、酿脓链球菌(streptococcuspyogenes)(例如,prsp)、链球菌属某些种(streptococcusspp.)、粪类圆线虫(strongyloidesstercoralis)、猪肉绦虫(taeniasolium)、tbe病毒、弓虫(toxoplasmagondii)、梅毒密螺旋体(treponemapallidum)、旋毛线虫(trichinellaspiralis)、阴道毛滴虫(trichomonasvaginalis)、发癣菌属某些种(trichophytonspp.)、毛孢子菌属某些种(trichosporonspp.)、鞭形鞭虫(trichuristrichiura)、冈比亚布氏锥虫(trypanosomabruceigambiense)、罗德西亚布氏锥虫(trypanosomabruceirhodesiense)、克氏锥虫(trypanosomacruzi)、乌苏土病毒(usutuvirus)、牛痘病毒(vacciniavirus)、水痘带状疱疹病毒(varicellazostervirus)、痘疮病毒(variolavirus)、霍乱弧菌(vibriocholerae)、西尼罗病毒(westnilevirus,wnv)、黄热病毒(yellowfevervirus)、小肠大肠炎耶尔辛氏菌(yersiniaenterocolitica)、鼠疫耶尔辛氏菌(yersiniapestis)、假结核病耶尔辛氏菌(yersiniapseudotuberculosis)及兹卡病毒(zikavirus)。14.在至少一个方面,本公开包括用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;选择分类为保守的氨基酸序列的部分,将所选择的保守序列与人蛋白序列进行比较,并进一步将所选择的保守序列分类为与人蛋白序列同一或不同一;以及将与人蛋白序列不同一的选择的保守序列归类为在针对病原体的疗法的开发中的候选抗原。在各种实施方案中,提取可包括例如通过选择序列端点来鉴定、划分或分离序列。在各种实施方案中,提取可包括向序列或序列的一部分分配一个或多个特定特征或状态,例如作为编码序列的状态。在各种实施方案中,提取可包括例如通过观察注释(例如,将参考的相应的序列和/或比对的序列注释为编码序列或非编码序列,和/或注释归类的序列的基因组位置)来鉴定序列、诸如已根据同一性的量度和覆盖率的量度归类的序列实际上是编码序列。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,将所选择的保守序列归类为候选抗原进一步包括确定所选择的保守序列中一个或多个氨基酸结构域的存在或不存在。在某些实施方案中,将所选择的保守序列归类为候选抗原进一步包括确定候选抗原是否对应于在病原体的膜和/或细胞壁内分泌或暴露的蛋白质。在某些实施方案中,将所选择的保守序列归类为候选抗原进一步包括确定所选择的保守序列中跨膜结构域的存在。在某些实施方案中,疗法包括疫苗,并且所述方法进一步包括非临床评价候选抗原的免疫原性。在某些实施方案中,评价步骤包括向动物施用包含候选抗原的多肽,例如,其中动物是人、非人灵长类、小鼠或大鼠。在某些实施方案中,疗法包括抗体疗法,并且所述方法进一步包括产生特异性结合至候选抗原上的表位的抗体或其片段。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(methicillin-resistantstaphylococcusaureus,mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。15.在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法包括产生靶向或结合候选抗原的治疗剂。在某些实施方案中,治疗剂是抗体或抑制剂。在某些实施方案中,治疗剂是shrna或sirna,其对应于核酸序列,诸如编码候选抗原的编码序列。16.在至少一个方面,本公开包括在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变的方法,所述方法包括:在向每个受试者施用治疗剂之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;在比对的氨基酸序列中鉴定在比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一个或多个推定的逃逸突变。在某些实施方案中,参考包括代表典型病原体序列的一种或多种完整或部分病原体基因组序列、病原体的一个或多个临床株系、来自施用治疗剂的一个或多个受试者的病原体的一个或多个早期样品或来自未施用治疗剂的受试者的病原体的一个或多个样品。在某些实施方案中,所述方法进一步包括确定一个或多个推定的逃逸突变是否降低治疗剂与参考多肽的结合亲和性的步骤。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,治疗剂是抗体或抑制剂。在某些实施方案中,治疗剂是shrna或sirna。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,治疗剂包括治疗covid-19的治疗剂。在某些实施方案中,治疗剂包括瑞德西韦(remdesivir)、克力芝(kaletra)、伊维菌素(ivermectin)、特敏福(tamiflu)、阿维根(avigan)、秋水仙碱(colcrys)、地塞米松(dexamethasone)、氯喹(chloroquine)、羟基氯喹、阿奇霉素(azithromycin)、il-6抑制剂(例如,托珠单抗(tocilizumab)和西鲁库单抗(sarilumab))、激酶抑制剂(例如,阿卡拉替尼(acalabrutinib)、依鲁替尼(ibrutinib)、泽布替尼(zanubrutinib)、巴瑞替尼(baricitinib)、鲁索替尼(ruxolitinib)和托法替尼(tofacitinib))、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法包括在鉴定一种或多种推定的逃逸突变之后,向一个或多个受试者施用不同治疗剂。在某些实施方案中,不同治疗剂包括治疗covid-19的治疗剂。在某些实施方案中,不同治疗剂包括瑞德西韦、克力芝、伊维菌素、特敏福、阿维根、秋水仙碱、地塞米松、氯喹、羟基氯喹、阿奇霉素、il-6抑制剂(例如,托珠单抗和西鲁库单抗)、激酶抑制剂(例如,阿卡拉替尼、依鲁替尼、泽布替尼、巴瑞替尼、鲁索替尼和托法替尼)、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。[0017]在至少一个方面,本公开包括向有需要的受试者施用用于治疗病原体感染的治疗剂的方法,所述方法包括:通过以下方式选择氨基酸序列的保守部分:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;以及选择比对的氨基酸序列的保守部分;以及如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的保守部分,则向受试者施用治疗剂,其中治疗剂选择性地结合氨基酸序列的保守部分。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,治疗剂包括治疗covid-19的治疗剂。在某些实施方案中,治疗剂包括瑞德西韦、克力芝、伊维菌素、特敏福、阿维根、秋水仙碱、地塞米松、氯喹、羟基氯喹、阿奇霉素、il-6抑制剂(例如,托珠单抗和西鲁库单抗)、激酶抑制剂(例如,阿卡拉替尼、依鲁替尼、泽布替尼、巴瑞替尼、鲁索替尼和托法替尼)、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0018]在至少一个方面,本公开包括选择用于治疗感染病原体的受试者的治疗剂的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平,将比对的氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表病原体的编码序列的保守部分;以及选择结合保守编码序列的治疗剂作为对感染病原体的受试者的治疗。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,所述方法进一步包括将治疗剂非临床评价为疫苗或它的组分。在某些实施方案中,评价步骤包括向动物施用治疗剂,例如,其中动物是人、非人灵长类、小鼠或大鼠。在某些实施方案中,所述方法进一步包括向感染病原体的受试者施用治疗剂。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,治疗剂包括治疗covid-19的治疗剂。在某些实施方案中,治疗剂包括瑞德西韦、克力芝、伊维菌素、特敏福、阿维根、秋水仙碱、地塞米松、氯喹、羟基氯喹、阿奇霉素、il-6抑制剂(例如,托珠单抗和西鲁库单抗)、激酶抑制剂(例如,阿卡拉替尼、依鲁替尼、泽布替尼、巴瑞替尼、鲁索替尼和托法替尼)、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0019]在至少一个方面,本公开包括用于评价代表病原体的氨基酸序列的部分的保守性的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及使用比对的氨基酸序列鉴定代表病原体的氨基酸序列的一个或多个部分的保守水平。在某些实施方案中,所述部分中的一个或多个被鉴定为在针对病原体的疗法的开发中的候选抗原。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,基因组序列是sars-cov-2基因组序列并且参考序列是sars-cov-2参考序列。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0020]在至少一个方面,本公开包括用于鉴定分离的病原体是否代表循环株系的方法,所述方法包括:从数据结构获得病原体的循环株系的多个完整或部分基因组序列;鉴定循环株系的序列的一个或多个保守部分;获得分离的病原体的多个完整或部分基因组序列;以及通过将分离的病原体的序列的至少一部分与循环株系的序列的鉴定的一个或多个保守部分进行比较来鉴定分离的病原体是否代表循环株系。在某些实施方案中,鉴定循环株系的序列的一个或多个保守部分包括:通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及根据所述部分在比对的氨基酸序列中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法进一步包括储存(例如冷冻)分离的病原体和/或循环株系的样品。在某些实施方案中,所述方法进一步包括从分离的病原体和/或循环株系分离基因组材料和/或储存(例如冷冻)从病原体和/或循环株系分离的基因组材料。在某些实施方案中,所述方法进一步包括如果分离的病原体代表循环株系,则利用和/或维持分离的病原体作为用于研究(例如,用于开发治疗病原体的治疗剂的研究,任选地其中治疗剂可为例如shrna、sirna、抑制剂或抗体)的株系。[0021]在至少一个方面,本公开包括用于鉴定代表病原体的肽的质荷比的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;提供处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;提供处理器将所选择的编码序列转换成相应的氨基酸序列;以及确定一个或多个氨基酸序列或它们的部分的质荷比。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法进一步包括对来自病原体的样品的一种或多种多肽进行质谱法和/或确定来自样品的多肽是否是或包括具有与所确定的质荷比匹配的质荷比的氨基酸序列。[0022]在至少一个方面,本公开包括用于将氨基酸序列鉴定为候选抗生素抗性标记物的方法,所述方法包括:从数据结构获得病原性细菌的多个完整或部分质粒序列;通过计算装置的处理器从质粒序列中提取编码序列;提供处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;提供处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在多个质粒序列中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;选择分类为保守的氨基酸序列部分;以及将所选择的保守序列归类为候选抗生素抗性标记物。在某些实施方案中,所述方法进一步包括根据一个或多个额外标准将候选抗生素抗性标记物鉴定为候选者,所述额外标准包括在所选择的序列中存在跨膜结构域。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得多个完整或部分质粒序列包括通过处理器合并重叠的重叠群以产生至少一些完整或部分质粒序列。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法进一步包括筛选来自一个或多个受试者的一个或多个样品中候选抗生素抗性标记物的存在或不存在,例如,其中一个或多个受试者感染病原性细菌。[0023]在至少一个方面,本公开包括用于鉴定代表质粒的编码序列的一个或多个保守部分的方法,所述方法包括:从数据结构获得病原性细菌的多个完整或部分质粒序列;通过计算装置的处理器从质粒序列中提取编码序列;提供处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;提供处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及根据所述部分在多个质粒序列中的保守水平对氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表质粒的编码序列的一个或多个保守部分。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得多个完整或部分质粒序列包括通过处理器合并重叠的重叠群以产生至少一些完整或部分质粒序列。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法进一步包括筛选来自一个或多个受试者的一个或多个样品中代表质粒的编码序列的保守部分的存在或不存在,例如,其中一个或多个受试者感染病原性细菌。[0024]在至少一个方面,本公开包括用于自动鉴定代表病原体的编码序列的一个或多个保守部分的系统,所述系统包括:处理器;以及其上具有指令的存储器,所述指令在由处理器执行时引起处理器:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表病原体的编码序列的一个或多个保守部分。在某些实施方案中,所述指令当由处理器执行时引起处理器对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,所述指令当由处理器执行时引起处理器创建相似性量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,数据结构包括重叠群,并且其中所述指令当由处理器执行时引起处理器通过处理器合并重叠的重叠群以产生至少一些完整或部分基因组序列来获得病原体的不同株系的多个完整或部分基因组序列。在某些实施方案中,所述指令当由处理器执行时引起处理器评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,所述指令当由处理器执行时引起处理器评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0025]在至少一个方面,本公开包括用于自动鉴定代表质粒的编码序列的一个或多个保守部分的系统,该系统包括:处理器;以及其上具有指令的存储器,所述指令当由处理器执行时引起处理器:从数据结构获得病原性细菌的多个完整或部分质粒序列;通过处理器从质粒序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及根据所述部分在多个质粒序列中的保守水平对氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表质粒的编码序列的一个或多个保守部分。在某些实施方案中,所述指令当由处理器执行时引起处理器对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,所述指令当由处理器执行时引起处理器创建相似性量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,数据结构包括重叠群,并且其中所述指令当由处理器执行时引起处理器通过处理器合并重叠的重叠群以产生至少一些完整或部分质粒序列来获得病原性细菌的多个完整或部分质粒序列。在某些实施方案中,所述指令当由处理器执行时引起处理器评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,所述指令当由处理器执行时引起处理器评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0026]在至少一个方面,本公开包括用于在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变的治疗剂,所述用途包括:在向每个受试者施用治疗剂之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;在比对的氨基酸序列中鉴定比对的氨基酸序列中比参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一种或多种推定的逃逸突变。在某些实施方案中,参考包括代表典型病原体序列的一种或多种完整或部分病原体基因组序列、病原体的一个或多个临床株系、来自施用治疗剂的一个或多个受试者的病原体的一个或多个早期样品或来自未施用治疗剂的受试者的病原体的一个或多个样品。在某些实施方案中,所述用途进一步包括确定一个或多个推定的逃逸突变是否降低治疗剂与参考多肽的结合亲和性的步骤。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述用途包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述用途包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0027]在至少一个方面,本公开包括用于治疗病原体感染的治疗剂,所述用途包括:通过以下方式选择氨基酸序列的保守部分:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;以及选择比对的氨基酸序列的保守部分;以及如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的保守部分,则向受试者施用治疗剂,其中治疗剂选择性地结合氨基酸序列的保守部分。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述用途包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0028]在至少一个方面,本公开包括治疗剂在制造药物中的用途,所述药物用于在向一个或多个受试者施用所述药物以治疗病原体感染之后鉴定一种或多种推定的逃逸突变,所述用途包括:在向每个受试者施用所述药物之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;提供处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;在比对的氨基酸序列中鉴定比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一种或多种推定的逃逸突变。在某些实施方案中,参考包括代表典型病原体序列的一种或多种完整或部分病原体基因组序列、病原体的一个或多个临床株系、来自施用治疗剂的一个或多个受试者的病原体的一个或多个早期样品或来自未施用治疗剂的受试者的病原体的一个或多个样品。在某些实施方案中,所述用途进一步包括确定一个或多个推定的逃逸突变是否降低治疗剂与参考多肽的结合亲和性的步骤。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述用途包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述用途包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0029]在至少一个方面,本公开包括治疗剂用于制造用于治疗病原体感染的药物的用途,所述用途包括:通过以下方式选择氨基酸序列的保守部分:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;以及选择比对的氨基酸序列的保守部分;以及如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的保守部分,则向受试者施用所述药物,其中治疗剂选择性地结合氨基酸序列的保守部分。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述用途包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0030]在至少一个方面,本公开包括确定由抗体结合的病原体表位是否保守的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;将编码序列与编码病原体表位的参考序列进行比较;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;将所选择的编码序列转换成相应的氨基酸序列;以及确定病原体的不同株系中的病原体表位的保守水平。附图说明[0031]本文包括的由下图构成的图仅用于说明目的,而非限制目的。[0032]图1是根据说明性实施方案的示出示例性序列分析工作流程的示意图。[0033]图2是示出当从可公开访问的数据库提取序列时或当手动提供序列以根据本公开的方法或系统进行分析时要提供的一组示例性信息的示意图。[0034]图3是示出根据本公开的方法或系统将数据组织到文件夹中用于分析的示例性系统的示意图。[0035]图4是根据说明性实施方案的示出从一个或多个可公开访问的数据库(例如,ncbi)下载到文件夹中的序列和/或注释信息的拷贝的示例性分布的示意图。如图4所示,将下载的序列和/或注释信息拷贝到三个文件夹中:参考序列文件夹、比对器数据库文件夹和注释文件夹。[0036]图5是根据说明性实施方案的示出用于从示例性可公开访问的数据库(ncbi)下载和策划序列的示例性步骤的示意图。[0037]图6是示出用于输入查询序列以供在本公开的方法或系统中使用的示例性步骤的示意图。[0038]图7是根据说明性实施方案的示出分别存储在查询序列文件夹和比对器数据库文件夹中的查询序列和主题序列(参考序列)的成对blast比较的示例性方法的示意图。[0039]图8是根据说明性实施方案的示出用于应用blast来执行查询序列和主题序列(参考序列)的成对序列比较的示例性步骤的示意图。[0040]图9是根据说明性实施方案的示出用于生成基因输出表(“got表”)的blast结果、序列信息和序列注释信息的示例性编译的示意图。[0041]图10是根据说明性实施方案的示出用于编译blast结果以包括在got表中的示例性步骤的示意图。[0042]图11是根据说明性实施方案的示出用于编译got表中与重叠群相关的信息的示例性步骤的示意图。[0043]图12是根据说明性实施方案的示出用于在成对比较之后鉴定匹配序列、计算匹配序列的突变百分比以及编译可公开访问的数据库(ncbi)中可获得的特征文件注释的示例性步骤的示意图。[0044]图13是根据说明性实施方案的示出got表的示例性内容的示意图。[0045]图14是根据说明性实施方案的示出用于为每个查询序列生成比较表的示例性步骤的示意图,所述比较表包括用于成对比较的相似性评分矩阵,所述相似性评分值基于覆盖率百分比和突变数量来分配。[0046]图15是根据说明性实施方案的示出用于在热图或条形图中表示相似性评分的示例性步骤的示意图。[0047]图16是根据说明性实施方案的示出用于提取编码序列的示例性步骤的示意图,所述提取的序列可经翻译和比对。步骤为重叠群提供一种示例性方法。步骤提供生成表的示例性方法,所述表包括提取的序列的独特型式的数量和频率。[0048]图17是根据说明性实施方案的示出用于从提取的编码序列生成系统发育的示例性方法的示意图。[0049]图18是根据说明性实施方案的示出用于产生got表和可从got表中存在的数据生成的示例性输出的示例性步骤的示意图。[0050]图19是示出在ncbi中代表的并且适合用于根据本文公开的方法和系统的分析的示例性细菌基因组的图形。[0051]图20是示出本文公开的示例性系统的示意图。[0052]图21是表示人感染乙型肝炎病毒(hbv)的示意图,所述感染可导致肝细胞癌。[0053]图22是示出示例性hbv环状基因组的示意图。[0054]图23是示出具有由括号鉴定的基因s的示例性hvc环状基因组的示意图。[0055]图24是示出hbv的基因型的示例性分布的示意图。[0056]图25是示出适合用于根据本公开的方法和系统进行分析的示例性序列结构的示意图,所述示例性序列结构包括手动提供和/或从诸如ncbi的可公开访问的数据库下载的环状、线性和片段化序列。[0057]图26是根据说明性实施方案的代表从基因组序列中提取编码序列的示意图。从基因组序列中提取的编码序列可在不同长度和取向的基因组序列中找到。[0058]图27是根据说明性实施方案的代表来自查询编码序列集合的单个编码序列与多个输入基因组序列中的每一个的示例性成对blast比较、例如来自提取的查询编码序列集合的提取的查询编码序列与作为参考基因组序列的多个主题序列中的每一个的比较的示意图。至少部分由于诸如参考序列的主题序列可在核苷酸序列和内容上变化,所以提取的查询序列与每个参考序列的比对可在比对的相对位置、覆盖长度和/或取向上变化。在一些实施方案中,将不会发现主题序列和参考序列具有相应的序列(即,比较可在一个或多个特定主题基因组序列中产生“无命中”)。在某些实施方案中,从主题基因组序列中提取编码序列,将每个主题编码序列与一个或多个查询基因组序列进行比较(例如,通过blast),并为每个比较确定一个或多个序列归类因子(例如,覆盖长度和同一性百分比)。在各种实施方案中,如果覆盖长度和同一性百分比各自大于各别的阈值,则提取相应的查询序列,并且可进一步对其进行分析或评价。应用阈值来确定每个查询基因组序列或它的部分是否与参考序列相似。本文提供的方法和系统适用于代表完整基因组的基因组序列以及代表完整基因组的一个或多个部分的基因组序列。[0059]图28是根据说明性实施方案的示出单个参考序列与多个输入查询基因组序列中的每一个的成对blast比较、例如多个查询编码序列与作为参考基因组序列的主题基因组序列的比较的结果的示例性汇总的示意图。汇总的第1列指示要与查询基因组序列进行比较的参考基因组序列(b_李(lee)_1940)。具体来说,所示表涉及参考基因组序列的特定基因,其编码在参考基因组序列中注释的特定已知产物血凝素。所述表显示将来自参考基因组的血凝素参考序列与9个查询基因组中的每一个进行比较。归类因子用于确定每个查询基因组中是否存在对应于血凝素的序列(是、否或部分,如“基因存在”列中所指示)。相应查询序列的取向(“链”)也包括在表中。对于每个比较,覆盖率百分比、突变数量(snp)和比对空位在表中注明。[0060]图29是根据说明性实施方案的示出四个示例图的示意图,每个示例图示出与四个查询序列之一相比具有指定数量和类型的变异的主题基因组的数量。[0061]图30是根据说明性实施方案的示出相似性评分的示例性热图的示意图,所述相似性评分代表作为参考基因组序列(x轴)的20个示例性主题序列中的每一个与八个示例性查询编码序列中的每一个之间的保守水平。[0062]图31是根据说明性实施方案的flua当代株系的全基因组系统发育的示例性表示。[0063]图32是根据说明性实施方案的示出呈矩形布局的示例性系统发育的示意图。[0064]图33是根据说明性实施方案的示出呈极地布局的示例性系统发育的示意图。[0065]图34是根据说明性实施方案的示出从基因组序列提取的示例性编码序列的示意图。[0066]图35是根据说明性实施方案的示出图34的示例性编码序列的翻译的示意图,并且包括所分析的基因组内的特定变体序列以及它们的频率的汇总。[0067]图36是根据说明性实施方案的示出源自8个不同的成对比较基因组的氨基酸序列的示例性比对的示意图。[0068]图37是用于提供本文所述的系统和方法的计算机网络环境的示意图。[0069]图38是可用于实现本文所述的系统和方法的计算装置和移动计算装置的示意图。[0070]图39是根据说明性实施方案的用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的示例性方法的方块流程图。[0071]图40是根据说明性实施方案的用于鉴定代表病原体的编码序列的一个或多个保守部分的示例性方法的方块流程图。[0072]图41是根据说明性实施方案的用于鉴定分离的病原体是否代表循环株系的示例性方法的方块流程图。[0073]图42是根据说明性实施方案的用于将氨基酸序列鉴定为候选抗生素抗性标记物的示例性方法的方块流程图。[0074]图43是根据说明性实施方案的用于鉴定代表质粒的编码序列的一个或多个保守部分的示例性方法的方块流程图。[0075]图44是根据说明性实施方案的用于鉴定代表病原体的肽的质荷比、例如用于鉴定所述代表病原体的肽的质谱法靶标的示例性方法的方块流程图。[0076]图45是根据说明性实施方案的用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的示例性方法的方块流程图。[0077]图46是根据说明性实施方案的用于将氨基酸序列鉴定为候选抗生素抗性标记物的示例性方法的方块流程图。[0078]图47是诸如sars-cov-2的示例性冠状病毒的示意图。冠状病毒结构具有外部脂质膜,其包括嵌入的跨膜蛋白,包括但不限于刺突蛋白、套膜蛋白和膜糖蛋白。所述示意图包括与核衣壳蛋白相关的冠状病毒rna病毒基因组的表示。[0079]图48是确定一组查询序列中的主题序列的氨基酸保守性的方法的示意性代表图。从查询序列和主题序列中提取编码序列。执行提取的查询编码序列和提取的主题编码序列的成对blast比较。来自成对blast的数据用于产生数据表,包括归类因子,诸如对于每个成对比较的同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度内的同一性百分比、e值、突变数量和突变百分比。然后,基于一个或多个归类因子的阈值对blast比较结果进行归类。从分析中去除不符合纳入阈值和/或符合排除阈值的类别中的比较。翻译剩余查询序列,并将所得氨基酸序列与相应的翻译的主题序列进行比对。从这些比对评价翻译的查询序列中翻译的主题序列的氨基酸保守性。[0080]图49是说明从参考基因组中提取刺突编码序列的示意图。提取是基于基因库(genbank)文件注释。[0081]图50是示出通过blast与参考刺突编码序列比较的刺突编码序列随时间的累积数量的图形。如取样序列的日期和数量所示,获取并分析大量序列,代表在欧洲、北美、亚洲、大洋洲、南美和非洲分离的序列。[0082]图51是说明刺突氨基酸序列的比对的示意图。基于突变数量和覆盖长度筛选过滤后保留用于分析的编码序列通过blast进行翻译和比对。然后可检查和/或比较比对的序列,以鉴定在参考刺突蛋白序列的每个比对位置存在的氨基酸范围。[0083]图52是部分说明通过分析的编码序列的氨基酸翻译本的比对鉴定的氨基酸变异的示意图。具体实施方式[0084]基因组和质粒序列信息[0085]本公开的方法和系统包括基因组序列和/或质粒序列的分析。基因组序列可包括完整和/或部分基因组序列。质粒序列可包括完整和/或部分质粒序列。不同生物体间的基因组的大小和结构不同。例如,真核基因组通常包括多个染色体,并且原核基因组通常包括单个环状核酸。原核生物另外可包括本领域中称为质粒的较小的独立分子。质粒可编码基因,例如,编码赋予抗生素抗性的蛋白质的基因(抗生素抗性标记物)。本文公开的适用于一种形式的遗传序列信息的各种实施方案也适用于其他形式,例如,关于基因组序列公开的实施方案也适用于质粒序列。[0086]完整基因组序列可包括代表生物体的整个基因组的单个序列。完整基因组序列可包括一起代表生物体的整个基因组的多个序列。部分基因组序列可指代表基因组序列的核酸的邻接亚组的任何单个序列。部分基因组序列可包括一起代表基因组序列的核酸的邻接亚组的多个序列。[0087]在各种实施方案中,基因组序列是病原体基因组的完整或部分序列,例如任何病原性细菌、酵母、原生动物或病毒的完整或部分基因组。例如,在一些实施方案中,基因组序列是冠状病毒(例如严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov2)或中东呼吸综合征相关冠状病毒(mers-cov))的基因组的完整或部分序列。[0088]完整质粒序列可包括代表生物体的整个基因组的单个序列。完整质粒序列可包括一起代表生物体的整个基因组的多个序列。部分质粒序列可指代表质粒序列的核酸的邻接亚组的任何单个序列。部分质粒序列可包括一起代表质粒序列的核酸的邻接亚组的多个序列。[0089]在一些实施方案中,一起代表较大核酸序列的个别序列可称为重叠群。在一些实施方案中,可组装重叠群以提供它们所代表的较大核酸序列的序列。[0090]在各种实施方案中,完整或部分基因组序列可包括至少(例如)约1kb、5kb、10kb、50kb、100kb、500kb、1mb、2mb、3mb、4mb、5mb、10mb、20mb、50mb、100mb、500mb、1,000mb、2,000mb、3,000mb或更多。在各种实施方案中,完整基因组序列可包括与相关生物体的基因组的核苷酸的典型数量相等的核苷酸数量。在各种实施方案中,完整基因组序列可包括在相关生物体的基因组典型的核苷酸数量范围内的核苷酸数量。[0091]在各种实施方案中,完整或部分质粒序列可包括至少(例如)约1kb、5kb、10kb、50kb、100kb、200kb或更多。在各种实施方案中,完整质粒序列可包括与相关质粒的序列的核苷酸的典型数量相等的核苷酸数量。在各种实施方案中,完整基因组序列可包括在相关质粒典型的核苷酸数量范围内的核苷酸数量。[0092]本公开的基因组序列或质粒序列可包括可在可公开访问的数据库获得的一个或多个序列。各种可公开访问的数据库包括可访问的基因组和质粒序列信息(例如,参见图19)。可公开访问的基因组和/或质粒序列信息的数据库的一个实例是国家生物技术信息中心(ncbi)的基因库。另一个可公开访问的基因组和/或质粒序列信息数据库是欧洲分子生物学实验室(europeanmolecularbiologylaboratory,embl)的国际协作核苷酸序列数据库(internationalnucleotidesequencedatabasecollaboration,insdc)(可在ncbi.nlm.nih.gov/sra/万维网上获得)、日本dna数据库(ddbj,dnadatabankofjapan)和ncbi。另一个实例是1000基因组计划(1000genomesproject)。[0093]为了提供可公开访问的基因组序列信息资源扩展的仅一个实例,从2010年8月至2017年8月,公共数据库从约19个金黄色葡萄球菌基因组序列扩展至约48,259个源自约4,155个独立研究的金黄色葡萄球菌基因组序列。大多数序列数据寄存在美国国家生物技术信息中心(ncbi)的序列读段档案中,ncbi是insdc的一部分。在金黄色葡萄球菌基因组序列中,约84%(约42,285)代表短的dna读段(reads)或小片段。剩余部分(约7,974;约16%)组装成更大的dna区段,且仅约2%(约166/7,974)是无空位的并且完全注释的。因此,完全组装和注释的完整基因组序列占ncbi中可获得的金黄色葡萄球菌基因组的一小部分。[0094]本公开的基因组序列或质粒序列可包括源自生物样品且未在可公开访问的数据库中发现的序列。生物样品可包括(例如)实验室样品或临床样品。基因组序列或质粒序列可(例如)通过本领域已知的各种dna测序方法(例如高通量测序和/或多重测序)中的任一种来确定。[0095]数据结构可包括(例如,存储)与本公开的基因组序列和/或质粒序列相关的信息,包括序列本身。因此,本公开的数据结构可包括(但不限于)基因组序列信息的可公开访问的数据库、包括序列信息的私有结构、包括从高通量测序系统直接输入的数据的结构及它们的组合。[0096]代表双链dna的基因组序列可以任一链(有时称为“watson”和“crick”链或称为“5′”和“3′”链)的形式提供。两条链通常应理解为是互补的,使得任一链的序列公开另一链的序列。[0097]可获取多个完整或部分基因组序列和/或质粒序列,包括在数据结构中,并根据本领域已知的各种技术从数据结构获得。从数据结构获得或可获得的基因组序列和/或质粒序列可为来自现有记录(例如,在公共数据库中)的序列和/或通过样品测序获取的序列。在各种实施方案中,数据结构可包括代表特定来源(例如,特定物种,例如,人或特定病原体物种)或与特定来源相关的不同序列。在各种实施方案中,代表特定来源或与特定来源相关的每个不同序列可称为株系。在各种实施方案中,从数据结构获得代表特定来源或与特定源来源相关的多个序列是有利的,使得例如根据本文公开的各种方法和系统可比较和/或对比所获得的序列。[0098]编码序列和编码的氨基酸序列的提取[0099]本公开的基因组和质粒序列可包括编码序列。各种基因组和质粒包括编码可从基因组或质粒表达的蛋白质的氨基酸的核苷酸序列(所述核苷酸序列可称为编码序列)和不编码可从所述序列表达的蛋白质的氨基酸的核苷酸序列(所述核苷酸序列可称为非编码序列)。编码序列可以称为密码子的三联体读取,其中所述密码子的每一个编码一个氨基酸。因此,本公开的编码序列是由密码子组成并编码蛋白质或它的一部分的序列。非编码序列(例如,启动子或内含子)在一些情况下与编码序列相邻和/或散布在编码序列中。可通过本领域已知的多种技术、包括但不限于通过编码氨基酸的邻接和/或框内密码子的数量和/或通过与已知序列(诸如已知编码序列或由编码序列编码的已知蛋白质)的比较,来区分编码序列与非编码序列。提取(鉴定和/或分离)编码序列的各种方法是本领域已知的。提取编码序列的各种方法包括分析为开放阅读框提供的序列,所述开放阅读框除了其他特征之外可包括不包括终止密码子的连续系列密码子,例如不包括终止密码子的至少约20个、30个、40个、50个、60个、70个、80个、90个、100个、125个、150个、175个、200个、250个或300个或更多个密码子的连续系列。在一些实施方案中,可公开访问的数据库中的序列与划分编码序列的位置的注释信息相关。因此,数据库注释和本领域已知的各种方法中的任一种或两种都可用于从基因组和质粒序列中提取编码序列。[0100]一旦提取了编码序列,就可通过应用遗传密码确定由编码序列编码的氨基酸序列。不是终止密码子的每个密码子对应于特定氨基酸。生物体之间的遗传密码可不同。因此,当将编码序列转换成氨基酸序列时,可应用适合于基因组序列或质粒编码序列的来源和/或上下文的遗传密码。通过应用遗传密码已经转换成氨基酸序列的核酸序列可称为核酸序列的翻译。[0101]人类遗传密码与其他遗传密码一样,可表示为dna密码子表,如表1中所见。大多数密码子编码特定氨基酸,而几个密码子编码不编码任何氨基酸的“终止(stop)”信号。表1包括应用于表示核酸和氨基酸序列的某些一般惯例。在提及核酸序列时,字母a、c、g和t分别指示腺嘌呤(a)、胞嘧啶(c)、鸟嘌呤(g)和胸腺嘧啶(t)。关于氨基酸序列,20个氨基酸中的每一个可由如下的特定字母或三个字母的组表示:丙氨酸(a;ala)、精氨酸(r;arg)、天冬酰胺(n;asn)、天冬氨酸(d;asp)、半胱氨酸(c;cys)、谷氨酸(e;glu)、谷氨酰胺(q;gln)、甘氨酸(g;gly)、组氨酸(h;his)、异亮氨酸(i;ile)、亮氨酸(l;leu)、赖氨酸(k;lys)、甲硫氨酸(m;met)、苯丙氨酸(f;phe)、脯氨酸(p;pro)、丝氨酸(s;ser)、苏氨酸(t;thr)、色氨酸(w;trp)、酪氨酸(y;tyr)、缬氨酸(v;val)。[0102]表1[0103][0104]从序列的成对比较生成的数据[0105]在某些实施方案中,本公开的方法和系统包括确定用于表征序列之间的比对的测量。示例性测量包括同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和系统发育(例如,系统发育分组和/或系统发育关系),所有这些都在本文中更详细地讨论。已发现,使用覆盖率的量度(例如,覆盖率百分比和/或覆盖长度)和同一性的量度(例如,同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比)两者来表征比对有效且高效地实现了大量的成对比较,所述成对比较可用于例如在保守性评价中鉴定适当匹配的序列。成对比较可用于评价聚合序列之间、例如核酸序列(例如dna分子和/或rna分子)之间和/或氨基酸序列之间的总体关联性。在本文提供的各种方法和系统中,成对比较用于评价所提取的编码序列和/或它们的翻译之间的总体关联性。在一些实施方案中,两个序列的成对比较是在查询序列和主题序列(例如,参考序列)之间进行,所述比较包括比对和确定同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系)中的一个或多个或全部。在各种实施方案中,诸如参考序列的主题序列可为查询序列与之比较的基线。通常,查询序列和主题序列分别是指一个或多个序列的集合,其中查询序列与主题序列成对比较。在一些实施方案中,不将查询序列与查询序列进行比较,并且不将主题序列与主题序列进行比较,除非查询序列和主题序列具有同一的序列(例如,在其中查询序列和主题序列是序列的同一集合的实施方案中)。主题序列可为或包括参考序列。参考序列可为完整或部分基因组序列,所述完整或部分基因组序列代表群体、物种、株系、生物体等的相应的完整或部分基因组序列,例如其包括一个或多个特定基因或它们的部分和/或编码一个或多个蛋白质或它们的部分。可基于但不限于序列可用性、公众可访问性、历史背景、惯例、准则、标准实践、统计分析、实际考虑或用户偏好中的一个或多个来选择参考序列和/或将其用作代表性序列。如本文所公开的,从序列的成对比较生成的数据可包括同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系)中的一个或多个或全部,其中的每一个提供与所分析的序列相关的不同信息。[0106]在执行查询序列与参考序列的成对比较时,本文发现可显著有效和高效地为给定成对比较确定同一性的测量和覆盖率的测量两者,然后在将查询序列(例如编码序列)归类为两组或多组时使用这两种测量,例如,用于在评价一个或多个氨基酸序列或它们的部分的保守性时鉴定适当可比较的序列部分。同一性的测量的实例包括同一性百分比;同一性百分比/预定覆盖长度;突变数量;和突变百分比(例如单核苷酸多型性snp/大小)。覆盖率的测量的实例包括覆盖率百分比和覆盖长度。[0107]用于比对两种提供的序列的方法包括算法和/或商业上可获得的计算机程序,诸如用于核苷酸序列的blastn和用于氨基酸序列的blastp、有空位的blast和psi-blast。在使用所述比对算法中的一种或多种对两个序列(或一个或两个序列的补体)进行比对后,可计算覆盖率和同一性的量度。在某些实施方案中,为了最佳比对,在第一序列和第二序列中的一个或两个中引入空位,并且为了比较的目的,可忽略不相同的序列。比对是指将两个或多个序列的核苷酸或氨基酸残基进行匹配,以达到最大同一性百分比水平、并且在一些实施方案中(例如,在氨基酸序列的比对中)使物理化学性质的保存最大化的过程或结果。[0108]比对后,可比较第一序列和第二序列的相应位置处的核苷酸或氨基酸。当第一序列中的一个位置被与第二序列中相应位置相同的残基(例如,核苷酸或氨基酸)占据时,则分子在该位置是相同的。两个序列之间的同一性百分比随着序列所共用的相同位置的数量变化,任选地考虑空位的数量和每个空位的长度,为了两个序列的最佳比对,可能需要引入这些空位。因此,确定同一性百分比需要确定比对位置的同一性或非同一性。使用计算算法(诸如blast(基本局部比对搜索工具)),可完成两个序列之间的同一性百分比的确定。[0109]同一性百分比可表示比对序列内两个比对序列中具有相同残基的位置的分数。在一些实施方案中,如果两个序列的相应残基的至少约50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更多在相关序列上同一,则认为两个序列基本上同一。如果序列因保守取代(例如,因不改变编码氨基酸序列的核苷酸取代,或因其中取代的氨基酸具有相似结构或功能特征的氨基酸取代(例如,用同一类型的不同氨基酸置换疏水、亲水、极性或非极性类型的氨基酸))而不同,则序列可基本上相似。[0110]在成对比较中分析的每个序列也可根据通过与第二序列的比对所覆盖的第一序列的百分比(即,与第二序列比对的第一序列的百分比,其可称为覆盖率或覆盖率百分比)(例如,与查询序列比对的主题序列长度的%或与主题序列比对的查询序列长度的%)来评价。[0111]两个序列的比对可产生覆盖长度和/或覆盖率百分比。在第一序列和第二序列的比对中,覆盖长度是指经比对的单元(例如核苷酸或氨基酸)的数量。为免生疑问,在计算覆盖长度时,一对相应的位置(即第一序列的核苷酸或氨基酸和第二序列的相应定位的核苷酸或氨基酸)计数为一个覆盖长度单元。在第一个序列和第二个序列的比对中,覆盖率百分比是指序列的比对中所包括的查询的百分比。覆盖率百分比可指主题序列中与查询序列的相应核苷酸或氨基酸比对的核苷酸或氨基酸的百分比,无论比对的核苷酸或氨基酸是同一的还是不同一的。覆盖率百分比也可指查询序列中与主题序列的相应核苷酸或氨基酸比对的核苷酸或氨基酸的百分比,无论比对的核苷酸或氨基酸是同一的还是不同一的。在本文提供的各种方法和系统中,覆盖率百分比具体来说是指主题序列中与查询序列的相应核苷酸或氨基酸比对的核苷酸或氨基酸的百分比,不管比对的核苷酸或氨基酸是同一的还是不同一的。可确定连续和有空位的比对的覆盖率百分比。[0112]在各种实施方案中,至少由于通过比较比对的核苷酸或氨基酸以确定每个比对的核苷酸或氨基酸对的同一性或非同一性来确定同一性百分比,所以序列空位不会降低同一性百分比。为了提供用于说明目的的一个实例,如果80个氨基酸的查询序列与100个氨基酸的主题序列比对,其中主题序列的前40个氨基酸与查询序列的前40个氨基酸比对具有完美同一性,并且主题序列的后40个氨基酸与查询序列的后40个氨基酸比对具有完美同一性,则同一性百分比将等于100%,但覆盖率百分比将是80%。因此,在一些实施方案中,尽管100%同一性,仍将查询序列归类为部分或“缺乏完整性”,落入70%至95%覆盖率的阈值范围内。[0113]在各种实施方案中,两个序列的比对可用于确定在预定覆盖长度上的同一性百分比。预定覆盖长度可为多个核苷酸和/或氨基酸,其中在预定覆盖长度上的同一性百分比可指查询序列与主题序列在其长度等于预定覆盖长度和/或大于预定覆盖长度的比对的任何部分上的同一性百分比。为免生疑问,比对的部分可为比对的核苷酸或氨基酸的任何足够长的亚组,使得单个比对可包括用于分析的多个足够长的部分,这些部分可为重叠的、不重叠的、相邻的或不相邻的。在各种实施方案中,两个序列的比对在预定覆盖长度上的同一性百分比可表示为与比对的任何足够长的部分相关的最高同一性百分比。[0114]计算同一性百分比的各种技术会产生预计(e)值。例如,使用blast确定同一性百分比会产生e值。e值代表比对偶然发生的可能性(例如,而非生物学意义上的相似性的结果)。一些来源将e值描述为基本上是背景噪声的描述。e值越接近零,比对就越显著。e值至少部分与确定的比对同一性百分比和比对长度相关。大致地,较短和较低的比对同一性百分比将比较长和较高的比对同一性百分比具有更高的e值。e值可用于对多个比对进行排序,或可单独或与其他标准组合选择作为用于归类比对的显著性阈值。[0115]在一些实施方案中,对于成对比较中分析的每个查询序列,可相对于主题序列确定比对内的序列变异的数量。变异可为第一序列和第二序列的比对位置之间的差异,其中所述序列是核酸序列或其中所述序列是氨基酸序列(例如,查询序列和诸如参考序列的主题序列之间的差异)。核酸序列的变异或氨基酸序列的变异在本文中可称为突变。核酸序列的变异可为单核苷酸多型性(“snp”)。[0116]在一些实施方案中,对于成对比较中分析的每个查询序列,查询序列和主题序列之间的序列变异的数量(即,不匹配的查询序列和主题序列之间的比对内序列位置的数量)可称为“突变数量”。在一些实施方案中,对于成对比较中分析的每个查询序列,可确定序列覆盖长度的每个核苷酸或氨基酸的序列变异的数量。此比率可为比对长度上比对内的序列变异的数量(“突变百分比”,在本文中或者称为“突变/大小”,其实例是“snp/大小”)。[0117]在一些实施方案中,成对比较的结果可用于产生一个或多个基因组、质粒、基因、编码序列或翻译的编码序列的系统发育。在一些实施方案中,系统发育可基于通过成对比较产生的同一性百分比数据。在一些实施方案中,系统发育可基于通过成对比较产生的突变百分比数据。用于从所提供的数据产生系统发育的工具和技术是本领域已知的。[0118]使用最保守的主题序列的同一性百分比或突变百分比成对比较结果,可产生基因组水平或质粒水平的系统发育。例如,基因组水平或质粒水平的系统发育可基于约前1个、前2个、前3个、前4个、前5个、前10个、前20个、前25个、前50个、前100个、前1%、前2%、前5%、前10%、前15%、前20%、前25%或前50%的保守的成对比较序列(例如,顶部基因、编码序列或翻译的编码序列氨基酸序列)。可使用例如同一性百分比或突变百分比数据,基于成对比较的结果对保守性进行排序。[0119]同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比中的一个或多个或全部中的任一个可代表核酸或氨基酸比对的全长或它的一个或多个部分。完整或部分基因组序列的示例性部分可包括例如基因、编码序列、个别核苷酸或一组邻接核苷酸(例如,约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、20个、30个、40个、50个、100个、150个、200个、250个、500个、1,000个、1,500个、2,000个、2,500个、3,000个、5,000个、10,000个或更多个核苷酸)。氨基酸序列的示例性部分可包括例如蛋白质、结构域、个别氨基酸或一组邻接氨基酸(例如,约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、20个、30个、40个、50个、100个、150个、200个、250个、300个、350个、400个、450个或500个或更多个氨基酸)。在一些实施方案中,核酸序列的一部分可包括具有约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、20个、30个、40个、50个、100个、150个、200个、250个、500个、1,000个、1,500个、2,000个、2,500个或3,000个核苷酸的下限和约50个、100个、150个、200个、250个、500个、1,000个、1,500个、2,000个、2,500个、3,000个、5,000个、10,000个或更多个核苷酸的上限的核苷酸数。在一些实施方案中,氨基酸序列的部分可包括具有约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、20个、30个、40个、50个、100个、150个、200个、250个或300个氨基酸的下限和约10个、20个、30个、40个、50个、100个、150个、200个、250个、300个、350个、400个、450个或500个或更多个氨基酸的上限的氨基酸数。在各种实施方案中,可个别地分析核酸或氨基酸序列的每个重叠或相邻的非重叠部分。因此,第一比对的核苷酸序列和第二比对的核苷酸序列可具有代表第一比对的序列和第二比对的序列的所有比对的核苷酸之间的同一性百分比的总同一性百分比,并且可具有代表第一比对的序列和第二比对的序列的比对的核苷酸的亚组之间的同一性百分比的一个或多个同一性百分比。第一比对的氨基酸序列和第二比对的氨基酸序列可具有代表第一比对的序列和第二比对的序列的所有比对的氨基酸之间的同一性百分比的总同一性百分比,并且可具有代表第一比对的序列和第二比对的序列的比对的氨基酸的亚组之间的同一性百分比的一个或多个同一性百分比。比对的核苷酸或氨基酸的亚组的同一性百分比可为不同于所有比对的核苷酸或氨基酸的总同一性百分比的百分比。[0120]在各种实施方案中,同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比中的一个或多个或全部中的任一个可显示为图形或热图。在各种实施方案中,图形或热图的至少一个轴包括序列的成对比较中包括的序列,并且至少一个额外轴包括通过序列的成对比较生成的数据。[0121]在一些实施方案中,分析基因组序列的单个集合或质粒序列的单个集合,其中以成对方式比较所分析集合的所有成员(即,单个集合用作查询序列集合和参考序列集合两者),以确定每个成对比较的同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比。在一些实施方案中,分析基因组序列的集合或质粒序列的集合,其中将所分析的集合的每个成员与主题序列进行比较,以确定每个比较的同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比。[0122]在一些实施方案中,集合的每个基因组或质粒序列可属于同一物种。在一些实施方案中,集合的每个基因组或质粒序列可为或包括代表同一属、科、目、纲、门、界或域的生物体的序列。在一些实施方案中,集合的每个基因组或质粒序列可为或包括代表同一基因或它的一部分的序列。在一些实施方案中,单个集合的每个基因组或质粒序列可为或包括代表同一编码序列或它的一部分的序列。[0123]某些实施方案中,分析包括两个集合,每个集合是基因组序列的集合或每个集合是质粒序列的集合。在所述情况下,第一个集合可称为主题集合,并且第二个集合可称为查询集合。在包括主题集合和查询集合的某些实施方案中,以成对方式将查询集合的每个序列与主题集合的每个序列进行比较,以确定每个比较的同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比。[0124]在一些实施方案中,分析包括序列的单个集合,并且以成对方式将每个序列彼此进行比较,使得在至少某些实施方案中,序列的单个集合是主题集合和查询集合两者。无论所分析的序列包括单个序列集合或多个集合(诸如主题集合和查询集合),分析中使用的所有序列都可累积在一起,或者关于它的任何亚组,称为输入序列。[0125]在一些实施方案中,主题集合和/或查询集合的每个基因组或质粒序列可属于同一物种。在一些实施方案中,主题集合和/或查询集合的每个基因组或质粒序列可为或包括代表同一属、科、目、纲、门、界或域的生物体的序列。在一些实施方案中,主题集合和/或查询集合的每个基因组或质粒序列可为或包括代表同一基因或它的一部分的序列。在一些实施方案中,主题集合和/或查询集合的每个基因组或质粒序列可为或包括代表同一编码序列或它的一部分的序列。[0126]在一些实施方案中,一个或多个或全部主题序列可与一个或多个查询序列相当,因为其代表同一物种。在一些实施方案中,一个或多个或全部主题序列可与一个或多个查询序列相当,因为其来自同一属、科、目、类、门、界或域的生物体。在一些实施方案中,一个或多个或全部主题序列可与一个或多个查询序列相当,因为其代表同一的基因或它的一部分。在一些实施方案中,一个或多个或全部主题序列可与一个或多个查询序列相当,因为其代表同一的编码序列或它的一部分。[0127]在一些实施方案中,一个或多个或全部主题序列可在可公开访问的数据库中获得和/或可从可公开访问的数据库中获得。在一些实施方案中,一个或多个或全部主题序列源自生物样品,并且在可公开访问的数据库中没有找到。在一些实施方案中,一个或多个或全部查询序列可在可公开访问的数据库中获得和/或可从可公开访问的数据库中获得。在一些实施方案中,一个或多个或全部查询序列源自生物样品,并且在可公开访问的数据库中没有找到。在一些实施方案中,一个或多个或全部主题序列可在可公开访问的数据库中获得和/或可从可公开访问的数据库中获得;并且一个或多个或全部查询序列源自生物样品,并且在可公开访问的数据库中没有找到。[0128]在一些实施方案中,比较最初输入的基因组或质粒序列。在某些实施方案中,比较最初输入的基因组或质粒序列的提取的编码序列。在某些实施方案中,比较最初输入的基因组或质粒序列的提取的编码序列的翻译。因此,在某些实施方案中,以成对方式将最初输入的查询基因组或质粒序列与最初输入的主题基因组或质粒序列进行比较。在某些实施方案中,以成对方式将最初输入的查询基因组或质粒序列的提取的编码序列与最初输入的主题基因组或质粒序列的提取的编码序列进行比较。在某些实施方案中,以成对方式将最初输入的查询基因组或质粒序列的提取的编码序列的翻译与最初输入的主题基因组或质粒序列的提取的编码序列的翻译进行比较。[0129]通过成对比较生成的数据的处理:用于序列的有效归类的多个序列归类因子的组合[0130]本公开包括使用从成对序列比较生成的数据来有效地对序列进行归类。在各种实施方案中,由成对序列比较得到的数据包括同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育,其中的任一个或全部可个别地或组合(例如以本文所示的组合)用作序列归类因子。因此,在各种实施方案中,可将序列归类到归类的序列组中,所述归类的序列组可基于一个或多个归类因子的一个或多个阈值。在各种实施方案中,归类因子可用于出于任何进一步分析的目的过滤出序列(或者以其他方式将序列排除在进一步考虑之外),例如,其中过滤是基于一个或多个归类因子的阈值和/或过滤出一个或多个归类的序列组,相反地,在各种实施方案中,归类因子可用于选择序列以包括在进一步的分析中,例如,其中选择是基于一个或多个归类因子的阈值和/或一个或多个归类的序列组的选择。在各种实施方案中,由成对序列比较得到的数据可任选地与所分析的序列的序列和/或可用注释(如果有的话)一起编译在例如got表中。[0131]如本文所公开的,成对序列比较可为核酸编码序列(例如,提取的编码序列)的比较或氨基酸序列(例如,提取的编码序列的翻译)的比较。因此,根据本公开的方法和系统归类的查询序列可包括核酸编码序列(例如,提取的编码序列)或氨基酸序列(例如,提取的编码序列的翻译)。[0132]在各种实施方案中,出于任何进一步分析的目的,可基于同一性百分比是否等于和/或低于阈值来对序列进行归类或过滤。在各种实施方案中,可基于同一性百分比是否等于和/或高于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值同一性百分比可等于或为至少约(例如)75%、80%、85%、90%、95%、96%、97%、98%、99%或100%。在各种实施方案中,阈值同一性百分比可在具有(例如)75%、80%、85%、90%或95%的下限和(例如)80%、85%、90%、95%、96%、97%、98%、99%或100%的上限的范围内。[0133]在各种实施方案中,出于任何进一步分析的目的,可基于覆盖率百分比是否等于和/或低于阈值来对序列进行归类或过滤。在各种实施方案中,可基于覆盖率百分比是否等于和/或高于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值覆盖率百分比可等于或为至少约(例如)75%、80%、85%、90%、95%、96%、97%、98%、99%或100%。在各种实施方案中,阈值覆盖率百分比可在具有(例如)75%、80%、85%、90%或95%的下限和(例如)80%、85%、90%、95%、96%、97%、98%、99%或100%的上限的范围内。[0134]在各种实施方案中,出于任何进一步分析的目的,可基于覆盖长度是否等于和/或低于阈值来对序列进行归类或过滤。在各种实施方案中,可基于覆盖长度是否等于和/或高于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值覆盖长度可等于或为至少约(例如)20个、25个、30个、35个、40个、45个、50个、75个、100个、125个、150个、175个或200个核苷酸或氨基酸。在各种实施方案中,阈值覆盖长度可在具有(例如)20个、25个、30个、35个、40个、45个、50个、75个、100个、125个、150个或175个核苷酸或氨基酸的下限和(例如)25个、30个、35个、40个、45个、50个、75个、100个、125个、150个、175个或200个核苷酸或氨基酸的上限的范围内。[0135]在各种实施方案中,出于任何进一步分析的目的,可基于预定覆盖长度上的同一性百分比是否等于和/或低于阈值来对序列进行分归或过滤。在各种实施方案中,可基于在预定覆盖长度上的同一性百分比是否等于和/或高于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,在预定覆盖长度上的示例性阈值同一性百分比可为(例如)在等于或为至少约20个、25个、30个、35个、40个、45个、50个、75个、100个、125个、150个、175个或200个核苷酸或氨基酸的预定覆盖长度上等于或为至少约75%、80%、85%、90%、95%、96%、97%、98%、99%或100%的同一性百分比。在各种实施方案中,在预定覆盖长度上的阈值同一性百分比可包括在具有(例如)75%、80%、85%、90%或95%的下限和(例如)80%、85%、90%、95%、96%、97%、98%、99%或100%的上限的范围内的同一性百分比,并且可包括在具有(例如)20个、25个、30个、35个、40个、45个、50个、75个、100个、125个、150个或175个核苷酸或氨基酸的下限和(例如)25个、30个、35个、40个、45个、50个、75个、100个、125个、150个、175个或200个核苷酸或氨基酸的上限的范围内的覆盖长度。[0136]在各种实施方案中,出于任何进一步分析的目的,可基于e值是否等于和/或高于阈值来对序列进行归类或过滤。在各种实施方案中,可基于e值是否等于和/或低于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值e值可等于或为至少约(例如)1e-50、1e-40、1e-30、1e-20、1e-10、1e-9、1e-8、1e-7、1e-6、1e-5、1e-4、1e-3或1e-2。在各种实施方案中,示例性阈值e值可在具有(例如)1e-50、1e-40、1e-30、1e-20、1e-10、1e-9、1e-8、1e-7、1e-6、1e-5、1e-4或1e-3的下限和(例如)1e-40、1e-30、1e-20、1e-10、1e-9、1e-8、1e-7、1e-6、1e-5、1e-4、1e-3或1e-2的上限的范围内。[0137]在各种实施方案中,出于任何进一步分析的目的,可基于突变数量是否等于和/或高于阈值来对序列进行归类或过滤。在各种实施方案中,可基于突变数量是否等于和/或低于阈值来对序列进行归类,或选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值突变数量可等于或为至少约(例如)1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、35、40、45或50。在各种实施方案中,阈值突变数量可在具有(例如)1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、35、40或45的下限和(例如)2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、35、40、45或50的上限的范围内。[0138]在各种实施方案中,出于任何进一步分析的目的,可基于突变百分比是否等于和/或高于阈值来对序列进行归类或过滤。在各种实施方案中,可基于突变百分比是否等于和/或低于阈值来对序列进行归类,或选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值突变百分比可等于或为至少约(例如)0%、1%、2%、3%、4%、5%、10%、15%、20%或25%。在各种实施方案中,阈值突变百分比可在具有(例如)0%、1%、2%、3%、4%、5%、10%、15%或20%的下限和(例如)1%、2%、3%、4%、5%、10%、15%、20%或25%的上限的范围内。[0139]在各种实施方案中,为了任何进一步分析的目的,可基于系统发育对序列进行归类或过滤。在各种实施方案中,出于任何进一步分析的目的,过滤掉一个或多个分支。在各种实施方案中,选择一个或多个分支以包括在进一步的分析中。[0140]本公开包括基于来自成对序列比较的两个或多个归类因子的序列的归类。在各种实施方案中,序列的归类基于两个或多个归类因子,所述归类因子选自同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比。本公开进一步包括其中基于两个或多个归类因子的参数(例如,一个或多个阈值)来产生归类的序列组的实施方案。在一些实施方案中,对每个序列类别分配一数值。在各种实施方案中,分配给序列类别的数值可为利用测量查询序列和主题序列之间的相似性的一个或多个归类因子来跟踪的值,并且/或可称为“相似性评分”。相似性评分可包括跨任何范围的任何系列数值,但在特定实施方案中可包括0至1、0至10或0至100的范围。本文提供了相似性评分的实例。[0141]在各种实施方案中,本公开基于两个或个多归类因子对序列进行归类,所述两个或多个归类因子包括作为同一性的测量的第一归类因子和作为覆盖率的测量的第二归类因子。在各种实施方案中,同一性的测量可选自同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比。在各种实施方案中,覆盖率的测量可选自覆盖率百分比和覆盖长度。[0142]在各种实施方案中,可基于定义的评分系统对成对比较中分析的每个序列分配相似性评分,其中成对比较中分析的每个序列根据覆盖率百分比和序列变异的数量来归类或排序。例如,可根据下表2对序列进行归类并分配相似性评分,其中基于来自查询序列与特定主题序列的比较的数据,将在与特定主题序列的成对比较中分析的每个查询序列分配到其所属的具有最高相似性评分的库:[0143]表2[0144]覆盖率百分比突变数量分配的相似性评分≥99%=01≥99%《100.95≥99%≥100.8≥90%(任何)0.5≥75%(任何)0.4》0%(任何)0.3=0%(任何)0[0145]表2中的值应进一步理解为提供了所提供值周围的范围,例如,如同表2中的每个值前面都有术语“约”。一些或所有成对比较的序列的相似性评分可以矩阵、热图或图形(诸如条形图)显示。例如,包括单元列和单元行的矩阵或热图可包括用于每个主题序列的列和用于每个查询序列的行,其中每个单元基于查询序列和主题序列的比较来显示相似性评分。[0146]在一些实施方案中,出于任何进一步分析的目的,可过滤出不能满足一个或多个阈值标准或值(例如,阈值相似性评分)的成对序列比较(和/或它们的查询序列)(或以其他方式从进一步考虑中排除)。在一些实施方案中,出于任何进一步分析的目的,可过滤出与特定查询序列和特定主题序列(和/或相关的查询序列)的成对序列比较相关的数据(其中所述数据不能满足一个或一个以上阈值标准或值(例如,阈值相似性评分))(或以其他方式从进一步考虑中排除)。[0147]在一些实施方案中,出于任何进一步分析的目的,可过滤出落入如本文所示的一个或多个特定归类的序列组中的成对序列比较(和/或它们的查询序列或主题序列)(或以其他方式从进一步考虑中排除)。在一些实施方案中,出于任何进一步分析的目的,可过滤出与特定查询序列和特定主题序列(和/或相关的查询序列)的成对序列比较相关的数据(其中所述数据和/或序列落入一个或多个特定归类的序列组)(或以其他方式从进一步考虑中排除)。[0148]表2提供了允许通过相似性评分过滤归类的序列组的示例性归类方案。如表2的示例性归类方案中所示,对导致至少约99%的覆盖率百分比的成对比较(其中突变数量为零)分配1的相似性评分;对导致至少约99%的覆盖率百分比的剩余成对比较(其中突变数量小于约10)分配0.95的相似性评分;对导致至少约99%的覆盖率百分比的剩余成对比较(其中突变数量为至少10)分配0.8的相似性评分;对导致至少约90%但小于约99%的覆盖率百分比的剩余成对比较(包括任何突变数量)分配0.5的相似性评分;对导致至少约75%但小于约90%的覆盖率百分比的剩余成对比较(包括任何突变数量)分配0.4的相似性评分;对导致至少约0%但小于约75%的覆盖率百分比的剩余成对比较(包括任何突变数量)分配0.3的相似性评分;对导致等于0%的覆盖率百分比的剩余成对比较(包括任何突变数量)分配0的相似性评分。[0149]在某些实施方案中,出于任何进一步分析的目的,例如通过过滤以排除具有小于1、小于0.95、小于0.8、小于0.5、小于0.4、小于0.3或0的分配的相似性评分的序列比较,可过滤出如表2中所示归类的(或如由覆盖率和同一性的另一组合量度归类的)一个或多个序列比较中的任一个(或以其他方式从进一步考虑中排除)。在某些实施方案中,在将一个或多个阈值分配给对应于如表2中所述的相似性评分(或作为覆盖率的量度和同一性的量度的组合的其他相似性评分)的类别之前或之后(或之前和之后两者),将所述一个或多个阈值应用于成对比较。在某些实施方案中,一个或多个阈值可包括(例如)最小覆盖长度、最小覆盖率百分比、最大e值、最小同一性百分比、覆盖长度上的最小同一性百分比、最大突变数量和/或最大突变百分比。在某些实施方案中,应用一个或多个阈值作为基于表2的过滤的替代。在某些实施方案中,一个或多个阈值可包括例如最小覆盖长度、最小覆盖率百分比、最大e值、最小同一性百分比、覆盖长度上的最小同一性百分比、最大突变数量和/或最大突变百分比。[0150]在一些实施方案中,除了基于表2的归类和/或过滤之外或作为基于表2的归类和/或过滤的替代,可包括展现在至少约51个核苷酸或氨基酸的覆盖长度上至少约80%同一性且e值为约0.001或低于约0.001的成对序列比较用于进一步分析,和/或自分析过滤出展现小于约80%同一性和/或约50个或更少核苷酸或氨基酸的比对匹配长度和/或大于约0.001的e值的成对序列比较。[0151]靶特征的确定和/或具有靶特征的序列的选择[0152]在各种实施方案中,本公开的方法和系统可用于确定一个或多个序列是否显示某些靶特征,和/或选择经确定具有一个或多个靶特征的序列。如本文进一步公开的,示例性靶特征可包括但不限于序列保守的靶水平、序列可变性的水平(例如,跨序列集合和/或与一个或多个主题序列相比)或系统发育分组。[0153]在各种实施方案中,归类和/或过滤步骤之后是用于分析靶特征的一个或多个其他步骤,任选地包括选择具有靶特征的序列。在其中比较和归类和/或过滤核酸序列(例如,提取的编码序列)的一些实施方案中,通过将核酸(例如,提取的编码序列)翻译成氨基酸序列并任选地进行氨基酸序列与一个或多个主题氨基酸序列的进一步成对比较来进行靶特征分析。在其中比较和归类和/或过滤核酸序列(例如,提取的编码序列)的一些实施方案中,通过分析来自成对核酸序列比较的数据进行靶特征分析。在比较、归类和/或过滤氨基酸序列的一些实施方案中,通过分析来自成对氨基酸序列比较的数据来进行靶特征分析。[0154]可相对于基因组、质粒、基因、编码序列或翻译的编码序列氨基酸序列中的任一种或多种来评价(例如,测量或确定)保守性和/或可变性。可关于编码序列的核苷酸位置的亚组(例如编码氨基酸结构域的编码序列的核苷酸位置的亚组)来评价保守性和/或可变性。可关于编码序列内的一个或多个核苷酸位置来评价保守性和/或可变性。可相对于翻译的编码序列氨基酸序列的氨基酸位置的亚组(例如包括氨基酸结构域的氨基酸位置的亚组)来评价保守性和/或可变性。可相对于翻译的编码序列氨基酸序列内的一个或多个氨基酸位置来评价保守性和/或可变性。[0155]多种方法可用于分析序列保守性和/或可变性。如本文所公开的,序列保守性和/或可变性可指在所比较的序列中的一个或多个相应位置处的核苷酸或氨基酸的同一性或非同一性的频率的量度。至少在序列保守性和序列可变性两者都是序列之间或之中的相似性的量度的范围内,用于测量一者的方法通常适用于测量两者。[0156]在一些实施方案中,可根据突变百分比测量序列保守性和/或可变性。在一些实施方案中,序列保守性和/或可变性可根据同一性百分比来测量。在各种实施方案中,保守性和/或可变性可通过同一性的量度和覆盖率的量度的组合来确定。例如,在各种实施方案中,如果序列同时满足同一性的量度的阈值和覆盖率的量度的阈值,则将所述序列鉴定为保守的。在一些实施方案中,可根据突变百分比与覆盖长度和/或覆盖率百分比的组合来测量序列保守性和/或可变性。在一些实施方案中,可根据同一性百分比与覆盖长度和/或覆盖率百分比的组合来测量序列保守性和/或可变性。在一些实施方案中,可根据相似性评分(例如,如表2中所例示)来测量序列保守性和/或可变性。[0157]在一些实施方案中,对应于特定主题编码序列的序列的保守性可通过平均每个序列与特定主题编码序列相比的同一性百分比来确定。在各种实施方案中,基于至少85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、99.5%、99.6%、99.7%、99.8%、99.9%或100%的平均同一性百分比选择具有高保守性(低可变性)的序列。在一些实施方案中,基于小于99%、98%、97%、96%、95%、94%、93%、92%、91%、90%、85%、80%、75%、70%、65%、60%、55%、50%、40%或30%的平均同一性百分比选择具有低保守性(高可变性)的序列。[0158]在各种实施方案中,可基于序列的测量的保守性和/或可变性水平选择序列。在一些实施方案中,选择具有高保守性(低可变性)的序列,例如,在根据保守性的量度对成对比较的序列进行排序之后,选择约前1个、前2个、前3个、前4个、前5个、前10个、前20个、前25个、前50个、前100个、前1%、前2%、前5%、前10%、前15%、前20%、前25%或前50%的保守的成对比较序列(例如,顶部基因、编码序列或翻译的编码序列)。在一些实施方案中,选择具有低保守性(高可变性)的序列,例如,在根据保守性的量度对成对比较的序列进行排序之后,选择约后1个、后2个、后3个、后4个、后5个、后10个、后20个、后25个、后50个、后100个、后1%、后2%、后5%、后10%、后15%、后20%、后25%或后50%的保守的成对比较序列(例如,底部基因、编码序列、翻译的编码序列)。[0159]在各种实施方案中,通过系统发育分析展现序列保守性。用于系统发育分析的各种方法和程序包括:ancestree、aligroove、ape、armadillo工作流程平台、bali-phy、batwing、bayesphylogenies、bayestraits、beast、bionumerics、bosque、bucky、canopy、citup、clustalw、dendroscope、ezeditor、fastdnaml、fasttree2、fitmodel、geneious、hyphy、iqpnni、iq-tree、jmodeltest2、lisbeth、mega、mesquite、metapiga2、modelgenerator、molphy、morphobank、mrbayes、network、nona、paml、paraphylo、partitionfinder、pastis、paup*、phangorn、phybase、phyclust、phylip、phylot、phyloquart、phylowgs、phyml、phyx、poy、prottest3、pycogent、quicktree、raxml-hpc、raxml-ng、semphy、sowhat、splitstree、tnt、topali、treegen、treealign、treefinder、tree-puzzle、t-rex(webserver)、ugene、winclada和xrate。[0160]网络环境和计算装置[0161]如图37所示,示出并描述用于提供如本文所述的系统、方法和架构的网络环境3700的实现。简而言之,现在参考图37,示出并描述了示例性云计算环境3700的方块图。云计算环境3700可包括一个或多个资源提供者3702a、3702b、3702c(统称为3702)。每个资源提供者3702可包括计算资源。在一些实施方式中,计算资源可包括用于处理数据的任何硬件和/或软件。例如,计算资源可包括能够执行算法、计算机程序和/或计算机应用的硬件和/或软件。在一些实施方式中,示例性计算资源可包括具有存储和检索能力的应用服务器和/或数据库。每个资源提供者3702可连接到云计算环境3700中的任何其他资源提供者3702。在一些实施方式中,资源提供者3702可通过计算机网络3708连接。每个资源提供者3702可通过计算机网络3708连接到一个或多个计算装置3704a、3704b、3704c(统称为3704)。[0162]云计算环境3700可包括资源管理器3706。资源管理器3706可通过计算机网络3708连接到资源提供者3702和计算装置3704。在一些实施方式中,资源管理器3706可促进由一个或多个资源提供者3702向一个或多个计算装置3704提供计算资源。资源管理器3706可从特定计算装置3704接收对计算资源的请求。资源管理器3706可鉴定能够提供由计算装置3704请求的计算资源的一个或多个资源提供者3702。资源管理器3706可选择资源提供者3702来提供计算资源。资源管理器3706可促进资源提供者3702和特定计算装置3704之间的连接。在一些实施方式中,资源管理器3706可在特定资源提供者3702和特定计算装置3704之间确立连接。在一些实施方式中,资源管理器3706可将特定计算装置3704重新定向到具有所请求的计算资源的特定资源提供者3702。[0163]图38示出了可用于实现本公开中描述的技术的计算装置3800和移动计算装置3850的实例。计算装置3800旨在代表各种形式的数字计算机,诸如膝上型计算机、台式计算机、工作站、个人数字助理、服务器、刀片服务器、大型机和其他适当的计算机。移动计算装置3850旨在代表各种形式的移动装置,诸如个人数字助理、蜂窝电话、智能电话和其他相似的计算装置。本文所示的组件、它们的连接和关系以及它们的功能意味着仅是实例,而非意味着具有限制性。[0164]计算装置3800包括处理器3802、存储器3804、存储装置3806、连接到存储器3804和多个高速扩展端口3810的高速接口3808,以及连接到低速扩展端口3814和存储装置3806的低速接口3812。处理器3802、存储器3804、存储装置3806、高速接口3808、高速扩展端口3810和低速接口3812中的每一个都使用各种总线互连,并且可安装在公共母板上或以其他适当的方式安装。处理器3802可处理用于在计算装置3800内执行的指令,包括存储在存储器3804中或存储装置3806上的指令,以在外部输入/输出装置(诸如耦合到高速接口3808的显示器3816)上为gui显示图形信息。在其他实施方式中,可适当地使用多个处理器和/或多条总线以及多个存储器和多个类型的存储器。此外,可连接多个计算装置,每个装置提供必要操作的部分(例如,作为服务器阵列、一组刀片服务器或多处理器系统)。因此,在将多个功能描述为由处理器执行的情况下,这涵盖其中多个功能由任何数量的计算装置(一个或多个)中的任何数量的处理器(一个或多个)执行的实施方案。此外,在将功能描述为由处理器执行的情况下,这涵盖其中功能由任何数量的计算装置(一个或多个)(例如,在分布的计算系统中)中的任何数量的处理器(一个或多个)执行的实施方案。[0165]存储器3804存储计算装置3800内的信息。在一些实施方式中,存储器3804是一个或多个易失性存储器单元。在一些实施方式中,存储器3804是一个或多个非易失性存储器单元。存储器3804也可为另一种形式的计算机可读媒体,诸如磁盘或光盘。[0166]存储装置3806能够为计算装置3800提供大容量存储。在一些实施方式中,存储装置3806可为或含有计算机可读媒体,诸如软盘装置、硬盘装置、光盘装置或磁带装置、快闪存储器或其他类似的固态存储装置或装置阵列,包括存储区域网络或其他配置中的装置。指令可存储在信息载体中。当由一个或多个处理装置(例如,处理器3802)执行时,所述指令执行一种或多种方法,诸如上述那些方法。指令也可由一个或多个存储装置(诸如计算机可读媒体或机器可读媒体(例如,存储器3804、存储装置3806或处理器3802上的存储器))存储。[0167]高速接口3808管理计算装置3800的带宽密集型操作,而低速接口3812管理较低带宽密集型操作。这种功能分配仅是一实例。在一些实施方式中,高速接口3808耦合到存储器3804、显示器3816(例如,通过图形处理器或加速器)以及可接受各种扩展卡(未示出)的高速扩展端口3810。在所述实施方式中,低速接口3812耦合到存储装置3806和低速扩展端口3814。可包括各种通信端口(例如,usb、以太网、无线以太网)的低速扩展端口3814可例如通过网络适配器耦合到一个或多个输入/输出装置,诸如键盘、定点装置、扫描仪或联网装置(诸如交换机或路由器)。[0168]如图所示,计算装置3800可以多种不同的形式实现。例如,其可实现为标准服务器3820,或者在一组这样的服务器中多次实现。此外,其可在诸如膝上型计算机3822的个人计算机中实现。其也可实现为机架服务器系统3824的一部分。或者,来自计算装置3800的组件可与移动装置(未示出)(诸如移动计算装置3850)中的其他组件相结合。所述装置中的每一个可含有计算装置3800和移动计算装置3850中的一个或多个,并且整个系统可由多个彼此通信的计算装置构成。[0169]移动计算装置3850包括处理器3852、存储器3864、诸如显示器3854的输入/输出设备、通信接口3866和收发器3868以及其他组件。移动计算装置3850还可提供有存储装置,诸如微驱动器或其他装置,以提供额外的存储。处理器3852、存储器3864、显示器3854、通信接口3866和收发器3868中的每一个都使用各种总线互连,并且这些组件中的几个可安装在公共母板上或者以其他适当的方式安装。[0170]处理器3852可执行移动计算装置3850内的指令,包括存储在存储器3864中的指令。处理器3852可实现为包括单独的和多个模拟和数字处理器的芯片的芯片组。处理器3852可提供(例如)移动计算装置3850的其他组件的协调,诸如控制用户接口、由移动计算装置3850运行的应用以及由移动计算装置3850的无线通信。[0171]处理器3852可通过控制接口3858和耦合到显示器3854的显示接口3856与用户通信。显示器3854可为例如tft(薄膜晶体管液晶显示器)显示器或oled(有机发光二极管)显示器,或其他适当的显示技术。显示器接口3856可包括用于驱动显示器3854向用户呈现图形和其他信息的适当电路。控制接口3858可从用户接收命令,并转换它们以提交给处理器3852。此外,外部接口3862可提供与处理器3852的通信,以便实现移动计算装置3850与其他装置的近区通信。例如,外部接口3862可在一些实施方式中提供有线通信,或者在其他实施方式中提供无线通信,并且也可使用多个接口。[0172]存储器3864存储移动计算装置3850内的信息。存储器3864可实现为一个或多个计算机可读媒体、一个或多个易失性存储器单元或一个或多个非易失性存储器单元中的一者或多者。还可提供扩展存储器3874,并通过扩展接口3872连接到移动计算装置3850,所述扩展接口可包括例如simm(单列直插式存储模块)卡接口。扩展存储器3874可为移动计算装置3850提供额外的存储空间,或者也可为移动计算装置3850存储应用或其他信息。具体地,扩展存储器3874可包括执行或补充上述过程的指令,并且还可包括安全信息。因此,例如,扩展存储器3874可提供作为移动计算装置3850的安全模块,并且可用允许安全使用移动计算装置3850的指令来编程。此外,可通过simm卡提供安全应用以及额外信息,诸如以不可破解的方式在simm卡上放置鉴定信息。[0173]存储器可包括例如快闪存储器和/或nvram存储器(非易失性随机存取存储器),如下所讨论的。在一些实施方式中,指令存储在信息载体中。当由一个或多个处理装置(例如,处理器3852)执行时,所述指令执行一个或多个方法,诸如上述那些方法。指令也可由一个或多个存储装置(诸如一个或多个计算机可读媒体或机器可读媒体(例如,存储器3864、扩展存储器3874或处理器3852上的存储器))存储。在一些实施方式中,可在传播的信号中例如通过收发器3868或外部接口3862接收指令。[0174]移动计算装置3850可通过通信接口3866进行无线通信,必要时所述通信接口可包括数字信号处理电路。通信接口3866可提供各种模式或协议下的通信,诸如gsm语音呼叫(全球移动通信系统)、sms(短消息服务)、ems(增强消息服务)或mms消息服务(多媒体消息服务)、cdma(码分多址)、tdma(时分多址)、pdc(个人数字蜂窝)、wcdma(宽带码分多址)、cdma2000或gprs(通用分组无线服务)等。例如,这种通信可通过使用射频的收发器3868进行。此外,短距离通信可诸如使用wi-fitm或其他此类收发器(未示出)进行。此外,gps(全球定位系统)接收器模块3870可向移动计算装置3850提供额外导航相关的和位置相关的无线数据,这些数据可由移动计算装置3850上运行的应用适当地使用。[0175]移动计算装置3850还可使用音频编译码器3860进行听觉通信,所述音频编译码器可从用户接收口头信息并将其转换成可用的数字信息。音频编译码器3860同样可为用户生成听觉声音,诸如通过例如移动计算装置3850的电话听筒中的扬声器。这种声音可包括来自语音电话呼叫的声音,可包括记录的声音(例如,语音消息、音乐文件等)并且还可包括由在移动计算装置3850上操作的应用生成的声音。[0176]如图所示,移动计算装置3850可以多种不同的形式实现。例如,其可实现为蜂窝电话3880。其也可实现为智能电话3882、个人数字助理或其他类似移动装置的一部分。[0177]图20中提供了包括示例性系统的某些组件的另一非限制性示意图。[0178]本文描述的系统和技术的各种实现可在数字电子电路、集成电路、专门设计的asic(应用专用集成电路)、计算机硬件、固件、软件和/或它们的组合中实现。这些各种实现可包括在一个或多个计算机程序中的实现,所述一个或多个计算机程序在可编程系统上是可执行的和/或可解释的,所述可编程系统包括至少一个可编程处理器、至少一个输入装置和至少一个输出装置,所述可编程处理器可为专用的或通用的,经耦合以从存储系统接收数据和指令,以及将数据和指令发送到存储系统。[0179]所述计算机程序(也称为程序、软件、软件应用或代码)包括用于可编程处理器的机器指令,并且可以高级程序语言和/或面向对象的编程语言和/或以汇编/机器语言来实现。机器可读媒体和计算机可读媒体可指用于向可编程处理器提供机器指令和/或数据的计算机程序产品、设备和/或装置(例如,磁盘、光盘、存储器、可编程逻辑装置(pld)),包括接收机器指令作为机器可读信号的机器可读媒体。机器可读信号可指用于向可编程处理器提供机器指令和/或数据的信号。[0180]在某些实施方案中,计算机程序包括一个或多个机器学习模块。机器学习模块可指实现一个或多个特定机器学习算法的计算机实现的过程(例如,功能)。机器学习模块可包括例如一个或多个人工神经网络。在某些实施方案中,两个或多个机器学习模块可组合并实现为单个模块和/或单个软件应用。在某些实施方案中,两个或多个机器学习模块也可单独实现,例如,作为单独软件应用。机器学习模块可为软件和/或硬件。例如,机器学习模块可完全实现为软件,或者机器学习模块的某些功能可经由专用硬件(例如,经由应用专用集成电路(asic))来执行。[0181]为了提供与用户的交互,本文描述的系统和技术可在计算机上实现,所述计算机具有用于向用户显示信息的显示装置(例如,crt(阴极射线管)或lcd(液晶显示器)监视器)以及用户可通过其向计算机提供输入的键盘和定点装置(例如,鼠标或轨迹球)。也可使用其他种类的装置来提供与用户的交互;例如,提供给用户的反馈可为任何形式的感觉反馈(例如,视觉反馈、听觉反馈或触觉反馈);并且可以任何形式接收来自用户的输入,包括听觉、语音或触觉输入。[0182]本文描述的系统和技术可在计算系统中实现,所述计算系统包括后端组件(例如,作为数据服务器),或者包括中间件组件(例如,应用服务器),或者包括前端组件(例如,具有用户可通过其与本文描述的系统和技术的实现交互的图形用户接口或网络浏览器的客户端计算机),或者这种后端组件、中间件组件或前端组件的任何组合。系统的组件可通过任何形式或媒体的数字数据通信(例如,通信网络)互连。通信网络的实例包括局域网(lan)、广域网(wan)和互联网。[0183]计算系统可包括客户端和服务器。客户端和服务器通常彼此远离,并且通常通过通信网络进行交互。客户端和服务器的关系是通过在相应计算机上运行并且彼此之间具有客户端-服务器关系的计算机程序产生。[0184]各种实施方案的方块流程图[0185]图39是用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的示例性方法的方块流程图3900。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0186]在步骤3910中,获得(访问)病原体的不同株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0187]在步骤3920中,从基因组序列中鉴定编码序列。在步骤3930中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0188]在步骤3940中,将编码序列转换成氨基酸序列,并且在步骤3950中,将氨基酸序列进行比对。在某些实施方案中,氨基酸序列凭借经比对的编码序列进行比对。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0189]在步骤3960中,根据在步骤3910中访问的多个基因组序列所代表的病原体的不同株系之间的序列部分的保守水平来对氨基酸序列的比对部分进行分类。特别感兴趣的是高度保守并且因此对于由在步骤3910中访问的多个基因组序列所代表的病原体的各种株系是常见的那些序列部分。[0190]在步骤3970中,检查被鉴定为高度保守的每个氨基酸序列部分,以确定其是否与人蛋白序列同一。由于毒性问题,任何与人蛋白序列同一的高度保守序列均被作为候选抗原消除。其他标准也可应用于在针对病原体的疗法的开发中鉴定一种或多种最终候选抗原,例如肽信号的存在、蛋白质注释(或其存在/不存在)、特定结构域结构和/或序列中跨膜结构域的存在,后者可指示候选抗原是否对应于在病原体的膜和/或细胞壁内分泌或暴露的蛋白质,从而增强其作为针对病原体的治疗剂的潜在价值。所述方法可另外包括将涵盖候选抗原的多肽施用于动物的步骤。此外,当疗法是疫苗的情况下,所述方法可包括非临床评价候选抗原的免疫原性的步骤。[0191]图40是用于鉴定代表病原体的编码序列的一个或多个保守部分的示例性方法的方块流程图4000。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0192]在步骤4010中,从数据结构获得(访问)病原体的不同株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0193]在步骤4020中,从基因组序列中鉴定编码序列。在步骤4030中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0194]在步骤4040中,将编码序列转换成氨基酸序列。在某些实施方案中,编码序列在根据同一性百分比和覆盖率百分比归类后转换成氨基酸序列。在其他实施方案中,编码序列在根据同一性百分比和覆盖率百分比归类之前转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0195]在步骤4050中,根据在步骤4010中访问的多个基因组序列所代表的病原体的不同株系之间的序列部分的保守水平来对氨基酸序列的部分进行分类。特别感兴趣的是高度保守并且因此对于由在步骤4010中访问的多个基因组序列所代表的病原体的各种株系是常见的那些序列部分。[0196]图41是用于鉴定分离的病原体是否代表循环株系的示例性方法的方块流程图4100。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0197]在步骤4110中,获得(访问)病原体的循环株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0198]在步骤4120中,鉴定循环株系的序列的一个或多个保守(例如高度保守)部分。在某些实施方案中,循环株系的序列根据同一性百分比和覆盖率百分比进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个(其中“查询”序列和“主题”序列两者都是病原体的循环株系的序列),计算查询编码序列和每个主题序列之间的相似性的量度,其中相似性的量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0199]在步骤4130中,获得(访问)分离的病原体的多个完整或部分基因组序列。例如,分离的病原体的序列可来自从头测序读段(例如,从患有感染的患者获得的生物样品的高通量测序读段)。在某些实施方案中,可如上分析这些序列以鉴定哪些部分是保守的并且适当地代表分离的病原体。[0200]在步骤4140中,将分离的病原体的一个或多个序列(或它们的部分)与步骤4120中鉴定的循环株系的序列的一个或多个保守(例如高度保守)部分进行比较,从而鉴定分离的病原体是否代表循环株系(例如,对于循环株系的发病率是常见的)。[0201]图42是根据说明性实施方案的用于将氨基酸序列鉴定为候选抗生素抗性标记物(例如,在针对病原性细菌的疗法的开发中)的示例性方法的方块流程图。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0202]在步骤4210中,从数据结构获得(访问)病原性细菌的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0203]在步骤4220中,从质粒序列中鉴定编码序列。在步骤4230中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0204]在步骤4240中,将编码序列转换成氨基酸序列,并且在步骤4250中,将氨基酸序列进行比对。在某些实施方案中,氨基酸序列凭借经比对的编码序列进行比对。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0205]在步骤4260中,根据在步骤4210中访问的多个质粒序列中的序列部分的保守水平来对氨基酸序列的比对部分进行分类。特别感兴趣的是高度保守并且因此对于由步骤4210中访问的多个基因组序列所代表的病原体的质粒是常见的那些序列部分。[0206]在步骤4270中,选择鉴定为保守(例如,高度保守)的一个或多个序列部分作为候选抗生素抗性标记物。其他标准也可应用于鉴定候选抗生素抗性标记物,例如肽信号的存在、蛋白质注释(或它的存在/不存在)、特定结构域结构和/或序列中跨膜结构域的存在。所述方法可另外包括向动物施用涵盖候选抗生素抗性标记物的多肽的步骤。此外,当疗法是疫苗的情况下,所述方法可包括非临床评价肽的免疫原性的步骤。[0207]图43是根据说明性实施方案的用于鉴定代表质粒的编码序列的一个或多个保守部分的示例性方法的方块流程图4300。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0208]在步骤4310中,从数据结构获得(访问)病原性细菌的多个完整或部分质粒序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0209]在步骤4320中,从质粒序列中鉴定编码序列。在步骤4330中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0210]在步骤4340中,将编码序列转换成氨基酸序列。在某些实施方案中,编码序列在根据同一性百分比和覆盖率百分比归类后转换成氨基酸序列。在其他实施方案中,编码序列在根据同一性百分比和覆盖率百分比归类之前转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0211]在步骤4350中,根据在步骤4310中访问的多个质粒序列中的序列部分的保守水平来对氨基酸序列的部分进行分类。特别感兴趣的是高度保守并且因此对于由步骤4310中访问的多个基因组序列所代表的病原体的质粒是常见的那些序列部分。[0212]图44是用于鉴定代表病原体的肽的质荷比、例如用于鉴定这种代表病原体的肽的质谱法靶标的示例性方法的方块流程图。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0213]在步骤4410中,获得(访问)病原体的不同株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0214]在步骤4420中,从基因组序列中鉴定编码序列,并且在步骤4430中,将编码序列转换成氨基酸序列。在步骤4440中,鉴定氨基酸序列的一个或多个保守部分。例如,序列可根据同一性百分比和覆盖率百分比进行归类。例如,对于与一组主题序列进行比较的一组查询序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0215]在步骤4450中,确定鉴定为保守的一个或多个序列部分的质荷比。例如,这有助于鉴定相应代表病原体的肽的质谱法靶标,从而可通过质谱法对其进行鉴定。[0216]图45是用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的示例性方法的方块流程图。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0217]在步骤4510中,获得(访问)病原体的不同株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0218]在步骤4520中,从基因组序列中鉴定编码序列。在步骤4530中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0219]在步骤4540中,将编码序列转换成氨基酸序列。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0220]在步骤4550中,根据在步骤4510中访问的多个基因组序列所代表的病原体的不同株系之间的序列部分的保守水平来对氨基酸序列的部分进行分类。特别感兴趣的是高度保守并且因此对于由在步骤4510中访问的多个基因组序列所代表的病原体的各种株系是共同的那些序列部分。[0221]在步骤4560中,检查被鉴定为高度保守的每个氨基酸序列部分,以确定其是否与人蛋白序列同一。由于毒性问题,任何与人蛋白序列同一的高度保守序列均被作为候选抗原消除。其他标准也可应用于在针对病原体的疗法的开发中鉴定一种或多种最终候选抗原,例如肽信号的存在、蛋白质注释(或其存在/不存在)、特定结构域结构和/或序列中跨膜结构域的存在,后者可指示候选抗原是否对应于在病原体的膜和/或细胞壁内分泌或暴露的蛋白质,从而增强其作为针对病原体的治疗剂的潜在价值。所述方法可另外包括将涵盖候选抗原的多肽施用于动物的步骤。此外,当疗法是疫苗的情况下,所述方法可包括非临床评价候选抗原的免疫原性的步骤。[0222]图46是根据说明性实施方案的用于将氨基酸序列鉴定为候选抗生素抗性标记物的示例性方法4600的方块流程图。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0223]在步骤4610中,从数据结构获得(访问)病原性细菌的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0224]在步骤4620中,从质粒序列中鉴定编码序列。在步骤4630中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0225]在步骤4640中,将编码序列转换成氨基酸序列。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0226]在步骤4650中,根据在步骤4610中访问的多个质粒序列中的序列部分的保守水平来对氨基酸序列的部分进行分类。特别感兴趣的是高度保守并且因此对于由步骤4610中访问的多个基因组序列所代表的病原体的质粒是常见的那些序列部分。[0227]在步骤4660中,选择鉴定为保守(例如,高度保守)的一个或多个序列部分作为候选抗生素抗性标记物。其他标准也可应用于鉴定候选抗生素抗性标记物,例如肽信号的存在、蛋白质注释(或它的存在/不存在)、特定结构域结构和/或序列中跨膜结构域的存在。所述方法可另外包括向动物施用涵盖候选抗生素抗性标记物的多肽的步骤。此外,当疗法是疫苗的情况下,所述方法可包括非临床评价肽的免疫原性的步骤。[0228]本文描述的不同实施方式的要素可经组合以形成上文没有具体阐述的其他实施方式。要素可排除在本文描述的方法、过程、计算机程序、数据库等之外,而不会不利地影响它们的操作。各种单独的要素可组合成一个或多个单独的要素,以执行本文描述的功能。[0229]考虑要求保护的本发明的系统、架构、装置、方法和过程涵盖使用来自本文描述的实施方案的信息开发的变异和适应。如本说明书所考虑,可执行本文描述的系统、架构、装置、方法和过程的适应和/或修改。[0230]在整个说明书中,当物品、装置、系统和架构描述为具有、包括或包含特定组件的情况下,或者当过程和方法描述为具有、包括或包含特定步骤时,考虑另外存在基本上由所述组件组成或由所述组件组成的本发明的物品、装置、系统和架构,并且存在基本上由所述处理步骤组成或由所述处理步骤组成的根据本发明的过程和方法。[0231]应理解,只要本发明仍然可操作,步骤的顺序或执行某些动作的顺序是无关紧要的。此外,两个或多个步骤或动作可同时进行。[0232]本文提及任何出版物,例如在背景部分,并非承认该出版物作为关于本文提出的任何权利要求的现有技术。背景部分是为了清楚的目的而呈现的,并不意味着对关于任何权利要求的现有技术的描述。[0233]提供标题是为了方便读者–标题的存在和/或放置不打算限制本文描述的主题物的范围。[0234]应用[0235]本公开的表征输入序列之间、之中和/或之内的残基亚组的序列保守性的方法和系统可用于各种分析和治疗应用中。本文提供表征序列保守性的方法和系统的各种用途。例如,本文公开的方法和系统可例如基于序列保守性特征用于鉴定未表征的序列的治疗相关性。提供了本文公开的方法和系统的效用的非限制性实例。[0236]用于选择抗抗原抗体的抗原的鉴定[0237]在特定物种(诸如病原体物种)的实例中,基因组和质粒核酸序列(包括编码序列)可变。在许多情况下,源自特定物种成员的核酸序列的可变性可通过分析可公开获得的基因组序列和/或其他基因组序列(诸如非公开测序数据)来揭示。随着单独寄存在可公开访问的数据库中的序列数量不断增加,成功分析不断增长的不同序列信息变得越来越具挑战性。本公开的方法和系统通过提供分析输入序列的保守性特征的系统性方法来解决这个困难。[0238]作为用于产生抗病原体治疗剂的抗原来源,病原体基因组的保守序列可优于病原体基因组的非保守序列。抗原的鉴定和/或表征可为或包括表位的鉴定和/或表征。抗原可为或包括表位,并且本文公开的用于鉴定抗原的一个或多个特征同样可用于鉴定表位。至少一个原因是,与相关病原体群体内相对保守的序列结合或以其他方式相互作用的治疗性抗体或其他药物分子将必然更可能在病原体物种的更宽范围的成员中具有治疗益处,并因此在遭受所述病原体物种困扰的患者中具有治疗益处。因此,将通过本公开的方法和系统鉴定的在相关病原体群体中保守的序列鉴定为用于开发治疗性抗体的候选抗原或鉴定为用于其他治疗模式的靶标,诸如小分子药物。用于开发针对治疗性抗原的抗体的某些方法是本领域已知的,并且可包括(仅提供一个实例)用感兴趣的抗原对产生抗体的生物体进行免疫。[0239]在各种实施方案中,通过次要考虑因素,鉴定为保守的序列可进一步缩小范围以鉴定治疗相关靶标。一个次要考虑因素是鉴定的候选治疗靶标是否与已知的人序列同一。可使用可公开获得的数据库和搜索工具来确定所鉴定的序列是否与已知的人序列同一。目前公开的方法和系统的各种实施方案包括从候选治疗靶标中(例如,从候选抗原列表中)去除与已知人序列同一的候选治疗靶标。去除与已知人序列同一的序列的至少一个原因是,靶向这种序列的药物(例如抗体)的开发可表现出与非靶向人细胞和/或蛋白质的临床上有害的或另外不希望的相互作用。[0240]次要考虑因素的其他实例包括蛋白质注释、功能和/或蛋白质结构域的存在或不存在。蛋白质结构域的实例包括信号序列、已知引起分泌或与分泌相关的结构域、细胞膜蛋白质的特征性结构域、指示序列在细胞膜或细胞壁处的细胞外暴露的特征或其他结构特征。序列的细胞外暴露促进治疗剂与序列的相互作用,并且因此是治疗靶标中可需要的特征。[0241]在某些实施方案中,上述信息(例如通过本文提供的方法鉴定候选抗原)用于开发一种或多种用于治疗病原体引起的疾病的组合物(或鉴定一种或多种新的和/或现有的组合物)。在某些实施方案中,鉴定和/或开发涉及多种药物组合物(例如,药物混合物)的疗法。例如,本文提供的方法可用于选择可用于供治疗病原体引起的疾病(诸如covid-19)的药物(例如,药物混合物)中的最佳一种或多种病原体中和抗体。在一些实施方案中,所述药物不是对疾病的治疗,而是例如用于疫情中的权宜之计,以增强人体(例如,免疫受损或以其他方式易受感染的个体)抵抗感染的能力,例如直到开发出疫苗。在一些实施方案中,所述药物干扰病原体(例如,诸如sars-cov2的病毒)的功能以防止或减少由所述病毒引起的对人体的损害,例如,由此减少患者使用呼吸机和/或其他呼吸装置的需要。在一些实施方案中,药物是为特定个体或个体组定制的治疗。在某些实施方案中,小鼠或其他动物可用于制造用于治疗病原体引起的疾病的组合物,其中在这种制造中使用通过本文提供的计算机实施的方法产生的信息。例如,可向小鼠或其他动物注射用于产生人抗体的病毒(或它的部分),所述人抗体可被制造并施用给一个或多个患者。在某些实施方案中,可从鉴定病毒或其他病原体的序列进行到产生抗体,所述抗体可使用本文所述的方法大规模生产。[0242]在某些实施方案中,本文提供的方法用于评价编码蛋白质的核酸的编码序列、编码蛋白质的核酸序列的保守序列、编码蛋白质的核酸的非保守序列(特征在于变异的序列)、特定蛋白质内的保守结构域和/或特定蛋白质内的非保守结构域(特征在于变异的部分),例如,其中所述蛋白质与病原体相关。这种评价然后用于开发用于治疗、预防或改善由病原体引起的疾病的抗体、进入抑制剂、疫苗和/或其他治疗剂。例如,在某些实施方案中,本文提供的方法用于评价与sars-cov2宿主细胞(诸如人或蝙蝠血管收缩肽-转化酶2(ace2)受体)上的受体结合以促进宿主细胞感染的sars-cov2刺突(s)蛋白或其受体结合结构域(rbd),或编码所述蛋白的核酸序列。因此,例如,本说明书包括使用本文提供的计算机实现的方法来分析sars-cov2刺突(s)蛋白或其rbd,以鉴定可用于开发抗体、进入抑制剂、疫苗和/或其他治疗剂以治疗、预防或改善由sars-cov2病毒(即covid-19)引起的疾病的序列。[0243]在某些实施方案中,本文提供的方法用于评价编码sars-cov2刺突蛋白或其受体结合结构域(rbd)的核酸的编码序列、编码sars-cov2刺突蛋白或其rbd蛋白的核酸序列的保守序列、编码sars-cov2刺突蛋白或其rbd蛋白的核酸的非保守结构域(特征在于变异的序列)、特定sars-cov2刺突蛋白或其rbd的保守结构域和/或sars-cov2刺突蛋白或其rbd的非保守结构域(特征在于变异的部分)。在某些实施方案中,本文提供的方法用于评价编码冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的核酸的编码序列、编码冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的核酸序列的保守序列、编码冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的核酸序列的非保守序列(特征在于变异的序列)、特定冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的保守结构域和/或冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的非保守结构域(特征在于变异的部分)。[0244]候选疫苗抗原的鉴定[0245]疫苗包括施用以刺激接受者产生针对病原体的抗体(疫苗抗原)的非病原性物质。疫苗抗原可为由病原体呈递的肽。疫苗功效要求,如果接受者稍后被感染,则接受者因应疫苗抗原产生的抗体能够结合病原体。由于病原体的株系可不同,所以当疫苗抗原具有保守序列或由保守序列编码时,疫苗提供针对最广泛范围的病原体株系的免疫性。如本文关于鉴定用于选择抗抗原抗体的抗原所公开,本公开的方法和系统可用于鉴定保守的病原体序列。因此,使用本公开的方法和系统鉴定的保守的病原体序列可用作疫苗抗原和/或候选疫苗抗原。候选疫苗抗原可在临床上适当的免疫和感染动物模型中验证,并在临床试验中例如针对安全性和功效进一步验证。[0246]代表性样品的鉴定[0247]尽管已知或可能在临床样品中存在多种病原体的许多株系,但出于实际和/或历史原因,研究往往聚焦于一种或少数株系。然而,在治疗剂的开发中,使用代表病原体的临床样品、优选许多或大多数临床样品的研究株系促进发现具有广泛临床功效的治疗剂。本公开提供可用于将一种或多种研究株系的序列与来自其他株系(例如,不同临床分离株)的不同序列集合进行比较以表征一种或多种研究株系的基因组与其他株系相比的保守性的方法和系统。研究株系的序列的保守性指示,分析的研究株系或研究株系序列代表所有或大量的比较株系。因此,根据本公开的方法和系统在分析中展现保守性的研究株系或研究株系序列适用于临床相关研究。相反,根据本公开的方法和系统在分析中未展现保守性的研究株系或研究株系序列对于临床相关研究可能不是最佳的。[0248]抗生素抗性标记物的鉴定[0249]病原性细菌的抗生素抗性成为临床上日益关注的问题。例如,抗性感染更有可能导致死亡。细菌通过两种主要途径获取对抗生素的抗性:染色体突变和通过水平基因转移获取可移动的遗传元件,诸如质粒。质粒是独立于染色体复制并能够通过接合在细菌之间水平转移的基因组外环状dna分子。因此,质粒在许多病原体中的抗生素抗性散播中起着重要作用。[0250]本文提供的方法和系统可应用于鉴定指示和/或引起病原性细菌抗体抗性的遗传和/或氨基酸序列(抗体抗性标记物)。本文提供的方法和系统可应用于质粒序列以鉴定保守序列。因此,质粒的保守序列鉴定为候选抗生素抗性标记物。此外,质粒的保守序列是开发破坏或中和质粒赋予的抗生素抗性的治疗剂的候选靶标。[0251]用于质谱法的肽发现资源的生成[0252]质谱法基于精确测量的质荷比鉴定分析的物质。肽的质荷比取决于肽序列。至少部分由于质荷比复杂,质谱法分析可通过将检测的质荷比与预计质荷比的集合进行比较来鉴定肽。因此,质谱法不能鉴定意外序列。由于特定物种的生物体(例如,临床相关的病原体分离株)的基因组和蛋白质组改变,因此无法鉴定意外肽可阻碍不同样品的分析。[0253]本公开的方法和系统可通过分析代表感兴趣的物种(例如,临床相关的病原体)的不同基因组的保守特征,为质谱法提供肽发现资源。例如,根据本公开的方法和系统的分析可鉴定序列多样性的区域,所述区域可用于修订用于查询质谱法数据的预计质荷比的集合。因此,掺入通过本公开的方法和系统鉴定的不同序列可增强质谱法发现样品中的肽的能力,例如发现临床相关的病原体肽。[0254]为了提供一个特定实例,主要组织相容性复合体i相关蛋白具有临床相关性,并且可通过质谱法发现,基于预计质荷比的适当集合来分析所提供的数据。主要组织相容性复合体(人的mhc或hla)在所有有核细胞的细胞表面上表达,并作为获得性免疫系统中t细胞抗原呈递的机制起作用。其用于在细胞表面上显示加工的自身和外源蛋白(抗原)的肽片段,以供由t淋巴细胞(对于mhci类为cd8 细胞毒性t淋巴细胞(ctl),并且对于mhcii类为cd4 辅助t淋巴细胞)检查。表征该过程中涉及的抗原有助于鉴定治疗上有用的靶标,例如作为用于开发治疗性抗体的抗原。质谱法是一种可用于鉴定mhc呈递的抗原的技术。然而,如果质谱法分析不是为了检测存在的抗原而设计的,则不能检测mhc呈递的抗原。本文公开的方法和系统可用于生成预计质荷比的包含性集合,以查询靶病原体的mhc呈递的抗原的质谱法数据。[0255]基因组、基因和蛋白质(例如抗原)中多样性区域的鉴定[0256]如本文所公开的,所提供的方法和系统可用于鉴定基因组、基因和蛋白质内的多样性区域。多样性区域(与其他区域相比保守性较低的区域)可指示可适合于更实质性的实验室操作(例如,实验室引入的序列修饰)的核苷酸或氨基酸位置。在某些生物学背景下,序列多样性的特征对生物学功能至关重要,例如在免疫球蛋白的可变区情况即如此。多样性还可指示可用于系统发育分析的区域,这是因为与分析相对更保守的序列相比,多样性区域可在相同或更短的时间内为系统发育分析提供更大数量的序列变异。多样性也可指示比保守序列更近期经历进化发展的序列。[0257]引起流行病学的病原体的系统发育的生成[0258]文公开的方法和系统可用于生成系统发育。系统发育对于分析来自病原体(例如,快速进化的病原体)的序列特别有用。系统发育可用于描述诸如人免疫缺失病毒(hiv)等病原体的分子流行病学和传播、严重急性呼吸综合征(sars)相关冠状病毒(例如,严重急性呼吸综合征相关冠状病毒(sars-cov);严重急性呼吸综合征冠状病毒2(sars-cov2),其是导致冠状病毒疾病(covid19)的病毒;中东呼吸综合征相关冠状病毒(mers-cov))的起源和随后的进化、禽流行性感冒以及季节性和疫情人流感的进化流行病学。可使用系统发育确定的信息的实例包括对新病原体株系的起源或其在新物种中出现的实际时间、病原体重组和重配事件、病原体流行中种群大小变化的速率以及病原体如何在特定种群和地理区域内传播和进化的估计(具有置信界限)。[0259]基因组研究确认,突变和可移动遗传元件的获取可显著影响微生物克隆的病理学。事实上,即使是不大的基因改变也可对宿主-病原体相互作用以及病原体的抗体鉴定产生巨大影响。宿主内进化不仅对患者有意义,而且对在医院出于流行病学目的确立区分株系中的关联性的阈值也有意义。微生物遗传多样性、免疫调节和由个别株系的损害可显著变化。因此,捕获克隆的宽度以解释基因组水平上宿主-病原体相互作用多样性的程序,将可能产生对微生物病原体生物学的独特理解。这种理解促进了更有效和个性化方法的开发,以预防感染和改善病原体管理。[0260]从系统发育获得的序列源信息可帮助设计和实现公共卫生和治疗干预措施。例如,如应用于hbv,本公开的方法和系统可用于确定特定株系(例如,实验室株系)属于哪个hbv谱系,确定跨hbv谱系的一个或多个hbv基因或蛋白质(例如,hbsag)的遗传多样性,确定自然界中存在的hbv或hbv基因或蛋白质(例如,hbsag)的遗传变体的数量和宽度,和/或确定hbv基因组或它的遗传或编码蛋白质序列(例如,hbsag的遗传或编码蛋白质序列)的哪个部分是遗传保守的。在另一个实例中,本文公开的方法和系统可用于确定特定患者感染哪种株系和/或这种株系的定义遗传特征和/或特定患者感染的株系的抗生素抗性特征。在另一个实例中,本文公开的方法和系统可用于确定病原体基因组(例如埃博拉病毒基因组)的遗传多样性,并确定所测量的变异是否具有临床分支。[0261]直系同源基因的鉴定[0262]直系同源物是由共同的祖先dna序列衍生而来的不同物种的同源序列。物种间的比较遗传学至少部分基于如下事实:认为直系同源物在功能上是物种间相关的。尽管详细的分析通常可确立直系同源物鉴定的准确度,但基因组信息的大量分析增加了直系同源物鉴定的误差率。因此,需要区分真实直系同源物和错误注释的直系同源物的改良的方法。如本文所公开的,本公开的方法和系统可用于表征序列保守性。因此,本公开的方法和系统可用于提高直系同源物鉴定的准确度,并且/或用于鉴定和校正现有的直系同源物错误注释。根据本文所公开的方法和系统的直系同源物的鉴定可用于通过将新序列或未表征的序列与先前注释的序列进行比对并将先前的注释应用于直向同源的新序列或未表征的序列来注释新序列或未表征的序列。[0263]用于选择抗体疗法、鉴定推定的逃逸突变体和个体化药物的表位序列变异的评价[0264]在各种实施方案中,评价特定基因或蛋白质或它的一部分的变异是有用的。例如,在抗体疗法的背景下,许多重要的问题可通过评价抗体的抗原和/或表位的变异来解决。[0265]本说明书的各种实施方案包括疗法和/或治疗剂。在各种实施方案中,疗法和/或治疗剂可为或包括小干扰rna(sirna)或短发夹rna(shrna)。在各种实施方案中,疗法和/或治疗剂可为或包括抗体。在各种实施方案中,疗法和/或治疗剂可为或包括治疗covid-19的疗法和/或治疗剂。治疗covid-19的示例性疗法和/或治疗剂可包括瑞德西韦、克力芝、伊维菌素、特敏福、阿维根、秋水仙碱、地塞米松、氯喹、羟基氯喹、阿奇霉素、il-6抑制剂(例如,托珠单抗和西鲁库单抗)、激酶抑制剂(例如,阿卡拉替尼、依鲁替尼、泽布替尼、巴瑞替尼、鲁索替尼和托法替尼)、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。示例性抗体可包括结合sars-cov-2的刺突蛋白用于covid-19疗法的抗体,例如,如美国专利第10,787,501号中所公开,所述专利全文通过引用、特别是关于covid-19治疗性抗体及它们的表位和其他性质并入本文。美国专利第10,787,501号的表1提供了示例性抗sars-cov-2-刺突蛋白(sars-cov-2-s)抗体和抗体序列,其全文通过引用具体并入本文。还参见下表3:[0266]表3[0267][0268][0269][0270][0271][0272][0273][0274][0275][0276][0277][0278][0279][0280][0281][0282][0283][0284][0285][0286][0287][0288]表1的抗体包括多特异性分子,例如抗体或抗原结合片段,其分别包括这些抗体的cdr-h和cdr-l、vh和vl或hc和lc(包括如在本文中所示的它们的变体)。[0289]在一实施方案中,可包括在多特异性分子中的特异性结合cov-s的抗原结合结构域包含:[0290](1)[0291](i)重链可变结构域序列,其包含表1中所示的cdr-h1、cdr-h2和cdr-h3氨基酸序列,和[0292](ii)轻链可变结构域序列,其包含表1中所示的cdr-l1、cdr-l2和cdr-l3氨基酸序列;[0293]或,[0294](2)[0295](i)重链可变结构域序列,其包含表1中所示的氨基酸序列,和[0296](ii)轻链可变结构域序列,其包含表1中所示的氨基酸序列;[0297]或,[0298](3)[0299](i)重链免疫球蛋白序列,其包含表1中所示的氨基酸序列,和[0300](ii)轻链免疫球蛋白序列,其包含表1中所示的氨基酸序列。[0301]在各种实施方案中,本公开提供特异性结合冠状病毒刺突蛋白(cov-s)的分离的重组抗体或它的抗原结合片段,其中所述抗体具有以下特征中的一个或多个:(a)以小于约10-9m的ec50结合cov-s;(b)展现与未进行施用的可比较的冠状病毒感染的动物相比,在向冠状病毒感染的动物施用后,所述冠状病毒感染的动物的存活率增加;和/或(c)包含包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(cdr-h1、cdr-h2和cdr-h3),所述重链可变区包含与表1的hcvr具有至少约90%序列同一性的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链cdr(cdr-l1、cdr-l2和cdr-l3),所述轻链可变区包含与表1的lcvr具有至少约90%序列同一性的氨基酸序列。[0302]在各种实施方案中,刺突蛋白与以下序列(seqidno:108)具有至少80%同一性(例如,至少80%、90%、95%、96%、97%、98%、99%或100%同一性):[0303]mfvflvllplvssqcvnlttrtqlppaytnsftrgvyypdkvfrssvlhstqdlflpffsnvtwfhaihvsgtngtkrfdnpvlpfndgvyfasteksniirgwifgttldsktqsllivnnatnvvikvcefqfcndpflgvyyhknnkswmesefrvyssannctfeyvsqpflmdlegkqgnfknlrefvfknidgyfkiyskhtpinlvrdlpqgfsaleplvdlpiginitrfqtllalhrsyltpgdsssgwtagaaayyvgylqprtfllkynengtitdavdcaldplsetkctlksftvekgiyqtsnfrvqptesivrfpnitnlcpfgevfnatrfasvyawnrkrisncvadysvlynsasfstfkcygvsptklndlcftnvyadsfvirgdevrqiapgqtgkiadynyklpddftgcviawnsnnldskvggnynylyrlfrksnlkpferdisteiyqagstpcngvegfncyfplqsygfqptngvgyqpyrvvvlsfellhapatvcgpkkstnlvknkcvnfnfngltgtgvltesnkkflpfqqfgrdiadttdavrdpqtleilditpcsfggvsvitpgtntsnqvavlyqdvnctevpvaihadqltptwrvystgsnvfqtragcligaehvnnsyecdipigagicasyqtqtnsprrarsvasqsiiaytmslgaensvaysnnsiaiptnftisvtteilpvsmtktsvdctmyicgdstecsnlllqygsfctqlnraltgiaveqdkntqevfaqvkqiyktppikdfggfnfsqilpdpskpskrsfiedllfnkvtladagfikqygdclgdiaardlicaqkfngltvlpplltdemiaqytsallagtitsgwtfgagaalqipfamqmayrfngigvtqnvlyenqklianqfnsaigkiqdslsstasalgklqdvvnqnaqalntlvkqlssnfgaissvlndilsrldkveaevqidrlitgrlqslqtyvtqqliraaeirasanlaatkmsecvlgqskrvdfcgkgyhlmsfpqsaphgvvflhvtyvpaqeknfttapaichdgkahfpregvfvsngthwfvtqrnfyepqiittdntfvsgncdvvigivnntvydplqpeldsfkeeldkyfknhtspdvdlgdisginasvvniqkeidrlnevaknlneslidlqelgkyeqyikwpwyiwlgfiagliaivmvtimlccmtsccsclkgccscgscckfdeddsepvlkgvklhyt[0304]在一些实施方案中,本公开提供结合sars-cov-2刺突蛋白的分离的抗体或它的抗原结合片段,所述sars-cov-2刺突蛋白包含seqidno:108中所示的氨基酸序列,其中所述分离的抗体或抗原结合片段包含包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(hcdr1、hcdr2和hcdr3),所述重链可变区包含seqidno:29中所示的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链互补决定区(cdr)(lcdr1、lcdr2和lcdr3),所述轻链可变区包含seqidno:33中所示的氨基酸序列。[0305]在一些实施方案中,hcdr1包含seqidno:30中所示的氨基酸序列,hcdr2包含seqidno:31中所示的氨基酸序列,hcdr3包含seqidno:32中所示的氨基酸序列,lcdr1包含seqidno:34中所示的氨基酸序列,lcdr2包含seqidno:35中所示的氨基酸序列,并且lcdr3包含seqidno:36中所示的氨基酸序列。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:29中所示的氨基酸序列的hcvr。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:33中所示的氨基酸序列的lcvr。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:29中所示的氨基酸序列的hcvr和包含seqidno:33中所示的氨基酸序列的lcvr。[0306]在一些实施方案中,本公开提供结合sars-cov-2刺突蛋白的分离的抗体,所述sars-cov-2刺突蛋白包含seqidno:108中所示的氨基酸序列,其中所述分离的抗体包含免疫球蛋白恒定区、包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(hcdr1、hcdr2和hcdr3),所述重链可变区包含seqidno:29中所示的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链互补决定区(cdr)(lcdr1、lcdr2和lcdr3),所述轻链可变区包含seqidno:33中所示的氨基酸序列。[0307]在一些实施方案中,hcdr1包含seqidno:30中所示的氨基酸序列,hcdr2包含seqidno:31中所示的氨基酸序列,hcdr3包含seqidno:32中所示的氨基酸序列,lcdr1包含seqidno:34中所示的氨基酸序列,lcdr2包含seqidno:35中所示的氨基酸序列,并且lcdr3包含seqidno:36中所示的氨基酸序列。在一些实施方案中,分离的抗体包含含有seqidno:29中所示的氨基酸序列的hcvr和包含seqidno:33中所示的氨基酸序列的lcvr。在一些实施方案中,分离的抗体包含含有seqidno:37中所示的氨基酸序列的重链和包含seqidno:38中所示的氨基酸序列的轻链。在一些情况下,免疫球蛋白恒定区是igg1恒定区。在一些情况下,分离的抗体是重组抗体。在一些情况下,分离的抗体是多特异性的。[0308]在一些实施方案中,本公开提供包含如上或本文所讨论的分离的抗体和药学上可接受的载体或稀释剂的药物组合物。[0309]在一些情况下,抗体或它的抗原结合片段包含包含在hcvr内的三个重链cdr(hcdr1、hcdr2和hcdr3),所述hcvr包含seqidno:69中所示的氨基酸序列;以及包含在lcvr内的三个轻链cdr(lcdr1、lcdr2和lcdr3),所述lcvr包含seqidno:73中所示的氨基酸序列。在一些情况下,抗体或它的抗原结合片段包含:hcdr1,包含seqidno:70中所示的氨基酸序列;hcdr2,包含seqidno:71中所示的氨基酸序列;hcdr3,包含seqidno:72中所示的氨基酸序列;lcdr1,包含seqidno:74中所示的氨基酸序列;lcdr2,包含seqidno:75中所示的氨基酸序列;以及lcdr3,包含seqidno:76中所示的氨基酸序列。在一些情况下,抗体或它的抗原结合片段包含含有seqidno:69中所示的氨基酸序列的hcvr和包含seqidno:73中所示的氨基酸序列的lcvr。在一些情况下,抗体或它的抗原结合片段包含含有seqidno:77中所示的氨基酸序列的重链和包含seqidno:78中所示的氨基酸序列的轻链。[0310]在一些实施方案中,本公开提供结合sars-cov-2刺突蛋白的分离的抗体或它的抗原结合片段,所述sars-cov-2刺突蛋白包含seqidno:108中所示的氨基酸序列,其中所述分离的抗体或抗原结合片段包含包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(hcdr1、hcdr2和hcdr3),所述重链可变区包含seqidno:69中所示的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链互补决定区(cdr)(lcdr1、lcdr2和lcdr3),所述轻链可变区包含seqidno:73中所示的氨基酸序列。[0311]在一些实施方案中,hcdr1包含seqidno:70中所示的氨基酸序列,hcdr2包含seqidno:71中所示的氨基酸序列,hcdr3包含seqidno:72中所示的氨基酸序列,lcdr1包含seqidno:74中所示的氨基酸序列,lcdr2包含seqidno:75中所示的氨基酸序列,并且lcdr3包含seqidno:76中所示的氨基酸序列。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:69中所示的氨基酸序列的hcvr。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:73中所示的氨基酸序列的lcvr。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:69中所示的氨基酸序列的hcvr和包含seqidno:73中所示的氨基酸序列的lcvr。[0312]在一些实施方案中,本公开提供结合sars-cov-2刺突蛋白的分离的抗体,所述sars-cov-2刺突蛋白包含seqidno:108中所示的氨基酸序列,其中所述分离的抗体包含免疫球蛋白恒定区、包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(hcdr1、hcdr2和hcdr3),所述重链可变区包含seqidno:69中所示的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链互补决定区(cdr)(lcdr1、lcdr2和lcdr3),所述轻链可变区包含seqidno:73中所示的氨基酸序列。[0313]在一些实施方案中,hcdr1包含seqidno:70中所示的氨基酸序列,hcdr2包含seqidno:71中所示的氨基酸序列,hcdr3包含seqidno:72中所示的氨基酸序列,lcdr1包含seqidno:74中所示的氨基酸序列,lcdr2包含seqidno:75中所示的氨基酸序列,并且lcdr3包含seqidno:76中所示的氨基酸序列。在一些实施方案中,分离的抗体包含含有seqidno:69中所示的氨基酸序列的hcvr和包含seqidno:73中所示的氨基酸序列的lcvr。在一些实施方案中,分离的抗体包含含有seqidno:77中所示的氨基酸序列的重链和包含seqidno:78中所示的氨基酸序列的轻链。在一些情况下,免疫球蛋白恒定区是igg1恒定区。在一些情况下,分离的抗体是重组抗体。在一些情况下,分离的抗体是多特异性的。[0314]在一些实施方案中,药物组合物进一步包含第二治疗剂。在一些情况下,第二治疗剂是选自由以下组成的组:结合包含seqidno:108中所示的氨基酸序列的sars-cov-2刺突蛋白的第二抗体或它的抗原结合片段、抗炎剂、抗疟疾剂以及结合tmprss2的抗体或它的抗原结合片段。[0315]在其中感兴趣的抗体的表位已知的某些实施方案中,表位的氨基酸的变异频率可用于确定包括与感兴趣的抗体结合或预计与感兴趣的抗体结合的表位的受试者的频率。例如,在临床环境中,可从受试者分离编码抗体的靶抗原的基因组,并分析分离的基因组是否编码抗体的表位(例如,抗体结合或预计结合的抗原序列)或不同的序列(例如,对应于表位但不是抗体结合或预计结合的序列的序列)。如果比较许多不同的表位,靶向治疗群体中更保守的表位的抗体通常可优于靶向治疗群体中更不保守的表位的抗体。[0316]可在已接受抗体疗法的受试者中评价治疗性抗体的抗原、特别是表位的变异,以评价推定的逃逸变异。例如通过抗体疗法的治疗性干预对不易受干预影响的变体(逃逸变体)产生选择性压力。逃逸变体的一个实例是选择病原体基因组突变,所述病原体基因组突变导致病原体不易受抗体疗法的治疗影响。例如,病原体基因组突变可为治疗性抗体表位的改变,使得抗体不再结合它的靶抗原。本公开的方法和系统可用于通过在治疗后从受试者中分离编码抗体的靶抗原的基因组并分析序列的抗原和/或表位的氨基酸序列中的变异,来评价已接受抗体疗法的受试者中的推定的逃逸变体选择。与抗体能够结合的主题序列(例如,参考序列)相比,表位的变异可鉴定为推定的逃逸变体。[0317]抗原或表位的变异的分析也可用于确定尚未接受特定抗体疗法的受试者是否可能对抗体疗法有响应。包括编码与抗体疗法结合或预计由抗体疗法结合的序列匹配的表位序列的基因组序列(例如,病原体基因组序列)的受试者可分类为可能对抗体疗法有响应的受试者。相反,具有编码对应于与抗体疗法结合或预计由抗体疗法结合的序列不匹配的表位序列的氨基酸的基因组序列(例如,病原体基因组序列)的受试者可分类为不可能对抗体疗法有响应的受试者。因此,本公开的方法和系统可用于个性化医学应用中,其中选择可能对抗体疗法有响应的受试者进行所述疗法的治疗,并且不选择可能对抗体疗法无响应的个体进行所述疗法的治疗。[0318]用于应用的示例性方法和系统[0319]从本公开将理解,本文提供的方法和系统至少部分通过改变查询序列、主题序列和/或分析查询序列和主题序列之间的成对比较,可用于各种应用。[0320]在各种实施方案中,本公开的方法和系统包括以下步骤:获得和/或选择查询序列和(如果不同于查询序列的话)主题序列;从查询序列和主题序列中提取编码序列;对所有查询提取的编码序列和所有主题提取的编码序列进行成对比较,产生每个比较的与一个或多个归类因子(例如,同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系))相关的数据;基于一个或多个归类因子的一个或多个阈值将比较的序列归类成一个或多个归类的序列组(例如,其中向每个归类的序列组分配相似性评分);从进一步分析中过滤一个或多个归类的序列组(例如,基于相似性评分阈值),将编码序列翻译成氨基酸序列;比对翻译的编码序列;以及确定一个或多个主题序列的每一个的保守性和/或可变性。[0321]在各种实施方案中,本公开的方法和系统包括以下步骤:获得和/或选择查询序列和(如果不同于查询序列的话)主题序列;从查询序列中提取编码序列;所有查询提取的编码序列和所有主题序列的成对比较,形成哪些主题序列编码序列没有被提取,为每个比较产生与一个或多个归类因子(例如,同一性百分比、覆盖率百分比率、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系))相关的数据;基于一个或多个归类因子的一个或多个阈值将比较的序列归类到一个或多个归类的序列组中(例如,其中每个归类的序列组被分配相似性评分);从进一步分析中过滤一个或多个归类的序列组(例如,基于相似性评分阈值),将编码序列翻译成氨基酸序列;比对翻译的编码序列;以及确定一个或多个主题序列或它们的部分中的每一个的保守性和/或可变性。[0322]示例性示意图提供于图48中。[0323]在各种实施方案中,本公开的方法和系统包括以下步骤:获得和/或选择查询序列和(如果不同于查询序列的话)主题序列;从查询序列和主题序列中提取编码序列;将编码序列翻译成氨基酸序列;对所有查询翻译的编码序列和所有主题翻译的编码序列进行成对比较,产生每个比较的与一个或多个归类因子(例如,同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系))相关的数据;基于一个或多个归类因子的一个或多个阈值将比较的序列归类成一个或多个归类的序列组中(例如,其中向每个归类的序列组分配相似性评分);从进一步分析中过滤一个或多个归类的序列组(例如,基于相似性评分阈值);以及确定每个主题序列的保守性和/或可变性。[0324]在各种实施方案中,编码序列的提取是基于参考基因组序列的注释。参考基因组序列的注释可包括编码序列的鉴定、划分或分离。注释的参考基因组序列可在可公开访问的数据库中获得和/或可由用户生成或修改。因此,在其中主题序列是参考基因组序列的各种实施方案中,查询编码序列的鉴定和/或提取可基于例如在参考基因组序列中编码序列的可用或用户定义的注释。在各种实施方案中,可通过将主题基因组序列和/或查询基因组序列与注释的参考基因组序列和/或它的编码序列进行比对来鉴定和/或提取主题基因组序列和/或查询基因组序列的编码序列。[0325]在各种实施方案中,从查询序列和主题序列中提取编码序列是基于检测编码至少约20个、30个、40个、50个、60个、70个、80个、90个、100个、125个、150个、175个、200个、250个或300个或更多个氨基酸的邻接框内密码子。[0326]在各种实施方案中,查询序列和主题序列的成对比较是基于blast算法。blast算法是本领域已知的,包括用于核苷酸序列的blastn和用于氨基酸序列的blastp、有空位的blast和psi-blast。blast算法对序列进行比对,并产生每次比对的各种数据,包括但不限于提供同一性百分比、突变数量、突变百分比、覆盖长度、覆盖率百分比和e值的数据。[0327]可根据如表2中所示的归类因子对比较的序列进行归类。表2基于覆盖率百分比和突变数量为归类的序列组分配相似性评分。在形成归类的序列组之后,可从进一步的分析中过滤出相似性评分小于特定阈值(例如,相似性评分小于1、小于0.95或小于0.8)的归类的序列组。[0328]编码序列(例如,编码序列的剩余归类的组)可通过应用相关遗传密码(例如,人遗传密码)翻译成氨基酸序列。可对翻译的编码序列进行比对。如上所述,可使用blast算法完成比对。然后可确定序列的保守性和/或可变性。本公开的方法和系统中所示的各种分析不需要在氨基酸序列的比对之后进行过滤或选择。没有进一步选择的比对提供了有价值的信息。例如,在各种实施方案中,氨基酸序列的比对提供诸如比对位置处的保守性(例如,在一个或多个比对位置的每一个处包括与参考同一的氨基酸的比对序列的百分比)和比对位置处的序列变异(例如,可在每个比对位置处出现的不同氨基酸的数量和频率)的信息。就在某些实施方案中在氨基酸比对之后选择序列而言,选择可由用户例如根据应用于通过氨基酸序列比对产生的信息的标准来进行。因此,在各种实施方案中,不对氨基酸序列应用过滤器,例如,不使用阈值来选择氨基酸序列或它们的部分。在一些实施方案中,可基于如本文公开的阈值选择保守序列或可变序列。[0329]在其中评价保守性和/或可变性的各种实施方案中,查询序列是序列的第一集合,并且主题序列是序列的第二不同集合。在各种实施方案中,查询序列是序列的第一集合,并且主题序列是序列的相同集合。在其中评价保守性和/或可变性的各种实施方案中,查询序列是序列的第一集合,并且主题序列是单个序列(例如,感兴趣的序列)。[0330]在某些实施方案中,可关于成对比较来评价保守性和/或可变性,在成对比较中,查询序列是来自特定物种(例如,特定病原体)的多个生物体的序列的第一集合,并且主题序列是序列的相同集合。各种所述实施方案可从成对比较产生数据,所述数据可用于确定特定物种的保守序列和/或特定物种的可变序列。保守序列可例如在抗体或疫苗开发中经选择或使用抗原或表位。保守序列可为在例如人类受试者中病原体的正选择(例如进化存活选择压力和/或抗生素抗性选择)下的性状。可变序列可例如选择作为实验室工程化(例如,基因工程化)的靶标,选择作为系统发育分析的靶标,并且/或鉴定为经历进化多样化的序列。序列的变异也可用于产生可能序列(例如,可能的氨基酸序列)的列表或数据库,其可用于例如产生可能的质量用于质谱法分析。[0331]在某些实施方案中,可关于成对比较来评价保守性和/或可变性,在成对比较中,查询序列是来自特定物种(例如,特定病原体)的多个生物体的序列的集合,并且主题序列包括来自特定株系或生物体的一个或多个序列。在各种实施方案中,查询序列包括来自不同样品(例如,病原体的多个临床分离株)的多个生物体的序列。在各种实施方案中,受试者是实验室株系。在某些实施方案中,主题序列和查询序列之间测量的保守性和/或可变性可用于确定主题株系或生物体在查询序列中的代表性如何。在各种实施方案中,在生物体水平和/或通过评价所有比对的序列来确定主题株系是否代表查询序列。在各种实施方案中,生物体水平的确定可基于系统发育分析。例如,系统发育分析可鉴定簇中一个或多个感兴趣的序列,并确定所有簇的大小。[0332]序列的变异也可用于产生可能序列(例如,可能的氨基酸序列)的列表或数据库,其可用于例如产生可能的质量的列表或数据库用于质谱法分析。[0333]为了提供一个特定实例,本公开的方法和系统可用于分析诸如sars-cov-2的病毒的序列的各种实施方案中。在各种实施方案中,将本公开的方法和系统应用于sars-cov-2序列的分析可包括作为主题序列的一个或多个参考sars-cov-2序列,诸如作为基因库登录号mn908947可公开获得的已知sars-cov-2参考基因组序列。在一些实施方案中,主题序列可为或包括sars-cov-2参考基因组序列的一部分(例如,基因库登录号mn908947的一部分),其编码氨基酸序列,例如,sars-cov-2刺突蛋白或它的一部分(例如,sars-cov-2刺突受体结合结构域(rbd))。在各种实施方案中,查询序列可为多个sars-cov-2基因组序列或从其提取的编码序列。例如,通过全球共享流行性感冒数据倡议(gisaid)数据库(https://www.gisaid.org/)可获得至少约120,000个sars-cov-2基因组序列。可从受感染的受试者中得出替代或额外查询序列。可例如根据图26中发现的一般示意图从sars-cov-2基因组序列中提取编码序列。可如图27中发现的一般示意图中所说明,执行所有查询提取的编码序列和所有主题提取的编码序列的成对比较。查询序列和主题sars-cov-2序列的成对比较产生与归类因子相关的数据,所述归类因子包括同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和系统发育(例如,每个比较的系统发育分组和/或系统发育关系)。这些数据允许进行各种进一步分析。可准备包括所得到的序列比较数据的汇总表,例如如图28的表中发现的总体布局所说明,其示出了归类因子的亚组。此外,查询sars-cov-2序列与参考sars-cov-2的每个比较可基于一个或多个归类因子的一个或多个阈值归类成一个或多个归类的序列组。在一些实施方案中,可例如通过如表2所说明的相似性评分的分配将一个或多个归类因子的一个或多个阈值集成到单个量度中。在一些实施方案中,一个或多个归类因子的阈值(或基于两个或多个这样的阈值确定的相似性评分的阈值)可用于将sars-cov-2序列比较结果归类成各类别,其中一个或多个类别包括与参考序列或它的一部分更相似的查询序列,并且一个或多个不同类别包括与参考序列或它的一部分不太相似的查询序列。因此,在各种实施方案中,可保留与参考序列更相似的序列,用于关于参考序列或它的一部分的进一步分析,并且可从进一步分析中排除与参考序列或它的一部分不太相似的序列。当在查询基因组序列中发现与参考序列或它的一部分更相似的序列时,该参考序列或它的一部分可称为“存在于”查询基因组序列中,如例如在图28中一般性地指示。保守性和/或可变性的量度可以图形、热图、系统发育、排序列表和其他格式显示(对于一般示例,参见例如图29-图33)。可翻译并比对每个参考序列或它的一部分的剩余sars-cov-2序列,并且可确定比对序列的氨基酸保守性和/或可变性的量度。[0334]在各种实施方案中,用于核酸序列的比较的blast参数可使用blast默认值或利用表4中提供的任何值进行。在各种实施方案中,用于氨基酸序列的比较的blast参数可使用blast默认值或利用表5中提供的任何值进行。使用本公开的系统和方法不需要任何参数或参数组合的值的特定组。[0335]表4核酸比较blastn参数[0336][0337]表5氨基酸比较blastp参数[0338][0339][0340]示例性实施方案[0341]本公开尤其包括以下示例性实施方案:[0342]1.一种用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的方法,所述方法包括:[0343]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0344]通过计算装置的处理器从所述基因组序列中提取编码序列;[0345]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0346]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0347]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0348]通过所述处理器对所述氨基酸序列进行比对;[0349]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;[0350]选择分类为保守的所述氨基酸序列的部分,将所述选择的保守序列与人蛋白序列进行比较,并进一步将所述选择的保守序列分类为与人蛋白序列同一或不同一;以及[0351]将与人蛋白序列不同一的选择的保守序列归类为在针对所述病原体的疗法的开发中的候选抗原。[0352]2.根据实施方案1所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0353]3.根据实施方案1或实施方案2所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0354]4.根据实施方案1至3中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0355]5.根据实施方案4所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0356]6.根据实施方案5所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0357]7.根据实施方案1至6中任一项所述的方法,其中所述同一性的量度包括突变数量。[0358]8.根据实施方案1至7中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0359]9.根据实施方案1至8中任一项所述的方法,其中所述同一性的量度包括计算e值。[0360]10.根据实施方案1至9中任一项所述的方法,其中将所述选择的保守序列归类为候选抗原进一步包括确定所述选择的保守序列中一个或多个氨基酸结构域的存在或不存在。[0361]11.根据实施方案1至10中任一项所述的方法,其中将所述选择的保守序列归类为候选抗原进一步包括确定所述候选抗原是否对应于在所述病原体的膜和/或细胞壁内分泌或暴露的蛋白质。[0362]12.根据实施方案1至11中任一项所述的方法,其中将所述选择的保守序列归类为候选抗原进一步包括确定所述选择的保守序列中跨膜结构域的存在。[0363]13.根据实施方案1至12中任一项所述的方法,其中所述疗法包括疫苗,并且所述方法进一步包括非临床评价所述候选抗原的免疫原性。[0364]14.根据实施方案13所述的方法,其中所述评价步骤包括向动物施用包含所述候选抗原的多肽。[0365]15.根据实施方案1至14中任一项所述的方法,其中所述疗法包括抗体疗法,并且所述方法进一步包括产生特异性结合至所述候选抗原上的表位的抗体或其片段。[0366]16.根据实施方案1至15中任一项所述的方法,其中所述病原体是病毒。[0367]17.根据实施方案16所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0368]18.根据实施方案16所述的方法,其中所述病毒是冠状病毒。[0369]19.根据实施方案18所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0370]20.根据实施方案1至15中任一项所述的方法,其中所述病原体是细菌。[0371]21.根据实施方案20所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0372]22.一种在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变的方法,所述方法包括:[0373]在向每个受试者施用所述治疗剂之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;[0374]通过计算装置的处理器从所述基因组序列中提取编码序列;[0375]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0376]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0377]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0378]通过所述处理器对所述氨基酸序列进行比对;[0379]在所述比对的氨基酸序列中鉴定在所述比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一个或多个推定的逃逸突变。[0380]23.根据实施方案22所述的方法,其中所述参考包括代表典型病原体序列的一种或多种完整或部分病原体基因组序列、所述病原体的一个或多个临床株系、来自施用所述治疗剂的一个或多个受试者的病原体的一个或多个早期样品或来自未施用所述治疗剂的受试者的病原体的一个或多个样品。[0381]24.根据实施方案22或实施方案23所述的方法,所述方法进一步包括确定所述一个或多个推定的逃逸突变是否降低所述治疗剂与参考多肽的结合亲和性的步骤。[0382]25.根据实施方案22至24中任一项所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0383]26.根据实施方案22至25中任一项所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0384]27.根据实施方案22至26中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0385]28.根据实施方案27所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0386]29.根据实施方案28所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0387]30.根据实施方案22至29中任一项所述的方法,其中所述同一性的量度包括突变数量。[0388]31.根据实施方案22至30中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0389]32.根据实施方案22至31中任一项所述的方法,其中所述同一性的量度包括计算e值。[0390]33.根据实施方案22至32中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0391]编码与所述病原体相关的蛋白质的核酸的编码序列;[0392]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0393]编码蛋白质的核酸的非保守序列;[0394]与所述病原体相关的特定蛋白质内的保守结构域;和[0395]与所述病原体相关的特定蛋白质内的非保守结构域。[0396]34.根据实施方案22至33中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0397]35.根据实施方案22至34中任一项所述的方法,其中所述病原体是病毒。[0398]36.根据实施方案35所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0399]37.根据实施方案35所述的方法,其中所述病毒是冠状病毒。[0400]38.根据实施方案37所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0401]39.根据实施方案38所述的方法,其中所述冠状病毒是sars-cov-2。[0402]40.根据实施方案22至39中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0403]41.根据实施方案22至40中任一项所述的方法,其中所述治疗剂包含抗体。[0404]42.根据实施方案41所述的方法,其中所述抗体结合sars-cov-2。[0405]43.根据实施方案42所述的方法,其中所述抗体结合sars-cov-2刺突蛋白。[0406]44.根据实施方案41至43中任一项所述的方法,其中根据表3,所述抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。[0407]45.根据实施方案22至34中任一项所述的方法,其中所述病原体是细菌。[0408]46.根据实施方案45所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0409]47.一种向有需要的受试者施用用于治疗病原体感染的治疗剂的方法,所述方法包括:[0410]通过以下方式选择氨基酸序列的保守部分:[0411]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0412]通过计算装置的处理器从所述基因组序列中提取编码序列;[0413]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0414]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0415]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0416]通过所述处理器对所述氨基酸序列进行比对;[0417]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;以及[0418]选择所述比对的氨基酸序列的保守部分:以及[0419]如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的所述保守部分,则向所述受试者施用所述治疗剂,其中所述治疗剂选择性地结合所述氨基酸序列的所述保守部分。[0420]48.根据实施方案47所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0421]49.根据实施方案47或实施方案48所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0422]50.根据实施方案47至49中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0423]51.根据实施方案50所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0424]52.根据实施方案51所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0425]53.根据实施方案47至52中任一项所述的方法,其中所述同一性的量度包括突变数量。[0426]54.根据实施方案47至53中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0427]55.根据实施方案47至54中任一项所述的方法,其中所述同一性的量度包括计算e值。[0428]56.根据实施方案47至55中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0429]编码与所述病原体相关的蛋白质的核酸的编码序列;[0430]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0431]编码蛋白质的核酸的非保守序列;[0432]与所述病原体相关的特定蛋白质内的保守结构域;和[0433]与所述病原体相关的特定蛋白质内的非保守结构域。[0434]57.根据实施方案47至56中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0435]58.根据实施方案47至57中任一项所述的方法,其中所述病原体是病毒。[0436]59.根据实施方案58所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0437]60.根据实施方案58所述的方法,其中所述病毒是冠状病毒。[0438]61.根据实施方案60所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0439]62.根据实施方案61所述的方法,其中所述冠状病毒是sars-cov-2。[0440]63.根据实施方案47至62中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0441]64.根据实施方案47至63中任一项所述的方法,其中所述治疗剂包含抗体。[0442]65.根据实施方案64所述的方法,其中所述抗体结合sars-cov-2。[0443]66.根据实施方案65所述的方法,其中所述抗体结合sars-cov-2刺突蛋白。[0444]67.根据实施方案64至66中任一项所述的方法,其中根据表3,所述抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。[0445]68.根据实施方案47至57中任一项所述的方法,其中所述病原体是细菌。[0446]69.根据实施方案68所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0447]70.一种用于选择用于治疗感染病原体的受试者的治疗剂的方法,所述方法包括:[0448]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0449]通过计算装置的处理器从所述基因组序列中提取编码序列;[0450]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0451]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0452]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0453]通过所述处理器对所述氨基酸序列进行比对;[0454]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表所述病原体的编码序列的保守部分;以及[0455]选择结合所述保守编码序列的治疗剂作为对感染所述病原体的受试者的治疗。[0456]71.根据实施方案70所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0457]72.根据实施方案70或实施方案71所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0458]73.根据实施方案70至72中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0459]74.根据实施方案73所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0460]75.根据实施方案74所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0461]76.根据实施方案70至75中任一项所述的方法,其中所述同一性的量度包括突变数量。[0462]77.根据实施方案70至76中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0463]78.根据实施方案70至77中任一项所述的方法,其中所述同一性的量度包括计算e值。[0464]79.根据实施方案70至78中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0465]编码与所述病原体相关的蛋白质的核酸的编码序列;[0466]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0467]编码蛋白质的核酸的非保守序列;[0468]与所述病原体相关的特定蛋白质内的保守结构域;和[0469]与所述病原体相关的特定蛋白质内的非保守结构域。[0470]80.根据实施方案70至79中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0471]81.根据实施方案80所述的方法,其中所述方法进一步包括将所述治疗剂非临床评价为疫苗或它的组分。[0472]82.根据实施方案81所述的方法,其中所述评价步骤包括向动物施用所述治疗剂。[0473]83.根据实施方案70至82中任一项所述的方法,其中所述病原体是病毒。[0474]84.根据实施方案83所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0475]85.根据实施方案83所述的方法,其中所述病毒是冠状病毒。[0476]86.根据实施方案85所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0477]87.根据实施方案86所述的方法,其中所述冠状病毒是sars-cov-2。[0478]88.根据实施方案70至87中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0479]89.根据实施方案70至88中任一项所述的方法,其中所述治疗剂包含抗体。[0480]90.根据实施方案89所述的方法,其中所述抗体结合sars-cov-2。[0481]91.根据实施方案90所述的方法,其中所述抗体结合sars-cov-2刺突蛋白。[0482]92.根据实施方案89至91中任一项所述的方法,其中根据表3,所述抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。[0483]93.根据实施方案70至82中任一项所述的方法,其中所述病原体是细菌。[0484]94.根据实施方案93所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0485]95.一种用于评价代表病原体的氨基酸序列的部分的保守性的方法,所述方法包括:[0486]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0487]通过计算装置的处理器从所述基因组序列中提取编码序列;[0488]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0489]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0490]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0491]通过所述处理器对所述氨基酸序列进行比对;以及[0492]使用所述比对的氨基酸序列鉴定代表所述病原体的氨基酸序列的一个或多个部分的保守水平。[0493]96.根据实施方案95所述的方法,其中所述部分中的一个或多个被鉴定为在针对所述病原体的疗法的开发中的候选抗原。[0494]97.根据实施方案95或实施方案96所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0495]98.根据实施方案95至97中任一项所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0496]99.根据实施方案95至98中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0497]100.根据实施方案99所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0498]101.根据实施方案100所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0499]102.根据实施方案95至101中任一项所述的方法,其中所述同一性的量度包括突变数量。[0500]103.根据实施方案95至102中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0501]104.根据实施方案95至103中任一项所述的方法,其中所述同一性的量度包括计算e值。[0502]105.根据实施方案95至104中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0503]编码与所述病原体相关的蛋白质的核酸的编码序列;[0504]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0505]编码蛋白质的核酸的非保守序列;[0506]与所述病原体相关的特定蛋白质内的保守结构域;和[0507]与所述病原体相关的特定蛋白质内的非保守结构域。[0508]106.根据实施方案95至105中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0509]107.根据实施方案95至106中任一项所述的方法,其中所述病原体是病毒。[0510]108.根据实施方案107所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0511]109.根据实施方案107所述的方法,其中所述病毒是冠状病毒。[0512]110.根据实施方案109所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0513]111.根据实施方案110所述的方法,其中所述冠状病毒是sars-cov-2。[0514]112.根据实施方案95至111中任一项所述的方法,其中所述基因组序列是sars-cov-2基因组序列,并且所述参考序列是sars-cov-2参考序列。[0515]113.根据实施方案95至112中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0516]114.根据实施方案95至106中任一项所述的方法,其中所述病原体是细菌。[0517]115.根据实施方案114所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0518]116.一种用于鉴定分离的病原体是否代表循环株系的方法,所述方法包括:[0519]从数据结构获得所述病原体的所述循环株系的多个完整或部分基因组序列;[0520]鉴定所述循环株系的所述序列的一个或多个保守部分;[0521]获得所述分离的病原体的多个完整或部分基因组序列;以及[0522]通过将所述分离的病原体的所述序列的至少一部分与所述循环株系的所述序列的所述鉴定的一个或多个保守部分进行比较来鉴定所述分离的病原体是否代表所述循环株系。[0523]117.根据实施方案116所述的方法,其中鉴定所述循环株系的所述序列的一个或多个保守部分包括:[0524]通过计算装置的处理器从所述基因组序列中提取编码序列;[0525]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0526]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0527]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0528]通过所述处理器对所述氨基酸序列进行比对;以及[0529]根据所述部分在所述比对的氨基酸序列中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类。[0530]118.根据实施方案116或实施方案117所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0531]119.根据实施方案116至118中任一项所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0532]120.根据实施方案116至119中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0533]121.根据实施方案120所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0534]122.根据实施方案121所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0535]123.根据实施方案116至122中任一项所述的方法,其中所述同一性的量度包括突变数量。[0536]124.根据实施方案116至123中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0537]125.根据实施方案116至124中任一项所述的方法,其中所述同一性的量度包括计算e值。[0538]126.根据实施方案116至125中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0539]编码与所述病原体相关的蛋白质的核酸的编码序列;[0540]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0541]编码蛋白质的核酸的非保守序列;[0542]与所述病原体相关的特定蛋白质内的保守结构域;和[0543]与所述病原体相关的特定蛋白质内的非保守结构域。[0544]127.根据实施方案116至126中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0545]128.根据实施方案116至127中任一项所述的方法,其中所述病原体是病毒。[0546]129.根据实施方案128所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0547]130.根据实施方案128所述的方法,其中所述病毒是冠状病毒。[0548]131.根据实施方案130所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0549]132.根据实施方案131所述的方法,其中所述冠状病毒是sars-cov-2。[0550]133.根据实施方案116至132中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0551]134.根据实施方案116至127中任一项所述的方法,其中所述病原体是细菌。[0552]135.根据实施方案134所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0553]136.一种用于鉴定代表病原体的肽的质荷比的方法,所述方法包括:[0554]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0555]通过计算装置的处理器从所述基因组序列中提取编码序列;[0556]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0557]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0558]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;以及[0559]确定所述一个或多个氨基酸序列或它们的部分的所述质荷比。[0560]137.根据实施方案136所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0561]138.根据实施方案136或实施方案137所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0562]139.根据实施方案136至138中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0563]140.根据实施方案139所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0564]141.根据实施方案140所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0565]142.根据实施方案136至141中任一项所述的方法,其中所述同一性的量度包括突变数量。[0566]143.根据实施方案136至142中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0567]144.根据实施方案136至143中任一项所述的方法,其中所述同一性的量度包括计算e值。[0568]145.根据实施方案136至144中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0569]编码与所述病原体相关的蛋白质的核酸的编码序列;[0570]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0571]编码蛋白质的核酸的非保守序列;[0572]与所述病原体相关的特定蛋白质内的保守结构域;和[0573]与所述病原体相关的特定蛋白质内的非保守结构域。[0574]146.根据实施方案136至145中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0575]147.根据实施方案136至146中任一项所述的方法,其中所述病原体是病毒。[0576]148.根据实施方案147所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0577]149.根据实施方案147所述的方法,其中所述病毒是冠状病毒。[0578]150.根据实施方案149所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0579]151.根据实施方案150所述的方法,其中所述冠状病毒是sars-cov-2。[0580]152.根据实施方案136至151中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0581]153.根据实施方案136至146中任一项所述的方法,其中所述病原体是细菌。[0582]154.根据实施方案153所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0583]155.一种用于将氨基酸序列鉴定为候选抗生素抗性标记物的方法,所述方法包括:[0584]从数据结构获得病原性细菌的多个完整或部分质粒序列;[0585]通过计算装置的处理器从所述质粒序列中提取编码序列;[0586]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0587]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0588]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0589]通过所述处理器对所述氨基酸序列进行比对;[0590]根据所述部分在所述多个质粒序列中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;[0591]选择分类为保守的所述氨基酸序列的部分;以及[0592]将所选择的保守序列归类为候选抗生素抗性标记物。[0593]156.根据实施方案155所述的方法,所述方法进一步包括根据一个或多个额外标准将所述候选抗生素抗性标记物鉴定为候选者,所述额外标准包括在所选择的序列中存在跨膜结构域。[0594]157.根据实施方案155或实施方案156所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述多个完整或部分质粒序列包括通过所述处理器合并重叠的重叠群以产生至少一些所述完整或部分质粒序列。[0595]158.根据实施方案155至157中任一项所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0596]159.根据实施方案155至158中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0597]160.根据实施方案159所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0598]161.根据实施方案160所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0599]162.根据实施方案155至161中任一项所述的方法,其中所述同一性的量度包括突变数量。[0600]163.根据实施方案155至162中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0601]164.根据实施方案155至163中任一项所述的方法,其中所述同一性的量度包括计算e值。[0602]165.根据实施方案155至164中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0603]编码与所述病原体相关的蛋白质的核酸的编码序列;[0604]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0605]编码蛋白质的核酸的非保守序列;[0606]与所述病原体相关的特定蛋白质内的保守结构域;和[0607]与所述病原体相关的特定蛋白质内的非保守结构域。[0608]166.根据实施方案155至165中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0609]167.根据实施方案155至166中任一项所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0610]168.一种用于鉴定代表质粒的编码序列的一个或多个保守部分的方法,所述方法包括:[0611]从数据结构获得病原性细菌的多个完整或部分质粒序列;[0612]通过计算装置的处理器从所述质粒序列中提取编码序列;[0613]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0614]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0615]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0616]通过所述处理器对所述氨基酸序列进行比对;以及[0617]根据所述部分在所述多个质粒序列中的保守水平对所述氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表所述质粒的编码序列的一个或多个保守部分。[0618]169.根据实施方案168所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述多个完整或部分质粒序列包括通过所述处理器合并重叠的重叠群以产生至少一些所述完整或部分质粒序列。[0619]170.根据实施方案168或实施方案169所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0620]171.根据实施方案168至170中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0621]172.根据实施方案171所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0622]173.根据实施方案172所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0623]174.根据实施方案168至173中任一项所述的方法,其中所述同一性的量度包括突变数量。[0624]175.根据实施方案168至174中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0625]176.根据实施方案168至175中任一项所述的方法,其中所述同一性的量度包括计算e值。[0626]177.根据实施方案168至176中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0627]编码与所述病原体相关的蛋白质的核酸的编码序列;[0628]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0629]编码蛋白质的核酸的非保守序列;[0630]与所述病原体相关的特定蛋白质内的保守结构域;和[0631]与所述病原体相关的特定蛋白质内的非保守结构域。[0632]178.根据实施方案168至177中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0633]179.根据实施方案168至178中任一项所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0634]180.一种用于自动鉴定代表病原体的编码序列的一个或多个保守部分的系统,所述系统包括:[0635]处理器;以及[0636]其上具有指令的存储器,所述指令在由所述处理器执行时引起所述处理器:[0637]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0638]通过所述处理器从所述基因组序列中提取编码序列;[0639]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0640]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0641]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0642]通过所述处理器对所述氨基酸序列进行比对;以及[0643]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表所述病原体的编码序列的一个或多个保守部分。[0644]181.根据实施方案180所述的系统,其中所述指令当由所述处理器执行时引起所述处理器对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0645]182.根据实施方案181所述的系统,其中所述指令当由所述处理器执行时引起所述处理器创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0646]183.根据实施方案182所述的系统,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0647]184.根据实施方案180至183中任一项所述的系统,其中所述数据结构包括重叠群,并且其中所述指令当由所述处理器执行时引起所述处理器通过所述处理器合并重叠的重叠群以产生至少一些所述完整或部分基因组序列来获得所述病原体的不同株系的所述多个完整或部分基因组序列。[0648]185.根据实施方案180至184中任一项所述的系统,其中所述指令当由所述处理器执行时引起所述处理器评价以下中的一种或多种:[0649]编码与所述病原体相关的蛋白质的核酸的编码序列;[0650]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0651]编码蛋白质的核酸的非保守序列;[0652]与所述病原体相关的特定蛋白质内的保守结构域;和[0653]与所述病原体相关的特定蛋白质内的非保守结构域。[0654]186.根据实施方案180至185中任一项所述的系统,其中所述指令当由所述处理器执行时引起所述处理器评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0655]187.根据实施方案180至186中任一项所述的系统,其中所述病原体是病毒。[0656]188.根据实施方案187所述的系统,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0657]189.根据实施方案187所述的系统,其中所述病毒是冠状病毒。[0658]190.根据实施方案189所述的系统,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0659]191.根据实施方案190所述的系统,其中所述冠状病毒是sars-cov-2。[0660]192.根据实施方案180至186中任一项所述的系统,其中所述病原体是细菌。[0661]193.根据实施方案192所述的系统,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0662]194.一种用于自动鉴定代表质粒的编码序列的一个或多个保守部分的系统,所述系统包括:[0663]处理器;以及[0664]其上具有指令的存储器,所述指令在由所述处理器执行时引起所述处理器:[0665]从数据结构获得病原性细菌的多个完整或部分质粒序列;[0666]通过所述处理器从所述质粒序列中提取编码序列;[0667]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0668]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0669]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0670]通过所述处理器对所述氨基酸序列进行比对;以及[0671]根据所述部分在所述多个质粒序列中的保守水平对所述氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表所述质粒的编码序列的一个或多个保守部分。[0672]195.根据实施方案194所述的系统,其中所述指令当由所述处理器执行时引起所述处理器对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0673]196.根据实施方案195所述的系统,其中所述指令当由所述处理器执行时引起所述处理器创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0674]197.根据实施方案196所述的系统,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0675]198.根据实施方案194至197中任一项所述的系统,其中所述数据结构包括重叠群,并且其中所述指令当由所述处理器执行时引起所述处理器通过所述处理器合并重叠的重叠群以产生至少一些所述完整或部分质粒序列来获得病原性细菌的所述多个完整或部分质粒序列。[0676]199.根据实施方案194至198中任一项所述的系统,其中所述指令当由所述处理器执行时引起所述处理器评价以下中的一种或多种:[0677]编码与所述病原体相关的蛋白质的核酸的编码序列;[0678]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0679]编码蛋白质的核酸的非保守序列;[0680]与所述病原体相关的特定蛋白质内的保守结构域;和[0681]与所述病原体相关的特定蛋白质内的非保守结构域。[0682]200.根据实施方案194至199中任一项所述的系统,其中所述指令当由所述处理器执行时引起所述处理器评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0683]201.根据实施方案194至200中任一项所述的系统,其中所述病原体是病毒。[0684]202.根据实施方案201所述的系统,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0685]203.根据实施方案201所述的系统,其中所述病毒是冠状病毒。[0686]204.根据实施方案203所述的系统,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0687]205.根据实施方案204所述的系统,其中所述冠状病毒是sars-cov-2。[0688]206.根据实施方案194至200中任一项所述的系统,其中所述病原体是细菌。[0689]207.根据实施方案206所述的系统,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0690]208.一种用于在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变的治疗剂,所述用途包括:[0691]在向每个受试者施用所述治疗剂之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;[0692]通过计算装置的处理器从所述基因组序列中提取编码序列;[0693]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0694]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0695]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0696]通过所述处理器对所述氨基酸序列进行比对;[0697]在所述比对的氨基酸序列中鉴定在所述比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一个或多个推定的逃逸突变。[0698]209.一种用于治疗病原体感染的治疗剂,所述用途包括:[0699]通过以下方式选择氨基酸序列的保守部分:[0700]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0701]通过计算装置的处理器从所述基因组序列中提取编码序列;[0702]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0703]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0704]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0705]通过所述处理器对所述氨基酸序列进行比对;[0706]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;以及[0707]选择所述比对的氨基酸序列的保守部分:以及[0708]如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的所述保守部分,则向所述受试者施用所述治疗剂,其中所述治疗剂选择性地结合所述氨基酸序列的所述保守部分。[0709]210.一种确定由抗体结合的病原体表位是否保守的方法,所述方法包括:[0710]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0711]通过计算装置的处理器从所述基因组序列中提取编码序列;[0712]将所述编码序列与编码所述病原体表位的参考序列进行比较;[0713]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0714]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0715]将所述选择的编码序列转换成相应的氨基酸序列;以及[0716]确定所述病原体的所述不同株系中的所述病原体表位的保守水平。[0717]210.治疗剂用于制造药物的用途,所述药物用于在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变,所述用途包括:[0718]在向每个受试者施用所述药物之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;[0719]通过计算装置的处理器从所述基因组序列中提取编码序列;[0720]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0721]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0722]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0723]通过所述处理器对所述氨基酸序列进行比对;[0724]在所述比对的氨基酸序列中鉴定在所述比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一个或多个推定的逃逸突变。[0725]211.治疗剂用于制造用于治疗病原体感染的药物的用途,所述用途包括:[0726]通过以下方式选择氨基酸序列的保守部分:[0727]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0728]通过计算装置的处理器从所述基因组序列中提取编码序列;[0729]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0730]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0731]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0732]通过所述处理器对所述氨基酸序列进行比对;[0733]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;以及[0734]选择所述比对的氨基酸序列的保守部分:以及[0735]如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的所述保守部分,则向所述受试者施用所述药物,其中所述治疗剂选择性地结合所述氨基酸序列的所述保守部分。[0736]实施例[0737]本实施例提供本公开的示例性方法和系统及它们的示例性用途。过去的十年见证了泛滥的测序的基因组,在最频繁测序的物种中有病毒和细菌,其中许多是病原性的。例如,根据对ncbi数据库中存在的超过约150万个基因组序列的一个审查,ncbi数据库包括约642,604个真核基因组序列、约757,524个细菌基因组序列和约176,471个病毒基因组序列。[0738]研究人员发现,在一些情况下,对大规模基因组数据集的分析可揭示病原体基因组的变化,这些变化在流行病学上与临床后果相关。在某些实例中,此类相关变化可对病原体表型有显著贡献。然而,随着每周可公开访问的基因组序列数量增加数千个基因组,管理不断扩大的测序信息量变得越来越困难。此外,访问序列数据对用户不友好;需要计算技能来将数据转换成可行的形式。本实施例提供提取和处理可公开访问的基因组序列的方法和系统。本文提供的方法和系统特别适于在用户友好的计算程序中使用,所述计算程序例如以低或最少的用户输入来执行可公开访问的基因组序列的分析。[0739]本实施例展现可公开获得的基因组序列的分析揭示影响或可能影响病原体表型(例如,宿主-病原体相互作用)、影响治疗性开发或为治疗性开发(例如,治疗抗体的开发)提供靶标的基因组的特定特征的能力。本实施例特别展现目前公开的方法和系统在鉴定尤其用于治疗剂的开发的保守序列中、例如作为用于治疗性抗体开发的抗原的效用。尽管常规疫苗学可需要约5年至约15年来进行疫苗抗原的选择和验证,并且使用基于基因组的方法的反向疫苗学可需要约1年至约2年来进行疫苗抗原的选择和验证,但本文公开的方法和系统可快速鉴定用于疫苗开发的抗原,例如促进在约1周至约2周内选择和验证疫苗抗原。[0740]实施例1:用于鉴定治疗目标的保守序列的示例性方法和系统[0741]本实施例提供用于鉴定治疗目标的保守序列的示例性方法和系统。本实施例利用以r编写的计算机程序(“got_gene”),所述程序使用本领域已知的blast算法和专有的r包来鉴定、比较和表征数千个输入基因组序列。本文公开的got_gene程序是用户友好型并且不需要计算技能。其自动讯问公共数据库,以表格、图形和视觉形式提供一组全面的信息。[0742]本实施例的程序包括约2500行代码和10个r包。本实施例的程序利用2至4个外部程序:blastn、phyml和quicktree中的一个或两个,以及可选的megahit。blast算法用于比对,并且可在万维网(worldwideweb)的ncbi.nlm.nih.gov上使用;quicktree用于系统发育分析,并且可例如在hypertexttransferprotocolgithub.com/tseemann/quicktree使用;megahit用于序列组装,并且可在例如万维网的metagenomics.wiki/tools/assembly/megahit上使用。所利用的r包包括:data.table;iranges;reutils;biofiles;ggplot2;cowplot;rcolorbrewer;reshape2;gridextra;decipher;shiny;colourpicker;以及plotly。[0743]不希望受任何特定示例或解释的限制,本实施例中使用的got_gene程序可被视为包括五个步骤(例如,参见图18):[0744](1)首先,用户指示关于从中提取感兴趣的一组基因的基因组的信息。这包括选择感兴趣的生物体,基于所述选择,可将基因组序列鉴定用作got_gene程序中的输入(例如,作为主题输入)。用户还可选择用于比较分析的查询序列列表。[0745](2)特征和序列文件自动从ncbi下载。这包括收集输入(例如,主题输入),例如通过从诸如ncbi的可公开访问的数据库下载相关序列,包括任选地与序列注释信息一起的序列;[0746](3)序列(例如,每个查询序列与每个主题序列)的成对blast比较提供确立跨所有基因组序列的每个感兴趣的基因的序列多样性水平的数据;[0747](4)将表示序列多样性信息(例如,序列保守性)的数据编译在例如生成的got表中。got表包括关于每个基因组中每个基因的存在或不存在、多样性水平、变异性质和基因组坐标的信息;以及[0748](5)got表用于生成表示编译的序列多样性信息的显示(例如,表、热图和/或图形)。生成的显示可为或包括序列多样性的图形、最大似然系统发育和/或比对文件。然后从所有基因组中提取基因序列并翻译,以创建核苷酸和氨基酸比对。将每个步骤保存到fasta文件中。最后,使用phyml程序创建基于基因组的系统发育和基于基因的系统发育,并保存到单独的文件中。[0749]这些步骤不旨在并且不限制、避免或要求在本公开的方法或系统中包括本文提供的任何步骤或一系列步骤。[0750]如图1中所提供,本发明的方法和系统可包括由用户手动提供的或从序列数据库获取的主题序列输入(以及特征信息,诸如gff、gbk、gtf),并且可包括由用户手动提供的或例如从从头测序数据(例如,illumina或其他高通量测序读段)组装的查询序列输入。比对查询序列和主题序列,每个查询序列针对每个主题序列。所得的数据用于生成got表。got表可用于生成信息显示,包括图形(graphics)(图形(graph)、热图)、序列比对、经翻译的序列比对和系统发育显示(包括基于基因组的系统发育和/或基于基因的系统发育)。可出于用户指定的目的、例如通过鉴定以下中的任何一个或多个或全部来选择基因或氨基酸序列:(i)最保守的基因;(ii)最不保守的基因(即,最多样或最可变的基因);(iii)毒力因子;(iv)抗生素抗性;(v)人序列同源性;(vi)分泌的蛋白质和/或包括分泌结构域的蛋白质;和(vii)跨膜或表面蛋白质,和/或包括跨膜或表面结构域的蛋白质。[0751]方法或系统的第一步骤可为确定要从一个或多个可公开访问的数据库(例如,ncbi)获取(例如,下载)(如果可用的话,连同注释信息)的主题序列的特征,并且确定是否将手动提供一个或多个查询序列用于与主题序列进行比较(图2)。got_gene程序可自动生成某些文件夹用于组织和/或存储数据,这些文件夹如图3所示。[0752]方法或系统的第二步骤可为从一个或多个可公开访问的数据库中获取主题序列和注释信息,其可被复制并存储在几个got_gene文件夹(参考序列、比对器数据库和注释文件夹)中(图4)。用于从一个或多个可公开访问的数据库获取序列和注释信息的步骤提供于图5中。r包reutils用于打开与ncbi数据库的服务器的通道。reutils是ncbientrez编程实用程序的接口,并且为与ncbi数据库(诸如pubmed、genbank或geo)交互的系统提供支持,所述编程接口的每个功能称为r功能。[0753]方法或系统的第三步骤可为手动提供查询序列或从可公开访问的数据库下载查询序列(图6)。[0754]方法或系统的第四步骤可为将查询序列与比对器数据库文件夹中的序列(即,主题序列)进行比对(图7)。使用blast的比对的步骤提供于图8中。例如,用于序列比较的blast参数可包括outfmt‘7stdsgistitle’;最小e值=约0.001;打开空位的成本=约5;延伸空位的成本=约2;最佳完美匹配的长度=约11;核苷酸匹配的加分=约2;核苷酸错配的加分=-约3(图8)。[0755]方法或系统的第五步骤可包括创建got表。got表可包括成对序列比较的blast结果、所分析序列的序列和可用注释(图9)。丢弃无结果的blast输出(即在特定的比较对之间未鉴定到匹配),包括无匹配的重叠群。也丢弃e值大于约0.001、同一性百分比低于约79%或覆盖长度小于约50个核苷酸的blast结果(图10)。未丢弃的成对序列比较称为匹配。在查询包括重叠群并且多个查询重叠群以重叠方式匹配特定参考序列的情况下,可能必需策划包括哪个重叠群用于分析(图11)。用于选择保留哪个查询重叠群作为参考序列的成对匹配的标准可包括图11(18)中提供的那些标准。在生成got表时,如果重叠的重叠群覆盖的基因百分比大于约95%,则可认为查询存在于参考序列中;如果重叠的重叠群覆盖的基因百分比大于约80%,则可认为查询部分存在于参考中;或者如果重叠的重叠群覆盖的基因百分比小于约79%或小于约80%,则可认为查询不存在参考序列(图12)。也可使用其他阈值。对于每个剩余的匹配,可计算snp/大小比(匹配中的突变数量与所述匹配的长度之间的比率)(图12)。选择覆盖参考序列的整个长度的单个重叠群,并且如果查询序列的多个此类重叠群相对于参考序列存在,则保留相对于参考具有最少突变的重叠群(图12)。在没有匹配的重叠群覆盖参考序列的整个长度的情况下,保留snp/大小比小于约0.5的所有重叠群(图12)。got表还可并入注释信息(图12)。got表可包括与参数相关的信息,包括图13所示的那些。对于每个查询序列生成一个got表(图13)。[0756]got表可用于生成各种信息分析并显示为输出。一个所述输出是比较表。为了生成比较表,将关于在got表中找到的每个查询序列与所有参考序列相比的序列相似性的信息转换成相似性评分(图15)。基于查询和主题之间的比对的覆盖率百分比以及查询和主题之间的突变数量来分配相似性评分。可例如根据表2来分配相似性评分(也参见图14)。可以矩阵编译相似度评分,所述矩阵是对比表(图14)。在比较表中发现的相似性数字也可呈现为热图,显示相关查询序列和每个主题序列之间的保守性(图15)。[0757]可基于got表中匹配的坐标和相关注释在查询核苷酸序列中鉴定编码序列。可提取和翻译经鉴定的编码序列(图16)。可比对经翻译的序列并保存在用于提取序列的got_gene文件夹中(图16)。在多个查询重叠群与参考编码序列匹配的情况下,重叠的重叠群合并成单个匹配序列。延伸超出参考编码序列的边界的查询重叠群可能需要策划(图16)。可将每个变体主题编码序列翻译的数量和频率制成表(图16)。也可例如使用quicktree对提取的序列进行系统发育分析(图17)。可使用参考核苷酸序列生成个别基因的基于参考的系统发育(图17)。可基于所有查询序列中最保守的主题序列生成个别基因组的基于基因组的系统发育,例如,其中主题序列一起包括不超过约40,000个核苷酸(图17)。[0758]本实施例展现本实施例的方法和系统可用于多种治疗相关的应用。这些尤其可包括:(1)确定抗原/表位的遗传保守性,以预测靶向抗体的临床潜力;(2)通过质谱法鉴定用于肽发现的氨基酸序列变体;(3)提取序列并创建比对以突出基因/抗原内的多样性区域;(4)鉴定基因组内的多样性/保护性的区域;(5)将基因组内未表征的感兴趣序列鉴定为潜在的治疗或疫苗靶标;(6)建立系统发育以鉴定引起流行病的病原体的基因型;(7)从错误注释的基因组中检索出一套直系同源基因;并且/或(8)出于流行病学目的区分株系中的关联性。[0759]实施例2:用于鉴定乙型肝炎病毒的新治疗性抗原的方法和系统的使用[0760]在本实施例中,根据本文所述的方法和系统,使用got_gene程序鉴定hcc肿瘤上mhc-1上存在的新乙型肝炎病毒肽。乙型肝炎病毒(hbv)是全球性健康问题并且是肝细胞癌(hcc)的主要原因(图21)。发生慢性感染的人通常用核苷类似物治疗以抑制病毒复制,但仍处于hcc的加强风险中。免疫系统无法清除感染的一个主要促成因素是,患有慢性hbv的患者的hbv特异性t细胞数量减少,并且那些患者中的许多保持显示衰竭表型。[0761]在肿瘤学领域,t细胞重定向抗体是通过利用那些细胞表面上的肿瘤特异性抗原靶向和杀死肿瘤细胞的常见方法。不幸的是,不存在在感染/肿瘤细胞的表面上表达的hbv蛋白。然而,与mhc-i复合的hbv肽呈递在细胞表面上。某些先前努力无法鉴定与mhc-i复合的临床有用的hbv肽呈递在细胞表面上。例如,分析来自hbv 患者的hcc肿瘤样品时,最初通过质谱法仅鉴定出细胞表面上呈递的少数hbv肽。这种失败至少部分是由于关于此类肽的预计序列的限制性假设。质谱法方案使用预先确立的一组来源于参考基因组的氨基酸序列来捕获实验设置中肽的存在。质谱法对肽序列变异高度敏感,并且用于鉴定所述肽的呈递的肽与参考序列之间的单个氨基酸变化会对信号检测产生显著影响。因此,确立一套用于质谱法分析的正确参考序列至关重要。[0762]进行本实施例中所述的工作以鉴定与mhc-i复合的hbv肽作为新的候选hbv抗原呈递在细胞表面上,用于治疗性抗体开发,例如,用于开发抗hbvpig/cd3双特异性抗体以驱动针对肿瘤/感染细胞的t细胞响应。[0763]hbv具有约3.1kb的环状基因组,其包括编码约4种多肽的约7个重叠编码序列(图22)。主要乙型肝炎表面抗原(hbsag)蛋白由基因s编码(图23)。hbsag是hbv的表面抗原,并且已知其指示当前的乙型肝炎感染。在世界各地发现了各种hbv基因组,并且已公布至少约7,108个hbv基因组序列(图24)。通过got_gene对hbv基因组的分析展现,该程序能够分析具有多种特征的序列,包括环状序列、线性序列、片段化序列、dna序列、rna序列、数据库序列和手动提供的序列(图25)。[0764]在本实施例中,对几个hbv样品进行rnaseq。序列读段用于构建每个样品的从头基因组病毒序列。从ncbi下载额外hbv基因组(例如,参见图18)。got_gene用于从所有hbv基因组中提取编码序列(图26)。通过blast成对比较所有查询hbv基因组和参考hbv基因组的编码序列(图27)。制备包括所得序列比较数据的汇总表(图28)。序列保守性以图形(图29)、热图(图30)和系统发育(参见图31和图32中的示例性系统发育显示)来显示。将提取的编码序列(例如,参见图34)翻译成氨基酸序列(例如,参见图35)并比对氨基酸序列(例如,参见图36)。分析比对的氨基酸序列的保存性(图36)。[0765]将本实施例中鉴定的氨基酸序列添加到上述质谱法分析方案中,从而能够检测先前意外的hbv肽。利用更新的参数相应地重新分析质谱法结果。这些分析导致发现了呈递在感染细胞表面上的新肽。这些肽特别令人感兴趣,这是因为它们显示与i类人hla结合的混杂性,进一步支持了它们是有希望的治疗开发靶标。[0766]got_gene还用于表征约7,000个hbv基因组中强效hbv抗原的多样性水平,以鉴定高度保守的表位区域。[0767]实施例3:方法和系统的用于确定样品基因组和参考基因组集合之间的相似性的用途[0768]出于历史原因以及与效率和一致性相关的原因,实验室或研究团体通常会使用一种或几种感兴趣的特定生物体株系进行实验。这些实验室株系通常被视为代表非实验室形式(例如,同一生物体的天然或野生实例)。然而,这种典型的方法有某些固有的缺点。具体来说,由于特定生物体的现实世界多样性远远大于(例如)给定实验中测试的实验室样品所代表的多样性,所以实验室结果不一定适用于相关生物体多样性的整个范围。为了提供来自临床背景的实例,病原体的特定株系可用于实验室实验,但临床分离株代表实验室株系可充分代表或不可充分代表的更大多样性的序列。[0769]本公开的方法和系统可用于确定所提供的序列(例如,实验室株系的基因组序列)是否由在非实验室形式中保守(或不保守)的序列表征。因此,例如,本公开的方法和系统可应用于基于所测量的序列保守性来确定实验室病原体株系是否代表病原体的临床分离株。在一种或几种实验室测试株系用于旨在代表更广泛的株系群体的实验中的情况下(例如,在一种或几种病原体株系可用于实验室、但在临床应用中可遇到许多不同株系的情况下),这种使用特别有价值。在所述情况下,重要的是实验室或测试株系代表参考基因组的集合(例如临床相关基因组的集合)。[0770]在本实施例中,got_gene用于确定样品基因组和参考基因组集合的相似性。更特定来说,got_gene用于确立金黄色葡萄球菌的一个特定实验室株系代表在社区中引起疾病的循环株系。got_gene应用基于基因组的系统发育,以出于流行病学目的容易地区分株系之间的关联性。相同方法成功地应用于确定绿脓假单胞菌和流感病毒的实验室株系是否临床上相关。[0771]实施例4:方法和系统的用于评价sars-cov-2受体结合结构域的保守性的用途[0772]2019年冠状病毒病(covid-19)全球疫情激发了广泛的努力以了解其病原性因子严重急性呼吸综合征冠状病毒2(sars-cov-2)的适应机制。因此,世界各地的科学家和医学专业人士已对来自患者分离株的sars-cov-2基因组进行了测序,并通过策划的数据存储库(诸如全球共享流感数据倡议(globalinitiativeonsharingallinfluenzadata,gisaid.https://www.gisaid.org))以前所未有的速度传播了他们的发现。这提供了可用于确定传播模式和鉴定可与毒力和疾病严重程度相关的sars-cov-2变体的独特的数据集。[0773]sars-cov-2的结构的示意图提供于图47中。它包括四种结构蛋白:核衣壳(n)蛋白、膜(m)蛋白、刺突(s)蛋白和包膜(e)蛋白以及几种非结构蛋白(nsp)。衣壳是病毒的蛋白外壳。在衣壳内,有核衣壳与病毒的病毒单阳性链rna基因组结合。冠状病毒基因组包括约30,000个核苷酸。使用计算技术和/或分子生物学技术,可容易地将呈rna形式的基因组序列转换或翻译成dna形式。[0774]为了确立复制生态位和对抗先天和适应性免疫响应,sars-cov-2必须适应宿主环境。一种常见的适应机制是抗原变异,其中抗体识别的病毒靶标产生逃逸突变,所述逃逸突变使病毒逃逸识别和消除。抗原变异的后果可包括持续性病毒感染、疾病大流行和恢复后的再感染。在covid-19治疗发展的背景下,抗原变异也影响治疗功效,这是因为突发突变可通过修饰其靶标的蛋白结构来混淆基于抗体的治疗的功效。[0775]病毒刺突蛋白(s)的sars-cov-2受体结合结构域(rbd)是covid-19患者血清或血浆样品中强效中和抗s抗体的重要靶标。因此,s是开发用于治疗covid-19的抗体的重要靶标。rbd的遗传保守性对于确保基于抗体的治疗成功(至少在包括抗s抗体的治疗方面)至关重要。在此背景下,got_gene用于评价rbd的遗传多样性。[0776]自2020年1月初报道第一个sars-cov-2基因组序列以来,截至2020年10月,已有约120,000个序列寄存到gisaid(https://www.gisaid.org/)。在本实施例中,使用got_gene算法来提取、过滤和比较从总共118,728个策划的基因组序列中检索到的刺突编码基因序列的同一性。在本实施例中,使用基因库文件注释(部分在图49的示意图中说明)从参考sars-cov-2基因组提取编码序列。使用用于序列的比对的blastn,在每个策划的基因组序列和刺突蛋白参考序列之间进行成对比较。图50中图示了所分析的查询序列的累积数量。在比对后,从策划的基因组序列中提取与刺突蛋白参考序列比对的编码序列。然后基于表2所示的覆盖长度和突变数量对与刺突蛋白参考序列比对的基因组序列进行归类。从进一步分析中去除来自与刺突蛋白参考序列比较的分配相似性评分小于0.8的序列。将分析中剩余的与刺突蛋白参考序列比对的序列翻译成氨基酸序列,并使用blastp对氨基酸序列进行比对(部分在图51的示意图中说明)。这个分析允许鉴定刺突蛋白的每个比对位置存在的氨基酸的范围(部分在图52的示意图中说明)。[0777]结果鉴定了sars-cov-2刺突蛋白中的965个可变氨基酸位置和总数为1782个独特的氨基酸变化。正如预计,在118,728个基因组中,大多数变体仅在一个给定的基因组(单例)中鉴定到。然而,鉴定到超过100种株系(高频变体或hfv)间共有的47种氨基酸变化。发现在刺突蛋白内鉴定的hfv在n-末端和s2结构域内积累。除了在与人ace2受体直接相互作用的受体结合基序内鉴定的两个hfv(n439k和s477n)之外,rbd没有hfv。总之,s蛋白显示相对较小的序列多样性。在本研究使用的118,728个株系中,仅观察到7个变体(l5f、l18f、r21i、a222v、s477n、d614g和d936y),频率大于0.6%。[0778]本实施例的一个重要发现是sars-cov-2表位保守性在这种高度成功的人病原体中是规则而不是例外的强有力证据。sars-cov-2rbd是covid-19患者血清或血浆样品中强效中和抗s抗体的主要靶标。因此,治疗性抗体施加的大部分选择性压力应靶向这个结构域。rbd保守性的仔细检查指示,几乎没有证据表明在所有sars-cov-2株系的》0.15%中繁殖的突变积累。虽然已在循环sars-cov-2分离株中鉴定出几种rbd变体,但如本研究中所测量,在病毒群体中,无一达到显著的频率。总之,这些数据表明rbd靶向抗体表位在循环sars-cov-2中的保守性;因此,基于s的治疗应该对所有循环sars-cov-2病毒有效是理所当然的。[0779]实施例5:方法和系统的用于评价表位变异的用途[0780]2019年末sars-cov-2的出现以及其随后对人类健康的有害影响导致数百万感染以及显著发病率和死亡率。在终止covid-19疫情的努力中,瑞泽恩制药公司(regeneronpharmaceuticals)已应用其最先进的技术来开发专用于对抗sars-cov-2病毒的单克隆抗体的混合物(例如,参见美国专利第10,787,501号,所述专利全文通过引用并且特别是关于covid-19治疗性抗体以及它们的表位和其他性质并入本文。美国专利第10,787,501号的表1提供了示例性抗sars-cov-2-刺突蛋白(sars-cov-2-s)抗体序列,其全文通过引用具体并入本文。)。瑞泽恩开始生产数百种病毒中和抗体,并从人covid-19幸存者中鉴定出表现相似的抗体。这些抗体特异性鉴定来自刺突蛋白的受体结合结构域(rbd)的表位。[0781]靶向相同抗原(例如,sars-cov-2刺突蛋白)的个别抗体可在抗原内具有不同的结构靶标(表位),且至少出于所述原因可具有不同特征,例如在个体受试者中和/或跨受试者群体的不同临床表现。根据至少一种方法,结合抗原的更保守表位的抗体优于结合抗原的更不保守表位的抗体,使得在任何给定株系或患者中或跨患者群体,抗体更可能有效地结合靶抗原和/或具有治疗效应。当可获得多种不同的抗体且可获得关于它们的不同表位的信息时,可使用序列分析来确定哪些抗体有利地结合更保守的表位。本实施例将该推理应用于用于治疗covid-19的抗体的开发。本公开的方法和系统用于评价多种抗体的sars-cov-2表位在数千个循环sars-cov-2株系中的保守性,其中靶向更保守的表位的抗体被选择或优选用于进一步的治疗评价。[0782]使用got_gene算法对数千个基因组中的表位遗传序列进行比较分析,所述got_gene算法允许将每个基因组序列针对独特的参考基因组进行快速成对比较。从全球共享流感数据倡议(gisaid)数据库中提取了超过120,000个sars-cov-2策划的基因组序列。[0783]在got_gene程序内使用blastn将来自gisaid的sars-cov-2核苷酸序列与sars-cov-2参考基因组核苷酸序列(基因库登录号:mn908947)进行比对。对每个策划的基因组序列和sars-cov-2参考基因组序列进行成对比较。比对后,评价与参考sars-cov-2基因组的刺突核酸序列比对的基因组序列,以验证刺突核酸序列的存在。got_gene根据某些阈值,基于关于刺突蛋白存在、缺乏完整性或不存在的确定,创建基因组的组类别。对于每个序列,如果与参考序列的比较产生大于95%的覆盖率百分比,则将刺突蛋白鉴定为存在;如果与参考的比较产生大于70%但小于95%的覆盖率百分比,则将刺突蛋白鉴定为部分存在或缺乏完整性;或如果与参考的比较产生低于70%的覆盖率百分比,则将刺突蛋白鉴定为不存在。如果与刺突蛋白参考序列的比较产生》95%的覆盖长度和》70%的同一性百分比,则验证刺突序列的存在。保留根据该阈值验证的序列用于进一步分析,并去除所有其他序列。got_gene从每个策划的基因组序列中提取刺突蛋白编码序列,并将每个策划的基因组序列的经验证的直系同源刺突序列翻译成氨基酸序列。然后使用blastp对氨基酸序列进行比对,并鉴定氨基酸变体。实施表位位置,并计算每个表位的变体的频率。[0784]实施例6:方法和系统的用于评价经治疗的受试者中推定的逃逸变体的选择的用途[0785]本实施例展现本公开的方法和系统的评估刺激对序列多样性的影响,特别是病毒疗法对病毒序列多样性的影响的用途。本实施例具体展现本公开的方法和系统的评估基于抗体的covid-19疗法对治疗接受者中的sars-cov-2序列多样性的影响的用途。[0786]两种强效瑞泽恩抗体(regn10933和regn10987)形成瑞泽恩的regn-cov2抗体疗法(也参见美国专利第10,787,501号,所述专利全文通过引用并且特别是关于covid-19治疗性抗体及它们的表位和其他性质并入本文。美国专利第10,787,501号的表1提供了示例性抗sars-cov-2-刺突蛋白(sars-cov-2-s)抗体序列,其全文通过引用具体并入。)。在9月,瑞泽恩公布了早期临床数据,所述临床数据显示regn-cov2抗体混合物对275例非住院covid-19患者中的病毒基因组序列的效应。本研究的一个目标是评估在治疗性施用regn-cov2治疗后来自患者的sars-cov-2分离株的推定的逃逸变体(对病毒有益的突变,因为它们使病毒逃避抗体识别)的选择。[0787]在本实施例中,对从接受regn-cov2治疗的患者中分离的病毒基因组进行测序,并使用got_gene程序鉴定分离的基因组中的新突变。使用用于序列的比对的blastn,在每个分离的基因组序列和编码刺突蛋白的参考序列之间进行成对比较。比对后,从策划的基因组序列中提取与编码刺突蛋白的参考序列比对的序列作为查询编码序列。然后基于表2所示的覆盖长度和突变数量对与刺突蛋白参考序列比对的基因组序列进行归类。从进一步分析中去除来自与刺突蛋白参考序列比较的分配相似性评分小于0.8的序列。将分析中剩余的与刺突蛋白参考序列比对的序列翻译成氨基酸序列,并使用blastp对氨基酸序列进行比对。这种分析允许鉴定刺突蛋白的每个比对位置处存在的氨基酸的范围。因此,使用got_gene从所有基因组中提取并翻译刺突编码基因序列,并将它们与参考序列进行比较,以鉴定其中新突变导致由中和抗体识别的区域中氨基酸变化的基因组。表位序列突变可为推定的逃逸变体。最终,分析评估了治疗是否会导致所有患者样品中的sars-cov-2s蛋白出现突变。[0788]实施例7:方法和系统在个性化医疗中的用途[0789]本实施例说明本公开的方法和系统可用于选择可能对感兴趣的治疗性治疗有利地响应的受试者。具体来说,本实施例公开了分析来自感染患者的病毒序列以确定患者是否可能受益于施用用于治疗病毒感染的抗体疗法。例如,got_gene程序可用于鉴定未治疗的患者中的推定的逃逸变体。got_gene程序也可用于鉴定具有推定的逃逸潜力的新突变。在这种情况下,如实施例6中所示,got_gene用于从自未治疗的患者分离的基因组中提取并翻译刺突编码基因序列,以鉴定与刺突蛋白参考序列相比的刺突蛋白突变。可将鉴定的刺突蛋白突变与已知或预计会负面地影响治疗功效的有害变体的预先确立的列表进行比较。该分析允许got_gene基于感染病毒株的遗传背景将患者分类成组(治疗易感组对比治疗抗性组)。[0790]其他实施方案[0791]虽然我们已经描述了许多实施方案,但是显而易见的是,我们的基本公开和实施例可提供利用本文所述的组合物和方法或由本文所述的组合物和方法涵盖的其他实施方案。因此,应理解,本发明的范围由可从本公开和所附权利要求中理解的内容来限定,而不是由已经通过实施例表示的具体实施方案来限定。[0792]本文引用的所有参考文献均以引用的方式并入本文中。当前第1页12当前第1页12
背景技术:
::5.近几十年来,基因组测序的速度和效率大大提高,从而能够收集到大量的基因组序列信息。可在可公开访问的数据库中获得超过100万个基因组序列,其中大部分是微生物基因组。例如,对于病原性冠状病毒sars-cov-2,大约160,000个基因组序列已寄存在可公开访问的数据库中。因此,有不断增长的不同基因组序列信息库。6.基因组序列信息的效用受到分析工具可用性的限制。分析所需的计算资源落后于序列数据的积累。例如,治疗和疫苗开发研究往往不能评估病原体群体的遗传多样性,从而导致临床试验失败。需要用于分析基因组序列信息的改进方法和系统,包括需要用于分析特定生物体、序列或基因的大量不同基因组序列的方法和系统。需要改进分析方法和系统,以为治疗开发提供信息并潜在地预测临床结果。另外,用于分析基因组序列信息的许多现有方法需要序列数据库的专用知识、序列分析软件的操作和/或数据输出的提炼。技术实现要素:7.本公开提供了用于分析基因组序列信息的方法和系统。近年来,基因组序列信息(包括微生物基因组序列信息)例如在可公开访问的数据库中激增。成本有效、高通量测序仪器和多重测序方案的开发已拓宽了基因组分析的吸引力,从而转变传染病的领域。然而,比较基因组分析通常由一组小的、有偏见的、经充分注释的库存基因组来指导,而没有考虑到公共数据库中可用的基因组多样性的广度。这些库存基因组通常被认为代表自然或相关多样性的广度,但实际上只代表自然群体的一微小部分。(例如)关于病原体的研究,这种鉴定、分析和/或代表自然多样性的问题尤其尖锐,其中所开发的治疗对不同病原体分离株的适用性是总体临床功效的重要组成部分。利用来自不同株系的可用序列在历史上需要计算技能和精心策划的最新基因组资源,其包括跨不同谱系(例如,跨病原体谱系)的基因组注释。至少部分由于大的可用基因组序列不是以这种方式完全组装,并且/或可用基因组序列(例如,病原体的不同株系的基因组序列)以不一致的方式注释,所以基因组分析(例如,种间或种内)在实践中是复杂的。随着测序的基因组数量的增加,对分析和计算工具的需要是确保优化利用这些资源的重要组成部分。8.本公开的方法和系统尤其提供了用于表征输入序列之中以及输入序列之间的序列保守性的方法和系统。如本文所讨论的,本公开的某些方法和系统包括在基于序列之间的比对的覆盖率百分比和序列之间的变异数量的多序列比较之后,将相似性或保守性评分分配给序列。9.在某些实施例中,本公开的方法和系统包括以下描述的一个或多个步骤。例如,在某些实施方案中,本文所述的方法和系统包括选择获取用于比较分析的基因组序列的生物体(例如病原体)的第一步骤。因此,在某些实施例中,用户在第一步骤中指示关于从中提取感兴趣序列的基因组的信息。第二步骤可包括例如通过从可公开访问的数据库获取序列数据,诸如通过从国家生物技术信息中心(nationalcenterforbiotechnologyinformationdatabase,ncbi)数据库下载,并且可选地从相同或不同来源获取序列注释和/或特征信息来提供序列。也可通过直接实验测量、例如从利用物理生物样品的高通量测序系统中读取来提供序列。因此,在某些实施例中,可通过直接测量、从ncbi数据库下载或两者来提供序列。序列和特征文件可从某些可公开访问的数据库(诸如ncbi数据库)自动下载。第三步骤可包括例如通过基本局部比对搜索工具(blast)成对比较分析序列。成对blast分析确立所有比较序列中每个分析的感兴趣序列的序列多样性水平。第四步骤可包括例如通过生成编译与序列保守性相关的信息的输出表编译与所有成对序列比较相关的信息。示例性表可包括关于特定序列的存在或不存在、特定序列基因座中的多样性水平、特定序列基因座中的变异性质和/或分析序列中特定特征的基因组坐标的信息。在各种实施方案中,可基于定义的评分系统为所分析的每个序列分配相似性评分,其中每个序列根据覆盖率百分比和序列变异数量进行归类。例如,在某些实施方案中,可根据表2对序列进行归类并分配相似性评分。在一些实施方案中,然后可从分析的序列中提取编码序列并翻译以产生核苷酸和氨基酸比对。可选的第五步骤可包括生成代表编译的序列保守性信息的视觉显示,例如,呈多样性图形、系统发育(例如,最大似然或简约系统发育)、热图和/或比对文件的形式。在某些实例中,基于基因组的系统发育和基因的系统发育是使用系统发育软件(诸如phyml或quicktree程序)生成,并保存到单独的文件中。10.在各种实施方案中,本文公开的方法和系统的步骤是通过使用计算机处理器和软件来实现。所述特定的专有软件在本文中称为“got_gene”,以r编程语言编写。got_gene使用blast算法和r包来鉴定、比较和表征一组序列的多样性,并可分析数千个序列间的多样性。11.在各种实施方案中,可用基因组序列的集合(主题序列,例如,参考序列)以成对方式与一个或多个用户选择的序列(查询序列)进行比较,以鉴定临床相关的序列特征。在各种实施方案中,本公开的方法和系统利用数据库(包括基因组序列信息的可公开访问的数据库)中可用的基因组序列信息的集合。在某些实施方案中,成对比较包括主题遗传序列和查询遗传序列(例如,主题编码遗传序列和查询编码遗传序列)的成对比较。在某些实施方案中,成对比较包括主题序列和查询序列编码的蛋白质的成对比较。12.在某些实施方案中,本公开的方法和系统可用于鉴定治疗效用的序列和序列特征。例如,本公开的方法和系统可用于鉴定候选抗原(例如,病原体抗原)以开发抗抗原治疗剂,诸如抗抗原治疗性抗体。在一些实施方案中,本公开的方法和系统可用于鉴定候选疫苗抗原。在一些实施方案中,本公开的方法和系统可用于确定一个或多个特定遗传序列(例如,实验室病原体株系的基因组)是否代表可比较的遗传序列的集合(例如,临床相关的病原体株系的基因组)。在一些实施方案中,本公开的方法和系统可用于鉴定抗生素抗性标记物。在一些实施方案中,本公开的方法和系统可用于生成肽发现资源,例如用于查询质谱法数据的预计肽和特征的列表。在一些实施方案中,本公开的方法和系统可用于鉴定序列内的多样性区域。在一些实施方案中,本公开的方法和系统可用于产生系统发育,例如,以增强对流行病(例如,病原体的传播)的临床理解。在一些实施方案中,本公开的方法和系统可用于鉴定物种之间或物种之中的直向同源序列。13.本公开的病原体可包括任何包括核酸或氨基酸序列或由核酸或氨基酸序列表征的病原体。本公开的病原体包括原核病原体和真核病原体。本发明的病原体的实例包括(但不限于)细菌、酵母、原生动物和病毒。在各种实施方案中,本公开的病原体选自鲍氏不动杆菌(acinetobacterbaumannii)、鲁氏不动杆菌(acinetobacterlwoffii)、不动杆菌属某些种(acinetobacterspp.)(例如,多药抗性不动杆菌(mdr-a))、放线菌(actinomycetes)、腺病毒(adenovirus)、气单胞菌属某些种(aeromonasspp.)、粪产碱菌(alcaligenesfaecalis)、产碱菌属某些种(alcaligenesspp.)/无色杆菌属某些种(achromobacterspp.)、木糖氧化产碱菌(alcaligenesxylosoxidans)(例如,广谱β-内酰胺酶(esbl)/多药抗性革兰氏阴性生物体(mrgn))、虫媒病毒(arbovirus)、人蛔虫(ascarislumbricoides)、曲菌属某些种(aspergillusspp.)、星状病毒(astrovirus)、炭疽芽孢杆菌(bacillusanthracis)、蜡状芽孢杆菌(bacilluscereus)、枯草杆菌(bacillussubtilis)、脆弱类杆菌(bacteriodesfragilis)、五日热巴尔通体(bartonellaquintana)、人芽囊原虫(blastocystishominis)、百日咳博德特氏菌(bordetellapertussis)、伯氏疏螺旋体(borreliaburgdorferi)、达氏疏螺旋体(borreliaduttoni)、回归热疏螺旋体(borreliarecurrentis)、缺陷短波单胞菌(brevundimonasdiminuta)、泡囊短波单胞菌(brevundimonasvesicularis)、布氏杆菌属某些种(brucellaspp.)、洋葱伯霍尔德杆菌(burkholderiacepacia)(例如,多药抗性(mdr))、鼻疽伯霍尔德杆菌(burkholderiamallei)、类鼻疽伯霍尔德杆菌(burkholderiapseudomallei)、空肠弯曲杆菌(campylobacterjejuni)/大肠弯曲杆菌(campylobactercoli)、白色念珠菌(candidaalbicans)、耳念珠菌(candidaauris)、克鲁斯念珠菌(candidakrusei)、近平滑念珠菌(candidaparapsilosis)、屈公病病毒(chikungunyavirus)(chikv)、肺炎衣原体(chlamydiapneumoniae)、鹦鹉衣原体(chlamydiapsittaci)、沙眼衣原体(chlamydiatrachomatis)、柠檬酸杆菌属某些种(citrobacterspp.)、肉毒梭菌(clostridiumbotulinum)、难养芽胞梭菌(clostridiumdifficile)、产气荚膜梭菌(clostridiumperfringens)、破伤风梭菌(clostridiumtetani)、冠状病毒(例如,严重急性呼吸综合征相关冠状病毒(sars-cov);严重急性呼吸综合征冠状病毒2(sars-cov2),其是引起冠状病毒疾病(covid-19)的病毒;以及中东呼吸综合征相关冠状病毒(mers-cov))、白喉棒状杆菌(corynebacteriumdiphtheriae)、假结核棒状杆菌(corynebacteriumpseudotuberculosis)、棒状杆菌属某些种(corynebacteriumspp.)、溃疡棒状杆菌(corynebacteriumulcerans)、伯内特考克斯体(coxiellaburnetii)、柯萨奇病毒(coxsackievirus)、克里米亚-刚果出血热病毒(crimean-congohaemorrhagicfevervirus)、新型隐球菌(cryptococcusneoformans)、人隐孢子虫(cryptosporidiumhominis)、小隐孢子虫(cryptosporidiumparvum)、卡耶塔环孢子虫(cyclosporacayetanensis)、巨细胞病毒(cytomegalovirus)、登革热病毒(denguevirus)、脆双核阿米species)、奇异变形杆菌(proteusmirabilis)(例如,esbl/mrgn)、普通变形杆菌(proteusvulgaris)、雷氏普罗威登斯菌(providenciarettgeri)、斯氏普罗威登斯菌(providenciastuartii)、绿脓假单胞菌(pseudomonasaeruginosa)、假单胞菌属某些种(pseudomonasspp.)、狂犬病病毒(rabiesvirus)、罗尔斯通氏菌属某些种(ralstoniaspp.)、呼吸道融合病毒、鼻病毒、普氏立克次体(rickettsiaprowazekii)、斑疹伤寒立克次体(rickettsiatyphi)、玫瑰单胞菌(roseomonasgilardii)、轮状病毒(rotavirus)、德国麻疹病毒(rubellavirus)、曼森氏住血吸虫(schistosomamansoni)、肠炎沙门氏菌(salmonellaenteritidis)、副伤寒沙门氏菌(salmonellaparatyphi)、沙门氏菌属某些种(salmonellaspp.)、伤寒沙门氏菌(salmonellatyphi)、鼠伤寒沙门氏菌(salmonellatyphimurium)、疥螨(sarcoptesscabiei,itchmite)、札如病毒(sapovirus)、粘质沙雷氏菌(serratiamarcescens)(例如,esbl/mrgn)、宋氏志贺氏菌(shigellasonnei)、假平胞菌属物种(sphingomonasspecies)、金黄色葡萄球菌(staphylococcusaureus)(例如,甲氧西林(methicillin)抗性金黄色葡萄球菌mrsa、万古霉素(vancomycin)抗性金黄色葡萄球菌(vrsa))、头葡萄球菌(staphylococcuscapitis)、表皮葡萄球菌(staphylococcusepidermidis)(例如,甲氧西林抗性表皮葡萄球菌(mrse))、溶血性葡萄球菌(staphylococcushaemolyticus)、人葡萄球菌(staphylococcushominis)、路邓葡萄球菌(staphylococcuslugdunensis)、巴斯德葡萄球菌(staphylococcuspasteuri)、腐生性葡萄球菌(staphylococcussaprophyticus)、嗜麦芽窄食单胞菌(stenotrophomonasmaltophilia)、肺炎链球菌(streptococcuspneumoniae)、酿脓链球菌(streptococcuspyogenes)(例如,prsp)、链球菌属某些种(streptococcusspp.)、粪类圆线虫(strongyloidesstercoralis)、猪肉绦虫(taeniasolium)、tbe病毒、弓虫(toxoplasmagondii)、梅毒密螺旋体(treponemapallidum)、旋毛线虫(trichinellaspiralis)、阴道毛滴虫(trichomonasvaginalis)、发癣菌属某些种(trichophytonspp.)、毛孢子菌属某些种(trichosporonspp.)、鞭形鞭虫(trichuristrichiura)、冈比亚布氏锥虫(trypanosomabruceigambiense)、罗德西亚布氏锥虫(trypanosomabruceirhodesiense)、克氏锥虫(trypanosomacruzi)、乌苏土病毒(usutuvirus)、牛痘病毒(vacciniavirus)、水痘带状疱疹病毒(varicellazostervirus)、痘疮病毒(variolavirus)、霍乱弧菌(vibriocholerae)、西尼罗病毒(westnilevirus,wnv)、黄热病毒(yellowfevervirus)、小肠大肠炎耶尔辛氏菌(yersiniaenterocolitica)、鼠疫耶尔辛氏菌(yersiniapestis)、假结核病耶尔辛氏菌(yersiniapseudotuberculosis)及兹卡病毒(zikavirus)。14.在至少一个方面,本公开包括用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;选择分类为保守的氨基酸序列的部分,将所选择的保守序列与人蛋白序列进行比较,并进一步将所选择的保守序列分类为与人蛋白序列同一或不同一;以及将与人蛋白序列不同一的选择的保守序列归类为在针对病原体的疗法的开发中的候选抗原。在各种实施方案中,提取可包括例如通过选择序列端点来鉴定、划分或分离序列。在各种实施方案中,提取可包括向序列或序列的一部分分配一个或多个特定特征或状态,例如作为编码序列的状态。在各种实施方案中,提取可包括例如通过观察注释(例如,将参考的相应的序列和/或比对的序列注释为编码序列或非编码序列,和/或注释归类的序列的基因组位置)来鉴定序列、诸如已根据同一性的量度和覆盖率的量度归类的序列实际上是编码序列。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,将所选择的保守序列归类为候选抗原进一步包括确定所选择的保守序列中一个或多个氨基酸结构域的存在或不存在。在某些实施方案中,将所选择的保守序列归类为候选抗原进一步包括确定候选抗原是否对应于在病原体的膜和/或细胞壁内分泌或暴露的蛋白质。在某些实施方案中,将所选择的保守序列归类为候选抗原进一步包括确定所选择的保守序列中跨膜结构域的存在。在某些实施方案中,疗法包括疫苗,并且所述方法进一步包括非临床评价候选抗原的免疫原性。在某些实施方案中,评价步骤包括向动物施用包含候选抗原的多肽,例如,其中动物是人、非人灵长类、小鼠或大鼠。在某些实施方案中,疗法包括抗体疗法,并且所述方法进一步包括产生特异性结合至候选抗原上的表位的抗体或其片段。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(methicillin-resistantstaphylococcusaureus,mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。15.在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法包括产生靶向或结合候选抗原的治疗剂。在某些实施方案中,治疗剂是抗体或抑制剂。在某些实施方案中,治疗剂是shrna或sirna,其对应于核酸序列,诸如编码候选抗原的编码序列。16.在至少一个方面,本公开包括在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变的方法,所述方法包括:在向每个受试者施用治疗剂之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;在比对的氨基酸序列中鉴定在比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一个或多个推定的逃逸突变。在某些实施方案中,参考包括代表典型病原体序列的一种或多种完整或部分病原体基因组序列、病原体的一个或多个临床株系、来自施用治疗剂的一个或多个受试者的病原体的一个或多个早期样品或来自未施用治疗剂的受试者的病原体的一个或多个样品。在某些实施方案中,所述方法进一步包括确定一个或多个推定的逃逸突变是否降低治疗剂与参考多肽的结合亲和性的步骤。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,治疗剂是抗体或抑制剂。在某些实施方案中,治疗剂是shrna或sirna。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,治疗剂包括治疗covid-19的治疗剂。在某些实施方案中,治疗剂包括瑞德西韦(remdesivir)、克力芝(kaletra)、伊维菌素(ivermectin)、特敏福(tamiflu)、阿维根(avigan)、秋水仙碱(colcrys)、地塞米松(dexamethasone)、氯喹(chloroquine)、羟基氯喹、阿奇霉素(azithromycin)、il-6抑制剂(例如,托珠单抗(tocilizumab)和西鲁库单抗(sarilumab))、激酶抑制剂(例如,阿卡拉替尼(acalabrutinib)、依鲁替尼(ibrutinib)、泽布替尼(zanubrutinib)、巴瑞替尼(baricitinib)、鲁索替尼(ruxolitinib)和托法替尼(tofacitinib))、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法包括在鉴定一种或多种推定的逃逸突变之后,向一个或多个受试者施用不同治疗剂。在某些实施方案中,不同治疗剂包括治疗covid-19的治疗剂。在某些实施方案中,不同治疗剂包括瑞德西韦、克力芝、伊维菌素、特敏福、阿维根、秋水仙碱、地塞米松、氯喹、羟基氯喹、阿奇霉素、il-6抑制剂(例如,托珠单抗和西鲁库单抗)、激酶抑制剂(例如,阿卡拉替尼、依鲁替尼、泽布替尼、巴瑞替尼、鲁索替尼和托法替尼)、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。[0017]在至少一个方面,本公开包括向有需要的受试者施用用于治疗病原体感染的治疗剂的方法,所述方法包括:通过以下方式选择氨基酸序列的保守部分:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;以及选择比对的氨基酸序列的保守部分;以及如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的保守部分,则向受试者施用治疗剂,其中治疗剂选择性地结合氨基酸序列的保守部分。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,治疗剂包括治疗covid-19的治疗剂。在某些实施方案中,治疗剂包括瑞德西韦、克力芝、伊维菌素、特敏福、阿维根、秋水仙碱、地塞米松、氯喹、羟基氯喹、阿奇霉素、il-6抑制剂(例如,托珠单抗和西鲁库单抗)、激酶抑制剂(例如,阿卡拉替尼、依鲁替尼、泽布替尼、巴瑞替尼、鲁索替尼和托法替尼)、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0018]在至少一个方面,本公开包括选择用于治疗感染病原体的受试者的治疗剂的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平,将比对的氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表病原体的编码序列的保守部分;以及选择结合保守编码序列的治疗剂作为对感染病原体的受试者的治疗。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,所述方法进一步包括将治疗剂非临床评价为疫苗或它的组分。在某些实施方案中,评价步骤包括向动物施用治疗剂,例如,其中动物是人、非人灵长类、小鼠或大鼠。在某些实施方案中,所述方法进一步包括向感染病原体的受试者施用治疗剂。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,治疗剂包括治疗covid-19的治疗剂。在某些实施方案中,治疗剂包括瑞德西韦、克力芝、伊维菌素、特敏福、阿维根、秋水仙碱、地塞米松、氯喹、羟基氯喹、阿奇霉素、il-6抑制剂(例如,托珠单抗和西鲁库单抗)、激酶抑制剂(例如,阿卡拉替尼、依鲁替尼、泽布替尼、巴瑞替尼、鲁索替尼和托法替尼)、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0019]在至少一个方面,本公开包括用于评价代表病原体的氨基酸序列的部分的保守性的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及使用比对的氨基酸序列鉴定代表病原体的氨基酸序列的一个或多个部分的保守水平。在某些实施方案中,所述部分中的一个或多个被鉴定为在针对病原体的疗法的开发中的候选抗原。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,基因组序列是sars-cov-2基因组序列并且参考序列是sars-cov-2参考序列。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0020]在至少一个方面,本公开包括用于鉴定分离的病原体是否代表循环株系的方法,所述方法包括:从数据结构获得病原体的循环株系的多个完整或部分基因组序列;鉴定循环株系的序列的一个或多个保守部分;获得分离的病原体的多个完整或部分基因组序列;以及通过将分离的病原体的序列的至少一部分与循环株系的序列的鉴定的一个或多个保守部分进行比较来鉴定分离的病原体是否代表循环株系。在某些实施方案中,鉴定循环株系的序列的一个或多个保守部分包括:通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及根据所述部分在比对的氨基酸序列中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法进一步包括储存(例如冷冻)分离的病原体和/或循环株系的样品。在某些实施方案中,所述方法进一步包括从分离的病原体和/或循环株系分离基因组材料和/或储存(例如冷冻)从病原体和/或循环株系分离的基因组材料。在某些实施方案中,所述方法进一步包括如果分离的病原体代表循环株系,则利用和/或维持分离的病原体作为用于研究(例如,用于开发治疗病原体的治疗剂的研究,任选地其中治疗剂可为例如shrna、sirna、抑制剂或抗体)的株系。[0021]在至少一个方面,本公开包括用于鉴定代表病原体的肽的质荷比的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;提供处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;提供处理器将所选择的编码序列转换成相应的氨基酸序列;以及确定一个或多个氨基酸序列或它们的部分的质荷比。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法进一步包括对来自病原体的样品的一种或多种多肽进行质谱法和/或确定来自样品的多肽是否是或包括具有与所确定的质荷比匹配的质荷比的氨基酸序列。[0022]在至少一个方面,本公开包括用于将氨基酸序列鉴定为候选抗生素抗性标记物的方法,所述方法包括:从数据结构获得病原性细菌的多个完整或部分质粒序列;通过计算装置的处理器从质粒序列中提取编码序列;提供处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;提供处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在多个质粒序列中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;选择分类为保守的氨基酸序列部分;以及将所选择的保守序列归类为候选抗生素抗性标记物。在某些实施方案中,所述方法进一步包括根据一个或多个额外标准将候选抗生素抗性标记物鉴定为候选者,所述额外标准包括在所选择的序列中存在跨膜结构域。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得多个完整或部分质粒序列包括通过处理器合并重叠的重叠群以产生至少一些完整或部分质粒序列。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法进一步包括筛选来自一个或多个受试者的一个或多个样品中候选抗生素抗性标记物的存在或不存在,例如,其中一个或多个受试者感染病原性细菌。[0023]在至少一个方面,本公开包括用于鉴定代表质粒的编码序列的一个或多个保守部分的方法,所述方法包括:从数据结构获得病原性细菌的多个完整或部分质粒序列;通过计算装置的处理器从质粒序列中提取编码序列;提供处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;提供处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及根据所述部分在多个质粒序列中的保守水平对氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表质粒的编码序列的一个或多个保守部分。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得多个完整或部分质粒序列包括通过处理器合并重叠的重叠群以产生至少一些完整或部分质粒序列。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述方法包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。在某些实施方案中,所述方法进一步包括筛选来自一个或多个受试者的一个或多个样品中代表质粒的编码序列的保守部分的存在或不存在,例如,其中一个或多个受试者感染病原性细菌。[0024]在至少一个方面,本公开包括用于自动鉴定代表病原体的编码序列的一个或多个保守部分的系统,所述系统包括:处理器;以及其上具有指令的存储器,所述指令在由处理器执行时引起处理器:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表病原体的编码序列的一个或多个保守部分。在某些实施方案中,所述指令当由处理器执行时引起处理器对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,所述指令当由处理器执行时引起处理器创建相似性量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,数据结构包括重叠群,并且其中所述指令当由处理器执行时引起处理器通过处理器合并重叠的重叠群以产生至少一些完整或部分基因组序列来获得病原体的不同株系的多个完整或部分基因组序列。在某些实施方案中,所述指令当由处理器执行时引起处理器评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,所述指令当由处理器执行时引起处理器评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0025]在至少一个方面,本公开包括用于自动鉴定代表质粒的编码序列的一个或多个保守部分的系统,该系统包括:处理器;以及其上具有指令的存储器,所述指令当由处理器执行时引起处理器:从数据结构获得病原性细菌的多个完整或部分质粒序列;通过处理器从质粒序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;以及根据所述部分在多个质粒序列中的保守水平对氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表质粒的编码序列的一个或多个保守部分。在某些实施方案中,所述指令当由处理器执行时引起处理器对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,所述指令当由处理器执行时引起处理器创建相似性量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,数据结构包括重叠群,并且其中所述指令当由处理器执行时引起处理器通过处理器合并重叠的重叠群以产生至少一些完整或部分质粒序列来获得病原性细菌的多个完整或部分质粒序列。在某些实施方案中,所述指令当由处理器执行时引起处理器评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,所述指令当由处理器执行时引起处理器评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0026]在至少一个方面,本公开包括用于在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变的治疗剂,所述用途包括:在向每个受试者施用治疗剂之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;在比对的氨基酸序列中鉴定比对的氨基酸序列中比参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一种或多种推定的逃逸突变。在某些实施方案中,参考包括代表典型病原体序列的一种或多种完整或部分病原体基因组序列、病原体的一个或多个临床株系、来自施用治疗剂的一个或多个受试者的病原体的一个或多个早期样品或来自未施用治疗剂的受试者的病原体的一个或多个样品。在某些实施方案中,所述用途进一步包括确定一个或多个推定的逃逸突变是否降低治疗剂与参考多肽的结合亲和性的步骤。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述用途包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述用途包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0027]在至少一个方面,本公开包括用于治疗病原体感染的治疗剂,所述用途包括:通过以下方式选择氨基酸序列的保守部分:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;以及选择比对的氨基酸序列的保守部分;以及如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的保守部分,则向受试者施用治疗剂,其中治疗剂选择性地结合氨基酸序列的保守部分。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述用途包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0028]在至少一个方面,本公开包括治疗剂在制造药物中的用途,所述药物用于在向一个或多个受试者施用所述药物以治疗病原体感染之后鉴定一种或多种推定的逃逸突变,所述用途包括:在向每个受试者施用所述药物之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;提供处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;在比对的氨基酸序列中鉴定比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一种或多种推定的逃逸突变。在某些实施方案中,参考包括代表典型病原体序列的一种或多种完整或部分病原体基因组序列、病原体的一个或多个临床株系、来自施用治疗剂的一个或多个受试者的病原体的一个或多个早期样品或来自未施用治疗剂的受试者的病原体的一个或多个样品。在某些实施方案中,所述用途进一步包括确定一个或多个推定的逃逸突变是否降低治疗剂与参考多肽的结合亲和性的步骤。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,所述用途包括评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述用途包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0029]在至少一个方面,本公开包括治疗剂用于制造用于治疗病原体感染的药物的用途,所述用途包括:通过以下方式选择氨基酸序列的保守部分:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;通过处理器将所选择的编码序列转换成相应的氨基酸序列;通过处理器对氨基酸序列进行比对;根据所述部分在病原体的不同株系中的保守水平对比对的氨基酸序列的多个部分中的每一个进行分类;以及选择比对的氨基酸序列的保守部分;以及如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的保守部分,则向受试者施用所述药物,其中治疗剂选择性地结合氨基酸序列的保守部分。在某些实施方案中,数据结构包括重叠群,并且其中从数据结构获得病原体的不同株系的多个完整或部分基因组序列包括通过处理器合并重叠的重叠群以产生完整或部分基因组序列的至少一部分。在某些实施方案中,归类步骤包括量化多个对中的每一个的同一性的量度和覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。在某些实施方案中,归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性的量度,相似性量度中的每一个是查询序列和主题序列之间的同一性的量度以及查询序列和主题序列之间的覆盖率的量度的函数。在某些实施方案中,计算步骤包括创建相似性的量度的矩阵,并呈现矩阵的图形表示,从而显示查询序列和主题序列之间的保守水平。在某些实施方案中,图形表示包括热图、图形和系统发育中的一个或多个。在某些实施方案中,同一性的量度包括突变数量。在某些实施方案中,覆盖率的量度包括覆盖率百分比。在某些实施方案中,同一性的量度包括计算e值。在某些实施方案中,评价以下中的一种或多种:编码与病原体相关的蛋白质的核酸的编码序列;编码与病原体相关的蛋白质的核酸序列的保守序列;编码蛋白质的核酸的非保守序列;与病原体相关的特定蛋白质内的保守结构域;和与病原体相关的特定蛋白质内的非保守结构域。在某些实施方案中,氨基酸序列的每个部分包括一个或多个氨基酸位置。在某些实施方案中,病原体是病毒。在某些实施方案中,病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。在某些实施方案中,病毒是冠状病毒。在某些实施方案中,冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。在某些实施方案中,冠状病毒是sars-cov-2。在某些实施方案中,所述用途包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。在某些实施方案中,治疗剂包含抗体。在某些实施方案中,抗体结合sars-cov-2。在某些实施方案中,抗体结合sars-cov-2刺突蛋白。在某些实施方案中,根据表3,抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。在某些实施方案中,病原体是细菌。在某些实施方案中,细菌是葡萄球菌属物种或假单胞菌属物种。[0030]在至少一个方面,本公开包括确定由抗体结合的病原体表位是否保守的方法,所述方法包括:从数据结构获得病原体的不同株系的多个完整或部分基因组序列;通过计算装置的处理器从基因组序列中提取编码序列;将编码序列与编码病原体表位的参考序列进行比较;通过处理器根据同一性的量度和覆盖率的量度对编码序列进行归类,其中同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;根据同一性的量度和覆盖率的量度在归类的编码序列中选择编码序列;将所选择的编码序列转换成相应的氨基酸序列;以及确定病原体的不同株系中的病原体表位的保守水平。附图说明[0031]本文包括的由下图构成的图仅用于说明目的,而非限制目的。[0032]图1是根据说明性实施方案的示出示例性序列分析工作流程的示意图。[0033]图2是示出当从可公开访问的数据库提取序列时或当手动提供序列以根据本公开的方法或系统进行分析时要提供的一组示例性信息的示意图。[0034]图3是示出根据本公开的方法或系统将数据组织到文件夹中用于分析的示例性系统的示意图。[0035]图4是根据说明性实施方案的示出从一个或多个可公开访问的数据库(例如,ncbi)下载到文件夹中的序列和/或注释信息的拷贝的示例性分布的示意图。如图4所示,将下载的序列和/或注释信息拷贝到三个文件夹中:参考序列文件夹、比对器数据库文件夹和注释文件夹。[0036]图5是根据说明性实施方案的示出用于从示例性可公开访问的数据库(ncbi)下载和策划序列的示例性步骤的示意图。[0037]图6是示出用于输入查询序列以供在本公开的方法或系统中使用的示例性步骤的示意图。[0038]图7是根据说明性实施方案的示出分别存储在查询序列文件夹和比对器数据库文件夹中的查询序列和主题序列(参考序列)的成对blast比较的示例性方法的示意图。[0039]图8是根据说明性实施方案的示出用于应用blast来执行查询序列和主题序列(参考序列)的成对序列比较的示例性步骤的示意图。[0040]图9是根据说明性实施方案的示出用于生成基因输出表(“got表”)的blast结果、序列信息和序列注释信息的示例性编译的示意图。[0041]图10是根据说明性实施方案的示出用于编译blast结果以包括在got表中的示例性步骤的示意图。[0042]图11是根据说明性实施方案的示出用于编译got表中与重叠群相关的信息的示例性步骤的示意图。[0043]图12是根据说明性实施方案的示出用于在成对比较之后鉴定匹配序列、计算匹配序列的突变百分比以及编译可公开访问的数据库(ncbi)中可获得的特征文件注释的示例性步骤的示意图。[0044]图13是根据说明性实施方案的示出got表的示例性内容的示意图。[0045]图14是根据说明性实施方案的示出用于为每个查询序列生成比较表的示例性步骤的示意图,所述比较表包括用于成对比较的相似性评分矩阵,所述相似性评分值基于覆盖率百分比和突变数量来分配。[0046]图15是根据说明性实施方案的示出用于在热图或条形图中表示相似性评分的示例性步骤的示意图。[0047]图16是根据说明性实施方案的示出用于提取编码序列的示例性步骤的示意图,所述提取的序列可经翻译和比对。步骤为重叠群提供一种示例性方法。步骤提供生成表的示例性方法,所述表包括提取的序列的独特型式的数量和频率。[0048]图17是根据说明性实施方案的示出用于从提取的编码序列生成系统发育的示例性方法的示意图。[0049]图18是根据说明性实施方案的示出用于产生got表和可从got表中存在的数据生成的示例性输出的示例性步骤的示意图。[0050]图19是示出在ncbi中代表的并且适合用于根据本文公开的方法和系统的分析的示例性细菌基因组的图形。[0051]图20是示出本文公开的示例性系统的示意图。[0052]图21是表示人感染乙型肝炎病毒(hbv)的示意图,所述感染可导致肝细胞癌。[0053]图22是示出示例性hbv环状基因组的示意图。[0054]图23是示出具有由括号鉴定的基因s的示例性hvc环状基因组的示意图。[0055]图24是示出hbv的基因型的示例性分布的示意图。[0056]图25是示出适合用于根据本公开的方法和系统进行分析的示例性序列结构的示意图,所述示例性序列结构包括手动提供和/或从诸如ncbi的可公开访问的数据库下载的环状、线性和片段化序列。[0057]图26是根据说明性实施方案的代表从基因组序列中提取编码序列的示意图。从基因组序列中提取的编码序列可在不同长度和取向的基因组序列中找到。[0058]图27是根据说明性实施方案的代表来自查询编码序列集合的单个编码序列与多个输入基因组序列中的每一个的示例性成对blast比较、例如来自提取的查询编码序列集合的提取的查询编码序列与作为参考基因组序列的多个主题序列中的每一个的比较的示意图。至少部分由于诸如参考序列的主题序列可在核苷酸序列和内容上变化,所以提取的查询序列与每个参考序列的比对可在比对的相对位置、覆盖长度和/或取向上变化。在一些实施方案中,将不会发现主题序列和参考序列具有相应的序列(即,比较可在一个或多个特定主题基因组序列中产生“无命中”)。在某些实施方案中,从主题基因组序列中提取编码序列,将每个主题编码序列与一个或多个查询基因组序列进行比较(例如,通过blast),并为每个比较确定一个或多个序列归类因子(例如,覆盖长度和同一性百分比)。在各种实施方案中,如果覆盖长度和同一性百分比各自大于各别的阈值,则提取相应的查询序列,并且可进一步对其进行分析或评价。应用阈值来确定每个查询基因组序列或它的部分是否与参考序列相似。本文提供的方法和系统适用于代表完整基因组的基因组序列以及代表完整基因组的一个或多个部分的基因组序列。[0059]图28是根据说明性实施方案的示出单个参考序列与多个输入查询基因组序列中的每一个的成对blast比较、例如多个查询编码序列与作为参考基因组序列的主题基因组序列的比较的结果的示例性汇总的示意图。汇总的第1列指示要与查询基因组序列进行比较的参考基因组序列(b_李(lee)_1940)。具体来说,所示表涉及参考基因组序列的特定基因,其编码在参考基因组序列中注释的特定已知产物血凝素。所述表显示将来自参考基因组的血凝素参考序列与9个查询基因组中的每一个进行比较。归类因子用于确定每个查询基因组中是否存在对应于血凝素的序列(是、否或部分,如“基因存在”列中所指示)。相应查询序列的取向(“链”)也包括在表中。对于每个比较,覆盖率百分比、突变数量(snp)和比对空位在表中注明。[0060]图29是根据说明性实施方案的示出四个示例图的示意图,每个示例图示出与四个查询序列之一相比具有指定数量和类型的变异的主题基因组的数量。[0061]图30是根据说明性实施方案的示出相似性评分的示例性热图的示意图,所述相似性评分代表作为参考基因组序列(x轴)的20个示例性主题序列中的每一个与八个示例性查询编码序列中的每一个之间的保守水平。[0062]图31是根据说明性实施方案的flua当代株系的全基因组系统发育的示例性表示。[0063]图32是根据说明性实施方案的示出呈矩形布局的示例性系统发育的示意图。[0064]图33是根据说明性实施方案的示出呈极地布局的示例性系统发育的示意图。[0065]图34是根据说明性实施方案的示出从基因组序列提取的示例性编码序列的示意图。[0066]图35是根据说明性实施方案的示出图34的示例性编码序列的翻译的示意图,并且包括所分析的基因组内的特定变体序列以及它们的频率的汇总。[0067]图36是根据说明性实施方案的示出源自8个不同的成对比较基因组的氨基酸序列的示例性比对的示意图。[0068]图37是用于提供本文所述的系统和方法的计算机网络环境的示意图。[0069]图38是可用于实现本文所述的系统和方法的计算装置和移动计算装置的示意图。[0070]图39是根据说明性实施方案的用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的示例性方法的方块流程图。[0071]图40是根据说明性实施方案的用于鉴定代表病原体的编码序列的一个或多个保守部分的示例性方法的方块流程图。[0072]图41是根据说明性实施方案的用于鉴定分离的病原体是否代表循环株系的示例性方法的方块流程图。[0073]图42是根据说明性实施方案的用于将氨基酸序列鉴定为候选抗生素抗性标记物的示例性方法的方块流程图。[0074]图43是根据说明性实施方案的用于鉴定代表质粒的编码序列的一个或多个保守部分的示例性方法的方块流程图。[0075]图44是根据说明性实施方案的用于鉴定代表病原体的肽的质荷比、例如用于鉴定所述代表病原体的肽的质谱法靶标的示例性方法的方块流程图。[0076]图45是根据说明性实施方案的用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的示例性方法的方块流程图。[0077]图46是根据说明性实施方案的用于将氨基酸序列鉴定为候选抗生素抗性标记物的示例性方法的方块流程图。[0078]图47是诸如sars-cov-2的示例性冠状病毒的示意图。冠状病毒结构具有外部脂质膜,其包括嵌入的跨膜蛋白,包括但不限于刺突蛋白、套膜蛋白和膜糖蛋白。所述示意图包括与核衣壳蛋白相关的冠状病毒rna病毒基因组的表示。[0079]图48是确定一组查询序列中的主题序列的氨基酸保守性的方法的示意性代表图。从查询序列和主题序列中提取编码序列。执行提取的查询编码序列和提取的主题编码序列的成对blast比较。来自成对blast的数据用于产生数据表,包括归类因子,诸如对于每个成对比较的同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度内的同一性百分比、e值、突变数量和突变百分比。然后,基于一个或多个归类因子的阈值对blast比较结果进行归类。从分析中去除不符合纳入阈值和/或符合排除阈值的类别中的比较。翻译剩余查询序列,并将所得氨基酸序列与相应的翻译的主题序列进行比对。从这些比对评价翻译的查询序列中翻译的主题序列的氨基酸保守性。[0080]图49是说明从参考基因组中提取刺突编码序列的示意图。提取是基于基因库(genbank)文件注释。[0081]图50是示出通过blast与参考刺突编码序列比较的刺突编码序列随时间的累积数量的图形。如取样序列的日期和数量所示,获取并分析大量序列,代表在欧洲、北美、亚洲、大洋洲、南美和非洲分离的序列。[0082]图51是说明刺突氨基酸序列的比对的示意图。基于突变数量和覆盖长度筛选过滤后保留用于分析的编码序列通过blast进行翻译和比对。然后可检查和/或比较比对的序列,以鉴定在参考刺突蛋白序列的每个比对位置存在的氨基酸范围。[0083]图52是部分说明通过分析的编码序列的氨基酸翻译本的比对鉴定的氨基酸变异的示意图。具体实施方式[0084]基因组和质粒序列信息[0085]本公开的方法和系统包括基因组序列和/或质粒序列的分析。基因组序列可包括完整和/或部分基因组序列。质粒序列可包括完整和/或部分质粒序列。不同生物体间的基因组的大小和结构不同。例如,真核基因组通常包括多个染色体,并且原核基因组通常包括单个环状核酸。原核生物另外可包括本领域中称为质粒的较小的独立分子。质粒可编码基因,例如,编码赋予抗生素抗性的蛋白质的基因(抗生素抗性标记物)。本文公开的适用于一种形式的遗传序列信息的各种实施方案也适用于其他形式,例如,关于基因组序列公开的实施方案也适用于质粒序列。[0086]完整基因组序列可包括代表生物体的整个基因组的单个序列。完整基因组序列可包括一起代表生物体的整个基因组的多个序列。部分基因组序列可指代表基因组序列的核酸的邻接亚组的任何单个序列。部分基因组序列可包括一起代表基因组序列的核酸的邻接亚组的多个序列。[0087]在各种实施方案中,基因组序列是病原体基因组的完整或部分序列,例如任何病原性细菌、酵母、原生动物或病毒的完整或部分基因组。例如,在一些实施方案中,基因组序列是冠状病毒(例如严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov2)或中东呼吸综合征相关冠状病毒(mers-cov))的基因组的完整或部分序列。[0088]完整质粒序列可包括代表生物体的整个基因组的单个序列。完整质粒序列可包括一起代表生物体的整个基因组的多个序列。部分质粒序列可指代表质粒序列的核酸的邻接亚组的任何单个序列。部分质粒序列可包括一起代表质粒序列的核酸的邻接亚组的多个序列。[0089]在一些实施方案中,一起代表较大核酸序列的个别序列可称为重叠群。在一些实施方案中,可组装重叠群以提供它们所代表的较大核酸序列的序列。[0090]在各种实施方案中,完整或部分基因组序列可包括至少(例如)约1kb、5kb、10kb、50kb、100kb、500kb、1mb、2mb、3mb、4mb、5mb、10mb、20mb、50mb、100mb、500mb、1,000mb、2,000mb、3,000mb或更多。在各种实施方案中,完整基因组序列可包括与相关生物体的基因组的核苷酸的典型数量相等的核苷酸数量。在各种实施方案中,完整基因组序列可包括在相关生物体的基因组典型的核苷酸数量范围内的核苷酸数量。[0091]在各种实施方案中,完整或部分质粒序列可包括至少(例如)约1kb、5kb、10kb、50kb、100kb、200kb或更多。在各种实施方案中,完整质粒序列可包括与相关质粒的序列的核苷酸的典型数量相等的核苷酸数量。在各种实施方案中,完整基因组序列可包括在相关质粒典型的核苷酸数量范围内的核苷酸数量。[0092]本公开的基因组序列或质粒序列可包括可在可公开访问的数据库获得的一个或多个序列。各种可公开访问的数据库包括可访问的基因组和质粒序列信息(例如,参见图19)。可公开访问的基因组和/或质粒序列信息的数据库的一个实例是国家生物技术信息中心(ncbi)的基因库。另一个可公开访问的基因组和/或质粒序列信息数据库是欧洲分子生物学实验室(europeanmolecularbiologylaboratory,embl)的国际协作核苷酸序列数据库(internationalnucleotidesequencedatabasecollaboration,insdc)(可在ncbi.nlm.nih.gov/sra/万维网上获得)、日本dna数据库(ddbj,dnadatabankofjapan)和ncbi。另一个实例是1000基因组计划(1000genomesproject)。[0093]为了提供可公开访问的基因组序列信息资源扩展的仅一个实例,从2010年8月至2017年8月,公共数据库从约19个金黄色葡萄球菌基因组序列扩展至约48,259个源自约4,155个独立研究的金黄色葡萄球菌基因组序列。大多数序列数据寄存在美国国家生物技术信息中心(ncbi)的序列读段档案中,ncbi是insdc的一部分。在金黄色葡萄球菌基因组序列中,约84%(约42,285)代表短的dna读段(reads)或小片段。剩余部分(约7,974;约16%)组装成更大的dna区段,且仅约2%(约166/7,974)是无空位的并且完全注释的。因此,完全组装和注释的完整基因组序列占ncbi中可获得的金黄色葡萄球菌基因组的一小部分。[0094]本公开的基因组序列或质粒序列可包括源自生物样品且未在可公开访问的数据库中发现的序列。生物样品可包括(例如)实验室样品或临床样品。基因组序列或质粒序列可(例如)通过本领域已知的各种dna测序方法(例如高通量测序和/或多重测序)中的任一种来确定。[0095]数据结构可包括(例如,存储)与本公开的基因组序列和/或质粒序列相关的信息,包括序列本身。因此,本公开的数据结构可包括(但不限于)基因组序列信息的可公开访问的数据库、包括序列信息的私有结构、包括从高通量测序系统直接输入的数据的结构及它们的组合。[0096]代表双链dna的基因组序列可以任一链(有时称为“watson”和“crick”链或称为“5′”和“3′”链)的形式提供。两条链通常应理解为是互补的,使得任一链的序列公开另一链的序列。[0097]可获取多个完整或部分基因组序列和/或质粒序列,包括在数据结构中,并根据本领域已知的各种技术从数据结构获得。从数据结构获得或可获得的基因组序列和/或质粒序列可为来自现有记录(例如,在公共数据库中)的序列和/或通过样品测序获取的序列。在各种实施方案中,数据结构可包括代表特定来源(例如,特定物种,例如,人或特定病原体物种)或与特定来源相关的不同序列。在各种实施方案中,代表特定来源或与特定来源相关的每个不同序列可称为株系。在各种实施方案中,从数据结构获得代表特定来源或与特定源来源相关的多个序列是有利的,使得例如根据本文公开的各种方法和系统可比较和/或对比所获得的序列。[0098]编码序列和编码的氨基酸序列的提取[0099]本公开的基因组和质粒序列可包括编码序列。各种基因组和质粒包括编码可从基因组或质粒表达的蛋白质的氨基酸的核苷酸序列(所述核苷酸序列可称为编码序列)和不编码可从所述序列表达的蛋白质的氨基酸的核苷酸序列(所述核苷酸序列可称为非编码序列)。编码序列可以称为密码子的三联体读取,其中所述密码子的每一个编码一个氨基酸。因此,本公开的编码序列是由密码子组成并编码蛋白质或它的一部分的序列。非编码序列(例如,启动子或内含子)在一些情况下与编码序列相邻和/或散布在编码序列中。可通过本领域已知的多种技术、包括但不限于通过编码氨基酸的邻接和/或框内密码子的数量和/或通过与已知序列(诸如已知编码序列或由编码序列编码的已知蛋白质)的比较,来区分编码序列与非编码序列。提取(鉴定和/或分离)编码序列的各种方法是本领域已知的。提取编码序列的各种方法包括分析为开放阅读框提供的序列,所述开放阅读框除了其他特征之外可包括不包括终止密码子的连续系列密码子,例如不包括终止密码子的至少约20个、30个、40个、50个、60个、70个、80个、90个、100个、125个、150个、175个、200个、250个或300个或更多个密码子的连续系列。在一些实施方案中,可公开访问的数据库中的序列与划分编码序列的位置的注释信息相关。因此,数据库注释和本领域已知的各种方法中的任一种或两种都可用于从基因组和质粒序列中提取编码序列。[0100]一旦提取了编码序列,就可通过应用遗传密码确定由编码序列编码的氨基酸序列。不是终止密码子的每个密码子对应于特定氨基酸。生物体之间的遗传密码可不同。因此,当将编码序列转换成氨基酸序列时,可应用适合于基因组序列或质粒编码序列的来源和/或上下文的遗传密码。通过应用遗传密码已经转换成氨基酸序列的核酸序列可称为核酸序列的翻译。[0101]人类遗传密码与其他遗传密码一样,可表示为dna密码子表,如表1中所见。大多数密码子编码特定氨基酸,而几个密码子编码不编码任何氨基酸的“终止(stop)”信号。表1包括应用于表示核酸和氨基酸序列的某些一般惯例。在提及核酸序列时,字母a、c、g和t分别指示腺嘌呤(a)、胞嘧啶(c)、鸟嘌呤(g)和胸腺嘧啶(t)。关于氨基酸序列,20个氨基酸中的每一个可由如下的特定字母或三个字母的组表示:丙氨酸(a;ala)、精氨酸(r;arg)、天冬酰胺(n;asn)、天冬氨酸(d;asp)、半胱氨酸(c;cys)、谷氨酸(e;glu)、谷氨酰胺(q;gln)、甘氨酸(g;gly)、组氨酸(h;his)、异亮氨酸(i;ile)、亮氨酸(l;leu)、赖氨酸(k;lys)、甲硫氨酸(m;met)、苯丙氨酸(f;phe)、脯氨酸(p;pro)、丝氨酸(s;ser)、苏氨酸(t;thr)、色氨酸(w;trp)、酪氨酸(y;tyr)、缬氨酸(v;val)。[0102]表1[0103][0104]从序列的成对比较生成的数据[0105]在某些实施方案中,本公开的方法和系统包括确定用于表征序列之间的比对的测量。示例性测量包括同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和系统发育(例如,系统发育分组和/或系统发育关系),所有这些都在本文中更详细地讨论。已发现,使用覆盖率的量度(例如,覆盖率百分比和/或覆盖长度)和同一性的量度(例如,同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比)两者来表征比对有效且高效地实现了大量的成对比较,所述成对比较可用于例如在保守性评价中鉴定适当匹配的序列。成对比较可用于评价聚合序列之间、例如核酸序列(例如dna分子和/或rna分子)之间和/或氨基酸序列之间的总体关联性。在本文提供的各种方法和系统中,成对比较用于评价所提取的编码序列和/或它们的翻译之间的总体关联性。在一些实施方案中,两个序列的成对比较是在查询序列和主题序列(例如,参考序列)之间进行,所述比较包括比对和确定同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系)中的一个或多个或全部。在各种实施方案中,诸如参考序列的主题序列可为查询序列与之比较的基线。通常,查询序列和主题序列分别是指一个或多个序列的集合,其中查询序列与主题序列成对比较。在一些实施方案中,不将查询序列与查询序列进行比较,并且不将主题序列与主题序列进行比较,除非查询序列和主题序列具有同一的序列(例如,在其中查询序列和主题序列是序列的同一集合的实施方案中)。主题序列可为或包括参考序列。参考序列可为完整或部分基因组序列,所述完整或部分基因组序列代表群体、物种、株系、生物体等的相应的完整或部分基因组序列,例如其包括一个或多个特定基因或它们的部分和/或编码一个或多个蛋白质或它们的部分。可基于但不限于序列可用性、公众可访问性、历史背景、惯例、准则、标准实践、统计分析、实际考虑或用户偏好中的一个或多个来选择参考序列和/或将其用作代表性序列。如本文所公开的,从序列的成对比较生成的数据可包括同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系)中的一个或多个或全部,其中的每一个提供与所分析的序列相关的不同信息。[0106]在执行查询序列与参考序列的成对比较时,本文发现可显著有效和高效地为给定成对比较确定同一性的测量和覆盖率的测量两者,然后在将查询序列(例如编码序列)归类为两组或多组时使用这两种测量,例如,用于在评价一个或多个氨基酸序列或它们的部分的保守性时鉴定适当可比较的序列部分。同一性的测量的实例包括同一性百分比;同一性百分比/预定覆盖长度;突变数量;和突变百分比(例如单核苷酸多型性snp/大小)。覆盖率的测量的实例包括覆盖率百分比和覆盖长度。[0107]用于比对两种提供的序列的方法包括算法和/或商业上可获得的计算机程序,诸如用于核苷酸序列的blastn和用于氨基酸序列的blastp、有空位的blast和psi-blast。在使用所述比对算法中的一种或多种对两个序列(或一个或两个序列的补体)进行比对后,可计算覆盖率和同一性的量度。在某些实施方案中,为了最佳比对,在第一序列和第二序列中的一个或两个中引入空位,并且为了比较的目的,可忽略不相同的序列。比对是指将两个或多个序列的核苷酸或氨基酸残基进行匹配,以达到最大同一性百分比水平、并且在一些实施方案中(例如,在氨基酸序列的比对中)使物理化学性质的保存最大化的过程或结果。[0108]比对后,可比较第一序列和第二序列的相应位置处的核苷酸或氨基酸。当第一序列中的一个位置被与第二序列中相应位置相同的残基(例如,核苷酸或氨基酸)占据时,则分子在该位置是相同的。两个序列之间的同一性百分比随着序列所共用的相同位置的数量变化,任选地考虑空位的数量和每个空位的长度,为了两个序列的最佳比对,可能需要引入这些空位。因此,确定同一性百分比需要确定比对位置的同一性或非同一性。使用计算算法(诸如blast(基本局部比对搜索工具)),可完成两个序列之间的同一性百分比的确定。[0109]同一性百分比可表示比对序列内两个比对序列中具有相同残基的位置的分数。在一些实施方案中,如果两个序列的相应残基的至少约50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更多在相关序列上同一,则认为两个序列基本上同一。如果序列因保守取代(例如,因不改变编码氨基酸序列的核苷酸取代,或因其中取代的氨基酸具有相似结构或功能特征的氨基酸取代(例如,用同一类型的不同氨基酸置换疏水、亲水、极性或非极性类型的氨基酸))而不同,则序列可基本上相似。[0110]在成对比较中分析的每个序列也可根据通过与第二序列的比对所覆盖的第一序列的百分比(即,与第二序列比对的第一序列的百分比,其可称为覆盖率或覆盖率百分比)(例如,与查询序列比对的主题序列长度的%或与主题序列比对的查询序列长度的%)来评价。[0111]两个序列的比对可产生覆盖长度和/或覆盖率百分比。在第一序列和第二序列的比对中,覆盖长度是指经比对的单元(例如核苷酸或氨基酸)的数量。为免生疑问,在计算覆盖长度时,一对相应的位置(即第一序列的核苷酸或氨基酸和第二序列的相应定位的核苷酸或氨基酸)计数为一个覆盖长度单元。在第一个序列和第二个序列的比对中,覆盖率百分比是指序列的比对中所包括的查询的百分比。覆盖率百分比可指主题序列中与查询序列的相应核苷酸或氨基酸比对的核苷酸或氨基酸的百分比,无论比对的核苷酸或氨基酸是同一的还是不同一的。覆盖率百分比也可指查询序列中与主题序列的相应核苷酸或氨基酸比对的核苷酸或氨基酸的百分比,无论比对的核苷酸或氨基酸是同一的还是不同一的。在本文提供的各种方法和系统中,覆盖率百分比具体来说是指主题序列中与查询序列的相应核苷酸或氨基酸比对的核苷酸或氨基酸的百分比,不管比对的核苷酸或氨基酸是同一的还是不同一的。可确定连续和有空位的比对的覆盖率百分比。[0112]在各种实施方案中,至少由于通过比较比对的核苷酸或氨基酸以确定每个比对的核苷酸或氨基酸对的同一性或非同一性来确定同一性百分比,所以序列空位不会降低同一性百分比。为了提供用于说明目的的一个实例,如果80个氨基酸的查询序列与100个氨基酸的主题序列比对,其中主题序列的前40个氨基酸与查询序列的前40个氨基酸比对具有完美同一性,并且主题序列的后40个氨基酸与查询序列的后40个氨基酸比对具有完美同一性,则同一性百分比将等于100%,但覆盖率百分比将是80%。因此,在一些实施方案中,尽管100%同一性,仍将查询序列归类为部分或“缺乏完整性”,落入70%至95%覆盖率的阈值范围内。[0113]在各种实施方案中,两个序列的比对可用于确定在预定覆盖长度上的同一性百分比。预定覆盖长度可为多个核苷酸和/或氨基酸,其中在预定覆盖长度上的同一性百分比可指查询序列与主题序列在其长度等于预定覆盖长度和/或大于预定覆盖长度的比对的任何部分上的同一性百分比。为免生疑问,比对的部分可为比对的核苷酸或氨基酸的任何足够长的亚组,使得单个比对可包括用于分析的多个足够长的部分,这些部分可为重叠的、不重叠的、相邻的或不相邻的。在各种实施方案中,两个序列的比对在预定覆盖长度上的同一性百分比可表示为与比对的任何足够长的部分相关的最高同一性百分比。[0114]计算同一性百分比的各种技术会产生预计(e)值。例如,使用blast确定同一性百分比会产生e值。e值代表比对偶然发生的可能性(例如,而非生物学意义上的相似性的结果)。一些来源将e值描述为基本上是背景噪声的描述。e值越接近零,比对就越显著。e值至少部分与确定的比对同一性百分比和比对长度相关。大致地,较短和较低的比对同一性百分比将比较长和较高的比对同一性百分比具有更高的e值。e值可用于对多个比对进行排序,或可单独或与其他标准组合选择作为用于归类比对的显著性阈值。[0115]在一些实施方案中,对于成对比较中分析的每个查询序列,可相对于主题序列确定比对内的序列变异的数量。变异可为第一序列和第二序列的比对位置之间的差异,其中所述序列是核酸序列或其中所述序列是氨基酸序列(例如,查询序列和诸如参考序列的主题序列之间的差异)。核酸序列的变异或氨基酸序列的变异在本文中可称为突变。核酸序列的变异可为单核苷酸多型性(“snp”)。[0116]在一些实施方案中,对于成对比较中分析的每个查询序列,查询序列和主题序列之间的序列变异的数量(即,不匹配的查询序列和主题序列之间的比对内序列位置的数量)可称为“突变数量”。在一些实施方案中,对于成对比较中分析的每个查询序列,可确定序列覆盖长度的每个核苷酸或氨基酸的序列变异的数量。此比率可为比对长度上比对内的序列变异的数量(“突变百分比”,在本文中或者称为“突变/大小”,其实例是“snp/大小”)。[0117]在一些实施方案中,成对比较的结果可用于产生一个或多个基因组、质粒、基因、编码序列或翻译的编码序列的系统发育。在一些实施方案中,系统发育可基于通过成对比较产生的同一性百分比数据。在一些实施方案中,系统发育可基于通过成对比较产生的突变百分比数据。用于从所提供的数据产生系统发育的工具和技术是本领域已知的。[0118]使用最保守的主题序列的同一性百分比或突变百分比成对比较结果,可产生基因组水平或质粒水平的系统发育。例如,基因组水平或质粒水平的系统发育可基于约前1个、前2个、前3个、前4个、前5个、前10个、前20个、前25个、前50个、前100个、前1%、前2%、前5%、前10%、前15%、前20%、前25%或前50%的保守的成对比较序列(例如,顶部基因、编码序列或翻译的编码序列氨基酸序列)。可使用例如同一性百分比或突变百分比数据,基于成对比较的结果对保守性进行排序。[0119]同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比中的一个或多个或全部中的任一个可代表核酸或氨基酸比对的全长或它的一个或多个部分。完整或部分基因组序列的示例性部分可包括例如基因、编码序列、个别核苷酸或一组邻接核苷酸(例如,约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、20个、30个、40个、50个、100个、150个、200个、250个、500个、1,000个、1,500个、2,000个、2,500个、3,000个、5,000个、10,000个或更多个核苷酸)。氨基酸序列的示例性部分可包括例如蛋白质、结构域、个别氨基酸或一组邻接氨基酸(例如,约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、20个、30个、40个、50个、100个、150个、200个、250个、300个、350个、400个、450个或500个或更多个氨基酸)。在一些实施方案中,核酸序列的一部分可包括具有约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、20个、30个、40个、50个、100个、150个、200个、250个、500个、1,000个、1,500个、2,000个、2,500个或3,000个核苷酸的下限和约50个、100个、150个、200个、250个、500个、1,000个、1,500个、2,000个、2,500个、3,000个、5,000个、10,000个或更多个核苷酸的上限的核苷酸数。在一些实施方案中,氨基酸序列的部分可包括具有约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、20个、30个、40个、50个、100个、150个、200个、250个或300个氨基酸的下限和约10个、20个、30个、40个、50个、100个、150个、200个、250个、300个、350个、400个、450个或500个或更多个氨基酸的上限的氨基酸数。在各种实施方案中,可个别地分析核酸或氨基酸序列的每个重叠或相邻的非重叠部分。因此,第一比对的核苷酸序列和第二比对的核苷酸序列可具有代表第一比对的序列和第二比对的序列的所有比对的核苷酸之间的同一性百分比的总同一性百分比,并且可具有代表第一比对的序列和第二比对的序列的比对的核苷酸的亚组之间的同一性百分比的一个或多个同一性百分比。第一比对的氨基酸序列和第二比对的氨基酸序列可具有代表第一比对的序列和第二比对的序列的所有比对的氨基酸之间的同一性百分比的总同一性百分比,并且可具有代表第一比对的序列和第二比对的序列的比对的氨基酸的亚组之间的同一性百分比的一个或多个同一性百分比。比对的核苷酸或氨基酸的亚组的同一性百分比可为不同于所有比对的核苷酸或氨基酸的总同一性百分比的百分比。[0120]在各种实施方案中,同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比中的一个或多个或全部中的任一个可显示为图形或热图。在各种实施方案中,图形或热图的至少一个轴包括序列的成对比较中包括的序列,并且至少一个额外轴包括通过序列的成对比较生成的数据。[0121]在一些实施方案中,分析基因组序列的单个集合或质粒序列的单个集合,其中以成对方式比较所分析集合的所有成员(即,单个集合用作查询序列集合和参考序列集合两者),以确定每个成对比较的同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比。在一些实施方案中,分析基因组序列的集合或质粒序列的集合,其中将所分析的集合的每个成员与主题序列进行比较,以确定每个比较的同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比。[0122]在一些实施方案中,集合的每个基因组或质粒序列可属于同一物种。在一些实施方案中,集合的每个基因组或质粒序列可为或包括代表同一属、科、目、纲、门、界或域的生物体的序列。在一些实施方案中,集合的每个基因组或质粒序列可为或包括代表同一基因或它的一部分的序列。在一些实施方案中,单个集合的每个基因组或质粒序列可为或包括代表同一编码序列或它的一部分的序列。[0123]某些实施方案中,分析包括两个集合,每个集合是基因组序列的集合或每个集合是质粒序列的集合。在所述情况下,第一个集合可称为主题集合,并且第二个集合可称为查询集合。在包括主题集合和查询集合的某些实施方案中,以成对方式将查询集合的每个序列与主题集合的每个序列进行比较,以确定每个比较的同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比。[0124]在一些实施方案中,分析包括序列的单个集合,并且以成对方式将每个序列彼此进行比较,使得在至少某些实施方案中,序列的单个集合是主题集合和查询集合两者。无论所分析的序列包括单个序列集合或多个集合(诸如主题集合和查询集合),分析中使用的所有序列都可累积在一起,或者关于它的任何亚组,称为输入序列。[0125]在一些实施方案中,主题集合和/或查询集合的每个基因组或质粒序列可属于同一物种。在一些实施方案中,主题集合和/或查询集合的每个基因组或质粒序列可为或包括代表同一属、科、目、纲、门、界或域的生物体的序列。在一些实施方案中,主题集合和/或查询集合的每个基因组或质粒序列可为或包括代表同一基因或它的一部分的序列。在一些实施方案中,主题集合和/或查询集合的每个基因组或质粒序列可为或包括代表同一编码序列或它的一部分的序列。[0126]在一些实施方案中,一个或多个或全部主题序列可与一个或多个查询序列相当,因为其代表同一物种。在一些实施方案中,一个或多个或全部主题序列可与一个或多个查询序列相当,因为其来自同一属、科、目、类、门、界或域的生物体。在一些实施方案中,一个或多个或全部主题序列可与一个或多个查询序列相当,因为其代表同一的基因或它的一部分。在一些实施方案中,一个或多个或全部主题序列可与一个或多个查询序列相当,因为其代表同一的编码序列或它的一部分。[0127]在一些实施方案中,一个或多个或全部主题序列可在可公开访问的数据库中获得和/或可从可公开访问的数据库中获得。在一些实施方案中,一个或多个或全部主题序列源自生物样品,并且在可公开访问的数据库中没有找到。在一些实施方案中,一个或多个或全部查询序列可在可公开访问的数据库中获得和/或可从可公开访问的数据库中获得。在一些实施方案中,一个或多个或全部查询序列源自生物样品,并且在可公开访问的数据库中没有找到。在一些实施方案中,一个或多个或全部主题序列可在可公开访问的数据库中获得和/或可从可公开访问的数据库中获得;并且一个或多个或全部查询序列源自生物样品,并且在可公开访问的数据库中没有找到。[0128]在一些实施方案中,比较最初输入的基因组或质粒序列。在某些实施方案中,比较最初输入的基因组或质粒序列的提取的编码序列。在某些实施方案中,比较最初输入的基因组或质粒序列的提取的编码序列的翻译。因此,在某些实施方案中,以成对方式将最初输入的查询基因组或质粒序列与最初输入的主题基因组或质粒序列进行比较。在某些实施方案中,以成对方式将最初输入的查询基因组或质粒序列的提取的编码序列与最初输入的主题基因组或质粒序列的提取的编码序列进行比较。在某些实施方案中,以成对方式将最初输入的查询基因组或质粒序列的提取的编码序列的翻译与最初输入的主题基因组或质粒序列的提取的编码序列的翻译进行比较。[0129]通过成对比较生成的数据的处理:用于序列的有效归类的多个序列归类因子的组合[0130]本公开包括使用从成对序列比较生成的数据来有效地对序列进行归类。在各种实施方案中,由成对序列比较得到的数据包括同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育,其中的任一个或全部可个别地或组合(例如以本文所示的组合)用作序列归类因子。因此,在各种实施方案中,可将序列归类到归类的序列组中,所述归类的序列组可基于一个或多个归类因子的一个或多个阈值。在各种实施方案中,归类因子可用于出于任何进一步分析的目的过滤出序列(或者以其他方式将序列排除在进一步考虑之外),例如,其中过滤是基于一个或多个归类因子的阈值和/或过滤出一个或多个归类的序列组,相反地,在各种实施方案中,归类因子可用于选择序列以包括在进一步的分析中,例如,其中选择是基于一个或多个归类因子的阈值和/或一个或多个归类的序列组的选择。在各种实施方案中,由成对序列比较得到的数据可任选地与所分析的序列的序列和/或可用注释(如果有的话)一起编译在例如got表中。[0131]如本文所公开的,成对序列比较可为核酸编码序列(例如,提取的编码序列)的比较或氨基酸序列(例如,提取的编码序列的翻译)的比较。因此,根据本公开的方法和系统归类的查询序列可包括核酸编码序列(例如,提取的编码序列)或氨基酸序列(例如,提取的编码序列的翻译)。[0132]在各种实施方案中,出于任何进一步分析的目的,可基于同一性百分比是否等于和/或低于阈值来对序列进行归类或过滤。在各种实施方案中,可基于同一性百分比是否等于和/或高于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值同一性百分比可等于或为至少约(例如)75%、80%、85%、90%、95%、96%、97%、98%、99%或100%。在各种实施方案中,阈值同一性百分比可在具有(例如)75%、80%、85%、90%或95%的下限和(例如)80%、85%、90%、95%、96%、97%、98%、99%或100%的上限的范围内。[0133]在各种实施方案中,出于任何进一步分析的目的,可基于覆盖率百分比是否等于和/或低于阈值来对序列进行归类或过滤。在各种实施方案中,可基于覆盖率百分比是否等于和/或高于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值覆盖率百分比可等于或为至少约(例如)75%、80%、85%、90%、95%、96%、97%、98%、99%或100%。在各种实施方案中,阈值覆盖率百分比可在具有(例如)75%、80%、85%、90%或95%的下限和(例如)80%、85%、90%、95%、96%、97%、98%、99%或100%的上限的范围内。[0134]在各种实施方案中,出于任何进一步分析的目的,可基于覆盖长度是否等于和/或低于阈值来对序列进行归类或过滤。在各种实施方案中,可基于覆盖长度是否等于和/或高于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值覆盖长度可等于或为至少约(例如)20个、25个、30个、35个、40个、45个、50个、75个、100个、125个、150个、175个或200个核苷酸或氨基酸。在各种实施方案中,阈值覆盖长度可在具有(例如)20个、25个、30个、35个、40个、45个、50个、75个、100个、125个、150个或175个核苷酸或氨基酸的下限和(例如)25个、30个、35个、40个、45个、50个、75个、100个、125个、150个、175个或200个核苷酸或氨基酸的上限的范围内。[0135]在各种实施方案中,出于任何进一步分析的目的,可基于预定覆盖长度上的同一性百分比是否等于和/或低于阈值来对序列进行分归或过滤。在各种实施方案中,可基于在预定覆盖长度上的同一性百分比是否等于和/或高于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,在预定覆盖长度上的示例性阈值同一性百分比可为(例如)在等于或为至少约20个、25个、30个、35个、40个、45个、50个、75个、100个、125个、150个、175个或200个核苷酸或氨基酸的预定覆盖长度上等于或为至少约75%、80%、85%、90%、95%、96%、97%、98%、99%或100%的同一性百分比。在各种实施方案中,在预定覆盖长度上的阈值同一性百分比可包括在具有(例如)75%、80%、85%、90%或95%的下限和(例如)80%、85%、90%、95%、96%、97%、98%、99%或100%的上限的范围内的同一性百分比,并且可包括在具有(例如)20个、25个、30个、35个、40个、45个、50个、75个、100个、125个、150个或175个核苷酸或氨基酸的下限和(例如)25个、30个、35个、40个、45个、50个、75个、100个、125个、150个、175个或200个核苷酸或氨基酸的上限的范围内的覆盖长度。[0136]在各种实施方案中,出于任何进一步分析的目的,可基于e值是否等于和/或高于阈值来对序列进行归类或过滤。在各种实施方案中,可基于e值是否等于和/或低于阈值来对序列进行归类,或者选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值e值可等于或为至少约(例如)1e-50、1e-40、1e-30、1e-20、1e-10、1e-9、1e-8、1e-7、1e-6、1e-5、1e-4、1e-3或1e-2。在各种实施方案中,示例性阈值e值可在具有(例如)1e-50、1e-40、1e-30、1e-20、1e-10、1e-9、1e-8、1e-7、1e-6、1e-5、1e-4或1e-3的下限和(例如)1e-40、1e-30、1e-20、1e-10、1e-9、1e-8、1e-7、1e-6、1e-5、1e-4、1e-3或1e-2的上限的范围内。[0137]在各种实施方案中,出于任何进一步分析的目的,可基于突变数量是否等于和/或高于阈值来对序列进行归类或过滤。在各种实施方案中,可基于突变数量是否等于和/或低于阈值来对序列进行归类,或选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值突变数量可等于或为至少约(例如)1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、35、40、45或50。在各种实施方案中,阈值突变数量可在具有(例如)1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、35、40或45的下限和(例如)2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、35、40、45或50的上限的范围内。[0138]在各种实施方案中,出于任何进一步分析的目的,可基于突变百分比是否等于和/或高于阈值来对序列进行归类或过滤。在各种实施方案中,可基于突变百分比是否等于和/或低于阈值来对序列进行归类,或选择序列以包括在进一步的分析中。在各种实施方案中,示例性阈值突变百分比可等于或为至少约(例如)0%、1%、2%、3%、4%、5%、10%、15%、20%或25%。在各种实施方案中,阈值突变百分比可在具有(例如)0%、1%、2%、3%、4%、5%、10%、15%或20%的下限和(例如)1%、2%、3%、4%、5%、10%、15%、20%或25%的上限的范围内。[0139]在各种实施方案中,为了任何进一步分析的目的,可基于系统发育对序列进行归类或过滤。在各种实施方案中,出于任何进一步分析的目的,过滤掉一个或多个分支。在各种实施方案中,选择一个或多个分支以包括在进一步的分析中。[0140]本公开包括基于来自成对序列比较的两个或多个归类因子的序列的归类。在各种实施方案中,序列的归类基于两个或多个归类因子,所述归类因子选自同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量和/或突变百分比。本公开进一步包括其中基于两个或多个归类因子的参数(例如,一个或多个阈值)来产生归类的序列组的实施方案。在一些实施方案中,对每个序列类别分配一数值。在各种实施方案中,分配给序列类别的数值可为利用测量查询序列和主题序列之间的相似性的一个或多个归类因子来跟踪的值,并且/或可称为“相似性评分”。相似性评分可包括跨任何范围的任何系列数值,但在特定实施方案中可包括0至1、0至10或0至100的范围。本文提供了相似性评分的实例。[0141]在各种实施方案中,本公开基于两个或个多归类因子对序列进行归类,所述两个或多个归类因子包括作为同一性的测量的第一归类因子和作为覆盖率的测量的第二归类因子。在各种实施方案中,同一性的测量可选自同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比。在各种实施方案中,覆盖率的测量可选自覆盖率百分比和覆盖长度。[0142]在各种实施方案中,可基于定义的评分系统对成对比较中分析的每个序列分配相似性评分,其中成对比较中分析的每个序列根据覆盖率百分比和序列变异的数量来归类或排序。例如,可根据下表2对序列进行归类并分配相似性评分,其中基于来自查询序列与特定主题序列的比较的数据,将在与特定主题序列的成对比较中分析的每个查询序列分配到其所属的具有最高相似性评分的库:[0143]表2[0144]覆盖率百分比突变数量分配的相似性评分≥99%=01≥99%《100.95≥99%≥100.8≥90%(任何)0.5≥75%(任何)0.4》0%(任何)0.3=0%(任何)0[0145]表2中的值应进一步理解为提供了所提供值周围的范围,例如,如同表2中的每个值前面都有术语“约”。一些或所有成对比较的序列的相似性评分可以矩阵、热图或图形(诸如条形图)显示。例如,包括单元列和单元行的矩阵或热图可包括用于每个主题序列的列和用于每个查询序列的行,其中每个单元基于查询序列和主题序列的比较来显示相似性评分。[0146]在一些实施方案中,出于任何进一步分析的目的,可过滤出不能满足一个或多个阈值标准或值(例如,阈值相似性评分)的成对序列比较(和/或它们的查询序列)(或以其他方式从进一步考虑中排除)。在一些实施方案中,出于任何进一步分析的目的,可过滤出与特定查询序列和特定主题序列(和/或相关的查询序列)的成对序列比较相关的数据(其中所述数据不能满足一个或一个以上阈值标准或值(例如,阈值相似性评分))(或以其他方式从进一步考虑中排除)。[0147]在一些实施方案中,出于任何进一步分析的目的,可过滤出落入如本文所示的一个或多个特定归类的序列组中的成对序列比较(和/或它们的查询序列或主题序列)(或以其他方式从进一步考虑中排除)。在一些实施方案中,出于任何进一步分析的目的,可过滤出与特定查询序列和特定主题序列(和/或相关的查询序列)的成对序列比较相关的数据(其中所述数据和/或序列落入一个或多个特定归类的序列组)(或以其他方式从进一步考虑中排除)。[0148]表2提供了允许通过相似性评分过滤归类的序列组的示例性归类方案。如表2的示例性归类方案中所示,对导致至少约99%的覆盖率百分比的成对比较(其中突变数量为零)分配1的相似性评分;对导致至少约99%的覆盖率百分比的剩余成对比较(其中突变数量小于约10)分配0.95的相似性评分;对导致至少约99%的覆盖率百分比的剩余成对比较(其中突变数量为至少10)分配0.8的相似性评分;对导致至少约90%但小于约99%的覆盖率百分比的剩余成对比较(包括任何突变数量)分配0.5的相似性评分;对导致至少约75%但小于约90%的覆盖率百分比的剩余成对比较(包括任何突变数量)分配0.4的相似性评分;对导致至少约0%但小于约75%的覆盖率百分比的剩余成对比较(包括任何突变数量)分配0.3的相似性评分;对导致等于0%的覆盖率百分比的剩余成对比较(包括任何突变数量)分配0的相似性评分。[0149]在某些实施方案中,出于任何进一步分析的目的,例如通过过滤以排除具有小于1、小于0.95、小于0.8、小于0.5、小于0.4、小于0.3或0的分配的相似性评分的序列比较,可过滤出如表2中所示归类的(或如由覆盖率和同一性的另一组合量度归类的)一个或多个序列比较中的任一个(或以其他方式从进一步考虑中排除)。在某些实施方案中,在将一个或多个阈值分配给对应于如表2中所述的相似性评分(或作为覆盖率的量度和同一性的量度的组合的其他相似性评分)的类别之前或之后(或之前和之后两者),将所述一个或多个阈值应用于成对比较。在某些实施方案中,一个或多个阈值可包括(例如)最小覆盖长度、最小覆盖率百分比、最大e值、最小同一性百分比、覆盖长度上的最小同一性百分比、最大突变数量和/或最大突变百分比。在某些实施方案中,应用一个或多个阈值作为基于表2的过滤的替代。在某些实施方案中,一个或多个阈值可包括例如最小覆盖长度、最小覆盖率百分比、最大e值、最小同一性百分比、覆盖长度上的最小同一性百分比、最大突变数量和/或最大突变百分比。[0150]在一些实施方案中,除了基于表2的归类和/或过滤之外或作为基于表2的归类和/或过滤的替代,可包括展现在至少约51个核苷酸或氨基酸的覆盖长度上至少约80%同一性且e值为约0.001或低于约0.001的成对序列比较用于进一步分析,和/或自分析过滤出展现小于约80%同一性和/或约50个或更少核苷酸或氨基酸的比对匹配长度和/或大于约0.001的e值的成对序列比较。[0151]靶特征的确定和/或具有靶特征的序列的选择[0152]在各种实施方案中,本公开的方法和系统可用于确定一个或多个序列是否显示某些靶特征,和/或选择经确定具有一个或多个靶特征的序列。如本文进一步公开的,示例性靶特征可包括但不限于序列保守的靶水平、序列可变性的水平(例如,跨序列集合和/或与一个或多个主题序列相比)或系统发育分组。[0153]在各种实施方案中,归类和/或过滤步骤之后是用于分析靶特征的一个或多个其他步骤,任选地包括选择具有靶特征的序列。在其中比较和归类和/或过滤核酸序列(例如,提取的编码序列)的一些实施方案中,通过将核酸(例如,提取的编码序列)翻译成氨基酸序列并任选地进行氨基酸序列与一个或多个主题氨基酸序列的进一步成对比较来进行靶特征分析。在其中比较和归类和/或过滤核酸序列(例如,提取的编码序列)的一些实施方案中,通过分析来自成对核酸序列比较的数据进行靶特征分析。在比较、归类和/或过滤氨基酸序列的一些实施方案中,通过分析来自成对氨基酸序列比较的数据来进行靶特征分析。[0154]可相对于基因组、质粒、基因、编码序列或翻译的编码序列氨基酸序列中的任一种或多种来评价(例如,测量或确定)保守性和/或可变性。可关于编码序列的核苷酸位置的亚组(例如编码氨基酸结构域的编码序列的核苷酸位置的亚组)来评价保守性和/或可变性。可关于编码序列内的一个或多个核苷酸位置来评价保守性和/或可变性。可相对于翻译的编码序列氨基酸序列的氨基酸位置的亚组(例如包括氨基酸结构域的氨基酸位置的亚组)来评价保守性和/或可变性。可相对于翻译的编码序列氨基酸序列内的一个或多个氨基酸位置来评价保守性和/或可变性。[0155]多种方法可用于分析序列保守性和/或可变性。如本文所公开的,序列保守性和/或可变性可指在所比较的序列中的一个或多个相应位置处的核苷酸或氨基酸的同一性或非同一性的频率的量度。至少在序列保守性和序列可变性两者都是序列之间或之中的相似性的量度的范围内,用于测量一者的方法通常适用于测量两者。[0156]在一些实施方案中,可根据突变百分比测量序列保守性和/或可变性。在一些实施方案中,序列保守性和/或可变性可根据同一性百分比来测量。在各种实施方案中,保守性和/或可变性可通过同一性的量度和覆盖率的量度的组合来确定。例如,在各种实施方案中,如果序列同时满足同一性的量度的阈值和覆盖率的量度的阈值,则将所述序列鉴定为保守的。在一些实施方案中,可根据突变百分比与覆盖长度和/或覆盖率百分比的组合来测量序列保守性和/或可变性。在一些实施方案中,可根据同一性百分比与覆盖长度和/或覆盖率百分比的组合来测量序列保守性和/或可变性。在一些实施方案中,可根据相似性评分(例如,如表2中所例示)来测量序列保守性和/或可变性。[0157]在一些实施方案中,对应于特定主题编码序列的序列的保守性可通过平均每个序列与特定主题编码序列相比的同一性百分比来确定。在各种实施方案中,基于至少85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、99.5%、99.6%、99.7%、99.8%、99.9%或100%的平均同一性百分比选择具有高保守性(低可变性)的序列。在一些实施方案中,基于小于99%、98%、97%、96%、95%、94%、93%、92%、91%、90%、85%、80%、75%、70%、65%、60%、55%、50%、40%或30%的平均同一性百分比选择具有低保守性(高可变性)的序列。[0158]在各种实施方案中,可基于序列的测量的保守性和/或可变性水平选择序列。在一些实施方案中,选择具有高保守性(低可变性)的序列,例如,在根据保守性的量度对成对比较的序列进行排序之后,选择约前1个、前2个、前3个、前4个、前5个、前10个、前20个、前25个、前50个、前100个、前1%、前2%、前5%、前10%、前15%、前20%、前25%或前50%的保守的成对比较序列(例如,顶部基因、编码序列或翻译的编码序列)。在一些实施方案中,选择具有低保守性(高可变性)的序列,例如,在根据保守性的量度对成对比较的序列进行排序之后,选择约后1个、后2个、后3个、后4个、后5个、后10个、后20个、后25个、后50个、后100个、后1%、后2%、后5%、后10%、后15%、后20%、后25%或后50%的保守的成对比较序列(例如,底部基因、编码序列、翻译的编码序列)。[0159]在各种实施方案中,通过系统发育分析展现序列保守性。用于系统发育分析的各种方法和程序包括:ancestree、aligroove、ape、armadillo工作流程平台、bali-phy、batwing、bayesphylogenies、bayestraits、beast、bionumerics、bosque、bucky、canopy、citup、clustalw、dendroscope、ezeditor、fastdnaml、fasttree2、fitmodel、geneious、hyphy、iqpnni、iq-tree、jmodeltest2、lisbeth、mega、mesquite、metapiga2、modelgenerator、molphy、morphobank、mrbayes、network、nona、paml、paraphylo、partitionfinder、pastis、paup*、phangorn、phybase、phyclust、phylip、phylot、phyloquart、phylowgs、phyml、phyx、poy、prottest3、pycogent、quicktree、raxml-hpc、raxml-ng、semphy、sowhat、splitstree、tnt、topali、treegen、treealign、treefinder、tree-puzzle、t-rex(webserver)、ugene、winclada和xrate。[0160]网络环境和计算装置[0161]如图37所示,示出并描述用于提供如本文所述的系统、方法和架构的网络环境3700的实现。简而言之,现在参考图37,示出并描述了示例性云计算环境3700的方块图。云计算环境3700可包括一个或多个资源提供者3702a、3702b、3702c(统称为3702)。每个资源提供者3702可包括计算资源。在一些实施方式中,计算资源可包括用于处理数据的任何硬件和/或软件。例如,计算资源可包括能够执行算法、计算机程序和/或计算机应用的硬件和/或软件。在一些实施方式中,示例性计算资源可包括具有存储和检索能力的应用服务器和/或数据库。每个资源提供者3702可连接到云计算环境3700中的任何其他资源提供者3702。在一些实施方式中,资源提供者3702可通过计算机网络3708连接。每个资源提供者3702可通过计算机网络3708连接到一个或多个计算装置3704a、3704b、3704c(统称为3704)。[0162]云计算环境3700可包括资源管理器3706。资源管理器3706可通过计算机网络3708连接到资源提供者3702和计算装置3704。在一些实施方式中,资源管理器3706可促进由一个或多个资源提供者3702向一个或多个计算装置3704提供计算资源。资源管理器3706可从特定计算装置3704接收对计算资源的请求。资源管理器3706可鉴定能够提供由计算装置3704请求的计算资源的一个或多个资源提供者3702。资源管理器3706可选择资源提供者3702来提供计算资源。资源管理器3706可促进资源提供者3702和特定计算装置3704之间的连接。在一些实施方式中,资源管理器3706可在特定资源提供者3702和特定计算装置3704之间确立连接。在一些实施方式中,资源管理器3706可将特定计算装置3704重新定向到具有所请求的计算资源的特定资源提供者3702。[0163]图38示出了可用于实现本公开中描述的技术的计算装置3800和移动计算装置3850的实例。计算装置3800旨在代表各种形式的数字计算机,诸如膝上型计算机、台式计算机、工作站、个人数字助理、服务器、刀片服务器、大型机和其他适当的计算机。移动计算装置3850旨在代表各种形式的移动装置,诸如个人数字助理、蜂窝电话、智能电话和其他相似的计算装置。本文所示的组件、它们的连接和关系以及它们的功能意味着仅是实例,而非意味着具有限制性。[0164]计算装置3800包括处理器3802、存储器3804、存储装置3806、连接到存储器3804和多个高速扩展端口3810的高速接口3808,以及连接到低速扩展端口3814和存储装置3806的低速接口3812。处理器3802、存储器3804、存储装置3806、高速接口3808、高速扩展端口3810和低速接口3812中的每一个都使用各种总线互连,并且可安装在公共母板上或以其他适当的方式安装。处理器3802可处理用于在计算装置3800内执行的指令,包括存储在存储器3804中或存储装置3806上的指令,以在外部输入/输出装置(诸如耦合到高速接口3808的显示器3816)上为gui显示图形信息。在其他实施方式中,可适当地使用多个处理器和/或多条总线以及多个存储器和多个类型的存储器。此外,可连接多个计算装置,每个装置提供必要操作的部分(例如,作为服务器阵列、一组刀片服务器或多处理器系统)。因此,在将多个功能描述为由处理器执行的情况下,这涵盖其中多个功能由任何数量的计算装置(一个或多个)中的任何数量的处理器(一个或多个)执行的实施方案。此外,在将功能描述为由处理器执行的情况下,这涵盖其中功能由任何数量的计算装置(一个或多个)(例如,在分布的计算系统中)中的任何数量的处理器(一个或多个)执行的实施方案。[0165]存储器3804存储计算装置3800内的信息。在一些实施方式中,存储器3804是一个或多个易失性存储器单元。在一些实施方式中,存储器3804是一个或多个非易失性存储器单元。存储器3804也可为另一种形式的计算机可读媒体,诸如磁盘或光盘。[0166]存储装置3806能够为计算装置3800提供大容量存储。在一些实施方式中,存储装置3806可为或含有计算机可读媒体,诸如软盘装置、硬盘装置、光盘装置或磁带装置、快闪存储器或其他类似的固态存储装置或装置阵列,包括存储区域网络或其他配置中的装置。指令可存储在信息载体中。当由一个或多个处理装置(例如,处理器3802)执行时,所述指令执行一种或多种方法,诸如上述那些方法。指令也可由一个或多个存储装置(诸如计算机可读媒体或机器可读媒体(例如,存储器3804、存储装置3806或处理器3802上的存储器))存储。[0167]高速接口3808管理计算装置3800的带宽密集型操作,而低速接口3812管理较低带宽密集型操作。这种功能分配仅是一实例。在一些实施方式中,高速接口3808耦合到存储器3804、显示器3816(例如,通过图形处理器或加速器)以及可接受各种扩展卡(未示出)的高速扩展端口3810。在所述实施方式中,低速接口3812耦合到存储装置3806和低速扩展端口3814。可包括各种通信端口(例如,usb、以太网、无线以太网)的低速扩展端口3814可例如通过网络适配器耦合到一个或多个输入/输出装置,诸如键盘、定点装置、扫描仪或联网装置(诸如交换机或路由器)。[0168]如图所示,计算装置3800可以多种不同的形式实现。例如,其可实现为标准服务器3820,或者在一组这样的服务器中多次实现。此外,其可在诸如膝上型计算机3822的个人计算机中实现。其也可实现为机架服务器系统3824的一部分。或者,来自计算装置3800的组件可与移动装置(未示出)(诸如移动计算装置3850)中的其他组件相结合。所述装置中的每一个可含有计算装置3800和移动计算装置3850中的一个或多个,并且整个系统可由多个彼此通信的计算装置构成。[0169]移动计算装置3850包括处理器3852、存储器3864、诸如显示器3854的输入/输出设备、通信接口3866和收发器3868以及其他组件。移动计算装置3850还可提供有存储装置,诸如微驱动器或其他装置,以提供额外的存储。处理器3852、存储器3864、显示器3854、通信接口3866和收发器3868中的每一个都使用各种总线互连,并且这些组件中的几个可安装在公共母板上或者以其他适当的方式安装。[0170]处理器3852可执行移动计算装置3850内的指令,包括存储在存储器3864中的指令。处理器3852可实现为包括单独的和多个模拟和数字处理器的芯片的芯片组。处理器3852可提供(例如)移动计算装置3850的其他组件的协调,诸如控制用户接口、由移动计算装置3850运行的应用以及由移动计算装置3850的无线通信。[0171]处理器3852可通过控制接口3858和耦合到显示器3854的显示接口3856与用户通信。显示器3854可为例如tft(薄膜晶体管液晶显示器)显示器或oled(有机发光二极管)显示器,或其他适当的显示技术。显示器接口3856可包括用于驱动显示器3854向用户呈现图形和其他信息的适当电路。控制接口3858可从用户接收命令,并转换它们以提交给处理器3852。此外,外部接口3862可提供与处理器3852的通信,以便实现移动计算装置3850与其他装置的近区通信。例如,外部接口3862可在一些实施方式中提供有线通信,或者在其他实施方式中提供无线通信,并且也可使用多个接口。[0172]存储器3864存储移动计算装置3850内的信息。存储器3864可实现为一个或多个计算机可读媒体、一个或多个易失性存储器单元或一个或多个非易失性存储器单元中的一者或多者。还可提供扩展存储器3874,并通过扩展接口3872连接到移动计算装置3850,所述扩展接口可包括例如simm(单列直插式存储模块)卡接口。扩展存储器3874可为移动计算装置3850提供额外的存储空间,或者也可为移动计算装置3850存储应用或其他信息。具体地,扩展存储器3874可包括执行或补充上述过程的指令,并且还可包括安全信息。因此,例如,扩展存储器3874可提供作为移动计算装置3850的安全模块,并且可用允许安全使用移动计算装置3850的指令来编程。此外,可通过simm卡提供安全应用以及额外信息,诸如以不可破解的方式在simm卡上放置鉴定信息。[0173]存储器可包括例如快闪存储器和/或nvram存储器(非易失性随机存取存储器),如下所讨论的。在一些实施方式中,指令存储在信息载体中。当由一个或多个处理装置(例如,处理器3852)执行时,所述指令执行一个或多个方法,诸如上述那些方法。指令也可由一个或多个存储装置(诸如一个或多个计算机可读媒体或机器可读媒体(例如,存储器3864、扩展存储器3874或处理器3852上的存储器))存储。在一些实施方式中,可在传播的信号中例如通过收发器3868或外部接口3862接收指令。[0174]移动计算装置3850可通过通信接口3866进行无线通信,必要时所述通信接口可包括数字信号处理电路。通信接口3866可提供各种模式或协议下的通信,诸如gsm语音呼叫(全球移动通信系统)、sms(短消息服务)、ems(增强消息服务)或mms消息服务(多媒体消息服务)、cdma(码分多址)、tdma(时分多址)、pdc(个人数字蜂窝)、wcdma(宽带码分多址)、cdma2000或gprs(通用分组无线服务)等。例如,这种通信可通过使用射频的收发器3868进行。此外,短距离通信可诸如使用wi-fitm或其他此类收发器(未示出)进行。此外,gps(全球定位系统)接收器模块3870可向移动计算装置3850提供额外导航相关的和位置相关的无线数据,这些数据可由移动计算装置3850上运行的应用适当地使用。[0175]移动计算装置3850还可使用音频编译码器3860进行听觉通信,所述音频编译码器可从用户接收口头信息并将其转换成可用的数字信息。音频编译码器3860同样可为用户生成听觉声音,诸如通过例如移动计算装置3850的电话听筒中的扬声器。这种声音可包括来自语音电话呼叫的声音,可包括记录的声音(例如,语音消息、音乐文件等)并且还可包括由在移动计算装置3850上操作的应用生成的声音。[0176]如图所示,移动计算装置3850可以多种不同的形式实现。例如,其可实现为蜂窝电话3880。其也可实现为智能电话3882、个人数字助理或其他类似移动装置的一部分。[0177]图20中提供了包括示例性系统的某些组件的另一非限制性示意图。[0178]本文描述的系统和技术的各种实现可在数字电子电路、集成电路、专门设计的asic(应用专用集成电路)、计算机硬件、固件、软件和/或它们的组合中实现。这些各种实现可包括在一个或多个计算机程序中的实现,所述一个或多个计算机程序在可编程系统上是可执行的和/或可解释的,所述可编程系统包括至少一个可编程处理器、至少一个输入装置和至少一个输出装置,所述可编程处理器可为专用的或通用的,经耦合以从存储系统接收数据和指令,以及将数据和指令发送到存储系统。[0179]所述计算机程序(也称为程序、软件、软件应用或代码)包括用于可编程处理器的机器指令,并且可以高级程序语言和/或面向对象的编程语言和/或以汇编/机器语言来实现。机器可读媒体和计算机可读媒体可指用于向可编程处理器提供机器指令和/或数据的计算机程序产品、设备和/或装置(例如,磁盘、光盘、存储器、可编程逻辑装置(pld)),包括接收机器指令作为机器可读信号的机器可读媒体。机器可读信号可指用于向可编程处理器提供机器指令和/或数据的信号。[0180]在某些实施方案中,计算机程序包括一个或多个机器学习模块。机器学习模块可指实现一个或多个特定机器学习算法的计算机实现的过程(例如,功能)。机器学习模块可包括例如一个或多个人工神经网络。在某些实施方案中,两个或多个机器学习模块可组合并实现为单个模块和/或单个软件应用。在某些实施方案中,两个或多个机器学习模块也可单独实现,例如,作为单独软件应用。机器学习模块可为软件和/或硬件。例如,机器学习模块可完全实现为软件,或者机器学习模块的某些功能可经由专用硬件(例如,经由应用专用集成电路(asic))来执行。[0181]为了提供与用户的交互,本文描述的系统和技术可在计算机上实现,所述计算机具有用于向用户显示信息的显示装置(例如,crt(阴极射线管)或lcd(液晶显示器)监视器)以及用户可通过其向计算机提供输入的键盘和定点装置(例如,鼠标或轨迹球)。也可使用其他种类的装置来提供与用户的交互;例如,提供给用户的反馈可为任何形式的感觉反馈(例如,视觉反馈、听觉反馈或触觉反馈);并且可以任何形式接收来自用户的输入,包括听觉、语音或触觉输入。[0182]本文描述的系统和技术可在计算系统中实现,所述计算系统包括后端组件(例如,作为数据服务器),或者包括中间件组件(例如,应用服务器),或者包括前端组件(例如,具有用户可通过其与本文描述的系统和技术的实现交互的图形用户接口或网络浏览器的客户端计算机),或者这种后端组件、中间件组件或前端组件的任何组合。系统的组件可通过任何形式或媒体的数字数据通信(例如,通信网络)互连。通信网络的实例包括局域网(lan)、广域网(wan)和互联网。[0183]计算系统可包括客户端和服务器。客户端和服务器通常彼此远离,并且通常通过通信网络进行交互。客户端和服务器的关系是通过在相应计算机上运行并且彼此之间具有客户端-服务器关系的计算机程序产生。[0184]各种实施方案的方块流程图[0185]图39是用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的示例性方法的方块流程图3900。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0186]在步骤3910中,获得(访问)病原体的不同株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0187]在步骤3920中,从基因组序列中鉴定编码序列。在步骤3930中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0188]在步骤3940中,将编码序列转换成氨基酸序列,并且在步骤3950中,将氨基酸序列进行比对。在某些实施方案中,氨基酸序列凭借经比对的编码序列进行比对。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0189]在步骤3960中,根据在步骤3910中访问的多个基因组序列所代表的病原体的不同株系之间的序列部分的保守水平来对氨基酸序列的比对部分进行分类。特别感兴趣的是高度保守并且因此对于由在步骤3910中访问的多个基因组序列所代表的病原体的各种株系是常见的那些序列部分。[0190]在步骤3970中,检查被鉴定为高度保守的每个氨基酸序列部分,以确定其是否与人蛋白序列同一。由于毒性问题,任何与人蛋白序列同一的高度保守序列均被作为候选抗原消除。其他标准也可应用于在针对病原体的疗法的开发中鉴定一种或多种最终候选抗原,例如肽信号的存在、蛋白质注释(或其存在/不存在)、特定结构域结构和/或序列中跨膜结构域的存在,后者可指示候选抗原是否对应于在病原体的膜和/或细胞壁内分泌或暴露的蛋白质,从而增强其作为针对病原体的治疗剂的潜在价值。所述方法可另外包括将涵盖候选抗原的多肽施用于动物的步骤。此外,当疗法是疫苗的情况下,所述方法可包括非临床评价候选抗原的免疫原性的步骤。[0191]图40是用于鉴定代表病原体的编码序列的一个或多个保守部分的示例性方法的方块流程图4000。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0192]在步骤4010中,从数据结构获得(访问)病原体的不同株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0193]在步骤4020中,从基因组序列中鉴定编码序列。在步骤4030中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0194]在步骤4040中,将编码序列转换成氨基酸序列。在某些实施方案中,编码序列在根据同一性百分比和覆盖率百分比归类后转换成氨基酸序列。在其他实施方案中,编码序列在根据同一性百分比和覆盖率百分比归类之前转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0195]在步骤4050中,根据在步骤4010中访问的多个基因组序列所代表的病原体的不同株系之间的序列部分的保守水平来对氨基酸序列的部分进行分类。特别感兴趣的是高度保守并且因此对于由在步骤4010中访问的多个基因组序列所代表的病原体的各种株系是常见的那些序列部分。[0196]图41是用于鉴定分离的病原体是否代表循环株系的示例性方法的方块流程图4100。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0197]在步骤4110中,获得(访问)病原体的循环株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0198]在步骤4120中,鉴定循环株系的序列的一个或多个保守(例如高度保守)部分。在某些实施方案中,循环株系的序列根据同一性百分比和覆盖率百分比进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个(其中“查询”序列和“主题”序列两者都是病原体的循环株系的序列),计算查询编码序列和每个主题序列之间的相似性的量度,其中相似性的量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0199]在步骤4130中,获得(访问)分离的病原体的多个完整或部分基因组序列。例如,分离的病原体的序列可来自从头测序读段(例如,从患有感染的患者获得的生物样品的高通量测序读段)。在某些实施方案中,可如上分析这些序列以鉴定哪些部分是保守的并且适当地代表分离的病原体。[0200]在步骤4140中,将分离的病原体的一个或多个序列(或它们的部分)与步骤4120中鉴定的循环株系的序列的一个或多个保守(例如高度保守)部分进行比较,从而鉴定分离的病原体是否代表循环株系(例如,对于循环株系的发病率是常见的)。[0201]图42是根据说明性实施方案的用于将氨基酸序列鉴定为候选抗生素抗性标记物(例如,在针对病原性细菌的疗法的开发中)的示例性方法的方块流程图。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0202]在步骤4210中,从数据结构获得(访问)病原性细菌的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0203]在步骤4220中,从质粒序列中鉴定编码序列。在步骤4230中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0204]在步骤4240中,将编码序列转换成氨基酸序列,并且在步骤4250中,将氨基酸序列进行比对。在某些实施方案中,氨基酸序列凭借经比对的编码序列进行比对。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0205]在步骤4260中,根据在步骤4210中访问的多个质粒序列中的序列部分的保守水平来对氨基酸序列的比对部分进行分类。特别感兴趣的是高度保守并且因此对于由步骤4210中访问的多个基因组序列所代表的病原体的质粒是常见的那些序列部分。[0206]在步骤4270中,选择鉴定为保守(例如,高度保守)的一个或多个序列部分作为候选抗生素抗性标记物。其他标准也可应用于鉴定候选抗生素抗性标记物,例如肽信号的存在、蛋白质注释(或它的存在/不存在)、特定结构域结构和/或序列中跨膜结构域的存在。所述方法可另外包括向动物施用涵盖候选抗生素抗性标记物的多肽的步骤。此外,当疗法是疫苗的情况下,所述方法可包括非临床评价肽的免疫原性的步骤。[0207]图43是根据说明性实施方案的用于鉴定代表质粒的编码序列的一个或多个保守部分的示例性方法的方块流程图4300。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0208]在步骤4310中,从数据结构获得(访问)病原性细菌的多个完整或部分质粒序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0209]在步骤4320中,从质粒序列中鉴定编码序列。在步骤4330中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0210]在步骤4340中,将编码序列转换成氨基酸序列。在某些实施方案中,编码序列在根据同一性百分比和覆盖率百分比归类后转换成氨基酸序列。在其他实施方案中,编码序列在根据同一性百分比和覆盖率百分比归类之前转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0211]在步骤4350中,根据在步骤4310中访问的多个质粒序列中的序列部分的保守水平来对氨基酸序列的部分进行分类。特别感兴趣的是高度保守并且因此对于由步骤4310中访问的多个基因组序列所代表的病原体的质粒是常见的那些序列部分。[0212]图44是用于鉴定代表病原体的肽的质荷比、例如用于鉴定这种代表病原体的肽的质谱法靶标的示例性方法的方块流程图。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0213]在步骤4410中,获得(访问)病原体的不同株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0214]在步骤4420中,从基因组序列中鉴定编码序列,并且在步骤4430中,将编码序列转换成氨基酸序列。在步骤4440中,鉴定氨基酸序列的一个或多个保守部分。例如,序列可根据同一性百分比和覆盖率百分比进行归类。例如,对于与一组主题序列进行比较的一组查询序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0215]在步骤4450中,确定鉴定为保守的一个或多个序列部分的质荷比。例如,这有助于鉴定相应代表病原体的肽的质谱法靶标,从而可通过质谱法对其进行鉴定。[0216]图45是用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的示例性方法的方块流程图。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0217]在步骤4510中,获得(访问)病原体的不同株系的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0218]在步骤4520中,从基因组序列中鉴定编码序列。在步骤4530中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0219]在步骤4540中,将编码序列转换成氨基酸序列。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0220]在步骤4550中,根据在步骤4510中访问的多个基因组序列所代表的病原体的不同株系之间的序列部分的保守水平来对氨基酸序列的部分进行分类。特别感兴趣的是高度保守并且因此对于由在步骤4510中访问的多个基因组序列所代表的病原体的各种株系是共同的那些序列部分。[0221]在步骤4560中,检查被鉴定为高度保守的每个氨基酸序列部分,以确定其是否与人蛋白序列同一。由于毒性问题,任何与人蛋白序列同一的高度保守序列均被作为候选抗原消除。其他标准也可应用于在针对病原体的疗法的开发中鉴定一种或多种最终候选抗原,例如肽信号的存在、蛋白质注释(或其存在/不存在)、特定结构域结构和/或序列中跨膜结构域的存在,后者可指示候选抗原是否对应于在病原体的膜和/或细胞壁内分泌或暴露的蛋白质,从而增强其作为针对病原体的治疗剂的潜在价值。所述方法可另外包括将涵盖候选抗原的多肽施用于动物的步骤。此外,当疗法是疫苗的情况下,所述方法可包括非临床评价候选抗原的免疫原性的步骤。[0222]图46是根据说明性实施方案的用于将氨基酸序列鉴定为候选抗生素抗性标记物的示例性方法4600的方块流程图。一些或所有步骤可全部或部分地由计算装置的处理器来进行(例如,执行软件指令)。[0223]在步骤4610中,从数据结构获得(访问)病原性细菌的多个完整或部分基因组序列。序列可来自公共或私人序列数据库,和/或来自从头测序读段。所述多个序列可包括经合并以产生至少一些完整或部分基因组序列的重叠群。[0224]在步骤4620中,从质粒序列中鉴定编码序列。在步骤4630中,根据同一性百分比和覆盖率百分比对编码序列进行归类。例如,对于与一组主题序列进行比较的一组查询编码序列中的每一个,计算查询编码序列和每个主题序列之间的相似性量度,其中相似性量度中的每一个是(i)查询序列和主题序列之间的同一性百分比和(ii)查询序列和主题序列之间的覆盖率百分比的函数。在某些实施方案中,应用涉及(i)和(ii)两者的阈值。在一些情况下,将绝对(相对于相对)突变数量视为等同于“同一性百分比”。查询序列组可与主题序列组同一,或者其可为不同的组或部分重叠的组。相似性的量度的矩阵可以图形方式呈现。例如,相似性测量的热图可以图形方式显示,例如,其中x轴和y轴代表序列,并且给定x-y位置中的强度或颜色代表相应的两个序列之间的相似性测量。[0225]在步骤4640中,将编码序列转换成氨基酸序列。在某些实施方案中,在计算相似性的量度之后将编码序列转换成氨基酸序列,并且在其他实施方案中,在计算相似性的量度之前将编码序列转换成氨基酸序列(例如,其中计算一组查询氨基酸序列中的每一个针对一组主题氨基酸序列的相似性的量度)。[0226]在步骤4650中,根据在步骤4610中访问的多个质粒序列中的序列部分的保守水平来对氨基酸序列的部分进行分类。特别感兴趣的是高度保守并且因此对于由步骤4610中访问的多个基因组序列所代表的病原体的质粒是常见的那些序列部分。[0227]在步骤4660中,选择鉴定为保守(例如,高度保守)的一个或多个序列部分作为候选抗生素抗性标记物。其他标准也可应用于鉴定候选抗生素抗性标记物,例如肽信号的存在、蛋白质注释(或它的存在/不存在)、特定结构域结构和/或序列中跨膜结构域的存在。所述方法可另外包括向动物施用涵盖候选抗生素抗性标记物的多肽的步骤。此外,当疗法是疫苗的情况下,所述方法可包括非临床评价肽的免疫原性的步骤。[0228]本文描述的不同实施方式的要素可经组合以形成上文没有具体阐述的其他实施方式。要素可排除在本文描述的方法、过程、计算机程序、数据库等之外,而不会不利地影响它们的操作。各种单独的要素可组合成一个或多个单独的要素,以执行本文描述的功能。[0229]考虑要求保护的本发明的系统、架构、装置、方法和过程涵盖使用来自本文描述的实施方案的信息开发的变异和适应。如本说明书所考虑,可执行本文描述的系统、架构、装置、方法和过程的适应和/或修改。[0230]在整个说明书中,当物品、装置、系统和架构描述为具有、包括或包含特定组件的情况下,或者当过程和方法描述为具有、包括或包含特定步骤时,考虑另外存在基本上由所述组件组成或由所述组件组成的本发明的物品、装置、系统和架构,并且存在基本上由所述处理步骤组成或由所述处理步骤组成的根据本发明的过程和方法。[0231]应理解,只要本发明仍然可操作,步骤的顺序或执行某些动作的顺序是无关紧要的。此外,两个或多个步骤或动作可同时进行。[0232]本文提及任何出版物,例如在背景部分,并非承认该出版物作为关于本文提出的任何权利要求的现有技术。背景部分是为了清楚的目的而呈现的,并不意味着对关于任何权利要求的现有技术的描述。[0233]提供标题是为了方便读者–标题的存在和/或放置不打算限制本文描述的主题物的范围。[0234]应用[0235]本公开的表征输入序列之间、之中和/或之内的残基亚组的序列保守性的方法和系统可用于各种分析和治疗应用中。本文提供表征序列保守性的方法和系统的各种用途。例如,本文公开的方法和系统可例如基于序列保守性特征用于鉴定未表征的序列的治疗相关性。提供了本文公开的方法和系统的效用的非限制性实例。[0236]用于选择抗抗原抗体的抗原的鉴定[0237]在特定物种(诸如病原体物种)的实例中,基因组和质粒核酸序列(包括编码序列)可变。在许多情况下,源自特定物种成员的核酸序列的可变性可通过分析可公开获得的基因组序列和/或其他基因组序列(诸如非公开测序数据)来揭示。随着单独寄存在可公开访问的数据库中的序列数量不断增加,成功分析不断增长的不同序列信息变得越来越具挑战性。本公开的方法和系统通过提供分析输入序列的保守性特征的系统性方法来解决这个困难。[0238]作为用于产生抗病原体治疗剂的抗原来源,病原体基因组的保守序列可优于病原体基因组的非保守序列。抗原的鉴定和/或表征可为或包括表位的鉴定和/或表征。抗原可为或包括表位,并且本文公开的用于鉴定抗原的一个或多个特征同样可用于鉴定表位。至少一个原因是,与相关病原体群体内相对保守的序列结合或以其他方式相互作用的治疗性抗体或其他药物分子将必然更可能在病原体物种的更宽范围的成员中具有治疗益处,并因此在遭受所述病原体物种困扰的患者中具有治疗益处。因此,将通过本公开的方法和系统鉴定的在相关病原体群体中保守的序列鉴定为用于开发治疗性抗体的候选抗原或鉴定为用于其他治疗模式的靶标,诸如小分子药物。用于开发针对治疗性抗原的抗体的某些方法是本领域已知的,并且可包括(仅提供一个实例)用感兴趣的抗原对产生抗体的生物体进行免疫。[0239]在各种实施方案中,通过次要考虑因素,鉴定为保守的序列可进一步缩小范围以鉴定治疗相关靶标。一个次要考虑因素是鉴定的候选治疗靶标是否与已知的人序列同一。可使用可公开获得的数据库和搜索工具来确定所鉴定的序列是否与已知的人序列同一。目前公开的方法和系统的各种实施方案包括从候选治疗靶标中(例如,从候选抗原列表中)去除与已知人序列同一的候选治疗靶标。去除与已知人序列同一的序列的至少一个原因是,靶向这种序列的药物(例如抗体)的开发可表现出与非靶向人细胞和/或蛋白质的临床上有害的或另外不希望的相互作用。[0240]次要考虑因素的其他实例包括蛋白质注释、功能和/或蛋白质结构域的存在或不存在。蛋白质结构域的实例包括信号序列、已知引起分泌或与分泌相关的结构域、细胞膜蛋白质的特征性结构域、指示序列在细胞膜或细胞壁处的细胞外暴露的特征或其他结构特征。序列的细胞外暴露促进治疗剂与序列的相互作用,并且因此是治疗靶标中可需要的特征。[0241]在某些实施方案中,上述信息(例如通过本文提供的方法鉴定候选抗原)用于开发一种或多种用于治疗病原体引起的疾病的组合物(或鉴定一种或多种新的和/或现有的组合物)。在某些实施方案中,鉴定和/或开发涉及多种药物组合物(例如,药物混合物)的疗法。例如,本文提供的方法可用于选择可用于供治疗病原体引起的疾病(诸如covid-19)的药物(例如,药物混合物)中的最佳一种或多种病原体中和抗体。在一些实施方案中,所述药物不是对疾病的治疗,而是例如用于疫情中的权宜之计,以增强人体(例如,免疫受损或以其他方式易受感染的个体)抵抗感染的能力,例如直到开发出疫苗。在一些实施方案中,所述药物干扰病原体(例如,诸如sars-cov2的病毒)的功能以防止或减少由所述病毒引起的对人体的损害,例如,由此减少患者使用呼吸机和/或其他呼吸装置的需要。在一些实施方案中,药物是为特定个体或个体组定制的治疗。在某些实施方案中,小鼠或其他动物可用于制造用于治疗病原体引起的疾病的组合物,其中在这种制造中使用通过本文提供的计算机实施的方法产生的信息。例如,可向小鼠或其他动物注射用于产生人抗体的病毒(或它的部分),所述人抗体可被制造并施用给一个或多个患者。在某些实施方案中,可从鉴定病毒或其他病原体的序列进行到产生抗体,所述抗体可使用本文所述的方法大规模生产。[0242]在某些实施方案中,本文提供的方法用于评价编码蛋白质的核酸的编码序列、编码蛋白质的核酸序列的保守序列、编码蛋白质的核酸的非保守序列(特征在于变异的序列)、特定蛋白质内的保守结构域和/或特定蛋白质内的非保守结构域(特征在于变异的部分),例如,其中所述蛋白质与病原体相关。这种评价然后用于开发用于治疗、预防或改善由病原体引起的疾病的抗体、进入抑制剂、疫苗和/或其他治疗剂。例如,在某些实施方案中,本文提供的方法用于评价与sars-cov2宿主细胞(诸如人或蝙蝠血管收缩肽-转化酶2(ace2)受体)上的受体结合以促进宿主细胞感染的sars-cov2刺突(s)蛋白或其受体结合结构域(rbd),或编码所述蛋白的核酸序列。因此,例如,本说明书包括使用本文提供的计算机实现的方法来分析sars-cov2刺突(s)蛋白或其rbd,以鉴定可用于开发抗体、进入抑制剂、疫苗和/或其他治疗剂以治疗、预防或改善由sars-cov2病毒(即covid-19)引起的疾病的序列。[0243]在某些实施方案中,本文提供的方法用于评价编码sars-cov2刺突蛋白或其受体结合结构域(rbd)的核酸的编码序列、编码sars-cov2刺突蛋白或其rbd蛋白的核酸序列的保守序列、编码sars-cov2刺突蛋白或其rbd蛋白的核酸的非保守结构域(特征在于变异的序列)、特定sars-cov2刺突蛋白或其rbd的保守结构域和/或sars-cov2刺突蛋白或其rbd的非保守结构域(特征在于变异的部分)。在某些实施方案中,本文提供的方法用于评价编码冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的核酸的编码序列、编码冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的核酸序列的保守序列、编码冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的核酸序列的非保守序列(特征在于变异的序列)、特定冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的保守结构域和/或冠状病毒刺突蛋白(例如,mers或sars-cov刺突蛋白)或其rbd的非保守结构域(特征在于变异的部分)。[0244]候选疫苗抗原的鉴定[0245]疫苗包括施用以刺激接受者产生针对病原体的抗体(疫苗抗原)的非病原性物质。疫苗抗原可为由病原体呈递的肽。疫苗功效要求,如果接受者稍后被感染,则接受者因应疫苗抗原产生的抗体能够结合病原体。由于病原体的株系可不同,所以当疫苗抗原具有保守序列或由保守序列编码时,疫苗提供针对最广泛范围的病原体株系的免疫性。如本文关于鉴定用于选择抗抗原抗体的抗原所公开,本公开的方法和系统可用于鉴定保守的病原体序列。因此,使用本公开的方法和系统鉴定的保守的病原体序列可用作疫苗抗原和/或候选疫苗抗原。候选疫苗抗原可在临床上适当的免疫和感染动物模型中验证,并在临床试验中例如针对安全性和功效进一步验证。[0246]代表性样品的鉴定[0247]尽管已知或可能在临床样品中存在多种病原体的许多株系,但出于实际和/或历史原因,研究往往聚焦于一种或少数株系。然而,在治疗剂的开发中,使用代表病原体的临床样品、优选许多或大多数临床样品的研究株系促进发现具有广泛临床功效的治疗剂。本公开提供可用于将一种或多种研究株系的序列与来自其他株系(例如,不同临床分离株)的不同序列集合进行比较以表征一种或多种研究株系的基因组与其他株系相比的保守性的方法和系统。研究株系的序列的保守性指示,分析的研究株系或研究株系序列代表所有或大量的比较株系。因此,根据本公开的方法和系统在分析中展现保守性的研究株系或研究株系序列适用于临床相关研究。相反,根据本公开的方法和系统在分析中未展现保守性的研究株系或研究株系序列对于临床相关研究可能不是最佳的。[0248]抗生素抗性标记物的鉴定[0249]病原性细菌的抗生素抗性成为临床上日益关注的问题。例如,抗性感染更有可能导致死亡。细菌通过两种主要途径获取对抗生素的抗性:染色体突变和通过水平基因转移获取可移动的遗传元件,诸如质粒。质粒是独立于染色体复制并能够通过接合在细菌之间水平转移的基因组外环状dna分子。因此,质粒在许多病原体中的抗生素抗性散播中起着重要作用。[0250]本文提供的方法和系统可应用于鉴定指示和/或引起病原性细菌抗体抗性的遗传和/或氨基酸序列(抗体抗性标记物)。本文提供的方法和系统可应用于质粒序列以鉴定保守序列。因此,质粒的保守序列鉴定为候选抗生素抗性标记物。此外,质粒的保守序列是开发破坏或中和质粒赋予的抗生素抗性的治疗剂的候选靶标。[0251]用于质谱法的肽发现资源的生成[0252]质谱法基于精确测量的质荷比鉴定分析的物质。肽的质荷比取决于肽序列。至少部分由于质荷比复杂,质谱法分析可通过将检测的质荷比与预计质荷比的集合进行比较来鉴定肽。因此,质谱法不能鉴定意外序列。由于特定物种的生物体(例如,临床相关的病原体分离株)的基因组和蛋白质组改变,因此无法鉴定意外肽可阻碍不同样品的分析。[0253]本公开的方法和系统可通过分析代表感兴趣的物种(例如,临床相关的病原体)的不同基因组的保守特征,为质谱法提供肽发现资源。例如,根据本公开的方法和系统的分析可鉴定序列多样性的区域,所述区域可用于修订用于查询质谱法数据的预计质荷比的集合。因此,掺入通过本公开的方法和系统鉴定的不同序列可增强质谱法发现样品中的肽的能力,例如发现临床相关的病原体肽。[0254]为了提供一个特定实例,主要组织相容性复合体i相关蛋白具有临床相关性,并且可通过质谱法发现,基于预计质荷比的适当集合来分析所提供的数据。主要组织相容性复合体(人的mhc或hla)在所有有核细胞的细胞表面上表达,并作为获得性免疫系统中t细胞抗原呈递的机制起作用。其用于在细胞表面上显示加工的自身和外源蛋白(抗原)的肽片段,以供由t淋巴细胞(对于mhci类为cd8 细胞毒性t淋巴细胞(ctl),并且对于mhcii类为cd4 辅助t淋巴细胞)检查。表征该过程中涉及的抗原有助于鉴定治疗上有用的靶标,例如作为用于开发治疗性抗体的抗原。质谱法是一种可用于鉴定mhc呈递的抗原的技术。然而,如果质谱法分析不是为了检测存在的抗原而设计的,则不能检测mhc呈递的抗原。本文公开的方法和系统可用于生成预计质荷比的包含性集合,以查询靶病原体的mhc呈递的抗原的质谱法数据。[0255]基因组、基因和蛋白质(例如抗原)中多样性区域的鉴定[0256]如本文所公开的,所提供的方法和系统可用于鉴定基因组、基因和蛋白质内的多样性区域。多样性区域(与其他区域相比保守性较低的区域)可指示可适合于更实质性的实验室操作(例如,实验室引入的序列修饰)的核苷酸或氨基酸位置。在某些生物学背景下,序列多样性的特征对生物学功能至关重要,例如在免疫球蛋白的可变区情况即如此。多样性还可指示可用于系统发育分析的区域,这是因为与分析相对更保守的序列相比,多样性区域可在相同或更短的时间内为系统发育分析提供更大数量的序列变异。多样性也可指示比保守序列更近期经历进化发展的序列。[0257]引起流行病学的病原体的系统发育的生成[0258]文公开的方法和系统可用于生成系统发育。系统发育对于分析来自病原体(例如,快速进化的病原体)的序列特别有用。系统发育可用于描述诸如人免疫缺失病毒(hiv)等病原体的分子流行病学和传播、严重急性呼吸综合征(sars)相关冠状病毒(例如,严重急性呼吸综合征相关冠状病毒(sars-cov);严重急性呼吸综合征冠状病毒2(sars-cov2),其是导致冠状病毒疾病(covid19)的病毒;中东呼吸综合征相关冠状病毒(mers-cov))的起源和随后的进化、禽流行性感冒以及季节性和疫情人流感的进化流行病学。可使用系统发育确定的信息的实例包括对新病原体株系的起源或其在新物种中出现的实际时间、病原体重组和重配事件、病原体流行中种群大小变化的速率以及病原体如何在特定种群和地理区域内传播和进化的估计(具有置信界限)。[0259]基因组研究确认,突变和可移动遗传元件的获取可显著影响微生物克隆的病理学。事实上,即使是不大的基因改变也可对宿主-病原体相互作用以及病原体的抗体鉴定产生巨大影响。宿主内进化不仅对患者有意义,而且对在医院出于流行病学目的确立区分株系中的关联性的阈值也有意义。微生物遗传多样性、免疫调节和由个别株系的损害可显著变化。因此,捕获克隆的宽度以解释基因组水平上宿主-病原体相互作用多样性的程序,将可能产生对微生物病原体生物学的独特理解。这种理解促进了更有效和个性化方法的开发,以预防感染和改善病原体管理。[0260]从系统发育获得的序列源信息可帮助设计和实现公共卫生和治疗干预措施。例如,如应用于hbv,本公开的方法和系统可用于确定特定株系(例如,实验室株系)属于哪个hbv谱系,确定跨hbv谱系的一个或多个hbv基因或蛋白质(例如,hbsag)的遗传多样性,确定自然界中存在的hbv或hbv基因或蛋白质(例如,hbsag)的遗传变体的数量和宽度,和/或确定hbv基因组或它的遗传或编码蛋白质序列(例如,hbsag的遗传或编码蛋白质序列)的哪个部分是遗传保守的。在另一个实例中,本文公开的方法和系统可用于确定特定患者感染哪种株系和/或这种株系的定义遗传特征和/或特定患者感染的株系的抗生素抗性特征。在另一个实例中,本文公开的方法和系统可用于确定病原体基因组(例如埃博拉病毒基因组)的遗传多样性,并确定所测量的变异是否具有临床分支。[0261]直系同源基因的鉴定[0262]直系同源物是由共同的祖先dna序列衍生而来的不同物种的同源序列。物种间的比较遗传学至少部分基于如下事实:认为直系同源物在功能上是物种间相关的。尽管详细的分析通常可确立直系同源物鉴定的准确度,但基因组信息的大量分析增加了直系同源物鉴定的误差率。因此,需要区分真实直系同源物和错误注释的直系同源物的改良的方法。如本文所公开的,本公开的方法和系统可用于表征序列保守性。因此,本公开的方法和系统可用于提高直系同源物鉴定的准确度,并且/或用于鉴定和校正现有的直系同源物错误注释。根据本文所公开的方法和系统的直系同源物的鉴定可用于通过将新序列或未表征的序列与先前注释的序列进行比对并将先前的注释应用于直向同源的新序列或未表征的序列来注释新序列或未表征的序列。[0263]用于选择抗体疗法、鉴定推定的逃逸突变体和个体化药物的表位序列变异的评价[0264]在各种实施方案中,评价特定基因或蛋白质或它的一部分的变异是有用的。例如,在抗体疗法的背景下,许多重要的问题可通过评价抗体的抗原和/或表位的变异来解决。[0265]本说明书的各种实施方案包括疗法和/或治疗剂。在各种实施方案中,疗法和/或治疗剂可为或包括小干扰rna(sirna)或短发夹rna(shrna)。在各种实施方案中,疗法和/或治疗剂可为或包括抗体。在各种实施方案中,疗法和/或治疗剂可为或包括治疗covid-19的疗法和/或治疗剂。治疗covid-19的示例性疗法和/或治疗剂可包括瑞德西韦、克力芝、伊维菌素、特敏福、阿维根、秋水仙碱、地塞米松、氯喹、羟基氯喹、阿奇霉素、il-6抑制剂(例如,托珠单抗和西鲁库单抗)、激酶抑制剂(例如,阿卡拉替尼、依鲁替尼、泽布替尼、巴瑞替尼、鲁索替尼和托法替尼)、干扰素、恢复期血浆、结合sars-cov-2刺突蛋白的抗体(抗sars-cov-2-刺突蛋白抗体)、mab10933(regeneron)、mab10934(regeneron)、mab10987(regeneron)、mab10989(regeneron)、regn-cov2(regeneron)、ly-cov555(elililly)、ly-cov016(elililly)和/或bnt162b2(pfizer)。示例性抗体可包括结合sars-cov-2的刺突蛋白用于covid-19疗法的抗体,例如,如美国专利第10,787,501号中所公开,所述专利全文通过引用、特别是关于covid-19治疗性抗体及它们的表位和其他性质并入本文。美国专利第10,787,501号的表1提供了示例性抗sars-cov-2-刺突蛋白(sars-cov-2-s)抗体和抗体序列,其全文通过引用具体并入本文。还参见下表3:[0266]表3[0267][0268][0269][0270][0271][0272][0273][0274][0275][0276][0277][0278][0279][0280][0281][0282][0283][0284][0285][0286][0287][0288]表1的抗体包括多特异性分子,例如抗体或抗原结合片段,其分别包括这些抗体的cdr-h和cdr-l、vh和vl或hc和lc(包括如在本文中所示的它们的变体)。[0289]在一实施方案中,可包括在多特异性分子中的特异性结合cov-s的抗原结合结构域包含:[0290](1)[0291](i)重链可变结构域序列,其包含表1中所示的cdr-h1、cdr-h2和cdr-h3氨基酸序列,和[0292](ii)轻链可变结构域序列,其包含表1中所示的cdr-l1、cdr-l2和cdr-l3氨基酸序列;[0293]或,[0294](2)[0295](i)重链可变结构域序列,其包含表1中所示的氨基酸序列,和[0296](ii)轻链可变结构域序列,其包含表1中所示的氨基酸序列;[0297]或,[0298](3)[0299](i)重链免疫球蛋白序列,其包含表1中所示的氨基酸序列,和[0300](ii)轻链免疫球蛋白序列,其包含表1中所示的氨基酸序列。[0301]在各种实施方案中,本公开提供特异性结合冠状病毒刺突蛋白(cov-s)的分离的重组抗体或它的抗原结合片段,其中所述抗体具有以下特征中的一个或多个:(a)以小于约10-9m的ec50结合cov-s;(b)展现与未进行施用的可比较的冠状病毒感染的动物相比,在向冠状病毒感染的动物施用后,所述冠状病毒感染的动物的存活率增加;和/或(c)包含包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(cdr-h1、cdr-h2和cdr-h3),所述重链可变区包含与表1的hcvr具有至少约90%序列同一性的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链cdr(cdr-l1、cdr-l2和cdr-l3),所述轻链可变区包含与表1的lcvr具有至少约90%序列同一性的氨基酸序列。[0302]在各种实施方案中,刺突蛋白与以下序列(seqidno:108)具有至少80%同一性(例如,至少80%、90%、95%、96%、97%、98%、99%或100%同一性):[0303]mfvflvllplvssqcvnlttrtqlppaytnsftrgvyypdkvfrssvlhstqdlflpffsnvtwfhaihvsgtngtkrfdnpvlpfndgvyfasteksniirgwifgttldsktqsllivnnatnvvikvcefqfcndpflgvyyhknnkswmesefrvyssannctfeyvsqpflmdlegkqgnfknlrefvfknidgyfkiyskhtpinlvrdlpqgfsaleplvdlpiginitrfqtllalhrsyltpgdsssgwtagaaayyvgylqprtfllkynengtitdavdcaldplsetkctlksftvekgiyqtsnfrvqptesivrfpnitnlcpfgevfnatrfasvyawnrkrisncvadysvlynsasfstfkcygvsptklndlcftnvyadsfvirgdevrqiapgqtgkiadynyklpddftgcviawnsnnldskvggnynylyrlfrksnlkpferdisteiyqagstpcngvegfncyfplqsygfqptngvgyqpyrvvvlsfellhapatvcgpkkstnlvknkcvnfnfngltgtgvltesnkkflpfqqfgrdiadttdavrdpqtleilditpcsfggvsvitpgtntsnqvavlyqdvnctevpvaihadqltptwrvystgsnvfqtragcligaehvnnsyecdipigagicasyqtqtnsprrarsvasqsiiaytmslgaensvaysnnsiaiptnftisvtteilpvsmtktsvdctmyicgdstecsnlllqygsfctqlnraltgiaveqdkntqevfaqvkqiyktppikdfggfnfsqilpdpskpskrsfiedllfnkvtladagfikqygdclgdiaardlicaqkfngltvlpplltdemiaqytsallagtitsgwtfgagaalqipfamqmayrfngigvtqnvlyenqklianqfnsaigkiqdslsstasalgklqdvvnqnaqalntlvkqlssnfgaissvlndilsrldkveaevqidrlitgrlqslqtyvtqqliraaeirasanlaatkmsecvlgqskrvdfcgkgyhlmsfpqsaphgvvflhvtyvpaqeknfttapaichdgkahfpregvfvsngthwfvtqrnfyepqiittdntfvsgncdvvigivnntvydplqpeldsfkeeldkyfknhtspdvdlgdisginasvvniqkeidrlnevaknlneslidlqelgkyeqyikwpwyiwlgfiagliaivmvtimlccmtsccsclkgccscgscckfdeddsepvlkgvklhyt[0304]在一些实施方案中,本公开提供结合sars-cov-2刺突蛋白的分离的抗体或它的抗原结合片段,所述sars-cov-2刺突蛋白包含seqidno:108中所示的氨基酸序列,其中所述分离的抗体或抗原结合片段包含包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(hcdr1、hcdr2和hcdr3),所述重链可变区包含seqidno:29中所示的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链互补决定区(cdr)(lcdr1、lcdr2和lcdr3),所述轻链可变区包含seqidno:33中所示的氨基酸序列。[0305]在一些实施方案中,hcdr1包含seqidno:30中所示的氨基酸序列,hcdr2包含seqidno:31中所示的氨基酸序列,hcdr3包含seqidno:32中所示的氨基酸序列,lcdr1包含seqidno:34中所示的氨基酸序列,lcdr2包含seqidno:35中所示的氨基酸序列,并且lcdr3包含seqidno:36中所示的氨基酸序列。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:29中所示的氨基酸序列的hcvr。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:33中所示的氨基酸序列的lcvr。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:29中所示的氨基酸序列的hcvr和包含seqidno:33中所示的氨基酸序列的lcvr。[0306]在一些实施方案中,本公开提供结合sars-cov-2刺突蛋白的分离的抗体,所述sars-cov-2刺突蛋白包含seqidno:108中所示的氨基酸序列,其中所述分离的抗体包含免疫球蛋白恒定区、包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(hcdr1、hcdr2和hcdr3),所述重链可变区包含seqidno:29中所示的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链互补决定区(cdr)(lcdr1、lcdr2和lcdr3),所述轻链可变区包含seqidno:33中所示的氨基酸序列。[0307]在一些实施方案中,hcdr1包含seqidno:30中所示的氨基酸序列,hcdr2包含seqidno:31中所示的氨基酸序列,hcdr3包含seqidno:32中所示的氨基酸序列,lcdr1包含seqidno:34中所示的氨基酸序列,lcdr2包含seqidno:35中所示的氨基酸序列,并且lcdr3包含seqidno:36中所示的氨基酸序列。在一些实施方案中,分离的抗体包含含有seqidno:29中所示的氨基酸序列的hcvr和包含seqidno:33中所示的氨基酸序列的lcvr。在一些实施方案中,分离的抗体包含含有seqidno:37中所示的氨基酸序列的重链和包含seqidno:38中所示的氨基酸序列的轻链。在一些情况下,免疫球蛋白恒定区是igg1恒定区。在一些情况下,分离的抗体是重组抗体。在一些情况下,分离的抗体是多特异性的。[0308]在一些实施方案中,本公开提供包含如上或本文所讨论的分离的抗体和药学上可接受的载体或稀释剂的药物组合物。[0309]在一些情况下,抗体或它的抗原结合片段包含包含在hcvr内的三个重链cdr(hcdr1、hcdr2和hcdr3),所述hcvr包含seqidno:69中所示的氨基酸序列;以及包含在lcvr内的三个轻链cdr(lcdr1、lcdr2和lcdr3),所述lcvr包含seqidno:73中所示的氨基酸序列。在一些情况下,抗体或它的抗原结合片段包含:hcdr1,包含seqidno:70中所示的氨基酸序列;hcdr2,包含seqidno:71中所示的氨基酸序列;hcdr3,包含seqidno:72中所示的氨基酸序列;lcdr1,包含seqidno:74中所示的氨基酸序列;lcdr2,包含seqidno:75中所示的氨基酸序列;以及lcdr3,包含seqidno:76中所示的氨基酸序列。在一些情况下,抗体或它的抗原结合片段包含含有seqidno:69中所示的氨基酸序列的hcvr和包含seqidno:73中所示的氨基酸序列的lcvr。在一些情况下,抗体或它的抗原结合片段包含含有seqidno:77中所示的氨基酸序列的重链和包含seqidno:78中所示的氨基酸序列的轻链。[0310]在一些实施方案中,本公开提供结合sars-cov-2刺突蛋白的分离的抗体或它的抗原结合片段,所述sars-cov-2刺突蛋白包含seqidno:108中所示的氨基酸序列,其中所述分离的抗体或抗原结合片段包含包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(hcdr1、hcdr2和hcdr3),所述重链可变区包含seqidno:69中所示的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链互补决定区(cdr)(lcdr1、lcdr2和lcdr3),所述轻链可变区包含seqidno:73中所示的氨基酸序列。[0311]在一些实施方案中,hcdr1包含seqidno:70中所示的氨基酸序列,hcdr2包含seqidno:71中所示的氨基酸序列,hcdr3包含seqidno:72中所示的氨基酸序列,lcdr1包含seqidno:74中所示的氨基酸序列,lcdr2包含seqidno:75中所示的氨基酸序列,并且lcdr3包含seqidno:76中所示的氨基酸序列。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:69中所示的氨基酸序列的hcvr。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:73中所示的氨基酸序列的lcvr。在一些实施方案中,分离的抗体或它的抗原结合片段包含含有seqidno:69中所示的氨基酸序列的hcvr和包含seqidno:73中所示的氨基酸序列的lcvr。[0312]在一些实施方案中,本公开提供结合sars-cov-2刺突蛋白的分离的抗体,所述sars-cov-2刺突蛋白包含seqidno:108中所示的氨基酸序列,其中所述分离的抗体包含免疫球蛋白恒定区、包含在重链可变区(hcvr)内的三个重链互补决定区(cdr)(hcdr1、hcdr2和hcdr3),所述重链可变区包含seqidno:69中所示的氨基酸序列;以及包含在轻链可变区(lcvr)内的三个轻链互补决定区(cdr)(lcdr1、lcdr2和lcdr3),所述轻链可变区包含seqidno:73中所示的氨基酸序列。[0313]在一些实施方案中,hcdr1包含seqidno:70中所示的氨基酸序列,hcdr2包含seqidno:71中所示的氨基酸序列,hcdr3包含seqidno:72中所示的氨基酸序列,lcdr1包含seqidno:74中所示的氨基酸序列,lcdr2包含seqidno:75中所示的氨基酸序列,并且lcdr3包含seqidno:76中所示的氨基酸序列。在一些实施方案中,分离的抗体包含含有seqidno:69中所示的氨基酸序列的hcvr和包含seqidno:73中所示的氨基酸序列的lcvr。在一些实施方案中,分离的抗体包含含有seqidno:77中所示的氨基酸序列的重链和包含seqidno:78中所示的氨基酸序列的轻链。在一些情况下,免疫球蛋白恒定区是igg1恒定区。在一些情况下,分离的抗体是重组抗体。在一些情况下,分离的抗体是多特异性的。[0314]在一些实施方案中,药物组合物进一步包含第二治疗剂。在一些情况下,第二治疗剂是选自由以下组成的组:结合包含seqidno:108中所示的氨基酸序列的sars-cov-2刺突蛋白的第二抗体或它的抗原结合片段、抗炎剂、抗疟疾剂以及结合tmprss2的抗体或它的抗原结合片段。[0315]在其中感兴趣的抗体的表位已知的某些实施方案中,表位的氨基酸的变异频率可用于确定包括与感兴趣的抗体结合或预计与感兴趣的抗体结合的表位的受试者的频率。例如,在临床环境中,可从受试者分离编码抗体的靶抗原的基因组,并分析分离的基因组是否编码抗体的表位(例如,抗体结合或预计结合的抗原序列)或不同的序列(例如,对应于表位但不是抗体结合或预计结合的序列的序列)。如果比较许多不同的表位,靶向治疗群体中更保守的表位的抗体通常可优于靶向治疗群体中更不保守的表位的抗体。[0316]可在已接受抗体疗法的受试者中评价治疗性抗体的抗原、特别是表位的变异,以评价推定的逃逸变异。例如通过抗体疗法的治疗性干预对不易受干预影响的变体(逃逸变体)产生选择性压力。逃逸变体的一个实例是选择病原体基因组突变,所述病原体基因组突变导致病原体不易受抗体疗法的治疗影响。例如,病原体基因组突变可为治疗性抗体表位的改变,使得抗体不再结合它的靶抗原。本公开的方法和系统可用于通过在治疗后从受试者中分离编码抗体的靶抗原的基因组并分析序列的抗原和/或表位的氨基酸序列中的变异,来评价已接受抗体疗法的受试者中的推定的逃逸变体选择。与抗体能够结合的主题序列(例如,参考序列)相比,表位的变异可鉴定为推定的逃逸变体。[0317]抗原或表位的变异的分析也可用于确定尚未接受特定抗体疗法的受试者是否可能对抗体疗法有响应。包括编码与抗体疗法结合或预计由抗体疗法结合的序列匹配的表位序列的基因组序列(例如,病原体基因组序列)的受试者可分类为可能对抗体疗法有响应的受试者。相反,具有编码对应于与抗体疗法结合或预计由抗体疗法结合的序列不匹配的表位序列的氨基酸的基因组序列(例如,病原体基因组序列)的受试者可分类为不可能对抗体疗法有响应的受试者。因此,本公开的方法和系统可用于个性化医学应用中,其中选择可能对抗体疗法有响应的受试者进行所述疗法的治疗,并且不选择可能对抗体疗法无响应的个体进行所述疗法的治疗。[0318]用于应用的示例性方法和系统[0319]从本公开将理解,本文提供的方法和系统至少部分通过改变查询序列、主题序列和/或分析查询序列和主题序列之间的成对比较,可用于各种应用。[0320]在各种实施方案中,本公开的方法和系统包括以下步骤:获得和/或选择查询序列和(如果不同于查询序列的话)主题序列;从查询序列和主题序列中提取编码序列;对所有查询提取的编码序列和所有主题提取的编码序列进行成对比较,产生每个比较的与一个或多个归类因子(例如,同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系))相关的数据;基于一个或多个归类因子的一个或多个阈值将比较的序列归类成一个或多个归类的序列组(例如,其中向每个归类的序列组分配相似性评分);从进一步分析中过滤一个或多个归类的序列组(例如,基于相似性评分阈值),将编码序列翻译成氨基酸序列;比对翻译的编码序列;以及确定一个或多个主题序列的每一个的保守性和/或可变性。[0321]在各种实施方案中,本公开的方法和系统包括以下步骤:获得和/或选择查询序列和(如果不同于查询序列的话)主题序列;从查询序列中提取编码序列;所有查询提取的编码序列和所有主题序列的成对比较,形成哪些主题序列编码序列没有被提取,为每个比较产生与一个或多个归类因子(例如,同一性百分比、覆盖率百分比率、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系))相关的数据;基于一个或多个归类因子的一个或多个阈值将比较的序列归类到一个或多个归类的序列组中(例如,其中每个归类的序列组被分配相似性评分);从进一步分析中过滤一个或多个归类的序列组(例如,基于相似性评分阈值),将编码序列翻译成氨基酸序列;比对翻译的编码序列;以及确定一个或多个主题序列或它们的部分中的每一个的保守性和/或可变性。[0322]示例性示意图提供于图48中。[0323]在各种实施方案中,本公开的方法和系统包括以下步骤:获得和/或选择查询序列和(如果不同于查询序列的话)主题序列;从查询序列和主题序列中提取编码序列;将编码序列翻译成氨基酸序列;对所有查询翻译的编码序列和所有主题翻译的编码序列进行成对比较,产生每个比较的与一个或多个归类因子(例如,同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和/或系统发育(例如,系统发育分组和/或系统发育关系))相关的数据;基于一个或多个归类因子的一个或多个阈值将比较的序列归类成一个或多个归类的序列组中(例如,其中向每个归类的序列组分配相似性评分);从进一步分析中过滤一个或多个归类的序列组(例如,基于相似性评分阈值);以及确定每个主题序列的保守性和/或可变性。[0324]在各种实施方案中,编码序列的提取是基于参考基因组序列的注释。参考基因组序列的注释可包括编码序列的鉴定、划分或分离。注释的参考基因组序列可在可公开访问的数据库中获得和/或可由用户生成或修改。因此,在其中主题序列是参考基因组序列的各种实施方案中,查询编码序列的鉴定和/或提取可基于例如在参考基因组序列中编码序列的可用或用户定义的注释。在各种实施方案中,可通过将主题基因组序列和/或查询基因组序列与注释的参考基因组序列和/或它的编码序列进行比对来鉴定和/或提取主题基因组序列和/或查询基因组序列的编码序列。[0325]在各种实施方案中,从查询序列和主题序列中提取编码序列是基于检测编码至少约20个、30个、40个、50个、60个、70个、80个、90个、100个、125个、150个、175个、200个、250个或300个或更多个氨基酸的邻接框内密码子。[0326]在各种实施方案中,查询序列和主题序列的成对比较是基于blast算法。blast算法是本领域已知的,包括用于核苷酸序列的blastn和用于氨基酸序列的blastp、有空位的blast和psi-blast。blast算法对序列进行比对,并产生每次比对的各种数据,包括但不限于提供同一性百分比、突变数量、突变百分比、覆盖长度、覆盖率百分比和e值的数据。[0327]可根据如表2中所示的归类因子对比较的序列进行归类。表2基于覆盖率百分比和突变数量为归类的序列组分配相似性评分。在形成归类的序列组之后,可从进一步的分析中过滤出相似性评分小于特定阈值(例如,相似性评分小于1、小于0.95或小于0.8)的归类的序列组。[0328]编码序列(例如,编码序列的剩余归类的组)可通过应用相关遗传密码(例如,人遗传密码)翻译成氨基酸序列。可对翻译的编码序列进行比对。如上所述,可使用blast算法完成比对。然后可确定序列的保守性和/或可变性。本公开的方法和系统中所示的各种分析不需要在氨基酸序列的比对之后进行过滤或选择。没有进一步选择的比对提供了有价值的信息。例如,在各种实施方案中,氨基酸序列的比对提供诸如比对位置处的保守性(例如,在一个或多个比对位置的每一个处包括与参考同一的氨基酸的比对序列的百分比)和比对位置处的序列变异(例如,可在每个比对位置处出现的不同氨基酸的数量和频率)的信息。就在某些实施方案中在氨基酸比对之后选择序列而言,选择可由用户例如根据应用于通过氨基酸序列比对产生的信息的标准来进行。因此,在各种实施方案中,不对氨基酸序列应用过滤器,例如,不使用阈值来选择氨基酸序列或它们的部分。在一些实施方案中,可基于如本文公开的阈值选择保守序列或可变序列。[0329]在其中评价保守性和/或可变性的各种实施方案中,查询序列是序列的第一集合,并且主题序列是序列的第二不同集合。在各种实施方案中,查询序列是序列的第一集合,并且主题序列是序列的相同集合。在其中评价保守性和/或可变性的各种实施方案中,查询序列是序列的第一集合,并且主题序列是单个序列(例如,感兴趣的序列)。[0330]在某些实施方案中,可关于成对比较来评价保守性和/或可变性,在成对比较中,查询序列是来自特定物种(例如,特定病原体)的多个生物体的序列的第一集合,并且主题序列是序列的相同集合。各种所述实施方案可从成对比较产生数据,所述数据可用于确定特定物种的保守序列和/或特定物种的可变序列。保守序列可例如在抗体或疫苗开发中经选择或使用抗原或表位。保守序列可为在例如人类受试者中病原体的正选择(例如进化存活选择压力和/或抗生素抗性选择)下的性状。可变序列可例如选择作为实验室工程化(例如,基因工程化)的靶标,选择作为系统发育分析的靶标,并且/或鉴定为经历进化多样化的序列。序列的变异也可用于产生可能序列(例如,可能的氨基酸序列)的列表或数据库,其可用于例如产生可能的质量用于质谱法分析。[0331]在某些实施方案中,可关于成对比较来评价保守性和/或可变性,在成对比较中,查询序列是来自特定物种(例如,特定病原体)的多个生物体的序列的集合,并且主题序列包括来自特定株系或生物体的一个或多个序列。在各种实施方案中,查询序列包括来自不同样品(例如,病原体的多个临床分离株)的多个生物体的序列。在各种实施方案中,受试者是实验室株系。在某些实施方案中,主题序列和查询序列之间测量的保守性和/或可变性可用于确定主题株系或生物体在查询序列中的代表性如何。在各种实施方案中,在生物体水平和/或通过评价所有比对的序列来确定主题株系是否代表查询序列。在各种实施方案中,生物体水平的确定可基于系统发育分析。例如,系统发育分析可鉴定簇中一个或多个感兴趣的序列,并确定所有簇的大小。[0332]序列的变异也可用于产生可能序列(例如,可能的氨基酸序列)的列表或数据库,其可用于例如产生可能的质量的列表或数据库用于质谱法分析。[0333]为了提供一个特定实例,本公开的方法和系统可用于分析诸如sars-cov-2的病毒的序列的各种实施方案中。在各种实施方案中,将本公开的方法和系统应用于sars-cov-2序列的分析可包括作为主题序列的一个或多个参考sars-cov-2序列,诸如作为基因库登录号mn908947可公开获得的已知sars-cov-2参考基因组序列。在一些实施方案中,主题序列可为或包括sars-cov-2参考基因组序列的一部分(例如,基因库登录号mn908947的一部分),其编码氨基酸序列,例如,sars-cov-2刺突蛋白或它的一部分(例如,sars-cov-2刺突受体结合结构域(rbd))。在各种实施方案中,查询序列可为多个sars-cov-2基因组序列或从其提取的编码序列。例如,通过全球共享流行性感冒数据倡议(gisaid)数据库(https://www.gisaid.org/)可获得至少约120,000个sars-cov-2基因组序列。可从受感染的受试者中得出替代或额外查询序列。可例如根据图26中发现的一般示意图从sars-cov-2基因组序列中提取编码序列。可如图27中发现的一般示意图中所说明,执行所有查询提取的编码序列和所有主题提取的编码序列的成对比较。查询序列和主题sars-cov-2序列的成对比较产生与归类因子相关的数据,所述归类因子包括同一性百分比、覆盖率百分比、覆盖长度、在预定覆盖长度上的同一性百分比、e值、突变数量、突变百分比和系统发育(例如,每个比较的系统发育分组和/或系统发育关系)。这些数据允许进行各种进一步分析。可准备包括所得到的序列比较数据的汇总表,例如如图28的表中发现的总体布局所说明,其示出了归类因子的亚组。此外,查询sars-cov-2序列与参考sars-cov-2的每个比较可基于一个或多个归类因子的一个或多个阈值归类成一个或多个归类的序列组。在一些实施方案中,可例如通过如表2所说明的相似性评分的分配将一个或多个归类因子的一个或多个阈值集成到单个量度中。在一些实施方案中,一个或多个归类因子的阈值(或基于两个或多个这样的阈值确定的相似性评分的阈值)可用于将sars-cov-2序列比较结果归类成各类别,其中一个或多个类别包括与参考序列或它的一部分更相似的查询序列,并且一个或多个不同类别包括与参考序列或它的一部分不太相似的查询序列。因此,在各种实施方案中,可保留与参考序列更相似的序列,用于关于参考序列或它的一部分的进一步分析,并且可从进一步分析中排除与参考序列或它的一部分不太相似的序列。当在查询基因组序列中发现与参考序列或它的一部分更相似的序列时,该参考序列或它的一部分可称为“存在于”查询基因组序列中,如例如在图28中一般性地指示。保守性和/或可变性的量度可以图形、热图、系统发育、排序列表和其他格式显示(对于一般示例,参见例如图29-图33)。可翻译并比对每个参考序列或它的一部分的剩余sars-cov-2序列,并且可确定比对序列的氨基酸保守性和/或可变性的量度。[0334]在各种实施方案中,用于核酸序列的比较的blast参数可使用blast默认值或利用表4中提供的任何值进行。在各种实施方案中,用于氨基酸序列的比较的blast参数可使用blast默认值或利用表5中提供的任何值进行。使用本公开的系统和方法不需要任何参数或参数组合的值的特定组。[0335]表4核酸比较blastn参数[0336][0337]表5氨基酸比较blastp参数[0338][0339][0340]示例性实施方案[0341]本公开尤其包括以下示例性实施方案:[0342]1.一种用于在针对病原体的疗法的开发中将氨基酸序列鉴定为候选抗原的方法,所述方法包括:[0343]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0344]通过计算装置的处理器从所述基因组序列中提取编码序列;[0345]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0346]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0347]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0348]通过所述处理器对所述氨基酸序列进行比对;[0349]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;[0350]选择分类为保守的所述氨基酸序列的部分,将所述选择的保守序列与人蛋白序列进行比较,并进一步将所述选择的保守序列分类为与人蛋白序列同一或不同一;以及[0351]将与人蛋白序列不同一的选择的保守序列归类为在针对所述病原体的疗法的开发中的候选抗原。[0352]2.根据实施方案1所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0353]3.根据实施方案1或实施方案2所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0354]4.根据实施方案1至3中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0355]5.根据实施方案4所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0356]6.根据实施方案5所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0357]7.根据实施方案1至6中任一项所述的方法,其中所述同一性的量度包括突变数量。[0358]8.根据实施方案1至7中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0359]9.根据实施方案1至8中任一项所述的方法,其中所述同一性的量度包括计算e值。[0360]10.根据实施方案1至9中任一项所述的方法,其中将所述选择的保守序列归类为候选抗原进一步包括确定所述选择的保守序列中一个或多个氨基酸结构域的存在或不存在。[0361]11.根据实施方案1至10中任一项所述的方法,其中将所述选择的保守序列归类为候选抗原进一步包括确定所述候选抗原是否对应于在所述病原体的膜和/或细胞壁内分泌或暴露的蛋白质。[0362]12.根据实施方案1至11中任一项所述的方法,其中将所述选择的保守序列归类为候选抗原进一步包括确定所述选择的保守序列中跨膜结构域的存在。[0363]13.根据实施方案1至12中任一项所述的方法,其中所述疗法包括疫苗,并且所述方法进一步包括非临床评价所述候选抗原的免疫原性。[0364]14.根据实施方案13所述的方法,其中所述评价步骤包括向动物施用包含所述候选抗原的多肽。[0365]15.根据实施方案1至14中任一项所述的方法,其中所述疗法包括抗体疗法,并且所述方法进一步包括产生特异性结合至所述候选抗原上的表位的抗体或其片段。[0366]16.根据实施方案1至15中任一项所述的方法,其中所述病原体是病毒。[0367]17.根据实施方案16所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0368]18.根据实施方案16所述的方法,其中所述病毒是冠状病毒。[0369]19.根据实施方案18所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0370]20.根据实施方案1至15中任一项所述的方法,其中所述病原体是细菌。[0371]21.根据实施方案20所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0372]22.一种在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变的方法,所述方法包括:[0373]在向每个受试者施用所述治疗剂之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;[0374]通过计算装置的处理器从所述基因组序列中提取编码序列;[0375]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0376]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0377]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0378]通过所述处理器对所述氨基酸序列进行比对;[0379]在所述比对的氨基酸序列中鉴定在所述比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一个或多个推定的逃逸突变。[0380]23.根据实施方案22所述的方法,其中所述参考包括代表典型病原体序列的一种或多种完整或部分病原体基因组序列、所述病原体的一个或多个临床株系、来自施用所述治疗剂的一个或多个受试者的病原体的一个或多个早期样品或来自未施用所述治疗剂的受试者的病原体的一个或多个样品。[0381]24.根据实施方案22或实施方案23所述的方法,所述方法进一步包括确定所述一个或多个推定的逃逸突变是否降低所述治疗剂与参考多肽的结合亲和性的步骤。[0382]25.根据实施方案22至24中任一项所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0383]26.根据实施方案22至25中任一项所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0384]27.根据实施方案22至26中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0385]28.根据实施方案27所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0386]29.根据实施方案28所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0387]30.根据实施方案22至29中任一项所述的方法,其中所述同一性的量度包括突变数量。[0388]31.根据实施方案22至30中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0389]32.根据实施方案22至31中任一项所述的方法,其中所述同一性的量度包括计算e值。[0390]33.根据实施方案22至32中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0391]编码与所述病原体相关的蛋白质的核酸的编码序列;[0392]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0393]编码蛋白质的核酸的非保守序列;[0394]与所述病原体相关的特定蛋白质内的保守结构域;和[0395]与所述病原体相关的特定蛋白质内的非保守结构域。[0396]34.根据实施方案22至33中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0397]35.根据实施方案22至34中任一项所述的方法,其中所述病原体是病毒。[0398]36.根据实施方案35所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0399]37.根据实施方案35所述的方法,其中所述病毒是冠状病毒。[0400]38.根据实施方案37所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0401]39.根据实施方案38所述的方法,其中所述冠状病毒是sars-cov-2。[0402]40.根据实施方案22至39中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0403]41.根据实施方案22至40中任一项所述的方法,其中所述治疗剂包含抗体。[0404]42.根据实施方案41所述的方法,其中所述抗体结合sars-cov-2。[0405]43.根据实施方案42所述的方法,其中所述抗体结合sars-cov-2刺突蛋白。[0406]44.根据实施方案41至43中任一项所述的方法,其中根据表3,所述抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。[0407]45.根据实施方案22至34中任一项所述的方法,其中所述病原体是细菌。[0408]46.根据实施方案45所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0409]47.一种向有需要的受试者施用用于治疗病原体感染的治疗剂的方法,所述方法包括:[0410]通过以下方式选择氨基酸序列的保守部分:[0411]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0412]通过计算装置的处理器从所述基因组序列中提取编码序列;[0413]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0414]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0415]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0416]通过所述处理器对所述氨基酸序列进行比对;[0417]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;以及[0418]选择所述比对的氨基酸序列的保守部分:以及[0419]如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的所述保守部分,则向所述受试者施用所述治疗剂,其中所述治疗剂选择性地结合所述氨基酸序列的所述保守部分。[0420]48.根据实施方案47所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0421]49.根据实施方案47或实施方案48所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0422]50.根据实施方案47至49中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0423]51.根据实施方案50所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0424]52.根据实施方案51所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0425]53.根据实施方案47至52中任一项所述的方法,其中所述同一性的量度包括突变数量。[0426]54.根据实施方案47至53中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0427]55.根据实施方案47至54中任一项所述的方法,其中所述同一性的量度包括计算e值。[0428]56.根据实施方案47至55中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0429]编码与所述病原体相关的蛋白质的核酸的编码序列;[0430]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0431]编码蛋白质的核酸的非保守序列;[0432]与所述病原体相关的特定蛋白质内的保守结构域;和[0433]与所述病原体相关的特定蛋白质内的非保守结构域。[0434]57.根据实施方案47至56中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0435]58.根据实施方案47至57中任一项所述的方法,其中所述病原体是病毒。[0436]59.根据实施方案58所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0437]60.根据实施方案58所述的方法,其中所述病毒是冠状病毒。[0438]61.根据实施方案60所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0439]62.根据实施方案61所述的方法,其中所述冠状病毒是sars-cov-2。[0440]63.根据实施方案47至62中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0441]64.根据实施方案47至63中任一项所述的方法,其中所述治疗剂包含抗体。[0442]65.根据实施方案64所述的方法,其中所述抗体结合sars-cov-2。[0443]66.根据实施方案65所述的方法,其中所述抗体结合sars-cov-2刺突蛋白。[0444]67.根据实施方案64至66中任一项所述的方法,其中根据表3,所述抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。[0445]68.根据实施方案47至57中任一项所述的方法,其中所述病原体是细菌。[0446]69.根据实施方案68所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0447]70.一种用于选择用于治疗感染病原体的受试者的治疗剂的方法,所述方法包括:[0448]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0449]通过计算装置的处理器从所述基因组序列中提取编码序列;[0450]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0451]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0452]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0453]通过所述处理器对所述氨基酸序列进行比对;[0454]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表所述病原体的编码序列的保守部分;以及[0455]选择结合所述保守编码序列的治疗剂作为对感染所述病原体的受试者的治疗。[0456]71.根据实施方案70所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0457]72.根据实施方案70或实施方案71所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0458]73.根据实施方案70至72中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0459]74.根据实施方案73所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0460]75.根据实施方案74所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0461]76.根据实施方案70至75中任一项所述的方法,其中所述同一性的量度包括突变数量。[0462]77.根据实施方案70至76中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0463]78.根据实施方案70至77中任一项所述的方法,其中所述同一性的量度包括计算e值。[0464]79.根据实施方案70至78中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0465]编码与所述病原体相关的蛋白质的核酸的编码序列;[0466]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0467]编码蛋白质的核酸的非保守序列;[0468]与所述病原体相关的特定蛋白质内的保守结构域;和[0469]与所述病原体相关的特定蛋白质内的非保守结构域。[0470]80.根据实施方案70至79中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0471]81.根据实施方案80所述的方法,其中所述方法进一步包括将所述治疗剂非临床评价为疫苗或它的组分。[0472]82.根据实施方案81所述的方法,其中所述评价步骤包括向动物施用所述治疗剂。[0473]83.根据实施方案70至82中任一项所述的方法,其中所述病原体是病毒。[0474]84.根据实施方案83所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0475]85.根据实施方案83所述的方法,其中所述病毒是冠状病毒。[0476]86.根据实施方案85所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0477]87.根据实施方案86所述的方法,其中所述冠状病毒是sars-cov-2。[0478]88.根据实施方案70至87中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0479]89.根据实施方案70至88中任一项所述的方法,其中所述治疗剂包含抗体。[0480]90.根据实施方案89所述的方法,其中所述抗体结合sars-cov-2。[0481]91.根据实施方案90所述的方法,其中所述抗体结合sars-cov-2刺突蛋白。[0482]92.根据实施方案89至91中任一项所述的方法,其中根据表3,所述抗体包括至少一种抗体、重链(hc)、轻链(lc)、重链可变区(hcvr)、轻链可变区(lcvr)、重链互补决定区(hcdr)或轻链cdr(lcdr)。[0483]93.根据实施方案70至82中任一项所述的方法,其中所述病原体是细菌。[0484]94.根据实施方案93所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0485]95.一种用于评价代表病原体的氨基酸序列的部分的保守性的方法,所述方法包括:[0486]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0487]通过计算装置的处理器从所述基因组序列中提取编码序列;[0488]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0489]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0490]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0491]通过所述处理器对所述氨基酸序列进行比对;以及[0492]使用所述比对的氨基酸序列鉴定代表所述病原体的氨基酸序列的一个或多个部分的保守水平。[0493]96.根据实施方案95所述的方法,其中所述部分中的一个或多个被鉴定为在针对所述病原体的疗法的开发中的候选抗原。[0494]97.根据实施方案95或实施方案96所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0495]98.根据实施方案95至97中任一项所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0496]99.根据实施方案95至98中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0497]100.根据实施方案99所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0498]101.根据实施方案100所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0499]102.根据实施方案95至101中任一项所述的方法,其中所述同一性的量度包括突变数量。[0500]103.根据实施方案95至102中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0501]104.根据实施方案95至103中任一项所述的方法,其中所述同一性的量度包括计算e值。[0502]105.根据实施方案95至104中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0503]编码与所述病原体相关的蛋白质的核酸的编码序列;[0504]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0505]编码蛋白质的核酸的非保守序列;[0506]与所述病原体相关的特定蛋白质内的保守结构域;和[0507]与所述病原体相关的特定蛋白质内的非保守结构域。[0508]106.根据实施方案95至105中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0509]107.根据实施方案95至106中任一项所述的方法,其中所述病原体是病毒。[0510]108.根据实施方案107所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0511]109.根据实施方案107所述的方法,其中所述病毒是冠状病毒。[0512]110.根据实施方案109所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0513]111.根据实施方案110所述的方法,其中所述冠状病毒是sars-cov-2。[0514]112.根据实施方案95至111中任一项所述的方法,其中所述基因组序列是sars-cov-2基因组序列,并且所述参考序列是sars-cov-2参考序列。[0515]113.根据实施方案95至112中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0516]114.根据实施方案95至106中任一项所述的方法,其中所述病原体是细菌。[0517]115.根据实施方案114所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0518]116.一种用于鉴定分离的病原体是否代表循环株系的方法,所述方法包括:[0519]从数据结构获得所述病原体的所述循环株系的多个完整或部分基因组序列;[0520]鉴定所述循环株系的所述序列的一个或多个保守部分;[0521]获得所述分离的病原体的多个完整或部分基因组序列;以及[0522]通过将所述分离的病原体的所述序列的至少一部分与所述循环株系的所述序列的所述鉴定的一个或多个保守部分进行比较来鉴定所述分离的病原体是否代表所述循环株系。[0523]117.根据实施方案116所述的方法,其中鉴定所述循环株系的所述序列的一个或多个保守部分包括:[0524]通过计算装置的处理器从所述基因组序列中提取编码序列;[0525]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0526]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0527]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0528]通过所述处理器对所述氨基酸序列进行比对;以及[0529]根据所述部分在所述比对的氨基酸序列中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类。[0530]118.根据实施方案116或实施方案117所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0531]119.根据实施方案116至118中任一项所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0532]120.根据实施方案116至119中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0533]121.根据实施方案120所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0534]122.根据实施方案121所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0535]123.根据实施方案116至122中任一项所述的方法,其中所述同一性的量度包括突变数量。[0536]124.根据实施方案116至123中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0537]125.根据实施方案116至124中任一项所述的方法,其中所述同一性的量度包括计算e值。[0538]126.根据实施方案116至125中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0539]编码与所述病原体相关的蛋白质的核酸的编码序列;[0540]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0541]编码蛋白质的核酸的非保守序列;[0542]与所述病原体相关的特定蛋白质内的保守结构域;和[0543]与所述病原体相关的特定蛋白质内的非保守结构域。[0544]127.根据实施方案116至126中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0545]128.根据实施方案116至127中任一项所述的方法,其中所述病原体是病毒。[0546]129.根据实施方案128所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0547]130.根据实施方案128所述的方法,其中所述病毒是冠状病毒。[0548]131.根据实施方案130所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0549]132.根据实施方案131所述的方法,其中所述冠状病毒是sars-cov-2。[0550]133.根据实施方案116至132中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0551]134.根据实施方案116至127中任一项所述的方法,其中所述病原体是细菌。[0552]135.根据实施方案134所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0553]136.一种用于鉴定代表病原体的肽的质荷比的方法,所述方法包括:[0554]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0555]通过计算装置的处理器从所述基因组序列中提取编码序列;[0556]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0557]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0558]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;以及[0559]确定所述一个或多个氨基酸序列或它们的部分的所述质荷比。[0560]137.根据实施方案136所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述病原体的不同株系的所述多个完整或部分基因组序列包括通过所述处理器合并重叠的重叠群以产生所述完整或部分基因组序列的至少一部分。[0561]138.根据实施方案136或实施方案137所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0562]139.根据实施方案136至138中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0563]140.根据实施方案139所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0564]141.根据实施方案140所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0565]142.根据实施方案136至141中任一项所述的方法,其中所述同一性的量度包括突变数量。[0566]143.根据实施方案136至142中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0567]144.根据实施方案136至143中任一项所述的方法,其中所述同一性的量度包括计算e值。[0568]145.根据实施方案136至144中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0569]编码与所述病原体相关的蛋白质的核酸的编码序列;[0570]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0571]编码蛋白质的核酸的非保守序列;[0572]与所述病原体相关的特定蛋白质内的保守结构域;和[0573]与所述病原体相关的特定蛋白质内的非保守结构域。[0574]146.根据实施方案136至145中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0575]147.根据实施方案136至146中任一项所述的方法,其中所述病原体是病毒。[0576]148.根据实施方案147所述的方法,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0577]149.根据实施方案147所述的方法,其中所述病毒是冠状病毒。[0578]150.根据实施方案149所述的方法,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0579]151.根据实施方案150所述的方法,其中所述冠状病毒是sars-cov-2。[0580]152.根据实施方案136至151中任一项所述的方法,所述方法包括评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0581]153.根据实施方案136至146中任一项所述的方法,其中所述病原体是细菌。[0582]154.根据实施方案153所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0583]155.一种用于将氨基酸序列鉴定为候选抗生素抗性标记物的方法,所述方法包括:[0584]从数据结构获得病原性细菌的多个完整或部分质粒序列;[0585]通过计算装置的处理器从所述质粒序列中提取编码序列;[0586]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0587]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0588]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0589]通过所述处理器对所述氨基酸序列进行比对;[0590]根据所述部分在所述多个质粒序列中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;[0591]选择分类为保守的所述氨基酸序列的部分;以及[0592]将所选择的保守序列归类为候选抗生素抗性标记物。[0593]156.根据实施方案155所述的方法,所述方法进一步包括根据一个或多个额外标准将所述候选抗生素抗性标记物鉴定为候选者,所述额外标准包括在所选择的序列中存在跨膜结构域。[0594]157.根据实施方案155或实施方案156所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述多个完整或部分质粒序列包括通过所述处理器合并重叠的重叠群以产生至少一些所述完整或部分质粒序列。[0595]158.根据实施方案155至157中任一项所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0596]159.根据实施方案155至158中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0597]160.根据实施方案159所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0598]161.根据实施方案160所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0599]162.根据实施方案155至161中任一项所述的方法,其中所述同一性的量度包括突变数量。[0600]163.根据实施方案155至162中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0601]164.根据实施方案155至163中任一项所述的方法,其中所述同一性的量度包括计算e值。[0602]165.根据实施方案155至164中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0603]编码与所述病原体相关的蛋白质的核酸的编码序列;[0604]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0605]编码蛋白质的核酸的非保守序列;[0606]与所述病原体相关的特定蛋白质内的保守结构域;和[0607]与所述病原体相关的特定蛋白质内的非保守结构域。[0608]166.根据实施方案155至165中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0609]167.根据实施方案155至166中任一项所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0610]168.一种用于鉴定代表质粒的编码序列的一个或多个保守部分的方法,所述方法包括:[0611]从数据结构获得病原性细菌的多个完整或部分质粒序列;[0612]通过计算装置的处理器从所述质粒序列中提取编码序列;[0613]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0614]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0615]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0616]通过所述处理器对所述氨基酸序列进行比对;以及[0617]根据所述部分在所述多个质粒序列中的保守水平对所述氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表所述质粒的编码序列的一个或多个保守部分。[0618]169.根据实施方案168所述的方法,其中所述数据结构包括重叠群,并且其中从所述数据结构获得所述多个完整或部分质粒序列包括通过所述处理器合并重叠的重叠群以产生至少一些所述完整或部分质粒序列。[0619]170.根据实施方案168或实施方案169所述的方法,其中所述归类步骤包括量化多个对中的每一个的所述同一性的量度和所述覆盖率的量度,所述对中的每一个包括提取的编码序列和参考序列。[0620]171.根据实施方案168至170中任一项所述的方法,其中所述归类步骤包括对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0621]172.根据实施方案171所述的方法,其中所述计算步骤包括创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0622]173.根据实施方案172所述的方法,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0623]174.根据实施方案168至173中任一项所述的方法,其中所述同一性的量度包括突变数量。[0624]175.根据实施方案168至174中任一项所述的方法,其中所述覆盖率的量度包括覆盖率百分比。[0625]176.根据实施方案168至175中任一项所述的方法,其中所述同一性的量度包括计算e值。[0626]177.根据实施方案168至176中任一项所述的方法,所述方法包括评价以下中的一种或多种:[0627]编码与所述病原体相关的蛋白质的核酸的编码序列;[0628]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0629]编码蛋白质的核酸的非保守序列;[0630]与所述病原体相关的特定蛋白质内的保守结构域;和[0631]与所述病原体相关的特定蛋白质内的非保守结构域。[0632]178.根据实施方案168至177中任一项所述的方法,其中氨基酸序列的每个部分包括一个或多个氨基酸位置。[0633]179.根据实施方案168至178中任一项所述的方法,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0634]180.一种用于自动鉴定代表病原体的编码序列的一个或多个保守部分的系统,所述系统包括:[0635]处理器;以及[0636]其上具有指令的存储器,所述指令在由所述处理器执行时引起所述处理器:[0637]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0638]通过所述处理器从所述基因组序列中提取编码序列;[0639]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0640]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0641]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0642]通过所述处理器对所述氨基酸序列进行比对;以及[0643]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表所述病原体的编码序列的一个或多个保守部分。[0644]181.根据实施方案180所述的系统,其中所述指令当由所述处理器执行时引起所述处理器对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0645]182.根据实施方案181所述的系统,其中所述指令当由所述处理器执行时引起所述处理器创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0646]183.根据实施方案182所述的系统,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0647]184.根据实施方案180至183中任一项所述的系统,其中所述数据结构包括重叠群,并且其中所述指令当由所述处理器执行时引起所述处理器通过所述处理器合并重叠的重叠群以产生至少一些所述完整或部分基因组序列来获得所述病原体的不同株系的所述多个完整或部分基因组序列。[0648]185.根据实施方案180至184中任一项所述的系统,其中所述指令当由所述处理器执行时引起所述处理器评价以下中的一种或多种:[0649]编码与所述病原体相关的蛋白质的核酸的编码序列;[0650]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0651]编码蛋白质的核酸的非保守序列;[0652]与所述病原体相关的特定蛋白质内的保守结构域;和[0653]与所述病原体相关的特定蛋白质内的非保守结构域。[0654]186.根据实施方案180至185中任一项所述的系统,其中所述指令当由所述处理器执行时引起所述处理器评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0655]187.根据实施方案180至186中任一项所述的系统,其中所述病原体是病毒。[0656]188.根据实施方案187所述的系统,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0657]189.根据实施方案187所述的系统,其中所述病毒是冠状病毒。[0658]190.根据实施方案189所述的系统,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0659]191.根据实施方案190所述的系统,其中所述冠状病毒是sars-cov-2。[0660]192.根据实施方案180至186中任一项所述的系统,其中所述病原体是细菌。[0661]193.根据实施方案192所述的系统,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0662]194.一种用于自动鉴定代表质粒的编码序列的一个或多个保守部分的系统,所述系统包括:[0663]处理器;以及[0664]其上具有指令的存储器,所述指令在由所述处理器执行时引起所述处理器:[0665]从数据结构获得病原性细菌的多个完整或部分质粒序列;[0666]通过所述处理器从所述质粒序列中提取编码序列;[0667]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0668]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0669]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0670]通过所述处理器对所述氨基酸序列进行比对;以及[0671]根据所述部分在所述多个质粒序列中的保守水平对所述氨基酸序列的多个部分中的每一个进行分类,从而鉴定代表所述质粒的编码序列的一个或多个保守部分。[0672]195.根据实施方案194所述的系统,其中所述指令当由所述处理器执行时引起所述处理器对于针对一组主题序列的一组查询编码序列中的每一个,计算所述查询编码序列和每个主题序列之间的相似性的量度,所述相似性量度中的每一个是所述查询序列和所述主题序列之间的同一性的量度以及所述查询序列和所述主题序列之间的覆盖率的量度的函数。[0673]196.根据实施方案195所述的系统,其中所述指令当由所述处理器执行时引起所述处理器创建所述相似性的量度的矩阵,并呈现所述矩阵的图形表示,从而显示所述查询序列和所述主题序列之间的保守水平。[0674]197.根据实施方案196所述的系统,其中所述图形表示包括热图、图形和系统发育中的一个或多个。[0675]198.根据实施方案194至197中任一项所述的系统,其中所述数据结构包括重叠群,并且其中所述指令当由所述处理器执行时引起所述处理器通过所述处理器合并重叠的重叠群以产生至少一些所述完整或部分质粒序列来获得病原性细菌的所述多个完整或部分质粒序列。[0676]199.根据实施方案194至198中任一项所述的系统,其中所述指令当由所述处理器执行时引起所述处理器评价以下中的一种或多种:[0677]编码与所述病原体相关的蛋白质的核酸的编码序列;[0678]编码与所述病原体相关的蛋白质的核酸序列的保守序列;[0679]编码蛋白质的核酸的非保守序列;[0680]与所述病原体相关的特定蛋白质内的保守结构域;和[0681]与所述病原体相关的特定蛋白质内的非保守结构域。[0682]200.根据实施方案194至199中任一项所述的系统,其中所述指令当由所述处理器执行时引起所述处理器评价冠状病毒刺突(s)蛋白[例如,mers、sars-cov或sars-cov2刺突(s)蛋白]或其受体结合结构域(rbd)。[0683]201.根据实施方案194至200中任一项所述的系统,其中所述病原体是病毒。[0684]202.根据实施方案201所述的系统,其中所述病毒是甲氧西林抗性金黄色葡萄球菌(mrsa)、乙型肝炎病毒(hbv)、流感或埃博拉病毒。[0685]203.根据实施方案201所述的系统,其中所述病毒是冠状病毒。[0686]204.根据实施方案203所述的系统,其中所述冠状病毒是严重急性呼吸综合征相关冠状病毒(sars-cov)、严重急性呼吸综合征冠状病毒2(sars-cov-2)或中东呼吸综合征相关冠状病毒(mers-cov)。[0687]205.根据实施方案204所述的系统,其中所述冠状病毒是sars-cov-2。[0688]206.根据实施方案194至200中任一项所述的系统,其中所述病原体是细菌。[0689]207.根据实施方案206所述的系统,其中所述细菌是葡萄球菌属物种或假单胞菌属物种。[0690]208.一种用于在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变的治疗剂,所述用途包括:[0691]在向每个受试者施用所述治疗剂之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;[0692]通过计算装置的处理器从所述基因组序列中提取编码序列;[0693]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0694]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0695]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0696]通过所述处理器对所述氨基酸序列进行比对;[0697]在所述比对的氨基酸序列中鉴定在所述比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一个或多个推定的逃逸突变。[0698]209.一种用于治疗病原体感染的治疗剂,所述用途包括:[0699]通过以下方式选择氨基酸序列的保守部分:[0700]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0701]通过计算装置的处理器从所述基因组序列中提取编码序列;[0702]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0703]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0704]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0705]通过所述处理器对所述氨基酸序列进行比对;[0706]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;以及[0707]选择所述比对的氨基酸序列的保守部分:以及[0708]如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的所述保守部分,则向所述受试者施用所述治疗剂,其中所述治疗剂选择性地结合所述氨基酸序列的所述保守部分。[0709]210.一种确定由抗体结合的病原体表位是否保守的方法,所述方法包括:[0710]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0711]通过计算装置的处理器从所述基因组序列中提取编码序列;[0712]将所述编码序列与编码所述病原体表位的参考序列进行比较;[0713]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0714]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0715]将所述选择的编码序列转换成相应的氨基酸序列;以及[0716]确定所述病原体的所述不同株系中的所述病原体表位的保守水平。[0717]210.治疗剂用于制造药物的用途,所述药物用于在向一个或多个受试者施用治疗剂以治疗病原体感染之后鉴定一个或多个推定的逃逸突变,所述用途包括:[0718]在向每个受试者施用所述药物之后获得从一个或多个受试者分离的多个完整或部分病原体基因组序列;[0719]通过计算装置的处理器从所述基因组序列中提取编码序列;[0720]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0721]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0722]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0723]通过所述处理器对所述氨基酸序列进行比对;[0724]在所述比对的氨基酸序列中鉴定在所述比对的氨基酸序列中比在参考中更频繁的一种或多种氨基酸变体,所述一种或多种氨基酸变体是一个或多个推定的逃逸突变。[0725]211.治疗剂用于制造用于治疗病原体感染的药物的用途,所述用途包括:[0726]通过以下方式选择氨基酸序列的保守部分:[0727]从数据结构获得所述病原体的不同株系的多个完整或部分基因组序列;[0728]通过计算装置的处理器从所述基因组序列中提取编码序列;[0729]通过所述处理器根据同一性的量度和覆盖率的量度对所述编码序列进行归类,其中所述同一性的量度包括同一性百分比、在预定覆盖长度上的同一性百分比、突变数量和突变百分比中的一个或多个,并且其中所述覆盖率的量度包括覆盖率百分比和覆盖长度中的一个或多个;[0730]根据所述同一性的量度和所述覆盖率的量度在所述归类的编码序列中选择编码序列;[0731]通过所述处理器将所述选择的编码序列转换成相应的氨基酸序列;[0732]通过所述处理器对所述氨基酸序列进行比对;[0733]根据所述部分在所述病原体的所述不同株系中的保守水平对所述比对的氨基酸序列的多个部分中的每一个进行分类;以及[0734]选择所述比对的氨基酸序列的保守部分:以及[0735]如果从所述受试者分离的完整或部分病原体基因组序列编码氨基酸序列的所述保守部分,则向所述受试者施用所述药物,其中所述治疗剂选择性地结合所述氨基酸序列的所述保守部分。[0736]实施例[0737]本实施例提供本公开的示例性方法和系统及它们的示例性用途。过去的十年见证了泛滥的测序的基因组,在最频繁测序的物种中有病毒和细菌,其中许多是病原性的。例如,根据对ncbi数据库中存在的超过约150万个基因组序列的一个审查,ncbi数据库包括约642,604个真核基因组序列、约757,524个细菌基因组序列和约176,471个病毒基因组序列。[0738]研究人员发现,在一些情况下,对大规模基因组数据集的分析可揭示病原体基因组的变化,这些变化在流行病学上与临床后果相关。在某些实例中,此类相关变化可对病原体表型有显著贡献。然而,随着每周可公开访问的基因组序列数量增加数千个基因组,管理不断扩大的测序信息量变得越来越困难。此外,访问序列数据对用户不友好;需要计算技能来将数据转换成可行的形式。本实施例提供提取和处理可公开访问的基因组序列的方法和系统。本文提供的方法和系统特别适于在用户友好的计算程序中使用,所述计算程序例如以低或最少的用户输入来执行可公开访问的基因组序列的分析。[0739]本实施例展现可公开获得的基因组序列的分析揭示影响或可能影响病原体表型(例如,宿主-病原体相互作用)、影响治疗性开发或为治疗性开发(例如,治疗抗体的开发)提供靶标的基因组的特定特征的能力。本实施例特别展现目前公开的方法和系统在鉴定尤其用于治疗剂的开发的保守序列中、例如作为用于治疗性抗体开发的抗原的效用。尽管常规疫苗学可需要约5年至约15年来进行疫苗抗原的选择和验证,并且使用基于基因组的方法的反向疫苗学可需要约1年至约2年来进行疫苗抗原的选择和验证,但本文公开的方法和系统可快速鉴定用于疫苗开发的抗原,例如促进在约1周至约2周内选择和验证疫苗抗原。[0740]实施例1:用于鉴定治疗目标的保守序列的示例性方法和系统[0741]本实施例提供用于鉴定治疗目标的保守序列的示例性方法和系统。本实施例利用以r编写的计算机程序(“got_gene”),所述程序使用本领域已知的blast算法和专有的r包来鉴定、比较和表征数千个输入基因组序列。本文公开的got_gene程序是用户友好型并且不需要计算技能。其自动讯问公共数据库,以表格、图形和视觉形式提供一组全面的信息。[0742]本实施例的程序包括约2500行代码和10个r包。本实施例的程序利用2至4个外部程序:blastn、phyml和quicktree中的一个或两个,以及可选的megahit。blast算法用于比对,并且可在万维网(worldwideweb)的ncbi.nlm.nih.gov上使用;quicktree用于系统发育分析,并且可例如在hypertexttransferprotocolgithub.com/tseemann/quicktree使用;megahit用于序列组装,并且可在例如万维网的metagenomics.wiki/tools/assembly/megahit上使用。所利用的r包包括:data.table;iranges;reutils;biofiles;ggplot2;cowplot;rcolorbrewer;reshape2;gridextra;decipher;shiny;colourpicker;以及plotly。[0743]不希望受任何特定示例或解释的限制,本实施例中使用的got_gene程序可被视为包括五个步骤(例如,参见图18):[0744](1)首先,用户指示关于从中提取感兴趣的一组基因的基因组的信息。这包括选择感兴趣的生物体,基于所述选择,可将基因组序列鉴定用作got_gene程序中的输入(例如,作为主题输入)。用户还可选择用于比较分析的查询序列列表。[0745](2)特征和序列文件自动从ncbi下载。这包括收集输入(例如,主题输入),例如通过从诸如ncbi的可公开访问的数据库下载相关序列,包括任选地与序列注释信息一起的序列;[0746](3)序列(例如,每个查询序列与每个主题序列)的成对blast比较提供确立跨所有基因组序列的每个感兴趣的基因的序列多样性水平的数据;[0747](4)将表示序列多样性信息(例如,序列保守性)的数据编译在例如生成的got表中。got表包括关于每个基因组中每个基因的存在或不存在、多样性水平、变异性质和基因组坐标的信息;以及[0748](5)got表用于生成表示编译的序列多样性信息的显示(例如,表、热图和/或图形)。生成的显示可为或包括序列多样性的图形、最大似然系统发育和/或比对文件。然后从所有基因组中提取基因序列并翻译,以创建核苷酸和氨基酸比对。将每个步骤保存到fasta文件中。最后,使用phyml程序创建基于基因组的系统发育和基于基因的系统发育,并保存到单独的文件中。[0749]这些步骤不旨在并且不限制、避免或要求在本公开的方法或系统中包括本文提供的任何步骤或一系列步骤。[0750]如图1中所提供,本发明的方法和系统可包括由用户手动提供的或从序列数据库获取的主题序列输入(以及特征信息,诸如gff、gbk、gtf),并且可包括由用户手动提供的或例如从从头测序数据(例如,illumina或其他高通量测序读段)组装的查询序列输入。比对查询序列和主题序列,每个查询序列针对每个主题序列。所得的数据用于生成got表。got表可用于生成信息显示,包括图形(graphics)(图形(graph)、热图)、序列比对、经翻译的序列比对和系统发育显示(包括基于基因组的系统发育和/或基于基因的系统发育)。可出于用户指定的目的、例如通过鉴定以下中的任何一个或多个或全部来选择基因或氨基酸序列:(i)最保守的基因;(ii)最不保守的基因(即,最多样或最可变的基因);(iii)毒力因子;(iv)抗生素抗性;(v)人序列同源性;(vi)分泌的蛋白质和/或包括分泌结构域的蛋白质;和(vii)跨膜或表面蛋白质,和/或包括跨膜或表面结构域的蛋白质。[0751]方法或系统的第一步骤可为确定要从一个或多个可公开访问的数据库(例如,ncbi)获取(例如,下载)(如果可用的话,连同注释信息)的主题序列的特征,并且确定是否将手动提供一个或多个查询序列用于与主题序列进行比较(图2)。got_gene程序可自动生成某些文件夹用于组织和/或存储数据,这些文件夹如图3所示。[0752]方法或系统的第二步骤可为从一个或多个可公开访问的数据库中获取主题序列和注释信息,其可被复制并存储在几个got_gene文件夹(参考序列、比对器数据库和注释文件夹)中(图4)。用于从一个或多个可公开访问的数据库获取序列和注释信息的步骤提供于图5中。r包reutils用于打开与ncbi数据库的服务器的通道。reutils是ncbientrez编程实用程序的接口,并且为与ncbi数据库(诸如pubmed、genbank或geo)交互的系统提供支持,所述编程接口的每个功能称为r功能。[0753]方法或系统的第三步骤可为手动提供查询序列或从可公开访问的数据库下载查询序列(图6)。[0754]方法或系统的第四步骤可为将查询序列与比对器数据库文件夹中的序列(即,主题序列)进行比对(图7)。使用blast的比对的步骤提供于图8中。例如,用于序列比较的blast参数可包括outfmt‘7stdsgistitle’;最小e值=约0.001;打开空位的成本=约5;延伸空位的成本=约2;最佳完美匹配的长度=约11;核苷酸匹配的加分=约2;核苷酸错配的加分=-约3(图8)。[0755]方法或系统的第五步骤可包括创建got表。got表可包括成对序列比较的blast结果、所分析序列的序列和可用注释(图9)。丢弃无结果的blast输出(即在特定的比较对之间未鉴定到匹配),包括无匹配的重叠群。也丢弃e值大于约0.001、同一性百分比低于约79%或覆盖长度小于约50个核苷酸的blast结果(图10)。未丢弃的成对序列比较称为匹配。在查询包括重叠群并且多个查询重叠群以重叠方式匹配特定参考序列的情况下,可能必需策划包括哪个重叠群用于分析(图11)。用于选择保留哪个查询重叠群作为参考序列的成对匹配的标准可包括图11(18)中提供的那些标准。在生成got表时,如果重叠的重叠群覆盖的基因百分比大于约95%,则可认为查询存在于参考序列中;如果重叠的重叠群覆盖的基因百分比大于约80%,则可认为查询部分存在于参考中;或者如果重叠的重叠群覆盖的基因百分比小于约79%或小于约80%,则可认为查询不存在参考序列(图12)。也可使用其他阈值。对于每个剩余的匹配,可计算snp/大小比(匹配中的突变数量与所述匹配的长度之间的比率)(图12)。选择覆盖参考序列的整个长度的单个重叠群,并且如果查询序列的多个此类重叠群相对于参考序列存在,则保留相对于参考具有最少突变的重叠群(图12)。在没有匹配的重叠群覆盖参考序列的整个长度的情况下,保留snp/大小比小于约0.5的所有重叠群(图12)。got表还可并入注释信息(图12)。got表可包括与参数相关的信息,包括图13所示的那些。对于每个查询序列生成一个got表(图13)。[0756]got表可用于生成各种信息分析并显示为输出。一个所述输出是比较表。为了生成比较表,将关于在got表中找到的每个查询序列与所有参考序列相比的序列相似性的信息转换成相似性评分(图15)。基于查询和主题之间的比对的覆盖率百分比以及查询和主题之间的突变数量来分配相似性评分。可例如根据表2来分配相似性评分(也参见图14)。可以矩阵编译相似度评分,所述矩阵是对比表(图14)。在比较表中发现的相似性数字也可呈现为热图,显示相关查询序列和每个主题序列之间的保守性(图15)。[0757]可基于got表中匹配的坐标和相关注释在查询核苷酸序列中鉴定编码序列。可提取和翻译经鉴定的编码序列(图16)。可比对经翻译的序列并保存在用于提取序列的got_gene文件夹中(图16)。在多个查询重叠群与参考编码序列匹配的情况下,重叠的重叠群合并成单个匹配序列。延伸超出参考编码序列的边界的查询重叠群可能需要策划(图16)。可将每个变体主题编码序列翻译的数量和频率制成表(图16)。也可例如使用quicktree对提取的序列进行系统发育分析(图17)。可使用参考核苷酸序列生成个别基因的基于参考的系统发育(图17)。可基于所有查询序列中最保守的主题序列生成个别基因组的基于基因组的系统发育,例如,其中主题序列一起包括不超过约40,000个核苷酸(图17)。[0758]本实施例展现本实施例的方法和系统可用于多种治疗相关的应用。这些尤其可包括:(1)确定抗原/表位的遗传保守性,以预测靶向抗体的临床潜力;(2)通过质谱法鉴定用于肽发现的氨基酸序列变体;(3)提取序列并创建比对以突出基因/抗原内的多样性区域;(4)鉴定基因组内的多样性/保护性的区域;(5)将基因组内未表征的感兴趣序列鉴定为潜在的治疗或疫苗靶标;(6)建立系统发育以鉴定引起流行病的病原体的基因型;(7)从错误注释的基因组中检索出一套直系同源基因;并且/或(8)出于流行病学目的区分株系中的关联性。[0759]实施例2:用于鉴定乙型肝炎病毒的新治疗性抗原的方法和系统的使用[0760]在本实施例中,根据本文所述的方法和系统,使用got_gene程序鉴定hcc肿瘤上mhc-1上存在的新乙型肝炎病毒肽。乙型肝炎病毒(hbv)是全球性健康问题并且是肝细胞癌(hcc)的主要原因(图21)。发生慢性感染的人通常用核苷类似物治疗以抑制病毒复制,但仍处于hcc的加强风险中。免疫系统无法清除感染的一个主要促成因素是,患有慢性hbv的患者的hbv特异性t细胞数量减少,并且那些患者中的许多保持显示衰竭表型。[0761]在肿瘤学领域,t细胞重定向抗体是通过利用那些细胞表面上的肿瘤特异性抗原靶向和杀死肿瘤细胞的常见方法。不幸的是,不存在在感染/肿瘤细胞的表面上表达的hbv蛋白。然而,与mhc-i复合的hbv肽呈递在细胞表面上。某些先前努力无法鉴定与mhc-i复合的临床有用的hbv肽呈递在细胞表面上。例如,分析来自hbv 患者的hcc肿瘤样品时,最初通过质谱法仅鉴定出细胞表面上呈递的少数hbv肽。这种失败至少部分是由于关于此类肽的预计序列的限制性假设。质谱法方案使用预先确立的一组来源于参考基因组的氨基酸序列来捕获实验设置中肽的存在。质谱法对肽序列变异高度敏感,并且用于鉴定所述肽的呈递的肽与参考序列之间的单个氨基酸变化会对信号检测产生显著影响。因此,确立一套用于质谱法分析的正确参考序列至关重要。[0762]进行本实施例中所述的工作以鉴定与mhc-i复合的hbv肽作为新的候选hbv抗原呈递在细胞表面上,用于治疗性抗体开发,例如,用于开发抗hbvpig/cd3双特异性抗体以驱动针对肿瘤/感染细胞的t细胞响应。[0763]hbv具有约3.1kb的环状基因组,其包括编码约4种多肽的约7个重叠编码序列(图22)。主要乙型肝炎表面抗原(hbsag)蛋白由基因s编码(图23)。hbsag是hbv的表面抗原,并且已知其指示当前的乙型肝炎感染。在世界各地发现了各种hbv基因组,并且已公布至少约7,108个hbv基因组序列(图24)。通过got_gene对hbv基因组的分析展现,该程序能够分析具有多种特征的序列,包括环状序列、线性序列、片段化序列、dna序列、rna序列、数据库序列和手动提供的序列(图25)。[0764]在本实施例中,对几个hbv样品进行rnaseq。序列读段用于构建每个样品的从头基因组病毒序列。从ncbi下载额外hbv基因组(例如,参见图18)。got_gene用于从所有hbv基因组中提取编码序列(图26)。通过blast成对比较所有查询hbv基因组和参考hbv基因组的编码序列(图27)。制备包括所得序列比较数据的汇总表(图28)。序列保守性以图形(图29)、热图(图30)和系统发育(参见图31和图32中的示例性系统发育显示)来显示。将提取的编码序列(例如,参见图34)翻译成氨基酸序列(例如,参见图35)并比对氨基酸序列(例如,参见图36)。分析比对的氨基酸序列的保存性(图36)。[0765]将本实施例中鉴定的氨基酸序列添加到上述质谱法分析方案中,从而能够检测先前意外的hbv肽。利用更新的参数相应地重新分析质谱法结果。这些分析导致发现了呈递在感染细胞表面上的新肽。这些肽特别令人感兴趣,这是因为它们显示与i类人hla结合的混杂性,进一步支持了它们是有希望的治疗开发靶标。[0766]got_gene还用于表征约7,000个hbv基因组中强效hbv抗原的多样性水平,以鉴定高度保守的表位区域。[0767]实施例3:方法和系统的用于确定样品基因组和参考基因组集合之间的相似性的用途[0768]出于历史原因以及与效率和一致性相关的原因,实验室或研究团体通常会使用一种或几种感兴趣的特定生物体株系进行实验。这些实验室株系通常被视为代表非实验室形式(例如,同一生物体的天然或野生实例)。然而,这种典型的方法有某些固有的缺点。具体来说,由于特定生物体的现实世界多样性远远大于(例如)给定实验中测试的实验室样品所代表的多样性,所以实验室结果不一定适用于相关生物体多样性的整个范围。为了提供来自临床背景的实例,病原体的特定株系可用于实验室实验,但临床分离株代表实验室株系可充分代表或不可充分代表的更大多样性的序列。[0769]本公开的方法和系统可用于确定所提供的序列(例如,实验室株系的基因组序列)是否由在非实验室形式中保守(或不保守)的序列表征。因此,例如,本公开的方法和系统可应用于基于所测量的序列保守性来确定实验室病原体株系是否代表病原体的临床分离株。在一种或几种实验室测试株系用于旨在代表更广泛的株系群体的实验中的情况下(例如,在一种或几种病原体株系可用于实验室、但在临床应用中可遇到许多不同株系的情况下),这种使用特别有价值。在所述情况下,重要的是实验室或测试株系代表参考基因组的集合(例如临床相关基因组的集合)。[0770]在本实施例中,got_gene用于确定样品基因组和参考基因组集合的相似性。更特定来说,got_gene用于确立金黄色葡萄球菌的一个特定实验室株系代表在社区中引起疾病的循环株系。got_gene应用基于基因组的系统发育,以出于流行病学目的容易地区分株系之间的关联性。相同方法成功地应用于确定绿脓假单胞菌和流感病毒的实验室株系是否临床上相关。[0771]实施例4:方法和系统的用于评价sars-cov-2受体结合结构域的保守性的用途[0772]2019年冠状病毒病(covid-19)全球疫情激发了广泛的努力以了解其病原性因子严重急性呼吸综合征冠状病毒2(sars-cov-2)的适应机制。因此,世界各地的科学家和医学专业人士已对来自患者分离株的sars-cov-2基因组进行了测序,并通过策划的数据存储库(诸如全球共享流感数据倡议(globalinitiativeonsharingallinfluenzadata,gisaid.https://www.gisaid.org))以前所未有的速度传播了他们的发现。这提供了可用于确定传播模式和鉴定可与毒力和疾病严重程度相关的sars-cov-2变体的独特的数据集。[0773]sars-cov-2的结构的示意图提供于图47中。它包括四种结构蛋白:核衣壳(n)蛋白、膜(m)蛋白、刺突(s)蛋白和包膜(e)蛋白以及几种非结构蛋白(nsp)。衣壳是病毒的蛋白外壳。在衣壳内,有核衣壳与病毒的病毒单阳性链rna基因组结合。冠状病毒基因组包括约30,000个核苷酸。使用计算技术和/或分子生物学技术,可容易地将呈rna形式的基因组序列转换或翻译成dna形式。[0774]为了确立复制生态位和对抗先天和适应性免疫响应,sars-cov-2必须适应宿主环境。一种常见的适应机制是抗原变异,其中抗体识别的病毒靶标产生逃逸突变,所述逃逸突变使病毒逃逸识别和消除。抗原变异的后果可包括持续性病毒感染、疾病大流行和恢复后的再感染。在covid-19治疗发展的背景下,抗原变异也影响治疗功效,这是因为突发突变可通过修饰其靶标的蛋白结构来混淆基于抗体的治疗的功效。[0775]病毒刺突蛋白(s)的sars-cov-2受体结合结构域(rbd)是covid-19患者血清或血浆样品中强效中和抗s抗体的重要靶标。因此,s是开发用于治疗covid-19的抗体的重要靶标。rbd的遗传保守性对于确保基于抗体的治疗成功(至少在包括抗s抗体的治疗方面)至关重要。在此背景下,got_gene用于评价rbd的遗传多样性。[0776]自2020年1月初报道第一个sars-cov-2基因组序列以来,截至2020年10月,已有约120,000个序列寄存到gisaid(https://www.gisaid.org/)。在本实施例中,使用got_gene算法来提取、过滤和比较从总共118,728个策划的基因组序列中检索到的刺突编码基因序列的同一性。在本实施例中,使用基因库文件注释(部分在图49的示意图中说明)从参考sars-cov-2基因组提取编码序列。使用用于序列的比对的blastn,在每个策划的基因组序列和刺突蛋白参考序列之间进行成对比较。图50中图示了所分析的查询序列的累积数量。在比对后,从策划的基因组序列中提取与刺突蛋白参考序列比对的编码序列。然后基于表2所示的覆盖长度和突变数量对与刺突蛋白参考序列比对的基因组序列进行归类。从进一步分析中去除来自与刺突蛋白参考序列比较的分配相似性评分小于0.8的序列。将分析中剩余的与刺突蛋白参考序列比对的序列翻译成氨基酸序列,并使用blastp对氨基酸序列进行比对(部分在图51的示意图中说明)。这个分析允许鉴定刺突蛋白的每个比对位置存在的氨基酸的范围(部分在图52的示意图中说明)。[0777]结果鉴定了sars-cov-2刺突蛋白中的965个可变氨基酸位置和总数为1782个独特的氨基酸变化。正如预计,在118,728个基因组中,大多数变体仅在一个给定的基因组(单例)中鉴定到。然而,鉴定到超过100种株系(高频变体或hfv)间共有的47种氨基酸变化。发现在刺突蛋白内鉴定的hfv在n-末端和s2结构域内积累。除了在与人ace2受体直接相互作用的受体结合基序内鉴定的两个hfv(n439k和s477n)之外,rbd没有hfv。总之,s蛋白显示相对较小的序列多样性。在本研究使用的118,728个株系中,仅观察到7个变体(l5f、l18f、r21i、a222v、s477n、d614g和d936y),频率大于0.6%。[0778]本实施例的一个重要发现是sars-cov-2表位保守性在这种高度成功的人病原体中是规则而不是例外的强有力证据。sars-cov-2rbd是covid-19患者血清或血浆样品中强效中和抗s抗体的主要靶标。因此,治疗性抗体施加的大部分选择性压力应靶向这个结构域。rbd保守性的仔细检查指示,几乎没有证据表明在所有sars-cov-2株系的》0.15%中繁殖的突变积累。虽然已在循环sars-cov-2分离株中鉴定出几种rbd变体,但如本研究中所测量,在病毒群体中,无一达到显著的频率。总之,这些数据表明rbd靶向抗体表位在循环sars-cov-2中的保守性;因此,基于s的治疗应该对所有循环sars-cov-2病毒有效是理所当然的。[0779]实施例5:方法和系统的用于评价表位变异的用途[0780]2019年末sars-cov-2的出现以及其随后对人类健康的有害影响导致数百万感染以及显著发病率和死亡率。在终止covid-19疫情的努力中,瑞泽恩制药公司(regeneronpharmaceuticals)已应用其最先进的技术来开发专用于对抗sars-cov-2病毒的单克隆抗体的混合物(例如,参见美国专利第10,787,501号,所述专利全文通过引用并且特别是关于covid-19治疗性抗体以及它们的表位和其他性质并入本文。美国专利第10,787,501号的表1提供了示例性抗sars-cov-2-刺突蛋白(sars-cov-2-s)抗体序列,其全文通过引用具体并入本文。)。瑞泽恩开始生产数百种病毒中和抗体,并从人covid-19幸存者中鉴定出表现相似的抗体。这些抗体特异性鉴定来自刺突蛋白的受体结合结构域(rbd)的表位。[0781]靶向相同抗原(例如,sars-cov-2刺突蛋白)的个别抗体可在抗原内具有不同的结构靶标(表位),且至少出于所述原因可具有不同特征,例如在个体受试者中和/或跨受试者群体的不同临床表现。根据至少一种方法,结合抗原的更保守表位的抗体优于结合抗原的更不保守表位的抗体,使得在任何给定株系或患者中或跨患者群体,抗体更可能有效地结合靶抗原和/或具有治疗效应。当可获得多种不同的抗体且可获得关于它们的不同表位的信息时,可使用序列分析来确定哪些抗体有利地结合更保守的表位。本实施例将该推理应用于用于治疗covid-19的抗体的开发。本公开的方法和系统用于评价多种抗体的sars-cov-2表位在数千个循环sars-cov-2株系中的保守性,其中靶向更保守的表位的抗体被选择或优选用于进一步的治疗评价。[0782]使用got_gene算法对数千个基因组中的表位遗传序列进行比较分析,所述got_gene算法允许将每个基因组序列针对独特的参考基因组进行快速成对比较。从全球共享流感数据倡议(gisaid)数据库中提取了超过120,000个sars-cov-2策划的基因组序列。[0783]在got_gene程序内使用blastn将来自gisaid的sars-cov-2核苷酸序列与sars-cov-2参考基因组核苷酸序列(基因库登录号:mn908947)进行比对。对每个策划的基因组序列和sars-cov-2参考基因组序列进行成对比较。比对后,评价与参考sars-cov-2基因组的刺突核酸序列比对的基因组序列,以验证刺突核酸序列的存在。got_gene根据某些阈值,基于关于刺突蛋白存在、缺乏完整性或不存在的确定,创建基因组的组类别。对于每个序列,如果与参考序列的比较产生大于95%的覆盖率百分比,则将刺突蛋白鉴定为存在;如果与参考的比较产生大于70%但小于95%的覆盖率百分比,则将刺突蛋白鉴定为部分存在或缺乏完整性;或如果与参考的比较产生低于70%的覆盖率百分比,则将刺突蛋白鉴定为不存在。如果与刺突蛋白参考序列的比较产生》95%的覆盖长度和》70%的同一性百分比,则验证刺突序列的存在。保留根据该阈值验证的序列用于进一步分析,并去除所有其他序列。got_gene从每个策划的基因组序列中提取刺突蛋白编码序列,并将每个策划的基因组序列的经验证的直系同源刺突序列翻译成氨基酸序列。然后使用blastp对氨基酸序列进行比对,并鉴定氨基酸变体。实施表位位置,并计算每个表位的变体的频率。[0784]实施例6:方法和系统的用于评价经治疗的受试者中推定的逃逸变体的选择的用途[0785]本实施例展现本公开的方法和系统的评估刺激对序列多样性的影响,特别是病毒疗法对病毒序列多样性的影响的用途。本实施例具体展现本公开的方法和系统的评估基于抗体的covid-19疗法对治疗接受者中的sars-cov-2序列多样性的影响的用途。[0786]两种强效瑞泽恩抗体(regn10933和regn10987)形成瑞泽恩的regn-cov2抗体疗法(也参见美国专利第10,787,501号,所述专利全文通过引用并且特别是关于covid-19治疗性抗体及它们的表位和其他性质并入本文。美国专利第10,787,501号的表1提供了示例性抗sars-cov-2-刺突蛋白(sars-cov-2-s)抗体序列,其全文通过引用具体并入。)。在9月,瑞泽恩公布了早期临床数据,所述临床数据显示regn-cov2抗体混合物对275例非住院covid-19患者中的病毒基因组序列的效应。本研究的一个目标是评估在治疗性施用regn-cov2治疗后来自患者的sars-cov-2分离株的推定的逃逸变体(对病毒有益的突变,因为它们使病毒逃避抗体识别)的选择。[0787]在本实施例中,对从接受regn-cov2治疗的患者中分离的病毒基因组进行测序,并使用got_gene程序鉴定分离的基因组中的新突变。使用用于序列的比对的blastn,在每个分离的基因组序列和编码刺突蛋白的参考序列之间进行成对比较。比对后,从策划的基因组序列中提取与编码刺突蛋白的参考序列比对的序列作为查询编码序列。然后基于表2所示的覆盖长度和突变数量对与刺突蛋白参考序列比对的基因组序列进行归类。从进一步分析中去除来自与刺突蛋白参考序列比较的分配相似性评分小于0.8的序列。将分析中剩余的与刺突蛋白参考序列比对的序列翻译成氨基酸序列,并使用blastp对氨基酸序列进行比对。这种分析允许鉴定刺突蛋白的每个比对位置处存在的氨基酸的范围。因此,使用got_gene从所有基因组中提取并翻译刺突编码基因序列,并将它们与参考序列进行比较,以鉴定其中新突变导致由中和抗体识别的区域中氨基酸变化的基因组。表位序列突变可为推定的逃逸变体。最终,分析评估了治疗是否会导致所有患者样品中的sars-cov-2s蛋白出现突变。[0788]实施例7:方法和系统在个性化医疗中的用途[0789]本实施例说明本公开的方法和系统可用于选择可能对感兴趣的治疗性治疗有利地响应的受试者。具体来说,本实施例公开了分析来自感染患者的病毒序列以确定患者是否可能受益于施用用于治疗病毒感染的抗体疗法。例如,got_gene程序可用于鉴定未治疗的患者中的推定的逃逸变体。got_gene程序也可用于鉴定具有推定的逃逸潜力的新突变。在这种情况下,如实施例6中所示,got_gene用于从自未治疗的患者分离的基因组中提取并翻译刺突编码基因序列,以鉴定与刺突蛋白参考序列相比的刺突蛋白突变。可将鉴定的刺突蛋白突变与已知或预计会负面地影响治疗功效的有害变体的预先确立的列表进行比较。该分析允许got_gene基于感染病毒株的遗传背景将患者分类成组(治疗易感组对比治疗抗性组)。[0790]其他实施方案[0791]虽然我们已经描述了许多实施方案,但是显而易见的是,我们的基本公开和实施例可提供利用本文所述的组合物和方法或由本文所述的组合物和方法涵盖的其他实施方案。因此,应理解,本发明的范围由可从本公开和所附权利要求中理解的内容来限定,而不是由已经通过实施例表示的具体实施方案来限定。[0792]本文引用的所有参考文献均以引用的方式并入本文中。当前第1页12当前第1页12
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。