1.本技术涉及生物医药领域,具体的涉及一组肿瘤检测标志物及其用途。
背景技术:
2.寻找更高效的肿瘤标志物一直是肿瘤相关研究中重要的一个方向,基于液体活检的血浆游离dna检测技术对于肿瘤标志物的发现有了更高的需求。其基本原理为:肿瘤细胞死亡后,释放游离dna进入血液,通过检测血液中的dna突变、dna甲基化、mirna、组蛋白修饰等肿瘤细胞相关信息,可探测到极早期的肿瘤信号。已有研究发现dna甲基化变化可能早于dna突变的发生,是肿瘤早期筛查的更行之有效的检测标志物。
3.但目前早期癌症中,释放入血的肿瘤细胞极少,并且本领域急需寻找到更多的对于肝肿瘤有效的检测标志物。因此,亟需开发一种方法和/或试剂盒,通过检测准确、稳定、有效的肝癌生物标志物或其组合,且可以从生物样品中数量极为有限的细胞外游离dna高效地读取到该表观遗传学信息,更优的,该方法应可以在医院检验科里很容易地配置并可靠地应用。
技术实现要素:
4.本技术提供的方法可以将更多高效的甲基化标志物,用于确认或辅助确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展,可以提高肿瘤,例如肝肿瘤的早筛早诊效率,解决肝癌早诊率低、临床治疗负担重等问题。
5.一方面,本技术提供了一种确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的方法,包含确定待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量,所述目标基因包含sept9和ikzf1。
6.另一方面,本技术还提供了一种评估肝肿瘤相关dna区域甲基化状态的方法,包含确定待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量,所述目标基因包含sept9和ikzf1。
7.另一方面,本技术还提供了一种确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的方法,包含确定待测样本中目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量,所述目标dna区域包含来源于人chr17:75368651-75370720和来源于人chr7:50343720-50344547定义的区域。
8.另一方面,本技术还提供了一种确定dna区域甲基化状态的方法,包含确定待测样本中目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量,所述目标dna区域包含来源于人chr17:75368651-75370720和来源于人chr7:50343720-50344547定义的区域。
9.另一方面,本技术还提供了一种核酸,所述核酸包含能够结合如本技术的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列;或者所述核酸包含能够结合如本技术的方法中所述目标dna区域、或其互补区域、或上
述的转化而来的区域、或上述的片段的序列。
10.另一方面,本技术还提供了一种制备核酸的方法,所述方法包含根据如本技术的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸;或者所述方法包含根据如本技术的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
11.另一方面,本技术还提供了一种核酸组,所述核酸组包含能够结合如本技术的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列;或者所述核酸组包含能够结合如本技术的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列。
12.另一方面,本技术还提供了一种制备核酸组的方法,所述方法包含根据如本技术的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸组;或者所述方法包含根据如本技术的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸组。
13.另一方面,本技术还提供了一种试剂盒,包含如本技术的核酸和/或本技术的核酸组。
14.另一方面,本技术还提供了如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,在制备确定dna区域或其片段的修饰状态的物质中的应用。
15.另一方面,本技术还提供了如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,在制备疾病检测产品中的应用。
16.另一方面,本技术还提供了如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,在制备确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的物质中的应用。
17.另一方面,本技术还提供了用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒,在制备用于确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的物质中的应用,所述用于确定的dna区域包含如本技术的方法中所述目标基因所在dna区域或其片段。
18.另一方面,本技术还提供了用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒,在制备用于确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的物质中的应用,所述用于确定的dna区域包含如本技术的方法中所述目标dna区域、或其互补区域、或上述的片段。
19.另一方面,本技术还提供了如本技术的方法中所述目标基因所在dna区域、或其转化而来的区域、或上述的片段的核酸或其组合,在制备用于确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的物质中的应用。
20.另一方面,本技术还提供了如本技术的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸或其组合,在制备用于确认疾病的存在、评
估疾病形成或形成风险和/或评估疾病的进展的物质中的应用。
21.另一方面,本技术还提供了一种储存介质,其记载可以运行本技术的方法的程序。
22.另一方面,本技术还提供了一种设备,其包含本技术的储存介质。
23.本领域技术人员能够从下文的详细描述中容易地洞察到本技术的其它方面和优势。下文的详细描述中仅显示和描述了本技术的示例性实施方式。如本领域技术人员将认识到的,本技术的内容使得本领域技术人员能够对所公开的具体实施方式进行改动而不脱离本技术所涉及发明的精神和范围。相应地,本技术的说明书中的描述仅仅是示例性的,而非为限制性的。
具体实施方式
24.以下由特定的具体实施例说明本技术发明的实施方式,熟悉此技术的人士可由本说明书所公开的内容容易地了解本技术发明的其他优点及效果。
25.术语定义
26.在本技术中,术语“sept9”通常是指一种基因或其表达产物。例如,sept9
27.(septin9)蛋白的uniprot登录号可以是q9uhd8。本技术中,sept9可以涵盖其未加工形式、任何的加工形式、其变体或包含其功能活性片段的物质。例如,本技术中sept9也可以表示检测位点,例如针对该基因特定核酸序列进行甲基化检测。例如,以sept9表示的检测位点可以是该基因下的一个或多个区域的核酸序列。
28.在本技术中,术语“ikzf1”通常是指一种基因或其表达产物。例如,ikzf1(dna-binding protein ikaros)蛋白的uniprot登录号可以是q13422。本技术中,ikzf1可以涵盖其未加工形式、任何的加工形式、其变体或包含其功能活性片段的物质。例如,本技术中ikzf1也可以表示检测位点,例如针对该基因特定核酸序列进行甲基化检测。例如,以ikzf1表示的检测位点可以是该基因下的一个或多个区域的核酸序列。
29.在本技术中,术语“best4”通常是指一种基因或其表达产物。例如,best4(bestrophin-4)蛋白的uniprot登录号可以是q8nfu0。本技术中,best4可以涵盖其未加工形式、任何的加工形式、其变体或包含其功能活性片段的物质。例如,本技术中best4也可以表示检测位点,例如针对该基因特定核酸序列进行甲基化检测。例如,以best4表示的检测位点可以是该基因下的一个或多个区域的核酸序列。
30.在本技术中,术语“b4galnt1”通常是指一种基因或其表达产物。例如,b4galnt1(beta-1,4n-acetylgalactosaminyltransferase 1)蛋白的uniprot登录号可以是q00973。本技术中,b4galnt1可以涵盖其未加工形式、任何的加工形式、其变体或包含其功能活性片段的物质。例如,本技术中b4galnt1也可以表示检测位点,例如针对该基因特定核酸序列进行甲基化检测。例如,以b4galnt1表示的检测位点可以是该基因下的一个或多个区域的核酸序列。
31.在本技术中,术语“grasp”通常是指一种基因或其表达产物。例如,grasp(grp1(general receptor for phosphoinositides 1)-associated scaffold protein)蛋白的uniprot登录号可以是k7chn5。本技术中,grasp可以涵盖其未加工形式、任何的加工形式、其变体或包含其功能活性片段的物质。例如,本技术中grasp也可以表示检测位点,例如针对该基因特定核酸序列进行甲基化检测。例如,以grasp表示的检测位点可以是该基因下的
一个或多个区域的核酸序列。
32.在本技术中,术语“irf4”通常是指一种基因或其表达产物。例如,irf4(interferon regulatory factor 4)蛋白的uniprot登录号可以是q15306。本技术中,irf4可以涵盖其未加工形式、任何的加工形式、其变体或包含其功能活性片段的物质。例如,本技术中irf4也可以表示检测位点,例如针对该基因特定核酸序列进行甲基化检测。例如,以irf4表示的检测位点可以是该基因下的一个或多个区域的核酸序列。
33.在本技术中,术语“bend4”通常是指一种基因或其表达产物。例如,bend4(ben domain-containing protein 4)蛋白的uniprot登录号可以是q6zu67。本技术中,bend4可以涵盖其未加工形式、任何的加工形式、其变体或包含其功能活性片段的物质。例如,本技术中bend4也可以表示检测位点,例如针对该基因特定核酸序列进行甲基化检测。例如,以bend4表示的检测位点可以是该基因下的一个或多个区域的核酸序列。
34.在本技术中,术语“待测样本”通常是指需要进行检测的样本。例如,可以检测待测样本上的一个或者多个基因区域是否存在有修饰状态。
35.在本技术中,术语“无细胞游离核酸”或“cfdna”通常是指样品中的dna,当采集时,该dna没有包含在细胞内。例如,无细胞游离核酸可以不是指通过细胞或组织的体外破裂而使其不在细胞内的dna。例如,cfdna可以包括正常细胞和源自癌细胞的dna两者。例如,cfdna可以获自血液或血浆(“循环系统”)。例如,cfdna可以通过分泌或细胞死亡过程,如细胞坏死或凋亡释放到循环系统中。
36.在本技术中,术语“互补核酸”通常是指与参考核苷酸序列相比具有互补的核苷酸序列。例如,互补核酸可以为任选地具有相反方向的核酸分子。例如,所述互补可以是指具有下面的互补性关联:鸟嘌呤和胞嘧啶;腺嘌呤和胸腺嘧啶;腺嘌呤和尿嘧啶。
37.在本技术中,术语“dna区域”通常是指两个或更多个共价键合的天然存在的或经修饰的脱氧核糖核苷酸的序列。例如,基因的dna区域可以是指该基因所位于的特定的脱氧核糖核苷酸的序列的位置,例如该脱氧核糖核苷酸的序列编码所述基因。例如,本技术的dna区域包含dna区域的全长、其互补区域,或者上述的片段。例如,本技术所提供的检测区域的上下游至少约20kb的序列可以作为检测的位点。例如,本技术所提供的区域的上下游至少约20kb、至少约15kb、至少约10kb、至少约5kb、至少约3kb、至少约2kb、至少约1kb、或至少约0.5kb的序列可以作为检测的位点。例如,可以根据所述微电脑设计合适的引物和探针进行样品的甲基化检测。
38.在本技术中,术语“修饰状态”通常是指本技术中基因片段、核苷酸或其碱基具有的修饰状态。例如,本技术中的修饰状态可以是指胞嘧啶的修饰状态。例如,本技术的具有修饰状态的基因片段可以具有改变的基因表达活性。例如,本技术的修饰状态可以是指碱基具有的甲基化修饰。例如,本技术的修饰状态可以是指在基因组dna的cpg区域的胞嘧啶5
′
碳位共价结合一个甲基基团,例如可以成为5-甲基胞嘧啶(5mc)。例如,修饰状态可以是指dna序列内存在或不存在5-甲基胞嘧啶(“5-mcyt”)。
39.在本技术中,术语“甲基化”通常是指本技术中基因片段、核苷酸或其碱基具有的甲基化状态。例如,本技术中基因所在的dna片段可以在一条链或多条链上具有甲基化。例如,本技术中基因所在的dna片段可以在一个位点或多个位点上具有甲基化。
40.在本技术中,术语“转化”通常是指将一种或多种结构转变为另一种结构。例如,本
申请的转化可以是具有特异性。例如,不具有甲基化修饰的胞嘧啶经过转化可以变为其它结构(例如尿嘧啶),且具有甲基化修饰的胞嘧啶经过转化可以基本不发生变化。例如,不具有甲基化修饰的胞嘧啶经过转化可以被剪切,且具有甲基化修饰的胞嘧啶经过转化可以基本不发生变化。
41.在本技术中,术语“脱氨基试剂”通常是指具有移除氨基能力的物质。例如,脱氨基试剂可以将未修饰的胞嘧啶的氨基脱除。
42.在本技术中,术语“亚硫酸氢盐”通常是指一种可以区分具有修饰状态和不具有修饰状态的dna区域的试剂。例如,亚硫酸氢盐可以包括亚硫酸氢盐、或其类似物或上述的组合。例如,亚硫酸氢盐可以使未修饰的胞嘧啶的氨基脱氨基化,以使其与修饰的胞嘧啶区分。在本技术中,术语“类似物”通常是指具有类似结构和/或功能的物质。例如亚硫酸氢盐的类似物可以与亚硫酸氢盐具有类似的结构。例如,亚硫酸氢盐的类似物可以是指一种同样可以区分具有修饰状态和不具有修饰状态的dna区域的试剂。
43.在本技术中,术语“甲基化敏感限制酶”通常是指一种根据其识别位点的甲基化状态而选择性消化核酸的酶。例如,对于当识别位点未被甲基化时才特异剪切的限制酶来说,当识别位点被甲基化时,可以不会发生剪切,或以显著降低的效率剪切。对于当识别位点被甲基化时才特异剪切的限制酶来说,当识别位点未被甲基化时,可以不会发生剪切,或以显著降低的效率剪切。例如,甲基化特异的限制酶可以识别含有cg二核苷酸(例如cgcg或cccggg)的序列。
44.在本技术中,术语“肿瘤”通常是指在正常生长和/或发育中呈现出至少部分失去控制的细胞和/或组织。例如,常见的肿瘤或癌细胞通常可以是失去了接触抑制并可能是入侵性的和/或具有转移的能力。例如,本技术的肿瘤可以是良性的,也可能是恶性的。
45.在本技术中,术语“进展”通常是指疾病从不太严重状态到较严重状态的变化。例如,肿瘤进展可以包括肿瘤的数量或严重性、癌细胞转移程度、癌症生长或扩散的速度等增大。例如,肿瘤进展可以包括这种癌症从不太严重状态到较严重状态的阶段时期,例如从i期到ii期、从ii期到iii期等的进展。
46.在本技术中,术语“形成”通常是指个体体内出现病灶。例如,当肿瘤形成时,可以将该个体确诊为肿瘤患者。
47.在本技术中,术语“荧光pcr”通常是指一种定量或半定量的pcr技术。例如,可以是实时定量聚合酶链反应、定量聚合酶链反应或动力学聚合酶链反应的pcr技术。例如,可以利用pcr扩增并借助嵌入性荧光染料或序列特异性探针定量检测起始的靶核酸量,所述序列特异性探针可以含有仅与靶核酸杂交才可检出的荧光报道分子。
48.在本技术中,术语“pcr扩增”通常是指聚合酶链扩增反应。例如,本技术中的pcr扩增可以包含目前已知的用于dna扩增的任意聚合酶链扩增反应。
49.在本技术中,术语“荧光ct值”通常是指一种定量或半定量评估靶核酸的测量值。例如,可以是指荧光信号到达设定的域值时所经历的扩增反应循环数。
50.发明详述
51.本技术可以找到多种高效的肝脏肿瘤相关甲基化标志物,提高肝肿瘤早筛早诊效率,解决肝癌早诊率低,临床治疗负担重等问题。同时本技术提供多组高效的肝脏肿瘤甲基化标志物,并列举高效的标志物组合。
52.一方面,本技术提供一种确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的方法,可以包含确定待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量,所述目标基因可以包含sept9和ikzf1。
53.例如,本技术的方法可以包含,根据待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量的确定结果,确认肝肿瘤是否存在。例如,本技术的方法可以包含,根据待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量的确定结果,评估是否确诊为肝肿瘤形成。例如,本技术的方法可以包含,根据待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量的确定结果,评估是否有确诊为肝肿瘤形成的风险和/或风险的高低。例如,本技术的方法可以包含,根据待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量的确定结果,评估肝肿瘤的进展情况。
54.另一方面,本技术提供一种评估肝肿瘤相关dna区域甲基化状态的方法,可以包含确定待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量,所述目标基因可以包含sept9和ikzf1。
55.例如,根据待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量的确定情况,评估肝肿瘤相关dna区域甲基化状态。例如,肝肿瘤相关dna区域甲基化状态可以是指该dna区域的甲基化的确认存在或相对于参考水平的数量提高,可以与肝肿瘤的发生有关联。
56.例如,本技术的基因可以通过它们的名称和它们的染色体坐标来描述。例如,染色体坐标可以与2009年2月发布的人类基因组数据库hg19版(或称作“hg19坐标”)一致。例如,本技术的dna区域可以是来源于由hg19坐标限定的区域。
57.例如,本技术的方法中,所述sept9的dna区域可以来源于人chr17:75276651-75496678。
58.例如,本技术的方法中,所述ikzf1的dna区域可以来源于人chr7:50343720-50472799。
59.例如,本技术的方法中,所述目标基因还可以包含选自以下组的基因:best4、b4galnt1、grasp、irf4和bend4。
60.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4和b4galnt1。例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、grasp和b4galnt1。例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、irf4、b4galnt1和bend4。
61.例如,本技术的方法中,所述目标基因可以包含至少2种基因。
62.例如,本技术的方法中,所述目标基因可以包含2种至7种基因。例如,本技术的目标基因可以包含2种、3种、4种、5种、6种、或7种本技术提供的目标基因。例如,对相同的目标基因,本技术可以选择该目标基因所在的一种或更多种的dna区域。例如,本技术的方法中,所述best4的dna区域可以来源于人chr1:45249257-45253377。例如,本技术的方法中,所述b4galnt1的dna区域可以来源于人chr12:58017193-58027138。例如,本技术的方法中,所述grasp的dna区域可以来源于人chr12:52400724-52409673。例如,本技术的方法中,所述irf4的dna区域可以来源于人chr6:391739-411447。例如,本技术的方法中,所述bend4的dna区域可以来源于人chr4:42112955-42154895。
63.例如,本技术的方法中,所述目标基因可以包含选自以下组的基因中的2种:
sept9、ikzf1、best4、b4galnt1、grasp、irf4和bend4。
64.例如,本技术的方法中,所述目标基因可以包含sept9和ikzf1。
65.例如,本技术的方法中,所述目标基因可以包含sept9和best4。
66.例如,本技术的方法中,所述目标基因可以包含sept9和b4galnt1。
67.例如,本技术的方法中,所述目标基因可以包含sept9和grasp。
68.例如,本技术的方法中,所述目标基因可以包含sept9和irf4。
69.例如,本技术的方法中,所述目标基因可以包含sept9和bend4。
70.例如,本技术的方法中,所述目标基因可以包含ikzf1和best4。
71.例如,本技术的方法中,所述目标基因可以包含ikzf1和b4galnt1。
72.例如,本技术的方法中,所述目标基因可以包含ikzf1和grasp。
73.例如,本技术的方法中,所述目标基因可以包含ikzf1和irf4。
74.例如,本技术的方法中,所述目标基因可以包含ikzf1和bend4。
75.例如,本技术的方法中,所述目标基因可以包含best4和b4galnt1。
76.例如,本技术的方法中,所述目标基因可以包含best4和grasp。
77.例如,本技术的方法中,所述目标基因可以包含best4和irf4。
78.例如,本技术的方法中,所述目标基因可以包含best4和bend4。
79.例如,本技术的方法中,所述目标基因可以包含b4galnt1和grasp。
80.例如,本技术的方法中,所述目标基因可以包含b4galnt1和irf4。
81.例如,本技术的方法中,所述目标基因可以包含b4galnt1和bend4。
82.例如,本技术的方法中,所述目标基因可以包含grasp和irf4。
83.例如,本技术的方法中,所述目标基因可以包含grasp和bend4。
84.例如,本技术的方法中,所述目标基因可以包含irf4和bend4。
85.例如,本技术的方法中,所述目标基因可以包含选自以下组的基因中的3种:sept9、ikzf1、best4、b4galnt1、grasp、irf4和bend4。
86.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1和best4。
87.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1和b4galnt1。
88.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1和grasp。
89.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1和irf4。
90.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1和bend4。
91.例如,本技术的方法中,所述目标基因可以包含sept9、best4和b4galnt1。
92.例如,本技术的方法中,所述目标基因可以包含sept9、best4和grasp。
93.例如,本技术的方法中,所述目标基因可以包含sept9、best4和irf4。
94.例如,本技术的方法中,所述目标基因可以包含sept9、best4和bend4。
95.例如,本技术的方法中,所述目标基因可以包含sept9、b4galnt1和grasp。
96.例如,本技术的方法中,所述目标基因可以包含sept9、b4galnt1和irf4。
97.例如,本技术的方法中,所述目标基因可以包含sept9、b4galnt1和bend4。
98.例如,本技术的方法中,所述目标基因可以包含sept9、grasp和irf4。
99.例如,本技术的方法中,所述目标基因可以包含sept9、grasp和bend4。
100.例如,本技术的方法中,所述目标基因可以包含sept9、irf4和bend4。
101.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4和b4galnt1。
102.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4和grasp。
103.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4和irf4。
104.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4和bend4。
105.例如,本技术的方法中,所述目标基因可以包含ikzf1、b4galnt1和grasp。
106.例如,本技术的方法中,所述目标基因可以包含ikzf1、b4galnt1和irf4。
107.例如,本技术的方法中,所述目标基因可以包含ikzf1、b4galnt1和bend4。
108.例如,本技术的方法中,所述目标基因可以包含ikzf1、grasp和irf4。
109.例如,本技术的方法中,所述目标基因可以包含ikzf1、grasp和bend4。
110.例如,本技术的方法中,所述目标基因可以包含ikzf1、irf4和bend4。
111.例如,本技术的方法中,所述目标基因可以包含best4、b4galnt1和grasp。
112.例如,本技术的方法中,所述目标基因可以包含best4、b4galnt1和irf4。
113.例如,本技术的方法中,所述目标基因可以包含best4、b4galnt1和bend4。
114.例如,本技术的方法中,所述目标基因可以包含best4、grasp和irf4。
115.例如,本技术的方法中,所述目标基因可以包含best4、grasp和bend4。
116.例如,本技术的方法中,所述目标基因可以包含best4、irf4和bend4。
117.例如,本技术的方法中,所述目标基因可以包含b4galnt1、grasp和irf4。
118.例如,本技术的方法中,所述目标基因可以包含b4galnt1、grasp和bend4。
119.例如,本技术的方法中,所述目标基因可以包含b4galnt1、irf4和bend4。
120.例如,本技术的方法中,所述目标基因可以包含grasp、irf4和bend4。
121.例如,本技术的方法中,所述目标基因可以包含选自以下组的基因中的4种:sept9、ikzf1、best4、b4galnt1、grasp、irf4和bend4。
122.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4和b4galnt1。
123.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4和grasp。
124.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4和irf4。
125.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4和bend4。
126.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、b4galnt1和grasp。
127.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、b4galnt1和irf4。
128.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、b4galnt1和bend4。
129.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、grasp和irf4。
130.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、grasp和bend4。
131.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、irf4和bend4。
132.例如,本技术的方法中,所述目标基因可以包含sept9、best4、b4galnt1和grasp。
133.例如,本技术的方法中,所述目标基因可以包含sept9、best4、b4galnt1和irf4。
134.例如,本技术的方法中,所述目标基因可以包含sept9、best4、b4galnt1和bend4。
135.例如,本技术的方法中,所述目标基因可以包含sept9、best4、grasp和irf4。
136.例如,本技术的方法中,所述目标基因可以包含sept9、best4、grasp和bend4。
137.例如,本技术的方法中,所述目标基因可以包含sept9、best4、irf4和bend4。
138.例如,本技术的方法中,所述目标基因可以包含sept9、b4galnt1、grasp和irf4。
139.例如,本技术的方法中,所述目标基因可以包含sept9、b4galnt1、grasp和bend4。
140.例如,本技术的方法中,所述目标基因可以包含sept9、b4galnt1、irf4和bend4。
141.例如,本技术的方法中,所述目标基因可以包含sept9、grasp、irf4和bend4。
142.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、b4galnt1和grasp。
143.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、b4galnt1和irf4。
144.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、b4galnt1和bend4。
145.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、grasp和irf4。
146.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、grasp和bend4。
147.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、irf4和bend4。
148.例如,本技术的方法中,所述目标基因可以包含ikzf1、b4galnt1、grasp和irf4。
149.例如,本技术的方法中,所述目标基因可以包含ikzf1、b4galnt1、grasp和bend4。
150.例如,本技术的方法中,所述目标基因可以包含ikzf1、b4galnt1、irf4和bend4。
151.例如,本技术的方法中,所述目标基因可以包含ikzf1、grasp、irf4和bend4。
152.例如,本技术的方法中,所述目标基因可以包含best4、b4galnt1、grasp和irf4。
153.例如,本技术的方法中,所述目标基因可以包含best4、b4galnt1、grasp和bend4。
154.例如,本技术的方法中,所述目标基因可以包含best4、b4galnt1、irf4和bend4。
155.例如,本技术的方法中,所述目标基因可以包含b4galnt1、grasp、irf4和bend4。
156.例如,本技术的方法中,所述目标基因可以包含选自以下组的基因中的5种:sept9、ikzf1、best4、b4galnt1、grasp、irf4和bend4。
157.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、b4galnt1和grasp。
158.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、b4galnt1和irf4。
159.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、b4galnt1和bend4。
160.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、grasp和irf4。
161.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、grasp和bend4。
162.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、irf4和bend4。
163.例如,本技术的方法中,所述目标基因可以包含sept9、best4、b4galnt1、grasp和irf4。
164.例如,本技术的方法中,所述目标基因可以包含sept9、best4、b4galnt1、grasp和bend4。
165.例如,本技术的方法中,所述目标基因可以包含sept9、best4、b4galnt1、irf4和bend4。
166.例如,本技术的方法中,所述目标基因可以包含sept9、b4galnt1、grasp、irf4和bend4。
167.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、b4galnt1、grasp和irf4。
168.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、b4galnt1、grasp和bend4。
169.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、b4galnt1、irf4和bend4。
170.例如,本技术的方法中,所述目标基因可以包含ikzf1、b4galnt1、grasp、irf4和bend4。
171.例如,本技术的方法中,所述目标基因可以包含best4、b4galnt1、grasp、irf4和bend4。
172.例如,本技术的方法中,所述目标基因可以包含选自以下组的基因中的6种:sept9、ikzf1、best4、b4galnt1、grasp和irf4和bend4。
173.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、b4galnt1、grasp和irf4。
174.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、b4galnt1、grasp和bend4。
175.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、b4galnt1、irf4和bend4。
176.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、grasp、irf4和bend4。
177.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、b4galnt1、grasp、irf4和bend4。
178.例如,本技术的方法中,所述目标基因可以包含sept9、best4、b4galnt1、grasp、irf4和bend4。
179.例如,本技术的方法中,所述目标基因可以包含ikzf1、best4、b4galnt1、grasp、irf4和bend4。
180.例如,本技术的方法中,所述目标基因可以包含选自以下组的基因中的7种:sept9、ikzf1、best4、b4galnt1、grasp、irf4和bend4。
181.例如,本技术的方法中,所述目标基因可以包含sept9、ikzf1、best4、b4galnt1、grasp、irf4和bend4。
182.例如,本技术的方法中位于sept9基因所在dna区域的目标dna区域可以包含来源于人chr17:75368651-75370720定义的区域。例如来源于人chr17:75369558-75369622定义的区域。例如,该目标dna区域可以通过sept(1)表示。
183.例如,本技术的方法中位于ikzf1基因所在dna区域的目标dna区域可以包含来源于人chr7:50343720-50344547定义的区域。例如来源于人chr7:50343793-50343896定义的区域。例如,该目标dna区域可以通过ikzf1(1)表示。
184.例如,本技术的方法中位于best4基因所在dna区域的目标dna区域可以包含来源于人chr1:45251728-45252477定义的区域。例如来源于人chr1:45252095-45252176定义的区域。例如,该目标dna区域可以通过best4(1)表示。
185.例如,本技术的方法中位于b4galnt1基因所在dna区域的目标dna区域可以包含来源于人chr12:58020498-58022962定义的区域。例如来源于人chr12:58021586-58021670定义的区域。例如,该目标dna区域可以通过b4galnt1(1)表示。
186.例如,本技术的方法中位于grasp基因所在dna区域的目标dna区域可以包含来源于人chr12:52400724-52401698定义的区域。例如来源于人chr12:52401083-52401169定义的区域。例如,该目标dna区域可以通过grasp(1)表示。
187.例如,本技术的方法中位于irf4基因所在dna区域的目标dna区域可以包含来源于人chr6:391739-394056定义的区域。例如来源于人chr6:392282-392377定义的区域。例如,该目标dna区域可以通过irf4(1)表示。
188.例如,本技术的方法中位于bend4基因所在dna区域的目标dna区域可以包含来源于人chr4:42152705-42154895定义的区域。例如来源于人chr4:42153816-42153921定义的区域。例如,该目标dna区域可以通过bend4(1)表示。
189.例如,本技术的方法中位于sept9基因所在dna区域的目标dna区域可以包含来源于人chr17:75368651-75370720定义的区域。例如来源于人chr17:75369603-75369693定义的区域。例如,该目标dna区域可以通过sept9(1a)表示。
190.另一方面,本技术提供一种确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的方法,可以包含确定待测样本中目标基因所在dna区域的特定的亚区域(例如目标dna区域)、或其互补区域、或上述的片段的修饰状态的存在和/或含量。
191.另一方面,本技术提供一种确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的方法,可以包含确定待测样本中目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量,所述目标dna区域可以包含来源于人chr17:75368651-75370720和来源于人chr7:50343720-50344547定义的区域。例如,本技术的方法可以包含,根据待测样本中所述目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量的确定结果,确认疾病是否存在。例如,本技术的方法可以包含,根据待测样本中所述目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量的确定结果,评估是否确诊为疾病形成。例如,本技术的方法可以包含,根据待测样本中所述目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量的确定结果,评估是否有确诊为疾病的风险和/或风险的高低。例如,本技术的方法可以包含,根据待测样本中所述目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量的确定结果,评估疾病的进展情况。
192.另一方面,本技术提供一种确定dna区域甲基化状态的方法,可以包含确定待测样本中目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量,所述目标dna区域可以包含来源于人chr17:75368651-75370720和来源于人chr7:50343720-50344547定义的区域。
193.例如,该目标dna区域的甲基化的确认存在或相对于参考水平的数量提高,可以与疾病的发生有关联。例如,本技术的目标dna区域可以是指目标基因组dna的一个或多个特定区段。例如,本技术的目标dna区域可以通过基因名称或一组染色体坐标来指定。例如,以sept9表示的目标dna区域可以是sept9基因所在dna区域的一个或多个特定区段。例如,以sept9表示的目标dna区域可以是本技术提供的sept9基因中目标dna区域的一个或多个特
定区段。例如,sept9的一个或多个不同的特定区段可以通过sept9(1)和sept9(1a)等可以区分的形式分别地表示。
194.例如,一个基因可以通过参考其名称获得其序列和染色体位置,或通过参考其染色体坐标确定其序列和染色体位置。本技术采用这些特定dna区域甲基化状态作为一个系列分析指标,可以在灵敏度和/或特异性方面提供显著的改进,并且可以简化筛查过程。例如,“灵敏度”可以指正确鉴定的阳性结果的比例,即,正确鉴定为具有所讨论疾病的个体的百分数;“特异性”可以指正确鉴定的阴性结果的比例,即,正确鉴定为不具有所讨论疾病的个体的百分数。
195.本技术的dna区域可以包含这些分子的全部形式及其片段或变体。例如,变体可以包含相对于本技术所述的dna区域共有至少80%、至少85%、至少90%、95%、98%、或99%序列同一性,变体可以包含一个或多个缺失、添加、置换、倒转序列等。例如,本技术所述变体的修饰状态可以实现相同的评估结果。本技术的dna区域可以包含全部形式的任何其他的突变、多态性变异或等位变异。
196.例如,本技术的方法可以包含提供能够结合包含seq id no:1所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
197.例如,本技术的方法中,所述目标区域可以包含来源于人chr17:75369558-75369622定义的区域。例如,该目标dna区域可以通过sept(1)表示。
198.例如,本技术的方法可以包含提供seq id no:2所示的核酸或其互补核酸、或上述的片段。例如,所述核酸可以用于检测目标区域。例如,所述核酸可以作为探针。
199.例如,本技术的方法可以包含提供seq id no:3与4所示的核酸组或其互补核酸组、或上述的片段。例如,所述核酸组可以用于扩增目标区域。例如,所述核酸组可以作为引物组。
200.例如,上述探针和/或引物组可以对来源于人chr17:75369558-75369622定义的区域的dna区域进行检测和/或扩增。
201.例如,本技术的方法可以包含提供能够结合包含seq id no:5所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
202.例如,本技术的方法中,所述目标区域可以包含来源于人chr7:50343793-50343896定义的区域。例如,该目标dna区域可以通过ikzf1(1)表示。
203.例如,本技术的方法可以包含提供seq id no:6所示的核酸或其互补核酸、或上述的片段。例如,所述核酸可以用于检测目标区域。例如,所述核酸可以作为探针。
204.例如,本技术的方法可以包含提供seq id no:7与8所示的核酸组或其互补核酸组、或上述的片段。例如,所述核酸组可以用于扩增目标区域。例如,所述核酸组可以作为引物组。
205.例如,上述探针和/或引物组可以对来源于人chr7:50343793-50343896定义的区域的dna区域进行检测和/或扩增。
206.例如,本技术的方法中,所述目标dna区域还可以包含选自以下组定义的区域:来源于人chr1:45251728-45252477、来源于人chr12:58020498-58022962、来源于人chr12:52400724-52401698、来源于人chr6:391739-394056、和来源于人chr4:42152705-42154895。
207.例如,本技术的方法中,所述目标dna区域可以包含至少2个区域。
208.例如,本技术的方法中,所述目标dna区域可以包含2个至8个区域。例如,本技术的方法中,所述目标dna区域可以包含2个、3个、4个、5个、6个、7个、或8个区域。
209.例如,本技术的方法中,所述目标dna区域可以包含来源于人chr17:75368651-75370720、来源于人chr7:50343720-50344547、来源于人chr1:45251728-45252477和来源于人chr12:58020498-58022962定义的区域。
210.例如,本技术的方法中,所述目标dna区域可以包含来源于人chr17:75368651-75370720、来源于人chr7:50343720-50344547、来源于人chr1:45251728-45252477、来源于人chr12:52400724-52401698和来源于人chr12:58020498-58022962定义的区域。
211.例如,本技术的方法中,所述目标dna区域可以包含来源于人chr6:391739-394056、来源于人chr7:50343720-50344547、来源于人chr1:45251728-45252477、来源于人chr12:58020498-58022962、来源于人chr4:42152705-42154895和来源于人chr17:75368651-75370720定义的区域。
212.例如,本技术的方法可以包含提供能够结合包含seq id no:9所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
213.例如,本技术的方法中,所述目标区域可以包含来源于人chr1:45252095-45252176定义的区域。例如,该目标dna区域可以通过best4(1)表示。
214.例如,本技术的方法可以包含提供seq id no:10所示的核酸或其互补核酸、或上述的片段。例如,所述核酸可以用于检测目标区域。例如,所述核酸可以作为探针。
215.例如,本技术的方法可以包含提供seq id no:11与12所示的核酸组或其互补核酸组、或上述的片段。例如,所述核酸组可以用于扩增目标区域。例如,所述核酸组可以作为引物组。
216.例如,上述探针和/或引物组可以对来源于人chr1:45252095-45252176定义的区域的dna区域进行检测和/或扩增。
217.例如,本技术的方法可以包含提供能够结合包含seq id no:13所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
218.例如,本技术的方法中,所述目标区域可以包含来源于人chr12:58021586-58021670定义的区域。例如,该目标dna区域可以通过b4galnt1(1)表示。
219.例如,本技术的方法可以包含提供seq id no:14所示的核酸或其互补核酸、或上述的片段。例如,所述核酸可以用于检测目标区域。例如,所述核酸可以作为探针。
220.例如,本技术的方法可以包含提供seq id no:15与16所示的核酸组或其互补核酸组、或上述的片段。例如,所述核酸组可以用于扩增目标区域。例如,所述核酸组可以作为引物组。
221.例如,上述探针和/或引物组可以对来源于人chr12:58021586-58021670定义的区域的dna区域进行检测和/或扩增。
222.例如,本技术的方法可以包含提供能够结合包含seq id no:17所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
223.例如,本技术的方法中,所述目标区域可以包含来源于人chr12:52401083-52401169定义的区域。例如,该目标dna区域可以通过grasp(1)表示。
224.例如,本技术的方法可以包含提供seq id no:18所示的核酸或其互补核酸、或上述的片段。例如,所述核酸可以用于检测目标区域。例如,所述核酸可以作为探针。
225.例如,本技术的方法可以包含提供seq id no:19与20所示的核酸组或其互补核酸组、或上述的片段。例如,所述核酸组可以用于扩增目标区域。例如,所述核酸组可以作为引物组。
226.例如,上述探针和/或引物组可以对来源于人chr12:52401083-52401169定义的区域的dna区域进行检测和/或扩增。
227.例如,本技术的方法可以包含提供能够结合包含seq id no:21所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
228.例如,本技术的方法中,所述目标区域可以包含来源于人chr6:392282-392377定义的区域。例如,该目标dna区域可以通过irf4(1)表示。
229.例如,本技术的方法可以包含提供seq id no:22所示的核酸或其互补核酸、或上述的片段。例如,所述核酸可以用于检测目标区域。例如,所述核酸可以作为探针。
230.例如,本技术的方法可以包含提供seq id no:23与24所示的核酸组或其互补核酸组、或上述的片段。例如,所述核酸组可以用于扩增目标区域。例如,所述核酸组可以作为引物组。
231.例如,上述探针和/或引物组可以对来源于人chr6:392282-392377定义的区域的dna区域进行检测和/或扩增。
232.例如,本技术的方法可以包含提供能够结合包含seq id no:25所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
233.例如,本技术的方法中,所述目标区域可以包含来源于人chr4:42153816-42153921定义的区域。例如,该目标dna区域可以通过bend4(1)表示。
234.例如,本技术的方法可以包含提供seq id no:26所示的核酸或其互补核酸、或上述的片段。例如,所述核酸可以用于检测目标区域。例如,所述核酸可以作为探针。
235.例如,本技术的方法可以包含提供seq id no:27与28所示的核酸组或其互补核酸组、或上述的片段。例如,所述核酸组可以用于扩增目标区域。例如,所述核酸组可以作为引物组。
236.例如,上述探针和/或引物组可以对来源于人chr4:42153816-42153921定义的区域的dna区域进行检测和/或扩增。
237.例如,本技术的方法可以包含提供能够结合包含seq id no:1所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
238.例如,本技术的方法中,所述目标区域可以包含来源于人chr17:75369603-75369693定义的区域。例如,该目标dna区域可以通过sept9(1a)表示。
239.例如,本技术的方法可以包含提供seq id no:29所示的核酸或其互补核酸、或上述的片段。例如,所述核酸可以用于检测目标区域。例如,所述核酸可以作为探针。
240.例如,本技术的方法可以包含提供seq id no:30与31所示的核酸组或其互补核酸组、或上述的片段。例如,所述核酸组可以用于扩增目标区域。例如,所述核酸组可以作为引物组。
241.例如,上述探针和/或引物组可以对来源于人chr17:75369603-75369693定义的区
域的dna区域进行检测和/或扩增。
242.例如,上述目标区域的一种或多种可以作为扩增区域和/或检测区域。
243.例如,所述疾病可以包含肿瘤。例如,所述疾病可以包含实体瘤。例如,所述疾病可以包含肝肿瘤等任意的肿瘤。
244.例如,“引物”可以是天然的或合成的寡核苷酸,其在与多核苷酸模板形成双链体后能够充当核酸合成的起始点并从其3
′
端沿模板延伸,从而形成延伸的双链体。在延伸过程中添加的核苷酸的序列由模板多核苷酸的序列决定。引物通常可以通过聚合酶如核酸聚合酶延伸。
245.例如,本技术的“互补的”和“基本上互补的”可以包括在核苷酸或核酸之间,例如在双链dna分子的两条链之间,或在寡核苷酸引物和单链核酸上的引物结合位点之间的杂交或碱基配对或双链体的形成。互补的核苷酸可以通常是a和t(或a和u)或c和g。对于两个单链rna或dna分子,当一条链的核苷酸在进行最佳比对和比较并且具有适当的核苷酸插入或缺失时与另一条链的至少约80%(通常至少约90%至约95%,甚至约98%至约100%)成对时,可以认为它们是基本互补的。在一个方面,两个互补的核苷酸序列能够杂交,并且可以在反向的核苷酸之间有小于25%的错配,更可以以小于15%的错配,可以以小于5%的错配,或不具有错配。例如,两个分子可以在高严格条件下杂交。
246.例如,本技术的修饰状态可以是指该修饰状态在dna区域内部一个特定核苷酸或多个核苷酸处的存在、不存在和/或其含量。例如,本技术的修饰状态可以是指特定dna序列中每个碱基或每个特定碱基(例如胞嘧啶)的修饰状态。例如,本技术的修饰状态可以是指特定dna序列中碱基对组合和/或碱基组合的修饰状态。例如,本技术的修饰状态可以是指特定dna序列(包括基因所在dna区域或其特定区域片段)中关于区域修饰密度的信息,而可以不提供该序列中何处发生修饰的精确位置信息。
247.例如,本技术的修饰状态可以是指甲基化状态或与甲基化类似的状态。例如,具有或具有较高的甲基化的状态可以是与特定区域的转录沉默相关的。例如,具有或具有较高的甲基化的状态可以是与能够被甲基化特异性转化试剂(例如脱氨基试剂和/或甲基化敏感限制酶)转化相关的。例如,转化可以是指被转变为其它物质和/或被剪切或消化。
248.例如,“甲基化状态(methylation state)”或“甲基化状态(methylation status)”可以是指在dna序列内的一个或多个cpg二核苷酸处存在或不存在5-甲基胞嘧啶(“5-mc”或“5-mcyt”)。dna序列内一个或多个特定cpg甲基化位点(每个都有两个cpg二核苷酸序列)处的甲基化状态包括“未甲基化”、“完全甲基化”和“半甲基化”。术语“半甲基化”可以是指双链dna的甲基化状态,其中所述双链dna仅有一条链被甲基化。术语“高甲基化”可以是指与以下情况相对应的平均甲基化状态:相对于正常对照dna样品中相应cpg二核苷酸处发现的5-mcyt量,测试dna样品的dna序列中的一个或多个cpg二核苷酸处5-mcyt的存在增加。术语“低甲基化”可以是指与以下情况相对应的平均甲基化状态:相对于正常对照dna样品中相应cpg二核苷酸处发现的5-mcyt量,测试dna样品的dna序列中的一个或多个cpg二核苷酸处5-mcyt的存在降低。
249.例如,所述方法还可以包含获取待测样本中的核酸。例如,所述核酸可以包含无细胞游离核酸。例如,所述待测样本可以包含组织、细胞和/或体液。例如,所述待测样本可以包含血浆。例如,本技术的检测方法可以对任何适合的生物样品进行。例如,待测样本可以
为生物材料的任何样品,例如其可以源自动物,但不限于细胞材料、生物流体(例如血液)、排出物、组织活组织检查标本、手术标本或已经导入动物身体中并且随后取出的流体。例如,本技术的待测样本可以包含在所述样本分离后经任何形式处理的样本。
250.例如,所述方法还可以包含转化所述dna区域或其片段。例如,通过本技术的转化步骤,具有所述修饰状态的碱基以及不具有所述修饰状态的所述碱基,在转化后可以形成不同的物质。例如,具有所述修饰状态的碱基在转化后基本不发生改变,且不具有所述修饰状态的所述碱基在转化后可以改变为与所述碱基不同的其它碱基(例如,所述其它碱基可以包含尿嘧啶)、或在转化后被剪切。例如,所述碱基可以包含胞嘧啶。例如,所述修饰状态可以包含甲基化修饰。例如,所述转化可以包含通过脱氨基试剂和/或甲基化敏感限制酶转化。例如,所述脱氨基试剂可以包含亚硫酸氢盐或其类似物。例如,亚硫酸氢钠或亚硫酸氢钾。
251.例如,所述方法还可以任选地包含在确定所述dna区域或其片段的修饰的存在和/或含量之前,扩增待测样本中所述dna区域或其片段。例如,所述扩增可以包含pcr扩增。例如,本技术的扩增可以包含已知的任意一种扩增系统。例如,本技术的扩增步骤可以是任选地。例如,“扩增”可以是指产生所需序列的多个拷贝的过程。“多个拷贝”可以是指至少两个拷贝。“拷贝”可以不意味着与模板序列具有完美的序列互补性或同一性。例如,拷贝可以包括核苷酸类似物如脱氧肌苷,有意的序列改变(例如通过包含与模板可杂交但不互补的序列的引物引入的序列改变),和/或在扩增过程中可以发生序列错误。
252.例如,所述确定修饰状态的存在和/或含量的方法可以包含,确认具有所述修饰状态的碱基在所述转化后形成的物质的存在和/或含量。例如,所述确定修饰状态的存在和/或含量的方法可以包含,确定具有所述修饰状态的dna区域或其片段的存在和/或含量。例如,可以直接检测具有所述修饰状态的dna区域或其片段的存在和/或含量。例如,可以通过以下方式检测:具有所述修饰状态的dna区域或其片段可以在反应(例如扩增反应)的过程中可以与不具有所述修饰状态的dna区域或其片段具有不同的特性。例如,在荧光pcr方法中,具有所述修饰状态的dna区域或其片段可以被特异性扩增,并发出荧光;不具有所述修饰状态的dna区域或其片段可以基本不被扩增,并基本不发出荧光。例如,确定具有所述修饰状态的碱基在所述转化后形成的物质的存在和/或含量的替代方法,可以包含在本技术的范围之内。
253.例如,可以通过所述荧光pcr方法检测的荧光ct值,确定具有所述修饰状态的dna区域或其片段的存在和/或含量。例如,可以通过所述dna区域或其片段的修饰状态的存在和/或所述dna区域或其片段相对于参考水平具有更高的修饰状态的含量,确定肝肿瘤的存在、或者有肝肿瘤形成或形成的风险。例如,当所述待测样本的荧光ct值相对于参考荧光ct值更低时,可以确定所述dna区域或其片段的修饰状态的存在和/或可以确定所述dna区域或其片段的修饰状态的含量高于参考样本中的修饰状态的含量。例如,可以通过检测参考样本确定所述参考荧光ct值。例如,当所述待测样本的荧光ct值相对于参考荧光ct值更高或基本相当时,也可以不排除所述dna区域或其片段的修饰状态的存在;当所述待测样本的荧光ct值相对于参考荧光ct值更高或基本相当时,可以确认所述dna区域或其片段的修饰状态的含量低于或基本等于参考样本中的修饰状态的含量。
254.例如,本技术可以通过循环阈值(即ct值)来表示特定dna区域或其片段的修饰状
态的存在和/或含量,例如包括待测样本的甲基化水平和参考水平。例如,ct值可以是指在背景信号以上可以检测到pcr产物的荧光的循环数。例如,ct值与样品中目标标记物的起始数量可以成负相关关系,即ct值越低,待测样品中dna区域或其片段的修饰状态的数量越多。
255.例如,当待测样品的ct值相对于其相应的参考ct值相同或更低可以确认为存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。例如,当待测样品的ct值相对于其相应的参考ct值低至少1个循环、至少2个循环、至少5个循环、至少10个循环、至少20个循环、或至少50个循环时,可以确认为存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。
256.例如,当细胞样本、组织样本或来源于受试者的样本的ct值相对于其相应的参考ct值相同或更高,可以确认为不存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。例如,当细胞样本、组织样本或来源于受试者的样本的ct值相对于其相应的参考ct值高至少1个循环、至少2个循环、至少5个循环、至少10个循环、至少20个循环、或至少50个循环时,可以确认为不存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。例如,当细胞样本、组织样本或来源于受试者的样本的ct值相对于其相应的参考ct值疾病相同时,可以确认为存在或不存在特定疾病、诊断为特定疾病的形成或未形成、具有或不具有形成风险或者评估为特定疾病的某种进展,并同时可以给出需要进一步检测的建议。
257.例如,本技术的参考水平或对照水平可以是指是正常水平或健康水平。例如,所述正常水平可以是来源于无所述疾病的细胞、组织或个体的样本dna区域的修饰状态水平。例如,当用于肿瘤的评估,所述正常水平可以是来源于无肿瘤的细胞、组织或个体的样本dna区域的修饰状态水平。例如,当用于肝肿瘤的评估,所述正常水平可以是来源于无肝肿瘤的细胞、组织或个体的样本dna区域的修饰状态水平。
258.例如,在本技术中参考水平可以是指将受试者或样本确认为存在或不存在特定疾病的阈值水平。例如,在本技术中参考水平可以是指将受试者诊断为特定疾病的形成或具有形成风险的阈值水平。例如,在本技术中参考水平可以是指将受试者评估为特定疾病的某种进展的阈值水平。例如,当细胞样本、组织样本或来源于受试者的样本中的dna区域的修饰状态高于或基本等于相应参考水平时,例如此处参考水平可以是指不具有特定疾病患者的dna区域的修饰状态,可以确认为存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。例如,本技术中的a与b“基本等于”可以是指a与b的差值为1%或更少、0.5%或更少、0.1%或更少、0.01%或更少、0.001%或更少或0.0001%或更少。例如,当细胞样本、组织样本或来源于受试者的样本中的dna区域的修饰状态高于相应参考水平至少1%、至少5%、至少10%、至少20%、至少50%、至少1倍、至少2倍、至少5倍、至少10倍、或至少20倍时,可以确认为存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。例如,当多次检测中的至少一次、至少两次、或至少三次的检测中,细胞样本、组织样本或来源于受试者的样本中的dna区域的修饰状态高于相应参考水平至少1%、至少5%、至少10%、至少20%、至少50%、至少1倍、至少2倍、至少5倍、至少10倍、或至少20倍时,可以确认为存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。
259.例如,当细胞样本、组织样本或来源于受试者的样本中的dna区域的修饰状态低于或基本等于相应参考水平时,例如此处参考水平可以是指具有特定疾病患者的dna区域的修饰状态,可以确认为不存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。例如,当细胞样本、组织样本或来源于受试者的样本中的dna区域的修饰状态低于相应参考水平至少1%、至少5%、至少10%、至少20%、至少50%、至少100%时,可以确认为不存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。
260.本领域技术人员可以根据期望的灵敏度和特异性来选择参考水平。例如,在本技术中各种情况下的参考水平可以是本领域人员容易确认的,如根据有限次尝试确认合适的参考水平和/或合适的获取参考水平的手段,例如,参考水平可以源自一个或多个参考样品,其中参考水平获自与检测目的样品的实验平行进行的实验。或者,也可以在数据库中获得参考水平,该数据库包括来自一个或多个参考样品或疾病参考样品的数据、标准或水平的集合。在一些实施方式中,数据、标准或水平的集合可以被标准化或归一化,以便可用于与来自一个或多个样品的数据进行比较,从而用于减少不同检测条件下产生的误差。
261.例如,参考水平可以来源于数据库,该数据库可以是参考数据库,例如包括来自一个或多个参考样品的目标标记物和/或其他实验室和临床数据的修饰状态水平。例如,可以通过汇总获自健康个体和/或非相应疾病患者个体(即已知没有该疾病的个体)的参考样品的参考水平数据来建立参考数据库。例如,可以通过汇总获自正在接受治疗的患有相应疾病个体的参考样品的参考水平数据来建立参考数据库。例如,可以通过汇总获自疾病不同阶段的个体的参考样品的数据来建立参考数据库。例如,例如不同阶段可以是通过本技术目标标记物的不同的修饰状态水平来证明的。本领域技术人员还可以基于各种因素,例如年龄、性别、病史、家族史、症状等,来确定个体是否患相应疾病或具有患相应疾病的风险。
262.例如,本技术的方法可以包含以下步骤:获取待测样本中的核酸;转化所述dna区域或其片段;确认具有所述修饰状态的碱基在所述转化后形成的物质的存在和/或含量。
263.例如,本技术的方法可以包含以下步骤:获取待测样本中的核酸;转化所述dna区域或其片段;扩增待测样本中所述dna区域或其片段;确认具有所述修饰状态的碱基在所述转化后形成的物质的存在和/或含量。
264.例如,本技术的方法可以包含以下步骤:获取待测样本中的核酸;用试剂处理从待测样品中获得的dna,所述试剂能够区分所述dna中的未甲基化位点和甲基化位点,从而获得经处理的dna;可选地扩增待测样本中所述dna区域或其片段;定量、半定量或定性分析待测样本中经处理的dna的甲基化状态的存在和/或含量;比较测样本中经处理的dna的甲基化水平以及相应的参考水平,当待测样本中的dna区域的甲基化状态高于或基本等于相应参考水平时,可以确认为存在特定疾病、诊断为特定疾病的形成或具有形成风险或者评估为特定疾病的某种进展。
265.另一方面,本技术提供一种核酸,所述核酸可以包含能够结合本技术目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列。例如,所述核酸可以是本技术的任一种探针。另一方面,本技术提供一种制备核酸的方法,可以包含根据本技术目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述dna区域、或其互补区域、或上述的转化而来的区域、或上述的
片段的核酸。例如,制备核酸的方法可以是本领域已知的任意合适的方法。
266.另一方面,本技术提供一种核酸组,所述核酸组可以包含能够结合本技术目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列。例如,所述核酸组可以是本技术的任一种引物组。另一方面,本技术提供一种制备核酸组的方法,可以包含根据本技术目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够扩增所述dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸组。例如,制备核酸组中的核酸的方法可以是本领域已知的任意合适的方法。例如,可以使用单个探针或引物评估靶多核苷酸的甲基化状态,所述单个探针或引物被配置成与所述靶多核苷酸杂交。例如,可以使用多个探针或引物评估靶多核苷酸的甲基化状态,所述多个探针或引物被配置成与所述靶多核苷酸杂交。
267.另一方面,本技术提供一种核酸,所述核酸包含能够结合如本技术所述的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列;另一方面,本技术提供一种核酸,所述核酸可以包含能够结合如本技术所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列。
268.另一方面,本技术提供一种制备核酸的方法,所述方法可以包含根据如本技术所述的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸;另一方面,本技术提供一种核酸,所述方法可以包含根据如本技术所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
269.另一方面,本技术提供一种核酸组,所述核酸组包含能够结合如本技术所述的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列;另一方面,本技术提供一种核酸,所述核酸组可以包含能够结合如本技术所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列。例如,制备核酸的方法可以是本领域已知的任意合适的方法。
270.另一方面,本技术提供一种制备核酸组的方法,所述方法可以包含根据如本技术所述的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸组;另一方面,本技术提供一种核酸,所述方法可以包含根据如本技术所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸组。例如,可以使用单个探针或引物评估靶多核苷酸的甲基化状态,所述单个探针或引物被配置成与所述靶多核苷酸杂交。例如,可以使用多个探针或引物评估靶多核苷酸的甲基化状态,所述多个探针或引物被配置成与所述靶多核苷酸杂交。
271.另一方面,本技术提供一种试剂盒,可以包含如本技术的核酸和/或本技术的核酸组。例如,本技术的试剂盒可以可选地包含相应用途的参考样本或提供相应用途的参考水平。
272.诊断方法、制备用途
273.例如,本技术的方法可以用于受试者中的癌症或肿瘤形成的诊断、预后、分层、风险评估或治疗监测。例如,“受试者”可以是指可以对之施用或施加本技术提供的方法、核酸、核酸组、试剂盒、装置和系统的生物体或所述生物体的一部分或组分。例如,所述受试者可以是哺乳动物或所述哺乳动物的细胞、组织、器官或一部分。如本文所用,“哺乳动物”是指任何种类的哺乳动物,优选人(包括人、人受试者或人患者)。受试者和哺乳动物包括,但不限于,农场动物、运动动物、宠物、灵长类动物、马、狗、猫和啮齿类动物如小鼠和大鼠。
274.另一方面,本技术所述方法可用于评估任何合适的受试者中的癌症或肿瘤形成。例如,本技术所述方法可用于评估哺乳动物中的癌症或肿瘤形成。所述哺乳动物可以是非人哺乳动物,例如宠物,农场动物,伴侣动物或实验动物。优选地,所述哺乳动物是人。例如,受试者可以是需要进行癌症或肿瘤形成风险筛查的人,高危人群中的人,被诊断为患有癌症或肿瘤形成但需要进一步分层或分级的人,被诊断为患有癌症或肿瘤形成且正在接受积极治疗的人,或患有癌症或肿瘤形成并正在缓解的人。
275.另一方面,本技术提供如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,在制备确定dna区域或其片段的修饰状态的物质中的应用。
276.另一方面,本技术提供一种确定所述dna区域或其片段的修饰状态的方法,可以包括提供本技术的核酸、如本技术的核酸组和/或本技术的试剂盒。
277.另一方面,本技术提供如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,其可以用于确定所述dna区域或其片段的修饰状态。
278.另一方面,本技术提供如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,在制备疾病检测产品中的应用。例如,本技术应用中,所述疾病可以包含肿瘤。例如实体瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。
279.另一方面,本技术提供一种疾病检测方法,可以包括提供本技术的核酸、如本技术的核酸组和/或本技术的试剂盒。例如,本技术应用中,所述疾病可以包含肿瘤。例如实体瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。
280.另一方面,本技术提供如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,其可以用于进行疾病检测。例如,本技术应用中,所述疾病可以包含肿瘤。例如实体瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。
281.另一方面,本技术提供如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,在制备确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的物质中的应用。例如,本技术应用中,所述疾病可以包含肿瘤。例如实体瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。
282.另一方面,本技术提供一种确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的方法,可以包括提供本技术的核酸、如本技术的核酸组和/或本技术的试剂盒。例如,本技术应用中,所述疾病可以包含肿瘤。例如实体瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。
283.另一方面,本技术提供如本技术的核酸、如本技术的核酸组和/或本技术的试剂盒,其可以用于确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展。例如,本技术应用中,所述疾病可以包含肿瘤。例如实体瘤。例如,本技术应用中,所述疾病可以包含
肝肿瘤。
284.另一方面,本技术提供用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒,在制备用于确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的物质中的应用,所述用于确定的dna区域可以包含如本技术的方法中所述目标基因所在dna区域或其片段。
285.另一方面,本技术提供确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的方法,可以包含提供用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒。所述用于确定的dna区域可以包含如本技术的方法中所述目标基因所在dna区域或其片段。
286.另一方面,本技术提供用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒,其可以用于确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展。所述用于确定的dna区域可以包含如本技术的方法中所述目标基因所在dna区域或其片段。
287.另一方面,本技术提供用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒,在制备用于确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的物质中的应用,所述用于确定的dna区域可以包含如本技术的方法中所述目标dna区域、或其互补区域、或上述的片段。
288.另一方面,本技术提供确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的方法,可以包含提供用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒。所述用于确定的dna区域可以包含如本技术的方法中所述目标dna区域、或其互补区域、或上述的片段。
289.另一方面,本技术提供用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒,其可以用于确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展。所述用于确定的dna区域可以包含如本技术的方法中所述目标dna区域、或其互补区域、或上述的片段。
290.例如,本技术应用中,所述疾病可以包含肿瘤。例如实体瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。例如,本技术应用中,所述修饰状态可以包含甲基化修饰。
291.另一方面,本技术提供如本技术的方法中所述目标基因所在dna区域、或其转化而来的区域、或上述的片段的核酸或其组合,在制备用于确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的物质中的应用。
292.另一方面,本技术提供确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的方法,可以包含提供本技术的方法中所述目标基因所在dna区域、或其转化而来的区域、或上述的片段的核酸或其组合。
293.另一方面,本技术提供如本技术的方法中所述目标基因所在dna区域、或其转化而来的区域、或上述的片段的核酸或其组合,其可以用于确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展。
294.另一方面,本技术提供如本技术的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸或其组合,在制备用于确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的物质中的应用。例如,本技术应用中,所述疾病可以包含肿瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。
295.另一方面,本技术提供确认疾病的存在、评估疾病形成或形成风险和/或评估疾病
的进展的方法,可以包含提供如本技术的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸或其组合。例如,本技术应用中,所述疾病可以包含肿瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。
296.另一方面,本技术提供如本技术的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸或其组合,其可以用于确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展。例如,本技术应用中,所述疾病可以包含肿瘤。例如,本技术应用中,所述疾病可以包含肝肿瘤。
297.另一方面,本技术提供一种设备,其可以包含本技术的储存介质。另一方面,本技术提供了一种非易失性计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行以实现本技术所述的任一种或多种的方法。例如,所述方法可以含有使用甲基化指标对具有不同癌性可能性的样品进行分类的方法。示例性分类算法可以是线性判别分析、逻辑回归、朴素贝叶斯分类、感知分类、二次分类、k近邻法、提升方法、决策树、随机森林、神经网络、学习向量量化或支持向量机、或其组合。例如,所述非易失性计算机可读存储介质可以包括软盘、柔性盘、硬盘、固态存储(sss)(例如固态驱动(ssd))、固态卡(ssc)、固态模块(ssm))、企业级闪存驱动、磁带或任何其他非临时性磁介质等。非易失性计算机可读存储介质还可以包括打孔卡、纸带、光标片(或任何其他具有孔型图案或其他光学可识别标记的物理介质)、压缩盘只读存储器(cd-rom)、可重写式光盘(cd-rw)、数字通用光盘(dvd)、蓝光光盘(bd)和/或任何其他非临时性光学介质。
298.例如,本技术的设备还可以包含耦接至所述储存介质的处理器,所述处理器被配置为基于存储在所述储存介质中的程序执行以实现本技术的方法。例如,所述设备可以实现各种机制以便确保在数据库系统上执行的本技术所述的方法产生正确的结果。在本技术中,所述设备可以使用磁盘作为永久性数据存储器。在本技术中,所述设备可以为多个数据库客户端提供数据库存储和处理服务。所述设备可以跨多个共享存储设备存储数据库数据,和/或可以利用具有多个执行节点的一个或更多个执行平台。所述设备可以被组织成使得存储和计算资源可以被有效地无限扩展。
299.本发明利用荧光pcr检测技术,检测白膜层(其中大多数为白细胞)、癌旁组织和肝癌组织相关标志物的甲基化情况。初步确认该标志物用于血液检测肝肿瘤的潜能。理想的肝癌甲基化检测标志物应该具备以下特征:1.白膜层dna甲基化水平低;2.肝癌组织dna甲基化水平高。
300.甲基化标志物检测可以采用甲基化特异pcr(msp)来进行,msp的基本原理是在检测中,只有甲基化的序列模板产生扩增信号,而非甲基化的序列模板不产生扩增信号。该方法可通过引物或者探针设计甲基化特异序列来实现,也可以同时设计甲基化特异的引物、探针对来实现。其主要步骤包括:1.根据标志物序列,在适合进行msp的检测区段(可以为cpg富集区域),设计检测引物和探针。2.进行白细胞和组织样本的核酸提取。3.对核酸进行亚硫酸氢盐处理,使未甲基化的胞嘧啶转化成尿嘧啶,而甲基化的胞嘧啶维持序列不变。4.进行荧光pcr检测。
301.经白细胞和组织样本检测,满足上述特征的标志物,证明其存在通过血液来检测肝癌的潜能。然后在血浆样本中,对这些标志物进行验证,检测样本包括对照组、肝癌组以及乙肝、肝硬化干扰人群。分析这些标志物的参考水平及标志物组合性能。因单个样本血浆
游离dna有限,可选地进行荧光pcr检测时,可以对靶点进行了预扩增,以使最少量的dna能够检测尽可能多的甲基化位点。其主要步骤包括:1.血浆样本的核酸提取。2.对核酸进行亚硫酸氢盐处理,使未甲基化的胞嘧啶转化成尿嘧啶,而甲基化的胞嘧啶维持序列不变。3.进行靶点的预扩增及稀释。4.进行荧光pcr检测。
302.在靶点组合分析时,也可在上述靶点组合基础上,再进行任意组合形式,或者在数据算法上引入机器学习的数学模型,如线性回归、支持向量回归、脊回归、随机森林等等,得到检测准确性高的肝癌甲基化检测标志物组合。
303.本申提供的内容:1.找到了众多具有优异性能的肝癌甲基化标志物;2.开发了检测准确性高的肝癌甲基化检测标志物组合;3.检测性能较已有的荧光pcr检测方法有大幅提升;4.可以较二代测序检测方法相比,具备易开展、易临床推广、成本低等众多优势。本技术提供的标志物以及标志物的组合,其应用方式可以不限于特定的检测方式。
304.实施方案
305.1.一种确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的方法,包含确定待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量,所述目标基因包含sept9和ikzf1。
306.2.一种评估肝肿瘤相关dna区域甲基化状态的方法,包含确定待测样本中目标基因所在dna区域或其片段的修饰状态的存在和/或含量,所述目标基因包含sept9和ikzf1。
307.3.如实施方案1-2中任一项所述的方法,所述sept9的dna区域来源于人chr17:75276651-75496678。
308.4.如实施方案1-3中任一项所述的方法,所述ikzf1的dna区域来源于人chr7:50343720-50472799。
309.5.如实施方案1-4中任一项所述的方法,所述目标基因还包含选自以下组的基因:best4、b4galnt1、grasp、irf4和bend4。
310.6.如实施方案1-5中任一项所述的方法,所述目标基因包含至少2种基因。
311.7.如实施方案1-6中任一项所述的方法,所述目标基因包含2种至7种基因。
312.8.如实施方案1-7中任一项所述的方法,所述目标基因包含sept9、ikzf1、best4和b4galnt1。
313.9.如实施方案1-8中任一项所述的方法,所述目标基因包含sept9、ikzf1、best4、grasp和b4galnt1。
314.10.如实施方案1-9中任一项所述的方法,所述目标基因包含sept9、ikzf1、best4、irf4、b4galnt1和bend4。
315.11.如实施方案5-10中任一项所述的方法,所述best4的dna区域来源于人chr1:45249257-45253377。
316.12.如实施方案5-11中任一项所述的方法,所述b4galnt1的dna区域来源于人chr12:58017193-58027138。
317.13.如实施方案5-12中任一项所述的方法,所述grasp的dna区域来源于人chr12:52400724-52409673。
318.14.如实施方案5-13中任一项所述的方法,所述irf4的dna区域来源于人chr6:391739-411447。
319.15.如实施方案5-14中任一项所述的方法,所述bend4的dna区域来源于人chr4:42112955-42154895。
320.16.一种确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的方法,包含确定待测样本中目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量,所述目标dna区域包含来源于人chr17:75368651-75370720和来源于人chr7:50343720-50344547定义的区域。
321.17.一种确定dna区域甲基化状态的方法,包含确定待测样本中目标dna区域、或其互补区域、或上述的片段的修饰状态的存在和/或含量,所述目标dna区域包含来源于人chr17:75368651-75370720和来源于人chr7:50343720-50344547定义的区域。
322.18.如实施方案1-17中任一项所述的方法,包含提供能够结合包含seq id no:1所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
323.19.如实施方案16-18中任一项所述的方法,所述目标区域包含来源于人chr17:75369558-75369622定义的区域。
324.20.如实施方案1-19中任一项所述的方法,包含提供seq id no:2所示的核酸或其互补核酸、或上述的片段。
325.21.如实施方案1-20中任一项所述的方法,包含提供seq id no:3与4所示的核酸组或其互补核酸组、或上述的片段。
326.22.如实施方案1-21中任一项所述的方法,包含提供能够结合包含seq id no:5所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
327.23.如实施方案16-22中任一项所述的方法,所述目标区域包含来源于人chr7:50343793-50343896定义的区域。
328.24.如实施方案1-23中任一项所述的方法,包含提供seq id no:6所示的核酸或其互补核酸、或上述的片段。
329.25.如实施方案1-24中任一项所述的方法,包含提供seq id no:7与8所示的核酸组或其互补核酸组、或上述的片段。
330.26.如实施方案16-25中任一项所述的方法,所述目标dna区域还包含选自以下组定义的区域:来源于人chr1:45251728-45252477、来源于人chr12:58020498-58022962、来源于人chr12:52400724-52401698、来源于人chr6:391739-394056、和来源于人chr4:42152705-42154895。
331.27.如实施方案16-26中任一项所述的方法,所述目标dna区域包含至少2个区域。
332.28.如实施方案16-27中任一项所述的方法,所述目标dna区域包含2个至8个区域。
333.29.如实施方案16-28中任一项所述的方法,所述目标dna区域包含来源于人chr17:75368651-75370720、来源于人chr7:50343720-50344547、来源于人chr1:45251728-45252477和来源于人chr12:58020498-58022962定义的区域。
334.30.如实施方案16-29中任一项所述的方法,所述目标dna区域包含来源于人chr17:75368651-75370720、来源于人chr7:50343720-50344547、来源于人chr1:45251728-45252477、来源于人chr12:52400724-52401698和来源于人chr12:58020498-58022962定义的区域。
335.31.如实施方案16-30中任一项所述的方法,所述目标dna区域包含来源于人chr6:
391739-394056、来源于人chr7:50343720-50344547、来源于人chr1:45251728-45252477、来源于人chr12:58020498-58022962、来源于人chr4:42152705-42154895和来源于人chr17:75368651-75370720定义的区域。
336.32.如实施方案1-31中任一项所述的方法,包含提供能够结合包含seq id no:9所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
337.33.如实施方案16-32中任一项所述的方法,所述目标区域包含来源于人chr1:45252095-45252176定义的区域。
338.34.如实施方案1-33中任一项所述的方法,包含提供seq id no:10所示的核酸或其互补核酸、或上述的片段。
339.35.如实施方案1-34中任一项所述的方法,包含提供seq id no:11与12所示的核酸组或其互补核酸组、或上述的片段。
340.36.如实施方案1-35中任一项所述的方法,包含提供能够结合包含seq id no:13所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
341.37.如实施方案16-36中任一项所述的方法,所述目标区域包含来源于人chr12:58021586-58021670定义的区域。
342.38.如实施方案1-37中任一项所述的方法,包含提供seq id no:14所示的核酸或其互补核酸、或上述的片段。
343.39.如实施方案1-38中任一项所述的方法,包含提供seq id no:15与16所示的核酸组或其互补核酸组、或上述的片段。
344.40.如实施方案1-39中任一项所述的方法,包含提供能够结合包含seq id no:17所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
345.41.如实施方案16-40中任一项所述的方法,所述目标区域包含来源于人chr12:52401083-52401169定义的区域。
346.42.如实施方案1-41中任一项所述的方法,包含提供seq id no:18所示的核酸或其互补核酸、或上述的片段。
347.43.如实施方案1-42中任一项所述的方法,包含提供seq id no:19与20所示的核酸组或其互补核酸组、或上述的片段。
348.44.如实施方案1-43中任一项所述的方法,包含提供能够结合包含seq id no:21所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
349.45.如实施方案16-44中任一项所述的方法,所述目标区域包含来源于人chr6:392282-392377定义的区域。
350.46.如实施方案1-45中任一项所述的方法,包含提供seq id no:22所示的核酸或其互补核酸、或上述的片段。
351.47.如实施方案1-46中任一项所述的方法,包含提供seq id no:23与24所示的核酸组或其互补核酸组、或上述的片段。
352.48.如实施方案1-47中任一项所述的方法,包含提供能够结合包含seq id no:25所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
353.49.如实施方案16-48中任一项所述的方法,所述目标区域包含来源于人chr4:42153816-42153921定义的区域。
354.50.如实施方案1-49中任一项所述的方法,包含提供seq id no:26所示的核酸或其互补核酸、或上述的片段。
355.51.如实施方案1-50中任一项所述的方法,包含提供seq id no:27与28所示的核酸组或其互补核酸组、或上述的片段。
356.52.如实施方案1-51中任一项所述的方法,包含提供能够结合包含seq id no:1所示的dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
357.53.如实施方案16-52中任一项所述的方法,所述目标区域包含来源于人chr17:75369603-75369693定义的区域。
358.54.如实施方案1-53中任一项所述的方法,包含提供seq id no:29所示的核酸或其互补核酸、或上述的片段。
359.55.如实施方案1-54中任一项所述的方法,包含提供seq id no:30与31所示的核酸组或其互补核酸组、或上述的片段。
360.56.如实施方案16-55中任一项所述的方法,所述疾病包含肿瘤。
361.57.如实施方案16-56中任一项所述的方法,所述疾病包含肝肿瘤。
362.58.如实施方案1-57中任一项所述的方法,所述方法还包含获取待测样本中的核酸。
363.59.如实施方案58所述的方法,所述核酸包含无细胞游离核酸。
364.60.如实施方案1-59中任一项所述的方法,所述待测样本包含组织、细胞和/或体液。
365.61.如实施方案1-60中任一项所述的方法,所述待测样本包含血浆。
366.62.如实施方案1-61中任一项所述的方法,所述方法还包含转化所述dna区域或其片段。
367.63.如实施方案62所述的方法,具有所述修饰状态的碱基以及不具有所述修饰状态的所述碱基,在转化后形成不同的物质。
368.64.如实施方案1-63中任一项所述的方法,具有所述修饰状态的碱基在转化后基本不发生改变,且不具有所述修饰状态的所述碱基在转化后改变为与所述碱基不同的其它碱基、或在转化后被剪切。
369.65.如实施方案63-64中任一项所述的方法,所述碱基包含胞嘧啶。
370.66.如实施方案1-65中任一项所述的方法,所述修饰状态包含甲基化修饰。
371.67.如实施方案64-66中任一项所述的方法,所述其它碱基包含尿嘧啶。
372.68.如实施方案62-67中任一项所述的方法,所述转化包含通过脱氨基试剂和/或甲基化敏感限制酶转化。
373.69.如实施方案68所述的方法,所述脱氨基试剂包含亚硫酸氢盐或其类似物。
374.70.如实施方案1-69中任一项所述的方法,所述确定修饰状态的存在和/或含量的方法包含,确认具有所述修饰状态的碱基在所述转化后形成的物质的存在和/或含量。
375.71.如实施方案1-70中任一项所述的方法,所述确定修饰状态的存在和/或含量的方法包含,确定具有所述修饰状态的dna区域或其片段的存在和/或含量。
376.72.如实施方案1-71中任一项所述的方法,通过所述荧光pcr方法检测的荧光ct值确定具有所述修饰状态的dna区域或其片段的存在和/或含量。
377.73.如实施方案1-72中任一项所述的方法,通过确认所述dna区域或其片段的修饰状态的存在和/或所述dna区域或其片段相对于参考水平具有更高的修饰状态的含量,确定肝肿瘤的存在、或者有肝肿瘤形成或形成的风险。
378.74.如实施方案1-73中任一项所述的方法,所述方法还包含在确定所述dna区域或其片段的修饰的存在和/或含量之前,扩增待测样本中所述dna区域或其片段。
379.75.如实施方案74所述的方法,所述扩增包含pcr扩增。
380.76.一种核酸,所述核酸包含能够结合如实施方案1-15和58-75中任一项所述的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列;或者所述核酸包含能够结合如实施方案16-75中任一项所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列。
381.77.一种制备核酸的方法,所述方法包含根据如实施方案1-15和58-75中任一项所述的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸;或者所述方法包含根据如实施方案16-75中任一项所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸。
382.78.一种核酸组,所述核酸组包含能够结合如实施方案1-15和58-75中任一项所述的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列;或者所述核酸组包含能够结合如实施方案16-75中任一项所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的序列。
383.79.一种制备核酸组的方法,所述方法包含根据如实施方案1-15和58-75中任一项所述的方法中所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标基因所在dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸组;或者所述方法包含根据如实施方案16-75中任一项所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的修饰状态,设计能够结合所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸组。
384.80.一种试剂盒,包含如实施方案76所述的核酸和/或实施方案78所述的核酸组。
385.81.如实施方案76所述的核酸、如实施方案78所述的核酸组和/或实施方案80所述的试剂盒,在制备确定dna区域或其片段的修饰状态的物质中的应用。
386.82.如实施方案76所述的核酸、如实施方案78所述的核酸组和/或实施方案80所述的试剂盒,在制备疾病检测产品中的应用。
387.83.如实施方案76所述的核酸、如实施方案78所述的核酸组和/或实施方案80所述的试剂盒,在制备确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的物质中的应用。
388.84.如实施方案82-83中任一项所述的应用,所述疾病包含肿瘤。
389.85.如实施方案82-84中任一项所述的应用,所述疾病包含肝肿瘤。
390.86.用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒,在制备用于确认肝肿
瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的物质中的应用,所述用于确定的dna区域包含如实施方案1-15和58-75中任一项所述的方法中所述目标基因所在dna区域或其片段。
391.87.用于确定dna区域修饰状态的核酸、核酸组和/或试剂盒,在制备用于确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的物质中的应用,所述用于确定的dna区域包含如实施方案16-75中任一项所述的方法中所述目标dna区域、或其互补区域、或上述的片段。
392.88.如实施方案87所述的应用,所述疾病包含肿瘤。
393.89.如实施方案87-88中任一项所述的应用,所述疾病包含肝肿瘤。
394.90.如实施方案81和86-89中任一项所述的应用,所述修饰状态包含甲基化修饰。
395.91.如实施方案1-15和58-75中任一项所述的方法中所述目标基因所在dna区域、或其转化而来的区域、或上述的片段的核酸或其组合,在制备用于确认肝肿瘤的存在、评估肝肿瘤形成或形成风险和/或评估肝肿瘤的进展的物质中的应用。
396.92.如实施方案16-75中任一项所述的方法中所述目标dna区域、或其互补区域、或上述的转化而来的区域、或上述的片段的核酸或其组合,在制备用于确认疾病的存在、评估疾病形成或形成风险和/或评估疾病的进展的物质中的应用。
397.93.如实施方案92所述的应用,所述疾病包含肿瘤。
398.94.如实施方案92-93中任一项所述的应用,所述疾病包含肝肿瘤。
399.95.一种储存介质,其记载可以运行实施方案1-75中任一项所述的方法的程序。
400.96.一种设备,其包含实施方案95所述的储存介质。
401.97.如实施方案96所述的设备,还包含耦接至所述储存介质的处理器,所述处理器被配置为基于存储在所述储存介质中的程序执行以实现实施方案1-75中任一项所述的方法。
402.不欲被任何理论所限,下文中的实施例仅仅是为了阐释本技术的方法和用途等,而不用于限制本技术发明的范围。
403.实施例
404.实施例1比较肝癌、癌旁组织及白膜层dna样本甲基化丰度
405.分别从来源于肝脏未见异常的健康人群的白膜层、来源于肝癌患者的癌组织中获得dna样品(其中白膜层样品10个,癌组织各10个),选择白膜层dna作为参考样本是因为血浆游离dna大多数来源于白膜层破裂后释放的dna,其本底背景可以是血浆游离dna该检测位点的一个基础背景信号。按照说明书的要求,用qiagen qiaamp dna mini kit提取白膜层dna,用qiagen qiaamp dna ffpe tissue kit提取组织dna。
406.将上述步骤中获得的dna取20ng样品用亚硫酸氢盐试剂(d5031,zymo research)处理,以获得转化的dna。
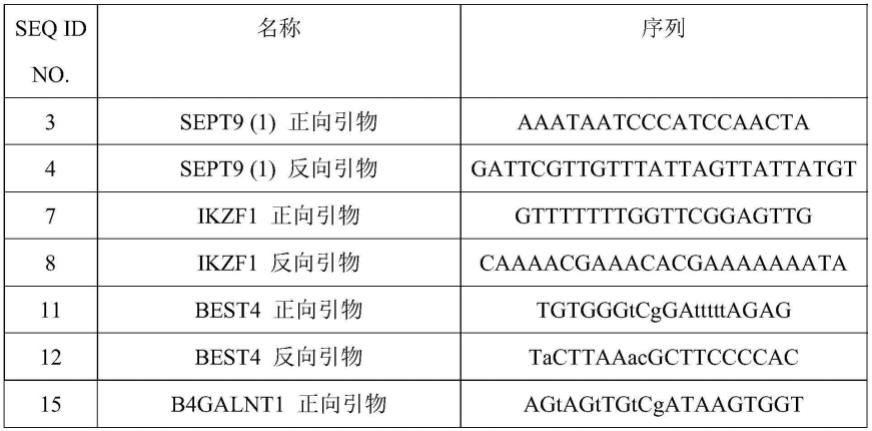
407.通过荧光pcr检测,获得标记物的检测荧光ct值。在荧光pcr反应体系中,每个引物的终浓度为500nm,每个检测探针的终浓度为200nm。pcr反应体系包含:10μl的预扩增稀释产物,2.5μl包含检测位点的引物和探针预混液;12.5μl pcr试剂(probe qpcr master mix(neb)。其中各个甲基化标志物的引物序列见表1,探针序列见表2。pcr反应条件如下:95℃5分钟;95℃30秒,56℃60秒(采集荧光),进行50个循环。使用
abi7500real-time pcr system在相应的荧光通道检测不同的荧光。计算并比较从白膜层、癌旁组织和癌组织获得的样品ct值,未检测到扩增信号的靶点ct值被设定为50。
408.表1引物序列
[0409][0410][0411]
表2检测探针序列
[0412]
seq id no.名称序列2sept9(1)探针ttaaccgcgaaatccgac6ikzf1探针cgccccgtcgccgaat10best4探针cggcgtatttgcgtttattacgt14b4galnt1探针agatttcgcgtatcgcgtttt18grasp探针tgtttttttttcggcgttcgcg22irf4探针atcgtacgtaaggttcggagcga26bend4探针taggacggcgacgacga
29sept9(1a)探针ttgttgcggtcgcggacg32actb探针accaccacccaacacacaataacaaacaca
[0413]
表3样本检测结果汇总
[0414][0415][0416]
上表结果显示,癌组织检测的平均ct值小,代表具有更强的甲基化信号。癌组织中甲基化信号检出率可以远高于白膜层,也代表甲基化信号强。白膜层大多数样本不能检出靶点甲基化信号。这些靶点都可以具备用于血液检测肝癌的潜能。证明所选目标标记物对肿瘤组织具有可行性和特异性。
[0417]
实施例2比较肝癌患者、肝脏未见异常人群血浆样本甲基化信号
[0418]
选取266个肝脏未见异常健康对照血浆、372个肝癌患者术前血浆(其中i期占比为86%)、37例乙肝患者血浆和39例肝硬化血浆样本进行检测。
[0419]
使用商业化qiagen qiaamp circulating nucleic acid kit抽提上述血浆样本中的细胞外游离dna。使用商业化亚硫酸氢盐转化试剂methylcodetm bisulfite conversion kit对抽提出的细胞外游离dna进行亚硫酸盐转化处理,得到转化后的dna。
[0420]
可选地,将上述转化后的dna用于预扩增,用含有表1所示的甲基化标志物特异性引物和内参引物(actb,正向引物和反向引物可以分别如表1所示,探针可以如表2所示)的引物池,以转化后的dna为模板,进行pcr扩增,每条引物的终浓度为100nm。pcr反应体系为10μl转化的dna,包含上述引物的预混液2.5μl;pcr试剂(probe qpcr master mix(neb)12.5μl。pcr反应条件如下:95℃5分钟;95℃30秒,56℃60秒,15个循环。
[0421]
将获得的预扩增产物稀释10倍后用于荧光pcr检测。使用如表1所示的各个甲基化标志物的引物、表2所示的检测探针序列,并且同时对内参基因actb进行检测,作为对照。引物终浓度为500nm,探针终浓度为200nm。pcr反应体系包含:10μl的预扩增稀释产物,包含检测位点的引物和探针预混液2.5μl;pcr试剂(probe qpcr master mix(neb)12.5μl。
[0422]
荧光pcr反应体系与上述实施例相同。pcr反应条件如下:95℃5分钟;95℃15秒,56
℃40秒(采集荧光),50个循环。针对不同基因探针修饰荧光,选择相应检测荧光通道。未检测到扩增信号的靶点ct值被设定为50。结果显示,本技术的各个靶点都可以具备用于血液检测肝癌的能力。
[0423]
在约90%特异性的情况下,检测位点的检测灵敏度统计如表4所示:
[0424]
表4检测位点的检测灵敏度
[0425]
位点特异性灵敏度best494.9%48.8%grasp91.8%40.2%b4galnt192.9%40.2%irf490.3%40.0%sept9(1a)91.8%40.0%sept9(1)92.3%40.0%ikzf198.3%35.1%bend499.1%17.8%
[0426]
上表结果显示,检测位点在对照血浆和肝癌血浆dna甲基化信号对比。证明所选目标标记物对肝癌患者血液样本具有较高的灵敏度。
[0427]
在对靶点进行组合分析时,数据分析可采用对单一靶点设定阳性判读阈值,联合靶点时,任一靶点阳性即样本综合判读为阳性的方式。以此方式验证了:
[0428]
联合sept9(1)、ikzf1,可使肝癌检测灵敏性64.5%,健康对照特异性高达95.9%,同时乙肝患者的阳性率为2.7%,肝硬化患者阳性率为20.5%;
[0429]
联合sept9(1)、ikzf1、best4、b4galnt1,可使肝癌检测灵敏性73.1%,健康对照特异性为91.0%,同时乙肝患者的阳性率为9.1%,肝硬化患者阳性率为20.5%;
[0430]
联合sept9(1)、sept9(1a)、ikzf1、best4、grasp、b4galnt1,可使肝癌检测灵敏性75.8%,健康对照特异性为90.2%,同时乙肝患者的阳性率为16.2%,肝硬化患者阳性率为20.5%;
[0431]
联合irf4、ikzf1、best4、b4galnt1、bend4、sept9(1)时,可使肝癌检测灵敏性76.9%,健康对照特异性为91.0%,同时乙肝患者的阳性率为9.1%,肝硬化患者阳性率为20.5%;以上靶点组合均证实区分健康对照与肝癌的性能优越,且在乙肝和肝硬化患者中特异性良好。
[0432]
前述详细说明是以解释和举例的方式提供的,并非要限制所附权利要求的范围。目前本技术所列举的实施方式的多种变化对本领域普通技术人员来说是显而易见的,且保留在所附的权利要求和其等同方案的范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
