1.本发明涉及生物技术领域,具体涉及一种基于全基因组关联分析筛选大白猪繁殖性能相关候选标记的方法。
背景技术:
2.中国是猪生产大国,猪肉也是人们日常消费的主要肉类,截止2021年我国生猪出栏量保持在4.0亿头以上,面对较大的出栏规模,只要对猪某一个重要性状进行改良就可能带来巨大的经济效益,因此迫切需要鉴定重要性状的主效基因或遗传标记并应用于育种中,可以加快遗传选育进度,提高经济效益。在猪一些重要性状中,繁殖性能特别是产仔数,属于低遗传力的数量性状,其遗传力约为0.1,因此通过传统的育种手段较难获得理想的遗传进展。随着分子生物学的发展,标记辅助选择(marker assisted selection,mas)、基因组选择(genome selection,gs)等技术被逐渐应用到动物育种实践中,比较有效地提高了育种工作的效率,但是缺乏可靠的分子标签这一现状也限制了分子育种效果的发挥。近年来,多基因标记筛选方法发展迅速,全基因组关联分析(genome-wide association study,gwas)已成为主流方法之一,其可在全基因组范围内筛选某个性状的相关变异,通过全基因组关联分析方法鉴定低遗传力性状的遗传标记已成为现代育种工作的常用手段。国内外许多研究人员针对猪重要性状展开gwas,鉴定出大量单核苷酸多态性(single nucleotide polymorphism,snp)标记、数量性状基因座(quantitative trait locus,qtl)和候选基因为猪育种提供必要的分子基础,同时有助于后续相关功能基因的研究和数量性状核苷酸(quantitative trait nucleotide,qtn)的鉴定。目前全基因组关联分析gwas技术在人类疾病和动物复杂性状中寻找候选基因或基因组区域得到广泛应用,关于猪gwas研究主要集中在肉质性状、生长、酮体性状以及疾病易感性等方面,而对大白猪总产仔数(total number of born,nba)、产活仔数(number of born alive,nba)、窝重(litter weight,lw)等重要繁殖性状性能相关遗传标记及候选基因缺乏系统的筛选与深入研究。
技术实现要素:
3.为了筛选大白猪产仔数、有效产仔数、窝重等繁殖性状的候选基因及snp标记,采用affymetrix porcine 55k芯片对695头单系大白母猪开展了基因分型和全基因组关联分析。结果显示:在总产仔数、产活仔数、断奶头数、出生窝重、校正21日龄窝重5个繁殖性状中共发现了7个显著snp和12个候选基因,通过功能注释,最终筛选出了stt3b、tusc3、thrb等作为产仔性状重要候选基因,为后续通过分子育种手段提升丹系大白猪繁殖性能奠定了基础。
4.有鉴于此,本发明实施例提供一种大白猪繁殖性能相关候选标记的筛选方法,主要目的是为其在培育优质高产大白猪的分子育种中的应用。
5.开展分子育种的前提是筛选某一性状的有效遗传标记,方可对其进行进一步的功
能试验及群体验证。因此本技术想通过对大白猪繁殖性能数据进行全基因组关联分析,以期获得繁殖性能相关的重要snps位点及候选基因。
6.为实现本发明的目的,本发明采用的技术方案是:
7.一种基于全基因组关联分析筛选大白猪繁殖性能相关候选标记的方法,所述方法包括以下步骤:
8.采集多个大白猪不同个体的dna样品,进行猪snp芯片基因型检测,将得到的数据
9.基于gemma全基因组关联分析方法进行分析;筛选与繁殖性状显著关联的单核苷酸多态snps以及候选基因,整合基因功能注释筛选大白猪繁殖性能相关的snp分子标记。
10.进一步的,所述繁殖性状包括:总产仔数、产活仔数、健仔数、断奶头数、出生窝重、断奶窝重、校正21日龄窝重。
11.进一步的,全基因组关联测序数据经过质控后,利用gemma软件基于glm模型进行关联分析,挖掘出与大白猪繁殖性能相关的snps,其中glm模型为y=x
α
q
β
k
γ
e,式中,y为性状值,x
α
为基因型值,q
β
为固定效应,k
γ
为随机效应,e为残差。
12.进一步的,群体分层对大白母猪繁殖性能的指标绘制q-q图,来判断关联分析中是否出现偏差和样本群体的分层现象。
13.进一步的,使用r语言绘制snp位点密度分布图、相关性热图、qq plot图以及曼哈顿图,计算遗传多样性分布。
14.进一步的,显著snps基于基因注释在获得全基因组关联分析的显著snps位点后,下载该位点上下游500kb的碱基序列,通过ncbi和ensembl数据库进行序列比对,对显著snps进行候选基因注释。
15.进一步的,剔除没有对应到猪sus scrofa11.1版本参考基因组位置的位点和y染色体上的位点;剔除snp检出率小于90%的位点、不符合哈代-温伯格平衡检验的位点、最小等位基因频率maf《5%的位点以及样本检出率小于90%的个体。
16.进一步的,采集大白猪的耳尾组织提取dna。
17.进一步的,利用紫外分光光度计检测dna浓度,利用1%的琼脂糖凝胶电泳检测dna的质量,将质量合格的dna稀释至50ng/μl进行测定。
18.本发明还提供上述基于全基因组关联分析筛选大白猪繁殖性能相关候选标记的方法在大白猪分子育种中的应用。
19.一种基于全基因组关联分析筛选大白猪繁殖性能相关候选标记的方法,包括以下步骤:
20.(1)试验动物选用的是695头丹系大白母猪(江苏康乐农牧有限公司),出生时期2012年~2016年范围内;
21.(2)样品采集及保存方式主要是剪取少量耳尾组织,置于离心管保存-20℃冰箱中;
22.(3)繁殖性能数据记录主要有第一胎产仔数(总产仔数、产活仔数、健仔数、断奶头数)以及出生窝重、断奶窝重、校正21日龄窝重;
23.(4)基因组dna提取采用dna提取试剂盒,按照说明书方法提起基因组dna,并对其进行完整性、纯度以及浓度的检测,符合要求的保留,不符合要求的淘汰或进行重新提取再检测;
24.(5)所有dna样本进行猪55k snp芯片基因型测定,由北京康普森生物技术有限公司完成;
25.(6)数据处理及统计分析,具体包括:
26.(1)繁殖性状描述性统计分析是通过ibm spss statistics 20.0软件中分析-描述统计分析获得,分析的繁殖性状指标有:总产仔数(tnb)、产活仔数(nba)、健仔数(mhs)、断奶头数(nwp)、出生窝重(balwt)、断奶窝重(wnwt)、校正21日龄窝重(w21),分析结果为有效记录数、最大值、最小值、平均值、标准差以及变异系数;
27.(2)全基因组关联测序数据经过质控后,利用gemma软件基于glm模型进行关联分析,挖掘出与大白猪繁殖性能相关的snps,其中glm模型为y=x
α
q
β
k
γ
e,式中,y为性状值,x
α
为基因型值,q
β
为固定效应(包括胎次、家系、年份),k
γ
为随机效应,e为残差。
28.(3)群体分层对大白母猪繁殖性能的7项指标绘制q-q图,来判断关联分析中是否出现偏差和样本群体的分层现象;
29.(4)使用r语言绘制曼哈顿图,将全基因组显著性阈值定义为0.05/n,其中n是snp的数目,将染色体范围显著性阈值定义为1/n;
30.(5)显著snps基于基因注释在获得全基因组关联分析的显著snps位点后,下载该位点上下游500kb的碱基序列,通过ncbi和ensembl数据库进行序列比对,对显著snps进行候选基因注释。
31.有益效果
32.对于猪生长、肉质和繁殖等重要性状的遗传改良进展直接影响生猪生产的经济效益,而gwas在鉴定这些重要性状的遗传标记或主效基因上具有强大优势,可显著促进遗传选育进展,本研究通过gwas技术系统筛选大白猪繁殖性状相关主效基因及snp,本发明方法方便快捷,准确率高,为后续通过分子育种手段提升大白猪繁殖性能和进一步挖掘其遗传潜力奠定基础。
附图说明
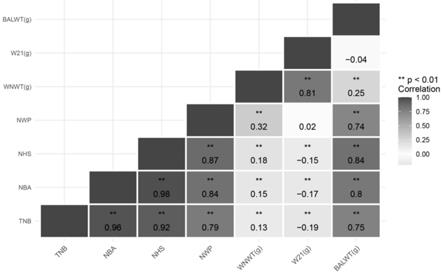
33.图1大白猪不同繁殖性能指标间的相关性分析热图;
34.图2大白猪群体snps密度分布图;
35.图3大白猪群体产仔性状相关snp全基因组关联分析曼哈顿和qq图;
36.图4大白猪群体窝重性状相关snp全基因组关联分析曼哈顿和qq图;
37.图5大白猪显著snps相关候选基因的go功能注释;
38.图6大白猪总产仔数性状相关snps基因型间差异显著性分析;
具体实施方式
39.1.材料与方法
40.1.1实验动物和样品采集
41.本研究中的800头纯种大白母猪群体来自江苏康乐农牧有限公司,采集该群体的耳尾组织,记录其总产仔数(total number of born,nba)、产活仔数(number of born alive,nba)、窝重(litter weight,lw)等性能指标。
42.1.2 55k snp芯片基因分型
43.采用动物组织dna提取试剂盒(上海捷瑞生物工程有限公司)提取耳尾组织的基因组dna。利用紫外分光光度计检测dna浓度,利用1%的琼脂糖凝胶电泳检测dna的质量。将质量合格的dna稀释至50ng/μl左右,送样至北京康普森生物技术有限公司(北京,中国)进行猪55k snp芯片基因型判定。
44.1.3基因型数据的质量控制
45.根据55k snp芯片11.1版本基因组(sus scrofa 11.1)的位置信息,利用plink 1.9对snp芯片检测的结果数据进行质量控制。质控标准为:剔除没有对应到猪sus scrofa11.1版本参考基因组位置的位点和y染色体上的位点;剔除snp检出率小于90%的位点、不符合哈代-温伯格平衡检验(p《10-6)的位点、最小等位基因频率maf《5%的位点以及样本检出率小于90%的个体。
46.1.4全基因组关联分析
47.利用gemma软件(v0.98.1)的gwas常用模型(glm)开展产仔数、有效仔猪数、窝重等繁殖性状的关联分析,筛选出显著效应的snp。glm模型如下:
48.y=x
α
q
β
k
γ
e
49.式中,y为性状值,x
α
为基因型值,q
β
为固定效应(包括胎次、家系、年份),k
γ
为随机效应,e为残差。
50.1.5候选基因鉴定及功能注释
51.选择各性状全基因组水平上显著snp位点上下游500kb作为候选区域,使用biomart(http://www.ensembl.org/biomart/martview)查询候选区域内编码基因,并在david在线网址(https://david.ncifcrf.gov/)进行go功能注释。
52.1.6r语言绘图及统计分析
53.利用使用r软件(v3.5.0)绘制snp位点密度分布图、相关性热图、qq plot图以及曼哈顿图。使用excel分析表型数据的平均值、基因型频率及相关系数,并计算遗传多样性分布(he:杂合度;ne:有效等位基因数;pic:多态信息含量)。
54.2结果与分析
55.2.1大白猪不同繁殖性状指标统计分析
56.由表1可知,对695头大白母猪总产仔数(tnb)、产活仔数(nba)、健仔数(mhs)、断奶头数(nwp)、出生窝重(balwt)、断奶窝重(wnwt)、校正21日龄窝重(w21)等指标平均值、最大值、最小值以及变异系数进行统计分析。此外,不同繁殖性能指标相关性分析显示(图1),balwt与tnb、nba、mhs、nwp存在明显的相关性。
57.表1.大白猪群体繁殖性能指标统计分析
[0058][0059]
注:tnb=总产仔数,nba=产活仔数,nwp=断奶头数,nhs=健仔数,balwt=出生
窝重(kg),wnwt=断奶窝重(kg),w21=校正21日龄窝重(kg).
[0060]
2.2大白猪不同繁殖性状gwas分析
[0061]
基于affymetrix porcine 55k snp芯片筛选出64812个snps用于下一步分析,发现这些snps均分布在猪不同染色体上且分布规律相似(图2),使用gwas常用模型glm统计分析,在5个繁殖性状总产仔数、产活仔数、断奶头数、出生窝重、校正21日龄窝重中分别发现了2、1、1、1、2个snps位点,同时发现12个候选基因(图3、图4、表2)。显著snps等位基因频率计算以及遗传多样性分布显示,一些snps(rs81374271,rs341645361,rs80851490)在大白猪群体中表现出极端偏态分布,而其他snps(rs81476831,rs81443336,rs81466107,rs329374863)表现出中度多态性(0.25《pic《0.5),可能作为今后重点关注的候选标记。
[0062]
表2.大白猪群体繁殖性能相关显著snps位点信息
[0063][0064]
表3.显著snps位点基因型频率以及遗传多样性分布
[0065][0066]
2.3繁殖性状相关候选基因鉴定及go功能注释
[0067]
为了进一步鉴定大白猪繁殖性能相关候选基因,发现位于7个显著snps附近或者位点上的候选功能基因有12个,分别为stt3b、pkdcc、tusc3、thrb、rarb、dchs2、sfrp2、tbx15、spag17、uroc1等(表2)。go功能注释显示,功能基因主要参与蛋白水解作用,肽酰-氨基酸和修饰,类固醇激素受体活性等(图5)。对于总产仔数性状而言,存在2个snps位点(rs81466107、rs329374863),不同基因型间总产仔数指标存在显著差异(图6),tusc3、thrb、rarb作为重要候选基因;对于出生窝重性状而言,dchs2、sfrp2作为重要候选基因;对于产活仔数而言,stt3b作为重要候选基因。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。