一种获得线性质粒dna断裂位置序列信息的方法
技术领域
1.本发明涉及一种获得线性质粒dna断裂位置序列信息的方法,可以有效检测线性质粒dna的断裂位置,属于分子生物学技术领域。
背景技术:
2.质粒dna有三种构象,大部分质粒dna通常呈现超螺旋的sc构型,其两条多核苷酸链均保持着完整的环形结构,也称为共价闭合环形dna(cccdna);当两条多核苷酸链中只有一条保持着完整的环形结构,另一条链出现有一至数个缺口时,也称为开环dna(ocdna),即oc构型;当质粒dna经过限制性内切酶等核酸酶切割之后,发生双链断裂形成线性分子(idna),即l构型。在琼脂糖凝胶电泳时,这三种不同状态的质粒dna泳动速率也不相同,超螺旋dna泳动速率最快,线性质粒dna其次,开环质粒dna最慢。
3.质粒dna(例如:puc19质粒)常作为检测未知蛋白质的核酸酶活性的作用底物,当被具有核酸酶活性的蛋白质处理后,会改变质粒dna的电泳特性,超螺旋形式的dna减少,而线性和开环形式的dna增多,因此根据线性和开环形式的dna的量可以验证未知核酸酶的活性高低。但是采用该方法仅能验证未知核酸酶的酶活,不能得出其识别位点的序列信息。目前常通过生物信息学软件预测核酸酶的识别位点,如www.zincfingers.org网站提供的多聚锌指识别序列设计软件,然而软件预测依据的是未知核酸酶的氨基酸序列信息,根据氨基酸序列信息(其仅决定了蛋白质的一级结构),还无法直接确定其所编码的蛋白质的最终功能,因此基于氨基酸序列分析推测的识别位点的准确性很难确定;仍需要经过实验验证。文献(胡晓倩,唐欣昀.螺旋藻胞内核酸酶种类初探[j].宿州学院学报,2006,021(003):128-129,123.)报道了一种螺旋藻胞内核酸酶,采用螺旋藻胞内核酸酶酶切底物λ-dna和质粒8760,然后电泳检测酶切产生的多个dna条带的大小,通过比较分析多个dna条带的电泳图片与p8760相关酶切图谱,得到螺旋藻胞内核酸酶的识别位点;然而该方法较复杂;且当底物质粒中具有多个相邻的酶切位点时,会导致酶切产生的dna片段大小较接近,易得出错误结果。因此,为了更好地研究未知核酸酶的功能特性,准确获得识别位点的序列信息至关重要。
技术实现要素:
[0004]
针对现有技术的缺陷,本发明提供了一种获得线性质粒dna断裂位置序列信息的方法,利用该方法可以准确推断出线性质粒的断裂位置以及断裂位置附近的dna序列信息,用于未知核酸酶的识别位点测定,具有操作简单快速、结果准确、成本低、耗时短等优点。
[0005]
为实现上述目的,本发明采用的技术方案是:
[0006]
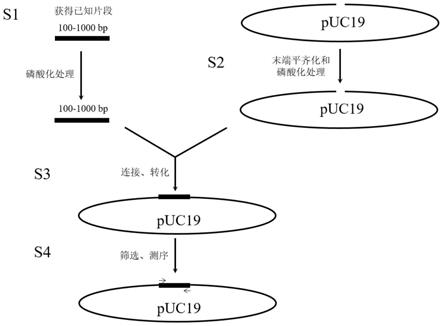
一种获得线性质粒dna断裂位置序列信息的方法,流程图如附图1所示,包括以下步骤:
[0007]
s1.对已知序列的dna片段进行磷酸化处理;
[0008]
s2.对待检测的线性质粒进行末端平齐化和磷酸化处理;
[0009]
s3.将步骤s1中处理得到的dna片段和步骤s2中处理得到的线性质粒进行平端连接得到连接产物,并将连接产物转化到宿主菌中;
[0010]
s4.筛选阳性重组子,再利用测序引物分别向连接位点方向进行测序,根据已知序列与紧邻的质粒序列得出线性质粒的断裂位置与断裂方式;
[0011]
所述测序引物为已知序列的dna片段上的任一段序列,且所述测序引物与所述连接位点间的距离为50~1000bp,以所述测序引物的第一个碱基距离所述连接位点的碱基起止。
[0012]
该方法的原理为:采用未知核酸酶切割连接产物(例如:puc19质粒),得到线性质粒,线性质粒的断裂位置即为未知核酸酶的识别位点。为获得线性质粒断裂位置的序列信息,对待检测的线性质粒纯化后进行末端平齐化和磷酸化处理,然后与一段经过磷酸化处理的已知序列的dna片段进行平端连接,得到连接产物-环状质粒,再将连接产物转化到宿主菌如大肠杆菌中,挑取单克隆,用已知序列的dna片段上的扩增引物进行pcr扩增检测,以筛选出阳性重组子,然后再用测序引物分别向连接位点(线性质粒与dna片段的连接位置,此位置也为线性质粒的断裂位置)方向进行测序,根据已知序列与紧邻的质粒序列信息,经过序列比对分析,即可推断出线性质粒dna的断裂位置与断裂方式。
[0013]
优选的,所述测序引物距离所述连接位点的距离为200~600bp。在该距离范围内,可保证断裂位置的序列信息位于测序结果的200~600bp范围内,结果更精确,可避免目前一代测序技术缺陷导致的误差,如一代测序对于0~100bp和800bp以后碱基的测序结果不准确,存在一定的错误;因此,将测序引物与连接位点的距离设定为200~600bp,可避开上述区域。
[0014]
优选的,所述已知序列的dna片段的长度为100~1000bp。已知序列的dna片段可根据需要选择,但一般以100~1000bp左右为宜,片段过长或过短都会降低连接效率。进一步的,可在已知序列的dna片段中加入与待检测的线性质粒不同的抗性基因(例如:氨苄抗性基因),以提高重组阳性率。
[0015]
采用上述的技术方案:当已知序列的dna片段的长度为100~600bp时,所述测序引物可以与筛选所述阳性克隆子用的扩增引物相同,也可以不同;而当已知序列的dna片段的长度为600~1000bp时,所述测序引物位于所述扩增引物的下游;以此保证所述测序引物距离所述连接位点的距离为200~600bp。
[0016]
进一步优选的,所述已知序列的dna片段的长度为400~600bp。此时可以将筛选阳性克隆子用的扩增引物和检测断裂位置用的测序引物设定为同一对,以简化程序,使操作更简单,并降低成本。
[0017]
优选的,步骤s1~s2处理后均需进行dna纯化。
[0018]
优选的,步骤s1中所述磷酸化处理采用多聚核苷酸激酶。
[0019]
优选的,步骤s2中所述末端平齐化和磷酸化处理采用快速末端平齐化试剂盒,将不平齐dna的5'或3'突出末端转变为带有5'磷酸的平末端。快速末端平齐化试剂盒是酶混合液,其中包含dna聚合酶和多聚核苷酸激酶,t4 dna聚合酶同时具有3
′→5′
外切酶活性和5
′→3′
聚合酶活性,可使dna末端平齐化;当末端为5'端突出末端时,t4 dna聚合酶可以利用其5'
→
3'dna聚合酶活性将末端补平;当末端为3'端突出末端时,t4 dna聚合酶可以利用其3'
→
5'dna外切酶活性将末端削平;t4多聚核苷酸激酶可将平末端dna的5
′
端磷酸化,以
用于连接反应。
[0020]
优选的,步骤s3中所述平端连接处理采用dna连接酶。
[0021]
优选的,步骤s4中所述筛选阳性克隆子的具体步骤为:挑取单克隆,用已知序列的dna片段上的扩增引物进行pcr扩增检测,得到阳性重组子后。
[0022]
本发明的优点在于:利用测序引物分别向连接产物的连接位点方向进行测序,使得测序结果覆盖线性质粒的断裂位置,然后根据已知序列与紧邻的质粒序列信息,经过序列比对分析,即可准确推断出线性质粒的断裂位置、断裂方式以及断裂位置附近的dna序列信息,因而可用于未知核酸酶的识别位点测定,具有操作简单快速、结果准确、成本低、耗时短等优点。
附图说明
[0023]
为了更清楚地说明本发明实施例中的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
[0024]
图1为一种获得线性质粒dna断裂位置序列信息的方法流程图;
[0025]
图2为实施例中检测puc19线性质粒dna断裂位置信息的方法流程图;
[0026]
图3为实施例中检测到的线性puc19质粒的三种断裂方式;图3-a为puc19质粒断裂产生5’突出末端;图3-b为puc19质粒断裂产生3’突出末端;图3-c为puc19质粒断裂产生平末端。
[0027]
图4为实施例中puc19质粒断裂产生三种末端断裂方式的测序结果;图中标注的加粗序列分别对应测序反应得出的断裂质粒上的序列,图中标注的斜体序列为末端补平的序列。
具体实施方式
[0028]
下面将结合本发明中的附图,对本发明的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动条件下所获得的所有其它实施例,都属于本发明保护的范围。
[0029]
实施例
[0030]
本实施例中采用一种未知功能的核酸酶,它可将超螺旋质粒dna处理为线性和开环质粒,为获得线性质粒dna断裂位置的序列信息,先将线性质粒切胶回收,再进行末端平齐化和磷酸化处理,然后与一段经过磷酸化处理已知序列的dna片段进行平端连接,得到连接产物,再将连接产物转化到再转化到大肠杆菌,挑取单克隆,用已知序列的dna片段上的扩增引物进行pcr扩增检测,以筛选出阳性重组子,然后选取10-20个阳性重组子再用测序引物分别向两端测序,得到与已知序列衔接处的序列信息,再与质粒进行序列比对,即可准确得出线性质粒的断裂位置的序列信息。本实施例流程如附图2所示,具体步骤如下:
[0031]
s1.对已知序列的dna片段进行磷酸化处理;
[0032]
s11.pcr扩增已知序列的dna片段:选取一段已知序列的dna片段,长度为600bp,采
用扩增引物f1、r1,进行pcr扩增得到扩增产物,然后纯化扩增产物,具体使用neb生产的dna纯化试剂盒(#t1030)进行dna纯化;
[0033]
s12.已知序列的dna片段的磷酸化处理:采用neb生产的t4多聚核苷酸激酶(#m0201)进行磷酸化处理,反应体系如表1所示:
[0034]
表1t4多聚核苷酸激酶磷酸化反应体系
[0035][0036]
磷酸化反应在37℃下进行,反应时间为30min,然后进行dna纯化;
[0037]
s2.对待检测的线性质粒进行末端平齐化和磷酸化处理;将与未知核酸酶反应后的puc19质粒进行电泳跑胶,再切胶回收,然后进行dna纯化;再采用neb生产的快速末端平齐化试剂盒(#e1201)进行末端平齐化和磷酸化处理,反应体系如表2所示:
[0038]
表2末端平齐化和磷酸化反应体系
[0039][0040][0041]
反应在室温下进行,反应时间为15min,然后进行dna纯化。
[0042]
s3.将步骤s1中处理得到的dna片段和步骤s2中处理得到的线性质粒通过neb生产的高浓度t4 dna连接酶(#m0202)进行平端连接,反应体系如表3所示,反应在室温下进行,反应时间为10min,得到连接产物-环状质粒,之后将连接产物转化大肠杆菌dh5α中;
[0043]
表3平端连接反应体系
[0044]
[0045]
s4.挑取步骤s3中的单克隆,用扩增引物f1、r1引物对进行pcr扩增后跑胶检测,筛选出阳性重组子,再利用测序引物分别向连接位点方向进行测序,根据已知序列与紧邻的质粒序列得出线性质粒的断裂位置与断裂方式;本实施例中,测序引物选择为f1、r1引物对,测序结果如附图3所示。本领域的技术人员应该知道,所述测序引物可以为已知序列的dna片段上的任一段序列,只要使用该测序引物得到的测序结果能够覆盖所述线性质粒的断裂位置,且能够保证该断裂位置附近的碱基序列的准确性,则该测序引物皆可实现本发明的技术方案,即,本实施例的测序引物的位置和序列信息不能作为对本发明保护范围的限定。
[0046]
根据附图3可知,本实施例使用的未知核酸酶,其识别位点无特异性,可能产生三种断裂情况,分别是5’突出末端、3’突出末端和平末端。在末端平齐化时,不同的断裂方式也会有不同的平齐化方式,5’突出末端时,线性质粒dna会被补齐;3’突出末端,线性质粒dna会被削平,平末端则无变化;这三种情况分别对应于附图3中的a~c,根据所获得的线性质粒与已知序列衔接处的序列信息加以确认。通过挑取不同的克隆去测序,结果发现共检测到了三种线性puc19质粒的断裂方式,分别为断裂产生5’突出末端、3’突出末端和平末端,如附图3所示;图3-a中,5’突出末端由于补平两端边界会产生重复序列;图3-b中,3’突出末端由于削平两端边界会产生序列缺失;图3-c中平末端的序列不发生变化。附图4示例性的展示了puc19质粒断裂产生三种末端断裂方式的测序结果。根据上述结果,可以得到本实施例中所用未知核酸酶的切割位点有三个,分别是:
[0047]
1)、正链5
’…
gcca/gtga
…3’
,负链5
’…
attc/actg
…3’
,切割造成dna双链断裂,断口为5’两个核苷酸突出(见附图4-a);
[0048]
2)、正链5
’…
agcg/ggtg
…3’
,负链5
’…
accc/gctg
…3’
,切割造成dna双链断裂,断口为3’一个核苷酸突出(见附图4-b);
[0049]
3)、正链5
’…
gaaa/cctg
…3’
,负链5
’…
cagg/tttc
…3’
,切割造成dna双链断裂,断口平齐无突出(见附图4-c)。
[0050]
采用本发明的方法检测线性质粒dna断裂位置的序列信息时,检测结果准确,不同的断裂方式都可用本方法检测出来,操作简单快速。
[0051]
尽管已经示出和描述了本发明的实施例,对于本领域的普通技术人员而言,可以理解在不脱离本发明的原理和精神的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由所附权利要求及其等同物限定。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。