1.本发明属于生物信息学和生物医药领域,具体涉及一种用于病毒基因组变异分析、监测方法和系统。
背景技术:
2.2019年底,由sars
‑
cov
‑
2引起的新冠疫情爆发,给世界公共卫生、经济、社会可持续发展带来了巨大危害及挑战。随着sars
‑
cov
‑
2在人群中的广泛流行,其基因组不断进化和变异。一些突变会赋予病毒新的遗传特征,如影响感染性和疫苗疗效等。前期相关研究表明,一些突变位点会影响病毒的感染特性以及对中和抗体甚至疫苗的免疫保护作用。
3.然而,疫苗的开发周期较长,从sars
‑
cov
‑
2出现到第一批在疫苗投入使用,大约历经了一年。目前,即将投入使用的疫苗是基于疫情初期的毒株开发的。在sars
‑
cov
‑
2大流行的这一年内,sars
‑
cov
‑
2基因组上经历了较多的变异,其中一些突变位点如s蛋白上的可导致感染性增强的d614g成为了优势变异且在全世界大范围流行。
4.因此,在病毒性疫情大流行期间,基于大尺度基因组序列数据来对病毒进行变异研究和监测,筛选并评估具有潜在功能的突变,对疫情的防控是至关重要的。
技术实现要素:
5.基于此,本发明通过序列比对后进行全基因注释获得编码基因信息,再进行翻译比对并根据遗传密码子表分析突变类型,获得基因组的突变位点、类型及频率,统计高频率突变位点,通过采集不同时间和区域的病毒株,分析高频率突变位点在时间和空间上的分布特征,并筛选位于免疫表位及其附近的高频率突变位点,以实现病毒基因组变异分析及监测。
6.本发明提供了一种用于病毒基因组变异分析方法,所述分析方法具体包括:
7.获取参考基因序列和待分析基因组序列,将所述待分析基因组序列进行质量控制后获得测序质量良好的全基因组序列集;
8.将所述测序质量良好的序列集进行多序列比对,获得alignment序列文件;
9.将所述alignment序列文件以字符串完全匹配方法匹配编码基因的起始字符串,并返回编码基因在全基因组序列中的位置下标,并以alignment序列文件中每个基因对应的位置下标,生成基因位置表;
10.根据所述基因位置表,循环遍历每条序列,截取每条序列对应的编码基因片段并存储;
11.根据所述编码基因序列进行翻译成氨基酸序列,并对氨基酸序列进行多序列比对,获得对齐的氨基酸序列文件,与对齐前的编码基因序列进行匹配,采用“密码子出现的顺序”作为映射关系进而进行“回译”,将对齐的氨基酸序列“回译”成对齐的核苷酸;
12.将所述对齐的核苷酸序列以每三个碱基扫描方式遍历序列的每个密码子位点,识别和记录序列中存在连续三个碱基插入位点的位置
“‑‑‑”
,标记为“插入位点”,并删除“插
入位点”,获取不含插入位点的突变分析序列;
13.将参考序列对应的密码子和/或其对应的氨基酸与所述突变分析序列的密码子和/或翻译氨基酸按照预设突变分析方法进行分析获得待分析基因组序列突变位点以及变异类型。
14.进一步的,所述质量控制具体包括:
15.循环扫描每条序列,统计位置碱基的数量,当序列中含有10个以上的连续未知碱基,则将对应的序列进行删除。
16.进一步的,所述预设突变分析方法具体包括:
17.将参考序列对应密码子变量名记作qury_seq_conding,突变分析序列的密码子变量名记作s;
18.如果s和qury_seq_conding相同,当二者均不为字符串
“‑‑‑”
,不变位点数加1,当二者都为字符串
“‑‑‑”
,则忽略;
19.如果s和qury_seq_conding不同,其中有一个为字符串
“‑‑‑”
,当qury_seq_conding为字符串
“‑‑‑”
,标记为碱基插入位点并计数,当s为第字符串
“‑‑‑”
,标记为碱基删除位点并计数;
20.如果s和qury_seq_conding不同,且都不为字符串
“‑‑‑”
,将二者翻译成氨基酸translate_s和translate_qury_seq,当氨基酸相同,则标记为同义突变并计数,当translate_s为字符“?”,标记为未知突变并计数,当translate_s为字符“*”,标记为提前终止并计数,其他属于非同义突变位点并计数;
21.所述非同义突变位点,比较并记录氨基酸性质的变化。
22.本发明实施例还提供了一种用于病毒基因组变异的监测方法,所述监测方法具体包括:
23.采集不同时间和区域的待分析病毒基因组,按照上述的突变分析方法进行分析获得所有的突变位点和突变类型;统计各突变位点的突变频率,将所述突变频率高于预设频率阈值对应的突变位点记为高频突变位点;
24.获取所述高频突变位点对应的基因组序列的采集时间计算高频突变在所有基因组中的占比,获得折线图,并进行拟合,从而获得具有增长趋势的突变位点和突变毒株;
25.获取所述高频突变位点对应的区域,构建聚类热图,用于不同地区疫苗设计提供参考;
26.针对所述高频突变位点进行免疫表位筛选,所述免疫表位筛选具体包括:b细胞表位预测和t细胞表位预测。
27.进一步的,所述预设频率阈值大于等于0.5。
28.进一步的,所述b细胞表位预测具体包括:
29.连续b细胞表位预测:预测氨基酸序列的线性b细胞表位,将存在6
‑
25个氨基酸的线性表位肽进行抗原评估,获得评分超过0.5对应的表位肽序列为连续b细胞表位;
30.离散b细胞表位预测:获取氨基酸序列的三级结构,并预测所述三级结构中的不连续b细胞表位,获得倾向性评分超过
‑
3.7的为离散b细胞表位。
31.进一步的,所述t细胞表位预测具体包括:
32.mhc
‑
i(cd8 t细胞)结合表位预测:根据人类hlai类等位基因预测mhc
‑
i结合表位,
获取评分超过0.85且vaxijen得分超过0.5的表位;所述人类hlai类等位基因为hla
‑
a01:01,hla
‑
a02:01,hla
‑
a03:01,hla
‑
a11:01,hla
‑
a24:02,hla
‑
b07:02,hla
‑
b08:01,hla
‑
b40:01;
33.mhc
‑ⅱ
(cd4 t细胞)结合表位预测:根据7个人类drb类等位基因预测mhc
‑ⅱ
结合表位,获得adjustedrank值低于1且vaxijen评分超过0.5的结合表位,所述人类drb类等位基因为drb103:01,drb107:01,drb115:01,drb301:01,drb302:02,drb401:01和drb501:01。
34.基于同一发明构思的,本发明实施例还提供了一种用于病毒基因组变异分析、检测系统,所述系统具体包括:
35.筛选模块,用于筛选待分析基因组序列,将所述待分析基因组序列进行质量控制获得测序质量良好的全基因组序列集;
36.序列比对模块,用于将所述测序质量良好的基因组序列集进行多序列比对,获得alignment序列文件;
37.基因组注释模块,用于将所述alignment序列文件以字符串完全匹配方法匹配编码基因的起始字符串,并返回编码基因在全基因组序列中的位置下标,并以alignment序列文件中每个基因对应的位置下标,生成基因位置表;根据所述基因位置表,循环遍历每条序列,截取每条序列对应的编码基因片段并存储;
38.翻译比对模块,用于根据所述编码基因序列进行翻译成氨基酸序列,并对氨基酸序列进行多序列比对,获得对齐的氨基酸序列文件,与对齐前的编码基因序列进行匹配,采用“密码子出现的顺序”作为映射关系进而进行“回译”,将对齐的氨基酸序列“回译”成对齐的核苷酸;
39.插入位点标记模块;将所述对齐的核苷酸序列以每三个碱基扫描方式遍历序列的每个密码子位点,识别和记录序列中存在连续三个碱基插入位点
“‑‑‑”
的位置,标记为“插入位点”,获得并删除“插入位点”,获取不含插入位点的突变分析序列;
40.编码基因突变分析模块,用于将参考序列对应的密码子和/或其对应的氨基酸与所述突变分析序列的密码子和/或翻译氨基酸按照预设突变分析方法进行分析获得待分析基因组序列突变位点以及变异类型;
41.监测模块,用于获取不同采集时间和区域的突变分析结果,统计高频率突变位点,并分析所述高频率突变位点在时间和区域上的规律以及免疫表位情况。
42.进一步的,所述监测模块具体包括:
43.高频率突变位点分析子模块,用于统计各突变位点的突变频率,将所述突变频率高于预设频率阈值对应的突变位点记为高频突变位点;
44.增长趋势监测子模块,用于获取所述高频突变位点对应的基因组序列的采集时间计算高频突变在所有基因组中的占比,获得折线图,并进行拟合,从而获得具有增长趋势的突变位点和突变毒株;
45.地域监测子模块,获取所述高频突变位点对应的区域,构建聚类热图,用于不同地区疫苗设计提供参考;
46.免疫表位筛选子模块,针对所述高频突变位点进行免疫表位筛选,所述免疫表位筛选具体包括:b细胞表位预测和t细胞表位预测。
47.有益效果:
48.该方法通过序列比对后以字符串完全匹配方式匹配编码基因的启示字符串并返回编码基因在全基因组序列中的位置下标,获得基因位置表,可以实现大量全基因组序列的快速批量注释,并采用编码基因翻译比对方法保留对齐后编码基因对应的核苷酸序列中的遗传信息,进行多种突变类型的分析,实现了病毒基因组变异的全面分析,并经过数据统计和免疫表位筛选进行突变监测,为疫苗设置提供了数据参考。
49.应当理解的是,以上的一般描述和后文的细节描述仅是示例性和解释性的,并不能限制本公开。
附图说明
50.为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
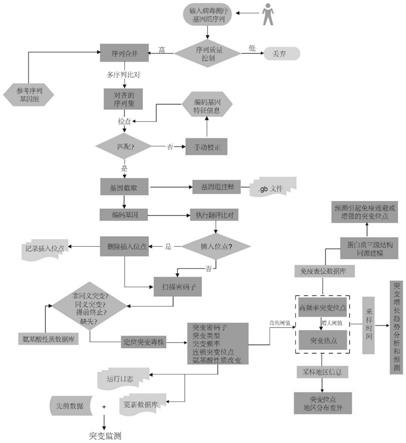
51.图1为本发明实施例提供的一种用于病毒基因组突变分析、监测方法的流程图;
52.图2为本发明实施例提供的新冠病毒的s基因序列(s_codon.fasta)示例的碱基位点突变频率分布图;
53.图3为本发明实施例提供的d614g a222v,d614g l18f a222v突变位点在种群中的突变频率趋势图;
54.图4为本发明实施例提供的40种高频突变位点的地区差异分布图。
具体实施方式
55.为了使本发明的目的、技术方案及优点更加清楚明白,以下结合附图及实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅用以解释本发明,并不用于限定本发明。
56.如图1所示,在本发明实施例中,提出了一种用于病毒基因组突变分析、监测方法的流程图,针对于突变分析过程具体包括以下步骤:
57.步骤s101,获取参考基因序列和待分析基因组序列,将所述待分析基因组序列进行质量控制后获得测序质量良好的全基因组序列集。在本发明实施例中,对获取的基因组序列循环扫描,统计未知碱基(n字符串)的数量,如果序列中含有10个及以上连续的未知碱基(n),则将该条序列剔除掉,不纳入后续的分析。过滤掉一些测序质量不好的序列。
58.步骤s102,将所述测序质量良好的基因组序列集进行多序列比对,获得alignment序列文件;在本发明实施例中,采用mafft程序进行多序列比对,获得alignment序列文件。
59.步骤s103,将所述alignment序列文件以字符串完全匹配方法匹配编码基因的起始字符串,并返回编码基因在全基因组序列中的位置下标,并以alignment序列文件中每个基因对应的位置下标,生成基因位置表,根据所述基因位置表,循环遍历每条序列,截取每条序列对应的编码基因片段并存储。
60.在本发明实施例中,以字符串完全匹配的方式匹配编码基因的起始字符串,返回编码基因在全基因组序列中的位置下标,进而对基因组进行序列注释。在进行注释前,需要先运行检查机制,判断用于识别的字符串是否在序列中存在。判断模式为:如果字符串存
在,则继续运行下一步,否则输出提示信息,提示更换字符串。基于用户提供的参考序列的每个编码基因的信息文件,该文件包含基因名称,起始字符串,结束字符串,分别以逗号隔开。使用find函数,基于完全匹配(两个字符串相等)的方式,获得alignment序列文件中每个基因的对应的下标值(在序列中的坐标),生成基因位置表。再循环遍历每条序列,基于基因位置表,截取出每条序列对应的编码基因片段,分别储存以每个编码基因名称命名的文件里。同时,对于每条序列,都输出一个*.gb格式的注释文件,里面包含了该全基因组序列的字符串信息,每个基因的基因名称和起始位置。该步骤可以实现大量全基因组序列数据的快速注释。
61.步骤s104,根据所述编码基因序列进行翻译比对,先将核苷酸序列翻译成氨基酸序列,并对氨基酸序列进行多序列比对,获得对齐的氨基酸序列文件,再“回译”成对齐的核苷酸序列。该比对过程可采用mafft程序。
62.步骤s105,根据所述的对齐的核苷酸序列。编码基因进行翻译比对后,对齐的序列文件中可能存在的“插入位点”(作为突变的一种),会在参考序列中引入gap(
‑
),因为gap的位置不确定性,在不同次分析中会影响后续对突变位置的统一标记,因此,需要将“插入位点”单独进行统计和记录,并删除插入位点再用于后续分析。
63.将所述对齐的核苷酸序列以每三个碱基扫描方式遍历序列的每个密码子位点,识别和记录序列中存在连续三个碱基插入位点gap(
‑‑‑
)的位置,标记为“插入位点”,并返回插入位点处其他非参考序列的密码子和翻译后的氨基酸,同时返回不含插入位点的突变分析序列。
64.步骤s106,将参考序列对应的密码子和/或其对应的氨基酸与所述突变分析序列密码子和/或翻译氨基酸按照预设突变分析方法进行分析获得待分析基因组序列突变位点以及变异类型。
65.在本发明实施例中,使用两个嵌套的循环,外循环的迭代数为密码子位点数量(总碱基数量除以3),内循环迭代数为序列的条数。对于每个内循环,每条序列都与序列集中的第一条序列(参考序列)进行比较,从而识别5种不同的突变类型,包括同义替换、非同义替换、提前终止、碱基插入和删除。具体识别模式和比较方法为:
66.1)将参考序列对应密码子变量名记作qury_seq_conding,用于突变分析的序列的密码子变量名记作s;
67.2)如果s和qury_seq_conding相同。a.如果qury_seq_conding和s都不为
“‑‑‑”
,不变位点数加1;b.如果s和qury_seq_conding都为
“‑‑‑”
,则忽略;
68.3)如果s和qury_seq_conding不相同,其中有一个为
“‑‑‑”
。a.如果qury_seq_conding为
“‑‑‑”
,标记为碱基插入位点并计数;b.如果s为"
‑‑‑
",标记为碱基删除位点并计数;
69.4)如果s和qury_seq_conding不相同,且两个都不为
“‑‑‑”
。将s和qury_seq_conding分别翻译成氨基酸,记作translate_s和translate_qury_seq。a.如果translate_s和translate_qury_seq相同,标记为同义突变并计数;b.如果translate_s为字符“?”,标记为未知突变并计数;c.如果translate_s为字符“*”,标记为提前终止并计数;d.如果都不符合上述条件,则标记为非同义突变并计数。
70.5)对于非同义突变位点,调用bioaider里内置的氨基酸性质表,判断并记录氨基
酸的性质是否改变,包括极性和带电性。
71.6)对于上述突变分析步骤,写入生成详细的日志文件(log文件)以及汇总文件(summary文件)。log文件记录每条序列每个位点上的变异,汇总文件记录突变的密码子位点,突变的碱基,突变类型,突变频率,突变氨基酸是否改变等汇总信息。
72.7)根据用户指定的突变频数分布表,生成同义或非同义非同义突变位点对应的频数分布图,以便了解序列数据集整体的变异情况,并以位图*.jpg和矢量图.pdf的格式输出。
73.对于关联突变分析,可逐一扫描所有核苷酸位点与参考序列进行对比,汇总每条序列所有的突变位点信息,生成汇总报告(summary文件),包括突变位点及突变频率、突变热点(高于指定的突变频率阈值),并生成详细的日志记录文件(log文件),详细记录每条序列每个位点上的变异,便于查询。
74.将上述编码基因进行关联突变分析,以每条序列作为一个整体标记所有的突变位点,如“l18f d614g a222v”,每条序列生成一个记录,并对它们进行统计得到这些关联突变位点的突变频率,最后根据指定的突变频率阈值输出高于阈值的突变热点。
75.所述监测方法具体包括:
76.采集不同时间和区域的待分析病毒基因组,按照上述突变分析方法进行分析获得所有的突变位点和突变类型;统计各突变位点的突变频率,将所述突变频率高于预设频率阈值对应的突变位点记为高频突变位点。如图2所示,上述基于密码子方式比对后的新冠病毒的s基因序列(s_codon.fasta)示例的碱基位点突变频率分布图。经关联突变分析获得40中突变频率大于0.1%的关联突变位点,如表1所示。
77.表1突变频率大于0.1%的关联突变位点
[0078][0079][0080]
读取summary文件中突变位点汇总信息,默认选取突变频率(突变序列的数量占
比)高于0.5%的突变位点作为用于时间分析的高频率突变位点。根据序列的采样时间,默认按月份为时间间隔,计算在每个时间间隔内这些高频率突变在群体中的占比,绘制折线图。此外,采用线性回归的方法进行拟合,计算回归系数,从而识别出具有增长趋势的突变位点和突变毒株。如图3示例中d614g a222v,d614g l18f a222v突变位点在种群中的突变频率趋势图。
[0081]
读取summary文件中突变位点汇总信息,默认选取突变频率(突变序列的数量占比)高于0.1%的突变位点作为用于地区分布分析的高频率突变位点。构建高频率突变位点及其在每个地区的数量矩阵,行为地区,列为突变位点,构建聚类热图。聚类热图采用欧式距离对行和列进行聚类,聚集在一起的行表示这些地区具有相似流行的突变毒株,聚集在一起的列表示这些突变位点具有相似地区分布。因为疫苗设计需要充分考虑现有毒株的流行情况,因此,对毒株基于高频率突变位点进行地理聚类,能为不同地区的疫苗设计提供参考,上述示例中的40种高频突变位点的地区差异分布图如图4所示。
[0082]
对所述突变频率高于0.5%的高频突变位点进行免疫表位筛选,所述免疫表位筛选具体包括:b细胞表位预测和t细胞表位预测。
[0083]
在本发明实施例中,连续b细胞表位预测:使用iedb(http://www.iedb.org/)中的bepipred 2.0程序来预测特定蛋白氨基酸序列的线性b细胞表位,使用默认阈值0.5。对于超过6个但小于25个氨基酸的线性表位肽,使用vaxijen2.0在线服务器进行抗原评估,仅考虑使用vaxijen预测得分超过0.5的表位肽。如果表位肽的长度超过25,则只考虑vaxijen评分超过0.5的子片段序列。离散b细胞表位预测:对于特定蛋白的氨基酸序列,如果存在该蛋白质序列的三级结构,则直接使用。如果不存在,通过同源建模(swiss
‑
model)或者从头预测(alphafold或rosettafold)方式获得其三级结构。再通过iedb中的discotope 2.0程序预测特定蛋白三级结构中的不连续b细胞表位,并且只考虑倾向性评分(propensity)和discotope超过默认阈值
‑
3.7的氨基酸残基。
[0084]
在本发明实施例中,所述t细胞表位预测具体包括:mhc
‑
i(cd8 t细胞)结合表位预测:使用iedb中的netmhcpan4.1方法基于8个最常见的人类hlai类等位基因(hla
‑
a01:01,hla
‑
a02:01,hla
‑
a03:01,hla
‑
a11:01,hla
‑
a24:02,hla
‑
b07:02,hla
‑
b08:01,hla
‑
b40:01)预测了mhc
‑
i结合表位,并且只考虑了评分超过0.85且vaxijen得分超过0.5的表位。mhc
‑ⅱ
(cd4 t细胞)结合表位预测:基于常见的7个人类等位基因(包括drb103:01,drb107:01,drb115:01,drb301:01,drb302:02,drb401:01和drb5*01:01)和iedb推荐的2.22算法,表位肽的长度设置为15。类似地,mhc
‑ⅱ
的表位预测仅考虑adjusted rank值低于1且vaxijen评分超过0.5的结合表位。以新冠病毒s蛋白氨基酸序列为例,采用上述方法进行b细胞表位,其结果如表2、3所示。
[0085]
区域开始终止免疫表位(bepipred)长度vaxijen得分ntd1337sqcvnlttrtqlppaytnsftrgvy250.6860ntd5981fsnvtwfhaihvsgtngtkrfdn230.6767ntd138154dpflgvyyhknnkswme170.5821ntd177189mdlegkqgnfknl131.2592ntd206221khtpinlvrdlpqgfs160.6403ntd253259dsssgwt70.6067
s1304322ksftvekgiyqtsnfrvqp190.5729rbd351360yawnrkrisn100.5855rbd369393ynsasfstfkcygvsptklndlcft251.4031rbd404426gdevrqiapgqtgkiadynyklp231.1017rbd441448ldskvggn80.8773rbd459464snlkpf60.5943rbd473478yqagst60.5812rbd487492ncyfpl60.996rbd528535kkstnlvk80.658s1555562snkkflpf81.3952s1627632dqltpt60.7329s1656666vnnsyecdipi110.6124s1,s2680687sprrarsv80.6844s2695710ytmslgaensvaysnn160.6434s2809814pskpsk61.1271s210351043gqskrvdfc91.779s211101118yepqiittd90.8297s211541169kyfknhtspdvdlgdi160.7333cp12551267kfdeddsepvlkg130.5066
[0086]
表2连续b细胞表位
[0087]
由表2可知,突变频率高于0.5%的高频率突变位点中,突变位点l18f、l18f和s477n突变热点位于连续b细胞免疫表位上,a222v突变热点位于连续b细胞免疫表位附近。l18f,a222v,s477n这3个高频率突变,值得后续进一步研究其突变对中和抗体及疫苗保护效果的影响。
[0088]
表3离散的b细胞表位
[0089]
[0090]
[0091][0092]
由表3可知,突变频率高于0.5%的高频率突变位点中,突变位点n439k,s477n位于离散b细胞免疫表位上。n439k,s477n这2个高频率突变,值得后续进一步研究其突变对中和抗体及疫苗保护效果的影响。
[0093]
本发明实施例还提供了一种用于病毒基因组变异分析、检测系统,所述系统具体包括:
[0094]
筛选模块,用于筛选待分析基因组序列,将所述待分析基因组序列进行质量控制获得测序质量良好的全基因组序列集。
[0095]
序列比对模块,用于将所述测序质量良好的基因组序列集进行多序列比对,获得alignment序列文件;
[0096]
基因组注释模块,用于将所述alignment序列文件以字符串完全匹配方法匹配编码基因的起始字符串,并返回编码基因在全基因组序列中的位置下标,并以alignment序列文件中每个基因对应的位置下标,生成基因位置表;根据所述基因位置表,循环遍历每条序列,截取每条序列对应的编码基因片段并存储;
[0097]
翻译比对模块,用于根据所述编码基因序列进行翻译成氨基酸序列,并对氨基酸序列进行多序列比对,获得对齐的氨基酸序列文件,与对齐前的编码基因序列进行匹配,采用“密码子出现的顺序”作为映射关系进而进行“回译”,将对齐的氨基酸序列“回译”成对齐的核苷酸;
[0098]
插入位点标记模块;将所述对齐的核苷酸序列以每三个碱基扫描方式遍历序列的每个密码子位点,识别和记录序列中存在连续三个碱基插入位点
“‑‑‑”
的位置,标记为“插入位点”,获得并删除“插入位点”,获取不含插入位点的突变分析序列;
[0099]
编码基因突变分析模块,用于将参考序列对应的密码子和/或其对应的氨基酸与所述突变分析序列的密码子和/或翻译氨基酸按照预设突变分析方法进行分析获得待分析基因组序列突变位点以及变异类型;
[0100]
监测模块,用于获取不同采集时间和区域的突变分析结果,统计高频率突变位点,并分析所述高频率突变位点在时间和区域上的规律以及免疫表位情况。
[0101]
所述监测模块具体包括:高频率突变位点分析子模块,用于统计各突变位点的突变频率,将所述突变频率高于预设频率阈值对应的突变位点记为高频突变位点;增长趋势监测子模块,用于获取所述高频突变位点对应的基因组序列的采集时间计算高频突变在所
有基因组中的占比,获得折线图,并进行拟合,从而获得具有增长趋势的突变位点和突变毒株;地域监测子模块,获取所述高频突变位点对应的区域,构建聚类热图,用于不同地区疫苗设计提供参考;免疫表位筛选子模块,对所述高频突变位点进行免疫表位筛选,所述免疫表位筛选具体包括:b细胞表位预测和t细胞表位预测。
[0102]
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
[0103]
本领域技术人员在考虑说明书及实践这里公开的发明后,将容易想到本公开的其它实施方案。本技术旨在涵盖本公开的任何变型、用途或者适应性变化,这些变型、用途或者适应性变化遵循本公开的一般性原理并包括本公开未公开的本技术领域中的公知常识或惯用技术手段。说明书和实施例仅被视为示例性的,本公开的真正范围和精神由权利要求指出。
[0104]
应该理解的是,虽然本发明各实施例的流程图中的各个步骤按照箭头的指示依次显示,但是这些步骤并不是必然按照箭头指示的顺序依次执行。除非本文中有明确的说明,这些步骤的执行并没有严格的顺序限制,这些步骤可以以其它的顺序执行。而且,各实施例中的至少一部分步骤可以包括多个子步骤或者多个阶段,这些子步骤或者阶段并不必然是在同一时刻执行完成,而是可以在不同的时刻执行,这些子步骤或者阶段的执行顺序也不必然是依次进行,而是可以与其它步骤或者其它步骤的子步骤或者阶段的至少一部分轮流或者交替地执行。
[0105]
以上所述实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例中的各个技术特征所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。