一种基于机器学习预测癌症的cfdna片段特征组合及系统
技术领域
1.本发明属于癌症基因组学技术领域,具体地,涉及一种基于机器学习预测癌症的 cfdna片段特征组合、系统及应用。
背景技术:
2.血液中的游离dna(cfdna,circulating free dna or cell free dna)能够随着组织损伤、癌症和炎症反应等发生浓度变化,在疾病的早期诊断、预后、监测等方面具有重要潜在价值。近年来,cfdna已被广泛用于癌症早筛等研究领域。研究表明,可以利用特定的cfdna片段特征对肿瘤组织来源进行分类,cfdna片段的长度也可以揭示组织起源或肿瘤来源。
3.然而,目前大多数液体活检方法都专注于检测血液中的基因突变或染色体异常,且已有的片段组学方法多依赖于全基因组测序(wgs)的方法,无法充分开发利用其他组学测序数据信息。
技术实现要素:
4.为解决上述技术问题中的至少一个,本发明开发了一种可基于多种组学数据分析片段组学的系统,以识别cfdna片段分布肿瘤标志物,进而鉴别样本是否为肿瘤样本。具体地,本发明采用的技术方案如下:
5.本发明第一方面提供一种cfdna片段特征组合,包括以下片段特征中的至少一个: 163-164bp、157-159bp、157-160bp、159-160bp、147-148bp、151-153bp、277-279bp、 277-278bp、137-138bp、283-284bp、142-144bp、107-108bp、141-144bp、267-268bp、 117-118bp、141-142bp、298-300bp、339-340bp、337-340bp、337-338bp、327-328bp、 375-376bp、217-218bp、382-384bp、383-384bp、383-389bp、385-387bp、386-390bp、 195-196bp、191-192bp、227-228bp、189-192bp、319-320bp、187-189bp、189-190bp、 61-62bp、64-66bp、239-240bp、67-68bp、69-70bp和67-72bp。
6.在本发明的一些实施方案中,所述cfdna片段特征组合包括以下片段特征中的至少一个:163-164bp、157-159bp、157-160bp、159-160bp、151-153bp、277-279bp、137-138bp、283-284bp、142-144bp、107-108bp、141-144bp、267-268bp、117-118bp、141-142bp、 298-300bp、339-340bp、375-376bp、217-218bp、383-384bp、383-389bp、385-387bp、 386-390bp、195-196bp、191-192bp、227-228bp、189-192bp、319-320bp、187-189bp、 61-62bp、64-66bp、239-240bp、67-68bp、69-70bp和67-72bp。
7.在本发明的另一些实施方案中,所述cfdna片段特征组合包括以下片段特征中的至少一个:163-164bp、157-159bp、159-160bp、147-148bp、151-153bp、277-279bp、 277-278bp、107-108bp、267-268bp、117-118bp、141-142bp、298-300bp、339-340bp、 337-340bp、337-338bp、327-328bp、217-218bp、382-384bp、383-384bp、195-196bp、 191-192bp、189-192bp、189-190bp、61-62bp、64-66bp、239-240bp、67-68bp、69-70bp。
8.在本发明的一些优选实施方案中,所述cfdna片段特征组合包括以下片段特征中的至少一个:163-164bp、157-159bp、159-160bp、151-153bp、277-279bp、107-108bp、 267-268bp、117-118bp、141-142bp、298-300bp、339-340bp、217-218bp、383-384bp、 195-196bp、191-192bp、189-192bp、61-62bp、64-66bp、239-240bp、67-68bp和69-70bp。
9.在本发明中,至少一个是指包括列出的cfdna片段特征中的一个、二个、三个或全部。这些cfdna片段特征组合之间由于拥有相同的性质,属于一个总的发明构思。
10.在本发明中,相关术语的定义如下:
11.片段特征:是指将cfdna片段按不同长度划分为不同的片段区间,每个片段区间内的所有cfdna片段即为一个片段特征。例如片段特征为:61-65bp,包括片段长度为61bp、62bp、63bp、64bp和65bp的cfdna片段。例如片段特征为:74-75bp,包括片段长度为74bp和75bp的cfdna片段。
12.片段数量比例:是指一个片段特征中的cfdna片段数占总片段数的比例。
13.在本发明中,所述cfdna片段长度和数量数据是指利用测序方法得到的数据,所述测序选自包括wgs测序、wes测序、medip和mbd-seq的组中的任意一种。事实上,本领域技术人员可能使用任意测序的或非测序的方法,只要能够获得cfdna片段的长度及数量即可。
14.本发明的第二方面提供一种预测受试者是否患有癌症或者是否具有患癌症的风险的系统,包括以下模块:
15.数据输入模块,用于输入受试者cfdna片段长度和数量数据;
16.分布谱分析模块,与所述数据输入模块连接,用于获得本发明第一方面所述 cfdna片段特征组合中各cfdna片段特征的片段数量比例;
17.癌症预测模块,与所述分布谱分析模块连接,用于根据所述cfdna片段特征的片段数量比例判断受试者是否患有癌症或者是否具有患癌症的风险。
18.在本发明的一些实施方案中,所述癌症预测模块中的利用机器学习模型判断所述受试者是否患有癌症或者是否具有患癌症的风险。
19.进一步,所述机器学习模型采用以下任意一项算法训练得到:随机森林算法、支持向量机算法、线性回归算法、逻辑回归算法、贝叶斯分类器和神经网络算法。
20.在本发明的一些实施方案中,所述机器学习模型采用广义线性回归的lasso回归算法得到。
21.在本发明的一些具体实施方案中,采用r语言中的glmnet进行所述lasso回归。 glmnet主要用于拟合广义线性模型。筛选可以使loss达到最小的正则化参数λ。该算法非常快,并且可以使用稀疏矩阵作为输入。lasso回归的特点是在拟合广义线性模型的同时进行变量筛选(variable selection)和复杂度调整(regularization)。因此,不论目标因变量(dependent/response varaible)是连续的(continuous),还是二元或者多元离散的(discrete),都可以用lasso回归建模然后预测。这里的变量筛选是指不把所有的变量都放入模型中进行拟合,而是有选择的把变量放入模型从而得到更好的性能参数。复杂度调整是指通过一系列参数控制模型的复杂度,从而避免过度拟合 (overfitting)。
22.进一步地,在本发明的一些实施方案中,利用lasso回归得到的lasso score进行预测。当lasso score超过预设阈值时,判断患有癌症或者具有患癌症的风险。
23.在本发明的一些实施方案中,所述预测阈值是根据群体癌症样本lasso score值
和 /或群体正常样本lasso score值进行确定的。
24.任选地,所述预测阈值是根据群体癌症样本lasso score值的代表值确定的。
25.任选地,所述预测阈值是根据群体正常样本lasso score值的代表值确定的。
26.任选地,所述预测阈值是根据群体癌症样本lasso score值相对于群体正常样本lasso score值的增加值的代表值确定的。这里的癌症样本和正常样本为配对样本,以使得增加值具有临床意义。
27.在本发明的一些具体实施方案中,所述群体癌症样本是指10个以上癌症样本,例如10个、20个、50个、100个、200个、500个或更多。
28.在本发明的一些具体实施方案中,所述代表值是指平均数、众数、中位数、1/4 分位数和3/4分位数中的一种。
29.在本发明中,所述癌症包括但不限于实体瘤和血癌,如纤维肉瘤、肌肉瘤、脂肪肉瘤、软骨肉瘤、成骨肉瘤、脊索瘤、血管肉瘤、内皮肉瘤、淋巴管肉瘤、淋巴管内皮肉瘤、滑膜瘤、间皮瘤、尤因瘤、平滑肌肉瘤、横纹肌肉瘤、结肠癌、胰腺癌、前列腺癌、鳞状细胞癌、基底细胞癌、腺癌、汗腺癌、皮脂腺癌、乳头状癌、乳头腺癌、囊腺癌、髓样癌、支气管癌、肝细胞瘤、胆管癌、绒毛膜癌、精原细胞瘤、胚胎癌、肾母细胞瘤、宫颈癌、睾丸瘤、肺癌、小细胞肺癌、上皮癌、胶质瘤、星形细胞瘤、髓母细胞瘤、颅咽管瘤、室管膜瘤、松果体瘤、成血管细胞瘤、听神经瘤、少突神经胶质瘤、脑膜瘤、黑素瘤、神经母细胞瘤、胶质母细胞瘤、视网膜母细胞瘤;白血病,如急性淋巴细胞性白血病和急性成髓细胞性白血病(成髓细胞、前髓细胞、髓单核细胞、单核细胞和红细胞白血病);慢性白血病(慢性髓细胞(粒细胞)白血病和慢性淋巴细胞性白血病);和真性红细胞增多、淋巴瘤(霍奇金病和非霍奇金病)、多发性骨髓瘤、瓦尔登斯特伦巨球蛋白血症和重链病。
30.在本发明中,所述识别、分类、判断、预测具有相同或相似的含义,均是指将癌症样本和正常样本区分开来。
31.本发明第三方面提供本发明第一方面所述的cfdna片段特征组合的检测试剂在制备用于预测受试者是否患有癌症或者是否具有患癌症的风险的试剂盒中的应用。
32.在本发明的一些实施方案中,所述检测试剂包括捕获试剂和/或测序试剂。
33.在本发明的一些实施方案中,所述试剂盒还包括cfdna提取试剂。
34.本发明的有益效果
35.相对于现有技术,本发明具有以下有效效果:
36.利用本发明的cfdna片段特征组合和系统进行癌症预测,不仅可以利用选自包括wgs测序、wes测序、medip和mbd-seq的组中的任意一种测序方法的数据,也可以使用任意测序的或非测序的方法得到的数据,只要能够获得cfdna片段的长度及数量即可。
37.利用本发明的cfdna片段特征组合和系统进行癌症预测,能够利用cfdna片段综合特征分析,对于癌症的预测性能更优。
38.利用本发明的cfdna片段特征组合和系统进行癌症预测,既降低了基于cfdna 片段分析预测癌症的方法对于上游实验端的要求和依赖,又显著拓宽了其他组学测序数据的可解读性和利用率,因此,极大的降低了基于cfdna诊断肿瘤的实验成本,同时提高了基于cfdna预测癌症的准确性。
附图说明
39.图1示出了本发明实施例2中lasso回归cvfit曲线。
40.图2示出了lambda.min模型在训练集和验证集中进行肿瘤识别的结果。
41.图3示出了lambda.1se模型在训练集和验证集中进行肿瘤识别的结果。
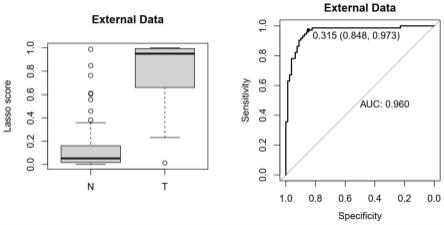
42.图4示出了lambda.min模型在外部数据集中进行肿瘤识别的结果。
43.图5示出了lambda.1se模型在外部数据集中进行肿瘤识别的结果。
44.图6示出了21特征分类模型在训练集和测试集中的分类效能。
45.图7示出了21特征分类模型在外部数据集中的分类效能。
具体实施方式
46.除非另有说明、从上下文暗示或属于现有技术的惯例,否则本技术中所有的份数和百分比都基于重量,且所用的测试和表征方法都是与本技术的提交日期同步的。在适用的情况下,本技术中涉及的任何专利、专利申请或公开的内容全部结合于此作为参考,且其等价的同族专利也引入作为参考,特别这些文献所披露的关于本领域中的技术术语等的定义。如果现有技术中披露的具体术语的定义与本技术中提供的任何定义不一致,则以本技术中提供的术语定义为准。
47.本技术中的数字范围是近似值,因此除非另有说明,否则其可包括范围以外的数值。数值范围包括以1个单位增加的从下限值到上限值的所有数值,条件是在任意较低值与任意较高值之间存在至少2个单位的间隔。这些仅仅是想要表达的内容的具体示例,并且所列举的最低值与最高值之间的数值的所有可能的组合都被认为清楚记载在本技术中。
48.为了使本发明所解决的技术问题、技术方案及有益效果更加清楚明白,以下结合实施例,对本发明进行进一步详细说明。
49.实施例
50.以下例子在此用于示范本发明的优选实施方案。本领域内的技术人员会明白,下述例子中披露的技术代表发明人发现的可以用于实施本发明的技术,因此可以视为实施本发明的优选方案。但是本领域内的技术人员根据本说明书应该明白,这里所公开的特定实施例可以做很多修改,仍然能得到相同的或者类似的结果,而非背离本发明的精神或范围。
51.除非另有定义,所有在此使用的技术和科学的术语,和本发明所属领域内的技术人员所通常理解的意思相同,在此公开引用及他们引用的材料都将以引用的方式被并入。
52.那些本领域内的技术人员将意识到或者通过常规试验就能了解许多这里所描述的发明的特定实施方案的许多等同技术。这些等同将被包含在权利要求书中。
53.下述实施例中未作具体说明的分子生物学实验方法,均按照《分子克隆实验指南》 (第四版)(j.萨姆布鲁克、m.r.格林,2017)一书中所列的具体方法进行,或者按照试剂盒和产品说明书进行。其他实验方法,如无特殊说明,均为常规方法。下述实施例中所用的仪器设备,如无特殊说明,均为实验室常规仪器设备;下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。
54.实施例1cfdna片段分布肿瘤标志物的识别
55.1.cfdna测序
56.为了获得cfdna片段分布肿瘤标志物,发明人获得了417个肿瘤患者(183个结直肠
72bp。
70.lambda.1se对应的模型包括28个片段特征,分别是:163-164bp、157-159bp、 159-160bp、147-148bp、151-153bp、277-279bp、277-278bp、107-108bp、267-268bp、 117-118bp、141-142bp、298-300bp、339-340bp、337-340bp、337-338bp、327-328bp、 217-218bp、382-384bp、383-384bp、195-196bp、191-192bp、189-192bp、189-190bp、 61-62bp、64-66bp、239-240bp、67-68bp、69-70bp。
71.两个分类模型在训练集和测试集中的分类效能如表1和图2~图3所示。
72.表1两种lasso分类模型的分类效能
[0073][0074]
由此可见,利用lambda.min模型和lambda.1se模型,均能够得到较好地识别肿瘤。
[0075]
实施例4肿瘤分类模型过拟合的判断
[0076]
对于线性模型来说,复杂度与模型的变量数有直接关系,变量数越多,模型复杂度就越高。更多的变量在拟合时往往可以给出一个看似更好的模型,但是同时也面临过度拟合的危险。此时如果用全新的数据去验证模型(validation),通常效果很差。一般来说,变量数大于数据点数量很多,或者某一个离散变量有太多独特值时,都有可能过度拟合。
[0077]
为了判断实施例3的肿瘤模型是否存在过拟合,发明人使用外部数据(即除训练集和测试集之外的数据,包括73例肿瘤样本和79例正常样本)进行判断,结果如表 2和图4~图5所示。
[0078]
表2两种lasso分类模型在外部数据中的分类效能
[0079][0080]
由此可见,实施例3的两个分类模型均不存在过拟合,并且具有非常好的判断效果。
[0081]
实施例5 21片段特征肿瘤识别模型
[0082]
实施例3中lambda.min对应的模型包括34个片段特征,lambda.1se对应的模型包括28个片段特征,他们之间一共有21个交集特征,分别为:163-164bp、157-159bp、159-160bp、151-153bp、277-279bp、107-108bp、267-268bp、117-118bp、141-142bp、 298-300bp、339-340bp、217-218bp、383-384bp、195-196bp、191-192bp、189-192bp、 61-62bp、64-66bp、
239-240bp、67-68bp、69-70bp。
[0083]
发明人检验单独使用21个交集特征,是否也有区分肿瘤和正常样本的能力。具体地:使用21个交集特征重新构建一个新的21特征lasso模型,训练集、测试集和外部数据集保持不变。
[0084]
21特征分类模型在训练集和测试集中的分类效能如表3和图6所示。
[0085]
表3 21特征lasso分类模型的分类效能
[0086][0087]
21特征分类模型在外部数据集中的分类效能如表4和图7所示。
[0088][0089][0090]
由此可见,仅仅利用21个片段特征,也能够很好的识别肿瘤,可以用于预测受试者是否患有肿瘤或者是否患肿瘤的风险。
[0091]
在本发明提及的所有文献都在本技术中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本技术所附权利要求书所限定的范围。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。