1.本发明涉及生物数据处理技术领域,尤其涉及一种生物序列标识符的压缩方法及装置、解压方法及装置。
背景技术:
2.自dna(脱氧核糖核酸)测序技术诞生以来,生物序列数据不断增长,且增长速度越来越快,随着数据积累,数据存储成本和传输成本不断提升,因此需要将生物序列数据进行数据压缩以降低成本。
3.fastq是用于保存生物序列和其测序质量信息的文本格式。fastq文件中包含多个生物序列片段(read),每四行为一个单位来表示一个生物序列片段。
4.每个生物序列片段包含四行:
5.1.第一行:以“@”开头,后面跟着唯一的序列id标识符,标识符可能包含若干序列描述内容;
6.2.第二行:碱基序列,通常是由acgtn组成的字符串,极少数情况下会出现其他字符;
7.3.第三行:以“ ”开头,后面什么也不加,或者后面跟着可选的id标识符和可选的描述内容,如果“ ”后面有内容,该内容必须与第一行“@”后的内容相同;
8.4.第四行:碱基的测序质量值,与第二行的碱基序列相对应,每一个碱基序列对应一个测序质量值。碱基的测序质量值指代每个碱基的测序可信度,有两种质量值系统,分别从!和@开始,范围都是40个字符左右。
9.起初,gzip和bzip2(均为压缩工具)等工具被用于压缩fastq格式文件,由于对fastq格式文件的特征利用不足,压缩率并不理想。后来出现了专门压缩fastq格式文件的工具,例如seqdb和g-sqz(均为压缩工具),它们都选择将fastq格式文件中的碱基序列和测序质量值结合编码,而对于fastq格式文件中的标识符还都是独立编码的,这种策略的压缩率很差,因而很快被摒弃。目前,将fastq格式文件中的标识符、碱基序列和测序质量值三个信息流独立压缩是主流做法。
10.由于fastq格式文件的标识符基本遵循固定字符与变量相结合的格式,规律性很强,所以主要有两类压缩方法:一是“总分法”,即在文件头记录总的格式,然后对每个标识符压缩时记录其变量值,如kungfq(一个压缩工具);二是“差分法”,即编码每个标识符跟上一个标识符的差异,如quip(一个压缩工具)。然而,总分法的适用性较差,只能识别特定格式的标识符;差分法对格式的拟合较差,压缩率更低。两类压缩方法都不能兼顾两者。
技术实现要素:
11.本发明为了克服现有技术中压缩fastq格式文件时无法平衡适用性和压缩率的缺陷(要么适用性较差,要么压缩率较低),提供一种生物序列标识符的压缩方法及装置、解压方法及装置。
12.本发明是通过下述技术方案来解决上述技术问题:
13.第一方面,提供一种生物序列标识符的压缩方法,包括:
14.对于基因测序文件中的每个标识符,将所述标识符拆分成若干子标识符;
15.定义若干窗口的编码规则,所述编码规则与所述子标识符的文本格式相匹配;
16.将指代含义相同的子标识符划分至相同的窗口;
17.对于各个窗口,根据对应的编码规则对所述窗口中的所有子标识符进行编码,并将各个窗口的编码结果汇总成所述标识符的压缩结果。
18.可选地,对于基因测序文件中的每个标识符,将所述标识符拆分成若干子标识符,包括:
19.确定所述标识符是基于单端测序得到的生物序列的标识符,还是基于双端测序得到的生物序列的标识符;
20.在确定所述标识符是基于单端测序得到的生物序列的标识符的情况下,根据预定义的拆分规则将所述标识符拆分成对应于各个窗口的子标识符;所述窗口的数量和/或各个窗口的属性由初始被拆分的标识符的子标识符的文本格式确定。
21.在确定所述标识符是基于双端测序得到的生物序列的标识符的情况下,根据预定义的拆分规则分别将所述标识符的第一片段和第二片段拆分成对应于各个窗口的子标识符。
22.可选地,若所述标识符是基于双端测序得到的生物序列的标识符,所述压缩方法还包括:
23.确定所述第一片段的各个子标识符与所述第二片段中相同序位的子标识符的差异信息;
24.将所述第一片段的子标识符的文本格式以及所述差异信息确定为所述标识符的子标识符的文本格式,或者将所述第二片段read2的子标识符的文本格式以及所述差异信息确定为所述标识符的子标识符的文本格式。
25.可选地,所述拆分规则包括:
26.按照连续符号及连续非符号进行拆分;
27.和/或,按照所述标识符与其他标识符的连续相同内容及差异内容进行拆分。
28.可选地,还包括:
29.根据各个子标识符的文本格式确定所述标识符的文本格式;
30.对具有相同文本格式的标识符进行共同编码。
31.第二方面,提供一种生物序列标识符的解压方法,包括:
32.确定用于对标识符的二进制流进行解压的解压信息;其中,所述二进制流由上述任一项所述的生物序列标识符的压缩方法对标识符进行压缩得到,所述解压信息根据对所述标识符进行压缩的过程中生成的文本格式所确定;
33.根据所述解压信息对所述二进制流进行解压,得到生物序列的标识符。
34.第三方面,提供一种生物序列标识符的压缩装置,包括:
35.拆分模块,用于对于基因测序文件中的每个标识符,将所述标识符拆分成若干子标识符;
36.定义模块,用于定义若干窗口的编码规则,所述编码规则与所述子标识符的文本
格式相匹配;
37.划分模块,用于将指代含义相同的子标识符划分至相同的窗口;
38.编码模块,用于对于各个窗口,根据对应的编码规则对所述窗口中的所有子标识符进行编码,并将各个窗口的编码结果汇总成所述标识符的压缩结果。
39.第四方面,提供一种生物序列标识符的解压装置,包括:
40.确定模块,用于确定用于对标识符的二进制流进行解压的解压信息;其中,所述二进制流由上述任一项所述的生物序列标识符的压缩方法对标识符进行压缩得到,所述解压信息根据对所述标识符进行压缩的过程中生成的文本格式所确定;
41.解压模块,用于根据所述解压信息对所述二进制流进行解压,得到生物序列的标识符。
42.第五方面,提供一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述计算机程序时实现上述任一项所述的方法。
43.第六方面,一种计算机可读存储介质,其上存储有计算机程序,所述程序被处理器执行时实现上述任一项所述的方法。
44.本发明的积极进步效果在于:
45.本发明实施例能够在尽量兼容特殊数据的前提下,尽量提升对所有标识符数据的压缩率,同时保证了编解码性能。
附图说明
46.图1是本发明一示例性实施例提供的一种生物序列标识符的压缩方法的流程图。
47.图2a是本发明一示例性实施例提供的另一种生物序列标识符的压缩方法的流程图。
48.图2b是本发明一示例性实施例提供的一种生物序列标识符的压缩方法对标识符进行格式判定时,窗口拆分的示意图。
49.图2c是本发明一示例性实施例提供的一种生物序列标识符的压缩方法在格式不匹配生成新的大编码器时,窗口拆分的示意图。
50.图3a是本发明一示例性实施例提供的另一种生物序列标识符的压缩方法的流程图。
51.图3b是采用本发明一示例性实施例提供的一种生物序列标识符的压缩方法对双端测序的生物序列的标识符进行编码的流程图。
52.图3c是采用本发明一示例性实施例提供的一种生物序列标识符的编码方法对非符号且非纯数字的窗口进行编码的流程图。
53.图3d是采用本发明一示例性实施例提供的一种生物序列标识符的编码方法对纯数字的窗口进行编码的流程图。
54.图4是本发明一示例性实施例提供的一种生物序列标识符的解压方法的流程图。
55.图5是本发明一示例性实施例提供的另一种生物序列标识符的解压方法的流程图。
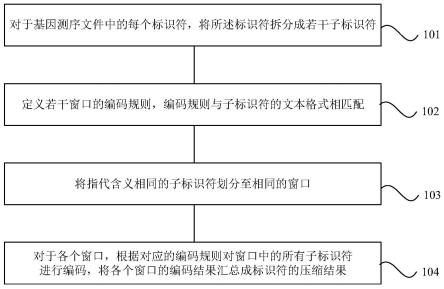
56.图6是本发明一示例实施例示出的一种电子设备的结构示意图。
具体实施方式
57.下面通过实施例的方式进一步说明本发明,但并不因此将本发明限制在所述的实施例范围之中。
58.本发明实施例提供一种生物序列标识符的压缩方法,下面详细介绍标识符的压缩过程。
59.图1是本发明一示例性实施例提供的一种生物序列标识符的压缩方法的流程图,该压缩方法包括以下步骤:
60.步骤101、对于基因测序文件中的每个标识符,将所述标识符拆分成若干子标识符。
61.基因测序类型包括单端序列(single-end sequencing)和双端序列,双端序列又包括paired-end sequencing和mate-paired sequencing。对于单端测序,单次测序只会产生单个fastq(基因测序)文件,而对于双端测序,单次测序会产生一对fastq文件,这两个fastq文件分别存放5’端测序生物序列片段(read1)和3’端测序生物序列片段(read2)的数据,这两个fastq文件的行数相等,每一对fastq文件在文件位置上都是一一对应的,且在基因组上的位置也是相邻的。对于双端序列来说,其成对的序列的标识符内容也基本相同,因此,同时压缩一对fastq格式文件能更好地去除数据的冗余,优化压缩率。
62.从而,在进行压缩之前,需基于fastq文件的文件个数判断生物序列是基于单端测序得到的,还是基于双端测序得到的,基于不同基因测序类型得到的生物序列采用不同的压缩策略,原始文件是单端测序或双端测序的判断信息会写入压缩文件中。下文中将基于单端测序得到的生物序列称为单端数据,将基于双端测序得到的生物序列称为双端数据。
63.由于标识符用于记录每条生物序列的测序信息,普遍遵从固定格式,子标识符可以分为两类:信息(一般是字母、数字或者二者的组合,各指代不同的含义,如测序仪的编号、该序列的测序坐标、该序列在文件中的编号等),以及用于分隔信息的分隔符(一般都是符号,如斜杠和下划线),指代含义相同的子标识符在标识符上的序位往往是固定的。另外,还会存在一个标识符上两个或多个信息相互有关联的情况(如标识符上两处信息的内容完全一致)。因此,将标识符拆分成各个子标识符,将子标识符对应各个窗口,使得一个窗口包含独立的信息或分隔符,可使得在不同标识符中指代含义相同的子标识符共同编码,指代含义相同的各个子标识符相似性高,因此这样能够提高压缩率。
64.在一个实施例中,若确定标识符是基于单端测序得到的生物序列的标识符,则根据预定义的拆分规则将标识符拆分成对应于各个窗口的子标识符。
65.在一个实施例中,若确定标识符是基于双端测序得到的生物序列的标识符,则根据预定义的拆分规则将标识符的第一片段和第二片段分别拆分成对应于各个窗口的子标识符。
66.其中,上述拆分规则包括:按照连续符号及连续非符号进行拆分。
67.步骤102、定义若干窗口的编码规则,编码规则与子标识符的文本格式相匹配。
68.按照上述拆分规则对标识符进行拆分之后,子标识符的文本格式包括:分隔符格式、纯数字格式和非纯数字(包含数字和字母)格式。不同文本格式的子标识符具有不同的特征,应采用不同的编码策略。应用不同的编码策略的方法,是声明大编码器。大编码器包含若干个窗口,每个窗口有独立的编码器,各个窗口的编码规则与子标识符的文本格式一
一匹配,由于子标识符的文本格式共同组成标识符的文本格式,因此同一类文本格式相同的标识符对应着一个大编码器。
69.具体的,编码规则包括:作为分隔符的连续符号,其内容一般来说是固定不变的,因此编码规则是不编码内容,只记录格式即可;作为信息的连续非符号,可以分成纯数字和非纯数字;作为信息的纯数字,编码规则是将其整体视作一个数字以编码;作为信息的非纯数字,其内容一般来说是不会改变长度的,因此编码规则是将其视作一个个字符加以编码。另外,还存在某窗口对应的子标识符与其他窗口的子标识符完全一致的情况,其编码规则是记录该窗口与对应窗口的关系,则省去重复编码的耗费。另外,还存在某纯数字窗口对应的子标识符表示的是该序列的长度信息,其编码规则是在编码纯数字之余记录该窗口是长度信息的特征,则在压缩测序序列的碱基信息和质量值时可省去重复编码的耗费。
70.步骤103、将指代含义相同的子标识符划分至相同的窗口。
71.各个标识符在得到子标识符的文本格式后,文本格式相同的标识符都用相同的大编码器进行编码,大编码器的各个窗口的编码规则都与这些标识符的子标识符一一对应。在大编码器内部,各个标识符的指代含义相同的子标识符都会相同的子标识符划分至相同的窗口,以便于共同编码。
72.具体的,确定标识符的各个子标识符的文本格式,并将其记录为标识符的文本格式,方式包括:由于具有相同文本格式的标识符都用同一个大编码器编码,因此其文本格式只需记录一次即可。
73.在出现文本格式与现有大编码器都不完全相同的标识符时,可以声明新的大编码器,而新的大编码器的窗口划分规则,除了根据所述标识符的子标识符外,还可以根据该标识符与已编码的标识符的比较结果。
74.另外,也可以使一个大编码器不只编码文本格式完全相同的标识符,只要额外记录文本格式不完全相同的标识符的文本格式差异即可。
75.其中,文本格式是否相同的判定与窗口的编码规则相关,包括:窗口的数量、相同序位上的窗口属性必须相同,连续非符号窗口的长度和内容必须一致;连续非符号非纯数字窗口的长度必须一致;连续纯数字窗口的长度必须一致等。
76.步骤104、对于各个窗口,根据对应的编码规则对窗口中的所有子标识符进行编码,将各个窗口的编码结果汇总成标识符的压缩结果。
77.如果选择将文本格式相同的标识符共同编码,则对于共同编码的数据,其各个窗口都是一一对应的,对于各个标识符在同一窗口中的内容,用该窗口的编码器根据窗口的编码规则一一进行编码。如果采取的方式是顺序为主的匹配,即顺序读取标识符,当标识符与前一个标识符的文本格式相同,则将其继续归入该批次标识符共同编码,否则将当前标识符视作新的批次,这种顺序匹配的方式无需记录标识符的顺序;如果采取的方式是批次为主的匹配,即顺序读取标识符,将标识符归入格式相同的批次进行编码,则需记录各个标识符在各个批次中的顺序,用于还原初始的标识符的顺序。
78.如果选择顺序编码标识符,并记录文本格式差异,则仍可对于各个标识符在同一窗口中的内容,用该窗口的编码器根据窗口的编码规则一一进行编码。
79.各个窗口编码完毕后,将各个窗口的编码结果二进制流汇总并输出,即为基因测序文件中的标识符的压缩结果。
80.本发明实施例中,通过对各个标识符进行拆分,分析其文本格式,根据该文本格式定义窗口的编码规则,并将指代含义相同的子标识符划分至相同的窗口进行共同编码,实现了标识符的整合压缩,最大程度地利用了信息,提高了总体规律的拟合,提高了提高压缩率,并且该压缩方法适用性较广,适用于各类fastq文件。
81.在一个实施例中,对基因测序文件中的所有标识符依次进行文本格式识别,并采取顺序为主的匹配方式,对文本格式相同的标识符共同编码,在编码过程中,各个标识符在同一窗口中的子标识符按照该窗口的编码规则进行编码,最后将所有窗口的编码结果输出,作为压缩结果。
82.在一个实施例中,标识符依次进行文本格式判定,当标识符与前一个标识符的文本格式相同,则将其继续归入该批次标识符共同编码,否则将当前标识符视作新的批次,每个批次独立进行编码。从基因测序文件中选取第一个标识符作为初始标识符,对其进行编码并记录其文本格式,然后再选取第二个标识符,判定其文本格式,确定第二个标识符的文本格式与初始标识符的文本格式是否相相同;在确定相同的情况下,第二个标识符的各个子标识符在初始标识符的各窗口中编码;在确定不相同的情况下,将初始标识符的各窗口的结果输出,第二个标识符作为初始标识符,对其进行编码并记录其文本格式;然后选取第三个标识符,判定其文本格式,根据其与初始标识符是否相同,重复第二个标识符的相应步骤。以此类推,直至对基因测序文件中的所有标识符完成压缩,最后再将初始标识符的各窗口的结果输出。
83.下面详细介绍对基因测序文件中的所有标识符依次进行文本格式识别,并采取顺序为主的匹配方式的场景下,生物序列标识符的压缩过程。
84.图2a是本发明一示例性实施例提供的另一种生物序列标识符的压缩方法的流程图,压缩方法包括以下步骤:
85.步骤201、基于基因测序文件是单个或者成对,将其认定为单端测序数据或双端测序数据。
86.基因测序类型包括单端序列(single-end sequencing)和双端序列,双端序列又包括paired-end sequencing和mate-paired sequencing。对于单端测序,单次测序只会产生单个fastq文件,而对于双端测序,单次测序会产生一对fastq文件,这两个fastq文件分别存放5’端测序生物序列片段(read1)和3’端测序生物序列片段(read2)的数据,这两个fastq文件的行数相等,每一对fastq文件在文件位置上都是一一对应的,且在基因组上的位置也是相邻的。对于双端序列来说,其成对的序列的标识符内容也基本相同,因此,同时压缩一对fastq格式文件能更好地去除数据的冗余,优化压缩率。
87.从而,在进行压缩之前,需基于基因测序文件的文件个数判断生物序列是基于单端测序得到的,还是基于双端测序得到的,基于不同基因测序类型得到的生物序列采用不同的压缩策略,标识符单端测序或双端测序的判断信息会写入压缩文件中。下文中将基于单端测序得到的生物序列称为单端数据,将基于双端测序得到的生物序列称为双端数据。
88.步骤202、对于单端数据,从尚未压缩的标识符中选取一个作为初始标识符,将初始标识符拆分成若干个子标识符,并记录初始标识符的文本格式;对于双端数据,从尚未压缩的标识符中选取一对作为初始标识符,将初始标识符拆分成若干个子标识符,并记录初始标识符的文本格式。
89.其中,标识符的获取顺序可以是标识符在基因测序文件中记录的顺序,也可以根据实际需求自行确定标识符的获取顺序,如果是后者,需要记录顺序以供解压时使用。由于基因测序文件中记录的原始标识码为文本格式,需进行编码。
90.对于单端测序得到的生物序列(单端数据)与双端测序得到的生物序列(双端数据),确定文本格式的具体方式有所不同。
91.下面介绍确定单端数据的文本格式的实现方式。
92.对于单端数据,对初始标识符,采用编码范围覆盖ascii(美国信息交换标准代码)码在[32,127]内所有字符的编码器进行编码,得到的编码结果记为init_bits;对初始标识符根据预定义的拆分规则,将初始标识符的编码结果拆分成各个独立且连续的子标识符,记录各个子标识符的文本格式init_format(init_format仅在内存中,不写入结果文件),并直接将初始标识符所含的各个子标识符的文本格式确定为初始标识符的文本格式。
[0093]
另外,记录初始标识符的文本格式也可以通过不直接记录init_bits,而选择记录init_format,并直接用各个窗口对初始标识符的各个子标识符进行编码的方式进行(编码方法与步骤206的方法相同)。
[0094]
图2b是本发明一示例性实施例提供的一种标识符拆分的结果示意图,图中以连续符号及连续非符号作为拆分规则,将标识符“@test123.4 4c009r042_292727length=717/1”拆分为8个符号窗口、5个纯数字窗口和3个非纯数字窗口,共16个窗口。
[0095]
预定义的拆分规则可以是但不限于将初始标识符按照连续符号及连续非符号拆分成各个子标识符,并记录各个子标识符的文本格式。文本格式可以包括但不限于以下参数:子标识符的属性,如纯符号、纯数字、无符号且非纯数字等;子标识符的字符长度;子标识符的字符内容;子标识符对应窗口的已处理字符串列表(记为window_str_list,该窗口处理过的子标识符)。其中,已处理字符串列表可以作为标识符压缩的参考,比如在一开始无法确定某个窗口的标识符用什么压缩方式合适,可以在处理了n条标识符后,根据已处理字符串列表中已处理的情况来确定压缩方式。
[0096]
本发明实施例中,基于标识符的数据特征将拆分规则设置为按照连续符号及连续非符号进行拆分,这样拆分较为高效,当然,除了上述的拆分规则之外,还可以采用其他的拆分规则,本发明对此不作特别限定。
[0097]
另外,若本步骤系205跳转而来,也可以对当前标识符与过往标识符进行比较,如图2c,在非符号非纯数字的子标识符在出现变长特征后,将其变长数字的部分与其他部分切割开,形成新的窗口。
[0098]
相对应的,子标识符的属性(文本格式)也可以被描述成其他类型,举例来说,属性可以是子标识符与其他子标识符的关系,如图2b的第四个和第六个窗口的子标识符完全相同,可以将第六个窗口对应的子标识符的属性标记为“与第四个窗口内容一致”,如果这样标记,则后续在编码“第四个窗口和第六个窗口的内容不完全相同”的标识符时,则判定为格式不相同。
[0099]
又如倒数第三个纯数字窗口的内容为717,由于它跟随在“length=”后面,其内容基本等同于碱基序列长度,可以对其进行标记,则在碱基序列压缩部分可以无需编码序列长度,以节省空间。
[0100]
下面介绍确定双端数据的文本格式的实现方式。
[0101]
对于双端数据,对初始标识符的第一片段和第二片段,用编码范围覆盖ascii码在[32,127]内所有字符的编码器进行编码,得到的编码结果记为init_bits。对初始标识符的第一片段和第二片段分别按照预设的拆分规则分别进行拆分,将第一片段的各个子标识符与第二片段中相同位序的子标识符进行比较,记录二者的差异信息,记为init_pe_diff。举例来说,假设第一片段为“@test123.4 4c009r042_292727length=717/1”,第二片段为“@test123.4 4c009r042_292727length=717/2”,两者唯一差异体现在末位字符相差1,差异信息为“末位字符相差1”。将第一片段(或者第二片段)所含的各个子标识符的文本格式记为init_format,并将init_format以及差异信息init_pe_diff确定为初始标识符的文本格式。
[0102]
确定标识符的文本格式的目的在于,将具有相同文本格式的标识符进行共同压缩。
[0103]
其中,确定标识符第一片段(或者第二片段)所含的各个子标识符的文本格式的具体实现方式与确定单端数据的子标识符的文本格式的具体实现方式类似,此处不再赘述。
[0104]
步骤203、对于单端数据,从尚未压缩的标识符中选取一个作为当前标识符,确定其文本格式;对于双端数据,从尚未压缩的标识符中选取一对作为当前标识符,确定其文本格式。其中,当前标识符的确定文本格式的具体实现方式与初始标识符的确定文本格式的具体实现方式类似,此处不再赘述具体实现过程。
[0105]
对于单端数据,将当前标识符的各个子标识符的文本格式记为current_format;对于双端数据,将当前标识符的第一片段(或者第二片段)的各个子标识符的文本格式current_format,以及第一片段与第二片段的差异信息记为current_pe_diff。
[0106]
步骤204、判断当前标识符的文本格式与初始标识符的文本格式是否相同。
[0107]
其中,文本格式包含多个参数,例如,窗口数量、子标识符的属性、子标识符的字符长度、子标识符对应窗口的字符串列表等,对于各个参数进行一一比较。当所有参数均相同时,则确定初始标识符的文本格式与当前标识符的文本格式相同;当存在一个参数不相同时,则确定两者的文本格式不相同。
[0108]
举例来说,若两者的窗口数量不同,则确定两者不相同,结束比较;若窗口数量相同,则对窗口对应的子标识符的属性两两进行比较,若子标识符的属性不同则确定两者不相同,结束比较;若子标识符的属性相同,在纯符号或非符号且非纯数字的窗口中,出现子标识符的字符长度不同,则确定两者不相同;在纯符号窗口中,出现子标识符内容不同,则确定两者不相同,结束比较;对于双端数据而言,若二者的差异信息不同,则确定两者不相同,结束比较;若比较完毕后以上情况都未出现,则确定两者相同。
[0109]
步骤204中,若判断结果为是,也即当前标识符的文本格式与初始标识符的文本格式相同,则执行步骤206,继续选取当前标识符进行文本格式判断。
[0110]
步骤204中,若判断结果为否,也即当前标识符的文本格式与初始标识符的文本格式不相同,则执行步骤205。
[0111]
步骤205、将当前标识符作为初始标识符。然后返回步骤202。
[0112]
步骤206、基于与初始标识符相同的拆分规则将当前标识符拆分成若干子标识符,并将当前标识符的各个子标识符划分至对应于初始标识符的各个窗口中,用初始标识符的各个窗口的编码器进行编码。
[0113]
初始标识符与当前标识符的文本格式相同意味着两者的子标识符遵从同样的格式,各个子标识符可以按照相同的编码规则编码,则将当前标识符与初始标识符中具有相同指代含义的子标识符划分至相同的窗口。
[0114]
在一个实施例中,每个子标识符对应一个窗口,每个窗口都配置有独立的编码器,已知当前标识符的文本格式与初始标识符的文本格式相同,也就是说,二者的子标识符的数量及属性都一一匹配,则子标识符会落入相应窗口,由窗口内的编码器编码。
[0115]
对各个窗口的编码结果汇总可以但不限于采用:用封装格式将各个窗口编码结果封装起来,便于解压时分辨不同窗口的结果。
[0116]
步骤207、判断fastq文件中的所有标识符是否均处理完毕。
[0117]
若判断结果为是,则执行步骤208。
[0118]
若判断结果为否,否则执行步骤203。
[0119]
步骤208、将init_bits和各个窗口的编码结果(二进制码流)输出,并得出成功压缩的提示。
[0120]
在一个实施例中,init_bits和每个窗口的二进制码流都经过封装格式的封装后输出,而在一次输出中,init_bits和各个窗口的二进制码流,这些信息在整体上还会进行一次封装然后输出。封装可以但不限于通过ebml(extensible binary meta language,一种编码语言)实现。
[0121]
图3a是本发明一示例性实施例提供的另一种生物序列标识符的压缩方法的流程图,该压缩方法包括以下步骤:
[0122]
步骤301、判断基因测序类型是单端测序还是双端测序。
[0123]
基于基因测序文件是单个或是成对,将其认定为单端数据或双端数据;若基因测序文件是单个,则确定基因测序类型是单端测序,得到的是单端数据;若基因测序文件是成对的,则确定基因测序类型是双端测序,得到的是双端数据。
[0124]
在步骤301中,单端测序或双端测序的判断信息会写入压缩文件中。
[0125]
在步骤301中,若判断为是,也即标识符是基于单端测序得到的,则执行步骤302。
[0126]
在步骤301中,若判断为否,也即标识符是基于双端测序得到的,标识符为标识符对,包含read1和read2,则执行步骤303。
[0127]
步骤302、从未压缩的标识符中读取第一个,作为初始标识符。然后执行步骤304。
[0128]
步骤303、从未压缩的标识符中读取第一对,作为初始标识符。然后执行步骤304。
[0129]
步骤304、用通用编码器编码初始标识符,得到init_bits。
[0130]
在步骤304中,若为单端数据,则对初始标识符用编码范围覆盖ascii码在[32,127]内所有字符的编码器进行编码,编码结果为init_bits。编码结束后,执行步骤305。
[0131]
在步骤304中,若为双端数据,则对read1和read2的初始标识符用编码范围覆盖ascii码在[32,127]内所有字符的编码器分别进行编码,得到两个二进制流,合称为init_bits。编码结束后,执行步骤306和307。
[0132]
步骤305、判定并记录初始标识符的文本格式,记为init_format。
[0133]
参见图3b,对单端测序得到的生物序列的标识符进行格式判定,与对双端测序中read1的标识符进行格式判定,其方法是一样的。这里依照连续符号或连续非符号的拆分规则进行拆分,得到init_format包括但不限于:窗口数量为16,包括8个符号窗口,5个纯数字
窗口,3个无符号非纯数字窗口。然后,第四和第六窗口都为纯数字且数值相同,记为相同内容窗口,只编码第四窗口。
[0134]
在一个实施例中,步骤305之后,判断是否已处理完所有标识符,也即fastq文件中是否还存在未被压缩的标识符,若判断结果为是,则执行步骤316,若判断结果为否,则执行步骤308。
[0135]
步骤306、判定并记录read1初始标识符的格式,记为init_format。
[0136]
在步骤306中,格式判定方法与步骤305类似,此处不再赘述。
[0137]
步骤307、判定并记录read2初始标识符与read1初始标识符的差异信息init_pe_diff。
[0138]
参见图3b,对read2和read1的标识符进行差异查找,将「末位1变为2」记为init_pe_diff。值得注意的是,由于read2和read1都包含长度信息,且read1与read2不等长,因此在规则中,对read2中形如「length=」后跟随的纯数字子标识符,将其放入init_format中,不计入init_pe_diff。
[0139]
在一个实施例中,执行完步骤306和307,判断是否已处理完所有标识符,也即fastq文件中是否还存在未被压缩的标识符,若判断结果为是,则执行步骤316,若判断结果为否,则执行步骤309。
[0140]
步骤308、从未压缩的标识符中读取下一个,作为当前标识符。
[0141]
在步骤308中,读取结束后执行步骤310。
[0142]
步骤309、从未压缩的标识符中读取下一对,作为当前标识符。
[0143]
在步骤309中,读取结束后执行步骤311。
[0144]
步骤310、确定当前标识符的文本格式current_format。
[0145]
在步骤310中,格式判定方法与步骤305类似,此处不再赘述。然后执行步骤314。
[0146]
步骤311、确定read1当前标识符的文本格式current_format。
[0147]
在步骤311中,文本格式的判定方法与步骤305类似,此处不再赘述。然后执行步骤312。
[0148]
步骤312、比较当前标识符的read2与read1的区别,记录差异信息current_pe_diff。
[0149]
在步骤312中,差异信息确定方法与步骤307类似,此处不再赘述。然后执行步骤313。
[0150]
步骤313、对init_pe_diff和current_pe_diff进行匹配。
[0151]
在步骤313中,只需要比较init_pe_diff和current_pe_diff是否相同即可。若匹配,执行步骤314,否则执行步骤316。
[0152]
步骤314、对init_format和current_format进行匹配。
[0153]
在步骤314中,对于init_format和current_format进行比较。主要规则包含但不限于:若两者的窗口数量不同,则确定两者不相同,结束比较;若窗口数量相同,则对窗口对应的子标识符的属性两两进行比较,若子标识符的属性不同则确定两者不相同,结束比较;若子标识符的属性相同,在纯符号或非符号且非纯数字的窗口中,出现子标识符的字符长度不同,则确定两者不相同;在纯符号窗口中,出现子标识符内容不同,则确定两者不相同,结束比较;对于重复内容窗口,其内容不完全相同,则确定两者不相同,结束比较。
[0154]
参见图3b,标识符“@test123.4 4c009r042_292727length=717/1”与“@test123.5 5c009r043_292735length=675/1”的文本格式相同,因为各个窗口的属性和内容都符合规则,但与标识符“@test123.10c010r023_310021length=589/1”的文本格式就不一致,因为其窗口数量不同。
[0155]
在步骤314中,若二者相同,执行步骤315,否则执行步骤316。
[0156]
步骤315、在各个窗口中编码各个子标识符。
[0157]
参见图3b,各个子标识符在其所属的窗口中编码,每个窗口都有独立的编码器,生成独立的二进制流。
[0158]
在编码的过程中,文本格式相同的标识符,其拆分出的子标识符用相同的编码器以相同编码规则编码,实现高压缩率。
[0159]
参见图3c,对于非符号且非纯数字的窗口,每位都用独立的编码器进行编码。图中的“c009r042”与“c009r043”表示一个窗口中的两个子标识符,两个子标识符存在差异特征,需要进行编码。
[0160]
参见图3d,图中的“292727”与“292735”表示一个纯数字窗口中的两个子标识符,用同一编码器按照纯数字的编码规则编码。对于纯数字的窗口,由于fastq文件中的纯数字通常较为接近,差值较小,因此使用一种新的编码方式,即将每个数字转化成它与该窗口的上一个数字的差值,然后对该差值的正负号、差值绝对值用n进制表示的长度、转为n进制后在各个位上的数值进行编码。
[0161]
在步骤315中,编码结束后,若已处理完所有标识符,则执行步骤316,否则执行步骤308(单端数据)或步骤309(双端数据)。
[0162]
步骤316、输出init_bits,各个窗口输出编码结果。
[0163]
每个窗口的编码结果为二进制流,将该二进制流依次完整输出到结果文件中,保证可完整解压即可。
[0164]
在一个实施例中,用封装格式进行封装后输出(即在每个窗口的二进制流前面加上该二进制流的长度和属性等信息),可以提升可靠性和可维护性。在步骤316中,若该步骤是由313或314触发的,则在输出结束后,执行步骤302(单端数据)或步骤303(双端数据);若该步骤是由315或305或307触发的,则压缩结束。
[0165]
对标识符的解压过程是对标识符的压缩过程的逆过程,首先确定用于对标识符的二进制流进行解压的解压信息;其中,二进制流由上述任一实施例提供的生物序列标识符的压缩方法对标识符进行压缩得到,解压信息根据对标识符进行压缩的过程中生成的文本格式所确定,根据该解压信息即可对二进制流进行解压,得到生物序列的标识符。
[0166]
下面详细介绍标识符的解码过程。
[0167]
图4是本发明一示例性实施例提供的一种生物序列标识符的解压方法的流程图,该解压方法包括以下步骤:
[0168]
步骤401、从压缩文件中解压出部分信息,获知基因测序文件是单端测序或双端测序。
[0169]
在步骤401中,解压的信息来自于步骤101中,被写入到了压缩文件中的「单端测序或双端测序」信息。
[0170]
步骤402、将init_bits解码得到初始标识符的文本,对初始标识符进行格式判定。
[0171]
在步骤402中,对于单端数据和双端数据,init_bits的解码方式是不同的。在单端数据中,init_bits只会有一段二进制流;在双端数据中,init_bits可以被视为两段二进制流。
[0172]
在步骤402中,若是单端数据,只需要判定得到init_format即可,而双端数据还需要判定init_pe_diff。
[0173]
在步骤402中,对初始标识符的文本格式判定与压缩是一致的,因此文本格式判定的结果也是一致的。
[0174]
另外,若编码时选择不记录init_bits,而是记录init_format,并直接用各个窗口对初始标识符的各个子标识符进行编码的方式进行,则在这一步不是对init_bits解码,而是对init_format进行解码。
[0175]
步骤403、基于文本格式,对各个窗口的二进制流进行解压,得到各个窗口的子标识符。
[0176]
步骤403中,也即基于文本格式确定对应的编码规则,使用该编码规则对各个窗口的二进制流进行解压,得到各个窗口的子标识符。
[0177]
在步骤403中,在已经获知编码格式文本格式的情况下,对于需要编码的窗口,相应地初始化各个编码器,然后对压缩文件中的各个窗口的二进制流进行解压,获得各个窗口的子标识符文本。
[0178]
步骤404、将解码得到的各个子标识符拼接成原始的生物序列标识符。
[0179]
步骤405、若文件未解压完毕,进入步骤402。
[0180]
在步骤405中,若文件未解压完毕,说明该文件在压缩时,下一个标识符出现了格式不相同的情况,因此要进入步骤402,重新开始解压init_bits和各个窗口的二进制流。
[0181]
图5是本发明一示例性实施例提供的另一种生物序列标识符的解压方法的流程图,该解压方法包括以下步骤:
[0182]
步骤501、判断是单端测序还是双端测序。
[0183]
其中,「是单端测序还是双端测序」信息在上述任一实施例提供的生物序列标识符的压缩方法中,在基于文件数量判定了单端或双端后,写入到了压缩文件中。
[0184]
在步骤501中,若是单端数据,执行步骤502,否则执行步骤503。
[0185]
步骤502、解码init_bits得到初始标识符的文本。
[0186]
在步骤502中,获取init_bits后用解码器进行解码,该解码器与压缩init_bits的编码器相对应即可。解码完毕后,执行步骤504。
[0187]
步骤503、解码init_bits得到初始标识符的文本。
[0188]
在步骤503中,获取init_bits后用解码器进行解码,read1和read2的二进制流基于封装格式进行区分,解码器与压缩init_bits的编码器相对应即可。解码完毕后,执行步骤505。
[0189]
步骤504、判定初始标识符的格式init_format。
[0190]
在步骤504中,格式判定方法与步骤305一致,不再赘述。判定完毕后,执行步骤507。
[0191]
步骤505、判定read1的初始标识符格式init_format。
[0192]
在步骤505中,格式判定方法与步骤504一致,不再赘述。判定完毕后,执行步骤
506。
[0193]
步骤506、判定init_pe_diff。
[0194]
在步骤506中,差异信息判定方法与步骤307一致,不再赘述。判定完毕后,执行步骤508。
[0195]
步骤507、对于各个需要编码的窗口,初始化编码器,从压缩文件中获取相应的二进制流,解压出各个子标识符的文本。
[0196]
其中,窗口信息已经在步骤504中得到,对于那些需要编码的窗口(如纯数字窗口、非符号且非纯数字窗口)初始化相应的解码器,从压缩文件中获取相应的二进制流(若各个二进制流在压缩时已基于封装格式保留,则此时获取会较为便捷),并经由各个解码器解码得到各个子标识符的文本。
[0197]
在步骤507中,解码完毕后执行步骤509。
[0198]
步骤508、对于各个需要编码的窗口,初始化编码器,从压缩文件中获取相应的二进制流,解压出各个子标识符的文本。
[0199]
在步骤508中,处理流程与步骤507一致,只是处理对象替换成read1,不再赘述。处理完毕后执行步骤510。
[0200]
步骤509、将各个子标识符拼接成原始的标识符。
[0201]
其中,由于窗口信息已经在步骤504中得到,对于无需编码的窗口,其信息在每条标识符上都是相同的,对于需要编码的窗口,其信息在每条标识符上是变化的。总的来说,拼接过程就是判定current_format的逆过程。
[0202]
在步骤509中,拼接完毕后执行步骤512。
[0203]
步骤510、将各个子标识符拼接成原始的read1标识符。
[0204]
在步骤510中,处理流程与步骤509一致,只是处理对象替换成read1,不再赘述。拼接完毕后,执行步骤511。
[0205]
步骤511、基于init_pe_diff和read1标识符,获得read2标识符。
[0206]
每个read2标识符,都是在其对应的read1标识符的基础上,基于init_pe_diff进行变换得到的。
[0207]
在步骤511中,read2标识符恢复完毕后执行步骤512。
[0208]
步骤512、将标识符文本输出到解压文件中。
[0209]
在步骤512中,在输出结束后,若压缩文件已解压完毕,则结束程序,否则执行步骤502(单端数据)或步骤503(双端数据)。
[0210]
与前述生物序列标识的压缩方法、解压方法实施例相对应,本发明还提供了生物序列标识的压缩装置、解压装置的实施例。
[0211]
本发明实施例提供一种生物序列标识符的编码装置,用于对生物序列fastq文件中包含的多段生物序列的标识符进行编码;所述编码装置包括:
[0212]
拆分模块,用于对于基因测序文件中的每个标识符,将所述标识符拆分成若干子标识符;
[0213]
定义模块,用于定义若干窗口的编码规则,所述编码规则与所述子标识符的文本格式相匹配;
[0214]
拆分模块,用于将具有相同文本格式的子标识符划分至相同的窗口;
[0215]
编码模块,用于对于各个窗口,根据对应的编码规则对所述窗口中的所有子标识符进行编码,并将各个窗口的编码结果汇总成所述标识符的压缩结果。
[0216]
可选地,所述拆分模块包括:
[0217]
确定单元,用于确定所述标识符是基于单端测序得到的生物序列的标识符,还是基于双端测序得到的生物序列的标识符;
[0218]
拆分单元,用于在确定所述标识符是基于单端测序得到的生物序列的标识符的情况下,根据预定义的拆分规则将所述标识符的拆分成对应于各个窗口的子标识符;所述窗口的数量和/或各个窗口的属性由初始被拆分的标识符的子标识符的文本格式确定。
[0219]
所述拆分单元,还用于在确定所述标识符是基于双端测序得到的生物序列的标识符的情况下,根据预定义的拆分规则分别将所述标识符的第一片段read1和第二片段read2拆分成对应于各个窗口的子标识符。
[0220]
可选地,若所述标识符是基于双端测序得到的生物序列的标识符,所述压缩装置还包括:
[0221]
确定模块,用于确定所述第一片段read1的各个子标识符与所述第二片段read2中相同位序的子标识符的差异信息;并将所述第一片段的子标识符的文本格式以及所述差异信息确定为所述标识符的文本格式,或者将所述第二片段的子标识符的文本格式以及所述差异信息确定为所述标识符的文本格式。
[0222]
可选地,所述拆分规则包括:按照连续符号及连续非符号进行拆分。
[0223]
可选地,所述编码模块用于:根据各个子标识符的文本格式确定所述标识符的文本格式,并对具有相同文本格式的标识符进行共同编码。
[0224]
本发明实施例还提供一种生物序列标识符的解码装置,包括:
[0225]
确定模块,用于确定用于对标识符的二进制流进行解压的解压信息;其中,所述二进制流由上述任一实施例所述的生物序列标识符的压缩方法对标识符进行压缩得到,所述解压信息根据对所述标识符进行雅压缩的过程中生成的文本格式所确定;
[0226]
解压模块,用于根据所述解压信息对所述二进制流进行解压,得到生物序列的标识符。
[0227]
对于装置实施例而言,由于其基本对应于方法实施例,所以相关之处参见方法实施例的部分说明即可。以上所描述的装置实施例仅仅是示意性的,其中所述作为分离部件说明的单元可以是或者也可以不是物理上分开的,作为单元显示的部件可以是或者也可以不是物理单元,即可以位于一个地方,或者也可以分布到多个网络单元上。可以根据实际的需要选择其中的部分或者全部模块来实现本发明方案的目的。本领域普通技术人员在不付出创造性劳动的情况下,即可以理解并实施。
[0228]
图6是本发明一示例实施例示出的一种电子设备的结构示意图,示出了适于用来实现本发明实施方式的示例性电子设备60的框图。图6显示的电子设备60仅仅是一个示例,不应对本发明实施例的功能和使用范围带来任何限制。
[0229]
如图6所示,电子设备60可以以通用计算设备的形式表现,例如其可以为服务器设备。电子设备60的组件可以包括但不限于:上述至少一个处理器61、上述至少一个存储器62、连接不同系统组件(包括存储器62和处理器61)的总线63。
[0230]
总线63包括数据总线、地址总线和控制总线。
[0231]
存储器62可以包括易失性存储器,例如随机存取存储器(ram)621和/或高速缓存存储器622,还可以进一步包括只读存储器(rom)623。
[0232]
存储器62还可以包括具有一组(至少一个)程序模块624的程序工具625(或实用工具),这样的程序模块624包括但不限于:操作系统、一个或者多个应用程序、其它程序模块以及程序数据,这些示例中的每一个或某种组合中可能包括网络环境的实现。
[0233]
处理器61通过运行存储在存储器62中的计算机程序,从而执行各种功能应用以及数据处理,例如上述任一实施例所提供的方法。
[0234]
电子设备60也可以与一个或多个外部设备64(例如键盘、指向设备等)通信。这种通信可以通过输入/输出(i/o)接口65进行。并且,模型生成的电子设备60还可以通过网络适配器66与一个或者多个网络(例如局域网(lan),广域网(wan)和/或公共网络,例如因特网)通信。如图所示,网络适配器66通过总线63与模型生成的电子设备60的其它模块通信。应当明白,尽管图中未示出,可以结合模型生成的电子设备60使用其它硬件和/或软件模块,包括但不限于:微代码、设备驱动器、冗余处理器、外部磁盘驱动阵列、raid(磁盘阵列)系统、磁带驱动器以及数据备份存储系统等。
[0235]
应当注意,尽管在上文详细描述中提及了电子设备的若干单元/模块或子单元/模块,但是这种拆分仅仅是示例性的并非强制性的。实际上,根据本发明的实施方式,上文描述的两个或更多单元/模块的特征和功能可以在一个单元/模块中具体化。反之,上文描述的一个单元/模块的特征和功能可以进一步拆分为由多个单元/模块来具体化。
[0236]
本发明实施例还提供一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现上述任一实施例所提供的方法。
[0237]
虽然以上描述了本发明的具体实施方式,但是本领域的技术人员应当理解,这仅是举例说明,本发明的保护范围是由所附权利要求书限定的。本领域的技术人员在不背离本发明的原理和实质的前提下,可以对这些实施方式做出多种变更或修改,但这些变更和修改均落入本发明的保护范围。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。
