一种snp变异率的批量组间对比分析方法
技术领域
1.本发明涉及检测疾病技术领域,具体来说,涉及一种snp变异率的批量组间对比分析方法。
背景技术:
2.新一代技术的迅猛发展在数据通量和成本上都显示出巨大的优势。尤其是全外显子组捕获测序技术wes针对外显子功能区域进行深度测序,可以更全面地检测编码区域的变异,美国医学遗传学与基因组学学会(acma)制定了序列变异指南,利用新一代测序技术,临床实验检测遗传性疾病的产品种类不断增加,包括基因分型、单基因、基因包、外显子组、基因组、转录组和表观遗传学检测。
3.在过去的十年中,随着新一代高通量测序的出现,测序技术有了快速发展,但随着技术的复杂性日益增加,基因检测在序列解读方面不断面临着新的挑战,虽然acmg工作组制定并不断修订了序列变异解读的标准和指南,但仍然存在大量临床意义不明确的变异,给临床医生的解读带来了困难。
4.针对上述问题,目前还没有有效的解决办法。
技术实现要素:
5.针对相关技术中的上述技术问题,本发明提出一种snp变异率的批量组间对比分析方法,利用基因组snp变异率筛查来寻找遗传疾病/性状易感基因,能够克服现有技术的上述不足。
6.为实现上述技术目的,本发明的技术方案是这样实现的:
7.一种snp变异率的批量组间对比分析方法,包括如下步骤:
8.s1病例分组:按照纳排标准将病历分为病例组和对照组;
9.s2组内snp变异数据整合:vcf文件储存病例组和对照组整个测序数据通过对比参考基因后得到的变异结果,并将病例组和对照组的snp变异数据分别进行整合;
10.s3计算整合数据中每个snp变异数:计算病例组和对照组的各snp变异在各组中所占变异数;
11.s4计算整合数据中每个snp变异频率:计算病例组和对照组的各snp变异在各组中所占变异频率;
12.s5组间snp变异频率差异分析:批量用卡方检验计算病例组和对照组的各snp变异频率的显著性差异。
13.进一步地,步骤s1中所述纳排标准包括纳入标准和排除标准。
14.进一步地,步骤s1中所述纳排标准为无规则的自由文本形式。
15.进一步地,步骤s5中所述卡方检验是统计样本的实际观测值与理论推断值之间的偏离程度,实际观测值与理论推断值之间的偏离程度决定卡方值的大小。
16.本发明的有益效果:本发明通过基因组snp变异率筛查来寻找遗传疾病/性状易感
基因,通过snp变异率的批量组间对比分析有效地提高了疾病风险基因的筛选效率,实现了低成本、更高效地找到遗传标记与疾病间的关联,为复杂疾病的发病机制提供了更多的线索。
附图说明
17.为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
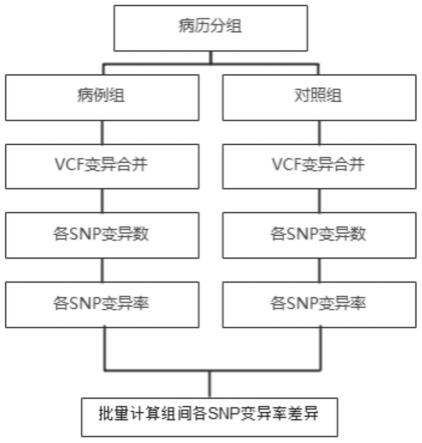
18.图1是根据本发明实施例所述的snp变异率的批量组间对比分析方法的操作流程示意图。
具体实施方式
19.下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员所获得的所有其他实施例,都属于本发明保护的范围。
20.如图1所示,根据本发明实施例所述的一种snp变异率的批量组间对比分析方法,包括如下步骤:
21.s1病例分组:按照纳排标准将病历分为病例组和对照组;
22.s2组内snp变异数据整合:vcf文件储存病例组和对照组整个测序数据通过对比参考基因后得到的变异结果,并将病例组和对照组的snp变异数据分别进行整合;
23.s3计算整合数据中每个snp变异数:计算病例组和对照组的各snp变异在各组中所占变异数;
24.s4计算整合数据中每个snp变异频率:计算病例组和对照组的各snp变异在各组中所占变异频率;
25.s5组间snp变异频率差异分析:批量用卡方检验计算病例组和对照组的各snp变异频率的显著性差异。
26.实施例,步骤s1中所述纳排标准包括纳入标准和排除标准。步骤s1中所述纳排标准为无规则的自由文本形式。
27.实施例,步骤s5中所述卡方检验是统计样本的实际观测值与理论推断值之间的偏离程度,实际观测值与理论推断值之间的偏离程度决定卡方值的大小。
28.为了方便理解本发明的上述技术方案,以下通过具体使用方式上对本发明的上述技术方案进行详细说明。
29.在具体使用时,根据本发明所述的一种snp变异率的批量组间对比分析方法,包括如下步骤:
30.1)病例分组
31.按照纳排标准进行病例组和对照组的分组
32.2)组内snp变异数据整合
33.vcf文件储存了整个测序数据通过对比参考基因后得到的变异结果,将病例组和对照组的snp变异数据分别进行整合。
34.3)计算整合数据中每一个snp变异数
35.计算病例组和对照组的各snp变异在各组中所占变异数。
36.4)计算整合数据中每一个snp变异频率
37.计算病例组和对照组的各snp变异在各组中所占变异频率。
38.5)组间snp变异频率差异分析
39.批量用卡方检验计算病例组和对照组的各snp变异频率的显著性差异。
40.具体实施时,snp变异率的批量组间对比分析步骤如下:
41.通过配置实验和对照组样本,使用本程序进行变异位点对比分析。
42.setting:设置case和control组的样本id和工作文件夹。
[0043][0044]
1.根据样本id获取储存的样本变异文件
[0045]
通过遍历对比储存库中的文件,获得文件名称进行拼接,用于vcf合并判断样本文件是否存在。
[0046][0047][0048]
2.使用bcftools合并两组vcf变异文件
[0049]
使用两个线程对control和case组的vcf文件进行合并。
[0050][0051][0052]
3.提取合并的变异文件中每个位点的数量
[0053]
[0054][0055]
4.合并两个位点突变计数文件,并进行计算每个位点的突变差值,根据差值大小进行排序
[0056]
[0057][0058]
5.计算突变位点的卡方检验结果,并筛选p小于0.5的位点
[0059]
[0060][0061]
6.计算完成
[0062]
在工作目录中保存了以下结果文件
[0063]
├──
casecounts.csv
[0064]
├──
casemerge.vcf.gz
[0065]
├──
controlcounts.csv
[0066]
├──
controlmerge.vcf.gz
[0067]
├──
merge.csv
[0068]
├──
merge_sort.csv
[0069]
├─
merge_sort_pv.csv
[0070]
综上所述,借助于本发明的上述技术方案,通过基因组snp变异率筛查来寻找遗传疾病/性状易感基因,通过snp变异率的批量组间对比分析有效地提高了疾病风险基因的筛选效率,实现了低成本、更高效地找到遗传标记与疾病间的关联,为复杂疾病的发病机制提供了更多的线索。
[0071]
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。
