1.本发明涉及生物技术领域,特别涉及一种基于核苷酸二聚体为单体实现核酸序列高通量合成测序方法。
背景技术:
2.dna测序技术是分子生物学研究中最常用的技术,它的出现极大地推动了生物学的发展。最近几年发展起来的第二代dna测序技术则使得dna测序进入了高通量、低成本的时代。高通量测序技术是对传统测序技术的一次革命性提高,一次能同时对几百万甚至几千万条dna序列进行测序。目前主流的二代高通量测序技术主要有四个代表:illumina公司的solexa测序合成技术、roche公司的454合成测序技术、abi公司iontorrent合成测序技术以及solid测序技术。然而,目前高通量dna测序平台、尤其是主流的高通量dna测序平台仍然存在着较大的错误率。高错误率使得检测单核苷酸多态性(snp)或低丰度突变极其困难,限制了高通量dna测序平台的临床应用,例如主要基于snp的药物基因组学研究和主要基于低丰度突变的早期临床诊断。目前,sanger测序因其准确度高(99.999%)仍被认为是金标准,高通量dna测序平台的结果需要在临床实践中通过桑格测序进行验证。
3.在现有的高通量dna测序平台中,虽然solid连接测序平台由于测序时间、测序长度等方面的原因使得其实际应用受到限制,但其原始测序错误效率则分别比nextseq、454gs flx和ion torrent的合成测序平台低13、16、30和倍,是高通量dna测序平台最高的,而在15
×
测序深度下准确率可达99.999%,达到桑格测序因其准确度。solid测序技术的测序错误效率低是由于所有位置的碱基都检测两次,这使得该方法具有纠错和发现snp/突变位点的特点,即碱基检测两次的信息可以提供固有的校对功能,从而减少原始数据中的错误。
4.针对现有二代高通量dna合成测序平台测序错误率高的问题,本发明以核苷酸二聚体作为测序单体,通过两轮dna测序,即所有碱基检测两次的信息提供校对功能,从而减少原始数据中的错误,提高测序的准确性。本发明有助于提高高通量dna测序的准确性、提升测序长度,拓展高通量dna测序技术在识别低丰度序列的能力,拓展高通量dna测序技术在生物科学研究和临床早期诊断的进一步应用。
技术实现要素:
5.发明目的:本发明目的是提供一种3’端羟基可逆封闭核苷酸二聚体合成测序法,该方法提高序列测定的长度及准确度,实现待测核酸序列的高通量检测。
6.技术方案:3’端羟基可逆封闭核苷酸二聚体合成测序法,在聚合酶的作用下,核苷酸二聚体如果与测序引物杂交的dna模板中两个碱基完全配对,则发生核苷酸二聚体的聚合反应,测序引物延伸两个碱基;如果与dna模板中两个碱基不完全配对,则不发生核苷酸二聚体的聚合反应。
7.进一步地,所述核苷酸二聚体的基本结构为两个相同或者不同的核苷酸通过磷酸
酯键相连接、其中5’端还包括一个三磷酸基团。核苷酸二聚体包括十六种具体形式、即aa、cc、gg、tt、ac、ca、gt、tg、ag、ga、ct、tc、at、ta、cg、gc。
8.进一步地,所述核苷酸二聚体3’端羟基是被包括3
’‑
o-烯丙基、3
’‑
o-氰乙基、3
’‑
o-叠氮甲基、3
’‑
o-氨基等基团封闭。这些3’端修饰的核苷酸二聚体能够参与合成测序反应、且最多只能发生一个核苷酸二聚体的合成。3’端封闭基团在合适的反应条件下可以被脱出、并活化出核苷酸的3’端羟基。
9.进一步地,所述核苷酸二聚体是用染料标记的,标记的位置可以是5’端碱基位置,5’端磷酸末端,或者3’端羟基位置。特别地,当3’端羟基位置时,同时可以用染料替代权利要求3封闭基团的作用。其中,aa、cc、gg、tt均用第一种染料标记,ac、ca、gt、tg均用第二种染料标记,ag、ga、ct、tc均用第三种染料标记,at、ta、cg、gc均用第四种染料标记。所述的四种染料的发射波长不能相护干扰,且这些标记的染料可以通过化学或者光化学方法切割,切割后不影响dna序列的生物化学功能。
10.进一步地,待测dna模板片段是指单分子,或者以单分子为模板扩增的相同序列产物。待测dna模板包括可以是一种序列,也可是不同dna模板的阵列。
11.进一步地,待测dna模板片段测序信息的获得是通过比较四种不同染料的荧光强度确定的,即四种染料中荧光强度最大者定为该dna模板的有效测序信息、并记录该dna的测序信息以及位置坐标信息。
12.进一步地,待测dna模板的具体碱基信息是通过两轮测序来实现的,每轮测序获得具体的染料(或者编码)信息。两轮测序的测序引物相差一个碱基,其中一轮测序的测序引物对应测序模板一个已知的碱基。待测dna模板具体碱基信息是通过测序模板一个已知的碱基而依次解码二组碱基片段编码信息而得到的。特别地,对于有参考序列的基因组测序的再测序,两组核苷酸实时合成dna测序得到的编码即可以直接用于基因组参考序列的比对,而不需要对编码进行解码,而实现对基因组序列的再测序。进一步地,待测dna模板信息的获得是通过比较染料的荧光强度与背景数值来确定的,当包含测序模板一个已知碱基中四种标记染料中最强的荧光强度明显高于背景数值时,可以确定该dna模板为有效测序模板、并记录该dna的荧光强度以及位置坐标信息。步骤为:
13.a:大肠杆菌基因组全基因组模板制备:将目标基因组用超声破碎成大小为100-1000bp碱基的片段,并在连接酶的作用下将这些片段化核酸序列用一对序列已知道的通用连接子进行连接,其中连接子1的序列为:ctg ctg tac cgt aca gcc ttg gcc g,连接子2的序列为:cgc ttt cct ctc tat ggg cag tcg gtgat;并进行预扩增10个循环;然后凝胶电泳切割200-800bp dna片段,并纯化;将这些200-800bp dna片段进行乳液并行pcr反应或者桥式pcr,扩增片段化的大肠杆菌基因组片段、构建大肠杆菌基因组测序dna模板芯片上。
14.b:第一轮测序
15.a.测序引物杂交:将5’端固定的模板与能和3’端连接子互补的第一个引物杂交,杂交引物作为所有大肠杆菌基因组dna模板的测序引物(参见图2);
16.b.测序
17.(1)将100um四色标记的16个3
’‑
o-叠氮甲基修饰的核苷酸二聚体(参见图1和表1)和包括9
°
dna聚合酶的测序体系加入到反应池中进行合成测序反应(60℃反应3分钟),然后用10mm乙二胺四乙酸二钠(edta)缓冲液(ph=7~8)洗涤未参与反应的标记核苷酸二聚体,
成像、记录包含测序模板一个已知碱基中四种标记染料中最强的荧光强度明显高于背景数值时、确定该dna模板为有效测序模板、并记录该dna的荧光强度以及位置坐标信息;
18.(2)加入100mm三(2-羧乙基)膦(ph 8.0)在55℃下反应3分钟,然后用10mm edta缓冲液(ph=7~8)洗涤;
19.(3)按照上述(1)~(2)步骤循环进行合成测序反应,得到一组由编码1、2、3、4构成的测序信息。然后进行第二轮测序反应。
20.c:第二轮测序
21.a.用8m尿素在65℃下处理5分钟共2次,将第一轮测序反应中的测序引物、及其测序引物合成链清除,重新得到单链dna模板;
22.b.测序引物杂交:将5’端固定的模板与能和3’端连接子互补的第二个引物杂交,杂交引物作为所有大肠杆菌基因组dna模板的测序引物(参见图3);
23.c.测序
24.(1)将100um四色标记的16个3
’‑o‑‑
叠氮甲基修饰的核苷酸二聚体和包括9
°
dna聚合酶的测序体系加入到反应池中进行合成测序反应(60℃反应3分钟),然后用10mm乙二胺四乙酸二钠(edta)缓冲液(ph=7~8)洗涤未参与反应的标记核苷酸二聚体,成像、记录包含测序模板一个已知碱基中四种标记染料中最强的荧光强度明显高于背景数值时、确定该dna模板为有效测序模板、并记录该dna的荧光强度以及位置坐标信息;
25.(2)加入100mm三(2-羧乙基)膦(ph 8.0)在55℃下反应3分钟,然后用10mm edta缓冲液(ph=7~8)洗涤;
26.(3)按照上述(1)~(2)步骤循环进行合成测序反应,得到一组由编码1、2、3、4构成的测序信息。然后进行第二轮测序反应。
27.d.解码
28.利用每个模板两轮测序中得到的核苷酸二聚体编码信息,并利用包含测序模板一个已知碱基的核苷酸二聚体编码信息、解码组装出每个模板相应的碱基序列信息(参见图4);
29.e.序列组装
30.利用所有模板的碱基序列信息、并利用纠错及其snp识别原理(参见图5),组装成大肠杆菌基因组序列。
31.有益效果:本发明与现有技术相比,具有如下优势:
32.1.本发明采用四色标记的十六种方法进行测序反应核苷酸二聚体作为合成测序原料的最大好处是,所有碱基均相差一个碱基的两个测序引物测定两次,当测序信息存在比对序列时(如参考序列信息或者2
×
以上测序深度),通过比对,如果测序信息与比对信息是只有一个编码发生变化,则这个测序编码判断为测序错误;而当测序信息连续二个与参考序列编码不一样的,则判断为snp。因为这个碱基与前一个碱基,后一个碱基组成核苷酸二聚体而被测序两次,使得测序信息具有校对功能,从而校正原始数据中的错误,提高测序信息的准确性。
33.2.采用四色标记的十六种方法进行测序反应核苷酸二聚体作为合成测序的原料,保证了每种dna模板对合成测序的要求,错误合成少,减少了原始测序的错误;采用核苷酸二聚体作为合成测序原料,每个测序反应延伸两个碱基,大幅度提高合成测序的长度。
附图说明
34.图1是本发明方法的一种核苷酸二聚体单体结构示意图;
35.图2是本发明方法的第一轮测序原理示意图;
36.图3是本发明方法的第二轮测序信息的获得原理示意图;
37.图4是本发明方法的的解码原理示意图;
38.图5是本发明方法的的的纠错及其snp识别原理。
具体实施方式
39.本实施例是基于3’端羟基可逆封闭核苷酸二聚体合成测序法测定大肠杆菌全基因组:
40.1.标记的核苷酸二聚体:分别合成或者市场购买下列3’端羟基可逆封闭核苷酸二聚体:aa、cc、gg、tt、ac、ca、gt、tg、ag、ga、ct、tc、at、ta、cg、gc(参见图1和表1)。
41.2.大肠杆菌基因组全基因组模板制备:将目标基因组用超声破碎成大小为100-1000bp碱基的片段,并在连接酶的作用下将这些片段化核酸序列用一对序列已知道的通用连接子进行连接,其中连接子1的序列为:ctg ctg tac cgt aca gcc ttg gcc g,连接子2的序列为:cgc ttt cct ctc tat ggg cag tcg gtgat;并进行预扩增10个循环;然后凝胶电泳切割200-800bp dna片段,并纯化;将这些200-800bp dna片段进行乳液并行pcr反应或者桥式pcr,扩增片段化的大肠杆菌基因组片段、构建大肠杆菌基因组测序dna模板芯片上。
42.b:第一轮测序
43.a.测序引物杂交:将5’端固定的模板与能和3’端连接子互补的第一个引物杂交,杂交引物作为所有大肠杆菌基因组dna模板的测序引物(参见图2);
44.b.测序
45.(1)将100um四色标记的16个3
’‑
o-叠氮甲基修饰的核苷酸二聚体(参见图1和表1)和包括9
°
dna聚合酶的测序体系加入到反应池中进行合成测序反应(60℃反应3分钟),然后用10mm乙二胺四乙酸二钠(edta)缓冲液(ph=7~8)洗涤未参与反应的标记核苷酸二聚体,成像、记录包含测序模板一个已知碱基中四种标记染料中最强的荧光强度明显高于背景数值时、确定该dna模板为有效测序模板、并记录最强荧光强度对应的染料编码记录及位置坐标信息;
46.(2)加入100mm三(2-羧乙基)膦(ph 8.0)在55℃下反应3分钟,然后用10mm edta缓冲液(ph=7~8)洗涤;
47.(3)按照上述(1)~(2)步骤循环进行合成测序反应,得到一组由编码1、2、3、4构成的测序信息。然后进行第二轮测序反应。
48.c:第二轮测序
49.a.用8m尿素在65℃下处理5分钟共2次,将第一轮测序反应中的测序引物、及其测序引物合成链清除,重新得到单链dna模板;
50.b.测序引物杂交:将5’端固定的模板与能和3’端连接子互补的第二个引物杂交,杂交引物作为所有大肠杆菌基因组dna模板的测序引物(参见图3);
51.c.测序
52.(1)将100um四色标记的16个3
’‑
o-叠氮甲基修饰的核苷酸二聚体和包括90dna聚
合酶的测序体系加入到反应池中进行合成测序反应(60℃反应3分钟),然后用10mm的edta缓冲液(ph=7~8)洗涤未参与反应的标记核苷酸二聚体,成像、记录包含测序模板一个已知碱基中四种标记染料中最强的荧光强度明显高于背景数值时、确定该dna模板为有效测序模板、并记录该dna的荧光强度以及位置坐标信息;
53.(2)加入100mm三(2-羧乙基)膦(ph 8.0)在55℃下反应3分钟,然后用10mm edta缓冲液(ph=7~8)洗涤;
54.(3)按照上述(1)~(2)步骤循环进行合成测序反应,得到一组由编码1、2、3、4构成的测序信息。然后进行第二轮测序反应。
55.d.解码
56.利用每个模板两轮测序中得到的核苷酸二聚体编码信息,并利用包含测序模板一个已知碱基的核苷酸二聚体编码信息、解码组装出每个模板相应的碱基序列信息(参见图4);
57.e.序列组装
58.利用所有模板的碱基序列信息、并利用纠错及其snp识别原理(参见图5),组装成大肠杆菌基因组序列。
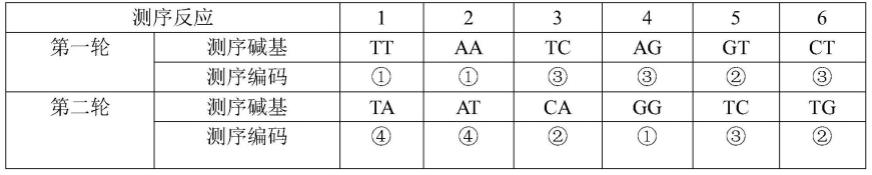
59.表1核苷酸二聚体的编码
60.编码核苷酸二聚体标记的染料1aa、cc、gg、ttfitc(fluorescein isothiocyanate):异硫氰酸荧光素2ac、ca、gt、tgcy3(cyanine 3):花青素33ag、ga、ct、tctexas red:德克萨斯红4at、ta、cg、gccy5(cyanine 5):花青素5
61.表2基于核苷酸二聚体为单体的合成测序方法对待测3
’‑
(t)taatcaggtctg-5’序列(其中括号中(t)为已知序列)获得测序编码信息
[0062][0063]
关于附图的解释说明:
[0064]
图1是一种基于核苷酸二聚体为单体的合成测序方法的一种核苷酸二聚体单体结构。其中3端羟基用叠氮基团可逆封闭,碱基2上连接可切割的荧光基团。
[0065]
图2是一种基于核苷酸二聚体为单体的合成测序方法第一轮测序原理。其中1为dna模板,1-1、1-2为dna模板两端连接的已知公用序列片段,2为基片,3为测序引物。测序引物杂交固定在基片3上的dna模板1构成测序芯片,加入16种标记四色染料的核苷酸二聚体,在dna聚合酶及其反应体系下发生聚合测序反应(1),洗涤未参与反应的标记核苷酸二聚体,成像、并记录四种染料的荧光强度,当四种染料中最强的荧光强度明显高于背景数值时、确定该dna模板为有效测序模板、并记录最强荧光强度对应的染料编码以及位置坐标信息。然后,加入切割试剂发生切割反应(2),切割标记染料以及活化出3’端羟基、以便发生下一个聚合测序反应(1)。循环进行(1)、(2),直到第一轮测序完成,获得每次测序反应dna模
板的测序信息。最后用8m尿素在65℃下处理5分钟(3)共2次,将第一轮测序反应中的测序引物、及其测序引物合成链清除,重新得到单链dna模板,以便进行第二轮测序。
[0066]
图3是一种基于核苷酸二聚体为单体的合成测序方法第二轮测序信息的获得原理。其中1为dna模板,1-1、1-2为dna模板两端连接的已知公用序列片段,2为基片,3为测序引物。测序引物杂交固定在基片3上的dna模板1构成测序芯片,加入16种标记四色染料的核苷酸二聚体,在dna聚合酶及其反应体系下发生聚合测序反应(1),洗涤未参与反应的标记核苷酸二聚体,成像、并记录四种染料的荧光强度,当四种染料中最强的荧光强度明显高于背景数值时、确定该dna模板为有效测序模板、并记录最强荧光强度对应的染料编码以及位置坐标信息。然后,加入切割试剂发生切割反应(2),切割标记染料以及活化出3’端羟基、以便发生下一个聚合测序反应(1)。循环进行(1)、(2),直到第二轮测序完成,获得每次测序反应dna模板的测序信息。
[0067]
图4是一种基于核苷酸二聚体为单体的合成测序方法解码原理。基于核苷酸二聚体为单体的合成测序方法对待测3
’‑
(t)taatcaggtctg-5
‘
序列(其中括号中(t)为已知序列)获得测序编码信息。其中,左图第一行为第一轮测序获得的染料编码信息,第二行为第一轮测序编码信息解码的核苷酸二聚体碱基信息,第三行为第一轮测序编码在已知第一个碱基信息的前提下,依次推知核苷酸二聚体碱基信息的顺序;第四行为第二轮测序编码信息解码的核苷酸二聚体碱基信息;第五行为第二轮测序获得的染料编码信息;第六行为第二轮测序编码在推知第一轮碱基信息的前提下,依次推知核苷酸二聚体碱基信息的顺序。右图为两轮测序合并的信息,第一行为待测dna模板序列;第二行为两轮测序获得的染料编码信息,其中,单数位为第一轮测序信息,双数位为第二轮测序信息;第三行为已知第一个碱基信息的前提下,一句两轮测序获得的染料编码信息解码出的碱基信息。
[0068]
图5是一种基于核苷酸二聚体为单体的合成测序方法纠错及其snp识别原理。参考信息是指在数据库中已知的信息,或者同一位置碱基在不同dna模板同时测到的信息左图为测序信息与参考信息比对结果发现一个编码不同(对比结果三角形显示处),这个测序不同的编码判断为测序错误(在2
×
测序深度下、可以判断是否存在测序错误)、并将这个测序错误编码
②
校正为正确编码
③
;右图测序信息与参考信息比对结果发现连续两个编码不同(对比结果三角形显示处),这个测序信息连续两个不同的编码判断为snp(在3
×
以上测序深度下,可以依据概率纠正测序错误),并确定这个snp由参考序列碱基g变成了测序序列的碱基c。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。