1.本发明涉及计算机技术领域,特别是指一种提取阿兹海默症的显著特征的电子设备。
背景技术:
2.阿尔茨海默症(ad)以一系列显著的痴呆症状为特征,常见于老年人。ad的发病机制复杂,其发病因素横跨宏观到微观多个层次,例如,大脑功能与结构的病变与基因的异常表达密切相关,因此,找到大脑宏观病变与微观变异的关联模式有助于揭示阿尔茨海默症的多层次发病机制。一些研究人员构建网络,将基因或大脑区域抽象为节点,以研究致病因素之间的功能相关性。huang等人定义了每个脑区的嵌入表示,并提出了一种新的度量评估两个区域之间的相似性,使测量结果更加合理。lella等人在分析大脑网络时引入了沟通距离,提供了新的3种方法发现与ad发展相关的隐藏模式。
3.然而,现有技术基于目前方法分析得到的脑区网络和基因网络进行ad诊断的准确率比较低,有必要提供一种更为精准确定ad的风险基因和病变脑区方法,以便助力对应的靶向药物开发和针对性经颅磁治疗。
技术实现要素:
4.有鉴于此,本发明的目的在于提出一种提取阿兹海默症的显著特征的电子设备,可以更为精准确定ad的显著特征,即ad的风险基因和病变脑区,以便助力对应的靶向药物开发和针对性经颅磁治疗。
5.基于上述目的,本发明提供一种提取阿兹海默症的显著特征的电子设备,包括存储器、处理器以及存储在存储器上并可在处理器上运行的计算机程序,其特征在于,所述处理器执行所述计算机程序时用于实现:
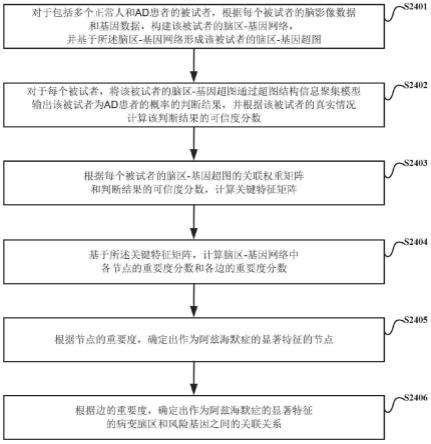
6.对于包括多个正常人和ad患者的被试者,根据每个被试者的脑影像数据和基因数据,构建该被试者的脑区-基因网络,并基于所述脑区-基因网络形成该被试者的脑区-基因超图;
7.对于每个被试者,将该被试者的脑区-基因超图通过超图结构信息聚集模型输出该被试者为ad患者的概率的判断结果,并根据该被试者的真实情况计算该判断结果的可信度分数;
8.根据每个被试者的脑区-基因超图的关联权重矩阵和判断结果的可信度分数,计算关键特征矩阵;
9.基于所述关键特征矩阵,计算脑区-基因网络中各节点的重要度分数;
10.根据节点的重要度,提取出作为阿兹海默症的显著特征的节点;
11.其中,所述超图结构信息聚集模型是以多个正常人的脑区-基因超图和ad患者的脑区-基因超图作为样本预先训练得到的。
12.进一步,所述处理器执行所述计算机程序还用于实现:
13.基于所述关键特征矩阵,计算脑区-基因网络中各边的重要度分数;
14.根据边的重要度,确定出作为阿兹海默症的显著特征的病变脑区和风险基因之间的关联关系。
15.较佳地,所述超图结构信息聚集模型包括:
16.生成器,用于将所述脑区-基因超图转换为脑区-基因边图和脑区-基因点图,并分别对所述脑区-基因边图和脑区-基因点图进行多次边级特征信息聚集与点级特征信息聚集,将信息聚集后的边图和点图进行融合得到低阶脑区-基因子超图;进而对所述低阶脑区-基因子超图进行结构信息扩散得到重构的脑区-基因超图;
17.判别器,用于对重构的脑区-基因超图进行多次边级-点级结构信息聚集得到高阶脑区-基因子超图;将所述高阶脑区-基因子超图和低阶脑区-基因子超图进行融合得到关键脑区-基因超图;进而根据所述关键脑区-基因超图输出该被试者为阿尔茨海默症ad患者的概率的判断结果。
18.较佳地,所述生成器中包括:
19.转换层模块,用于将所述脑区-基因超图转换为脑区-基因边图和脑区-基因点图,并输出所述脑区-基因边图的边权重矩阵和脑区-基因点图的点权重矩阵;
20.边卷积层模块,用于对所述脑区-基因边图的边权重矩阵进行多次边级特征信息聚集,得到关键脑区-基因边图的边权重矩阵;
21.点卷积层模块,用于对所脑区-基因点图的点权重矩阵进行多次点级特征信息聚集,得到关键脑区-基因点图的点权重矩阵;
22.第一融合层模块,用于将所述关键脑区-基因边图的边权重矩阵和关键脑区-基因点图的点权重矩阵进行融合得到低阶脑区-基因子超图的关联权重矩阵;
23.反卷积模块,用于对低阶脑区-基因子超图的关联权重矩阵通过反卷积操作进行结构信息扩散,得到重构的脑区-基因超图的关联权重矩阵。
24.较佳地,所述生成器中包括:
25.转换层模块,用于将所述脑区-基因超图转换为脑区-基因边图和脑区-基因点图,并输出所述脑区-基因边图的边权重矩阵和脑区-基因点图的点权重矩阵;
26.边卷积层模块,用于对所述脑区-基因边图的边权重矩阵进行多次边级特征信息聚集,得到关键脑区-基因边图的边权重矩阵;
27.点卷积层模块,用于对所脑区-基因点图的点权重矩阵进行多次点级特征信息聚集,得到关键脑区-基因点图的点权重矩阵;
28.第一融合层模块,用于将所述关键脑区-基因边图的边权重矩阵和关键脑区-基因点图的点权重矩阵进行融合得到低阶脑区-基因子超图的关联权重矩阵;
29.反卷积模块,用于对低阶脑区-基因子超图的关联权重矩阵通过反卷积操作进行结构信息扩散,得到重构的脑区-基因超图的关联权重矩阵。
30.较佳地,所述处理器执行所述计算机程序实现所述根据该被试者的真实情况计算该判断结果的可信度分数时,具体为:
31.根据如下公式29计算所述判断结果的可信度分数:
32.ri=ti×
piꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(公式29)
33.其中,pi为一个二维列向量,是超图结构信息聚集模型输出的第i个被试者为ad患
者的概率的判断结果;ti为一个二维行向量,是第i个被试者的真实标签,反映了第i爪被试者的真实情况;ri为计算得到的pi的可信度分数。
34.较佳地,所述处理器执行所述计算机程序实现所述根据每个被试者的脑区-基因超图的关联权重矩阵和判断结果的可信度分数,计算关键特征矩阵时,具体为:
35.根据如下公式30,计算关键特征矩阵:
[0036][0037]
其中,表示第i个被试者的关键脑区-基因超图的关联权重矩阵;ri表示超图结构信息聚集模型对第i个被试者的判断结果的可信度分数;m表示被试者的总数。
[0038]
较佳地,所述处理器执行所述计算机程序实现所述基于所述关键特征矩阵,计算脑区-基因网络中各节点的重要度分数时,具体为:
[0039]
根据如下公式32计算各节点的重要度分数:
[0040][0041]
其中表示关键特征矩阵hf第p行k列的值;表示节点v
p
的重要度分数。
[0042]
本发明还提供一种计算机可读存储介质,所述计算机可读存储介质内存储有计算机程序,所述计算机程序可被至少一个处理器所执行,以使所述至少一个处理器执行上述的电子设备的操作。
[0043]
本发明的技术方案中,对于包括多个正常人和ad患者的被试者,根据每个被试者的脑影像数据和基因数据,构建该被试者的脑区-基因网络,并基于所述脑区-基因网络形成该被试者的脑区-基因超图;对于每个被试者,将该被试者的脑区-基因超图通过超图结构信息聚集模型输出该被试者为ad患者的概率的判断结果,并根据该被试者的真实情况计算该判断结果的可信度分数;根据每个被试者的脑区-基因超图的关联权重矩阵和判断结果的可信度分数,计算关键特征矩阵;基于所述关键特征矩阵,计算脑区-基因网络中各节点的重要度分数;根据节点的重要度,提取出作为阿兹海默症的显著特征的节点;其中,所述超图结构信息聚集模型是以多个正常人的脑区-基因超图和ad患者的脑区-基因超图作为样本预先训练得到的。由于对脑区-基因超图进行低阶结构信息和高阶结构信息的聚集得到的关键脑区-基因超图,更能体现ad与正常个体的判断性特征;因此,根据关键脑区-基因超图判断被试者是正常人还是ad的概率更为准确,进而,根据超图结构信息聚集模型输出的更精准的判断,可以更为精准确定ad的显著特征,即ad的风险基因和病变脑区,以便助力对应的靶向药物开发和针对性经颅磁治疗。
附图说明
[0044]
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
[0045]
图1为本发明提供的超图结构信息聚集的过程的示意图;
[0046]
图2为本发明实施例一提供的超图结构信息聚集模型的内部结构框图;
[0047]
图3为本发明提供一的脑网络构建示意图;
[0048]
图4为本发明提供一的基因网络构建示意图;
[0049]
图5为本发明提供一的脑区-基因超图构建示意图;
[0050]
图6为本发明实施例一提供的生成器的内部结构示意图;
[0051]
图7为本发明实施例一提供的脑区-基因边图转换示意图;
[0052]
图8为本发明实施例一提供的脑区-基因点图转换示意图;
[0053]
图9为本发明实施例一提供的第t阶边卷积层的边卷积操作示意图;
[0054]
图10为本发明实施例一提供的t阶边卷积操作示意图;
[0055]
图11为本发明实施例一提供的第t阶边卷积层的点卷积操作示意图;
[0056]
图12为本发明实施例一提供的t阶点卷积操作示意图;
[0057]
图13为本发明实施例一提供的t阶扩散反卷积操作示意图;
[0058]
图14为本发明实施例一提供的扩散反卷积操作的意义的示意图;
[0059]
图15为本发明实施例一提供的判别器的内部结构示意图;
[0060]
图16为本发明实施例一提供的t阶边点卷积操作示意图;
[0061]
图17为本发明实施例一提供的边点卷积操作的意义的示意图;
[0062]
图18为本发明实施例一提供的全连接层模块的内部结构示意图;
[0063]
图19为本发明实施例二提供的超图结构信息聚集模型的训练方法流程图;
[0064]
图20为本发明实施例二提供的对所述生成器和判别器进行一次迭代的优化和训练的方法流程图;
[0065]
图21为本发明实施例二提供的对判别器进行优化的方法流程图;
[0066]
图22为本发明实施例二提供的对生成器进行训练的方法流程图;
[0067]
图23为本发明实施例三提供的基于结构信息聚集模型的阿尔茨海默症的判断方法的流程图;
[0068]
图24为本发明实施例四提供的一种提取阿尔茨海默症的显著特征的方法流程;
[0069]
图25为本发明实施例四提供的显著特征提取示意图;
[0070]
图26为本发明实施例五提供的电子设备硬件结构示意图。
具体实施方式
[0071]
为使本发明的目的、技术方案和优点更加清楚明白,以下结合具体实施例,并参照附图,对本发明进一步详细说明。
[0072]
需要说明的是,除非另外定义,本发明实施例使用的技术术语或者科学术语应当为本公开所属领域内具有一般技能的人士所理解的通常意义。本公开中使用的“第一”、“第二”以及类似的词语并不表示任何顺序、数量或者重要性,而只是用来区分不同的组成部分。“包括”或者“包含”等类似的词语意指出现该词前面的元件或者物件涵盖出现在该词后面列举的元件或者物件及其等同,而不排除其他元件或者物件。“连接”或者“相连”等类似的词语并非限定于物理的或者机械的连接,而是可以包括电性的连接,不管是直接的还是间接的。“上”、“下”、“左”、“右”等仅用于表示相对位置关系,当被描述对象的绝对位置改变后,则该相对位置关系也可能相应地改变。
[0073]
本发明的发明人考虑到,脑区-基因超图中的每个节点代表着一个脑区或基因,脑区或基因越异常,对应的节点及连接这些节点的超边越能代表网络的特征性结构信息,也越能作为区分ad与正常个体的判断性特征。通过超图结构信息聚集模型对脑区-基因超图进行低阶结构信息和高阶结构信息的聚集得到的关键脑区-基因超图,更能体现ad与正常个体的判断性特征;因此,根据关键脑区-基因超图判断被试者是正常人还是ad的概率更为准确,可以辅助于对阿尔茨海默症做更精准的诊断。
[0074]
对于低阶结构信息,首先,将脑区-基因超图转换为脑区-基因边图和脑区-基因点图;其次,分别使边图与点图经过多次(比如t1次)边级特征信息聚集与点级特征信息聚集;最后,将信息聚集后的边图与点图融合,得到代表低阶结构信息的低阶脑区-基因子超图。对于高阶结构信息,对脑区-基因超图进行多次(比如t2次)边级-点级结构信息聚集,得到代表高阶结构信息的高阶脑区-基因子超图;最后,将低阶脑区-基因子超图和高阶脑区-基因子超图融合,得到关键脑区-基因超图。
[0075]
下面结合附图详细说明本发明实施例的技术方案。
[0076]
对脑区-基因超图进行超图结构信息聚集的过程,如图1所示。一次超图结构信息聚集可分为高阶结构信息聚集和低阶结构信息聚集两个部分。图1中(a)和(b)分别展示的是高阶结构信息聚集和低阶结构信息聚集的过程。具体地,首先,在脑区-基因超图的基础上进行多次高阶边级-点级结构信息聚集,得到代表着高阶结构信息的高阶脑区-基因子超图;其次,将初始的脑区-基因超图转换为脑区-基因边图和脑区-基因点图,并分别让它们经过多次边级结构信息聚集和点级结构信息聚集,得到关键脑区-基因边图和关键脑区-基因点图,融合关键边图和点图得到代表低阶结构信息的低阶脑区-基因子超图;最后,将低阶脑区-基因子超图和高阶脑区-基因子超图融合得到关键脑区-基因超图。在得到关键脑区-基因超图演变的过程中,原本的超图中所含的高阶和低阶结构信息得到聚集,从而更能体现ad与正常个体的判断性特征;因此,根据关键脑区-基因超图可以找到致病基因和病变脑区及其间的关联模式,根据关键脑区-基因超图判断被试者是正常人还是ad的概率更为准确,可以辅助于对阿尔茨海默症做更精准的诊断。
[0077]
下面针对脑区-基因超图建立超图结构信息聚集模型,模型的要素包括如下:
[0078]
(1)脑区-基因超图:hi表示第i个被试者的脑区-基因超图,由第i个被试者的基因数据和脑影像数据构建,i=1,...,m,其中m为被试者的个数。
[0079]
(2)脑区-基因边图:表示第i个被试者的脑区-基因边图,由第i个被试者的脑区-基因超图hi进行边图转换得到。边图中每一个节点对应超图中的一条超边,边权重的计算方法后述。
[0080]
(3)脑区-基因点图:表示第i个被试者的脑区-基因点图,由第i个被试的脑区-基因超图hi进行点图转换得到。点图中的节点与超图中一致,但边权重需重新计算,具体公式后述。
[0081]
(4)脑区-基因边图中连边的权重:其中n为脑区-基因超图中节点总数,
边表示的是超边e
p
和eq之间的边,表示初始脑区-基因边图中边的权重,通过计算脑区-基因超图中同时与超边e
p
和eq相连的节点与两超边所连节点总数的占比得到。表示第i个被试者在第t1次边级结构信息聚集后边的权重,权重越大表明超边e
p
和eq之间的联系程度越紧密。t1为边级特征信息聚集次数总数。
[0082]
(5)脑区-基因点图中连边的权重:其中n为脑区-基因超图中节点总数,也是脑区-基因点图中节点总数;边表示的是脑区-基因点图中节点v
p
和vq之间的边,表示初始脑区-基因点图中边的权重,通过计算脑区-基因超图中同时与节点v
p
和vq共连的超边与两节点所连超边总数的占比得到。表示第i个被试者第t1次点级结构信息聚集后边的权重,权重越大表明节点v
p
和vq之间的联系程度越紧密。t1为点级特征信息聚集次数总数。
[0083]
(6)低阶脑区-基因子超图:表示第i个被试者的低阶脑区-基因子超图,由第i个被试者的脑区-基因点图权重和脑区-基因边图权重融合得到,表示节点v
p
和超边eq之间的边。表示第i个被试者的低阶脑区-基因子超图中节点v
p
与超边eq之间的关联权重。
[0084]
(7)脑区-基因超图中节点v
p
与超边eq间的关联权重:表示节点v
p
与超边eq的关联权重,表示初始脑区-基因超图中v
p
与eq间的关联权重,值为1时表示v
p
与eq存在关联,值为0时表示v
p
与eq没有关联。表示在第t2次信息聚集后v
p
与eq的关联权重。t2为边级-点级结构信息聚集次数总数。
[0085]
(8)高阶脑区-基因子超图:表示第i个被试者的高阶脑区-基因子超图,由第i个被试者的脑区-基因超图hgi经过t2次边级-点级结构信息聚集得到,表示节点v
p
和超边eq之间的边,表示第i个被试者的高阶脑区-基因子超图中节点v
p
和超边eq之间的关联权重。
[0086]
(9)关键脑区-基因超图:表示第i个被试者的关键脑区-基因超图,由高阶脑区-基因子超图和低阶脑区-基因子超图
融合得到,表示节点v
p
和超边eq之间的边,表示第i个被试者的关键脑区-基因超图中节点v
p
和超边eq之间的关联权重。
[0087]
根据以上模型要素,超图结构信息聚集模型可分为以下四步:(1)脑区-基因超图进行低阶结构信息聚集得到低阶脑区-基因子超图;(2)脑区-基因超图进行高阶结构信息聚集得到高阶脑区-基因子超图;(3)融合低阶和高阶脑区-基因子超图得到关键脑区-基因超图;(4)分析关键脑区-基因超图,提取判断性特征。
[0088]
第一步:被试的脑区-基因超图进行低阶结构信息聚集得到低阶脑区-基因子超图。首先,脑区-基因超图进行边图和点图转换得到脑区-基因边图和脑区-基因点图,分别提取低阶的边级和点级结构信息,第i个超图转化的公式如公式1所示:
[0089][0090]
其中hi表示第i个被试者的脑区-基因超图;conv_v(
·
)表示点图转换函数;conv_e(
·
)表示边图转换函数;表示脑区-基因点图;表示脑区-基因边图。
[0091]
其次,脑区-基因边图和脑区-基因点图分别进行边级结构信息聚集和点级结构信息聚集,得到关键脑区-基因边图和关键脑区-基因点图。每次边级结构信息聚集过程中,脑区-基因边图中每条边聚集其邻域内的边级结构信息,进而更新自身边的权重,经过多次边级结构信息聚集后,原始的脑区-基因边图逐渐演变为关键脑区-基因边图。在第n次边级结构信息聚集过程中,第i个被试的脑区-基因边图中边权重的计算方式如公式2所示:
[0092][0093]
其中表示聚集脑区-基因边图中超边eq的边级结构信息。
[0094]
类似地,每次点级结构信息聚集过程中,脑区-基因点图中每条边聚集其邻域内的点级结构信息并更新自身边权重,多次信息聚集后,原始的脑区-基因点图演变为关键脑区-基因点图。在第t1次点级结构信息聚集过程中,第i个脑区-基因点图中边权重的计算方式如公式3所示:
[0095][0096]
其中表示聚集脑区-基因点图中节点v
p
的点级结构信息。
[0097]
最后,将经过了t1次特征信息更新后的权重与融合得到低阶脑区-基因子超图的关联权重公式如公式4所示:
[0098]
[0099]
其中,fuse(
·
)表示图融合函数;表示低阶脑区-基因子超图的关联权重。
[0100]
第二步:被试的脑区-基因超图进行高阶结构信息聚集得到高阶脑区-基因子超图。对每一个脑区-基因超图,在每次高阶结构信息聚集过程中,每条超边聚集其邻域内的高阶结构信息并更新自身的权重,经过多次信息聚集后,原始的脑区-基因超图逐渐演变为高阶脑区-基因子超图。在第t2次高阶结构信息聚集过程中第i个脑区-基因超图中边权重的计算方式如公式5所示:
[0101][0102]
其中,表示聚集所连接超边e
p
一阶邻域内的边级结构信息;聚集所连接节点v
p
一阶邻域内的点级结构信息;表示边聚集了其t2阶邻域内的结构信息。
[0103]
经过t2次高阶结构信息聚集后,得到高阶脑区-基因子超图的关联权重如公式6所示:
[0104][0105]
第三步:被试者的低阶脑区-基因子超图和高阶脑区-基因子超图融合得到关键脑区-基因超图。将聚集了低阶结构信息的低阶脑区-基因子超图关联权重与聚集了高阶结构信息的高阶脑区-基因子超图的关联权重进行融合,进而得到关键脑区-基因超图的关联权重
[0106][0107]
其中,fuse(*)表示图融合函数;表示关键脑区-基因超图的关联权重。
[0108]
第四步:经过结构信息聚集得到的关键脑区-基因超图蕴含着ad患者与正常人之间的区别性信息,可以完成正常人和ad患者的分类任务,公式如下:
[0109][0110]
其中,表示经过超图结构信息聚集后的关键脑区-基因超图的关联权重;classify(
·
)表示分类函数;pi为判断结果,含义为第i个被试者为正常人(nc)或ad的概率,是一个二维向量。
[0111]
根据判断结果提取出正常人和ad患者之间的判断性特征,即致病基因和病变脑区以及它们的关联模式,特征提取的公式如公式9所示:
[0112][0113]
其中,pi表示判断结果;表示关键脑区-基因超图的关联权重;feat_extract表示特征提取函数;pg、pb和am分别表示致病基因、病变脑区和它们之间的关联模式;score(
·
)为重要度分数,分数越大表明提取的特征在正常人和ad之间差异越大。
[0114]
综上所述,将公式1~公式9联立,用以下方程组表示脑区-基因超图的超图结构信息聚集模型和应用:
[0115][0116]
在这些公式中,公式1表示脑区-基因超图进行边图和点图转换得到脑区-基因边图和脑区-基因点图,公式2表示脑区-基因边图中的边聚集其邻域内的边级结构信息之和,公式3表示脑区-基因点图中的点聚集其邻域内的点级结构信息之和,公式4表示利用和融合构建低阶脑区-基因子超图关联权重公式5表示脑区-基因超图中的边聚集其邻域内的高阶边级-点级结构信息之和,公式7表示融合低阶脑区-基因子超图关联权重和高阶脑区-基因子超图关联权重h
(i,n)
得到关键脑区-基因超图关联权重公式8表示基于关键脑区-基因超图对正常人和ad患者分类,公式9表示利用判断结果和关键脑区-基因超图提取ad患者的致病基因和病变脑区及其间的关联模式。
[0117]
实施例一
[0118]
基于上述的超图结构信息聚集模型的要素,本发明实施例一提供了一种基于生成对抗网络的超图结构信息聚集模型,其内部结构如图2所示,包括:生成器201和判别器202;
[0119]
其中,生成器201用于将输入的脑区-基因超图转换为脑区-基因边图和脑区-基因点图,并分别对所述脑区-基因边图和脑区-基因点图进行多次边级特征信息聚集与点级特征信息聚集,将信息聚集后的边图和点图进行融合得到低阶脑区-基因子超图;进而对所述低阶脑区-基因子超图进行结构信息扩散得到重构的脑区-基因超图;
[0120]
判别器202用于对重构的脑区-基因超图进行多次边级-点级结构信息聚集得到高阶脑区-基因子超图;将所述高阶脑区-基因子超图和低阶脑区-基因子超图进行融合得到关键脑区-基因超图;进而根据所述关键脑区-基因超图输出该被试者为阿尔茨海默症ad患者的概率的判断结果。
[0121]
在由生成器201和判别器202组成的超图结构信息聚集模型中,生成器的输入是正常人或ad患者的脑区-基因超图,并由卷积部分和反卷积部分组成。卷积部分的作用是将脑区-基因超图转换为脑区-基因边图和脑区-基因点图,通过点卷积与边卷积分别聚集边级和点级结构信息,并将卷积后的边图与点图融合得到低阶脑区-基因子超图;反卷积部分的作用是通过扩散反卷积操作将低阶脑区-基因子超图逐步重构得到生成脑区-基因超图作为生成器的输出。
[0122]
判别器的输入有两种:由样本的原始数据构建的真实的脑区-基因超图和通过生成器得到的生成的脑区-基因超图,判别器的作用有三个,第一,通过边点卷积操作聚集超图中的高阶边级-点级结构信息得到高阶脑区-基因超图,并与低阶脑区-基因超图融合为关键脑区-基因超图;第二,判断输入的脑区-基因超图真假,即判断超图是由原始数据得到的还是通过生成器生成的;第三,预测样本的类别,即预测一个样本为ad患者或正常个体的概率。根据生成对抗网络的原理,在模型训练的过程中,判别器的目标是尽可能地提升超图的分类准确率,而生成器的目标是使生成的超图与真实的脑区-基因超图尽可能接近,从而使判别器难以分辨真假。训练过程中的二者存在相互对抗关系,同时网络中的参数会不断调优,直至整个网络收敛。训练结束后,生成对抗网络提取出的关键脑区-基因超图能够准确聚集ad疾病的特征信息,进而可以提取出致病基因和病变脑区以及它们之间的关联模式。
[0123]
如图3所示,以脑区为节点,脑区之间的连接为边,脑区和脑区之间的欧氏距离为边的权重。脑区和脑区之间欧式距离计算公式如公式10所示:
[0124][0125]
其中,dis(bi,bj)表示脑区节点bi和bj之间的欧式距离;l1表示脑区的时间序列长度;表示节点bi的第k个特征(脑区时间序列值);n1为脑网络中节点的总数。
[0126]
如图4所示,以基因为节点,基因之间的连接为边,基因和基因之间的欧式距离为边的权重。基因和基因之间欧式距离计算公式如下:
[0127][0128]
其中,dis(gi,gj)表示基因节点gi和gj之间的欧式距离;l2表示基因节点特征信息的长度;表示节点gi的第k个特征(基因序列值);n2为基因网络中节点的总数。
[0129]
将被试者(可以是正常人,也可以是ad患者)的脑网络和基因网络进行融合,得到该被试者的脑区-基因网络:以脑区和基因为节点,采用现有的皮尔逊相关分析方法计算节点之间边的权重,若边权重大于阈值则保留该边,反之去掉该边,以此构建脑区-基因网络,实现脑网络和基因网络的融合。
[0130]
如图5所示,基于被试者的脑网络和基因网络构建的脑区-基因网络,以每一个脑区节点或基因节点为中心节点,选择该中心节点的最近的k个邻居节点,形成一条超边:即选择k个欧式距离上与中心节点最近的邻居节点,将所述中心节点与选择的邻居节点之间的连接构成一条超边,即所述中心节点与选择的邻居节点共同作为所述超边所连接的节点。其中,k为本领域技术人员根据经验设置,比如,设置为3或5等。由于脑区-基因网络中有n个节点,因此,构建的脑区-基因超图含有n条超边,并对应一个关联权重矩阵h∈rn×n,其中关联权重矩阵的行表示脑区或基因节点,列表示超边,关联权重矩阵中的元素表示节点v
p
与超边eq的关联权重;若节点v
p
是超边eq上的一个节点,则表示v
p
与eq存在关联,值为1;否则,值为0,表示v
p
与eq没有关联。最后,通过关联权重矩阵h构建脑区-基因超图。相比于脑区-基因网络,在构建的脑区-基因超图中脑区或基因越异常,对应的节点及连接这些节点的超边越能代表网络的特征性结构信息,也越能作为区分ad与正常个体的判断性特征。因此,基于脑区-基因超图进行是否为ad的判断,相比于基于脑区-基因网络的ad的判断,具有更高的准确性。
[0131]
在一个较优的实施方式中,如图6所示,生成器201包括转换部分、卷积部分、融合部分和反卷积部分;具体地,生成器201中的转换部分包括:转换层模块601;生成器201中的卷积部分包括:边卷积层模块602、点卷积层模块603;生成器201中的融合部分包括:第一融合层模块604;生成器201中的反卷积部分包括:反卷积模块605。
[0132]
其中,转换层模块601用于将所述脑区-基因超图转换为脑区-基因边图和脑区-基因点图;
[0133]
边卷积层模块602用于对所述脑区-基因边图进行多次边级特征信息聚集,得到关键脑区-基因边图;
[0134]
点卷积层模块603用于对所脑区-基因点图进行多次点级特征信息聚集,得到关键脑区-基因点图;
[0135]
第一融合层模块604用于将所述关键脑区-基因边图和关键脑区-基因点图进行融合得到低阶脑区-基因子超图;
[0136]
反卷积模块605用于对低阶脑区-基因子超图通过反卷积操作进行结构信息扩散,得到重构的脑区-基因超图。
[0137]
生成器201中的边卷积和点卷积实现从脑区-基因边图和脑区-基因点图逐步演化并融合为低阶脑区-基因子超图的过程。如前文所述,卷积部分包括边卷积和点卷积两部分。每经过一次边卷积,脑区-基因边图的权重矩阵都会更新,进而使脑区-基因边图的拓扑结构发生变化;每经过一次点卷积,脑区-基因点图的权重矩阵都会更新,进而使脑区-基因点图的拓扑结构发生变化。下面分别描述边卷积和点卷积的具体过程。
[0138]
图7展示了转换层模块601中将脑区-基因超图转换得到脑区-基因边图,并确定脑区-基因边图的边权重矩阵的过程。具体地,脑区-基因超图中的每条超边ei(i=1,...,n)都对应脑区-基因边图中的一个节点ei;对于脑区-基因超图中任意的两条超边,若此两条超边至少存在一个公共节点,则将此两条超边在脑区-基因边图中所对应的两个节点之间连接一条边,并根据此两条超边的公共节点个数,确定这两个节点之间连接的边的权重值。得到的脑区-基因边图中的节点集合表示为ve={e1,...,en},边集合表示为
脑区-基因边图中每条边对应的权重的计算公式如公式12所示:
[0139][0140]
其中,e
p
∩eq表示超边e
p
连接的节点的集合与eq连接的节点的集合的交集;e
p
∪eq表示超边e
p
连接的节点的集合与eq连接的节点的集合的并集;
[0141]
根据脑区-基因边图的每条边的权重值构建脑区-基因边图的边权重矩阵we∈rn×n,则we可以表示为公式13:
[0142][0143]
图8展示了转换层模块601中将脑区-基因超图通过点图转换得到脑区-基因点图,并确定脑区-基因点图的点权重矩阵的过程。具体地,转换层模块601中将脑区-基因超图中的每个节点vi(i=1,...,n)在脑区-基因点图中创建对应节点vi;对于脑区-基因超图中任意的两个节点,若此两个节点至少存在一个公共超边,则将此两个节点在脑区-基因点图中所对应的节点之间连接一条边,并根据脑区-基因超图中此两个节点的公共超边个数,确定脑区-基因点图中所对应的节点之间连接的边的权重值。得到的脑区-基因点图的节点集合vv={v1,...,vn},边集合脑区-基因点图中每条边对应的权重的计算方式如公式14所示:
[0144][0145]
根据脑区-基因点图的每条边的权重值构建脑区-基因点图的点权重矩阵wv∈rn×n,则wv可以表示为公式15所示:
[0146][0147]
边卷积层模块602中具体可以包括t1阶边卷积层,用于对所述脑区-基因边图的边权重矩阵进行t1次边卷积,从而实现对所述脑区-基因边图的t1次边级特征信息聚集,得到关键脑区-基因边图的边权重矩阵;其中,t1为大于1的自然数。
[0148]
其中,第t阶边卷积层用于对输入的边权重矩阵进行一次边卷积,得到并
输出更新后的边权重矩阵如公式16所示:
[0149][0150]
其中,i∈rn×n为n阶单位矩阵;表示对归一化后得到的权重矩阵;a
(t)
表示第t阶边卷积层的卷积核,为可学习的参数矩阵;relu(
·
)是激活函数;表示边权重矩阵经过t-1阶边卷积层的t-1次边卷积后得到的边权重矩阵;表示由所述转换层模块601输出到边卷积层模块602中的所述脑区-基因边图的边权重矩阵。其中,t1为大于1的自然数,t为小于等于t1的自然数。
[0151]
脑区-基因边图的边权重矩阵,经过t阶边卷积后,在脑区-基因边图中作为节点的每条超边将聚集其t阶邻域内的边级结构信息,得到更新后的边权重矩阵
[0152]
下面以一个具有6个节点的脑区-基因边图为例介绍第t阶边卷积层的边卷积的具体操作。如图9所示,以图9中(a)所示的脑区-基因边图的e1为中心超边,e2、e3、e4和e6为该中心超边的邻接超边,图9中(b)是第t阶边卷积层的卷积核a
(t)
,图9中(c)是对超边e1的t阶边卷积操作。权重矩阵的第一列表示超边e1与其邻接超边的联系程度,权重越大表示联系越紧密。将的第一列中元素以卷积核a
(t)
的第一行中的元素作为权重进行加权求和,表示超边e1从相邻超边聚集到的边级结构信息总量。以此类推,计算出脑区-基因超图中每条超边聚集到的结构信息。最终通过de进行归一化,得到最终的边权重矩阵
[0153]
t阶边卷积的意义:在脑区-基因边图中,节点为脑区-基因超图中的超边,边图中的超边互相连接,形成能区分ad与正常个体的边级结构信息。具体地,超图中的超边越异常,则该超边在边图中就越能代表疾病的特征性结构信息。在t阶边卷积的过程中,脑区-基因边图中每条超边能够从相邻的超边中聚集到边级结构信息,而由于此时每条超边已通过t-1次边卷积聚集到了t-1阶邻域内的边级结构信息,因此在t阶边卷积过程中,边图中的每条超边能够聚集到t阶邻域内的边级结构信息。本发明设计的边卷积操作能通过脑区-基因边图捕捉超边与其它超边之间的联系,从而高效实现边级结构信息的聚集。图10展示了脑区-基因边图的t阶边卷积操作,以图中超边e1为例,虚框内为超边e1的一阶邻域。由于图中的每条超边都聚集了t-1阶邻域内的边级结构信息,因此在经过此次边卷积后,超边e1将聚集到t阶邻域内的边级结构信息。
[0154]
点卷积层模块603中具体可以包括t1阶点卷积层,用于对所述脑区-基因点图的点权重矩阵进行t1次点卷积,从而实现对所述脑区-基因点图的t1次点级特征信息聚集,得到关键脑区-基因点图的点权重矩阵;其中,t1为大于1的自然数。
[0155]
其中,第t阶点卷积层用于对输入的点权重矩阵进行一次点卷积,得到并输出更新后的点权重矩阵如公式17所示:
[0156][0157]
其中,i∈rn×n为n阶单位矩阵;表示对归一化后得到的矩阵;b
(n)
表示第t阶点卷积层的卷积核,为可学习的参数矩阵;relu(
·
)是激活函数;表示点权重矩阵经过t-1阶点卷积层的t-1次点卷积后得到的点权重矩阵;表示由所述转换层模块601输出到点卷积层模块603中的所述脑区-基因点图的点权重矩阵。
[0158]
所述脑区-基因点图的点权重矩阵,经过第t次点卷积后,每个节点将聚集其t阶邻域内的点级结构信息,得到更新后的点权重矩阵
[0159]
下面以一个具有6个节点的脑区-基因点图为例解释t阶点卷积操作。如图11所示,图11中(a)所示的脑区-基因点图的节点v3为将要进行点卷积操作的节点,v1、v2、v4、v5为该中心节点的一阶邻接节点,图11中(b)是第t阶点卷积层的卷积核b
(t)
。图11中(c)是对节点v3的t阶点卷积操作。脑区-基因点图的点权重矩阵的第三行表示节点v3与邻接节点的联系程度,值越大表示联系越紧密,将卷积核b
(t)
的第三列作为权重一一对应赋予给的第三行,再进行加权求和得到节点v3相应的值,完成对节点v3的t阶点卷积操作。以此类推,对每个节点进行t阶点卷积操作,最终进行归一化操作得到更新的权重矩阵
[0160]
t阶点卷积的意义:脑区-基因点图中的节点,与脑区-基因超图中的节点一致,节点之间的低阶联系形成了能够区分ad与正常个体的点级结构信息。具体地,一个节点越异常,则该节点在脑区-基因点图中就越能代表疾病的特征性结构信息。在t阶点卷积的过程中,脑区-基因点图中每个节点从相邻的节点中聚集到点级结构信息,而由于此时每个节点已通过之前的t-1次点卷积聚集到了t-1阶邻域内的点级结构信息,因此在t阶点卷积过程中,脑区-基因点图中的每个节点能够聚集到其t阶邻域内的点级结构信息。本发明设计的点卷积操作能通过脑区-基因点图捕捉节点与其它节点之间的低阶关联,从而高效实现低阶点级结构信息的聚集。图12展示了脑区-基因点图的t阶点卷积操作,以图中节点v3为例,虚框区域为节点v3的一阶邻域。由于图中的每各节点都聚集了t-1阶邻域内的边级结构信息,因此在经过此次点卷积后,节点v3将聚集到t阶邻域内的点级结构信息。
[0161]
第一融合层模块604具体用于将所述关键脑区-基因边图的边权重矩阵和关键脑
区-基因点图的点权重矩阵进行融合得到低阶脑区-基因子超图的关联权重矩阵,如公式18所示:
[0162][0163]
其中,表示脑区-基因点图的初始的点权重矩阵;表示脑区-基因边图的初始的边权重矩阵;h表示脑区-基因超图的关联矩阵;表示点卷积层模块603经过t1次点卷积得到的关键脑区-基因点图的点权重矩阵;表示边卷积层模块602经过t1次边卷积得到的关键脑区-基因边图的边权重矩阵;h
l
表示计算得到的低阶脑区-基因子超图的关联权重矩阵。
[0164]
本发明设计扩散反卷积实现基于低阶脑区-基因子超图得到生成脑区-基因超图的过程,如图6所示。反卷积模块605中具体可以包括t1阶反卷积层,用于对低阶脑区-基因子超图的关联权重矩阵,进行t1次反卷积操作,实现对低阶脑区-基因子超图的多次结构信息扩散,得到重构的t1阶生成的脑区-基因超图的关联权重矩阵。
[0165]
每经过一次扩散反卷积操作,低阶脑区-基因子超图的关联权重矩阵都会得到更新,进而使其拓扑结构发生变化。下面以t-1阶生成脑区-基因超图经过第t次扩散反卷积得到t阶生成脑区-基因超图为例,描述扩散反卷积的具体过程。
[0166]
经过t1次扩散反卷积后,低阶脑区-基因子超图的关联权重矩阵得到了更新。我们将低阶脑区-基因子超图定义为一种结构信息的压缩表示,并认为即使节点和超边在低阶脑区-基因子超图中不存在关联关系,其间也存在潜在的联系。我们定义“潜在边”描述潜在的节点-超边的联系。作为对比,对于在超图中存在连接的节点和超边,定义“真实边”描述这种节点-超边的联系。在第t次扩散反卷积的过程中,低阶脑区-基因子超图中每条真实边将自身的结构信息扩散到其t阶邻域内的真实边(真实的节点-超边的关联关系)和潜在边(潜在的节点-超边的关联关系)上,进而更新这些边(节点-超边的关联关系)的权重。当潜在边更新后的信息量高于一定阈值时,就会转换为真实边;同样,当真实边更新后的信息量低于一定阈值时,就会转换为潜在边。因此,扩散反卷积能够引起网络拓扑结构的更新。在结构信息扩散的过程中,待更新边的结构信息变化量等于该边一阶邻域内所有真实边扩散到该边的总和,具体可以通过反卷积层中的矩阵运算得到。
[0167]
反卷积模块605中第t阶反卷积层,用于对输入的关联权重矩阵进行一次反卷积操作,输出更新的关联权重矩阵在此过程中,对t-1阶生成的脑区-基因超图的关联权重矩阵进行t阶扩散反卷积使每条真实边和潜在边聚集到其t阶邻域内的结构信息,得到更新的关联权重矩阵
[0168]
其中,是关联权重矩阵经过t-1阶反卷积层的t-1次反卷积后得到的关联权重矩阵;表示由第一融合层模块604融合得到并输出至反卷积模块605的低阶脑
区-基因子超图的关联权重矩阵。
[0169]
其中,的计算公式如公式19所示:
[0170][0171]
其中,b
(t)
是第t阶反卷积层的偏置,为第t阶反卷积层中可学习的参数;c
(t)
∈rn×n是第t阶反卷积层的扩散反卷积核,为第t阶反卷积层中可学习的参数矩阵;表示关联权重矩阵中第r行第i列的值;表示c
(t)
中第r行第i列的值;表示关联权重矩阵中第i行第c列的值;表示c
(t)
中第i行第c列的值;
[0172]
其中,表示位于第r行第c列的元素接收到的信息总量,计算方法是将和c
(t)
中的第r行和第c列对应相乘然后求和;f(
·
)是激活函数,本文采用的是relu函数,即当某条边更新后的信息值小于或等于0时,将该边的权重置为0;是经过t阶扩散反卷积更新后的值。换言之,每个元素更新后的值由该元素所在行与所在列的所有元素相加得到。
[0173]
如图13所示,图13中(a)为脑区-基因超图以及它的关联权重矩阵图中是将要进行t阶扩散反卷积操作的边,图13中(b)是第t阶反卷积层的扩散反卷积核c
(t)
。图13中(c)展示了边的t阶扩散反边卷积操作,的元素表示节点v1和超边e3的关联程度。一方面,若将边视为信息发送者,t阶反卷积核c
(t)
的第一行第三列的值作为权重赋予给中对应位置的上,再将其加到的第一行和第三列中的每一个元素上,即扩散到其一阶邻域内的所有边上,完成对边的t阶边扩散反卷积操作。另一方面,将边视为信息接收者,将接收到的第一行和第三列中的每个元素传播的信息,由此完成自身的信息更新。
[0174]
t阶扩散反卷积的意义:在t阶扩散反卷积的过程中,t-1阶生成脑区-基因超图中的每条潜在边与真实边吸收其一阶邻域内的结构信息,由于此时网络图中的每条潜在边与真实边已经接收到了t-1阶邻域内的信息,因此t阶扩散反卷积能使每条边聚集到其t阶邻域内的结构信息。在低阶脑区-基因子超图中,超边与脑区或基因等节点互相连接形成疾病独有的结构信息,本文设计的扩散反卷积能够很好地捕捉低阶脑区-基因子超图中边与其邻域内其他边之间的关系。图14表示的是t-1阶生成脑区-基因超图的t阶扩散反卷积操作,此时网络中的每条边都吸收了t-1阶邻域内的结构信息。以图14中边为例,节点v2~
1阶边点卷积层的t-1次边点卷积后得到的关联权重矩阵;h
(0)
为输入判别器202的脑区-基因超图的关联权重矩阵;和分别表示第t阶边点卷积层中的边卷积核与点卷积核,二者均为可学习的参数矩阵;relu(
·
)是激活函数;h
(t)
为t阶边点卷积的输出结果。
[0184]
下面以一个具有6个节点的脑区-基因超图为例解释t阶边点卷积操作。如图16所示,图16中(a)为脑区-基因超图及其关联权重矩阵h
(t-1)
,图中边是将要进行t阶边点卷积操作的边,图16中(b)是t阶边卷积核和t阶点卷积核图(c)是对边的t阶边点卷积操作。脑区-基因超图的关联权重矩阵h
(t-1)
的表示节点v1和超边e1的关联权重,值越大表示联系越紧密。首先,将t阶边卷积核的第一行作为权重赋给h
(t-1)
的第一列得到聚集边级结构信息的中间矩阵u,其次,将t阶点卷积核的第一列作为权重赋给中间矩阵u的第一行,完成对边的t阶边点卷积操作。以此类推,对每个节点进行上述卷积操作,得到更新的关联权重矩阵h
(t)
。
[0185]
t阶边点卷积的意义:在t阶边点卷积过程中,t-1阶脑区-基因超图中每条超边聚集到其一阶邻域内的边级-点级结构信息,而由于此时所有的超边已经在它们各自的t-1阶邻域内聚集到了边级-点级结构信息,所以t阶边点卷积过程可以使每条超边聚集其t阶邻域内的边级-点级结构信息。在疾病的脑区-基因超图中,超边将异常的节点(脑区或基因)连接起来形成疾病独有的高阶边级-点级结构信息,脑区或基因节点越异常,连接它们的边就越能够代表该疾病特征的边级-点级结构信息。本文设计的边点卷积能够很好地捕捉脑区-基因超图中节点与超边之间的高阶关联。图17展示的是t阶边点卷积操作,此时图中的每条边都聚集了t-1阶边级-点级结构信息。以边为例,经过t阶边点卷积,边能够分别通过超边e1和节点v1聚集到t阶边级结构信息与点级结构信息,图17中(a)展示的是超边e1的一阶邻域内的边级结构信息聚集过程,图17中(b)展示的是节点v1的一阶邻域内的点级结构信息聚集过程。
[0186]
判别器的边点卷积层模块1501中,边点卷积层共有t2层,即总共进行t2次边级-点级结构信息聚集。t2阶点边卷积的输出结果即为高阶脑区-基因子超图的关联矩阵,如公式21所示:
[0187][0188]
判别器的第二融合层模块1502用于对的边点卷积层模块1501输出的高阶脑区-基因子超图和生成器生成的低阶脑区-基因子超图进行融合,输出为融合了高阶和低阶结构信息的关键脑区-基因超图。
[0189]
具体地,第二融合层模块1502对边点卷积层模块1501输出的高阶脑区-基因子超图的关联权重矩阵hh和生成器输出的低阶脑区-基因子超图的关联权重矩阵h
l
进行融合,输出为融合了高阶和低阶结构信息的关键脑区-基因超图的关联权重矩阵hk,如公式22所示:
[0190]hk
=θhh (1-θ)h
l
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(公式22)
[0191]
其中,θ为设定的融合因子。
[0192]
如图18所示,全连接层模块1503中包括平坦层、全连接层和输出层;全连接层模块1503的输入为关键脑区-基因超图,并输出针对输入判别器202的脑区-基因超图的真假判断结果与属于ad或nc的概率的判断结果。首先,全连接层模块1503中的平坦层将经过融合层得到的关键脑区-基因超图的关联权重矩阵hk平铺为一维特征向量x
(0)
;全连接层模块1503中的全连接层包含多层,都使用的是激活函数rplu(
·
),用于对平铺后的一维特征向量x
(0)
提取特征;最后,全连接层模块1503中的输出层根据提取的特征通过激活函数sigmoid(
·
)和softmax(
·
)分别输出真假和nc/ad的判断结果,上述过程如公式23-25所示:
[0193]
x
(t 1)
=relu(w
(t)
x
(t)
b
(t)
),t≤l-1
ꢀꢀꢀꢀꢀꢀ
(公式23)
[0194]
p=softmax(w
(t)
x
(t)
b
(t)
),t=l
ꢀꢀꢀꢀꢀꢀꢀꢀ
(公式24)
[0195]
q=sigma(w
(t)
x
(t)
b
(t)
),t=l
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(公式25)
[0196]
其中,全连接层的输入为x
(0)
=ran(hk),ran(
·
)为矩阵的按行平铺函数,ran(hk)表示将关键脑区-基因超图二维关联权重矩阵hk平铺得到一维向量。q是全连接层模块1503中的输出层输出的有关超图的真假的判断结果,使用的激活函数为sigmoid(
·
),p是全连接层模块1503中的输出层输出的ad或nc类别的概率的判断结果,使用的激活函数为softmax(
·
);w
(t)
表示第t层全连接层的权重矩阵;x
(t)
和x
(t 1)
分别表示第t层和第t 1层全连接层的一维向量;b
(t)
表示第t层全连接层的偏置;l表示全连接层的总层数。
[0197]
实施例二
[0198]
在对基于生成对抗网络的超图结构信息聚集模型进行训练时,判别器的输入有两种:真实的脑区-基因超图和生成器生成的(重构的)脑区-基因超图;判别器的作用有两个,第一是判断输入的脑区-基因超图的真假性,即判断输入的脑区-基因超图是真实的,还是生成器生成的;第二是判断输入的脑区-基因超图是正常人(nc)的还是ad的。判别器和生成器互相训练,不断调优,直至生成对抗网络收敛。此时,关键结构子网络能够聚集被试脑区-基因网络的特征信息,从而根据关键结构子网络进行ad的判断,可以辅助于对阿尔茨海默症做精准的诊断。
[0199]
本发明实施例二提供的一种训练上述基于生成对抗网络的超图结构信息聚集模型的具体方法,流程如图19所示,包括如下步骤:
[0200]
步骤s1901:生成训练样本。
[0201]
具体地,以正常人的脑影像(fmri)数据和基因数据分别构建脑网络和基因网络,进而将构建的脑网络和基因网络根据现有方法进行融合,得到正常人的脑区-基因网络;具体地,以脑区和基因为节点,采用现有的皮尔逊相关分析方法计算节点之间边的权重,若边权重大于阈值则保留该边,反之去掉该边,以此构建脑区-基因网络,实现脑网络和基因网络的融合;进而基于构建的脑区-基因网络生成正常人的脑区-基因超图。
[0202]
以ad患者的脑影像(fmri)数据和基因数据分别构建脑网络和基因网络,进而将构建的脑网络和基因网络进行融合,得到ad患者的脑区-基因网络;基于得到的脑区-基因网络,生成ad患者的脑区-基因超图。
[0203]
本步骤中,以多个正常人的脑影像(fmri)数据和基因数据,分别构建多个正常人的脑区-基因超图作为样本;以及以多个ad患者的脑影像(fmri)数据和基因数据,分别构建多个ad患者的脑区-基因超图作为样本。
[0204]
步骤s1902:根据所述样本对所述生成器和判别器进行多次迭代的优化和训练;
[0205]
具体地,在模型训练过程中,判别器和生成器始终维持着对立与抗争关系,前者期盼重构的脑区-基因网络非常接近真实的脑区-基因网络,以此瞒过后者,而后者期盼能够精准判断生成的脑区-基因网络和真实的脑区-基因网络;由此,本步骤中对所述生成器和判别器进行多次迭代的优化和训练,使得二者不断进行对抗博弈,生成能力和判断能力分别得到增强,直到模型收敛,最终使得生成器能够更好地完成生成任务。
[0206]
其中,对所述生成器和判别器进行一次迭代的优化和训练的过程,具体流程如图20所示包括如下子步骤:
[0207]
子步骤s2001:固定所述生成器的参数后,根据所述样本对所述判别器进行优化;
[0208]
子步骤s2002:固定所述判别器的参数后,根据所述样本对所述生成器进行训练。
[0209]
步骤s1903:在对所述生成器和判别器进行多次迭代的优化和训练后,将由所述生成器和判别器组成的生成对抗网络作为训练得到的结构信息聚集模型。
[0210]
具体地,上述子步骤s2001中提到的固定所述生成器的参数后,根据所述样本对所述判别器进行优化的具体方法流程,如图21所示,包括如下步骤:
[0211]
步骤s2101:固定所述生成器的参数后,将样本中的正常人的脑区-基因超图或ad患者的脑区-基因超图输入到所述生成器中,由所述生成器根据输入的脑区-基因超图重构变化的脑区-基因超图;
[0212]
步骤s2102:将所述生成器重构的脑区-基因超图或者样本中的正常人或ad患者的脑区-基因超图输入判别器;
[0213]
步骤s2103:根据所述判别器输出的判断结果,对所述判别器的参数进行优化。
[0214]
在根据所述判别器输出的判断结果与真实标签之间的差异,对所述判别器的参数进行优化时,是通过损失函数的计算结果进行判别器的参数优化,使得判别器区分真实脑区-基因超图和生成脑区-基因超图能力达到最佳。其中,所述真实标签反映的是输入到判别器的脑区-基因超图的真实情况。
[0215]
例如,pi是判别器根据输入其的第i个脑区-基因网络输出的判断结果;pi为一个二维列向量表示判断结果为正常人的概率,表示判断结果为ad患者的概率。
[0216]
ti是对应于pi的真实标签;ti为一个二维行向量,当值取(1,0)时表示输入到判别器的脑区-基因超图的真实标签为正常人,当值取(0,1)时表示输入到判别器的脑区-基因超图的真实标签为ad患者,当值取(0,0)时表示输入到判别器的脑区-基因超图的真实标签为重构的脑区-基因超图。
[0217]
根据pi与ti之间的差异,对所述判别器的参数进行优化。
[0218]
损失函数是评估模型的性能与预期的结果接近程度,损失函数值的大小与模型预测性能成反比。
[0219]
本发明定义的损失函数loss,如公式26所示:
[0220]
[0221]
其中,生成器和判别器分别用g和d表示;e(
·
)表示求取变量的期望值;x表示正常人或ad患者的脑区-基因超图(即真实的脑区-基因超图),p
data
(x)表示x的分布,d(x)表示判别器判断x为真实脑区-基因超图的概率;g(z)表示生成器生成的(重构的)脑区-基因超图,pz(z)表示数据z的分布,d(g(z))表示判断g(z)为真实脑区-基因超图的概率。
[0222]
本步骤中,对所述判别器的参数进行优化时,使公式27达到最佳:
[0223][0224]
判别器的优化目标是区分真实脑区-基因超图和生成脑区-基因超图,公式中d(x)表示x为真实脑区-基因超图的概率,1-d(g(z))表示g(z)为生成脑区-基因超图的概率。log(
·
)为单调增函数,当d(x)和1-d(g(z))趋近1时,公式27达到最大值,判别器的区分能力达到最佳。
[0225]
具体地,上述子步骤s2002中提到的固定所述判别器的参数后,根据所述样本对所述生成器进行训练的具体方法流程,如图22所示,包括如下步骤:
[0226]
步骤s2201:在固定所述判别器的参数后,将样本中的正常人或ad患者的脑区-基因网络输入到所述生成器中,由所述生成器根据输入的脑区-基因网络重构变化的脑区-基因网络;
[0227]
步骤s2202:将所述生成器重构的脑区-基因网络输入到所述判别器;
[0228]
步骤s2203:根据所述判别器输出的判断结果对所述生成器的参数进行调整。
[0229]
本步骤中,根据判别器的判断结果与真实标签之间的差异,对所述生成器的参数进行调整,使生成器重构尽可能真实的脑区-基因网络来欺骗判别器。
[0230]
在本步骤中固定判别器优化生成器的参数时,使公式28达到最佳:
[0231][0232]
生成器g的优化目标是使生成器能够拟合真实脑区-基因超图的分布。公式中的g(z)表示生成脑区-基因超图,d(g(z))表示判别器判断g(z)为真实脑区-基因超图的概率,log(
·
)为单调增函数,当d(g(z))接近于1时,1-d(g(z))接近于0,公式28达到最小值,此时真实脑区-基因超图分布足够接近生成脑区-基因超图分布。
[0233]
通过反复迭代上述图21、图22所示的流程过程,判别器的判断能力和生成器的重构能力不断提高,直至生成对抗网络收敛。
[0234]
实施例三
[0235]
基于预先训练得到的超图结构信息聚集模型,本发明实施例三提供的一种阿尔茨海默症的判断方法,具体流程如图23所示,包括如下步骤:
[0236]
步骤s2301:根据被试者的脑影像数据和基因数据,构建该被试者的脑区-基因超图。
[0237]
本步骤中,根据被试者的脑影像数据和基因数据,分别构建脑网络和基因网络,进而将构建的脑网络和基因网络根据现有方法进行融合,得到被试者的脑区-基因网络;具体地,以脑区和基因为节点,采用现有的皮尔逊相关分析方法计算节点之间边的权重,若边权重大于阈值则保留该边,反之去掉该边,以此构建脑区-基因网络,实现脑网络和基因网络的融合;进而,基于所述脑区-基因网络生成被试者的脑区-基因超图。
[0238]
步骤s2302:将该脑区-基因超图输入到通过上述方法预先训练得到的超图结构信
息聚集模型中。
[0239]
步骤s2303:通过超图结构信息聚集模型进行该被试者是否为阿尔茨海默症ad患者的判断。
[0240]
具体地,通过超图结构信息聚集模型中上述的生成器,将被试者的脑区-基因超图转换为脑区-基因边图和脑区-基因点图,并分别对所述脑区-基因边图和脑区-基因点图进行多次边级特征信息聚集与点级特征信息聚集,将信息聚集后的边图和点图进行融合得到低阶脑区-基因子超图;以及
[0241]
通过超图结构信息聚集模型中上述的判别器对所述脑区-基因超图进行多次高阶边级-点级结构信息聚集,得到高阶脑区-基因子超图;将低阶脑区-基因子超图和高阶脑区-基因子超图融合得到关键脑区-基因超图;根据所述关键脑区-基因超图输出该被试者为阿尔茨海默症ad患者的概率的判断结果。
[0242]
例如,超图结构信息聚集模型输出的判断结果为pi,为一个二维列向量,为一个二维列向量表示判断结果为正常人的概率,表示判断结果为ad患者的概率。
[0243]
实施例四
[0244]
基于预先训练得到的超图结构信息聚集模型,本发明实施例四提供的一种提取阿尔茨海默症的显著特征的方法,具体流程如图24所示,包括如下步骤:
[0245]
步骤s2401:对于包括多个正常人和ad患者的被试者,根据每个被试者的脑影像数据和基因数据,构建该被试者的脑区-基因网络,并基于所述脑区-基因网络形成该被试者的脑区-基因超图。
[0246]
步骤s2402:对于每个被试者,将该被试者的脑区-基因超图通过超图结构信息聚集模型输出该被试者为ad患者的概率的判断结果,并根据该被试者的真实情况计算该判断结果的可信度分数。
[0247]
本步骤中,对于每个被试者,将该被试者的脑区-基因超图通过超图结构信息聚集模型输出该被试者为ad患者的概率的判断结果:超图结构信息聚集模型对脑区-基因超图进行低阶结构信息和高阶结构信息的聚集得到的关键脑区-基因超图,根据关键脑区-基因超图输出该被试者为ad患者的概率的判断结果。
[0248]
进一步,根据该被试者的真实情况计算该判断结果的可信度分数,计算方式如公式29所示:
[0249]ri
=ti×
piꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(公式29)
[0250]
其中,pi是超图结构信息聚集模型输出的第i个被试者为ad患者的概率的判断结果,ti是第i个被试者的真实标签,反映了第i个被试者的真实情况;ti∈r1*2为一个二维行向量,当ti=(1,0)时表示正常人,当fi=(0,1)时表示ad患者;pi为一个二维列向量表示判断结果中为正常人的概率,表示判断结果中为ad患者的概率。经过运算得到的ri为pi的可信度分数,为一个具体的数值,它可以反映预测情况和真实情况的接近程度。
[0251]
若判别器输出的判断结果pi为(0.9,0.1)
t
,则此判断结果表明该被试者为正常个体的概率更大;若该被试者的实际情况为正常人,则ti为(1,0),此时ri=ti×
pi=0.9,表示判断结果和真实情况的接近程度较大,即该结果的可信度较高;若被试者为ad患者,则ti为
(0,1),此时ri=ti×
pi=0.1,表示判断结果为ad患者的概率是0.1,该判断结果和真实情况的接近程度较小,因此,该判断结果并不可信,应尽量减小其对于显著特征提取的影响。
[0252]
步骤s2403:根据每个被试者的脑区-基因超图的关联权重矩阵和判断结果的可信度分数,计算关键特征矩阵。
[0253]
具体地,根据如下公式30,计算关键特征矩阵:
[0254][0255]
其中,表示第i个被试者的关键脑区-基因超图的关联权重矩阵;ri表示超图结构信息聚集模型对第i个被试者的判断结果的可信度分数;表示将第i个被试者的判断结果的可信度分数作为第i个被试者所对应的关联权重矩阵的权重;m表示被试者的总数。
[0256]
步骤s2404:基于所述关键特征矩阵,计算脑区-基因网络中各节点的重要度分数和各边的重要度分数。
[0257]
本步骤中,基于关键特征矩阵hf,计算并排序每个点级特征和边级特征的重要度分数:
[0258]
对于边级特征,关键特征矩阵hf中的每一行对应一个节点,因此,将对应p、q节点的两行的元素对应相乘并累加,即可得到边的重要度分数重要度分数越大表明这两个节点之间的连接对区分ad患者与正常个体的贡献越大。边级判别性特征重要度分数的计算公式如公式31所示:
[0259][0260]
其中,和分别表示关键特征矩阵hf中第p行k列和第q行k列的值;表示边的重要度分数。由于hf为对称矩阵,且无需考虑节点与自身的关联,因此边级特征共有个,即共计算有个边的重要度分数。
[0261]
对于点级特征,将关键特征矩阵hf中第p行的每个元素累加得到点v
p
的重要度分数重要度分数越大表明该节点对区分ad患者与正常个体的贡献越大。点级判别性特征重要度分数的计算公式如公式32所示:
[0262][0263]
其中表示关键特征矩阵hf第p行k列的值;表示节点v
p
的重要度分数。
[0264]
进而,将和分别进行降序排序得到点级特征集合featurev和边级特征集合featuree,集合中越靠前的边和节点表明其对区分ad患者与正常个体的贡献越大,如公式33所示:
[0265][0266]
步骤s2405:根据节点的重要度,确定出作为阿兹海默症的显著特征的节点。
[0267]
具体地,一般而言,重要度分数越大的节点,对区分ad患者与正常个体的贡献越大,因此,可以将点级特征集合featurev中靠前的节点,即重要度分数最大的若干节点,提取为阿兹海默症的显著特征的节点。
[0268]
作为一种更优的实施方式,还可使用增量搜索法确定出阿兹海默症的显著特征的节点:
[0269]
对点级特征集合featurev中各脑区节点进行降序排序,得到一个大小为n1=116的脑区特征集;对点级特征集合featurev中各基因节点进行降序排序,得到一个大小为n2=45的基因特征集;
[0270]
从脑区特征集中选取第1个节点组成第1个脑区特征子集以1为增量,依次选择前1,...,n1个节点组成多个脑区特征子集,最终得到n1个脑区特征子集
[0271]
从基因特征集中选取第1个节点组成第1个基因特征子集,以1为增量,依次选择前1,...,n2个节点组成多个基因特征子集,最终得到n2个基因特征子集
[0272]
将n1个脑区特征子集和n2个基因特征子集两两任意组合得到n1×
n2个组合,用超图结构信息聚集模型测试这n1×
n2个组合的分类准确率acc,最高准确率对应的组合即为最优脑区特征子集和基因特征子集,寻找最优脑区特征子集和基因特征子集的函数表示如公式34所示:
[0273][0274]
该公式表示寻找最优脑区特征子集和基因特征子集使得分类准确率acc最大;s.t.表示受限条件,i表示脑区特征子集的编号,j表示基因特征子集的编号。最优脑区特征子集中的节点对应的脑区作为提取出的阿兹海默症的显著特征中的病变脑区;最优基因特征子集中的节点对应的基因作为提取出的阿兹海默症的显著特征中的致病基因。
[0275]
步骤s2406:根据边的重要度,确定出作为阿兹海默症的显著特征的病变脑区和风险基因之间的关联关系。
[0276]
具体地,一般而言,重要度分数越大的边,对区分ad患者与正常个体的贡献越大,因此,可以将边级特征集合featuree中靠前的边,即重要度分数最大的若干边,确定为阿兹海默症的显著特征之间的关联关系,即病变脑区和风险基因之间的关联关系。
[0277]
作为一种更优的实施方式,还可使用增量搜索法确定出阿兹海默症的显著特征之间的关联关系:
[0278]
选取边级特征集台feature的前5个边组成第1个边级特征子集以1为增
量,依次选择前个边组成多个边级特征子集,最终得到个边级特征子集用超图结构信息聚集模型测试这个子集的分类准确率acc,最高准确率对应的子集即为最优边级特征子集,寻找最优边级特征子集的函数表示如公式35所示:
[0279][0280]
该公式表示寻找最优边级特征子集sub
edge
,使得分类准确率acc最大;i表示边级特征子集的编号。最优边级特征子集中的边即为确定出的阿兹海默症的显著特征之间的关联关系,即病变脑区和风险基因之间的关联关系。
[0281]
以3个具有4个节点的关键脑区-基因超图的关联权重矩阵为例解释显著特征提取的过程。假设被试者的真实标签t1=(1,0);t2=t3=(0,1),假设模型输出的判断结果p1=(0.9,0.1)
t
,p2=(0.2,0.8)
t
,p3=(0.4,0.6)
t
,三个被试者的关键脑区-基因超图的关联权重矩阵分别为首先,计算第i个被试者的判断结果的可信度分数probi=pi×
ti,进而计算3个关键脑区-基因超图的关联权重矩阵中每条边的平均重要度得到关键特征矩阵hf,如图25中(a)所示。其次,根据hf计算重要度分数和并进行降序排序,得到点级特征集合sub
vertex
和边级特征集合sub
edge
,如图25中(b)和(c)所示。最后使用增量搜索法寻找最优点级特征子集和最优边特征子集。寻找最优点级特征子集如下:首先,对所有节点的重要度按照脑区和基因分别进行降序排序,脑区排序结果为:v2、v1、v3,基因排序结果为:v4。选取第1个脑区组成第1个脑区特征子集,前2个脑区组成第2个脑区特征子集,前3个脑区组成第3个脑区特征子集;再选取第1个基因组成第1个基因特征子集。将3个脑区特征子集和1个基因特征子集两两组合得到3个组合。用提出的超图结构信息聚集模型测试这3个组合的分类准确率,假设第2个组合的准确率最高,则第2个脑区特征子集中的节点v2和v1对应的脑区即为模型捕获的病变脑区,第1个基因特征子集中的节点v4对应的基因即为模型捕获的致病基因。寻找最优边特征子集的过程如下:选取第1个边特征组成第1个边特征子集,再选取前2个边特征组成第2个边特征子集,直到包括全部的边特征。用提出的超图生成对抗网络测试这所有边特征子集的分类准确率,假设第2个边特征子集的准确率最高,那么该子集中的边和即为模型捕获的关键边特征,反映了病变脑区和致病基因的关联模式。
[0282]
实施例五
[0283]
图26示出了本实施例所提供的一种更为具体的电子设备硬件结构示意图,该设备可以包括:处理器1010、存储器1020、输入/输出接口1030、通信接口1040和总线1050。其中处理器1010、存储器1020、输入/输出接口1030和通信接口1040通过总线1050实现彼此之间在设备内部的通信连接。
[0284]
处理器1010可以采用通用的cpu(central processing unit,中央处理单元)、微
处理器、应用专用集成电路(application specific integrated circuit,asic)、或者一个或多个集成电路等方式实现,用于执行相关程序,以实现本说明书实施例所提供的提取阿尔茨海默症的显著特征的方法。
[0285]
此外,所述处理器执行所述计算机程序时还可实现本说明书实施例所提供的超图结构信息聚集模型的训练方法。
[0286]
存储器1020可以采用rom(read only memory,只读存储器)、ram(random access memory,随机存取存储器)、静态存储设备,动态存储设备等形式实现。存储器1020可以存储操作系统和其他应用程序,在通过软件或者固件来实现本说明书实施例所提供的技术方案时,相关的程序代码保存在存储器1020中,并由处理器1010来调用执行。
[0287]
输入/输出接口1030用于连接输入/输出模块,可以与非线性接收机相连,从非线性接收机接收信息,实现信息输入及输出。输入输出/模块可以作为组件配置在设备中(图中未示出),也可以外接于设备以提供相应功能。其中输入设备可以包括键盘、鼠标、触摸屏、麦克风、各类传感器等,输出设备可以包括显示器、扬声器、振动器、指示灯等。
[0288]
通信接口1040用于连接通信模块(图中未示出),以实现本设备与其他设备的通信交互。其中通信模块可以通过有线方式(例如usb、网线等)实现通信,也可以通过无线方式(例如移动网络、wifi、蓝牙等)实现通信。
[0289]
总线1050包括一通路,在设备的各个组件(例如处理器1010、存储器1020、输入/输出接口1030和通信接口1040)之间传输信息。
[0290]
需要说明的是,尽管上述设备仅示出了处理器1010、存储器1020、输入/输出接口1030、通信接口1040以及总线1050,但是在具体实施过程中,该设备还可以包括实现正常运行所必需的其他组件。此外,本领域的技术人员可以理解的是,上述设备中也可以仅包含实现本说明书实施例方案所必需的组件,而不必包含图中所示的全部组件。
[0291]
本技术实施例还提供一种计算机可读存储介质,计算机可读存储介质其上存储有计算机程序,所述计算机程序可被至少一个处理器所执行,以使所述至少一个处理器执行实现实施例中的电子设备的操作。
[0292]
本实施例中,计算机可读存储介质包括闪存、硬盘、多媒体卡、卡型存储器(例如,sd或dx存储器等)、随机访问存储器(ram)、静态随机访问存储器(sram)、只读存储器(rom)、电可擦除可编程只读存储器(eeprom)、可编程只读存储器(prom)、磁性存储器、磁盘、光盘等。在一些实施例中,计算机可读存储介质可以是计算机设备的内部存储单元,例如该计算机设备的硬盘或内存。在另一些实施例中,计算机可读存储介质也可以是计算机设备的外部存储设备,例如该计算机设备上配备的插接式硬盘,智能存储卡(smart media card,简称为smc),安全数字(secure digital,简称为sd)卡,闪存卡(flash card)等。当然,计算机可读存储介质还可以既包括计算机设备的内部存储单元也包括其外部存储设备。本实施例中,计算机可读存储介质通常用于存储安装于计算机设备的操作系统和各类应用软件,例如实施例中语音视频连线方法的程序代码等。此外,计算机可读存储介质还可以用于暂时地存储已经输出或者将要输出的各类数据。
[0293]
本发明的技术方案中,对于包括多个正常人和ad患者的被试者,根据每个被试者的脑影像数据和基因数据,构建该被试者的脑区-基因网络,并基于所述脑区-基因网络形成该被试者的脑区-基因超图;对于每个被试者,将该被试者的脑区-基因超图通过超图结
构信息聚集模型输出该被试者为ad患者的概率的判断结果,并根据该被试者的真实情况计算该判断结果的可信度分数;根据每个被试者的脑区-基因超图的关联权重矩阵和判断结果的可信度分数,计算关键特征矩阵;基于所述关键特征矩阵,计算脑区-基因网络中各节点的重要度分数;根据节点的重要度,确定出作为阿兹海默症的显著特征的节点;其中,所述超图结构信息聚集模型是以多个正常人的脑区-基因超图和ad患者的脑区-基因超图作为样本预先训练得到的。由于对脑区-基因超图进行低阶结构信息和高阶结构信息的聚集得到的关键脑区-基因超图,更能体现ad与正常个体的判断性特征;因此,根据关键脑区-基因超图判断被试者是正常人还是ad的概率更为准确,进而,根据超图结构信息聚集模型输出的更精准的判断,可以更为精准确定ad的显著特征,即ad的风险基因和病变脑区,以便助力对应的靶向药物开发和针对性经颅磁治疗。
[0294]
本实施例的计算机可读介质包括永久性和非永久性、可移动和非可移动媒体可以由任何方法或技术来实现信息存储。信息可以是计算机可读指令、数据结构、程序的模块或其他数据。计算机的存储介质的例子包括,但不限于相变内存(pram)、静态随机存取存储器(sram)、动态随机存取存储器(dram)、其他类型的随机存取存储器(ram)、只读存储器(rom)、电可擦除可编程只读存储器(eeprom)、快闪记忆体或其他内存技术、只读光盘只读存储器(cd-rom)、数字多功能光盘(dvd)或其他光学存储、磁盒式磁带,磁带磁磁盘存储或其他磁性存储设备或任何其他非传输介质,可用于存储可以被计算设备访问的信息。
[0295]
所属领域的普通技术人员应当理解:以上任何实施例的讨论仅为示例性的,并非旨在暗示本公开的范围(包括权利要求)被限于这些例子;在本发明的思路下,以上实施例或者不同实施例中的技术特征之间也可以进行组合,步骤可以以任意顺序实现,并存在如上所述的本发明的不同方面的许多其它变化,为了简明它们没有在细节中提供。
[0296]
另外,为简化说明和讨论,并且为了不会使本发明难以理解,在所提供的附图中可以示出或可以不示出与集成电路(ic)芯片和其它部件的公知的电源/接地连接。此外,可以以框图的形式示出装置,以便避免使本发明难以理解,并且这也考虑了以下事实,即关于这些框图装置的实施方式的细节是高度取决于将要实施本发明的平台的(即,这些细节应当完全处于本领域技术人员的理解范围内)。在阐述了具体细节(例如,电路)以描述本发明的示例性实施例的情况下,对本领域技术人员来说显而易见的是,可以在没有这些具体细节的情况下或者这些具体细节有变化的情况下实施本发明。因此,这些描述应被认为是说明性的而不是限制性的。
[0297]
尽管已经结合了本发明的具体实施例对本发明进行了描述,但是根据前面的描述,这些实施例的很多替换、修改和变型对本领域普通技术人员来说将是显而易见的。例如,其它存储器架构(例如,动态ram(dram))可以使用所讨论的实施例。
[0298]
本发明的实施例旨在涵盖落入所附权利要求的宽泛范围之内的所有这样的替换、修改和变型。因此,凡在本发明的精神和原则之内,所做的任何省略、修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。