植物产品中感染的预测
1.相关申请的交叉引用
2.本技术要求于2019年11月5日提交的美国临时专利申请号62/930,999的权益,该申请的全部内容通过引用并入本文。
3.背景
4.许多植物产品例如水果维持高感染率。当植物产品在生长过程中被病原体感染时,发生植物产品的收获前感染。例如,据估计,鳄梨园中大约20-40%的鳄梨树以及它们生产的鳄梨感染了炭疽病。未成熟的、收获前的植物产品通常对于这种感染是无症状的。然而,在植物产品被收获、成熟并经历衰老之后,植物产品变得有感染症状。感染症状可能包括茎端腐烂、霉、脉管/内部褐变和其他导致植物产品的有害质量损失的特征。55.植物产品感染可通过表现出感染症状的植物产品高度传播。此外,在收获后加工中,不同的处理方法和储存条件使植物产品容易受到在植物产品表面的攻击。感染及其相关症状极大地影响了提供给消费者的植物产品的质量。结果,受到感染的植物产品经常被扔掉,使植物产品的生物攻击成为导致收获后植物产品浪费率高的重要原因。虽然已经开发了许多不同的技术,包括清洗剂和杀虫剂,以减轻植物产品因感染而造成的损失,但供应商仍然难以防止因植物产品收获前感染而造成的损失。
6.由于植物产品感染引起的这些问题,需要在受感染的植物产品出现症状并能够将感染传播给其他植物产品之前鉴定受感染的植物产品。受感染的植物产品的早期检测能够对受感染的植物产品与未受感染的植物产品进行处理和隔离,从而减少感染的传播,减少植物产品的浪费,并提高消费者接受度。通过减少植物产品浪费和提高消费者接受度,供应商可以保证更高质量的植物产品,从而使供应商能够从市场上的其他供应商中脱颖而出。
7.目前用于鉴定未成熟、无症状植物产品中感染的方法包括通过量化引起感染的病原体的生物标志物(例如dna、rna等)来直接检测感染。
1-4
然而,由于引起感染的病原体通常在植物产品中以痕量存在,通过量化病原体生物标志物直接检测植物产品中的感染可能是困难且不可靠的。
8.概述
9.在一个方面,本公开内容提供了一种预测n个相似来源的植物产品的集合中感染的可能性的方法,其中n是大于10的整数。该方法包括从植物产品的集合中选择m个植物产品的子集,其中m为大于1且小于n/2的整数。然后,对于所选植物产品的子集的每个植物产品,确定一种或多种感染生物标志物的表达水平。对于所选植物产品的子集,基于子集的每个植物产品的一种或多种感染生物标志物的确定的表达水平确定生物标志物表达统计数据集。至少部分基于为植物产品子集确定的生物标志物表达统计数据集,预测植物产品的集合中感染的可能性。最后,返回植物产品的集合中预测的感染可能性。
10.在另一个方面,本公开内容提供了预测n个相似来源的植物产品的集合中感染的可能性的另一种方法,其中n是大于10的整数。该方法包括从植物产品的集合中选择m个植物产品的子集,其中m是大于2且小于n/2的整数。然后将子集的m个植物产品划分为p个子组,其中p是大于1且小于m的整数,并且其中p个子组中的每一个包含该子集的植物产品中
的至少1个。接下来,对于p个子组中的每一个,将来自该子组的每个植物产品的植物物质组合以形成汇集的植物物质组,其中p个汇集的植物物质组形成汇集的植物物质组的集合。接下来,对于汇集的植物物质组的集合,基于确定的植物物质组集合的每个汇集的植物物质组的一种或多种感染生物标志物的表达水平确定生物标志物表达统计数据集。至少部分基于为汇集的植物物质组的集合确定的生物标志物表达统计数据集,预测植物产品的集合中感染的可能性。最后,返回植物产品的集合中预测的感染可能性。
11.本文所述的任何方法可以包括单独或彼此组合的一个或多个以下步骤或特征。该方法可以进一步包括确定该子集的每个植物产品或每个汇集的植物物质组的一种或多种管家生物标志物的表达水平,其中至少基于子集的每个植物产品或每个汇集的植物物质组的一种或多种感染生物标志物的表达水平和一种或多种管家生物标志物的表达水平确定植物产品的子集或汇集的植物物质组集合的生物标志物表达统计数据集。一种或多种感染生物标志物可以选自表1。一种或多种管家生物标志物可以选自表2。每种生物标志物可以是小分子,每种生物标志物的表达水平可以包括或可以是对应的小分子的定量的量。
12.可以通过机器学习感染预测模型来预测植物产品的集合中感染的可能性。机器学习感染预测模型包括多个参数和一个函数。部分包含机器学习感染预测模型的函数表示作为机器学习感染预测模型的输入接收的植物产品子集或汇集的植物物质组的集合的生物标志物表达统计数据集与作为机器学习感染预测模型的输出生成的植物产品的集合中的预测的感染可能性之间的关系。
13.在使用机器学习感染预测模型来预测植物产品的集合中感染的可能性之前,机器学习感染预测模型的参数在一个或多个训练阶段期间被学习。具体地,在训练期间,感染预测模型的参数至少部分地基于包括多个训练样品的训练数据集来鉴定。训练数据集中的每个训练样品与相似来源的植物产品的回顾性集合相关联,并且包括来自相似来源植物产品的回顾性集合的植物产品子集或汇集的植物物质组集合的生物标志物表达统计数据集。如上所述,对于来自植物产品的回顾性集合的植物产品子集的每个植物产品或汇集的植物物质组集合的每个汇集的植物物质组,生物标志物表达统计数据集基于一种或多种感染生物标志物的表达水平和任选地基于一种或多种管家生物标志物的表达水平。此外,训练数据集中的每个训练样品还包括植物产品回顾集合中的实际已知感染率。下面更详细地讨论使用训练数据集训练感染预测模型。
14.在机器学习感染预测模型的训练和在一些实施方案中的验证之后,感染预测模型可以如上所述用于预测植物产品的集合中感染的可能性。为了预测植物产品的集合中感染的可能性,将针对植物产品子集或汇集的植物物质组的集合确定的生物标志物表达统计数据集输入到机器学习感染预测模型中。然后,至少部分基于植物产品子集或汇集的植物物质组的集合的生物标志物表达统计数据集以及至少基于训练数据集在训练期间鉴定的多个参数,机器学习感染预测模型输出植物产品的集合中预测的感染可能性。
15.在通过机器学习感染预测系统预测相似来源的植物产品的集合中感染的可能性的实施方案中,机器学习感染预测系统的函数可以包括以下的一个或多个:二元逻辑回归模型、逻辑模型树、逻辑模型回归器、随机森林分类器、逻辑模型回归器、l2正则化、偏最小二乘法分类、朴素贝叶斯分类器、多变量样条、一个或多个神经网络和k最近邻分类。在机器学习感染预测模型的函数包括k最近邻分类和随机森林分类器之一的实施方案中,机器学
习感染预测模型能够预测植物产品的集合中感染的可能性为大于至少5%的阈值(例如,至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%或95%),并且具有至少90%的准确率、至少80%的召回率和至少80%的精确率之一。
16.确定植物产品子集的生物标志物表达统计数据集可以包括,对于植物产品子集的每个植物产品或汇集的植物物质组的集合的每个汇集的植物物质组,确定一种或多种感染生物标志物的标准化表达水平并确定一种或多种感染生物标志物的特征缩放表达水平。植物产品或汇集的植物物质组的一种或多种感染生物标志物的标准化表达水平可以基于一种或多种感染生物标志物的表达水平并且任选地还基于一种或多种管家生物标志物的表达水平。例如,在一些实施方案中,确定植物产品或汇集的植物物质组的一种或多种感染生物标志物的标准化表达水平可以包括确定一种或多种感染生物标志物的表达水平与一种或多种管家生物标志物的表达水平的比率。植物产品或汇集的植物物质组的一种或多种感染生物标志物的特征缩放表达水平进而基于植物产品或汇集的植物物质组的一种或多种感染生物标志物的标准化表达水平。在一些实施方案中,确定植物产品或汇集的植物物质组的一种或多种感染生物标志物的特征缩放表达水平可以包括执行植物产品或汇集的植物物质组的一种或多种感染生物标志物的标准化表达水平的最小-最大标准化6和对数转换中的至少一种。
17.植物产品子集或汇集的植物物质组的集合的生物标志物表达统计数据集可以基于子集中每个植物产品或汇集的植物物质组集合中的每个汇集的植物物质组的一种或多种感染生物标志物的特征缩放表达水平。例如,在一些实施方案中,通过确定植物产品子集或汇集的植物物质组集合中的汇集的植物物质组的一种或多种感染生物标志物的特征缩放表达水平的平均值、中位数、最小值、最大值、标准偏差、第5个百分位数、第10个百分位数、第15个百分位数、第20个百分位数、第25个百分位数、第50个百分位数、第75个百分位数、第80个百分位数、第90个百分位数、第95个百分位数和第99个百分位数中的至少一种,和确定植物产品子集中的每个植物产品或每个汇集的植物物质组的一种或多种感染生物标志物中的至少一种的特征缩放表达水平与一种或多种感染生物标志物中的至少另一种的特征缩放表达水平的比率和/或植物产品子集中的每个植物产品或汇集的植物物质组集合中的每个汇集的植物物质组的一种或多种感染生物标志物中的至少一种的特征缩放表达水平与一种或多种感染生物标志物中的至少另一种的特征缩放表达水平的乘积,来确定植物产品子集和汇集的植物物质组集合的生物标志物表达统计数据集。
18.在某些实施方案中,感染包括真菌感染。在这样的实施方案中,真菌感染可以由一种或多种病原体引起,所述病原体选自炭疽菌属(colletotrichum)(即胶孢炭疽菌(c.gloeosporioides)、尖孢炭疽菌(c.acutatum))、溃疡病菌属(dothiorella)(即d.iberica、杨树溃疡病菌(d.gregaria)、d.aromatica)、葡萄座腔菌属(neofusicoccum)(即n.luteum、小新壳梭孢菌(n.parvum)、n.australe)、间座壳属(diaporthe)(即d.neotheicola、d.cinnamomi)、可可毛色二孢菌属(lasiodiplodia)(即l.pseudotheobromae、l.theobromae)、腐病菌属(diplodia)(即d.mutila、d.pseuodoseriata、d.seriata)和葡萄座腔菌科(botryosphaeria)(即b.dothidea)。例如,感染可表现为茎端腐烂、霉和/或脉管/内部褐变等。
19.在其中预测感染可能性的相似来源的植物产品的集合可以是收获的或未收获的植物产品。此外,在其中预测感染可能性的相似来源的植物产品的集合可以是成熟或未成熟的植物产品。在某些情况下,植物产品没有可见的感染症状。植物产品可以例如包括鳄梨、石榴、柿子、苹果、梨、葡萄、柑橘类水果、木瓜、樱桃、甜瓜、番石榴、芒果或核果等。
20.为了确定植物产品中生物标志物的表达水平,可以从植物产品的外果皮、内果皮和中果皮中的一种或多种提取材料,并且可以对提取的材料进行生物标志物分析以确定生物标志物的表达水平。此外,生物标志物表达水平的确定可以例如通过进行一种或多种选自以下的分析来确定:qpcr、pcr、rt-pcr、核糖核酸(rna)测序(rna-seq)、tag-seq、使用测序的转座酶可及染色质测定(atac-seq)、cytof/scop、e-ms/abseq、mirna-seq、cite-seq、质谱(ms)、气相色谱串联质谱(gc-ms)、综合二维气相色谱(gcxgc)、固相微萃取(spme)串联gcxgc(spme-gcxgc)、基质辅助激光解吸/电离(maldi)和maldi-tof。
21.生物标志物可以例如是基因。在生物标志物是基因的实施方案中,生物标志物的表达水平可以对应于与基因相关的rna序列的表达水平。在这样的实施方案中,与基因相关的rna序列的表达水平可以进一步包括与基因相关的rna序列的拷贝数。
22.在本文公开的任何方法中,预测植物产品的集合中感染的可能性可需要少于6小时(例如,少于5小时、少于4小时、少于3小时、少于2小时,或少于1小时)。在植物产品的集合中感染的可能性的预测之后,可以通过将预测的感染可能性自动呈现给查看用户来返回预测的感染可能性。此外,相似来源的植物产品的集合可以基于针对相似来源的植物产品的集合预测的感染可能性进行处理。具体而言,可以基于预测的感染可能性来鉴定具有感染风险的相似来源的植物产品的集合中的一个或多个植物产品。
23.在本文公开的任何方法中,可以基于预测的感染可能性为相似来源的植物产品的集合提供或开具抗微生物处理。可以根据预测的感染可能性选择性地收获相似来源的植物产品的集合。在本文公开的任何方法中,可以基于预测的感染可能性确定相似来源的植物产品的集合的质量保证。此外,可以基于预测的感染可能性为相似来源的植物产品的集合鉴定消费者和地理目的地中的至少一个。此外,基于预测的感染可能性,可以为相似来源的植物产品的集合暂停或开具乙烯处理。乙烯处理可以包括,例如,外源气态乙烯的应用、乙烯吸收剂的使用或化学乙烯阻断剂(即1-mcp)的应用。在一些情况下,可以基于预测的感染可能性来鉴定相似来源的植物产品的集合的一个或多个储存条件。例如,储存条件可以包括储存温度和储存湿度中的至少一种。此外,可以根据预测的感染可能性为相似来源的植物产品的集合提供收获后处理或开具收获后处理的剂量。此外,可以基于预测的感染可能性向用户提供基于预测的感染可能性来指导用户执行任何一种上述植物加工方法的指令。
24.在另一个方面,本公开内容提供了一种存储计算机程序指令的非暂时性计算机可读存储介质,当由计算机处理器执行时,其使计算机处理器通过执行上述方法步骤的任意组合来预测相似来源的植物产品的集合中感染的可能性。
25.在另一个方面,本公开内容可以包括一种用于鉴定植物产品中的潜伏感染的方法。在一些实施方案中,该方法可以包括通过一个或多个计算机获得描述植物产品中一种或多种感染生物标志物的表达水平的数据、通过一个或多个计算机将获得的数据编码成用于输入机器学习模型的数据结构、通过一个或多个计算机将编码的数据结构作为输入提供给机器学习模型,该机器学习模型已被训练以基于处理编码的数据结构生成指示植物产品
具有潜伏感染的可能性的输出数据、通过一个或多个计算机获得指示植物产品具有潜伏感染的可能性的生成的输出数据,通过一个或多个计算机并基于生成的输出数据确定植物产品具有潜伏感染,并且通过一个或多个计算机执行一种或多种操作以减轻植物产品中的潜伏感染。
26.其他方面包括相应的系统、装置和计算机程序,以执行如本文所公开的方法的动作,如由编码在计算机可读存储设备上的指令所定义的。
27.这些和其他方面可以可选地包括以下特征中的一个或多个。在一些实施方案中,通过一个或多个计算机执行一种或多种操作以减轻植物产品中的潜伏感染可包括通过一个或多个计算机并基于输出数据确定对一个或多个其他相似来源的植物产品开具抗微生物处理。
28.在一些实施方案中,通过一个或多个计算机执行一种或多种操作以减轻植物产品中的潜伏感染可包括通过一个或多个计算机并基于输出数据对一个或多个其他相似来源的植物产品施用抗微生物处理。
29.在一些实施方案中,通过一个或多个计算机执行一个或多个操作以减轻植物产品中的潜伏感染可以包括通过一个或多个计算机并基于输出数据确定运输一个或多个其他相似来源的植物产品的车辆将被改程到不同的目的地。
30.在一些实施方案中,通过一个或多个计算机执行一个或多个操作以减轻植物产品中的潜伏感染可以包括通过一个或多个计算机基于输出数据生成警报消息,当由用户设备处理时,其使用户设备输出通知用户设备的用户车辆将被改程到不同的目的地的警报;和通过一个或多个计算机将生成的警报传输到用户设备。
31.在一些实施方案中,通过一个或多个计算机执行一个或多个操作以减轻植物产品中的潜伏感染可以包括基于输出数据并基于输出数据生成警报消息,当由用户设备处理时,其使用户设备输出通知用户设备的用户一个或多个其他相似来源的植物产品的警报;和通过一个或多个计算机将警报消息传输到用户设备。
32.在一些实施方案中,描述植物产品中一种或多种感染生物标志物的表达水平的数据可以包括一种或多种变体的列表,其中一种或多种变体描述植物产品的读数序列和健康植物产品的参考基因组之间的差异。
33.在一些实施方案中,机器学习模型可以包括二元逻辑回归模型、逻辑模型树、随机森林分类器、l2正则化、偏最小二乘中的一种或多种。
34.在一些实施方案中,植物产品不包括任何可见的感染迹象。
35.在一些实施方案中,该方法可以包括通过一个或多个计算机从植物产品的集合中选择m个植物产品的子集,其中m是大于1且小于n/2的整数,并且对于子集的每个植物产品,通过一个或多个计算机确定一种或多种感染生物标志物的表达水平。在这样的实施方案中,通过一个或多个计算机获得描述植物产品中一种或多种生物标志物表达水平的数据可以包括通过一个或多个计算机并且对于子集的每个植物产品获得描述一种或多种感染生物标志物的所确定的表达水平的数据。同样地,在这样的实施方案中,通过一个或多个计算机将获得的数据编码成用于输入到机器学习模型的数据结构可以包括通过一个或多个计算机将获得的描述一种或多种感染生物标志物的确定的表达水平的数据编码成用于输入机器学习模型的一种或多种数据结构。
36.附图的简要说明
37.本发明的这些和其他特征、方面和优点将通过以下描述和附图得到更好的理解,其中:
38.图1是用于预测相似来源的植物产品的集合中感染的可能性的方法的流程图。
39.图2a是系统环境的框图,其中从植物产品的集合中选择植物产品的子集以用于预测植物产品的集合中感染的可能性。
40.图2b是系统环境的框图,其中为相似来源的植物产品的集合预测感染的可能性。
41.图2c是系统环境的框图,其中为植物产品的子集确定生物标志物表达统计数据集。
42.图2d是系统环境的框图,其中为植物产品的子集确定生物标志物表达统计数据集。
43.图3是感染预测系统的系统环境的框图,该感染预测系统被配置为预测相似来源的植物产品的集合中感染的可能性。
44.图4是配置成预测相似来源的植物产品的集合中感染的可能性的感染预测系统的架构的框图。
45.图5a是其中训练、验证和使用感染预测系统的系统环境的框图。
46.图5b是其中训练感染预测系统的系统环境的框图。
47.图5c是其中验证感染预测系统的系统环境的框图。
48.图5d是其中使用感染预测系统的系统环境的框图。
49.图6是描绘多个受感染的鳄梨和多个对照鳄梨的每个候选感染生物标志物的平均标准化表达水平的图。
50.图7a是描绘每批鳄梨a-k中茎端腐烂发生率的图表。
51.图7b是比较从每批鳄梨测试的六个鳄梨的每个鳄梨中的pal基因的标准化表达水平与鳄梨起源的那批鳄梨中茎端腐烂的发生率的图。
52.图8是描述对于mx28和mx29批次的鳄梨,在包装后第7-12天的每一天测试的六个鳄梨中每一个中pal基因的标准化表达水平的图表。
53.图9是描绘感染预测系统的预测性能的图表。
54.图10a是用于训练、验证和测试感染预测系统的示例数据流水线的概览的框图。
55.图10b是用于训练、验证和测试感染预测系统的示例性数据流水线的框图,该感染预测系统被配置为预测相似来源的植物产品的集合是否具有大于或小于5%的感染可能性。
56.图10c是用于测试和评估感染预测系统的性能的示例性方案的框图。
57.图11图示了用于实现图1中描述的方法的示例计算机。
58.图12是用于预测相似来源的植物产品的集合中感染可能性的方法的流程图。
59.图13是系统环境的框图,其中为相似来源的植物产品的集合预测感染的可能性。
60.详细描述
61.本公开内容涉及用于检测植物中的潜伏感染的系统、方法和计算机程序。在一个方面,本公开内容涉及一种经过训练的机器学习模型,该模型可以基于代表植物产品的特征的输入数据的机器学习模型处理来生成指示植物产品具有潜伏感染的可能性的输出数
据。在一些实施方案中,植物产品的特征可以包括代表在植物产品样品中检测到的一种或多种生物标志物的表达水平的数据。如果本公开内容基于所生成的输出数据确定植物产品具有潜伏感染,则本公开内容可以导致执行一种或多种旨在减轻检测到的潜伏感染的操作。
62.本公开内容提供了多种技术优势。例如,在一些实施方案中,本公开内容可以通过仅对相似来源的植物产品的集合的子集进行采样来减少需要破坏以检测相似来源的植物产品的集合中的潜伏感染的植物产品的量。在这些或其他实施方案中,本公开内容还通过检测潜伏感染并启动一种或多种补救措施以在潜伏感染表现之前减轻甚至消除潜伏感染来提供节省植物产品的益处,从而允许植物产品出售。这为本公开内容的用户提供了显著的经济利益。这些和其他优点从本文的公开内容中显而易见。
63.i.定义
64.通常,权利要求和说明书中使用的术语旨在被解释为具有本领域普通技术人员所理解的简单含义。下面定义了某些术语以提供额外的澄清。如果简单含义与提供的定义发生冲突,则应使用提供的定义。
65.本文未直接定义的任何术语应被理解为具有与它们通常相关的含义,如在本公开内容的领域内所理解的。本文讨论了某些术语以在描述本公开内容的方面的组合物、装置、方法等以及如何制造或使用它们时向从业者提供额外的指导。可以理解的是,可以以不止一种方式说同样的事情。因此,替代语言和同义词可用于本文讨论的任何一个或多个术语。此处是否详细阐述或讨论了一个术语并不重要。提供了一些同义词或可替代的方法、材料等。一个或几个同义词或等同词的叙述不排除使用其他同义词或等同词,除非明确说明。示例的使用,包括术语的示例,仅用于说明目的并且不限制本文中本公开内容的方面的范围和含义。
66.如本文所提及的,术语“植物产品”是指由植物产生的任何产品。植物产品包括例如果实、蔬菜、种子、花、块茎、鳞茎和植物的任何其他产品。例如,在一些实施方案中,植物产品可以包括鳄梨、石榴、柿子、苹果、梨、葡萄、柑橘类水果、木瓜、樱桃、甜瓜、番石榴、芒果或核果。
67.如上所述,本公开内容讨论了相似来源的植物产品的集合中的感染的预测。相似来源的植物产品的集合包括多个(即多于一个)植物产品。如上所述,关于植物产品的术语“相似来源”是指位于同一地理区域的多个植物产品。地理区域的界限可能会有所不同。例如,如上所述,相似来源的植物产品的集合可以包括在同一果园中种植的植物产品。在另一个示例中,相似来源的植物产品的集合可以包括在同一卡车上运输的植物产品的集合。在又一个示例中,相似来源的植物产品的集合可以包括在同一杂货店出售的植物产品。在另一个示例中,相似来源的植物产品的集合可以包括属于同一商业批次的植物产品的集合。相似来源的植物产品的地理定义的基础是将能够造成相同感染的植物产品的集合在一起,无论是植物产品的收获前还是收获后。
68.如本文所提及的,关于从n个植物产品的相似来源的集合中取样植物产品的术语“子集”(其中n是大于10的整数)是指m个植物产品的代表性样品,其中m是大于1且小于n/2的整数。该子集从植物产品的集合中随机选择,使得该子集的测量的生物标志物统计数据可推广到该集合。
69.如本文所用,术语“植物物质”是指植物的任何部分,包括例如果实(在植物意义上,包括果皮和果汁囊)、蔬菜、叶子、茎、树皮、种子、花、果皮、坚果、仁、果肉或根。植物物质包括收获前植物或其部分以及收获后植物或其部分,包括例如收获的果实和蔬菜、收获的根和浆果以及采摘的花。
70.如本文所述,关于植物产品的术语“感染”是指存在于植物产品中的任何病原感染。在一些实施方案中,植物产品感染可以是潜伏感染,使得受感染的植物产品不呈现可见的感染症状。在替代实施方案中,植物产品感染可以表现出明显的症状,包括下面讨论的那些。
71.植物产品感染可由任何病原体引起并且可包括例如细菌感染、病毒感染、真菌感染和卵菌感染或其任何组合。在植物产品感染包括真菌感染的实施方案中,真菌感染可以例如由选自以下的病原体引起:炭疽菌属(colletotrichum)(即胶孢炭疽菌(c.gloeosporioides)、尖孢炭疽菌(c.acutatum))、溃疡病菌属(dothiorella)(即d.iberica、杨树溃疡病菌(d.gregaria)、d.aromatica)、葡萄座腔菌属(neofusicoccum)(即n.luteum、小新壳梭孢菌(n.parvum)、n.australe)、间座壳属(diaporthe)(即d.neotheicola、d.cinnamomi)、可可毛色二孢菌属(lasiodiplodia)(即l.pseudotheobromae、l.theobromae)、腐病菌属(diplodia)(即d.mutila、d.pseuodoseriata、d.seriata)和葡萄座腔菌科(botryosphaeria)(即b.dothidea)和任何其他真菌病原体。
72.植物产品感染可以以任何方式表现出来。例如,在一些实施方案中,植物产品感染可表现为茎端腐烂、霉和/或脉管/内部褐变。作为具体示例,在一些实施方案中,感染可在鳄梨、苹果、梨和/或核果中表现为脉管/内部褐变。作为另一个示例,在一些实施方案中,感染可以在鳄梨和/或芒果中表现为茎端腐烂。
73.如上所述,植物产品感染可以是潜伏感染,使得受感染的植物产品在很长一段时间内没有可见的感染症状。在这个潜伏期结束时,植物产品感染可能表现为茎端腐烂、霉和/或脉管/内部褐变之一。作为一个具体的例子,在一些实施方案中,来自炭疽菌属的真菌的潜伏感染可以在鳄梨和/或芒果中表现为茎端腐烂。
74.如本文所提及,关于一种或多种植物产品的术语“感染的可能性”是指一种或多种植物产品中感染的可能性的预测。相反,如本文所述,关于一种或多种植物产品的术语“感染率”是指一种或多种植物产品中实际的、已知的感染发生率。如下文进一步详细讨论的,植物产品中的感染率可部分用于训练感染预测模型以预测其他植物产品中感染的可能性。
75.对于本文所述的任何实施方案,可以在植物产品生命周期的任何阶段针对相似来源的植物产品的集合预测植物产品的感染的可能性。例如,可以预测收获或未收获的植物产品的集合的感染可能性。作为另一个例子,可以预测成熟或未成熟的植物产品的集合的感染可能性。未成熟植物产品的感染预测可能特别有用,因为感染通常潜伏在未成熟植物产品中,并且直到植物产品成熟后才能目视检测到。因此,预测未成熟植物产品中的感染可以在肉眼可检测到之前暴露感染。
76.如本文所述,关于植物产品的术语“生物标志物”是指存在于植物产品中的任何分子。例如,生物标志物可以包括核酸,包括dna、修饰的(例如,甲基化的)dna、cdna和rna,包括编码(例如,mrna、trna)和非编码rna(例如,sncrna、mirna、pirna、incrna),蛋白质,包括
转录后修饰的蛋白质(例如,磷酸化、糖基化、肉豆蔻化等的蛋白质),核苷酸(例如,三磷酸腺苷(atp)、二磷酸腺苷(adp)和一磷酸腺苷(amp)),包括环状核苷酸,例如环状腺苷一磷酸(camp)和环状鸟苷一磷酸(cgmp),生物制剂,adc,小分子,例如氧化和还原形式的烟酰胺腺嘌呤二核苷酸(nadp/nadph),挥发性化合物,及其任意组合。
77.如本文所提及的,关于生物标志物的术语“表达水平”是指作为生物标志物表达的代表的任何物质的量度。该量度可以是定量的、定性的、绝对的和/或相对的。例如,在生物标志物包含基因的实施方案中,生物标志物的表达水平可以包括与基因相关的rna转录物的量化。下文进一步详细讨论了生物标志物表达水平的测定。
78.如本文所用,术语“感染生物标志物”是指适用于预测植物产品中感染的可能性的任何生物标志物,包括已确定在受感染的植物产品中与未感染的植物产品相比差异表达的生物标志物。换言之,适用于预测植物产品中感染可能性的感染生物标志物包括已确定与植物产品中的感染相关的生物标志物。例如,感染生物标志物可以包括与未感染的植物产品相比在受感染的植物产品中以阈值百分比差异表达的生物标志物。例如,感染生物标志物可以包括已确定在受感染的植物产品中与未受感染的植物产品相比差异表达至少0.1倍变化(例如,至少0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9、1、2、3、4、5、10或20倍变化)的生物标志物。表1包含示例性感染生物标志物的列表。
79.如本文所用,“管家生物标志物”是指适用于植物产品中的已被确定与植物产品中的感染不相关的生物标志物。换言之,管家生物标志物的表达在受感染和未受感染的植物产品中是一致的(例如,表现出小于0.5、0.4、0.3、0.2或0.1倍的变化)。表2包含示例性管家生物标志物的列表。
80.如本文所用,关于植物产品的术语“乙烯处理”是指改变或旨在改变收获后植物产品中的乙烯组成、消耗或检测的任何产品的应用或使用。在一些实施方案中,乙烯处理包括在收获后将外源气态乙烯应用于植物产品以改变或控制成熟速率。在其他实施方案中,乙烯处理包括在收获后将乙烯抑制剂、阻断剂或吸收剂(例如,1-甲基环丁二烯,本文中为“1-mcp”)使用或应用于植物产品以控制成熟率或延长植物产品的货架期。
81.ii.方法概述
82.图1是用于预测相似来源的植物产品的集合中感染可能性的示例性方法100的流程图。在一些实施方案中,设置了多于阈值数量的植物产品的相似来源的植物产品。例如,在一些实施方案中,植物产品的阈值数量可以大于10个植物产品。然而,本公开内容不限于这样的阈值数量,并且相似来源的植物产品的集合可以包括多于或少于10个植物产品。尽管显示某些步骤以特定顺序发生,但在某些情况下,该方法的步骤可以以与结合图1描述的顺序不同的顺序执行。此外,在某些情况下,某些步骤可以省略和/或由其他步骤代替,和/或可以添加额外的步骤。
83.为了预测相似来源的植物产品的集合中感染的可能性,如图1所示,从相似来源的植物产品的集合中选择植物产品的子集101。该子集通常包括至少2个植物产品,并且少于相似来源的植物产品的集合中产品总数的一半。从相似来源的植物产品的集合中选择植物产品的子集101将在下文关于图2a更详细地讨论。
84.确定所选植物产品子集的每个植物产品的至少一种感染生物标志物和任选地至少一种管家生物标志物的表达水平102。适用于预测植物产品中感染可能性的感染生物标
志物和管家生物标志物的鉴定将在下文进一步详细讨论。然而,简而言之,适用于预测植物产品中感染可能性的感染生物标志物包括已确定与未受感染的植物产品相比在受感染的植物产品中差异表达的生物标志物。换言之,适用于预测植物产品中感染可能性的感染生物标志物包括已确定与植物产品中的感染相关的生物标志物。在优选的实施方案中,感染生物标志物包括与未感染的植物产品相比在受感染的植物产品中以阈值百分比差异表达的生物标志物。例如,感染生物标志物可以包括已确定在受感染的植物产品中与未受感染的植物产品相比差异表达至少0.1倍变化(例如,至少0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9、1、2、3、4、5、10或20倍变化)的生物标志物。
85.适用于预测植物产品中感染可能性的管家生物标志物包括已确定与植物产品中的感染不相关的生物标志物。换言之,管家生物标志物的表达在受感染和未受感染的植物产品中是一致的(例如,表现出小于0.5、0.4、0.3、0.2或0.1倍的变化)。如下文详细讨论的,管家生物标志物表达可用于使受感染和未受感染的植物产品之间的感染生物标志物表达标准化。
86.在一些实施方案中,植物产品中的生物标志物表达水平可以通过使用被配置为检测植物产品中生物标志物表达水平的阵列和/或试剂盒测试植物产品来确定。在一些进一步的实施方案中,确定植物产品中的生物标志物表达水平还可以包括制备植物产品以用于生物标志物分析。例如,确定植物产品中的生物标志物表达水平可以包括从植物产品中提取材料用于生物标志物分析。下文详细讨论了用于生物标志物分析的植物产品的制备。
87.接下来,基于子集中每个植物产品的确定的至少一种感染生物标志物和任选地至少一种管家生物标志物的表达水平确定植物产品子集的生物标志物表达统计数据集103。如下面关于图2c详细讨论,在一些实施方案中,植物产品子集的生物标志物表达统计数据集是基于植物产品子集中每个植物产品的至少一种感染生物标志物的特征缩放表达水平确定的,该特征缩放表达水平进而基于植物产品子集中每个植物产品的至少一种感染生物标志物的标准化表达水平确定。此外,如下面关于图2d详细讨论的,植物产品子集的生物标志物表达统计数据集可以包括至少一种感染生物标志物的特征缩放表达水平的平均值、中位数、最小值、最大值、标准偏差、第5个百分位数、第10个百分位数、第15个百分位数、第20个百分位数、第25个百分位数、第50个百分位数、第75个百分位数、第80个百分位数、第90个百分位数、第95个百分位数和第99个百分位数中的至少一种,植物产品子集的每个植物产品的至少一种感染生物标志物中的至少一种的特征缩放表达水平与至少一种感染生物标志物中的至少另一种的特征缩放表达水平的比率,和植物产品子集的每个植物产品的至少一种感染生物标志物中的至少一种的特征缩放表达水平与至少一种感染生物标志物中的至少另一种的特征缩放表达水平的乘积。
88.最后,至少部分地基于所确定的植物产品子集的生物标志物表达统计数据集来预测相似来源的植物产品的集合中感染的可能性104。在一些实施方案中,如下面关于图3-5d详细讨论的,可以使用机器学习感染预测系统至少部分地基于植物产品子集的确定的生物标志物表达统计数据集来预测相似来源的植物产品的集合中感染的可能性。在某些实施方案中,可在少于6小时内完成对相似来源的植物产品的集合中感染可能性的预测。
89.然后可以返回在相似来源的植物产品的集合中感染的预测的可能性,并且在一些实施方案中,其被利用来故意处理相似来源的植物产品的集合。例如,相似来源的植物产品
的集合中潜伏感染的预测的可能性可用于确定何时何地销售植物产品的集合。下面详细讨论基于感染预测的植物产品处理的其他实施方案。
90.iii.用于生物标志物分析的植物产品的选择
91.转向图2a,图2a是示例性系统环境的框图200a,其中从植物产品的集合201中选择植物产品子集202以用于预测植物产品的集合201中感染的可能性。
92.在一些实施方案中,相似来源的植物产品的集合包括总共n个植物产品,其中n是大于10的整数。例如,如图2a所示,要预测感染可能性的植物产品的集合201包括16个单独的植物产品,每个单独的植物产品由正方形表示。然而,在替代实施方案中,相似来源的植物产品的集合可以包括任何数量的单独的植物产品。
93.在进一步的实施方案中,从相似来源的植物产品的集合中选择的植物产品子集可以包括m个植物产品,其中m是大于1且小于n/2的整数。在一些实施方案中,可以随机选择从相似来源的植物产品的集合中选择的植物产品子集。例如,如图2a所示,植物产品子集202是从相似来源的植物产品的集合201中随机选择的,并且包括4个单独的植物产品。然而,在替代实施方案中,从相似来源的植物产品的集合中选择的植物产品子集可以包括任何数量的单独的植物产品。
94.通过分析来自相似来源的n个植物产品的集合201的m个植物产品的代表性子集202(其中m大于1且小于n/2),以预测在n个植物产品的整个集合中感染的可能性,需要更少的时间和来源以预测n个植物产品的集合中的感染。例如,通过分析m个植物产品的子集,更少的植物产品被剔除以进行此预测,从而实现更高的产品质量,但仍具有较高的产品产率。
95.iv.用于生物标志物分析的植物产品的制备
96.如上所述,在从相似来源的植物产品的集合中选择植物产品子集后,确定该植物产品子集的每个单独植物产品中感染生物标志物和任选地管家生物标志物的表达以预测植物产品的集合中感染的可能性。然而,也如上所述,在一些情况下,在确定子集中每个植物产品中的生物标志物表达水平之前,将子集的每个植物产品制备用于生物标志物分析。
97.用于生物标志物分析的植物产品的制备可以取决于待测量的特定生物标志物。取决于待测量的特定生物标志物,植物产品中的生物标志物表达可以根据许多不同的方法来确定。例如,生物标志物表达可以通过聚合酶链反应(pcr)、定量聚合酶链反应(qpcr)、逆转录聚合酶链反应(rt-pcr)、逆转录定量聚合酶链反应(rt-qpcr)、核糖核酸(rna)测序(rna-seq)、tag-seq、使用测序的转座酶可及染色质测定(atac-seq)、cytof/scop、e-ms/abseq、mirna-seq、cite-seq、质谱(ms)、气相色谱串联质谱(gc-ms)、综合二维气相色谱(gcxgc)、固相微萃取(spme)串联gcxgc(spme-gcxgc)、基质辅助激光解吸/电离(maldi)和maldi-tof以及它们的任何组合来测量。可以基于待测量的生物标志物来选择测量方法。例如,pcr和qpcr测量dna表达。rt-pcr、rt-qpcr、rna-seq、tag-seq和mirna-seq测量rna表达。具体来说,rt-pcr、rt-qpcr和rna-seq测量rna转录物的表达,tag-seq允许检测稀有mrna种类,而mirna-seq测量micro-rna的表达。cytof/scop和e-ms/abseq测量蛋白质表达。cite-seq同时测量核酸表达和蛋白质表达。atac-seq测量染色质构象。
98.可以根据被测量的生物标志物和根据生物标志物测量的方法制备植物产品以用于生物标志物分析。更具体地,可以基于目标生物标志物和目标生物标志物测量方法从植物产品中提取材料(例如,植物物质)用于生物标志物测量。例如,在生物标志物包含基因并
且使用rt-qpcr来量化植物产品中基因的rna表达的情况下,可以使用一种或多种rna提取方法从植物产品中提取rna,然后使用rt-qpcr测序。
99.此外,取决于进行生物标志物分析的植物产品的类型,可以从植物产品的特定部分提取植物物质材料用于生物标志物测量。例如,在一些实施方案中,可以从植物产品的外果皮、中果皮和内果皮中的一种或多种提取材料以用于生物标志物测量。
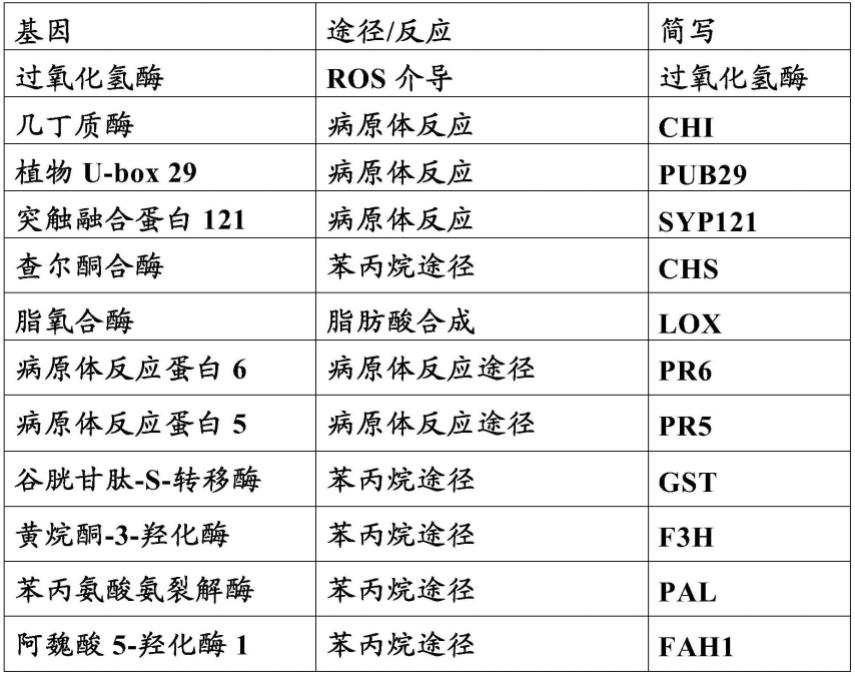
100.v.植物产品的生物标志物分析
101.接下来转到图2b,图2b是系统环境200b的框图,其中针对相似来源的植物产品的集合预测感染可能性206。图2b是图2a的延伸。具体地,图2b中描绘的植物产品的子集202是从图2a中相似来源的植物产品的集合201中选择的植物产品的子集202。因此,图2b是系统环境200b的框图,其中为图2a的相似来源的植物产品的集合201预测感染的可能性206。
102.如图2b所示,为了清楚起见,植物产品子集202的每个单独的植物产品被标记为植物产品202a、植物产品202b、植物产品202c和植物产品202d之一。此外,针对植物产品子集202中的每个植物产品确定至少一种感染生物标志物的表达水平和任选地至少一种管家生物标志物的表达水平。具体而言,确定植物产品202a的感染生物标志物表达203a和任选地管家生物标志物表达204a,确定植物产品202b的感染生物标志物表达203b和任选地管家生物标志物表达204b,确定植物产品202c的感染生物标志物表达203c和任选地管家生物标志物表达204c,和确定植物产品202d的感染生物标志物表达203d和任选地管家生物标志物表达204d。
103.如上所述,生物标志物的表达可以通过根据任何测量方法测量充当生物标志物表达的替代物的任何物质来确定。例如,当生物标志物包含基因时,该基因的rna转录物的拷贝数可以代表该基因的表达,并且rt-qpcr可用于量化该基因的rna转录物的拷贝数。在一些进一步的实施方案中,可以使用配置为检测生物标志物表达的阵列和/或试剂盒来确定生物标志物表达。因此,植物产品子集202的每个植物产品202a-d的至少一种感染生物标志物和任选地至少一种管家生物标志物的表达水平通过使用任何合适的测量方法测量用作生物标志物表达的替代物的任何物质来确定。下文详细讨论了在每个植物产品中测量其表达水平的至少一种感染生物标志物和任选地至少一种管家生物标志物的鉴定。
104.简要地回到图2b中的示例,在确定植物产品202a-d的感染生物标志物表达203a-d和任选地管家生物标志物表达204a-d之后,基于植物产品子集202中每个植物产品202a-d的感染生物标志物表达203a-d和任选地管家生物标志物表达204a-d确定植物产品子集202的生物标志物表达统计数据集205。如上所述,下面关于图2c-2d详细讨论了植物产品子集202的生物标志物表达统计数据集205的确定。
105.最后,如图2b所示,至少部分基于为植物产品子集202确定的生物标志物表达统计数据集205,针对图2a的相似来源的植物产品的集合201预测感染的可能性206。如上所述,感染可能性的预测206在下文关于图3-5d更详细地讨论。
106.植物产品的集合的子集的生物标志物表达数据(例如,生物标志物表达的水平)、植物产品的集合的子集的生物标志物表达统计数据集以及植物产品的集合的感染的可能性可以各自在任意位置组合处确定。作为一个例子,植物产品的集合的子集的生物标志物表达数据(例如,生物标志物表达的水平)、植物产品的集合子集的生物标志物表达统计数据集以及植物产品的集合的感染的可能性可以在植物产品的位置处确定。在另一个示例
中,可以在植物产品的位置处确定植物产品的集合的子集的生物标志物表达数据(例如,生物标志物表达水平),然后将其传输到另一个位置(例如,传输到远程计算系统)以确定植物产品的集合子集的生物标志物表达统计数据集和植物产品的集合的感染的可能性。在替代实施方案中,可以在位置的任何替代组合处确定植物产品的集合中感染可能性的预测。
107.v.a.感染生物标志物
108.如整个本公开内容所讨论的,感染生物标志物和管家生物标志物在选自相似来源的植物产品的集合的植物产品子集的每个植物产品中的表达水平用于预测植物产品的集合中感染的可能性。也如上面关于图1简要讨论的那样,感染生物标志物包括已确定在受感染的植物产品中与未受感染的植物产品相比表达差异的生物标志物。并且在更进一步的优选实施方案中,感染生物标志物包括与未受感染的植物产品相比在受感染的植物产品中以阈值百分比差异表达的生物标志物。例如,在优选的实施方案中,感染生物标志物包括已确定在受感染的植物产品中与未受感染的植物产品相比表达差异至少0.1倍的生物标志物。
109.如下所示,表1描述了满足该差异表达标准的一组示例性生物标志物。具体来说,表1描述了一组基因,这些基因在受感染的植物产品中的表达与在未感染的植物产品中的表达不同。此外,在一些实施方案中,表1中列出的基因还满足感染和未感染的植物产品之间至少0.1倍的阈值百分比的差异表达的优选标准。表1还显示了每个基因的途径/反应和简写。基因的途径/反应表明该基因参与的代谢途径,或基因表达的下游效应。基因的简写表示基因名称的首字母缩写词。表1中列出的感染生物标志物可以单独使用,也可以与下文讨论的管家生物标志物一起使用,以预测相似来源的植物产品的集合中感染的可能性。
110.表1:示例性感染生物标志物
111.112.[0113][0114]
为了解释植物产品之间生物标志物表达的变化,如下文实施例1中进一步详细讨论的,感染生物标志物的标准化表达水平可以基于感染生物标志物和管家生物标志物或感染生物标志物和另一种感染生物标志物的表达水平来确定。通过将感染生物标志物表达标准化至管家生物标志物的表达或另一种感染生物标志物的表达,可以控制植物产品之间基线感染生物标志物表达的变化。
[0115]
在一些实施方案中,可以针对特定植物产品检测表1中感染生物标志物的某些子集的表达水平。例如,如果要评估潜伏感染的植物或植物子集包括鳄梨,则表1中感染生物标志物的第一特定子集的表达水平的检测可以指示潜伏感染的存在。在一些实施方案中,感染生物标志物的第一特定子集可以包括pal、gst、comt、wrky75、f3h、pr6、pr5、lox、prbl-2或过氧化氢酶基因中的一个或多个。作为另一个例子,如果要评估潜伏感染的植物或植物子集包括应用,则表1中感染生物标志物的第二特定子集的表达水平的检测可以指示存在或不存在潜伏感染。在一些实施方案中,表1中的感染生物标志物的第二子集可以包括α-法呢烯、fa过氧化物裂解酶、乙烯响应传感器1、乙烯响应转录因子1、tiny、ac合酶、ac氧化酶、pr10、受体样蛋白激酶7几丁质诱导剂、苹果酸脱氢酶、丙酮酸脱羧酶、富马酸裂解酶、pal或c4h中的一种或多种。在其他实施方案中,表1中的感染生物标志物的第二子集可以包括植物u-box29、壳聚糖酶、突触融合蛋白121、阿魏酸5-羟化酶1、肉桂酸-4-羟化酶、苯丙氨酸氨裂解酶、膨胀素a8、九-顺式-环氧类胡萝卜素双加氧酶9、wrky31、lox5、acs1、aco5或ap2。
[0116]
表1中的感染生物标志物列表和上述生物标志物子集并非详尽无遗。具体而言,满足感染的植物产品与未感染的植物产品相比的差异表达的标准的其他生物标志物可包含在表1中。
[0117]
在一些实施方案中,不同于未受感染的植物中的基因表达的受感染的植物产品中的基因集合可以通过对由核酸测序仪产生的读数序列进行变体检出分析来鉴定。例如,核酸序列可用于对获自植物产品的集合的样品进行测序。基于获得的样品,由核酸测序仪产生的读数序列可以与健康植物的参考序列进行比对。然后可以获得比对的读数序列和参考序列之间的变体或差异,并用作不同于未受感染的植物中基因集合的表达的受感染的植物的基因集合的表达。
[0118]
v.b.管家生物标志物
[0119]
接下来转到用于预测植物产品中感染可能性的管家生物标志物,如上文关于图1
简要讨论的,管家生物标志物包括已确定与植物产品中的感染无关的生物标志物。换言之,在受感染和未受感染的植物产品中,管家生物标志物表达的变化小于0.5、0.4、0.3、0.2或0.1倍。因此,管家生物标志物表达可用于使感染和未受感染的植物产品之间的感染生物标志物表达标准化。具体而言,管家生物标志物表达可用于控制植物产品之间基线代谢活性的变化。
[0120]
许多植物产品管家生物标志物是本领域技术人员已知的。下面的表2描述了已知是植物产品中的管家生物标志物的一组基因,它们可用于使受感染和未受感染的植物产品之间的感染生物标志物表达标准化。表2还显示了每个基因的途径/反应和简写。基因的途径/反应指示该基因参与的代谢途径,或基因表达的下游效应。基因的简写指示基因名称的首字母缩写词。
[0121]
表2:示例性管家生物标志物
[0122][0123]
除了表2中列出的已知管家生物标志物之外,任何其他管家生物标志物都可以用于预测植物产品中的感染,如在本公开内容全文中所讨论的。鉴定另外的管家生物标志物的方法也是本领域技术人员已知的。例如,以下出版物讨论了在植物产品中鉴定管家基因的已知方法:chandna r,augustine r,bisht nc(2012)evaluation of candidate reference genes for gene expression normalization in brassica juncea using real time quantitative rt-pcr.plos one 7(5):e36918.https://doi.org/10.1371/journal.pone.0036918。除了表2中列出的那些之外,该出版物以及类似的出版物可用于鉴定其他管家生物标志物用于预测植物产品感染。
[0124]
表2中列出的管家生物标志物,以及根据已知方法鉴定的另外的管家生物标志物,可以与上面讨论的感染生物标志物一起使用,以预测相似来源的植物产品的集合中感染的可能性。
[0125]
vi.生物标志物表达的统计分析
[0126]
在确定选自相似来源的植物产品的集合的植物产品子集的每个植物产品中至少一种感染生物标志物和任选地至少一种管家生物标志物的表达水平之后,对这些生物标志
物表达水平进行统计分析。具体地,如上面关于图2b简要讨论的那样,植物产品子集202的生物标志物表达统计数据集205是基于植物产品子集202中每个植物产品202a-d的感染生物标志物表达203a-d和任选地管家生物标志物表达204a-d确定的。这个生物标志物表达统计数据集205用于预测在相似来源的植物产品的集合中感染的可能性206,如下面关于图3-5d详细讨论的。
[0127]
或者,在一些实施方案中,一种或多种生物标志物的绝对定量使用例如真实浓度标准、内标或制备的已知浓度的标准曲线来确定。在这些情况下,可能不需要确定管家生物标志物的表达。
[0128]
vi.a.生物标志物表达统计数据集
[0129]
图2c是示例性系统环境200c的框图,其中为植物产品的子集确定生物标志物表达统计数据集。图2c是图2a-b的应用的扩展,其中针对植物产品子集的每个植物产品测量一种或多种管家生物标志物的表达。具体地,图2c中描绘的植物产品子集202和为植物产品的子集202的每个植物产品202a-d确定的相关的感染生物标志物表达203a-d和管家生物标志物表达204a-d从图2b进行。因此,图2c是系统环境200c的框图,其中为图2a-b的植物产品的子集202确定生物标志物表达统计数据集205。
[0130]
如图2c所示,在一些实施方案中,通过确定植物产品子集202的每个植物产品202a-d的至少一种感染生物标志物的标准化表达水平来确定植物产品子集202的生物标志物表达统计数据集205,然后确定植物产品子集202的每个植物产品202a-d的至少一种感染生物标志物的特征缩放表达水平。具体地,在图2c所示的示例性实施方案中,基于为每个植物产品202a-d确定的感染生物标志物表达203a-d和管家生物标志物表达204a-d,确定植物产品子集202的每个植物产品202a-d的至少一种感染生物标志物的标准化表达水平207a-d。特别地,基于植物产品202a的感染生物标志物表达203a和管家生物标志物表达204a,确定植物产品202a的至少一种感染生物标志物的标准化表达水平207a。类似地,基于植物产品202b的感染生物标志物表达203b和管家生物标志物表达204b,确定植物产品202b的至少一种感染生物标志物的标准化表达水平207b。基于植物产品202c的感染生物标志物表达203c和管家生物标志物表达204c,确定植物产品202c的至少一种感染生物标志物的标准化表达水平207c。最后,基于植物产品202d的感染生物标志物表达203d和管家生物标志物表达204d,确定植物产品202d的至少一种感染生物标志物的标准化表达水平207d。
[0131]
在某些实施方案中,植物产品的感染生物标志物的标准化表达水平可以通过确定植物产品中感染生物标志物的表达水平与植物产品中管家生物标志物的表达水平的比率来确定。例如,在感染生物标志物包含表1所示的pal基因且管家生物标志物包含表2所示的肌动蛋白基因的实施方案中,植物产品的pal基因的标准化表达水平可以包括植物产品中pal基因的表达水平与植物产品中肌动蛋白基因的表达水平的比率。如上所述,通过将感染生物标志物的表达水平标准化至管家生物标志物,可以控制植物产品之间基线代谢活性的变化。
[0132]
在确定植物产品子集202的每个植物产品202a-d的至少一种感染生物标志物的标准化表达水平207a-d后,可以确定植物产品子集202的每个植物产品202a-d的至少一种感染生物标志物的特征缩放表达水平208a-d。具体地,在图2c所示的实施方案中,基于每个植物产品202a-d的至少一种感染生物标志物的标准化表达水平207a-d,确定植物产品子集
202的每个植物产品202a-d的至少一种感染生物标志物的特征缩放表达水平208a-d。特别地,基于植物产品202a的至少一种感染生物标志物的标准化表达207a水平,确定植物产品202a的至少一种感染生物标志物的特征缩放的表达水平208a。类似地,基于植物产品202b的至少一种感染生物标志物的标准化表达水平207b,确定植物产品202b的至少一种感染生物标志物的特征缩放表达水平208b。基于植物产品202c的至少一种感染生物标志物的标准化表达水平207c,确定植物产品202c的至少一种感染生物标志物的特征缩放表达水平208c。最后,基于植物产品202d的至少一种感染生物标志物的标准化表达水平207d,确定植物产品202d的至少一种感染生物标志物的特征缩放表达水平208d。
[0133]
一般而言,植物产品的感染生物标志物的特征缩放表达水平可以例如通过进行植物产品的感染生物标志物的标准化表达水平的最小-最大标准化6和对数转换中的至少一种来确定。
[0134]
最后,如图2c所示,基于每个植物产品202a-d的至少一种感染生物标志物的特征缩放表达水平208a-d,确定植物产品子集202的生物标志物表达统计数据集205。关于生物标志物表达统计数据集205的这种确定的一种实施方案在图2d中被示出和详细讨论。
[0135]
图2d是示例性系统环境200d的框图,其中为植物产品的子集确定生物标志物表达统计数据集。图2d是图2a-c的延伸。具体地,图2d中描绘的植物产品202a-d的至少一种感染生物标志物208a-d的特征缩放表达水平从图2c进行。因此,图2d是系统环境200d的框图,其中为图2a-d的植物产品的子集202确定生物标志物表达统计数据集205。
[0136]
如图2d所示并且如上文关于图2c所讨论的那样,在一些实施方案中,基于每个植物产品202a-d的至少一种感染生物标志物的特征缩放表达水平208a-d,确定植物产品子集202的生物标志物表达统计数据集205。此外,在图2d中描述的实施方案中,为植物产品子集202确定的生物标志物表达统计数据集205包括至少一种感染生物标志物的特征缩放表达水平208a-d的平均值、中位数、最小值、最大值、标准偏差、第5个百分位数、第10个百分位数、第15个百分位数、第20个百分位数、第25个百分位数、第50个百分位数、第75个百分位数、第80个百分位数、第90个百分位数、第95个百分位数和第99个百分位数中的至少一种(图2d中的209),植物产品子集202的每个植物产品202a-d的至少一种感染生物标志物中的至少一种的特征缩放表达水平与至少一种感染生物标志物中的至少另一种的特征缩放表达水平的比率210,和植物产品子集202的每个植物产品202a-d的至少一种感染生物标志物的至少一种的特征缩放表达水平与至少一种感染生物标志物的至少另一种的特征缩放表达水平的乘积210。
[0137]
例如,在图2d的实施方案中,其中感染生物标志物包括表1中所示的pal基因和f3h基因,植物产品子集202的生物标志物表达统计数据集205可以包括pal基因208a-d的特征缩放表达水平的平均值209,f3h基因的特征缩放表达水平208a-d的平均值209,以及植物产品202a的pal基因的特征缩放表达水平208a与f3h基因的特征缩放表达水平208a的比率210,植物产品202b的pal基因的特征缩放表达水平208b与f3h基因的特征缩放表达水平208b的比率210,植物产品202c的pal基因的特征缩放表达水平208c与f3h基因的特征缩放表达水平280c的比率210,以及植物产品202d的pal基因的特征缩放表达水平208d与f3h基因的特征缩放表达水平208d的比率210。
[0138]
在替代实施方案中,植物产品子集202的生物标志物表达统计数据集205可以包括
基于植物产品202a-d的感染生物标志物表达203a-d和管家生物标志物表达204a-d和/或基于植物产品202a-d的至少一种感染生物标志物的特征缩放表达208a-d的任何统计数据。
[0139]
在进一步的实施方案中,植物产品子集202的生物标志物表达统计数据集205可以包括基于植物产品202a-d的感染生物标志物表达203a-d和管家生物标志物表达204a-d和/或基于植物产品202a-d的至少一种感染生物标志物的特征缩放表达208a-d和/或基于使用例如内标或制备的标准曲线(已知浓度)确定的绝对表达值的统计数据的组合。
[0140]
最后,如图2d所示,植物产品子集202的生物标志物表达统计数据集205用于预测相似来源的植物产品的集合201的感染可能性206。感染可能性206的预测将在下文中关于图3-5d详细讨论。
[0141]
viii.其他方法的概述
[0142]
用于预测相似来源的植物产品的集合中感染的可能性的另一种方法1200在图12的流程图中示意性地示出。相似来源的植物产品的集合通常包括足够多的植物产品(例如,大于10个植物产品),以使本文所述的分析方法在统计上有效。尽管显示某些步骤以特定顺序发生,但在某些情况下,该方法的步骤可以以与结合图12描述的顺序不同的顺序执行。此外,在某些情况下,某些步骤可以省略和/或由其他步骤代替,和/或可以添加额外的步骤。
[0143]
为了预测相似来源的植物产品的集合中感染的可能性,如图12所示,从相似来源的植物产品的集合中选择植物产品的子集1201。该子集通常包括至少3个植物产品,并且少于相似来源的植物产品的集合中产品总数的一半。从相似来源的植物产品的集合中选择植物产品子集1201可以遵循与上面参考图1的方法100讨论的相同的过程。然后将子集的植物产品划分成子组,每个子组包含子集的一个或多个植物产品(步骤1202)。对于每个子组,来自该子组的每个植物产品的植物物质然后被组合以形成汇集的植物物质组(步骤1203)。组合来自子组的每个植物产品的植物物质可以例如包括将子组的所有植物产品一起放置在搅拌机中并将它们混合以形成汇集的植物物质组。
[0144]
然后确定每个汇集的植物物质组的至少一种感染生物标志物和任选地至少一种管家生物标志物的表达水平(步骤1204)。用于生物标志物分析的植物物质的制备,以及适用于预测植物产品中感染的可能性的感染生物标志物和管家生物标志物的鉴定,可以遵循与以上参照图1的方法100描述的那些相同的过程。
[0145]
接下来,基于所确定的每个汇集的植物物质组的至少一种感染生物标志物和任选地至少一种管家生物标志物的表达水平,确定汇集的植物物质组的集合的生物标志物表达统计数据集(步骤1205)。上文详细描述的方法100中的用于确定生物标志物表达统计数据集的相同方法和度量也可以与图12的方法1200一起使用。
[0146]
最后,至少部分地基于汇集的植物物质组的集合的确定的生物标志物表达统计数据集来预测相似来源的植物产品的集合中感染的可能性(步骤1206)。相似来源的植物产品的集合中感染的可能性的预测可以遵循上面参考图1的方法100描述的相同过程和度量。然后可以返回相似来源的植物产品的集合中的预测的感染可能性,并且在一些实施方案中,可以利用它来刻意处理相似来源的植物产品的集合。例如,相似来源的植物产品的集合中潜伏感染的预测的可能性可用于确定何时何地销售植物产品的集合。
[0147]
图13是系统环境1300的框图,其中基于图12的方法1200为相似来源的植物产品的集合预测感染的可能性1306。具体地,图13中描绘的植物产品的子集1302对应于在图12的
步骤1201中从相似来源的植物产品的集合中选择的植物产品子集。如图13所示,为了清楚起见,植物产品子集1302的每个单独的植物产品被标记为植物产品1302a、植物产品1302b、植物产品1302c和植物产品1302d之一。尽管图13中的植物产品子集1302显示包括四个植物产品,但该子集可以包括3、4或多于4个植物产品。
[0148]
仍然参考图13,植物产品1302a和1302b然后被分组成第一子组1308a并且植物产品1302c和1302d然后被分组成第二子组1308b,对应于图12中的步骤1202。虽然在图13中每个子组包含相同数量的植物产品,通常子组不必具有相同数量的植物产品,但每个子组应包括子集1302的至少1个植物产品。对于每个子组(例如,1308a和1308b),然后将来自子组中的每个植物产品的植物物质组合以分别形成汇集的植物物质组1310a和1310b,对应于图12中的步骤1203。例如,子组1308a的植物产品1302a和1302b可以放置在搅拌机中并混合以形成汇集的植物物质组1310a,并且子组1308b的植物产品1302c和1302d可以放置在另一个混合机中并混合以形成汇集的植物物质组1310b。
[0149]
接下来,确定每个汇集的植物物质组1310a和1310d的至少一种感染生物标志物的表达水平和任选地至少一种管家生物标志物的表达水平,对应于图12的步骤1204。具体地,针对汇集的植物物质组1310a确定感染生物标志物表达1303a和任选地管家生物标志物表达1304a,并且针对汇集的植物物质组1310b确定感染生物标志物表达1303b和任选地管家生物标志物表达1304b。如上所述,生物标志物的表达可以通过根据任何测量方法测量充当生物标志物表达的替代物的任何物质来确定。例如,当生物标志物包含基因时,该基因的rna转录物的拷贝数可以代表该基因的表达,并且rt-qpcr可用于量化该基因的rna转录物的拷贝数。在一些进一步的实施方案中,可以使用配置为检测生物标志物表达的阵列和/或试剂盒来确定生物标志物表达。因此,通过使用任何合适的测量方法测量充当生物标志物表达的替代物的任何物质来确定汇集的植物物质组1310a和1310b中的每一个的至少一种感染生物标志物和任选地至少一种管家生物标志物的表达水平。
[0150]
在确定汇集的植物物质组1310a-b的感染生物标志物表达1303a-b和任选地管家生物标志物表达1304a-b之后,基于感染生物标志物表达1303a-b和任选地管家生物标志物表达1304a-b确定汇集的植物物质组的集合的生物标志物表达统计数据集1305,对应于图12中的步骤1205。最后,至少部分基于为植物产品子集1302确定的生物标志物表达统计数据集1305,为从中选择子集1302的植物产品的相似来源的植物产品的集合预测感染可能性1306,对应于图12中的步骤1206。
[0151]
ix.感染预测系统
[0152]
如上所述,可以基于从相似来源的植物产品的集合中选择的植物产品子集的生物标志物表达统计数据集使用机器学习感染预测系统来预测相似来源的植物产品的集合中感染的可能性。图3是感染预测系统302的系统环境300的框图,该感染预测系统302被配置为预测相似来源的植物产品的集合中感染的可能性303。如下文关于图4更详细讨论的,感染预测系统302至少部分地包括机器学习感染预测模型。在替代配置中,系统环境300中可以包括不同的和/或附加的组件。
[0153]
如图3所示,感染预测系统302接收从相似来源的植物产品的集合中选择的植物产品子集的生物标志物表达统计数据集301的输入。可以根据上述实施方案之一确定生物标志物表达统计数据集。
[0154]
在一些实施方案中,在将生物标志物表达统计数据集301输入到包括感染预测系统302的感染预测模型中之前,对生物标志物表达统计数据集301进行编码。具体地,在一些实施方案中,生物标志物表达统计数据集301在被输入感染预测系统302之前被编码。在替代实施方案中,感染预测系统302包含编码模块,并且在输入到感染预测系统302之后,但在输入到感染预测系统302的感染预测模型之前通过编码模块编码生物标志物表达统计数据集301。在一些实施方案中,例如,生物标志物表达统计数据集301可以被编码到包括位数组(array of bits)的数据结构中。例如,包含200,000个拷贝的基因的rna转录物的中位特征缩放表达水平的生物标志物表达统计数据集可以在位数组中编码为[110000110101000000]。也可以使用在输入感染预测系统302的感染预测模型之前编码生物标志物表达统计数据集301的替代方法。
[0155]
生物标志物表达统计数据集301的输入由感染预测系统302处理以生成和输出相似来源的植物产品的集合中感染的可能性303。感染预测系统302输出的感染可能性303包括对相似来源的植物产品的集合中感染可能性的预测。在一些实施方案中,感染预测系统302输出的感染可能性包括对相似来源的植物产品的集合中感染可能性的二元预测。例如,感染预测系统302输出的感染可能性可以是“1”,代表相似来源的植物产品的集合被感染的二元预测,或者“0”,代表相似来源的植物产品的集合未受感染的二元预测。
[0156]
在替代实施方案中,感染预测系统302输出的感染可能性包括对相似来源的植物产品的集合的受感染的植物产品分数的预测。例如,感染预测系统302输出的感染可能性可以是0.27,代表植物产品的集合中27%的植物产品被感染的预测。
[0157]
在一些实施方案中,感染预测系统302输出的感染可能性包括对相似来源的植物产品的集合的受感染的阈值分数的可能性(例如,可能性百分比)的预测。例如,感染预测系统302输出的感染可能性可以是0.25(即25%),即植物产品的集合中至少1/5(即20%)的植物产品被感染。
[0158]
在进一步的实施方案中,由感染预测系统302输出的感染可能性可以包括在相似来源的植物产品的集合中感染的可能性的任何其他预测。
[0159]
ix.a.感染预测系统架构
[0160]
接下来转向图4,图4是被配置为预测相似来源的植物产品的集合中感染的可能性的感染预测系统400的架构的框图。感染预测系统400包括训练模块401、数据存储402、数据管理模块403和感染预测模型404。感染预测模型404还包括函数405和参数集406。在其他实施方案中,感染预测系统400可以包括用于各种应用的附加的、更少的或不同的组件。类似地,函数可以以不同于此处描述的方式分布在模块之间。未示出诸如网络接口、安全功能、负载平衡器、故障转移服务器、管理和网络操作控制台等的常规组件,以免混淆系统架构的细节。
[0161]
ix.a.1.训练模块
[0162]
训练模块401基于训练数据集构建感染预测模型404。通常,感染预测模型404包括函数405,该函数405捕获训练数据集中的自变量(例如,生物标志物表达统计数据集)和因变量(例如,感染率)之间的关系,从而使损失函数最小化.
[0163]
为了使用训练数据集构建感染预测模型404,来自训练数据集的每个训练样品i被输入到感染预测模型404中。感染预测模型404处理这些输入,如同模型被常规使用以在相
似来源的植物产品的集合中产生感染的可能性。然而,与感染预测模型404的常规使用不同,在感染预测模型404的训练期间,来自训练数据集的已知的回顾性感染率也被输入到模型中。具体来说,在训练过程中,还将已知对于相似来源的植物产品的集合准确的回顾性感染率输入到模型中。
[0164]
在使用训练数据集中的训练样品i对感染预测模型404进行每次迭代之后,该模型确定相似来源的植物产品的集合中的预测的感染可能性与相似来源的植物产品的集合中实际的回顾性感染率之间的差异。然后,感染预测模型404寻求最小化这种差异。具体而言,该模型试图最小化相似来源的植物产品的集合中预测的感染可能性与相似来源的植物产品的集合中的实际的回顾性感染率之间的差异。
[0165]
为了最小化这种差异,感染预测模型404最小化感染预测模型404的损失函数。损失函数表示训练数据s中的一个或多个训练样品i的因变量u
i∈s
(例如,已知的回顾性感染率)与模型404生成的训练样品i的因变量y
i∈s
(例如,预测的感染可能性)的值之间的差异。简单来说,损失函数表示模型404输出的预测的感染可能性与训练数据集中已知的回顾性感染率之间的差异。本领域技术人员已知有多种损失函数,并且这些损失函数中的任何一种都可以用于生成感染预测模型404。
[0166]
通过最小化关于θ的损失函数,可以确定参数集θ的值。在一些实施方案中,感染预测模型404可以是参数模型,其中参数集θ包括参数406并且在数学上修改函数405以指定自变量(例如,生物标志物表达统计数据集)和因变量(例如,感染率)之间的依赖性。换言之,通过最小化损失函数确定的参数集θ可以包括参数集406并且可以用于修改感染预测模型404的函数405,从而优化感染预测模型404的准确性。通常,最小化损失函数的参数型模型的参数是通过基于梯度的数值优化算法确定的,例如批量梯度算法、随机梯度算法等。备选地,感染预测模型404可以是非参数模型,其中模型结构是从训练数据集确定的并且不严格基于固定的参数集。
[0167]
在感染预测模型404包括参数模型的实施方案中,模型通常可以表示为:
[0168]
y=f(xk;θ)
ꢀꢀꢀꢀ
(1)
[0169]
其中y表示由感染预测模型404确定的相似来源的植物产品的集合中感染的可能性,xk表示相似来源的植物产品的集合的子集的生物标志物表达统计数据,θ表示通过最小化关于θ的损失函数确定的参数集406,并且f(
·
)是函数405。在一些实施方案中,生物标志物表达统计数据xk在被输入到函数f(
·
)之前被组合。在替代实施方案中,生物标志物表达统计数据xk在输入到函数f(
·
)之前不被组合。
[0170]
函数405可以是任何函数。例如,在一些实施方案中,函数405可以包括二元逻辑回归模型、逻辑模型树、随机森林分类器、l2正则化、偏最小二乘法分类、朴素贝叶斯分类器、多变量自适应回归样条、一个或多个神经网络和k最近邻分类之一。
[0171]
在替代实施方案中,感染预测模型404包括生物标志物表达统计数据集xk中的每种生物标志物表达统计数据的不同函数405和不同参数集406。例如,如上所述,生物标志物表达统计数据集xk可以包括至少一种感染生物标志物的特征缩放表达水平或绝对表达水平的平均值、中位数、最小值、最大值、标准偏差、第5个百分位数、第10个百分位数、第15个百分位数、第20个百分位数、第25个百分位数、第50个百分位数、第75个百分位数、第80个百分位数、第90个百分位数、第95个百分位数和第99个百分位数中的多于一种,至少一种感染
生物标志物的特征缩放表达水平与至少一种其他感染生物标志物的特征缩放表达水平的比率,以及至少一种感染生物标志物的特征缩放表达水平和至少一种其他感染生物标志物的特征缩放表达水平的乘积。此外,生物标志物表达统计数据集xk可以包括描述多于一种感染生物标志物的统计数据。在这样的实施方案中,可以为每种生物标志物表达统计数据和/或每种感染生物标志物确定单独的参数集θ。例如,在其中生物标志物表达统计数据集xk包括自变量x1(感染生物标志物a的特征缩放表达水平的第25个百分位数)和x2(感染生物标志物a的特征缩放表达水平与感染生物标志物b的特征缩放表达水平的比率)的实施方案中,可以分别为每个自变量x1和x2确定单独的参数集θ1和θ2。参数集θ1的值是通过最小化关于θ1的损失函数来确定的,而参数集θ2的值是通过最小化关于θ2的损失函数来确定的。参数集θ1然后用于修改第一函数f(x1;θ1),并且参数集θ2用于修改第二函数f(x2;θ2)。最后,这些由不同参数集修改的不同函数可以组合起来,以在相似来源的植物产品的集合中产生感染的可能性。在这样的实施方案中,感染预测模型404可以表示为:
[0172]
y=f(x1;θ1) f(x2;θ2)
ꢀꢀꢀꢀ
(2)
[0173]
其中y表示由感染预测模型404确定的相似来源的植物产品的集合中感染的可能性,x1表示第一自变量(例如,感染生物标志物a的特征缩放表达水平的第25个百分位数),x2表示第二自变量(例如,感染生物标志物a的特征缩放表达水平与感染生物标志物b的特征缩放表达水平的比率),θ1表示通过最小化关于θ1的损失函数确定的第一参数集406,θ2表示通过最小化关于θ2的损失函数确定的第二组参数406,并且f(
·
)是函数405。如上面关于等式1所讨论的,函数f(
·
)可以是任何函数。此外,公式2中表示的f(
·
)函数不需要是相同的函数。
[0174]
当感染预测模型404达到预测准确率的阈值水平时(例如,当损失函数被充分最小化时),该模型准备好用于使用。为了确定感染预测模型404何时已经达到足以使用的预测准确率的阈值水平,可以执行感染预测模型404的验证。感染预测模型404的验证将在下文关于图5c更详细地讨论。
[0175]
一旦感染预测模型404已经被验证为已经达到足以使用的预测准确率的阈值水平,在一些实施方案中,这并不排除模型继续训练。事实上,在优选实施方案中,尽管经过验证,感染预测模型404继续被训练,使得模型的参数集406被连续更新,使得损失函数继续减小并且模型的准确率继续改善。
[0176]
ix.a.2.数据存储
[0177]
在一些实施方案中,数据存储402存储用于如上文关于训练模块401所讨论的训练感染预测模型404的训练数据集。训练数据集包括多个训练样品。来自训练数据集的每个训练样品i与相似来源的植物产品的回顾性集合相关联。具体地,来自训练数据集的每个训练样品i与相似来源的植物产品的回顾性集合相关联,其中实际的已知感染率是已知的。每个训练样品i包括相似来源的植物产品的回顾集合的子集的生物标志物表达统计数据集,以及相似来源的植物产品的回顾集合中的实际已知感染率。在某些实施方案中,如上文关于输入到感染预测模型404中的生物标志物表达统计数据集的编码所讨论的,编码在训练期间输入到感染预测模型404中的相似来源的植物产品的回顾集合的实际已知感染率。
[0178]
在下面关于图5a和5c详细讨论的一些实施方案中,来自训练数据集的一个或多个训练样品可以从训练中保留,并用于验证感染预测模型404。
[0179]
ix.a.3.数据管理模块
[0180]
数据管理模块403生成用于训练感染预测模型404的训练数据集。如上所述,来自训练数据集的每个训练样品i与相似来源的植物产品的回顾集合和相似来源的植物产品的回顾性集合中实际的已知感染率相关联。因此,由数据管理模块403用来生成训练数据集的数据可以源自回顾性数据源。
[0181]
在训练数据集由数据存储402存储的实施方案中,数据管理模块403将生成的训练数据集存储在数据存储402中。在还验证感染预测模型404的实施方案中,数据管理模块403还可以从训练数据集中保留训练样品以用于验证感染预测模型404。
[0182]
ix.a.4.感染预测模型
[0183]
感染预测模型404是机器学习模型,其被配置为接收从相似来源的植物产品的集合中选择的植物产品子集的生物标志物表达统计数据集的输入,并预测相似来源的植物产品的集合中感染的可能性。如上所述,一般而言,感染预测模型404包括由参数集406修改的函数405,以准确地捕捉训练数据集中自变量(例如,生物标志物表达统计数据集)和因变量(例如,感染率)之间的关系。如上所述,在一些实施方案中,函数405包括二元逻辑回归模型、逻辑模型树、随机森林分类器、l2正则化、偏最小二乘法分类、朴素贝叶斯分类器、多变量自适应回归样条、一个或多个神经网络和k最近邻分类之一。
[0184]
在一些实施方案中,感染预测模型404包括被配置为预测相似来源的植物产品的集合中感染的可能性的单个模型。然而,在替代实施方案中,感染预测模型404可以包括多个不同的模型,每个模型被配置为执行特定任务。例如,在一种实施方案中,感染预测模型404可以包括多个模型,每个模型被配置为预测特定类型感染的可能性。
[0185]
x.感染预测系统的训练、验证和使用
[0186]
图5a是其中训练、验证和使用感染预测系统的系统环境500a的框图。图5a包括训练阶段501、验证阶段502、使用阶段503、回顾性数据存储504和前瞻性数据存储505。因此,图5a描绘了如何使用回顾性和前瞻性数据来训练、验证和测试感染预测系统。
[0187]
如上面关于图4所讨论的,在使用感染预测系统之前,对系统进行训练。如图5a所示,使用从回顾性数据源504接收的回顾性数据来完成感染预测系统的训练。回顾性数据源504包含训练数据集,该训练数据集包括如上文关于图4的数据存储402所讨论的训练数据样品。换言之,回顾性数据源504包含描述相似来源的植物产品的集合中的过去感染率的数据。回顾性数据源504中包含的数据可以包括私有数据、公开可用数据、商业可用数据、在使用阶段503使用感染预测模型404进行测试后回收的测试数据和/或任何其他回顾性数据源。
[0188]
在一些实施方案中,在训练之后或结合训练,感染预测系统还可以在验证阶段502中进行验证以确定系统是否已经达到预测准确率的阈值水平并且准备好用于使用。如上面简要讨论的,在验证感染预测系统的实施方案中,来自回顾性数据源504的一个或多个训练样品可以从训练阶段501中保留出来,并用于验证感染预测系统。
[0189]
一旦感染预测系统已经被验证为已经达到足以使用的预测准确率的阈值水平,系统就准备好用于使用阶段503。然而,在一些实施方案中,这并不排除系统的继续训练。事实上,在优选实施方案中,尽管经过验证,感染预测系统仍继续接受训练,从而系统不断更新,并且系统的准确性继续提高。
[0190]
转向使用阶段503,感染预测系统用于预测与从预期数据源505接收的预期数据相关联的相似来源的植物产品的集合中感染的可能性。预期数据源505包含描述待预测感染可能性的相似来源的植物产品的集合的子集的自变量(例如,生物标志物表达统计数据集)的数据。预期数据源505中包含的数据可以包括公开可用数据、商业可用数据、从私人实体(例如,植物产品生产商)接收的数据和/或任何其他预期数据源。
[0191]
在使用阶段503期间使用感染预测系统预测相似来源的植物产品的集合中感染的可能性之后,在使用阶段503期间通过感染预测系统从预期数据源505接收的自变量(例如,生物标志物表达统计数据集)以及相似来源的植物产品的集合中的实际回顾性感染率可以用作回顾性数据来训练或验证系统。换言之,在使用阶段503期间感染预测系统使用的预期数据505可以成为用于分别在训练阶段501或验证阶段502期间训练或验证感染预测系统的回顾性数据504。通过这种方式,感染预测系统可以得到持续的训练和验证。
[0192]
x.a.训练
[0193]
图5b是其中训练感染预测系统508的系统环境500b的框图。如图5b所示,为了训练感染预测系统508,相似来源的植物产品的回顾性集合的子集的生物标志物表达统计数据的回顾性集合506以及相似来源的植物产品的回顾性集合中的实际的回顾性感染率507被输入到感染预测系统508。
[0194]
在将生物标志物表达统计数据的回顾性集合506和实际的回顾性感染率507输入到感染预测系统508中之后,感染预测系统508确定并输出相似来源的植物产品的集合中感染的可能性509,基于生物标志物表达统计数据的回顾性集合506和实际的回顾性感染率507。感染预测系统508输出的相似来源的植物产品的集合中感染的可能性509不基于输入到感染预测系统508中的实际的回顾性感染率507。相反,将由感染预测系统508确定和输出的感染可能性509与实际的回顾性感染率507进行比较。
[0195]
由感染预测系统508确定和输出的感染可能性509与实际的回顾性感染率507的这种比较使得感染预测系统508能够确定优化感染预测系统508的准确性的参数,如上文关于图4详细讨论的。换言之,这种比较使得感染预测系统508能够被训练。
[0196]
x.b.验证
[0197]
如上所述,在一些实施方案中,在训练之后或结合训练,感染预测系统508还可以经历验证以确定系统是否已经达到预测准确率的阈值水平并且准备好用于使用。图5c是验证感染预测系统508的系统环境500c的框图。
[0198]
如上面简要讨论的,在感染预测系统508的训练期间,一个或多个训练样品可以从训练中保留出来并用于验证感染预测系统。具体地,如图5c所示,为了验证感染预测系统508,将包含相似来源的植物产品的回顾集合的子集的生物标志物表达统计数据的回顾集合506的保留训练样品输入感染预测系统508。然而,与训练不同,相似来源的植物产品的集合中的实际回顾性感染率不输入到感染预测系统508中。
[0199]
在将生物标志物表达统计数据的回顾性集合506输入感染预测系统508之后,感染预测系统508基于生物标志物表达统计数据的回顾性集合506确定并输出相似来源的植物产品的集合中感染的可能性509。然后,将感染预测系统508输出的感染可能性509与未输入感染预测系统508的相似来源的植物产品的集合中的实际回顾性感染率507进行比较。
[0200]
感染预测系统508确定和输出的感染可能性509与实际的回顾性感染率507的比较
能够确定感染预测系统508是否已经达到预测准确率的阈值水平。如果基于该比较确定感染预测系统508已经达到预测准确率的阈值水平,则可以认为感染预测系统508已经过验证,并且准备好用于使用。在一些实施方案中,感染预测系统508的验证导致感染预测系统508的训练结束。然而,在替代的优选实施方案中,感染预测系统508的验证不排除感染预测系统508的训练,并且感染预测系统508在其整个使用过程中继续接受训练。
[0201]
在基于比较确定感染预测系统508没有达到预测准确率的阈值水平的实施方案中,可以在使用之前进一步训练感染预测系统508。
[0202]
x.c.使用
[0203]
一旦感染预测系统508已经被验证为已经达到预测准确率的阈值水平,则该系统准备好用于使用。图5d是其中使用感染预测系统508的系统环境500d的框图。如图5d所示并且如上文详细讨论的,为了使用感染预测系统508,将相似来源的植物产品的集合的子集的生物标志物表达统计数据集510输入感染预测系统508。与训练和验证不同,生物标志物表达统计数据集510不是相似来源的植物产品的集合的回顾性数据。相反,生物标志物表达统计数据集510是针对其实际的回顾性感染率尚不清楚的相似来源的植物产品的集合的子集。
[0204]
在将生物标志物表达统计数据集510输入到感染预测系统508中之后,感染预测系统508基于生物标志物表达统计数据集510确定并输出相似来源的植物产品的集合中感染的可能性509。在使用期间,这种感染可能性509不与相似来源的植物产品的集合中的实际回顾性感染率进行比较,因为实际的回顾性感染率尚不知道。相反,基于感染预测系统508的先前训练和验证,假设感染预测系统508输出的感染可能性509足够准确。
[0205]
然而,在一些实施方案中,如上面关于图5a所讨论的,一旦已知相似来源的植物产品的集合中的实际的回顾性感染率,则相似来源植物产品的集合中的实际回顾性感染率和感染预测系统508输出的感染可能性509可以用于训练和/或验证系统。以这种方式,感染预测系统508可以在整个使用过程中被不断地训练和验证。
[0206]
xi.基于感染预测的植物产品处理
[0207]
在相似来源的植物产品的集合中感染的可能性的预测之后,可以以任何形式提供预测的感染可能性。在一些实施方案中,感染的可能性被自动呈现给查看用户(例如,以数字方式显示)。在进一步的实施方案中,感染的可能性可以自动以电子方式存储,自动无线传输到远程系统,和/或通过任何其他方法返回。
[0208]
在一些实施方案中,对其预测感染可能性的相似来源的植物产品的集合可以基于预测的感染可能性进行处理。例如,在一种实施方案中,可以基于预测的感染可能性来鉴定具有高感染风险的相似来源的植物产品的集合。
[0209]
在另一个实施方案中,可以基于预测的感染可能性向相似来源的植物产品的集合提供抗微生物处理或开具抗微生物处理的剂量。具体而言,可以为具有相对较高的预测的感染可能性的植物产品的集合提供或开具抗微生物处理,以避免感染的进展。或者,可以对具有相对较低的预测的感染可能性的植物产品的集合开具低剂量或零剂量的抗微生物处理,从而实现资源优化并可能降低抗微生物药物的总使用量。
[0210]
在一些实施方案中,本公开内容的主题可用于评估运输中的植物,该植物使用车辆从诸如植物存储仓库的第一位置运输到诸如另一个植物存储仓库、其他配送中心或市场
的第二位置。在这样的实施方案中,如果检测到超过阈值水平的感染,则可以生成警报,使车辆重定向到第三位置,该第三位置在地理上比第二位置更靠近车辆的当前位置。在一些实施方案中,这可以包括通知车辆(例如卡车、飞机或船)的驾驶员、飞行员或海军上将驶向第三位置。例如,可以进行这样的重定向,以便可以更快地将一种或多种受感染的植物放入冷藏中,以努力减缓检测到的感染的进展。在一些实施方案中,这可以在潜伏感染导致一种或多种受感染的植物发生腐烂例如茎腐烂之前挽救一种或多种受感染的植物。然而,本公开内容不限于此。相反,在其他实施方案中,第三位置可以是抗微生物处理设施,其使一个或多个受感染的植物能够在允许车辆继续驶向第二位置之前用抗微生物处理进行处理,如上所述。或者,如果确定检测到一种或多种植物的感染低于阈值水平,则不产生重新引导车辆的警报并且允许车辆继续沿着其当前导航路径。
[0211]
在尚未收获相似来源的植物产品的集合的实施方案中,可以基于预测的感染可能性选择性地收获植物产品的集合。例如,在一个实施方案中,植物产品的集合的收获时间可以基于植物产品的集合的感染的预测可能性来确定。在另一示例性实施方案中,可基于植物产品的集合的预测的感染可能性来确定植物产品的集合的收获方法。
[0212]
在另一个实施方案中,可以基于预测的感染可能性来确定相似来源的植物产品的集合的质量保证。例如,可以确定相似来源的植物产品的集合的质量保证,其中预测的感染可能性低于某个阈值。例如,可以确定任何相似来源的植物产品的集合的质量保证,其中预测的感染可能性低于5%、10%、15%、20%或25%。
[0213]
在另一实施方案中,可以基于预测的感染可能性来鉴定相似来源的植物产品的集合的消费者和地理目的地中的至少一个。例如,可以将具有较高的预测的感染可能性的植物产品的集合发送给附近地理目的地的消费者,以避免感染在长距离运输中的进展。类似地,具有相对较高的预测的感染可能性的植物产品的集合可以以相对较低的产品标准发送给消费者。
[0214]
在另一个实施方案中,基于预测的感染可能性,可以对相似来源的植物产品的集合扣除乙烯处理或向相似来源的植物产品的集合提供或开具一定剂量的乙烯处理。具体来说,乙烯处理用于改变植物产品的成熟率。然而,例如,外源乙烯的应用或乙烯阻滞剂/途径抑制剂的应用也可以改变某些植物产品中某些感染的进展。因此,在具有较高的预测的感染可能性的植物产品的集合中,可以避免或替代地提供或适当地开具乙烯处理以防止植物产品中感染进展的加速。
[0215]
在另一实施方案中,可以基于植物产品中感染的预测可能性来鉴定用于相似来源的植物产品的集合的一个或多个储存条件。储存条件可以包括储存温度和/或储存湿度。例如,在具有较高的预测的感染可能性的植物产品的集合中,植物产品的集合可以在低温和/或低湿度下储存以减缓植物产品中的感染进展。
[0216]
在又一实施方案中,可以基于植物产品的集合中感染的预测可能性向相似来源的植物产品的集合提供收获后处理。例如,可以为具有较高的预测的感染可能性的相似来源的植物产品的集合提供收获后处理。收获后处理可以包括apeel处理和任何其他收获后植物产品处理。
[0217]
除了上述感染后预测处理步骤之外,相似来源的植物产品的集合可以基于为植物产品的集合确定的预测的感染可能性进行任何类型的处理。此外,除了提供预测的感染可
能性之外,本文公开的方法还可以包括基于预测的感染可能性向用户提供植物产品处理指令。这些指令可以指导用户执行任何植物产品处理步骤,包括上述任何植物处理步骤。在进一步的实施方案中,可以自动执行基于相似来源的植物产品的集合中预测的感染可能性的植物处理步骤。
[0218]
xii.实施例1
–
预测鳄梨中的胶孢炭疽菌感染
[0219]
以下实施例验证了上文介绍的感染预测方法。更具体地,以下实施例描述了在相似来源的鳄梨的集合中预测胶孢炭疽菌感染的可能性。
[0220]
xii.a.外源感染的鳄梨中感染生物标志物的鉴定
[0221]
如上所述,植物产品中的感染生物标志物包括与未受感染的植物产品相比在受感染的植物产品中差异表达的生物标志物。此外,感染生物标志物可包括已确定在受感染的植物产品中与未受感染的植物产品相比以例如至少0.1倍变化的阈值差异表达的生物标志物。为了鉴定鳄梨中的胶孢炭疽菌感染生物标志物(例如,与未感染胶孢炭疽菌的鳄梨相比,在感染胶孢炭疽菌的鳄梨中差异表达的鳄梨生物标志物),选择pal、gst、comt、wrky75、f3h、pr6、pr5、chib、chia、lox、prbl-2和cat基因用于筛选。选择用于作为感染生物标志物筛选的这些基因在本文中将被称为“候选感染生物标志物”。
[0222]
30个鳄梨中的每一个被外源接种胶孢炭疽菌的100个孢子,并且30个鳄梨中的每一个被外源接种水。用水接种的鳄梨作为实验对照。48小时后,将每个鳄梨去核(core)并提取rna。然后,使用rt-qpcr,确定多个受感染鳄梨中的每个鳄梨和多个对照鳄梨中的每个鳄梨的每个候选感染生物标志物的表达水平。对于多个受感染的鳄梨中的每个鳄梨和多个对照鳄梨中的每个鳄梨,还使用rt-qpcr确定管家生物标志物肌动蛋白基因的表达水平。用于执行qpcr以确定pal和肌动蛋白基因表达水平的pal和肌动蛋白基因探针序列和相关荧光团分别在下表3中描述。用于感染生物标志物acs1和aco1基因的探针序列和相关qpcr荧光团也在下表3中描述。
[0223]
表3:基因探针序列和荧光团
[0224]
基因探针(5
’‑3’
)荧光团palacttcccagaggagaaccaagcaafam肌动蛋白tgaagactggcagtggatgaghexacs1ttgtggagaatttcctggccgagaabyaco1ttgtggagaatttcctggccgagajun
[0225]
基于候选感染生物标志物和肌动蛋白基因的表达水平确定多个受感染鳄梨中的每个鳄梨和多个对照鳄梨中的每个鳄梨中的每个候选感染生物标志物的标准化表达水平,如上面关于图2c所描述的。具体而言,对于每个鳄梨,通过确定鳄梨中候选感染生物标志物的log10表达水平与鳄梨中肌动蛋白基因的log10表达水平的比率来确定每个候选感染生物标志物的标准化表达水平。最后,确定多个受感染鳄梨和多个对照鳄梨的每个候选感染生物标志物的平均标准化表达水平。图6是描绘多个受感染鳄梨和多个对照鳄梨的每个候选感染生物标志物的平均标准化表达水平的图600。
[0226]
通过比较多个受感染鳄梨中候选感染生物标志物的平均标准化表达水平与多个对照鳄梨中候选感染生物标志物的平均标准化表达水平,鉴定了与对照鳄梨相比在受感染鳄梨中差异表达的生物标志物。具体而言,与对照鳄梨相比,每个候选感染生物标志物被确
定为在受感染的鳄梨中表现出至少0.1倍的差异表达(即,每个候选感染生物标志物在受感染的鳄梨中的表达是对照(未感染)鳄梨中的至少1.1倍)。因此,候选感染生物标志物,包括pal、gst、comt、wrky75、f3h、pr6、pr5、chib、chia、lox、prbl-2和cat基因,被确定为鳄梨中真正的感染生物标志物。
[0227]
此外,与对照鳄梨相比,苯丙烷途径(pal和gst)中的两个基因和三个病原体反应基因(pr6,pr5,和chib)被鉴定为在受感染的鳄梨中以至少1倍差异表达(即,这些基因各自在受感染的鳄梨中的表达是对照(未感染)鳄梨中的至少2倍)。换句话说,在鉴定的感染基因中,pal、gst、pr6、pr5和chib基因被确定在早期时间点(接种后24小时或48小时)在鳄梨中表现出至少1倍的优选阈值差异表达。也就是说,在许多情况下,受感染的鳄梨中这些基因的表达的至少1倍(即100%)的增加可以在接种后不到50小时、不到40小时、不到30小时或不到25小时内检测到。
[0228]
与本实施例中鉴定的感染生物标志物一样,可以使用与本实施例中描述的方法相似的方法鉴定其他感染生物标志物并将其列于表1中。类似地,在除鳄梨之外的实例中,可以使用与本实施例中描述的方法类似的方法鉴定表现出在受感染和未受感染的植物产品之间至少0.1倍变化的差异表达的其他感染生物标志物并将其列在表1中。
[0229]
xii.b.内源性感染的鳄梨中的感染生物标志物的鉴定
[0230]
除了鉴定外源性感染的鳄梨中的感染生物标志物外,上文鉴定的感染生物标志物也被证实与内源性感染的鳄梨中的感染相关。具体而言,将上文被鉴定为感染生物标志物的pal基因的表达在具有高内源性感染率的鳄梨中与具有低内源性感染率的鳄梨中进行比较,以验证pal基因表达的增加与鳄梨中的高内源性感染率相关。
[0231]
为了验证pal基因的表达增加与鳄梨中的高内源性感染率相关,在十一周内研究了十一批鳄梨a-k(例如,具有相同包装hue号的相似来源的鳄梨的集合)。具体来说,每周研究一批鳄梨。从每批中,三十个鳄梨被留出用于成熟。一旦成熟,这三十个鳄梨就会被检查是否有表现为茎端腐烂的感染。基于该检查,确定了三十个鳄梨的每个集合的茎端腐烂发生率,并推断出这三十个鳄梨所源自的鳄梨批次。图7a是描绘鳄梨a-k的每个批次中茎端腐烂发生率的图700a。鳄梨a-k的每个批次被进一步分类为具有茎端腐烂的高(例如,大于5%、10%或15%)或低(例如,小于5%、10%或15%)发生率。具有高茎端腐烂发生率(在这种情况下为15%)的鳄梨批次在图7a中由具有条纹填充的条形图描绘。相反,具有低茎端腐烂发生率的鳄梨批次在图7a中由没有图案填充的条形图描绘。
[0232]
除了在成熟后检查来自每个批次a-k的三十个鳄梨的茎端腐烂以外,还在成熟之前从每个批次中去核六个未成熟的鳄梨并提取rna。然后,使用rt-qpcr,确定每个鳄梨的pal基因表达水平。具体而言,针对每个批次a-k的六个鳄梨中的每一个,确定了与pal基因相关的rna转录物的拷贝数。还使用rt-qpcr确定了每个鳄梨的管家生物标志物肌动蛋白基因的表达水平。具体来说,对于每个批次a-k的六个鳄梨中的每一个都确定了与肌动蛋白基因相关的rna转录物的拷贝数。用于执行qpcr以确定pal和肌动蛋白基因表达水平的pal和肌动蛋白基因探针序列和相关荧光团如上表3所示。
[0233]
基于pal基因和肌动蛋白基因的表达水平确定每个鳄梨的pal基因的标准化表达水平。具体地,对于每个鳄梨,通过确定鳄梨中pal基因的表达水平与鳄梨中肌动蛋白基因的表达水平的比率来确定pal基因的标准化表达水平。
[0234]
图7b是图表700b,其比较了从每批鳄梨测试的六个鳄梨中的每个鳄梨中的pal基因的标准化表达水平与鳄梨所源自的鳄梨批次中茎端腐烂的发生率。其中确定了pal基因的标准化表达水平的每个鳄梨在图7b中表示为一个点。
[0235]
如图7b所示,一般而言,源自被归类为具有高茎端腐烂发生率的鳄梨批次的鳄梨中pal基因的标准化表达水平高于源自被归类为具有低茎端腐烂发生率的鳄梨批次的鳄梨中的水平。具体来说,图7b中表示的66个鳄梨中有61个(例如92.4%)表现出茎端腐烂的高发生率和大于1的标准化pal表达水平,或表现出茎端腐烂的低发生率和小于1的标准化pal表达水平。因此,图7b证实了增加的pal基因表达水平与鳄梨中较高的内源性感染率相关。
[0236]
xii.c.鳄梨中的年龄依赖性感染生物标志物
[0237]
在随后的实验中,其结果在图8中描绘,确定了pal基因在鳄梨中随着鳄梨年龄的增长而表达增加。在实验中,对两批不同的鳄梨进行了评估。第一批鳄梨(标记为“mx28”并在图8中用纯灰色表示)具有低感染发生率(5%)。第二批鳄梨(标记为“mx29”并在图8中用方格图案表示)具有高感染发生率(17%)。这些感染发生率是通过如上文关于图7a所述的在批次成熟后检查鳄梨批次的茎端腐烂来确定的。
[0238]
除了在成熟后检查来自两个批次的鳄梨的茎端腐烂以外,还在鳄梨包装(例如收获)后的第7-12天的每一天测试来自每个批次的六个鳄梨的pal基因表达。换言之,在第7-12天(含)的每一天,在包装后,从两个批次的鳄梨mx28和mx29中的每一个中去核六个鳄梨并提取rna。然后,使用rt-qpcr,确定每个鳄梨的pal基因表达水平。具体而言,确定了每个鳄梨的与pal基因相关的rna转录物的拷贝数。还使用rt-qpcr确定了每个鳄梨的管家生物标志物肌动蛋白基因的表达水平。具体而言,确定了每个鳄梨的与肌动蛋白基因相关的rna转录物的拷贝数。用于执行qpcr以确定pal和肌动蛋白基因表达水平的pal和肌动蛋白基因探针序列和相关荧光团如上表3所示。
[0239]
基于pal基因和肌动蛋白基因的表达水平确定每个鳄梨的pal基因的标准化表达水平。具体地,对于每个鳄梨,通过确定鳄梨中pal基因的表达水平与鳄梨中肌动蛋白基因的表达水平的比率来确定pal基因的标准化表达水平。
[0240]
图8是描述对于mx28和mx29批次的鳄梨在包装后第7-12天的每一天测试的六个鳄梨中的每一个中的pal基因的标准化表达水平的图800。如图8所示,对于mx28和mx29批次的鳄梨,pal基因的表达水平在包装后7-12天之间随时间增加。换句话说,随着鳄梨老化,鳄梨中pal基因的表达水平随着时间的推移而增加。然而,也如图8所示,平均而言,与来自mx28批次的鳄梨(例如具有低感染发生率的鳄梨批次)的鳄梨相比,来自mx29批次的鳄梨(例如具有高感染发生率的鳄梨批次)的鳄梨中pal基因的表达水平随时间增加更多。此外,平均而言,与来自mx28批次的鳄梨(例如具有低感染发生率的鳄梨批次)的鳄梨相比,来自mx29批次的鳄梨(例如具有高感染发生率的鳄梨批次)的鳄梨中pal基因的表达水平更早地增加。
[0241]
基于这一观察,与具有低感染发生率的鳄梨相比,具有高感染发生率的成熟鳄梨中一些感染生物标志物(例如pal基因)的表达随着时间的推移更早且更多地增加,为了考虑到植物产品之间感染生物标志物表达的变化,在一些实施方案中,可以基于感染生物标志物的表达水平和另一种感染生物标志物的表达水平来确定感染生物标志物的标准化表达水平。具体而言,在一些实施方案中,对于给定的植物产品,感染生物标志物的标准化表
达水平可以通过计算感染生物标志物的表达水平与另一种感染生物标志物的表达水平的比率和/或感染生物标志物的表达水平和另一种感染生物标志物的表达水平的乘积来确定。通过将感染生物标志物表达标准化至其他感染生物标志物表达,可以控制植物产品之间基线感染生物标志物表达的变化。
[0242]
xii.d.感染预测系统的训练数据集生成的优化
[0243]
为了充分训练上述感染预测系统以准确预测感染的可能性,强健的训练数据集是必要的。为了生成如此强健的训练数据集以充分训练感染预测系统,对生成训练数据集的训练样品的方法进行了优化。特别地,制备用于生物标志物分析的植物产品的方法如前所述进行了优化。
[0244]
用于制备用于生物标志物分析的植物产品的方法的优化使得能够有效地生成用于训练感染预测系统的大型训练数据集。具体而言,通过采用上述优化的植物产品制备方法,可以在6小时内确定植物产品中生物标志物的表达水平,而不是没有使用本文所述的优化植物产品制备方法时的1-2天。通过将确定植物产品中生物标志物表达水平所需的时间从1-2天缩短到6小时,植物产品样品的重新测试可以在一天的时间点进行多次,并且可以使用这种增加的数据(multiplicative data)为感染预测系统快速生成大型训练数据集。
[0245]
例如,由于确定植物产品中的生物标志物表达水平所需的时间缩短,重新测试在第0天的时间点的来自62个批次的384个鳄梨的感染生物标志物表达水平,以关联感染生物标志物在每个鳄梨中的表达与鳄梨所源自的批次中的感染发生率,用于在被配置为预测鳄梨中感染的可能性的感染预测系统的训练数据集中使用。具体来说,使用rt-qpcr测试了第0天时间点的384个鳄梨的pal、肌动蛋白、acs1和aco1基因表达。确定了每个鳄梨的与pal、肌动蛋白、acs1和aco1基因相关的rna转录物的拷贝数。用于执行qpcr的pal、肌动蛋白、acs1和aco1基因探针序列和相关荧光团在上面的表3和表4中进行了描述。
[0246]
对于每个鳄梨,基于pal、acs1和aco1基因和肌动蛋白基因的表达水平确定pal、acs1和aco1基因的标准化表达水平。具体而言,对于每个鳄梨,通过确定鳄梨中pal、acs1和aco1基因的表达水平与鳄梨中肌动蛋白基因的表达水平的比率来确定pal、acs1和aco1基因的标准化表达水平。然后,对于每个鳄梨,通过对pal、acs1和aco1基因的标准化表达水平进行最小-最大标准化6和对数转换来确定pal、acs1和aco1基因的特征缩放表达水平。接下来,通过确定每个鳄梨的pal、acs1和aco1基因的特征缩放表达水平的比率以及每个鳄梨的pal、acs1和aco1基因的特征缩放表达水平的乘积来确定384个测试的鳄梨的生物标志物表达统计数据集。此外,在384个鳄梨的成熟后,确定了每个训练批次的鳄梨的实际感染(表现为茎端腐烂)发生率。
[0247]
384个鳄梨的每一个中的pal、acs1和aco1基因的特征缩放表达水平的比率和乘积以及384个鳄梨所源自的每个批次的实际感染发生率被用于创建训练数据集以训练包含二元逻辑回归模型的感染预测系统,以预测个体鳄梨中感染的可能性。
[0248]
在感染预测系统的训练期间,感染预测系统基于每个鳄梨中pal、acs1和aco1基因的特征缩放表达水平的比率和乘积预测与训练数据集相关联的384个鳄梨的每一个中感染的可能性。然后,对于训练数据集中的每个鳄梨,将感染预测系统确定的预测的感染可能性与鳄梨所源自的批次的实际感染发生率进行比较。训练数据集中每个鳄梨的预测的感染可能性和实际感染发生率的比较在图9中描述,并在下面进一步详细讨论。
[0249]
在使用基于384个鳄梨的训练数据集训练感染预测系统之后,感染预测系统根据与上文所述类似的方法测试12批测试鳄梨中的每个鳄梨。更具体地说,在感染预测系统的训练之后,感染预测系统预测了每个测试鳄梨中感染的可能性。在测试的鳄梨成熟后,确定每个测试批次的鳄梨的实际感染发生率,表现为茎端腐烂。
[0250]
与训练数据集中的鳄梨一样,对于来自测试数据集的每个鳄梨,将由感染预测系统确定的预测的感染可能性与鳄梨所源自的批次的实际感染发生率进行比较。测试数据集中每个鳄梨的预测的感染可能性和实际感染发生率的比较在图9中描述。
[0251]
图9是描绘感染预测系统的预测性能的图表900。在图9中,黑点代表来自训练数据集的鳄梨,灰点代表来自测试数据集的鳄梨。图9将感染预测系统预测的每个鳄梨的感染可能性与鳄梨所源自的鳄梨批次中的实际感染发生率进行比较。通过图9画出的水平虚线表示50%的预测的感染可能性,并且通过图9画出的垂直虚线表示5%的感染发生率阈值。
[0252]
感染预测系统能够以至少85%的准确率预测来自训练数据集的未成熟鳄梨是否属于在成熟时具有大于5%的茎端腐烂发生率的(相似来源的)鳄梨批次。此外,感染预测系统能够以至少85%的准确率预测来自测试数据集中的未成熟鳄梨是否属于在成熟时具有大于5%的茎端腐烂发生率的(相似来源)鳄梨批次。
[0253]
xii.e.感染预测系统优化
[0254]
除了如上所述的用于为感染预测系统生成训练数据集的优化方法之外,感染预测系统本身也通过使用有效的数据流水线训练、验证和测试每个感染预测系统来将各种不同的感染预测系统相互比较来优化。用于训练、验证和测试感染预测系统的数据流水线在本公开内容全文中进行了描述,特别是上文关于图1-5d所述的。此外,图10a-c描绘了用于训练、验证和测试感染预测系统的示例数据流水线的不同粒度。
[0255]
图10a是用于训练、验证和测试感染预测系统的示例性数据流水线的概述1000a的框图。在图10a中描述的实施方案中,相似来源的植物产品(例如,一批鳄梨)的74个集合用于训练、验证和测试感染预测系统。如下文关于图10b更详细讨论的,在图10a所示的实施方案中,选择80%的相似来源的植物产品的集合来训练和验证感染预测系统,而选择20%的相似来源的植物产品的集合来测试感染预测系统。因此,在相似来源的植物产品的74个集合中选择62个集合来训练和验证感染预测系统,在相似来源的植物产品的74个集合中选择12个集合来测试感染预测系统。
[0256]
在用于训练和验证感染预测系统的相似来源的植物产品的62个集合中,相似来源的植物产品的62个集合中的51个集合用于训练感染预测系统,相似来源的植物产品的62个集合中的5个集合用于验证感染预测系统。如图10a所示,感染预测系统的验证作为5折交叉验证来执行,这意味着感染预测系统在总共25折数据上得到验证。
[0257]
在感染预测系统的训练和验证之后,使用如上所述选择的12个测试数据集来测试感染预测系统。如下文关于图10c详细描述的,可以确定感染预测系统在这些测试数据集上的表现并将其用于鉴定表现最佳的感染预测系统以供将来使用。
[0258]
接下来转向图10b,图10b是用于训练、验证和测试感染预测系统的示例性数据流水线1000b的框图,该感染预测系统被配置为预测相似来源的植物产品的集合是否具有大于或小于5%的感染可能性。在图10b中描述的实施方案中,感染预测系统的数据流水线包括一个起始输入和五个不同的步骤。同样在图10b中描述的实施方案中,植物产品包括鳄
梨,并且相似来源的植物产品的集合包括一批鳄梨。然而,在替代的实施方案中,图10b的步骤可以应用于任何替代植物产品。
[0259]
如图10b所示,起始输入包括从一批相似来源的鳄梨获得的鳄梨。起始输入还包括鳄梨中一种或多种感染生物标志物和一种或多种管家生物标志物的表达水平。在图10b中的数据流水线的实施方案中,pal、aco和acs基因是所述一种或多种感染生物标志物,肌动蛋白基因是所述一种或多种管家生物标志物。在一些实施方案中,基因的表达水平可以包括与该基因相关的rna转录物的拷贝数。特别地,如图10b所示,鳄梨中肌动蛋白、pal、aco和acs基因的表达水平包括与鳄梨中的基因相关的rna转录物的拷贝数。
[0260]
感染预测系统的数据流水线的步骤1包括基于感染生物标志物和管家生物标志物的表达水平确定鳄梨中每个感染生物标志物的标准化表达水平。更具体地说,在步骤1中,通过确定鳄梨中与感染生物标志物相关的rna转录物的拷贝数与鳄梨中与管家生物标志物相关的rna转录物的拷贝数的比率来确定鳄梨中每个感染生物标志物的标准化表达水平。因此,如图10b所示,确定了与pal基因相关的rna转录物的拷贝数与与肌动蛋白基因相关的rna转录物的拷贝数的比率(例如,pal/肌动蛋白),确定了与aco基因相关的rna转录物的拷贝数与与肌动蛋白基因相关的rna转录物的拷贝数的比率(例如,aco/肌动蛋白),并确定了与acs基因相关的rna转录物的拷贝数与与肌动蛋白基因相关的rna转录物的拷贝数的比率(例如,acs/肌动蛋白)。
[0261]
接下来,在图10b所示的数据流水线的步骤2中,确定在步骤1中确定的每种感染生物标志物的标准化表达水平的特征缩放表达水平。为了确定感染生物标志物的标准化表达水平的特征缩放表达水平,将数学标准化函数应用于感染生物标志物的标准化表达水平。例如,在某些实施方案中,感染生物标志物的标准化表达水平的特征缩放表达水平通过执行感染生物标志物的标准化表达水平的最小-最大标准化6和对数转换中的至少一种来确定。在步骤2中,针对pal、aco和acs基因的标准化表达水平确定特征缩放表达水平。
[0262]
然后,在步骤3中,已经历上述步骤1-2的个体鳄梨根据它们源自的批次分组在一起。换句话说,来自相似来源的鳄梨的共同集合的个体鳄梨的子集被组合在一起。然后,基于为子集的每个鳄梨确定的每个感染生物标志物的特征缩放表达水平,为每个鳄梨子集确定生物标志物表达统计数据集。在一些实施方案中,鳄梨子集的生物标志物表达统计数据集包括为子集中的鳄梨确定的每个感染生物标志物的特征缩放表达水平的平均值、中值、最小值、最大值、标准偏差、第5个百分位数、第10个百分位数、第15个百分位数、第20个百分位数、第25个百分位数、第50个百分位数、第75个百分位数、第80个百分位数、第90个百分位数、第95个百分位数和第99个百分位数的至少一种,鳄梨子集的每个鳄梨的每个感染生物标志物的特征缩放表达水平与每个其他感染生物标志物的特征缩放表达水平的比率,以及鳄梨子集的每个鳄梨的每个感染生物标志物的特征缩放表达水平和每个其他感染生物标志物的特征缩放表达水平的乘积。
[0263]
最后,在步骤3中,在鳄梨子集所源自的相似来源的鳄梨的集合的成熟之后,检查相似来源的鳄梨集合的表现为茎端腐烂的感染。基于该检查,确定相似来源的鳄梨的每个集合的茎端腐烂的实际发生率,并且将相似来源的鳄梨的每个集合分类为例如具有茎端腐烂的大于5%或小于5%的发生率。
[0264]
接下来转到步骤4,随机选择其生物标志物表达统计数据集和茎端腐烂发生率已
被确定的鳄梨的子集以训练感染预测系统或测试感染预测系统。在图10b所示的实施方案中,选择步骤3中处理的80%的鳄梨子集来训练感染预测系统,而选择步骤3中处理的20%的植物产品子集来测试感染预测系统。
[0265]
最后,在图10b中描绘的数据流水线的步骤5中,使用在步骤4中选择的训练数据集训练感染预测系统。如上文关于图4和5b所详细讨论的,训练感染预测系统涉及优化系统参数以最小化损失函数。除了在步骤5中训练感染预测系统以外,感染预测系统还使用保留的训练数据样品进行验证,如上文关于图5c和10a详细讨论的那样。在步骤5中对感染预测系统进行训练和验证之后,感染预测系统准备好使用在步骤4中选择的测试数据集进行测试,如下面关于图10c所讨论的。
[0266]
转到图10c,图10c是用于测试和评估图10b的感染预测系统的性能的示例性方案1000c的框图。如图10c所示,来自图10b的经过训练和验证的感染预测系统使用在图10b的数据流水线的步骤4中选择的测试数据集如上面关于图5d详细讨论的那样进行测试。具体而言,对于包括在测试数据集中的每个鳄梨子集,在图10b的步骤3中为该子集确定的生物标志物表达统计数据被输入到感染预测系统中。然后,感染预测系统输出鳄梨子集所源自的相似来源的鳄梨集合是否具有大于或小于例如5%的感染可能性的预测。
[0267]
最后,对于测试的每个鳄梨子集,将感染预测系统对鳄梨子集所源自的相似来源鳄梨集合中感染可能性的预测与如在图10b的步骤3中确定的相似来源的鳄梨集合中已知的感染发生率进行比较。基于该比较,可以确定感染预测系统的准确率、精确率和召回率。
[0268]
可以比较不同感染预测系统的性能以选择用于未来感染预测的最佳系统。换言之,上述图10a-c中描述的数据流水线使得能够实现高效且可重复的感染预测系统开发、比较和优化。例如,上述数据流水线促进了各种不同感染预测系统的开发和评估,包括二元逻辑回归模型(blm)、逻辑模型树(lmt)、随机森林分类器(rf)、l2正则化(l2)、朴素贝叶斯分类器(nb)、多变量自适应回归样条(mars)、一个或多个神经网络和k最近邻分类(knn)。下面的表4描述了对于这些不同的感染预测系统中的每一个,关于图10b描述的训练数据集上的平均验证准确率和关于图10a-b描述的测试数据集上的平均准确率、精确率和召回率。
[0269]
表4:感染预测系统的比较
[0270][0271]
如表4所示,最准确的感染预测系统包括基于rf和knn的系统。包括rf和knn感染预
测系统的感染预测系统在使用如上文关于图10c所述的测试数据集进行测试时均达到92%的平均准确率。
[0272]
xiii.示例计算机
[0273]
图11图示了用于实现图1中描述的方法的示例计算机1100。计算机1100包括耦合到芯片组1104的至少一个处理器1102。芯片组1104包括存储控制器集线器1120和输入/输出(i/o)控制器集线器1122。存储器1106和图形适配器1112耦合到存储控制器集线器1120,并且显示器1118耦合到图形适配器1112。存储设备1108、输入设备1114和网络适配器1116耦合到i/o控制器集线器1122。计算机1100的其他实施方案具有不同的架构。
[0274]
存储设备1108是非暂时性计算机可读存储介质,例如硬盘驱动器、光盘只读存储器(cd-rom)、dvd或固态存储设备。存储器1106保存处理器1102使用的指令和数据。输入接口1114是触摸屏接口、鼠标、轨迹球或其他类型的定点设备、键盘或它们的某种组合,并用于输入数据到计算机1100中。在一些实施方案中,计算机1100可以被配置为通过来自用户的手势从输入接口1114接收输入(例如,命令)。图形适配器1112在显示器1118上显示图像和其他信息。网络适配器1116将计算机1100耦合到一个或多个计算机网络。
[0275]
计算机1100适应于执行计算机程序模块以提供本文所述的功能。如本文所用,术语“模块”是指用于提供指定功能的计算机程序逻辑。因此,可以在硬件、固件和/或软件中实现模块。在一种实施方案中,程序模块存储在存储设备1108上,加载到存储器1106中,并由处理器1102执行。
[0276]
用于实现图1的方法的计算机1100的类型可以取决于实体所需的实施和处理能力而有所不同。例如,演示鉴定系统160可以在单个计算机1100或通过诸如服务器群中的网络相互通信的多个计算机1100中运行。计算机1100可能缺少一些上述组件,例如图形适配器1112和显示器1118。
[0277]
xiv.其他注意事项
[0278]
应当注意,如在说明书和所附权利要求中使用的,单数形式“一个”、“一种”和“该”包括复数指示物,除非上下文另有明确说明。
[0279]
出于所有目的,在说明书正文中引用的所有参考文献、授权专利和专利申请均通过引用整体并入本文。
[0280]
出于说明的目的,已经呈现了本公开内容的实施方案的前述描述;它不旨在穷举或将本公开内容限制为所公开的精确形式。相关领域的技术人员可以理解,鉴于上述公开内容,许多修改和变化是可能的。
[0281]
本说明书的一些部分在对信息的操作的算法和符号表示方面描述本公开内容的实施方案。这些算法描述和表示通常被数据处理领域的技术人员用来将他们工作的主要内容有效地传达给本领域的其他技术人员。这些操作虽然在功能上、计算上或逻辑上进行了描述,但被理解为通过计算机程序或等效电路、微代码等来实现。
[0282]
本文中描述的任何步骤、操作或过程可以单独或与其他设备结合地使用一个或多个硬件或软件模块来执行或实现。在一种实现方式中,软件模块用包括包含计算机程序代码的计算机可读非暂时性介质的计算机程序产品实现,该产品可以由计算机处理器执行以执行所描述的任何或所有步骤、操作或过程。
[0283]
实施方案还可以涉及用于执行这里的操作的装置。该装置可以为所需目的专门构
造,和/或它可以包括由存储在计算机中的计算机程序选择性地激活或重新配置的通用计算设备。这样的计算机程序可以存储在可以耦合到计算机系统总线的非暂时性的、有形的计算机可读存储介质或适合存储电子指令的任何类型的介质中。此外,本说明书中提及的任何计算系统可以包括单个处理器或者可以是采用多个处理器设计以增加计算能力的架构。
[0284]
本公开内容的实施方案还可以涉及由本文描述的计算过程产生的产品。这样的产品可以包括由计算过程产生的信息,其中该信息存储在非暂时的、有形的计算机可读存储介质上,并且可以包括计算机程序产品的或本文描述的其他数据组合的任何实施方案。
[0285]
最后,在说明书中使用的语言主要是为了可读性和指导目的而选择的,并且可能不是为了描述或限制本发明的主题而选择的。因此,本公开内容的范围意在不受该详细描述的限制,而是受在基于本文的申请上发布的任何权利要求限制。因此,本公开内容的实施方案的公开旨在说明而非限制本公开内容的范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。