基于多源数据融合和矩阵补全的mirna-疾病关联关系预测方法
技术领域
1.本发明涉及生物信息学领域,具体为一种基于多源数据融合和矩阵补全的mirna-疾病关联关系预测方法。
背景技术:
2.mirna(microrna)是一种内源rna,是由22-24个核苷酸组成的小rna。mirna通过切断靶基因的mrna与靶基因互补结合或者通过抑制靶基因的翻译来完成基因调控功能。近年来,越来越多的研究表明mirna的表达与许多复杂疾病可能有潜在的关系。首次确认mirna是从线虫中发现了lin-4和let-7,自那时起研究者们就不断在推进对mirna的探索。现如今已有大量证据表明mirna参与了人类的一系列生命活动进程,人体细胞的早期发育、增殖分化、死亡都与mirna有很大的关系。
3.早期的研究者们是通过生物实验的方法来探索mirna与疾病的关系,但是由于生物实验成本高、实验周期长等特点,在目前看来日益增长的大数据量的情况来看,生物实验越来越不适用于mirna和疾病关系预测的高效模型。但是经过大量研究者的付出,生物实验的方法已经为探究mirna和疾病的关系做出了巨大的贡献。
4.后来一些研究者们提出了基于网络的方法。例如jiang等人通过观察mirna靶基因的相似性,预测mirna之间的相似性。他们提出了首个超几何分布的计算模型用来对关联关系进行预测,对整个人类mirna组进行了优先排序,来推测潜在的mirna-疾病关联。但是,该计算方法具有较高的假阳性和假阴性,严重影响了该方法的可靠性和鲁棒性。
5.还有一些研究者们提出用基于机器学习的预测方法。例如shi等人使用了一种带有自启动的随机游走方法来识别mirna-疾病关联关系,致力于从蛋白质-蛋白质相互作用网络中探寻mirna靶标和疾病基因之间的关系。通过实验观察到,在同一共调控模块中大部分疾病都属于同一疾病类别,这说明了同一模块的疾病可能具有相似的mirna调控机制。chen等人通过整合mirna的功能相似性和疾病的语义相似性以及mirna和疾病的高斯核相似性来计算关联性得分,从而对二者的关联关系进行预测,也得到了较好的预测结果。但是,这些方法在计算时使用了较为稀疏的关系矩阵进行计算,影响了最终的预测结果。
技术实现要素:
6.本发明的目的在于提供了一种基于多源数据融合和矩阵补全的mirna-疾病关联关系预测方法。
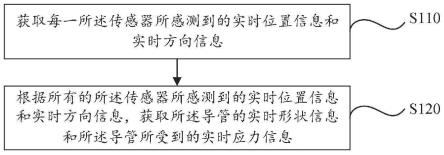
7.实现本发明目的的技术方案为:一种基于多源数据融合和矩阵补全的mirna-疾病关联关系预测方法,具体步骤为:
8.步骤1,从多个数据源中获取mirna-疾病关系、疾病自相关数据和mirna自相关数据;
9.步骤2,根据步骤1中获取的关联数据,计算出疾病语义相似度和mirna功能相似
度;
10.步骤3,根据步骤1中获取的mirna-疾病关系构建原始邻接矩阵,计算mirna高斯相互作用谱核相似度矩阵和疾病高斯相互作用谱核相似度矩阵;
11.步骤4,对步骤2中提取的疾病语义相似度矩阵和mirna功能相似度矩阵进行矩阵补全处理,得到补全后的疾病语义相似度矩阵和补全后的mirna功能相似度矩阵;
12.步骤5,将mirna高斯相互作用谱核相似度矩阵和补全后的mirna功能相似度矩阵整合成新的mirna相似度矩阵;将疾病高斯相互作用谱核相似度矩阵和补全后的疾病语义相似度矩阵整合成新的疾病相似度矩阵,并将整合后的mirna相似度矩阵与疾病相似度矩阵构建成网络;
13.步骤6,在构建的网络上使用标签传播算法,得到最终的mirna-疾病关联关系预测结果。
14.优选地获取hmdd数据库,从中得到经生物实验验证的mirna-疾病关联数据,构建mirna-疾病关系矩阵;获取mesh数据库,从中提取出疾病语义相似度矩阵;获取基因本体数据库,从中提取mirna功能相似度矩阵。
15.优选地,提取疾病语义相似度矩阵的具体方法为:
16.首先用下列公式计算每种疾病d对于其他疾病di的语义贡献值scv。
[0017][0018]
其中,δ为语义贡献因子,本方法中设置δ=0.5。疾病d对其本身的语义贡献值为1;而对于其他疾病di来说,d与di在dag中的距离越远,d对di的语义贡献值就越小。由疾病d和其所有祖先节点疾病的关系可以定义疾病d的语义关系值dv。
[0019]
dv(d)=∑
t∈t(d)
scvd(t)
[0020]
t(d)表示d的所有祖先节点疾病的集合。根据两疾病对其祖先节点的语义贡献,可以计算出两疾病的相似度。
[0021][0022]
根据上式,可以计算出所有疾病之间的语义相似度,从而构建成一个疾病语义相似度矩阵。
[0023]
优选地,高斯相互作用谱核相似度的具体计算方法为:
[0024]
从hmdd中得到了疾病与mirna的关联矩阵dm,dm的第i行定义其为向量ip(d(i)),表示疾病d(i)的相互作用谱。有了疾病d(i)和d(j)的相互作用谱,则可以通过下式可以计算出疾病d(i)和d(j)的高斯核相似度gd,具体计算公式如下。
[0025]
gd(d(i),d(j))=exp(-γd(ip(d(i))-ip(d(j)))2)
[0026]
其中,γd为疾病核带宽参数,计算公式为:
[0027][0028]
δd为疾病带宽系数,nd表示所有疾病的个数;
[0029]
通过比较不同mirna对疾病的关联关系,得到m(i)和m(j)这两种mirna的高斯核相似度gm,具体公式为:
[0030]
gm(m(i),m(j))=exp(-γm(ip(m(i))-ip(m(j)))2)
[0031][0032]
式中,定义的相互作用谱ip(m(i))表示m(i)与每种疾病的关联信息,γm由mirna带宽系数δm与所有mirna的个数nm进行归一化计算得到。
[0033]
优选地,所述的预测方法使用了矩阵补全算法对原始的疾病语义相似度矩阵和mirna功能相似度矩阵进行处理,以弥补数据噪声和数据不完整的问题。
[0034]
矩阵补全算法的过程如下:
[0035]
(1)首先定义因子分解矩阵u和v。u是以1/k为标准差的m*k的正态分布矩阵,v是以1/k为标准差的n*k的正态分布矩阵,其中m和n分别为原始矩阵的长和宽,k为秩参数。
[0036]
(2)初始化偏差u,v和b。u和v分别是长度为m和n的零向量,b为原始矩阵的所有元素的均值。
[0037]
(3)随机打乱样本,对原矩阵进行随机梯度下降的分解运算。
[0038]
在迭代中,对于每一组i(1≤i≤m)和j(1≤j≤n),可以根据下式进行计算。
[0039][0040]
其中,p表示预测值,ui表示u向量的第i位,vj表示v向量的第j位,u
ivjt
表示u矩阵的第i行与v矩阵的第j行的矩阵乘积。
[0041]
(4)更新偏差u、v和因子分解矩阵u、v,可以根据下列式子进行偏差的更新。
[0042]ut 1
=u
t
α(2e-βu
t
)
[0043]vt 1
=v
t
α(2e-βv
t
)
[0044][0045][0046]
其中,α为学习率,β为正则化系数,本方法中均设置为0.1。e表示误差,可由原始矩阵元素的均值和预测值p相减得到。t和t 1表示迭代次数。
[0047]
(5)重复步骤(3)和(4)直到分解矩阵u和v收敛,计算补全后的矩阵x’。
[0048]
x'=u
·vt
[0049]
通过上述基于随机梯度下降的矩阵补全方法,对mirna功能相似度矩阵和疾病语义相似度矩阵这两个稀疏矩阵进行矩阵补全,得到补全后的mirna矩阵和补全后的疾病矩阵。
[0050]
随后将疾病高斯相互作用谱核相似度矩阵、疾病语义相似度矩阵和补全后的疾病相似度矩阵进行整合,得到疾病整合相似度矩阵dis;将mirna高斯相互作用谱核相似度矩阵、mirna语义相似度矩阵和补全后的mirna相似度矩阵进行整合,得到mirna整合相似度矩阵mis。
[0051][0052][0053]
其中,dg和mg分别表示疾病和mirna高斯相互作用谱核相似度矩阵,dc和mc分别表示疾病和mirna补全后的相似度矩阵,dss和mfs表示疾病和mirna原始的相似度矩阵。整合后的fis和mis用来推断疾病与mirna的关联关系。
[0054]
优选地,在整合后的mirna相似度矩阵和疾病相似度矩阵上分别使用标签传播方法来预测二者的关联,标签传播的计算公式如下:
[0055]zt 1
=α
×
wz
t
(1-α)
×y[0056]
其中t表示迭代次数,z
t 1
表示标签传播在t 1步后的迭代结果,α为一个(0,1)范围内的参数,y是一个0和1组成的二进制矩阵,表示每个类的标签信息,经过不断的迭代,最终收敛到一个最优解。
[0057]
使用标签传播公式来更新每个数据对象直至其收敛,通过下式从疾病空间和mirna空间得到预测的关联关系:
[0058]
pd
t 1
=α
×
dis
×
pd
t
(1-α)
×r[0059]
pm
t 1
=α
×
mis
×
pm
t
(1-α)
×rt
[0060]
其中pd和pm分别表示疾病空间的预测结果和mirna空间的预测结果,指数t和t 1表示迭代次数,pd和pm在t=0时是零矩阵。α为一个(0,1)范围内的参数,用来表示邻居信息的权重,(1-α)就可以表示接受初始标签信息的概率。dis和mis表示整合疾病相似度矩阵和整合mirna相似度矩阵。r表示原始的疾病和mirna的关联矩阵。
[0061]
不断迭代当满足|pd
t 1-pd
t
|的误差小于10-6
时迭代完成,最终整合疾病空间和mirna空间的预测结果,可以得到最终的预测结果f。
[0062]
f=β
×
pd (1-β)
×
pm
t
[0063]
其中β表示疾病空间的预测结果和mirna空间的预测结果的占比,将其设置为0.5。
[0064]
本发明与现有技术相比,其显著优点为:
[0065]
本发明使用了多源数据融合的方法,原始数据源更多,生物信息更加全面;并且在使用多个关系矩阵整合时,使用了基于随机梯度下降的矩阵补全算法对稀疏矩阵进行处理,挖掘出一些稀疏矩阵中的隐藏信息,随后将补全后的矩阵进行整合,在整合的矩阵空间中使用随机游走算法得到最终的预测得分结果;本发明较于传统的生物实验的方法,成本低、实验周期短;较于过去的基于机器学习的预测方法,有准确率高、时间复杂度低等优点;本发明提供的这一新的mirna-疾病的预测方法,可以为发掘复杂疾病的发病机理和相关药物的研发提供支持。
[0066]
下面结合附图对本发明作进一步详细描述。
附图说明
[0067]
图1为本发明基于多源数据融合和矩阵补全的mirna-疾病关联关系预测方法的流程图。
[0068]
图2为本发明使用的方法与不使用矩阵补全的方法在全局留一交叉验证下的roc曲线对比图。
[0069]
图3为本发明使用的方法与不使用矩阵补全的方法在五折交叉验证下的roc曲线对比图。
具体实施方式
[0070]
下面对照附图,对本发明的具体实施方式作进一步详细的说明:
[0071]
如图1所示,本发明提出了一种基于多源数据融合和矩阵补全的mirna-疾病关联关系预测方法来预测mirna与疾病的关联关系,首先通过多源数据集获取与mirna和疾病相关的数据并构建多个相似性矩阵;接着构建了mirna高斯相互作用谱相似度矩阵和疾病高斯相互作用谱相似度矩阵;然后使用了随机梯度下降法来进行稀疏矩阵的补全;随后把mirna的多个相似性矩阵进行整合得到mirna空间的相似度关系,把疾病的多个相似性矩阵进行整合得到疾病空间的相似度关系;最后在整合的mirna空间和整合的疾病空间以及原始的mirna与疾病的关联矩阵上使用标签传播算法对mirna-疾病的关联关系进行最终的预测,得到最终预测的评分结果。具体包括以下步骤:
[0072]
步骤1,获取原始mirna-疾病关系矩阵。
[0073]
从hmdd数据库中获取经生物实验验证的mirna-疾病关联数据,构建了原始的mirna和疾病的关联矩阵,是一个由495种mirna和383种疾病构成的邻接矩阵。若某mirna和某疾病有关联关系,则矩阵中对应的位置为1;否则为0,构建的矩阵中共有经生物实验验证的5430对关联关系。
[0074]
步骤2,计算疾病语义相似度矩阵、mirna功能相似度矩阵。
[0075]
从mesh数据库中获取由生物术语构建的有向无环图dag,在dag中获取疾病之间的语义相似度,dag中距离越远的术语之间的语义相似度越小,两疾病d和di的语义贡献值scv可由下式计算:
[0076][0077]
其中,δ为语义贡献因子。由疾病d和其祖先疾病的关系可以定义疾病d的语义关系dv。
[0078]
dv(d)=∑
t∈t(d)
scvd(t)
[0079]
t(d)表示d的所有祖先节点的集合。根据两疾病对其祖先节点的语义贡献,可以计算出两疾病的相似度。
[0080][0081]
根据上式,计算出所有疾病之间的语义相似性,从而构建成一个疾病语义相似性矩阵。
[0082]
两个mirna对应的靶基因集合如果有重叠的现象,认为这两个mirna在功能上有相似性。基于功能相似度的mirna往往作用于类似疾病,有学者设计了misim方法来计算mirna的功能相似性,使用misim方法获取mirna功能相似度矩阵。
[0083]
步骤3,根据原始的mirna-疾病的关联矩阵,计算出疾病高斯相互作用谱核相似度和mirna高斯相互作用谱核相似度。
[0084]
得到的疾病与mirna的关联矩阵是一个由383种疾病和495种mirna构成的二进制矩阵,将该矩阵的第i行看做是疾病di的相互作用谱,根据两疾病的相互作用谱的对比可以计算出两疾病的高斯核相似度gd。
[0085]
gd(d(i),d(j))=exp(-γdip(d(i))-ip(d(j))2)
[0086]
其中,γd为疾病核带宽参数,可以通过下式来计算。
[0087][0088]
δd为疾病带宽系数,nd表示所有疾病的个数,通过归一化计算得到和带宽参数γd。
[0089]
与疾病的高斯相互作用谱核相似度类似,通过比较不同mirna对疾病的关联关系,可以得到m(i)和m(j)的高斯核相似度gm。
[0090]
gm(m(i),m(j))=exp(-γmip(m(i))-ip(m(j))2)
[0091][0092]
上式中定义的相互作用谱ip(m(i))表示m(i)与每种疾病的关联信息,γm由mirna带宽系数δm与所有mirna的个数进行归一化计算得到。
[0093]
步骤4,mirna功能相似度和疾病语义相似度是较为稀疏的矩阵,有很多隐藏信息未被挖掘出来,所以采用了随机梯度下降的方法对原稀疏矩阵进行矩阵补全处理,用来消除数据不完整带来的问题以及能提高预测的准确率。
[0094]
矩阵补全算法的过程如下:
[0095]
首先定义因子分解矩阵u和v。u是以1/k为标准差的m*k的正态分布矩阵,v是以1/k为标准差的n*k的正态分布矩阵,其中m和n分别为原始矩阵的长和宽,k为秩参数。
[0096]
随后进行偏差u,v和b的初始化操作。u和v分别是长度为m和n的零向量,b为原始矩阵的所有元素的均值。
[0097]
随机打乱样本,对原矩阵进行随机梯度下降的分解运算。
[0098]
在迭代中,对于每一组i(1≤i≤m)和j(1≤j≤n),可以根据下式进行计算。
[0099][0100]
其中,p表示预测值,ui表示u向量的第i位,vj表示v向量的第j位,u
ivjt
表示u矩阵的第i行与v矩阵的第j行的矩阵乘积。
[0101]
更新偏差u、v和因子分解矩阵u、v,可以根据下列式子进行偏差的更新。
[0102]ut 1
=u
t
α(2e-βu
t
)
[0103]vt 1
=v
t
α(2e-βv
t
)
[0104][0105][0106]
其中,α为学习率,β为正则化系数,本方法中均设置为0.1。e表示误差,可由原始矩阵元素的均值和预测值p相减得到。t和t 1表示迭代次数。
[0107]
重复上述计算预测值和更新偏差的操作,当误差小于e或者迭代次数大于iterations时停止迭代,得到补全后的矩阵x’。
[0108]
x'=u
·vt
[0109]
在mirna功能相似度矩阵上进行矩阵补全得到补全后的矩阵mc,在疾病语义相似度矩阵上进行矩阵补全得到补全后的矩阵dc。
[0110]
步骤5,将疾病相似度和mirna相似度进行整合,得到疾病整合相似度矩阵dis和mirna整合相似度矩阵mis。
[0111][0112][0113]
其中,dg和mg分别表示疾病和mirna高斯相互作用谱核相似度矩阵,dc和mc分别表示疾病和mirna补全后的相似度矩阵,dss和mfs表示疾病和mirna原始的相似度矩阵。整合后的fis和mis用来推断疾病与mirna的关联关系。
[0114]
步骤6,在整合完毕的疾病空间和mirna空间中使用标签传播算法对mirna和疾病的关系进行预测。
[0115]zt 1
=α
×
wz
t
(1-α)
×y[0116]
其中t表示迭代次数,z
t 1
表示标签传播在t 1步后的迭代结果,α为一个(0,1)范围内的参数,y是一个0和1组成的二进制矩阵,表示每个类的标签信息,经过不断的迭代,最终收敛到一个最优解。
[0117]
利用标签传播算法得到疾病空间的预测迭代公式为:
[0118]
pd
t 1
=α
×
dis
×
pd
t
(1-α)
×r[0119]
mirna空间的预测迭代公式为:
[0120]
pm
t 1
=α
×
mis
×
pm
t
(1-α)
×rt
[0121]
其中pd和pm分别表示疾病空间的预测结果和mirna空间的预测结果,指数t和t 1表示迭代次数,pd和pm在t=0时是零矩阵。α为一个(0,1)范围内的参数,用来表示邻居信息的权重,(1-α)就可以表示接受初始标签信息的概率。dis和mis表示整合疾病相似度矩阵和整合mirna相似度矩阵。r表示原始的疾病和mirna的关联矩阵。
[0122]
不断迭代当满足|pd
t 1-pd
t
|的误差小于10-6
时迭代完成,最终整合疾病空间和mirna空间的预测结果,可以得到最终的预测结果f。
[0123]
f=β
×
pd (1-β)
×
pm
t
[0124]
其中β表示疾病空间的预测结果和mirna空间的预测结果的占比,将其设置为0.5。
[0125]
在预测结果f中,同一列数据为同一mirna与不同疾病之间的预测得分,将预测结果进行排名,得分越高说明该mirna与该疾病越存在关联关系。
[0126]
根据的方法对mirna和疾病的关联关系进行预测,通过使用全局留一疾病交叉验证和五折交叉验证记录了在不同阈值下的tpr和fpr的关系,绘制出roc工作特征曲线。其中,测试的mirna和疾病的关系对在排名中高于给定阈值认为这样的关系为真,tpr指的是真阳性率,表示高于给定阈值的关系对所占的百分比;fpr指的是假阳性率,表示低于阈值的关系对所占的百分比。roc曲线的横轴表示假阳性率,纵轴表示真阳性率,roc曲线下的面积为auc,auc值越接近1表示本方法的性能越好,auc值越接近0.5表示方法具有随机性能。结果表明,在全局留一交叉验证中,的方法与不使用矩阵补全的方法、glnmda方法、pbmda方法的auc值分别为0.9347、0.9119、0.9291、0.9218;而在五折交叉验证中,的方法与不使用矩阵补全的方法、glnmda方法、pbmda方法的auc值分别为0.9351、0.9186、0.9264、0.9187。可以看出的方法比起其他的方法的预测性能更高,本发明对mirna和疾病的关联关系的预测比其他方法更可靠,可以为发掘复杂疾病的发病机理提供支持。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。