新型crispr dna靶向酶及系统
1.相关申请
2.本技术要求于2019年9月9日提交的美国临时申请62/897,859的优先权,该临时申请的全部内容通过援引特此并入。
3.序列表
4.本技术含有已以ascii格式以电子方式提交且特此通过援引以其全文并入的序列表。创建于2020年9月9日的所述ascii副本被命名为a2186-7028wo_sl.txt,并且大小是475,511字节。
技术领域
5.本披露涉及用于使用新型成簇规律间隔短回文重复序列(crispr)和crispr相关(cas)基因来基因组编辑和调节基因表达的系统和方法。
背景技术:
6.基因组测序技术和分析最近进展对自然界许多不同领域的生物活动(范围从原核生物合成途径到人类病理)的遗传基础产生了重要的见解。为了充分理解和评价所产生的巨量信息,需要在基因组和表观基因组操纵的序列技术的规模、效率和易用性方面进行相应的提高。这些新型技术将加速许多领域(包括生物技术、农业和人类治疗学)的新型应用的发展。
7.成簇规律间隔短回文重复序列(crispr)和crispr相关(cas)基因(统称为crispr-cas或crispr/cas系统)是古细菌和细菌中针对外来遗传元件而防御特定物种的适应性免疫系统。crispr-cas系统包括一组极其多样的蛋白质效应子、非编码元件以及基因座架构,其中一些实例已经被工程化并且适于产生重要的生物技术进展。
8.参与宿主防御的系统的组分包括一种或多种能够修饰核酸的效应子蛋白和负责将一种或多种效应子蛋白靶向至噬菌体核酸上的特定序列的rna指导元件。rna指导物由crispr rna(crrna)构成,并且可能需要另外的反式激活rna(tracrrna)以使得能够通过一种或多种效应子蛋白进行靶向核酸操纵。crrna由负责蛋白质与crrna结合的直接重复和与所期望核酸靶序列互补的间隔子序列组成。crispr系统可以通过修饰crrna的间隔子序列来重新编程以靶向替代性dna或rna靶标。
9.crispr-cas系统可以广义地分为两类:1类系统由多种效应子蛋白构成,该多种效应子蛋白一起围绕crrna形成复合物,并且2类系统由一种效应子蛋白构成,该一种效应子蛋白与rna指导物复合以靶向核酸底物。2类系统的单亚基效应子组成为工程化和应用转换提供了更简单的组分集合,并且迄今为止一直是可编程效应子的重要来源。然而,除了当前的通过其独特特性实现了新型应用的crispr-cas系统(诸如较小效应子和/或具有独特pam序列要求的效应子)之外,仍然需要用于修饰核酸和多核苷酸(即,dna、rna或任何杂合体、衍生物或修饰物)的另外的可编程效应子和系统。
技术实现要素:
10.本披露提供了用于新型单效应子2类crispr-cas系统的非天然存在的工程化系统和组合物,首先将这些crispr-cas系统从基因组数据库中计算鉴定出且随后对其进行工程化和实验验证。特别地,对这些crispr-cas系统的组分的鉴定允许其在非天然环境中,例如在除这些系统最初发现于其中的那些之外的细菌中或在真核细胞(诸如哺乳动物细胞)中使用。这些新的效应子在序列和功能上与现有2类crispr效应子的直系同源物和同源物相比是不同的。
11.在一方面,本披露提供了工程化的非天然存在的clust.091979成簇规律间隔短回文重复序列(crispr)-cas系统,这些系统包括:crispr相关蛋白,其中该crispr相关蛋白包含与seq id no:1-56中任一个中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列;以及rna指导物,该rna指导物包含直接重复序列和能够与靶核酸杂交的间隔子序列;其中该crispr相关蛋白能够结合该rna指导物并且能够修饰与该间隔子序列互补的该靶核酸序列。在一方面,本披露提供了工程化的非天然存在的clust.091979成簇规律间隔短回文重复序列(crispr)-cas系统,这些系统包括:crispr相关蛋白或编码该crispr相关蛋白的核酸,其中该crispr相关蛋白包含与seq id no:1-56中任一个中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列;以及rna指导物或编码该rna指导物的核酸,该rna指导物包含直接重复序列和能够与靶核酸杂交的间隔子序列;其中该crispr相关蛋白能够结合该rna指导物并且能够修饰与该间隔子序列互补的该靶核酸序列。
12.在一方面,本披露提供了一种工程化的非天然存在的clust.091979成簇规律间隔短回文重复序列(crispr)-cas系统,该系统包括crispr相关蛋白或编码该crispr相关蛋白的核酸,其中该crispr相关蛋白包含seq id no:241的氨基酸序列;以及rna指导物,该rna指导物包含直接重复序列和能够与靶核酸杂交的间隔子序列;其中该crispr相关蛋白能够结合该rna指导物并且能够修饰与该间隔子序列互补的该靶核酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:4、seq id no:10、seq id no:12或seq id no:14中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列。
13.在本文所述的任一系统的一些实施例中,该crispr相关蛋白包含至少一个(例如,一个、两个或三个)ruvc结构域或至少一个拆分型ruvc结构域。
14.在本文所述的任一系统的一些实施例中,该crispr相关蛋白包含以下序列中的一种或多种:(a)px1x2x3x4f(seq id no:216),其中x1是l或m或i或c或f,x2是y或w或f,x3是k或t或c或r或w或y或h或v,并且x4是i或l或m;(b)rx1x2x3l(seq id no:217),其中x1是i或l或m或y或t或f,x2是r或q或k或e或s或t,并且x3是l或i或t或c或m或k;(c)nx1yx2(seq id no:218),其中x1是i或l或f并且x2是k或r或v或e;(d)kx1x2x3fax4x5kd(seq id no:219),其中x1是t或i或n或a或s或f或v,x2是i或v或l或s,x3是h或s或g或r,x4是d或s或e,并且x5是i或v或m或t或n;(e)lx1nx2(seq id no:220),其中x1是g或s或c或t并且x2是n或y或k或s;(f)px1x2x3x4sqx5ds(seq id no:221),其中x1是s或p或a,x2是y或s或a或p或e或y或q或n,x3是f
或y或h,x4是t或s,并且x5是m或t或i;(g)kx1x2vrx3x4qex5h(seq id no:222),其中x1是n或k或w或r或e或t或y,x2是m或r或l或s或k或v或e或t或i或d,x3是l或r或h或p或t或k或q或p或s或a,x4是g或q或n或r或k或e或i或t或s或c,并且x5是r或w或y或k或t或f或s或q;以及(h)x1ngx2x3x4dx5nx6x7x8n(seq id no:223),其中x1是i或k或v或l,x2是l或m,x3是n或h或p,x4是a或s或c,x5是v或y或i或f或t或n,x6是a或s,x7是s或a或p,并且x8是m或c或l或r或n或s或k或l。在本文所述的任一系统的一些实施例中,seq id no:216的序列是n末端序列。在本文所述的任一系统的一些实施例中,seq id no:219的序列是c末端序列。在本文所述的任一系统的一些实施例中,seq id no:220的序列是c末端序列。在本文所述的任一系统的一些实施例中,seq id no:221的序列是c末端序列。在本文所述的任一系统的一些实施例中,seq id no:222的序列是c末端序列。在本文所述的任一系统的一些实施例中,seq id no:223的序列是c末端序列。
15.在本文所述的任一系统的一些实施例中,该crispr相关蛋白包含以下序列中的一种或多种:(a)ecpitkdvineyk(seq id no:290);(b)nltsitig(seq id no:231);(c)nyrtkirtln(seq id no:232);(d)isyienven(seq id no:233);(e)ellsveqlk(seq id no:234);(f)hinsmtiniqdfkie(seq id no:235);(g)kenslgfil(seq id no:236);(h)gnrqikkg(seq id no:237);(i)dvnfkha(seq id no:238);(j)gyinlykylleh(seq id no:239);(k)keqvlsklly(seq id no:240);(l)eyiyvscvnklrakyvsyfilkekyyekqkeydiemgf(seq id no:241);(m)ddsteskesmdkrr(seq id no:242);(n)nvqqdingclkniiny(seq id no:243);(o)alenlensnfek(seq id no:244);(p)qvlptiksll(seq id no:245);(q)yhklenqn(seq id no:246);(r)asdkvkeyie(seq id no:247);(s)tnenneivdakyt(seq id no:248);(t)anffnlmmkslhfas(seq id no:249);(u)llsnngktqialvpse(seq id no:250);(v)hinglnadfnaannikyi(seq id no:251);或相对于任一前述项具有不超过1、2或3个序列差异(例如,取代)的序列。在一些实施例中,该crispr相关蛋白具有与seq id no:4至少70%相同的序列。在一些实施例中,该crispr相关蛋白具有与seq id no:10至少70%相同的序列。
16.在本文所述的任一系统的一些实施例中,该直接重复序列包含与seq id no:57-90、seq id no:118-151或seq id no:213中任一个中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一系统的一些实施例中,该直接重复序列包含与seq id no:57-90、seq id no:118-151或seq id no:213中任一个中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。
17.在本文所述的任一系统的一些实施例中,该直接重复序列包含以下序列中的一种或多种:(a)x1x2tx3x4x5x6x7x8(seq id no:224),其中x1是a或c或g,x2是t或c或a,x3是t或g或a,x4是t或g,x5是t或g或a,x6是g或t或a,x7是t或g或a,并且x8是a或g或t(例如,attgttgda(seq id no:225));(b)x1x2x3x4x5x6x7x8x9(seq id no:226),其中x1是t或c或a,x2是t或a或g,x3是t或c或a,x4是t或a,x5是t或a或g,x6是t或a,x7是a或t,x8是a或g或c或t,并且x9是g或a或c(例如,ttttwtarg(seq id no:227));以及(c)x1x2x3ac(seq id no:228),其中x1是a或c或g,x2是c或a,并且x3是a或c(例如,acaac(seq id no:229))。在本文所述的任一系统的一
些实施例中,seq id no:224在该直接重复的5’端的近侧。在本文所述的任一系统的一些实施例中,seq id no:228在该直接重复的3’端的近侧。
18.在本文所述的任一系统的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam),其中该pam包括核酸序列,包括作为5
’‑
nttn-3’、5
’‑
nttr-3’、5
’‑
rttr-3’、5
’‑
tnnt-3’、5
’‑
tnrt-3’、5
’‑
tsrt-3’、5
’‑
tgrt-3’、5
’‑
tnry-3’、5
’‑
ttnr-3’、5
’‑
ttyr-3’、5
’‑
tttr-3’、5
’‑
ttcv-3’、5
’‑
dtyr-3’、5
’‑
wttr-3’、5
’‑
nnr-3’、5
’‑
nyr-3’、5
’‑
yyr-3’、5
’‑
tyr-3’、5
’‑
ttn-3’、5
’‑
ttr-3’、5
’‑
cnt-3’、5
’‑
ngg-3’、5
’‑
bgg-3’或5
’‑
r-3’列出的核酸序列,其中“n”是任何核苷酸,“b”是c或g或t,“d”是a或g或t,“r”是a或g,“s”是g或c,“v”是a或c或g,“w”是a或t,并且“y”是c或t。
19.在本文所述的任一系统的一些实施例中,该crispr相关蛋白是与seq id no:1中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:57中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一系统的一些实施例中,该crispr相关蛋白是与seq id no:1中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:57中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一系统的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
tnnt-3’或5
’‑
tnrt-3’列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
20.在本文所述的任一系统的一些实施例中,该crispr相关蛋白是与seq id no:4中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:60中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一系统的一些实施例中,该crispr相关蛋白是与seq id no:4中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:60中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一系统的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
nttn-3’、5
’‑
nttr-3’(例如,5
’‑
tttg-3’)或5
’‑
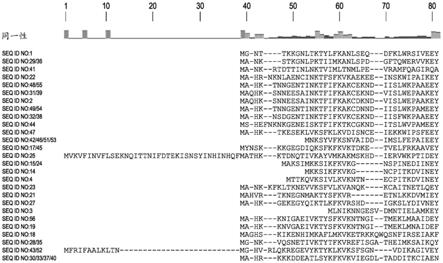
nnr-3’列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
21.在本文所述的任一系统的一些实施例中,该crispr相关蛋白是与seq id no:10中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:62或seq id no:213中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一系统的
一些实施例中,该crispr相关蛋白是与seq id no:10中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:62或seq id no:213中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一系统的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
nttn-3’或5
’‑
rttr-3’(例如,5
’‑
attg-3’或5
’‑
gtta-3’)列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
22.在本文所述的任一系统的一些实施例中,该rna指导物的该间隔子序列包含约15个核苷酸至约55个核苷酸。在本文所述的任一系统的一些实施例中,该rna指导物的该间隔子序列包含20至45个核苷酸。
23.在本文所述的任一系统的一些实施例中,该crispr相关蛋白包含催化残基(例如,天冬氨酸或谷氨酸)。在本文所述的任一系统的一些实施例中,该crispr相关蛋白切割该靶核酸。在本文所述的任一系统的一些实施例中,该crispr相关蛋白进一步包含肽标签、荧光蛋白、碱基编辑结构域、dna甲基化结构域、组蛋白残基修饰结构域、定位因子、转录修饰因子、光门控因子、化学诱导型因子或染色质可视化因子。
24.在本文所述的任一系统的一些实施例中,编码该crispr相关蛋白的该核酸经密码子优化以在细胞,例如真核细胞,例如哺乳动物细胞,例如人细胞中表达。在本文所述的任一系统的一些实施例中,编码该crispr相关蛋白的该核酸可操作地连接至启动子。在本文所述的任一系统的一些实施例中,编码该crispr相关蛋白的该核酸在载体中。在一些实施例中,该载体包括逆转录病毒载体、慢病毒载体、噬菌体载体、腺病毒载体、腺相关载体或单纯疱疹载体。
25.在本文所述的任一系统的一些实施例中,该靶核酸是dna分子。在本文所述的任一系统的一些实施例中,该靶核酸包含pam序列。
26.在本文所述的任一系统的一些实施例中,该crispr相关蛋白具有非特异性核酸酶活性。
27.在本文所述的任一系统的一些实施例中,通过该crispr相关蛋白和rna指导物对该靶核酸的识别导致对该靶核酸的修饰。在本文所述的任一系统的一些实施例中,对该靶核酸的该修饰是双链切割事件。在本文所述的任一系统的一些实施例中,对该靶核酸的该修饰是单链切割事件。在本文所述的任一系统的一些实施例中,对该靶核酸的该修饰导致插入事件。在本文所述的任一系统的一些实施例中,对该靶核酸的该修饰导致缺失事件。在本文所述的任一系统的一些实施例中,对该靶核酸的该修饰导致细胞毒性或细胞死亡。
28.在本文所述的任一系统的一些实施例中,该系统进一步包括供体模板核酸。在本文所述的任一系统的一些实施例中,该供体模板核酸是dna分子。在本文所述的任一系统的一些实施例中,其中该供体模板核酸是rna分子。
29.在本文所述的任一系统的一些实施例中,该rna指导物任选地包括tracrrna和/或调节子rna。在本文所述的任一系统的一些实施例中,该系统进一步包括tracrrna。在本文所述的任一系统的一些实施例中,该系统不包括tracrrna。在本文所述的任一系统的一些实施例中,该crispr相关蛋白是自加工的。在本文所述的任一系统的一些实施例中,该系统进一步包括调节子rna。
30.在本文所述的任一系统的一些实施例中,该crispr相关蛋白包含与seq id no:1的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:152、seq id no:153或seq id no:154的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。
31.在本文所述的任一系统的一些实施例中,该系统存在于递送组合物中,该递送组合物包含纳米颗粒、脂质体、外来体、微泡或基因枪。
32.在本文所述的任一系统的一些实施例中,这些系统在细胞内。在一些实施例中,该细胞是真核细胞。在一些实施例中,该细胞是哺乳动物细胞。在一些实施例中,该细胞是人细胞。在一些实施例中,该细胞是原核细胞。
33.在另一方面,本披露提供了一种细胞,其中该细胞包含:crispr相关蛋白,其中该crispr相关蛋白包含与seq id no:1-56中任一个中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列;以及rna指导物,该rna指导物包含直接重复序列和能够与靶核酸杂交的间隔子序列。在另一方面,本披露提供了一种细胞,其中该细胞包含:crispr相关蛋白或编码该crispr相关蛋白的核酸,其中该crispr相关蛋白包含与seq id no:1-56中任一个中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列;以及rna指导物或编码该rna指导物的核酸,该rna指导物包含直接重复序列和能够与靶核酸杂交的间隔子序列。
34.在本文所述的任一细胞的一些实施例中,该crispr相关蛋白包含至少一个(例如,一个、两个或三个)ruvc结构域或至少一个拆分型ruvc结构域。
35.在本文所述的任一细胞的一些实施例中,该crispr相关蛋白包含以下序列中的一种或多种:(a)px1x2x3x4f(seq id no:216),其中x1是l或m或i或c或f,x2是y或w或f,x3是k或t或c或r或w或y或h或v,并且x4是i或l或m;(b)rx1x2x3l(seq id no:217),其中x1是i或l或m或y或t或f,x2是r或q或k或e或s或t,并且x3是l或i或t或c或m或k;(c)nx1yx2(seq id no:218),其中x1是i或l或f并且x2是k或r或v或e;(d)kx1x2x3fax4x5kd(seq id no:219),其中x1是t或i或n或a或s或f或v,x2是i或v或l或s,x3是h或s或g或r,x4是d或s或e,并且x5是i或v或m或t或n;(e)lx1nx2(seq id no:220),其中x1是g或s或c或t并且x2是n或y或k或s;(f)px1x2x3x4sqx5ds(seq id no:221),其中x1是s或p或a,x2是y或s或a或p或e或y或q或n,x3是f或y或h,x4是t或s,并且x5是m或t或i;(g)kx1x2vrx3x4qex5h(seq id no:222),其中x1是n或k或w或r或e或t或y,x2是m或r或l或s或k或v或e或t或i或d,x3是l或r或h或p或t或k或q或p或s或a,x4是g或q或n或r或k或e或i或t或s或c,并且x5是r或w或y或k或t或f或s或q;以及(h)x1ngx2x3x4dx5nx6x7x8n(seq id no:223),其中x1是i或k或v或l,x2是l或m,x3是n或h或p,x4是a或s或c,x5是v或y或i或f或t或n,x6是a或s,x7是s或a或p,并且x8是m或c或l或r或n或s或k或l。在本文所述的任一细胞的一些实施例中,seq id no:216的序列是n末端序列。在本文所述的任一细胞的一些实施例中,seq id no:219的序列是c末端序列。在本文所述的任一细胞的一些实施例中,seq id no:220的序列是c末端序列。在本文所述的任一细胞的一些
实施例中,seq id no:221的序列是c末端序列。在本文所述的任一细胞的一些实施例中,seq id no:222的序列是c末端序列。在本文所述的任一细胞的一些实施例中,seq id no:223的序列是c末端序列。
36.在本文所述的任一细胞的一些实施例中,该直接重复序列包含与seq id no:57-90、seq id no:118-151或seq id no:213中任一个中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一细胞的一些实施例中,该直接重复序列包含与seq id no:57-90、seq id no:118-151或seq id no:213中任一个中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。
37.在本文所述的任一细胞的一些实施例中,该直接重复序列包含以下序列中的一种或多种:(a)x1x2tx3x4x5x6x7x8(seq id no:224),其中x1是a或c或g,x2是t或c或a,x3是t或g或a,x4是t或g,x5是t或g或a,x6是g或t或a,x7是t或g或a,并且x8是a或g或t(例如,attgttgda(seq id no:225));(b)x1x2x3x4x5x6x7x8x9(seq id no:226),其中x1是t或c或a,x2是t或a或g,x3是t或c或a,x4是t或a,x5是t或a或g,x6是t或a,x7是a或t,x8是a或g或c或t,并且x9是g或a或c(例如,ttttwtarg(seq id no:227));以及(c)x1x2x3ac(seq id no:228),其中x1是a或c或g,x2是c或a,并且x3是a或c(例如,acaac(seq id no:229))。在本文所述的任一细胞的一些实施例中,seq id no:224在该直接重复的5’端的近侧。在本文所述的任一细胞的一些实施例中,seq id no:228在该直接重复的3’端的近侧。
38.在本文所述的任一细胞的一些实施例中,该crispr相关蛋白是与seq id no:1中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:57中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白是与seq id no:1中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:57中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
tnnt-3’或5
’‑
tnrt-3’列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
39.在本文所述的任一细胞的一些实施例中,该crispr相关蛋白是与seq id no:4中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:60中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白是与seq id no:4中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:60
中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
nttn-3’、5
’‑
nttr-3’(例如,5
’‑
tttg-3’)或5
’‑
nnr-3’列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
40.在本文所述的任一细胞的一些实施例中,该crispr相关蛋白是与seq id no:10中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:62或seq id no:213中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白是与seq id no:10中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:62或seq id no:213中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
nttn-3’或5
’‑
rttr-3’(例如,5
’‑
attg-3’或5
’‑
gtta-3’)列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
41.在本文所述的任一细胞的一些实施例中,该间隔子序列包含约15个核苷酸至约55个核苷酸。在本文所述的任一细胞的一些实施例中,该间隔子序列包含20至45个核苷酸。
42.在本文所述的任一细胞的一些实施例中,该crispr相关蛋白包含催化残基(例如,天冬氨酸或谷氨酸)。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白切割该靶核酸。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白进一步包含肽标签、荧光蛋白、碱基编辑结构域、dna甲基化结构域、组蛋白残基修饰结构域、定位因子、转录修饰因子、光门控因子、化学诱导型因子或染色质可视化因子。
43.在本文所述的任一细胞的一些实施例中,编码该crispr相关蛋白的该核酸经密码子优化以在细胞,例如真核细胞,例如哺乳动物细胞,例如人细胞中表达。在本文所述的任一细胞的一些实施例中,编码该crispr相关蛋白的该核酸可操作地连接至启动子。在本文所述的任一细胞的一些实施例中,编码该crispr相关蛋白的该核酸在载体中。在一些实施例中,该载体包括逆转录病毒载体、慢病毒载体、噬菌体载体、腺病毒载体、腺相关载体或单纯疱疹载体。
44.在本文所述的任一细胞的一些实施例中,该rna指导物任选地包括tracrrna和/或调节子rna。在本文所述的任一细胞的一些实施例中,该细胞进一步包含tracrrna。在本文所述的任一细胞的一些实施例中,该细胞不包含tracrrna。在本文所述的任一细胞的一些实施例中,该crispr相关蛋白是自加工的。在本文所述的任一细胞的一些实施例中,该细胞进一步包含调节子rna。
45.在本文所述的任一细胞的一些实施例中,该细胞是真核细胞。在本文所述的任一细胞的一些实施例中,该细胞是哺乳动物细胞。在本文所述的任一细胞的一些实施例中,该细胞是人细胞。在本文所述的任一细胞的一些实施例中,该细胞是原核细胞。
46.在本文所述的任一细胞的一些实施例中,该靶核酸是dna分子。在本文所述的任一
细胞的一些实施例中,该靶核酸包含pam序列。
47.在本文所述的任一细胞的一些实施例中,该crispr相关蛋白具有非特异性核酸酶活性。
48.在本文所述的任一细胞的一些实施例中,通过该crispr相关蛋白和rna指导物对该靶核酸的识别导致对该靶核酸的修饰。在本文所述的任一细胞的一些实施例中,对该靶核酸的该修饰是双链切割事件。在本文所述的任一细胞的一些实施例中,对该靶核酸的该修饰是单链切割事件。在本文所述的任一细胞的一些实施例中,对该靶核酸的该修饰导致插入事件。在本文所述的任一细胞的一些实施例中,对该靶核酸的该修饰导致缺失事件。在本文所述的任一细胞的一些实施例中,对该靶核酸的该修饰导致细胞毒性或细胞死亡。
49.在另一方面,本披露提供了一种使本文所述的系统与细胞中的靶核酸结合的方法,该方法包括:(a)提供该系统;以及(b)将该系统递送至该细胞,其中该细胞包含该靶核酸,其中该crispr相关蛋白结合该rna指导物,并且其中该间隔子序列结合该靶核酸。在一些实施例中,该细胞是真核细胞,例如哺乳动物细胞,例如人细胞。
50.在另一方面,本披露提供了修饰靶核酸的方法,该方法包括向该靶核酸递送工程化的非天然存在的crispr-cas系统,该系统包括:crispr相关蛋白,其中该crispr相关蛋白包含与seq id no:1-56中任一个中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列;以及rna指导物,该rna指导物包含直接重复序列和能够与该靶核酸杂交的间隔子序列;其中该crispr相关蛋白能够结合该rna指导物;并且其中通过该crispr相关蛋白和rna指导物对该靶核酸的识别导致对该靶核酸的修饰。在另一方面,本披露提供了修饰靶核酸的方法,该方法包括向该靶核酸递送工程化的非天然存在的crispr-cas系统,该系统包括:crispr相关蛋白或编码该crispr相关蛋白的核酸,其中该crispr相关蛋白包含与seq id no:1-56中任一个中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列;以及rna指导物,该rna指导物包含直接重复序列和能够与该靶核酸杂交的间隔子序列;其中该crispr相关蛋白能够结合该rna指导物;并且其中通过该crispr相关蛋白和rna指导物对该靶核酸的识别导致对该靶核酸的修饰。
51.在本文所述的任一方法的一些实施例中,该crispr相关蛋白包含以下序列中的一种或多种:(a)px1x2x3x4f(seq id no:216),其中x1是l或m或i或c或f,x2是y或w或f,x3是k或t或c或r或w或y或h或v,并且x4是i或l或m;(b)rx1x2x3l(seq id no:217),其中x1是i或l或m或y或t或f,x2是r或q或k或e或s或t,并且x3是l或i或t或c或m或k;(c)nx1yx2(seq id no:218),其中x1是i或l或f并且x2是k或r或v或e;(d)kx1x2x3fax4x5kd(seq id no:219),其中x1是t或i或n或a或s或f或v,x2是i或v或l或s,x3是h或s或g或r,x4是d或s或e,并且x5是i或v或m或t或n;(e)lx1nx2(seq id no:220),其中x1是g或s或c或t并且x2是n或y或k或s;(f)px1x2x3x4sqx5ds(seq id no:221),其中x1是s或p或a,x2是y或s或a或p或e或y或q或n,x3是f或y或h,x4是t或s,并且x5是m或t或i;(g)kx1x2vrx3x4qex5h(seq id no:222),其中x1是n或k或w或r或e或t或y,x2是m或r或l或s或k或v或e或t或i或d,x3是l或r或h或p或t或k或q或p或s或a,x4是g或q或n或r或k或e或i或t或s或c,并且x5是r或w或y或k或t或f或s或q;以及(h)
x1ngx2x3x4dx5nx6x7x8n(seq id no:223),其中x1是i或k或v或l,x2是l或m,x3是n或h或p,x4是a或s或c,x5是v或y或i或f或t或n,x6是a或s,x7是s或a或p,并且x8是m或c或l或r或n或s或k或l。在本文所述的任一方法的一些实施例中,seq id no:216的序列是n末端序列。在本文所述的任一方法的一些实施例中,seq id no:219的序列是c末端序列。在本文所述的任一方法的一些实施例中,seq id no:220的序列是c末端序列。在本文所述的任一方法的一些实施例中,seq id no:221的序列是c末端序列。在本文所述的任一方法的一些实施例中,seq id no:222的序列是c末端序列。在本文所述的任一方法的一些实施例中,seq id no:223的序列是c末端序列。
52.在本文所述的任一方法的一些实施例中,该直接重复序列包含与seq id no:57-90、seq id no:118-151或seq id no:213中任一个中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一方法的一些实施例中,该直接重复序列包含与seq id no:57-90、seq id no:118-151或seq id no:213中任一个中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。
53.在本文所述的任一方法的一些实施例中,该直接重复序列包含以下序列中的一种或多种:(a)x1x2tx3x4x5x6x7x8(seq id no:224),其中x1是a或c或g,x2是t或c或a,x3是t或g或a,x4是t或g,x5是t或g或a,x6是g或t或a,x7是t或g或a,并且x8是a或g或t(例如,attgttgda(seq id no:225));(b)x1x2x3x4x5x6x7x8x9(seq id no:226),其中x1是t或c或a,x2是t或a或g,x3是t或c或a,x4是t或a,x5是t或a或g,x6是t或a,x7是a或t,x8是a或g或c或t,并且x9是g或a或c(例如,ttttwtarg(seq id no:227));以及(c)x1x2x3ac(seq id no:228),其中x1是a或c或g,x2是c或a,并且x3是a或c(例如,acaac(seq id no:229))。在本文所述的任一方法的一些实施例中,seq id no:224在该直接重复的5’端的近侧。在本文所述的任一方法的一些实施例中,seq id no:228在该直接重复的3’端的近侧。
54.在本文所述的任一方法的一些实施例中,该crispr相关蛋白是与seq id no:1中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:57中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一方法的一些实施例中,该crispr相关蛋白是与seq id no:1中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:57中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一方法的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
tnnt-3’或5
’‑
tnrt-3’列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
55.在本文所述的任一方法的一些实施例中,该crispr相关蛋白是与seq id no:4中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,
并且其中该直接重复序列包含与seq id no:60中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一方法的一些实施例中,该crispr相关蛋白是与seq id no:4中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:60中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一方法的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
nttn-3’、5
’‑
nttr-3’(例如,5
’‑
tttg-3’)或5
’‑
nnr-3’列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
56.在本文所述的任一方法的一些实施例中,该crispr相关蛋白是与seq id no:10中列出的氨基酸序列具有至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:62或seq id no:213中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一方法的一些实施例中,该crispr相关蛋白是与seq id no:10中列出的氨基酸序列具有至少95%(例如,95%、96%、97%、98%、99%或100%)同一性的蛋白质,并且其中该直接重复序列包含与seq id no:62或seq id no:213中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所述的任一方法的一些实施例中,该crispr相关蛋白能够识别原型间隔子相邻基序(pam)序列,其中该pam序列包括作为5
’‑
nttn-3’或5
’‑
rttr-3’(例如,5
’‑
attg-3’或5
’‑
gtta-3’)列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
57.在本文所述的任一方法的一些实施例中,该间隔子序列包含约15个核苷酸至约55个核苷酸。在本文所述的任一方法的一些实施例中,该间隔子序列包含20至45个核苷酸。
58.在本文所述的任一方法的一些实施例中,该rna指导物任选地包括tracrrna和/或调节子rna。在本文所述的任一方法的一些实施例中,该系统进一步包括tracrrna。在本文所述的任一方法的一些实施例中,该系统不包括tracrrna。在本文所述的任一方法的一些实施例中,该crispr相关蛋白是自加工的。在本文所述的任一方法的一些实施例中,该系统进一步包括调节子rna。
59.在本文所述的任一方法的一些实施例中,该靶核酸是dna分子。在本文所述的任一方法的一些实施例中,该靶核酸包含pam序列。
60.在本文所述的任一方法的一些实施例中,该crispr相关蛋白具有非特异性核酸酶活性。
61.在本文所述的任一方法的一些实施例中,对该靶核酸的该修饰是双链切割事件。在本文所述的任一方法的一些实施例中,对该靶核酸的该修饰是单链切割事件。在本文所述的任一方法的一些实施例中,对该靶核酸的该修饰导致插入事件。在本文所述的任一方法的一些实施例中,对该靶核酸的该修饰导致缺失事件。在本文所述的任一方法的一些实施例中,对该靶核酸的该修饰导致细胞毒性或细胞死亡。
62.在另一方面,本披露提供了一种编辑靶核酸的方法,该方法包括使该靶核酸与本
文所述的系统接触。在另一方面,本披露提供了一种修饰靶核酸的表达的方法,该方法包括使该靶核酸与本文所述的系统接触。在另一方面,本披露提供了一种使有效载荷核酸在靶核酸的位点处靶向插入的方法,该方法包括使该靶核酸与本文所述的系统接触。在另一方面,本披露提供了一种从靶核酸处的位点靶向切除有效载荷核酸的方法,该方法包括使该靶核酸与本文所述的系统接触。在另一方面,本披露提供了一种在识别dna靶核酸后非特异性降解单链dna的方法,该方法包括使该靶核酸与本文所述的系统接触。
63.在本文所提供的任一系统或方法的一些实施例中,该接触包括直接接触或间接接触。在本文所提供的任一系统或方法的一些实施例中,间接接触包括在这样的条件下施用编码本文所述的rna指导物或crispr相关蛋白的一种或多种核酸,这些条件允许产生该rna指导物和/或crispr相关蛋白。在本文所提供的任一系统或方法的一些实施例中,接触包括体内接触或体外接触。在本文所提供的任一系统或方法的一些实施例中,使靶核酸与该系统接触包括在这样的条件下使包含该核酸的细胞与该系统接触,这些条件允许该crispr相关蛋白和该指导rna到达该靶核酸。在本文所提供的任一系统或方法的一些实施例中,使体内细胞与该系统接触包括在这样的条件下将该系统施用至包括该细胞的受试者,这些条件允许该crispr相关蛋白和该指导rna到达该细胞或在该细胞中产生。
64.在另一方面,本披露提供了一种本文所提供的系统,用于在以下体外或离体方法中使用:(a)靶向并编辑靶核酸;(b)在识别该核酸后非特异性降解单链核酸;(c)在识别双链靶标的间隔子互补链后靶向该双链靶标的非间隔子互补链并使其产生切口;(d)靶向并切割双链靶核酸;(e)检测样品中的靶核酸;(f)特异性编辑双链核酸;(g)对双链核酸进行碱基编辑;(h)在细胞中诱导基因型特异性或转录状态特异性的细胞死亡或休眠;(i)在双链核酸靶标中产生插入缺失;(j)将序列插入至双链核酸靶标中;或(k)缺失或倒置双链核酸靶标中的序列。
65.在另一方面,本披露提供了将插入或缺失引入至哺乳动物细胞中的靶核酸中的方法,该方法包括以下的转染:(a)编码crispr相关蛋白的核酸序列,其中该crispr相关蛋白包含与seq id no:1-56中任一个中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列;以及(b)rna指导物(或编码该rna指导物的核酸),该rna指导物包含直接重复序列和能够与该靶核酸杂交的间隔子序列;其中该crispr相关蛋白能够结合该rna指导物;并且其中通过该crispr相关蛋白和rna指导物对该靶核酸的识别导致对该靶核酸的修饰。
66.在本文所提供的任一方法的一些实施例中,该crispr相关蛋白包含与seq id no:4中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列。在本文所提供的任一方法的一些实施例中,该crispr相关蛋白包含与seq id no:4中列出的氨基酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的氨基酸序列。在本文所提供的任一方法的一些实施例中,该直接重复包含与seq id no:60中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所提供的任一方法的一些实施例中,其中该直接重复包含与seq id no:60中列出的核苷酸序列至
少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所提供的任一方法的一些实施例中,该靶核酸相邻于pam序列,并且该pam序列包括作为5
’‑
nttn-3’、5
’‑
nttr-3’(例如,5
’‑
tttg-3’)或5
’‑
nnr-3’列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
67.在本文所提供的任一方法的一些实施例中,该crispr相关蛋白包含与seq id no:10中列出的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列。在本文所提供的任一方法的一些实施例中,该crispr相关蛋白包含与seq id no:10中列出的氨基酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的氨基酸序列。在本文所提供的任一方法的一些实施例中,该直接重复包含与seq id no:62或seq id no:213中列出的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所提供的任一方法的一些实施例中,其中该直接重复包含与seq id no:62或seq id no:213中列出的核苷酸序列至少95%(例如,95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在本文所提供的任一方法的一些实施例中,该靶核酸相邻于pam序列,并且该pam序列包括作为5
’‑
nttn-3’或5
’‑
rttr-3’(例如,5
’‑
attg-3’或5
’‑
gtta-3’)列出的核酸序列,其中“n”是任何核苷酸并且“r”是a或g。
68.在本文所提供的任一方法的一些实施例中,该转染是瞬时转染。在本文所提供的任一方法的一些实施例中,该细胞是人细胞。
69.在另一方面,本披露提供了一种组合物,该组合物包含:(a)crispr相关蛋白或编码该crispr相关蛋白的核酸;以及(b)rna指导物,该rna指导物包含直接重复序列和间隔子序列;其中该crispr相关蛋白包含以下氨基酸序列中的一种或多种:(i)px1x2x3x4f(seq id no:216),其中x1是l或m或i或c或f,x2是y或w或f,x3是k或t或c或r或w或y或h或v,并且x4是i或l或m;(ii)rx1x2x3l(seq id no:217),其中x1是i或l或m或y或t或f,x2是r或q或k或e或s或t,并且x3是l或i或t或c或m或k;(iii)nx1yx2(seq id no:218),其中x1是i或l或f并且x2是k或r或v或e;(iv)kx1x2x3fax4x5kd(seq id no:219),其中x1是t或i或n或a或s或f或v,x2是i或v或l或s,x3是h或s或g或r,x4是d或s或e,并且x5是i或v或m或t或n;(v)lx1nx2(seq id no:220),其中x1是g或s或c或t并且x2是n或y或k或s;(vi)px1x2x3x4sqx5ds(seq id no:221),其中x1是s或p或a,x2是y或s或a或p或e或y或q或n,x3是f或y或h,x4是t或s,并且x5是m或t或i;(vii)kx1x2vrx3x4qex5h(seq id no:222),其中x1是n或k或w或r或e或t或y,x2是m或r或l或s或k或v或e或t或i或d,x3是l或r或h或p或t或k或q或p或s或a,x4是g或q或n或r或k或e或i或t或s或c,并且x5是r或w或y或k或t或f或s或q;以及(viii)x1ngx2x3x4dx5nx6x7x8n(seq id no:223),其中x1是i或k或v或l,x2是l或m,x3是n或h或p,x4是a或s或c,x5是v或y或i或f或t或n,x6是a或s,x7是s或a或p,并且x8是m或c或l或r或n或s或k或l;并且其中该crispr相关蛋白能够结合该rna指导物并且能够修饰与该间隔子序列互补的该靶核酸序列。
70.在本文所述的任一组合物的一些实施例中,该直接重复序列包含以下序列中的一种或多种:(a)x1x2tx3x4x5x6x7x8(seq id no:224),其中x1是a或c或g,x2是t或c或a,x3是t或g或a,x4是t或g,x5是t或g或a,x6是g或t或a,x7是t或g或a,并且x8是a或g或t(例如,attgttgda(seq id no:225));(b)x1x2x3x4x5x6x7x8x9(seq id no:226),其中x1是t或c或a,x2是t或a或
g,x3是t或c或a,x4是t或a,x5是t或a或g,x6是t或a,x7是a或t,x8是a或g或c或t,并且x9是g或a或c(例如,ttttwtarg(seq id no:227));以及(c)x1x2x3ac(seq id no:228),其中x1是a或c或g,x2是c或a,并且x3是a或c(例如,acaac(seq id no:229))。在本文所述的任一组合物的一些实施例中,seq id no:224在该直接重复的5’端的近侧。在本文所述的任一组合物的一些实施例中,seq id no:228在该直接重复的3’端的近侧。
71.在本文所述的任一组合物的一些实施例中,该crispr相关蛋白包含至少一个(例如,一个、两个或三个)ruvc结构域或至少一个拆分型ruvc结构域。
72.在本文所述的任一组合物的一些实施例中,该rna指导物的该间隔子序列包含约15个核苷酸至约55个核苷酸。在本文所述的任一组合物的一些实施例中,该rna指导物的该间隔子序列包含20至45个核苷酸。
73.在本文所述的任一组合物的一些实施例中,该crispr相关蛋白包含催化残基(例如,天冬氨酸或谷氨酸)。在本文所述的任一组合物的一些实施例中,该crispr相关蛋白切割该靶核酸。在本文所述的任一组合物的一些实施例中,该crispr相关蛋白进一步包含肽标签、荧光蛋白、碱基编辑结构域、dna甲基化结构域、组蛋白残基修饰结构域、定位因子、转录修饰因子、光门控因子、化学诱导型因子或染色质可视化因子。
74.在本文所述的任一组合物的一些实施例中,编码该crispr相关蛋白的该核酸经密码子优化以在细胞,例如真核细胞,例如哺乳动物细胞,例如人细胞中表达。在本文所述的任一组合物的一些实施例中,编码该crispr相关蛋白的该核酸可操作地连接至启动子。在本文所述的任一组合物的一些实施例中,编码该crispr相关蛋白的该核酸在载体中。在一些实施例中,该载体包括逆转录病毒载体、慢病毒载体、噬菌体载体、腺病毒载体、腺相关载体或单纯疱疹载体。
75.在本文所述的任一组合物的一些实施例中,该靶核酸是dna分子。在本文所述的任一组合物的一些实施例中,该靶核酸包含pam序列。
76.在本文所述的任一组合物的一些实施例中,该crispr相关蛋白具有非特异性核酸酶活性。
77.在本文所述的任一组合物的一些实施例中,通过该crispr相关蛋白和rna指导物对该靶核酸的识别导致对该靶核酸的修饰。在本文所述的任一组合物的一些实施例中,对该靶核酸的该修饰是双链切割事件。在本文所述的任一组合物的一些实施例中,对该靶核酸的该修饰是单链切割事件。在本文所述的任一组合物的一些实施例中,对该靶核酸的该修饰导致插入事件。在本文所述的任一组合物的一些实施例中,对该靶核酸的该修饰导致缺失事件。在本文所述的任一组合物的一些实施例中,对该靶核酸的该修饰导致细胞毒性或细胞死亡。
78.在本文所述的任一组合物的一些实施例中,该系统进一步包括供体模板核酸。在本文所述的任一组合物的一些实施例中,该供体模板核酸是dna分子。在本文所述的任一组合物的一些实施例中,其中该供体模板核酸是rna分子。
79.在本文所述的任一组合物的一些实施例中,该rna指导物任选地包括tracrrna。在本文所述的任一组合物的一些实施例中,该系统进一步包括tracrrna。在本文所述的任一组合物的一些实施例中,该系统不包括tracrrna。在本文所述的任一组合物的一些实施例中,该crispr相关蛋白是自加工的。
80.在本文所述的任一组合物的一些实施例中,该系统存在于递送组合物中,该递送组合物包含纳米颗粒、脂质体、外来体、微泡或基因枪。
81.在本文所述的任一组合物的一些实施例中,这些组合物在细胞内。在一些实施例中,该细胞是真核细胞。在一些实施例中,该细胞是哺乳动物细胞。在一些实施例中,该细胞是人细胞。在一些实施例中,该细胞是原核细胞。
82.本文所述的效应子提供了另外的特征,这些另外的特征包括但不限于1)新型核酸编辑特性和控制机制,2)更小的大小以便在递送策略中具有更大的通用性,3)基因型触发的细胞过程诸如细胞死亡,和4)可编程的rna指导的dna插入、切除和转移,以及5)先前存在的免疫力通过非人共生来源产生的分化谱。参见例如实例1、4和5以及图1-3和5-11d。将本文所述的新型dna靶向系统添加至基因组和表观基因组操纵的技术工具箱中使得能够广泛应用于特定的编程扰动。
83.本发明的其他特征和优点通过以下详细描述以及权利要求将会变得清楚。
附图说明
84.这些图是表示被称为clust.091979的蛋白质聚簇的分析结果的一系列示意图。
85.图1a、图1b、图1c、图1d、图1e、图1f、图1g、图1h、图1i、图1j、图1k和图1l共同示出了seq id no:1-4、14、15、17-19、21-25、27-33、35-49、51-56的效应子的比对。
86.图2是示出了clust.091979效应子的ruvc结构域的示意图,该示意图是基于表6中所示序列的共有序列。
87.图3示出了seq id no:57、58、60、62、63、70、72-74、76、77、80、83、84、86-88、90、128、130、139和213的直接重复序列的比对。共有序列(seq id no:230)示出在比对的顶部。
88.图4a是实例4中所述的体内阴性选择筛选测定的组分的示意图。设计了crispr阵列文库,这些阵列文库包括侧接有两个dr并由j23119表达、从pacyc184或大肠杆菌必需基因的两条链上均匀取样的非代表性间隔子。
89.图4b是实例4中所述的体内阴性选择筛选工作流的示意图。将crispr阵列文库克隆至效应子质粒中。将效应子质粒和非编码质粒转化至大肠杆菌中,之后进行生长以便阴性选择赋予对来自pacyc184或大肠杆菌必需基因的转录物的干扰的crispr阵列。使用效应子质粒的靶向测序来鉴定耗减的crispr阵列。进一步进行小型rna测序以鉴定成熟crrna和潜在tracrrna要求。
90.图5是clust.091979auxo013988882(seq id no:1中列出的效应子)的图,该图示出了在具有非编码序列的情况下,工程化组合物针对靶向pacyc184的间隔子和直接重复转录取向的耗减活性程度。描绘了在呈“正向”取向(5
’‑
acta
…
aact-[间隔子]-3’)的直接重复的情况下和在呈“反向”取向(5
’‑
agtt
…
tagt-[间隔子]-3’)的直接重复的情况下的耗减程度。
[0091]
图6a是按pacyc184质粒上的位置示出了clust.091979auxo013988882(具有非编码序列)的耗减靶标和非耗减靶标的密度的图形表示。图6b是按大肠杆菌菌株e.cloni上的位置示出了clust.091979auxo013988882(具有非编码序列)的耗减靶标和非耗减靶标的密度的图形表示。单独地并与注释基因的取向相关地示出了顶链和底链上的靶标。谱带的幅度指示耗减程度,其中较轻的谱带接近于3的命中阈值。梯度是示出了相对转录物丰度的
rna测序的热图。
[0092]
图7是作为clust.091979auxo013988882(具有非编码序列)的pam序列的预测的e.cloni中侧接耗减靶标的序列的weblogo。
[0093]
图8是clust.091979srr3181151(seq id no:4中列出的效应子)的图,该图示出了在具有非编码序列的情况下,工程化组合物针对靶向pacyc184的间隔子和直接重复转录取向的耗减活性程度。描绘了在呈“正向”取向(5
’‑
gttg
…
cagg-[间隔子]-3’)的直接重复的情况下和在呈“反向”取向(5
’‑
cctg
…
caac-[间隔子]-3’)的直接重复的情况下的耗减程度。
[0094]
图9a是按pacyc184质粒上的位置示出了clust.091979srr3181151(具有非编码序列)的耗减靶标和非耗减靶标的密度的图形表示。图9b是按大肠杆菌菌株e.cloni上的位置示出了clust.091979srr3181151(具有非编码序列)的耗减靶标和非耗减靶标的密度的图形表示。单独地并与注释基因的取向相关地示出了顶链和底链上的靶标。谱带的幅度指示耗减程度,其中较轻的谱带接近于3的命中阈值。梯度是示出了相对转录物丰度的rna测序的热图。
[0095]
图10是作为clust.091979srr3181151(具有非编码序列)的pam序列的预测的e.cloni中侧接耗减靶标的序列的weblogo。
[0096]
图11a示出了在hek293细胞中seq id no:4的效应子在seq id no:206的aavs1靶基因座和seq id no:208的vegfa靶基因座处诱导的插入缺失。图11b示出了在hek293细胞中seq id no:4的效应子在seq id no:253、255、257、259和275的aavs1靶基因座、seq id no:263、265、267、269、271、273和277的vegfa靶基因座以及seq id no:261的emx1靶基因座处诱导的插入缺失。图11c示出了在hek293细胞中seq id no:10的效应子在seq id no:210的aavs1靶基因座、seq id no:212的aavs1靶基因座以及seq id no:215的vegfa靶基因座处诱导的插入缺失。图11d示出了在hek293细胞中seq id no:10的效应子在seq id no:279、281、285和287的aavs1靶基因座、seq id no:283的vegfa靶基因座以及seq id no:289的emx1靶基因座处诱导的插入缺失。
具体实施方式
[0097]
天然多样性的crispr-cas系统包括广泛范围的可用于可编程生物技术的活性机制和功能性元件。在自然界中,这些系统能够有效地防御外来dna和病毒,同时提供自我与非自我的区别以避免自我靶向。在工程化环境中,这些系统提供了分子技术的多样性工具箱,并且定义了靶向空间的边界。本文所述的方法已被用于发现单亚基2类效应子系统内的另外机制和参数,这些另外机制和参数扩展了rna可编程核酸操纵的能力。
[0098]
除非另有定义,否则本文所用的所有技术和科学术语均具有与本发明所属领域普通技术人员所通常理解的相同含义。虽然类似于或等同于本文所述的那些的方法和材料可以用于本发明的实践或测试中,但是下面描述了合适的方法和材料。本文所提及的所有出版物、专利申请、专利和其他参考文献均通过援引以其全文并入。在有矛盾的情况下,将以本说明书(包括定义)为准。此外,材料、方法和实例仅为说明性的,而并非旨在为限制性的。根据专利法中的标准惯例,申请人使用过渡性短语“包含”、“基本上由
……
组成”或“由
……
组成”保留替代性地要求保护任何所披露的发明的权利。
[0099]
除非上下文另有明确规定,否则如本文所用,单数形式“一种/一个(a/an)”和“该”包括复数指示物。例如,对“一种核酸”的提及意指一种或多种核酸。
[0100]
应指出,术语如“优选地”、“合适地”、“通常”和“典型地”在本文中不用于限制所要求保护的发明的范围或者暗示某些特征对于所要求保护的发明的结构或功能是关键、必要的或甚至重要的。相反,这些术语仅仅旨在强调在本发明的特定实施例中可以利用或可以不利用的替代性或其另外的特征。
[0101]
出于描述和限定本发明的目的,应指出术语“基本上”在本文中用于表示可以归因于任何定量比较、值、测量值或其他表示的固有不确定性程度。术语“基本上”在本文中还用于表示定量表示可以不同于所述参考而不导致讨论中的主题的基本功能的改变的程度。
[0102]
如本文所用,术语“crispr-cas系统”是指参与crispr效应子的表达或指导其活性的核酸和/或蛋白质,包括编码crispr效应子的序列、rna指导物以及来自crispr基因座的其他序列和转录物。
[0103]
如本文可互换使用的术语“crispr相关蛋白”、“crispr-cas效应子”、“crispr效应子”、“效应子”、“效应子蛋白”、“crispr酶”等是指完成酶活性或结合由rna指导物指定的核酸上的靶位点的蛋白质。在一些实施例中,crispr效应子具有核酸内切酶活性、切口酶活性和/或核酸外切酶活性。
[0104]
如本文所用,术语“rna指导物”、“指导rna”、“grna”和“指导序列”是指有利于将本文所述的效应子靶向至靶核酸(诸如dna和/或rna)的任何rna分子。示例性“rna指导物”包括但不限于crrna,以及与tracrrna和/或调节子rna杂交或融合的crrna。在一些实施例中,rna指导物包括融合成单rna分子或者作为单独的rna分子的crrna和tracrrna两者。在一些实施例中,rna指导物包括融合成单rna分子或者作为单独的rna分子的crrna和调节子rna。在一些实施例中,rna指导物包括融合成单rna分子或者作为单独的rna分子的crrna、tracrrna和调节子rna。
[0105]
如本文使用,术语“crispr效应子复合物”、“效应子复合物”或“监视复合物”是指含有crispr效应子和rna指导物的复合物。crispr效应子复合物可以进一步包含一种或多种辅助蛋白。一种或多种辅助蛋白可以是非催化性和/或非靶标结合的。
[0106]
如本文所用,术语“crispr rna”和“crrna”是指包含由crispr效应子用于特异性识别核酸序列的指导序列的rna分子。crrna“间隔子”序列与核酸靶序列互补并且能够部分或完全结合核酸靶序列。crrna可以包含与tracrrna杂交的序列。进而,crrna:tracrrna双链体可以结合crispr效应子。如本文所用,术语“前crrna”是指包含dr-间隔子-dr序列的未加工rna分子。如本文所用,术语“成熟crrna”是指前crrna的加工形式;成熟crrna可以包含dr-间隔子序列,其中dr是前crrna的dr的截短形式和/或间隔子是前crrna的间隔子的截短形式。
[0107]
如本文所用,术语“反式激活crrna”或“tracrrna”是指这样的rna分子,该rna分子包含形成为crispr效应子结合特定靶核酸所需的结构和/或序列基序的序列。
[0108]
如本文所用,术语“crispr阵列”是指包含crispr重复和间隔子的核酸(例如,dna)区段,该核酸区段从第一个crispr重复的第一个核苷酸开始并以最后一个(末端)crispr重复的最后一个核苷酸结束。典型地,crispr阵列中的每个间隔子位于两个重复之间。如本文所用,术语“crispr重复”、“crispr直接重复”和“直接重复”是指多个短的直接重复序列,这
protocols in molecular biology[分子生物学实验指南],greene publishing associates and wiley interscience[格林出版协会和威利跨学科出版社],new york[纽约];以及pcr protocols:a guide to methods and applications[pcr方案:方法和应用指南](innis等人.,1990,academic press[学术出版社],san diego,calif.[加利福尼亚州圣迭哥])。
[0117]
术语“基因修饰”或“基因工程化”广义地是指对细胞的基因组或核酸的操纵。同样,术语“基因工程化的”和“工程化的”是指包含经操纵的基因组或核酸的细胞。基因修饰的方法包括例如异源基因表达、基因或启动子插入或缺失、核酸突变、改变基因表达或失活、酶工程化、定向演化、基于知识的设计、随机诱变方法、基因改组和密码子优化。
[0118]
术语“重组体”表明核酸、蛋白质或细胞是基因修饰、工程化或重组的产物。一般来讲,术语“重组体”是指含有来源于多种来源的遗传物质或由其编码的核酸、蛋白质或细胞。如本文所用,术语“重组体”还可以用于描述包含突变的核酸或蛋白质(包括内源性核酸或蛋白质的突变形式)的细胞。术语“重组细胞”和“重组宿主”可以互换使用。在一些实施例中,重组细胞包含本文所披露的crispr效应子。crispr效应子可以经密码子优化以在重组细胞中表达。在一些实施例中,本文所披露的重组细胞进一步包含rna指导物。在一些实施例中,本文所披露的重组细胞的rna指导物包含tracrrna。在一些实施例中,本文所披露的重组细胞包含调节子rna。在一些实施例中,重组细胞可以是原核细胞,诸如大肠杆菌细胞。在一些实施例中,重组细胞是真核细胞,诸如哺乳动物细胞,包括人细胞。
[0119]
对clust.091979的鉴定
[0120]
本技术涉及在本文中称为“clust.091979”的新型蛋白质家族的鉴定、工程化和用途。如图2中所示,clust.091979的蛋白质包含ruvc结构域(表示为ruvc i、ruvc ii和ruvc iii)。如表5中所示,clust.091979的效应子的大小范围为从约700个氨基酸至约800个氨基酸。因此,clust.091979的效应子小于如下面所示的本领域已知的效应子。参见例如表1。
[0121]
表1.已知crispr-cas系统效应子的大小。
[0122][0123][0124]
使用计算方法和算法鉴定clust.091979的效应子以搜索和鉴定展现出与某些其他特征的强共现模式的蛋白质。在某些实施例中,这些计算方法涉及鉴定与crispr阵列很
靠近地共现的蛋白质。本文所披露的方法还可用于鉴定天然很靠近其他特征(非编码特征和蛋白质编码特征(例如,细菌基因座的非编码区域中的噬菌体序列片段;或crispr cas1蛋白质))出现的蛋白质。应理解,本文所述的方法和计算可以在一个或多个计算设备上执行。
[0125]
从基因组或宏基因组数据库获得基因组序列的集合。数据库包括短读段、或重叠群水平数据、或组装的支架、或生物体的完整基因组序列。同样,数据库可以包括来自原核生物或真核生物的基因组序列数据,或可以包括来自宏基因组环境样品的数据。数据库储存库的实例包括国家生物技术信息中心(national center for biotechnology information)(ncbi)refseq、ncbi genbank、ncbi全基因组鸟枪(wgs)和联合基因组研究所(joint genome institute)(jgi)综合微生物基因组(img)。
[0126]
在一些实施例中,施加最小大小要求以选择具有指定的最小长度的基因组序列数据。在某些示例性实施例中,最小重叠群长度可以是100个核苷酸、500nt、1kb、1.5kb、2kb、3kb、4kb、5kb、10kb、20kb、40kb或50kb。
[0127]
在一些实施例中,从完整的或选择的基因组序列数据集合中提取已知的或预测的蛋白质。在一些实施例中,已知或预测的蛋白质获取自提取源数据库提供的编码序列(cds)注释。在一些实施例中,通过应用计算方法从核苷酸序列中鉴定蛋白质来确定预测的蛋白质。在一些实施例中,使用genemark套件来从基因组序列预测蛋白质。在一些实施例中,使用prodigal来从基因组序列预测蛋白质。在一些实施例中,可以在相同的序列数据集合上使用多个蛋白质预测算法,其中所得的蛋白质集合被去重复。
[0128]
在一些实施例中,从基因组序列数据中鉴定crispr阵列。在一些实施例中,使用piler-cr来鉴定crispr阵列。在一些实施例中,使用crispr识别工具(crt)来鉴定crispr阵列。在一些实施例中,通过鉴定重复最小次数(例如,2、3或4次)的核苷酸基序的启发式方法来鉴定crispr阵列,其中重复基序的连续出现之间的间隔不超过指定长度(例如,50、100或150个核苷酸)。在一些实施例中,可以在相同的序列数据集合上使用多个crispr阵列鉴定工具,其中所得的crispr阵列集合被去重复。
[0129]
在一些实施例中,鉴定出与crispr阵列很靠近的蛋白质(在本文中称为“crispr近侧蛋白质聚簇”)。在一些实施例中,靠近性被定义为核苷酸距离,并且可以在20kb、15kb或5kb内。在一些实施例中,靠近性被定义为蛋白质与crispr阵列之间的开放阅读框(orf)的数目,并且某些示例性距离可以是10、5、4、3、2、1或0个orf。然后将被鉴定为与crispr阵列很靠近的蛋白质分组到同源蛋白质聚簇中。在一些实施例中,使用blastclust来形成crispr近侧蛋白质聚簇。在某些其他实施例中,使用mmseqs2来形成crispr近侧蛋白质聚簇。
[0130]
为了建立crispr近侧蛋白质聚簇的成员之间的强共现模式,可以在先前编译的已知和预测的蛋白质的完整集合上执行对蛋白质聚簇的每个成员的blast搜索。在一些实施例中,可以使用ublast或mmseqs2来搜索类似蛋白质。在一些实施例中,可以仅对家族中的代表性蛋白质子集进行搜索。
[0131]
在一些实施例中,通过度量对crispr近侧蛋白质聚簇进行排序或过滤,以确定共现。一个示例性度量是蛋白质聚簇中的元件数目与达到某个e值阈值的blast匹配数目的比率。在一些实施例中,可以使用恒定的e值阈值。在其他实施例中,e值阈值可以由蛋白质聚
簇的距离最远成员确定。在一些实施例中,将蛋白质的总体集合聚簇,并且共现度量是crispr近侧蛋白质聚簇的元件数目与一个或多个包含的总体聚簇的元件数目的比率。
[0132]
在一些实施例中,基于聚簇中蛋白质的天然存在的基因座结构,使用人工审查过程来评价工程化系统的潜在功能性和最小的组分集合。在一些实施例中,蛋白质聚簇的图形表示可以有助于人工审查,并且可以包含信息,该信息包括成对序列相似性、系统发育树、源生物体/环境、预测的功能性结构域和基因座结构的图形描绘。在一些实施例中,基因座结构的图形描绘可以针对具有高表示的附近蛋白质家族过滤。在一些实施例中,可以通过相关的附近蛋白质的数目与一个或多个包含的总体聚簇的一个或多个大小的比率来计算表示。在某些示例性实施例中,蛋白质聚簇的图形表示可以包含对天然存在的基因座的crispr阵列结构的描绘。在一些实施例中,蛋白质聚簇的图形表示可以包含保守的直接重复的数目相对于推定crispr阵列的长度的描绘,或者独特的间隔子序列的数目相对于推定crispr阵列的长度的描绘。在一些实施例中,蛋白质聚簇的图形表示可以包含对预测新的crispr-cas系统并鉴定其组分的推定效应子与crispr阵列共现的各种度量的描绘。
[0133]
clust.091979的池化筛选
[0134]
为了有效地验证本文鉴定的工程化clust.091979crispr-cas系统的活性、机制和功能参数,在大肠杆菌中使用了池化筛选方法,如实例4中所述。首先,根据clust.091979crispr系统的保守蛋白和非编码元件的计算鉴定,使用dna合成和分子克隆将单独的组分组装成单一人工表达载体,在一个实施例中,该单一人工表达载体是基于pet-28a 骨架。在第二实施例中,将效应子和非编码元件在mrna转录物上转录,并且使用不同的核糖体结合位点来翻译单独效应子。
[0135]
其次,用含有靶向第二质粒pacyc184的非天然间隔子的未加工crrna的文库替换天然crrna和靶向间隔子。将此crrna文库克隆至包含效应子和非编码元件的载体骨架(例如,pet-28a )中,并且随后将文库连同pacyc184质粒靶标转化至大肠杆菌中。因此,每个所得的大肠杆菌细胞含有不超过一个靶向阵列。在替代性实施例中,含有非天然间隔子的未加工crrna的文库另外靶向大肠杆菌必需基因,这些大肠杆菌必需基因从诸如在以下文献中所述的那些的资源中提取:baba等人(2006)mol.syst.biol.[分子系统生物学]2:2006.0008;和gerdes等人(2003)j.bacteriol.[细菌学杂志]185(19):5673-84,将这些文献中的每一个的全部内容通过援引并入本文。在此实施例中,新型crispr-cas系统的破坏必需基因功能的阳性靶向活性导致细胞死亡或生长停滞。在一些实施例中,可以将必需基因靶向间隔子与pacyc184靶标组合。
[0136]
第三,将大肠杆菌在抗生素选择下生长。在一个实施例中,使用三重抗生素选择:卡那霉素(用于确保成功转化含有工程化crispr效应子系统的pet-28a 载体)以及氯霉素和四环素(用于确保成功共转化pacyc184靶标载体)。由于pacyc184通常赋予对氯霉素和四环素的抗性,因此在抗生素选择下,靶向质粒的新型crispr-cas系统的阳性活性将消除主动表达crrna文库的效应子、非编码元件和特异性活性元件的细胞。典型地,在转化后12-14h分析存活细胞的群体。在一些实施例中,在转化后6-8h、在转化后8-12h、在转化后长达24h或在转化后超过24h进行对存活细胞的分析。与较早时间点相比,在较晚时间点检查存活细胞的群体产生与非活性crrna相比耗减的信号。
[0137]
在一些实施例中,使用双重抗生素选择。撤除氯霉素或四环素以去除选择压力可
以提供关于靶向底物、序列特异性和效力的新型信息。例如,对选择或未选择的基因中dsdna的切割可以导致大肠杆菌中的阴性选择,其中观察到选择的基因和未选择的基因两者的耗减。如果crispr-cas系统干扰转录或翻译(例如,通过结合或通过转录物切割),则将仅观察到对于选择的抗性基因中而非选择的抗性基因中的靶标的选择。
[0138]
在一些实施例中,仅使用卡那霉素来确保成功转化包含工程化crispr-cas系统的pet-28a 载体。此实施例适用于含有靶向大肠杆菌必需基因的间隔子的文库,因为除了卡那霉素之外,不需要另外的选择来观察生长变化。在此实施例中,去除氯霉素和四环素依赖性,并且它们在文库中的靶标(如果有的话)提供了关于靶向底物、序列特异性和效力的阴性或阳性信息的另外来源。
[0139]
由于pacyc184质粒包含一组可能影响crispr-cas系统活性的不同特征和序列,因此将来自池化筛选的活性crrna映射至pacyc184上提供了活性模式,该活性模式可以提示不同的活性机制和功能参数。这样,为在异源原核物种中重构新型crispr-cas系统所需的特征可以得到更全面的测试和研究。
[0140]
本文所述的体内池化筛选的关键优点包括:
[0141]
(1)通用性-质粒设计允许表达多种效应子和/或非编码元件;文库克隆策略使得计算预测的crrna的两个转录方向都能表达;
[0142]
(2)活性机制和功能参数的全面测试-评价多样性干扰机制,包括核酸切割;检查特征诸如转录、质粒dna复制的共现;以及可以使用crrna文库的侧接序列来可靠地确定复杂度等价为4n的pam;
[0143]
(3)敏感性-pacyc184是低拷贝质粒,能够对crispr-cas活性具有高敏感性,这是由于即使适度的干扰率也可以消除质粒编码的抗生素抗性;以及
[0144]
(4)效率-优化分子生物学步骤,使得实现速度更快且通量更大的rna测序,并且蛋白质表达样品可以直接从筛选中的存活细胞中获得。
[0145]
将本文所述的新型clust.091979crispr-cas家族使用体内池化筛选进行评价,以评价其操作元件、机制和参数,以及其在其内源细胞环境之外的工程化系统中具有活性和重新编程的能力。
[0146]
crispr效应子活性和修饰
[0147]
在一些实施例中,clust.091979的crispr效应子和rna指导物形成可以包含其他组分的“二元”复合物。在结合与rna指导物中的间隔子序列互补的核酸底物(即,序列特异性底物或靶核酸)后,二元复合物被激活。在一些实施例中,序列特异性底物是双链dna。在一些实施例中,序列特异性底物是单链dna。在一些实施例中,序列特异性底物是单链rna。在一些实施例中,序列特异性底物是双链rna。在一些实施例中,序列特异性要求rna指导物(例如,crrna)中的间隔子序列与靶底物完全匹配。在其他实施例中,序列特异性要求rna指导物(例如,crrna)中的间隔子序列与靶底物的部分(连续或非连续)匹配。
[0148]
在一些实施例中,本发明的crispr效应子在宽范围的ph条件下具有酶活性,例如核酸酶活性。在一些实施例中,核酸酶在从约3.0至约12.0的ph下具有酶活性,例如核酸酶活性。在一些实施例中,crispr效应子在从约4.0至约10.5的ph下具有酶活性。在一些实施例中,crispr效应子在从约5.5至约8.5的ph下具有酶活性。在一些实施例中,crispr效应子在从约6.0至约8.0的ph下具有酶活性。在一些实施例中,crispr效应子在约7.0的ph下具有
酶活性。
[0149]
在一些实施例中,本发明的crispr效应子在从约10℃至约100℃的温度范围下具有酶活性,例如核酸酶活性。在一些实施例中,本发明的crispr效应子在从约20℃至约90℃的温度范围下具有酶活性。在一些实施例中,本发明的crispr效应子在约20℃至约25℃的温度下或在约37℃的温度下具有酶活性。
[0150]
在一些实施例中,二元复合物在结合靶底物后变为激活的。在一些实施例中,激活的复合物展现出“多次周转”活性,由此在作用于(例如,切割)靶底物后,激活的复合物保持在激活状态。在一些实施例中,激活的二元复合物展现出“单周转”活性,由此在作用于靶底物后,二元复合物恢复到非活性状态。在一些实施例中,激活的二元复合物展现出非特异性(即,“伴”)切割活性,由此该复合物切割非靶标核酸。在一些实施例中,非靶标核酸是dna分子(例如,单链或双链dna)。在一些实施例中,非靶标核酸是rna分子(例如,单链或双链rna)。
[0151]
在其中本发明的crispr效应子在靶核酸(例如基因组dna)中诱导双链断裂或单链断裂的一些实施例中,双链断裂可以刺激细胞内源dna修复途径,包括同源定向重组(hdr)、非同源末端连接(nhej)或选择性非同源末端连接(a-nhej)。nhej可以修复裂解的靶核酸,而无需同源模板。此举可以导致一个或多个核苷酸在靶基因座处的缺失或插入。hdr可以用同源模板,诸如供体dna发生。同源模板可以包含与侧接靶核酸裂解位点的序列同源的序列。在一些情况下,hdr可以将外源多核苷酸序列插入至切割靶基因座中。由于nhej和/或hdr引起的对靶dna的修饰可以导致例如突变、缺失、改变、整合、基因矫正、基因替换、基因标记、转基因敲入、基因破坏和/或基因敲除。
[0152]
在一些实施例中,可以将本文所述的crispr效应子融合至一个或多个肽标签,包括his-标签、gst-标签、flag-标签或myc-标签。在一些实施例中,可以将本文所述的crispr效应子融合至可检测部分,诸如荧光蛋白(例如,绿色荧光蛋白或黄色荧光蛋白)。在一些实施例中,将本披露的crispr效应子和/或辅助蛋白融合至肽或非肽部分,该肽或非肽部分允许蛋白质进入或定位至组织、细胞或细胞区域。例如,本披露的crispr效应子可以包含核定位序列(nls),诸如sv40(猿猴病毒40)nls、c-myc nls或其他合适的单颗粒nls。可以将nls融合至crispr效应子的n末端和/或c末端,并且可以将其单独融合(即,单一nls)或串联(例如,2、3、4等个nls的链)。
[0153]
在一些实施例中,将至少一个核输出信号(nes)附接至编码crispr效应子的核酸序列。在一些实施例中,附接c末端和/或n末端nls或nes以用于在真核细胞(例如,人细胞)中的最佳表达和核靶向。
[0154]
在将标签融合至crispr效应子的那些实施例中,这种标签可以有利于crispr效应子的基于亲和力或基于电荷的纯化,该纯化例如通过利用固定的亲和力或离子交换试剂的液相色谱或珠分离进行。作为非限制性实例,本披露的重组crispr效应子包含多组氨酸(his)标签,并且为了纯化,将其装载至包含固定化金属离子的色谱柱上(例如由固定在树脂上的螯合配体螯合的zn
2
、ni
2
、cu
2
离子,该树脂可以是单独制备的树脂或可商购获得的树脂或即用型柱,诸如由马萨诸塞州马尔堡(marlborough)的通用医疗生命科学公司(ge healthcare life sciences)市售的histrap ff柱)。在装载步骤后,任选地例如使用一种或多种合适的缓冲溶液冲洗柱,并且然后使用合适的洗脱缓冲液洗脱加his标签的蛋白质。
替代性地或另外地,如果本披露的重组crispr效应子利用flag-标签,则可以使用业内已知的免疫沉淀方法纯化这种蛋白质。对于本领域技术人员来说,本披露的加标签的crispr效应子或辅助蛋白的其他合适的纯化方法将是明显的。
[0155]
本文所述的蛋白质(例如,crispr效应子或辅助蛋白)可以作为核酸分子或者多肽递送或使用。当使用核酸分子时,编码crispr效应子的核酸分子可以是密码子优化的。核酸可以经密码子优化以用于在任何目的生物体(特别是人细胞或细菌)中使用。例如,核酸可以经密码子优化以用于任何非人真核生物(包括小鼠、大鼠、兔、狗、家畜或非人灵长类动物)。密码子使用表是易于获得的,例如在www.kazusa.orjp/codon/上可获得的“密码子使用数据库(codon usage database)”中,并且这些表可以按多种方式进行改编。参见nakamura等人nucl.acids res.[核酸研究]28:292(2000),将该文献通过援引以其全文并入本文。用于密码子优化特定序列以在特定宿主细胞中表达的计算机算法也是可得的,诸如基因制造(gene forge)(aptagen公司;宾夕法尼亚州雅各布斯(jacobus,pa))。
[0156]
在一些情况下,本披露的编码用于在真核生物(例如,人或其他哺乳动物细胞)细胞中表达的crispr效应子的核酸包含一个或多个内含子,即一个或多个非编码序列,该一个或多个非编码序列在第一端(例如,5’端)包含剪接供体序列并且在第二端(例如,3’端)包含剪接受体序列。任何合适的剪接供体/剪接受体可以用于本披露的各种实施例中,包括但不限于猿猴病毒40(sv40)内含子、β-珠蛋白内含子和合成内含子。替代性地或另外地,本披露的编码crispr效应子或辅助蛋白的核酸可以在dna编码序列的3’端包含转录终止信号,诸如聚腺苷酸化(聚a)信号。在一些情况下,聚a信号很靠近或相邻于内含子(诸如sv40内含子)定位。
[0157]
去激活/失活的crispr效应子
[0158]
可以将本文所述的crispr效应子修饰为具有减弱的核酸酶活性,例如与野生型crispr效应子相比,至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少97%或100%的核酸酶失活。核酸酶活性可以通过本领域已知的若干种方法,例如将突变引入至蛋白质的核酸酶结构域中来减弱。在一些实施例中,鉴定出核酸酶活性的催化残基,并且这些氨基酸残基可以被不同的氨基酸残基(例如,甘氨酸或丙氨酸)取代以减弱核酸酶活性。
[0159]
失活的crispr效应子可以包含一个或多个功能性结构域或与一个或多个功能性结构域相关联(例如,经由融合蛋白、接头肽、“gs”接头等)。这些功能性结构域可以具有各种活性,例如甲基化酶活性、去甲基化酶活性、转录激活活性、转录阻遏活性、转录释放因子活性、组蛋白修饰活性、rna切割活性、dna切割活性、核酸结合活性和开关活性(例如,光诱导的)。在一些实施例中,功能性结构域是kr
ü
ppel相关框(krab)、vp64、vp16、fok1、p65、hsf1、myod1和生物素-apex。
[0160]
一个或多个功能性结构域在失活的crispr效应子上的定位允许功能性结构域的正确空间取向,从而以所归属的功能效应影响靶标。例如,如果功能性结构域是转录激活因子(例如,vp16、vp64或p65),则转录激活因子被放置成允许其影响靶标的转录的空间取向。同样,转录阻遏子被定位成影响靶标的转录,并且核酸酶(例如,fok1)被定位成切割或部分切割靶标。在一些实施例中,功能性结构域位于crispr效应子的n末端。在一些实施例中,功能性结构域位于crispr效应子的c末端。在一些实施例中,将失活的crispr效应子修饰为包
含在n末端处的第一功能性结构域和在c末端处的第二功能性结构域。
[0161]
拆分型酶
[0162]
本披露还提供了一种本文所述的crispr效应子的拆分形式。crispr效应子的拆分形式可能对于递送是有利的。在一些实施例中,crispr效应子被拆分为酶的两个部分,该两个部分一起基本上构成功能性crispr效应子。
[0163]
拆分可以以一个或多个催化结构域不受影响的方式进行。crispr效应子可以起核酸酶的作用,或者可以是失活的酶,这些失活的酶基本上是具有很少或没有催化活性(例如,由于其催化结构域中的一个或多个突变)的rna结合蛋白。
[0164]
在一些实施例中,核酸酶裂片和α-螺旋裂片表达为单独的多肽。尽管这些裂片本身并不相互作用,但rna指导物将它们招募至三元复合物中,该三元复合物重现全长crispr效应子的活性,并且催化位点特异性dna切割。使用经修饰的rna指导物通过阻止二聚化来消除拆分型酶的活性,从而允许诱导型二聚化系统的开发。拆分型酶描述于例如wright等人.“rational design of a split-cas9 enzyme complex[拆分型cas9酶复合物的合理设计],”proc.natl.acad.sci.[美国国家科学院院刊],112.10(2015):2984-2989中,将该文献通过援引以其全文并入本文。
[0165]
在一些实施例中,可以例如通过采用雷帕霉素敏感性二聚化结构域将拆分型酶融合至二聚化配偶体。这允许产生化学诱导型crispr效应子用于crispr效应子活性的时间控制。因此,crispr效应子可以通过拆分成两个片段而成为化学诱导型,并且雷帕霉素敏感性二聚化结构域可以用于crispr效应子的受控重组。
[0166]
拆分点典型地是经由计算机模拟设计的并且克隆至构建体中。在此过程中,可以将突变引入至拆分型酶,并且可以去除非功能性结构域。在一些实施例中,拆分型crispr效应子的两个部分或片段(即,n末端和c末端片段)可以形成包含野生型crispr效应子序列的例如至少70%、至少80%、至少90%、至少95%或至少99%的完全crispr效应子。
[0167]
自激活或自失活酶
[0168]
本文所述的crispr效应子可以被设计为自激活或自失活的。在一些实施例中,crispr效应子是自失活的。例如,可以将靶序列引入至crispr效应子编码构建体中。因此,crispr效应子可以切割靶序列以及编码该酶的构建体,从而自失活其表达。构建自失活crispr系统的方法描述于例如epstein等人,“engineering a self-inactivating crispr system for aav vectors[工程化用于aav载体的自失活crispr系统],”mol.ther.[分子疗法],24(2016):s50中,将该文献通过援引以其全文并入本文。
[0169]
在一些其他实施例中,在弱启动子(例如,7sk启动子)控制下表达的另外rna指导物可以靶向编码crispr效应子的核酸序列以阻止和/或阻断其表达(例如,通过阻止核酸的转录和/或翻译)。用表达crispr效应子、rna指导物和靶向编码crispr效应子的核酸的rna指导物的载体转染细胞可以导致编码crispr效应子的核酸的有效破坏并降低crispr效应子的水平,从而限制基因组编辑活性。
[0170]
在一些实施例中,crispr效应子的基因组编辑活性可以通过哺乳动物细胞中的内源rna特征(例如,mirna)来调节。crispr效应子开关可以通过在编码crispr效应子的mrna的5
′‑
utr中使用mirna互补序列来实现。开关选择性地且有效地响应靶细胞中的mirna。因此,开关可以通过感测异质细胞群体内的内源mirna活性来差异地控制基因组编辑。因此,
开关系统可以为基于细胞内mirna信息的细胞类型选择性基因组编辑和细胞工程化提供框架(hirosawa等人“cell-type-specific genome editing with a microrna-responsive crispr-cas9 switch[利用微rna响应性crispr-cas9开关的细胞类型特异性基因组编辑],”nucl.acids res.[核酸研究],2017年7月27日;45(13):e118)。
[0171]
诱导型crispr效应子
[0172]
crispr效应子可以是诱导型的,例如,光诱导型的或化学诱导型的。此机制允许激活crispr效应子中的功能性结构域。光诱导性可以通过本领域已知的各种方法来实现,例如通过设计这样的融合复合物来实现,其中将cry2phr/cibn配对用于拆分型crispr效应子中(参见例如,konermann等人,“optical control of mammalian endogenous transcription and epigenetic states[哺乳动物内源转录和表观遗传状态的光学控制],”nature[自然],500.7463(2013):472)。化学诱导性可以例如通过设计这样的融合复合物来实现,其中将fkbp/frb(fk506结合蛋白/fkbp雷帕霉素结合结构域)配对用于拆分型crispr效应子中。雷帕霉素是形成融合复合物从而激活crispr效应子所需的(参见例如,zetsche等人,“a split-cas9 architecture for inducible genome editing and transcription modulation[用于诱导型基因组编辑和转录调节的拆分型cas9架构],”nature biotech.[自然生物技术],33.2(2015):139-142)。
[0173]
此外,crispr效应子的表达可以通过诱导型启动子,例如四环素或强力霉素控制的转录激活(tet-on和tet-off表达系统)、激素诱导型基因表达系统(例如,蜕皮激素诱导型基因表达系统)和阿拉伯糖诱导型基因表达系统来调节。当作为rna递送时,rna靶向效应子蛋白的表达可以经由核糖开关来调节,该核糖开关可以感测小分子如四环素(参见例如,goldfless等人,“direct and specific chemical control of eukaryotic translation with a synthetic rna-protein interaction[通过合成的rna-蛋白质相互作用直接和特异性地化学控制真核翻译],”nucl.acids res.[核酸研究],40.9(2012):e64-e64)。
[0174]
诱导型crispr效应子和诱导型crispr系统的各种实施例描述于例如us 8871445、us 20160208243和wo 2016205764中,将这些文献中的每一个通过援引以其全文并入本文。
[0175]
功能性突变
[0176]
可以将各种突变或修饰引入至如本文所述的crispr效应子中,以改善特异性和/或稳健性。在一些实施例中,鉴定出识别原型间隔子相邻基序(pam)的氨基酸残基。可以进一步将本文所述的crispr效应子修饰以识别不同的pam,例如通过用其他氨基酸残基取代识别pam的氨基酸残基。在一些实施例中,crispr效应子可以识别例如5
’‑
nttn-3’、5
’‑
nttr-3’、5
’‑
rttr-3’、5
’‑
tnnt-3’、5
’‑
tnrt-3’、5
’‑
tsrt-3’、5
’‑
tgrt-3’、5
’‑
tnry-3’、5
’‑
ttnr-3’、5
’‑
ttyr-3’、5
’‑
tttr-3’、5
’‑
ttcv-3’、5
’‑
dtyr-3’、5
’‑
wttr-3’、5
’‑
nnr-3’、5
’‑
nyr-3’、5
’‑
yyr-3’、5
’‑
tyr-3’、5
’‑
ttn-3’、5
’‑
ttr-3’、5
’‑
cnt-3’、5
’‑
ngg-3’、5
’‑
bgg-3’或5
’‑
r-3’,其中“n”是任何核苷酸,“b”是c或g或t,“d”是a或g或t,“r”是a或g,“s”是g或c,“v”是a或c或g,“w”是a或t,并且“y”是c或t。
[0177]
在一些实施例中,可以使本文所述的crispr效应子在一个或多个氨基酸残基处突变以修饰一种或多种功能活性。例如,在一些实施例中,使crispr效应子在一个或多个氨基酸残基处突变以修饰其解旋酶活性。在一些实施例中,使crispr效应子在一个或多个氨基酸残基处突变以修饰其核酸酶活性(例如,核酸内切酶活性或核酸外切酶活性)。在一些实
施例中,使crispr效应子在一个或多个氨基酸残基处突变以修饰其与rna指导物功能上相关联的能力。在一些实施例中,使crispr效应子在一个或多个氨基酸残基处突变以改变其与靶核酸功能上相关联的能力。
[0178]
在一些实施例中,本文所述的crispr效应子能够切割靶核酸分子。在一些实施例中,crispr效应子切割靶核酸分子的两条链。然而,在一些实施例中,使crispr效应子在一个或多个氨基酸残基处突变以修饰其切割活性。例如,在一些实施例中,crispr效应子可以包含增加crispr效应子切割靶核酸的能力的一个或多个突变。在另一实例中,在一些实施例中,crispr效应子可以包含使该酶不能切割靶核酸的一个或多个突变。在其他实施例中,crispr效应子可以包含一个或多个突变,由此使得该酶能够切割靶核酸的链(即,切口酶活性)。在一些实施例中,crispr效应子能够切割靶核酸的与跟rna指导物杂交的链互补的链。在一些实施例中,crispr效应子能够切割靶核酸的与rna指导物杂交的链。
[0179]
在一些实施例中,将本文所披露的crispr效应子的一个或多个残基突变为精氨酸部分。在一些实施例中,将本文所披露的crispr效应子的一个或多个残基突变为甘氨酸部分。在一些实施例中,基于本文所披露的crispr效应子的系统发育比对的共有残基对本文所披露的crispr效应子的一个或多个残基进行突变。
[0180]
在一些实施例中,本文所述的crispr效应子可以经工程化以包含一个或多个氨基酸残基的缺失,以减小酶的大小,同时保留一个或多个所期望的功能活性(例如,核酸酶活性和与rna指导物功能上相互作用的能力)。截短型crispr效应子可以有利地与具有负载限制的递送系统组合使用。
[0181]
在一方面,本披露提供了与本文所述的核酸序列至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%相同,同时保持图2中所示的结构域架构的核酸序列。在另一方面,本披露还提供了与本文所述的氨基酸序列至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%相同,同时保持图2中所示的结构域架构的氨基酸序列。
[0182]
在一些实施例中,核酸序列具有至少一部分(例如,至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、20、30、40、50、60、70、80、90或100个核苷酸,例如,连续或非连续核苷酸)与本文所述的序列相同。在一些实施例中,核酸序列具有至少一部分(例如,至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、20、30、40、50、60、70、80、90或100个核苷酸,例如,连续或非连续核苷酸)与本文所述的序列不同。
[0183]
在一些实施例中,氨基酸序列具有至少一部分(例如,至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、20、30、40、50、60、70、80、90或100个氨基酸残基,例如,连续或非连续氨基酸残基)与本文所述的序列相同。在一些实施例中,氨基酸序列具有至少一部分(例如,至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、20、30、40、50、60、70、80、90或100个氨基酸残基,例如,连续或非连续氨基酸残基)与本文所述的序列不同。
[0184]
为了确定两个氨基酸序列、或两个核酸序列的同一性百分比,出于最佳比对的目的将序列比对(例如,可以在第一和第二氨基酸或核酸序列中的一者或两者中引入空位以用于最佳比对,并且出于比较目的可以忽略非同源序列)。一般来讲,出于比较目的而比对
的参考序列的长度应是参考序列长度的至少80%,并且在一些实施例中是参考序列长度的至少90%、95%或100%。然后比较在相应氨基酸位置或核苷酸位置处的氨基酸残基或核苷酸。当第一序列中的位置被与在第二序列中的相应位置相同的氨基酸残基或核苷酸占据时,则这些分子在那个位置是相同的。两个序列之间的同一性百分比是这些序列共享的相同位置的数目的函数,考虑了空位数目和每个空位的长度,需要引入它们以用于两个序列的最佳比对。出于本披露的目的,序列的比较和两个序列之间的一致性百分比的确定可以使用具有12的空位罚分、4的空位延伸罚分和5的移码空位罚分的blossum 62评分矩阵来完成。
[0185]
在一些实施例中,核酸酶包含作为px1x2x3x4f(seq id no:216)列出的序列,其中x1是l或m或i或c或f,x2是y或w或f,x3是k或t或c或r或w或y或h或v,并且x4是i或l或m。在一些实施例中,seq id no:216中列出的序列是n末端序列。在一些实施例中,核酸酶包含作为rx1x2x3l(seq id no:217)列出的序列,其中x1是i或l或m或y或t或f,x2是r或q或k或e或s或t,并且x3是l或i或t或c或m或k。在一些实施例中,核酸酶包含作为nx1yx2(seq id no:218)列出的序列,其中x1是i或l或f并且x2是k或r或v或e。在一些实施例中,核酸酶包含作为kx1x2x3fax4x5kd(seq id no:219)列出的序列,其中x1是t或i或n或a或s或f或v,x2是i或v或l或s,x3是h或s或g或r,x4是d或s或e,并且x5是i或v或m或t或n。在本文所述的任一系统的一些实施例中,seq id no:219的序列是c末端序列。在一些实施例中,核酸酶包含作为lx1nx2(seq id no:220)列出的序列,其中x1是g或s或c或t并且x2是n或y或k或s。在本文所述的任一系统的一些实施例中,seq id no:220的序列是c末端序列。在一些实施例中,核酸酶包含作为px1x2x3x4sqx5ds(seq id no:221)列出的序列,其中x1是s或p或a,x2是y或s或a或p或e或y或q或n,x3是f或y或h,x4是t或s,并且x5是m或t或i。在本文所述的任一系统的一些实施例中,seq id no:221的序列是c末端序列。在一些实施例中,核酸酶包含作为kx1x2vrx3x4qex5h(seq id no:222)列出的序列,其中x1是n或k或w或r或e或t或y,x2是m或r或l或s或k或v或e或t或i或d,x3是l或r或h或p或t或k或q或p或s或a,x4是g或q或n或r或k或e或i或t或s或c,并且x5是r或w或y或k或t或f或s或q。在本文所述的任一系统的一些实施例中,seq id no:222的序列是c末端序列。在一些实施例中,核酸酶包含作为x1ngx2x3x4dx5nx6x7x8n(seq id no:223)列出的序列,其中x1是i或k或v或l,x2是l或m,x3是n或h或p,x4是a或s或c,x5是v或y或i或f或t或n,x6是a或s,x7是s或a或p,并且x8是m或c或l或r或n或s或k或l。在本文所述的任一系统的一些实施例中,seq id no:223的序列是c末端序列。
[0186]
rna和rna指导物修饰
[0187]
在一些实施例中,本文所述的rna指导物包含尿嘧啶(u)。在一些实施例中,本文所述的rna指导物包含胸腺嘧啶(t)。在一些实施例中,本文所述的rna指导物的直接重复序列包含尿嘧啶(u)。在一些实施例中,本文所述的rna指导物的直接重复序列包含胸腺嘧啶(t)。在一些实施例中,根据表2或表8的直接重复序列包含这样的序列,该序列在表2或表8中相应序列中指示为胸腺嘧啶的一个或多个位置中包含尿嘧啶。
[0188]
在一些实施例中,直接重复仅包含在内源crispr阵列中重复的序列的一个拷贝。在一些实施例中,直接重复是相邻于(例如,侧接)内源crispr阵列中发现的一个或多个间隔子序列的全长序列。在一些实施例中,直接重复是相邻于(例如,侧接)内源crispr阵列中
发现的一个或多个间隔子序列的全长序列的一部分(例如,加工部分)。
[0189]
间隔子和直接重复
[0190]
rna指导物的间隔子长度可以在从约15至55个核苷酸的范围内。rna指导物的间隔子长度可以在从约20至45个核苷酸的范围内。在一些实施例中,rna指导物的间隔子长度是至少15个核苷酸、至少16个核苷酸、至少17个核苷酸、至少18个核苷酸、至少19个核苷酸、至少20个核苷酸、至少21个核苷酸或至少22个核苷酸。在一些实施例中,间隔子长度是从15至17个核苷酸、从15至23个核苷酸、从16至22个核苷酸、从17至20个核苷酸、从20至24个核苷酸(例如,20、21、22、23或24个核苷酸)、从23至25个核苷酸(例如,23、24或25个核苷酸)、从24至27个核苷酸、从27至30个核苷酸、从30至45个核苷酸(例如,30、31、32、33、34、35、40或45个核苷酸)、从30或35至40个核苷酸、从41至45个核苷酸、从45至50个核苷酸或更长。
[0191]
在一些实施例中,rna指导物的直接重复长度是至少16个核苷酸,或是从16至20个核苷酸(例如,16、17、18、19或20个核苷酸)。在一些实施例中,rna指导物的直接重复长度是约19至约40个核苷酸。
[0192]
示例性直接重复序列(例如,前crrna(例如,未加工crrna)的直接重复序列或成熟crrna的直接重复序列(例如,加工crrna的直接重复序列))示出于表2中。还参见表8。
[0193]
表2.crrna序列的示例性直接重复序列。
[0194][0195][0196]
在一些实施例中,该crispr相关蛋白包含与seq id no:1的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:57的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:2的氨基酸序列至少80%(例如,
81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:58的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:3的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:59的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:4的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:60的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:10的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:62或seq id no:213的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:14的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:128的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:15的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:63的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:17的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:130的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:18的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:70的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序
列。在一些实施例中,该crispr相关蛋白包含与seq id no:21的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:72的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:22的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:73的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:23的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:74的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:24的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:63的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:27的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:76的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:28的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:77的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:29的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:139的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:31的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:58的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、
id no:86的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:45的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:130的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:46的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:84的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:47的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:87的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:48的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:88的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:51的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:84的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:53的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:84的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:55的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:88的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:56的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、
95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该直接重复序列包含与seq id no:90的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。
[0197]
在一些实施例中,rna指导物包含图3中列出的直接重复序列。例如,在一些实施例中,rna指导物包含图3中所示的共有序列的直接重复或图3中所示的共有序列的一部分。在一些实施例中,rna指导物包含具有作为x1x2tx3x4x5x6x7x8(seq id no:224)列出的序列的直接重复,其中x1是a或c或g,x2是t或c或a,x3是t或g或a,x4是t或g,x5是t或g或a,x6是g或t或a,x7是t或g或a,并且x8是a或g或t。例如,在一些实施例中,rna指导物包含具有作为attgttgda(seq id no:225)列出的序列的直接重复。在一些实施例中,seq id no:224在直接重复的5’端的近侧。在一些实施例中,seq id no:225在直接重复的5’端的近侧。在一些实施例中,rna指导物包含具有作为x1x2x3x4x5x6x7x8x9(seq id no:226)列出的序列的直接重复,其中x1是t或c或a,x2是t或a或g,x3是t或c或a,x4是t或a,x5是t或a或g,x6是t或a,x7是a或t,x8是a或g或c或t,并且x9是g或a或c。例如,在一些实施例中,rna指导物包含具有作为ttttwtarg(seq id no:227)列出的序列的直接重复。在一些实施例中,rna指导物包含具有作为x1x2x3ac(seq id no:228)列出的序列的直接重复,其中x1是a或c或g,x2是c或a,并且x3是a或c。例如,在一些实施例中,rna指导物包含具有作为acaac(seq id no:229)列出的序列的直接重复。在一些实施例中,seq id no:228在直接重复的3’端的近侧。在一些实施例中,seq id no:229在直接重复的3’端的近侧。
[0198]
在一些实施例中,rna指导物的间隔子结合相邻于表3的pam序列的靶核酸。例如,在一些实施例中,本文所披露的效应子和rna指导物的复合物结合相邻于如表3中所指示的pam序列的靶核酸。
[0199]
表3.对应于clust.091979效应子的pam序列。
[0200]
[0201]
[0202][0203]
在一些实施例中,rna指导物进一步包含tracrrna。在一些实施例中,不需要tracrrna(例如,tracrrna是任选的)。在一些实施例中,tracrrna是表9中所示的非编码序列的一部分。例如,在一些实施例中,tracrrna是表4的序列。
[0204]
表4.示例性tracrrna序列。
[0205]
[0206]
[0207]
[0208]
[0209]
[0210][0211]
在一些实施例中,该crispr相关蛋白包含与seq id no:1的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:152、seq id no:153或seq id no:154的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:2的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:155、seq id no:156、seq id no:157或seq id no:158的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:3的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:159、seq id no:160或seq id no:161的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:14的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:162的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:17的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:163、seq id no:164、seq id no:165或seq id no:166的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:18的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、
98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:167或seq id no:168的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:21的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:169、seq id no:170或seq id no:171的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:22的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:172、seq id no:173、seq id no:174或seq id no:175的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:23的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:176、seq id no:177、seq id no:178或seq id no:179的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:27的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:180或seq id no:181的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:29的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:182、seq id no:183或seq id no:184的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:31的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:185、seq id no:186、seq id no:187或seq id no:188的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:32的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:189或seq id no:190的核苷酸序列至少
80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:36的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:182、seq id no:183或seq id no:184的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:38的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:189或seq id no:190的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:39的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:185、seq id no:186、seq id no:187或seq id no:188的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:41的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:191、seq id no:192、seq id no:193或seq id no:194的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:43的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:197、seq id no:198或seq id no:199的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:44的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:195或seq id no:196的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:45的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:163、seq id no:164、seq id no:165或seq id no:166的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同
的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:48的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:200、seq id no:201或seq id no:202的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:52的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:197、seq id no:198或seq id no:199的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:55的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:200、seq id no:201或seq id no:202的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。在一些实施例中,该crispr相关蛋白包含与seq id no:56的氨基酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的氨基酸序列,并且该tracrrna序列包含与seq id no:203或seq id no:204的核苷酸序列至少80%(例如,81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)相同的核苷酸序列。
[0212]
rna指导序列可以按这样的方式进行修饰,该方式允许形成crispr复合物并成功结合靶标,而同时不允许成功的核酸酶活性(即,没有核酸酶活性/没有引起插入缺失)。这些经修饰的指导序列被称为“死指导物”或“死指导序列”。这些死指导物或死指导序列对于核酸酶活性而言可以是催化上失活的或构象上失活的。死指导序列典型地比导致活性rna切割的对应指导序列短。在一些实施例中,死指导物比具有核酸酶活性的对应rna指导物短5%、10%、20%、30%、40%或50%。rna指导物的死指导序列可以具有从13至15个核苷酸的长度(例如,13、14或15个核苷酸的长度)、从15至19个核苷酸的长度、或从17至18个核苷酸的长度(例如,17个核苷酸的长度)。
[0213]
因此,在一方面,本披露提供了非天然存在的或工程化的crispr系统,这些系统包括如本文所述的功能性clust.091979crispr效应子、和rna指导物,其中rna指导物包含死指导序列,由此rna指导物能够与靶序列杂交,使得将crispr系统指导至细胞中目的基因组基因座而没有可检测的切割活性。对死指导物的详细描述是描述于例如wo 2016094872中,将该文献通过援引以其全文并入本文。
[0214]
诱导型rna指导物
[0215]
rna指导物可以作为诱导型系统的组分产生。这些系统的诱导型性质允许对基因编辑或基因表达的时空控制。在一些实施例中,用于诱导型系统的刺激包括例如电磁辐射、声能、化学能和/或热能。
[0216]
在一些实施例中,rna指导物的转录可以通过诱导型启动子,例如四环素或强力霉素控制的转录激活(tet-on和tet-off表达系统)、激素诱导型基因表达系统(例如,蜕皮激素诱导型基因表达系统)和阿拉伯糖诱导型基因表达系统来调节。诱导型系统的其他实例包括例如小分子双杂交转录激活系统(fkbp,aba等)、光诱导型系统(光敏色素、lov结构域或隐花色素)或光诱导型转录效应子(lite)。这些诱导型系统描述于例如wo 2016205764和us 8795965中,将这些文献中的每一个通过援引以其全文并入本文。
[0217]
化学修饰
[0218]
可以将化学修饰应用于rna指导物的磷酸骨架、糖和/或碱基。骨架修饰(诸如硫代磷酸酯)修饰磷酸骨架上的电荷并且有助于寡核苷酸的递送和核酸酶抗性(参见例如,eckstein,“phosphorothioates,essential components of therapeutic oligonucleotides[硫代磷酸酯,治疗性寡核苷酸的基本组分],”nucl.acid ther.[核酸疗法],24(2014),第374-387页);糖的修饰(诸如2
’‑
o-甲基(2
’‑
ome)、2
’‑
f和锁核酸(lna))增强碱基配对和核酸酶抗性两者(参见例如,allerson等人“fully 2
‘‑
modified oligonucleotide duplexes with improved in vitro potency and stability compared to unmodified small interfering rna[与未修饰的小干扰rna相比,完全2
‘
饰的寡核苷酸双链体具有改善的体外效力和稳定性],”j.med.chem.[医药化学杂志],48.4(2005):901-904)。化学修饰的碱基(诸如2-硫尿苷或n6-甲基腺苷等)可以允许更强或更弱的碱基配对(参见例如,bramsen等人,“development of therapeutic-grade small interfering rnas by chemical engineering[通过化学工程化开发治疗级小分子干扰rna],”front.genet.[遗传学前沿],2012年8月20日;3:154)。另外,rna适于5’端和3’端两者与多种功能性部分缀合,该多种功能性部分包括荧光染料、聚乙二醇或蛋白质。
[0219]
可以将广泛多种的修饰应用于化学合成的rna指导物分子。例如,用2
’‑
ome修饰寡核苷酸以改善核酸酶抗性可以改变沃森-克里克碱基配对的结合能。此外,2
’‑
ome修饰可以影响寡核苷酸与转染试剂、蛋白质或细胞中任何其他分子相互作用的方式。这些修饰的效果可以通过经验测试来确定。
[0220]
在一些实施例中,rna指导物包含一个或多个硫代磷酸酯修饰。在一些实施例中,rna指导物包含一个或多个锁核酸以用于增强碱基配对和/或增加核酸酶抗性。
[0221]
这些化学修饰的概述可以见于例如kelley等人,“versatility of chemically synthesized guide rnas for crispr-cas9 genome editing[用于crispr-cas9基因组编辑的化学合成的指导rna的通用性],”j.biotechnol.[生物技术杂志]2016年9月10日;233:74-83;wo 2016205764;以及us 8795965中,将这些文献中的每一个通过援引以其全文并入。
[0222]
序列修饰
[0223]
本文所述的rna指导物、tracrrna和crrna的序列和长度可以是优化的。在一些实施例中,rna指导物的优化长度可以通过鉴定tracrrna和/或crrna的加工形式,或者通过rna指导物、tracrrna、crrna和tracrrna四元环的经验长度研究来确定。
[0224]
rna指导物还可以包含一个或多个适配体序列。适配体是可以结合特定靶分子的寡核苷酸或肽分子。适配体可以对基因效应子、基因激活子或基因阻遏子具有特异性。在一些实施例中,适配体可以对蛋白质具有特异性,该蛋白质又特异性于并招募/结合特定基因
效应子、基因激活子或基因阻遏子。效应子、激活子或阻遏子可以以融合蛋白的形式存在。在一些实施例中,rna指导物具有两个或更多个对相同衔接蛋白具有特异性的适配体序列。在一些实施例中,两个或更多个适配体序列对不同的衔接蛋白具有特异性。衔接蛋白可以包括例如ms2、pp7、qβ、f2、ga、fr、jp501、m12、r17、bz13、jp34、jp500、ku1、m11、mx1、tw18、vk、sp、fi、id2、nl95、tw19、ap205、φcb5、φcb8r、φcb12r、φcb23r、7s和prr1。因此,在一些实施例中,适配体选自特异性结合如本文所述的任一种衔接蛋白的结合蛋白。在一些实施例中,适配体序列是ms2环。适配体的详细描述可以见于例如nowak等人,“guide rna engineering for versatile cas9 functionality[针对通用性cas9功能的指导rna工程化],”nucl.acid.res.[核酸研究],2016年11月16日;44(20):9555-9564;和wo 2016205764中,将这些文献中的每一个通过援引以其全文并入本文。
[0225]
指导物:靶序列匹配要求
[0226]
在crispr系统中,指导序列与其相应的靶序列之间的互补程度可以是约50%、60%、75%、80%、85%、90%、95%、97.5%、99%或100%。为了减少脱靶相互作用,例如为了减少指导物与具有低互补性的靶序列相互作用,可以将突变引入至crispr系统,使得crispr系统可以区分具有大于80%、85%、90%或95%互补性的靶序列和脱靶序列。在一些实施例中,互补程度是从80%至95%、例如约83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%或95%(例如,区分具有18个核苷酸的靶标与具有1、2或3个错配的18个核苷酸的脱靶)。因此,在一些实施例中,指导序列与其相应的靶序列之间的互补程度大于94.5%、95%、95.5%、96%、96.5%、97%、97.5%、98%、98.5%、99%、99.5%或99.9%。在一些实施例中,互补程度是100%。
[0227]
在本领域已知,不需要完全的互补性,前提是有足够的互补性发挥作用。可以通过引入错配(例如,间隔子序列与靶序列之间的一个或多个错配,诸如1或2个错配(包括沿着间隔子/靶标的错配的位置))来利用对切割效率的调节。错配(例如,双错配)位于越中心(即,不在3’或5’端处);切割效率受到的影响越大。因此,通过选择沿着间隔子序列的错配位置,可以调节切割效率。例如,如果期望靶标的小于100%的切割率(例如,在细胞群体中),则间隔子序列中可以引入间隔子序列与靶序列之间的1或2个错配。
[0228]
crispr系统的使用方法
[0229]
本文所述的crispr系统具有广泛多种的效用,包括修饰(例如,缺失、插入、易位、失活或激活)多种细胞类型中的靶多核苷酸。crispr系统在以下中具有广谱的应用:例如dna/rna检测(例如,特异性高敏感性酶促报告子解锁(sherlock))、跟踪和标记核酸、富集测定(从背景中提取所期望序列)、检测循环肿瘤dna、制备下一代文库、药物筛选、疾病诊断和预后以及治疗各种遗传障碍。
[0230]
dna/rna检测
[0231]
在一方面,本文所述的crispr系统可以用于dna/rna检测中。单效应子rna指导的dna酶可以用crispr rna(crrna)重新编程,以为特异性单链dna(ssdna)感测提供平台。激活的v型单效应子dna指导的dna酶在识别其dna靶标后参与附近的非靶向ssdna的“伴”切割。此crrna编程的伴切割活性允许crispr系统通过经标记的ssdna的非特异性降解来检测特定dna的存在。
[0232]
伴ssdna活性可以在dna检测应用中与报告子组合,这些检测应用诸如称为dna核
酸内切酶靶向crispr反式报告子(detectr)方法的方法,该方法实现了对dna检测的渺摩尔敏感性(参见例如,chen等人,science[科学],360(6387):436-439,2018),将该文献通过援引以其全文并入本文。使用本文所述的酶的一个应用是在体外环境中降解非特异性ssdna。也可以将连接荧光团和淬灭剂的“报告子”ssdna分子与未知dna样品(单链或者双链)一起添加至体外系统中。在识别未知dna片段中的靶序列后,效应子复合物切割报告子ssdna,从而产生荧光读出。
[0233]
在其他实施例中,sherlock方法(特异性高敏感性酶促报告子解锁)还提供基于报告子ssdna的核酸扩增和伴切割的具有渺摩尔(或单分子)敏感性的体外核酸检测平台,从而允许实时检测靶标。在sherlock中使用crispr的方法详细描述于例如gootenberg等人“nucleic acid detection with crispr-cas13a/c2c2[使用crispr-cas13a/c2c2进行核酸检测],”science[科学],356(6336):438-442(2017)中,将该文献通过援引以其全文并入本文。
[0234]
在一些实施例中,本文所述的crispr系统可以用于多重复用的误差稳健性荧光原位杂交(merfish)中。这些方法描述于例如chen等人,“spatially resolved,highly multiplexed rna profiling in single cells[在单一细胞中进行空间分辨的高度复用的rna谱分析],”science[科学],2015年4月24日;348(6233):aaa6090中,将该文献通过援引以其全文并入本文。
[0235]
核酸的跟踪和标记
[0236]
细胞过程依赖于蛋白质、rna和dna之间的分子相互作用网络。准确检测蛋白质-dna和蛋白质-rna的相互作用是理解此类过程的关键。体外邻近标记技术采用亲和标签与报告基团(例如,可光激活基团)的组合,以在体外标记目的蛋白或rna附近的多肽和rna。在uv辐照后,可光激活基团与跟加标签的分子很靠近的蛋白质和其他分子反应,从而标记它们。随后可以回收和鉴定经标记的相互作用分子。rna靶向效应子蛋白例如可以用于将探针靶向至选择的rna序列。这些应用还可以应用于疾病或难以培养的细胞类型的体内成像的动物模型中。核酸的跟踪和标记方法描述于例如us 8795965;wo 2016205764;和wo 2017070605中,将这些文献中的每一个通过援引以其全文并入本文。
[0237]
高通量筛选
[0238]
本文所述的crispr系统可以用于制备下一代测序(ngs)文库。例如,为了产生具有成本效益的ngs文库,可以将crispr系统用于破坏靶基因的编码序列,并且可以同时通过下一代测序(例如,在离子激流(ion torrent)pgm系统上)筛选crispr效应子转染的克隆。关于如何制备ngs文库的详细描述可见于例如bell等人,“a high-throughput screening strategy for detecting crispr-cas9 induced mutations using next-generation sequencing[用于使用下一代测序检测crispr-cas9诱导的突变的高通量筛选策略],”bmc genomics[bmc基因组学],15.1(2014):1002中,将该文献通过援引以其全文并入本文。
[0239]
工程化细胞
[0240]
微生物(例如,大肠杆菌、酵母和微藻)广泛用于合成生物学。合成生物学的发展具有广泛的效用,包括各种临床应用。例如,可编程的crispr系统可以用于拆分具有用于靶向细胞死亡的毒性结构域的蛋白质,例如使用癌症相关联rna作为靶转录物。此外,涉及蛋白质-蛋白质相互作用的途径可以在使用例如具有适当效应子诸如激酶或酶的融合复合物的
合成生物系统中受到影响。
[0241]
在一些实施例中,可以将靶向噬菌体序列的rna指导序列引入至微生物中。因此,本披露还提供了针对噬菌体感染“接种”微生物(例如,生产菌株)的方法。
[0242]
在一些实施例中,本文所提供的crispr系统可以用于工程化微生物,例如以改善产率或改善发酵效率。例如,本文所述的crispr系统可以用于工程化微生物(诸如酵母),以从可发酵糖产生生物燃料或生物聚合物,或降解来源于作为可发酵糖来源的农业废弃物的植物来源木质纤维素。更具体地,本文所述的方法可以用于修饰生物燃料生产所需的内源基因的表达和/或修饰可能干扰生物燃料合成的内源基因。这些工程化微生物的方法描述于例如verwaal等人,“crispr/cpf1 enables fast and simple genome editing of saccharomyces cerevisiae[crispr/cpf1实现对酿酒酵母的快速且简单的基因组编辑],”yeast[酵母],2017年9月8日doi:10.1002/yea.3278;以及hlavova等人,“improving microalgae for biotechnology—from genetics to synthetic biology[改善生物技术中的微藻-从遗传学到合成生物学],”biotechnol.adv.[生物技术进展],2015年11月1日;33:1194-203中,将这些文献中的每一个通过援引以其全文并入本文。
[0243]
在一些实施例中,本文所提供的crispr系统可以用于工程化真核细胞或真核生物。例如,本文所述的crispr系统可以用于工程化真核细胞,这些真核细胞不限于植物细胞、真菌细胞、哺乳动物细胞、爬行动物细胞、昆虫细胞、禽细胞、鱼细胞、寄生虫细胞、节肢动物细胞、无脊椎动物细胞、脊椎动物细胞、啮齿动物细胞、小鼠细胞、大鼠细胞、灵长类动物细胞、非人灵长类细胞或人细胞。在一些实施例中,真核细胞在体外培养物中。在一些实施例中,真核细胞是体内的。在一些实施例中,真核细胞是离体的。
[0244]
在一些实施例中,细胞来源于细胞系。用于组织培养的多种多样的细胞系是本领域已知的。细胞系的实例包括但不限于293t、mf7、k562、hela及其转基因品种。细胞系可从本领域技术人员已知的多种来源获得(例如,参见美国典型培养物保藏中心(atcc)(弗吉尼亚州马纳萨斯(manassas,va.)))。在一些实施例中,将用一种或多种核酸(诸如编码核酸酶多肽的载体和rna指导物)转染的细胞用于建立包含一种或多种载体来源序列的新细胞系以建立包含对靶核酸或靶基因座的修饰的新细胞系。在一些实施例中,细胞是永生或永生化细胞。
[0245]
在一些实施例中,细胞是原代细胞。在一些实施例中,细胞是干细胞,诸如全能干细胞(例如,万能)、多能干细胞、多潜能干细胞、寡能干细胞或单能干细胞。在一些实施例中,细胞是诱导多能干细胞(ipsc)或来源于ipsc。在一些实施例中,细胞是分化细胞。例如,在一些实施例中,分化细胞是肌肉细胞(例如,肌细胞)、脂肪细胞(例如,脂细胞)、骨细胞(例如,成骨细胞、骨细胞、破骨细胞)、血细胞(例如,单核细胞、淋巴细胞、嗜中性粒细胞、嗜酸性粒细胞、嗜碱性粒细胞、巨噬细胞、红细胞或血小板)、神经细胞(例如,神经元)、上皮细胞、免疫细胞(例如,淋巴细胞、嗜中性粒细胞、单核细胞或巨噬细胞)、肝脏细胞(例如,肝细胞)、成纤维细胞或性细胞。在一些实施例中,细胞是终末分化细胞。例如,在一些实施例中,终末分化细胞是神经元细胞、脂细胞、心肌细胞、骨骼肌细胞、表皮细胞或肠道细胞。在一些实施例中,细胞是哺乳动物细胞(例如,人细胞或鼠类细胞)。在一些实施例中,鼠类细胞来源于野生型小鼠、免疫抑制小鼠或疾病特异性小鼠模型。
[0246]
基因驱动
[0247]
基因驱动是一种现象,在这种现象中,特定的基因或一组基因的遗传被有利地偏向。本文所述的crispr系统可以用于构建基因驱动。例如,crispr系统可以被设计为靶向并破坏基因的特定等位基因,从而引起细胞拷贝第二等位基因以固定序列。因为拷贝,第一等位基因将转换成第二等位基因,增加了第二等位基因传给后代的机会。关于如何使用本文所述的crispr系统以构建基因驱动的详细方法描述于例如hammond等人,“a crispr-cas9 gene drive system targeting female reproduction in the malaria mosquito vector anopheles gambiae[靶向疟蚊载体冈比亚按蚊中雌性繁殖的crispr-cas9基因驱动系统],”nat.biotechnol.[自然生物技术],2016年1月;34(1):78-83中,将该文献通过援引以其全文并入本文。
[0248]
池化筛选
[0249]
如本文所述,池化crispr筛选是用于鉴定参与诸如细胞增殖、药物抗性和病毒感染等生物学机制的基因的有力工具。用本文所述的rna指导物编码载体文库成批转导细胞,并且在应用选择性激发之前和之后测量grna的分布。池化crispr筛选对于影响细胞存活和增殖的机制工作良好,它们可以扩展至测量单独基因的活性(例如,通过使用工程化报告细胞系)。阵列crispr筛选(其中一次只靶向一个基因)使得能够使用rna-seq作为读出。在一些实施例中,如本文所述的crispr系统可以用于单细胞crispr筛选中。关于池化crispr筛选的详细描述可见于例如datlinger等人,“pooled crispr screening with single-cell transcriptome read-out[具有单细胞转录组读出的池化crispr筛选],”nat.methods.[自然方法],2017年3月;14(3):297-301中,将该文献通过援引以其全文并入本文。
[0250]
饱和诱变(“过度攻击(bashing)”)
[0251]
本文所述的crispr系统可以用于原位饱和诱变。在一些实施例中,池化的rna指导物文库可以用于对特定基因或调控元件执行原位饱和诱变。此类方法可以揭示这些基因或调控元件(例如,增强子)的关键的最小特征和离散脆弱性。这些方法描述于例如canver等人,“bcl11a enhancer dissection by cas9-mediated in situ saturating mutagenesis[通过cas9介导的原位饱和诱变进行bcl11a增强子解析],”nature[自然],2015年11月12日;527(7577):192-7中,将该文献通过援引以其全文并入本文。
[0252]
治疗性应用
[0253]
在一些实施例中,本文所述的crispr系统可以用于编辑靶核酸以修饰靶核酸(例如,通过插入、缺失或突变一个或多个氨基酸残基)。例如,在一些实施例中,本文所述的crispr系统包括包含所期望核酸序列的外源供体模板核酸(例如,dna分子或rna分子)。在解析到用本文所述的crispr系统诱导的切割事件后,细胞的分子机器可以在修复和/或解决切割事件中利用外源供体模板核酸。替代性地,细胞的分子机器可以在修复和/或解决切割事件中利用内源模板。在一些实施例中,本文所述的crispr系统可以用于修饰靶核酸,从而导致插入、缺失和/或点突变。在一些实施例中,插入是无痕插入(即,在解析到切割事件后将预期的核酸序列插入至靶核酸中未导致另外的非预期核酸序列)。供体模板核酸可以是双链或单链核酸分子(例如,dna或rna)。设计外源供体模板核酸的方法描述于例如wo 2016094874中,将该文献的全部内容通过援引明确并入本文。
[0254]
在另一方面,本披露提供了本文所述的系统在选自下组的方法中的用途,该组由以下组成:rna序列特异性干扰;rna序列特异性基因调控;筛选rna、rna产物、lncrna、非编
码rna、核rna或mrna;诱变;抑制rna剪接;荧光原位杂交;育种;诱导细胞休眠;诱导细胞周期停滞;减少细胞生长和/或细胞增殖;诱导细胞无变应性;诱导细胞凋亡;诱导细胞坏死;诱导细胞死亡;或诱导程序化细胞死亡。
[0255]
本文所述的crispr系统可以具有各种治疗性应用。在一些实施例中,新的crispr系统可以用于治疗各种疾病和障碍,例如遗传障碍(例如,单基因疾病)或可以通过核酸酶活性(例如,pcsk9靶向或bcl11a靶向)治疗的疾病。在一些实施例中,将在此所述的方法用于治疗受试者,诸如哺乳动物,诸如人患者。哺乳动物受试者也可以是驯养哺乳动物,诸如狗、猫、马、猴、兔、大鼠、小鼠、牛、山羊或绵羊。
[0256]
这些方法可以包括具有感染性的病症或疾病,并且其中感染原选自下组,该组由以下组成:人类免疫缺陷病毒(hiv)、单纯疱疹病毒-1(hsv1)和单纯疱疹病毒-2(hsv2)。
[0257]
在一方面,本文所述的crispr系统可以用于治疗由rna、毒性rna和/或突变型rna(例如,剪接缺陷或截短)的过表达引起的疾病。例如,毒性rna的表达可以与核内含物的形成以及脑、心脏或骨骼肌的迟发型退行性变化相关联。在一些实施例中,障碍是肌强直性营养不良。在肌强直性营养不良中,毒性rna的主要致病作用是螯合结合蛋白并且损害选择性剪接的调控(参见例如,osborne等人,“rna-dominant diseases[rna显性疾病],”hum.mol.genet.[人类分子遗传学],2009年4月15日;18(8):1471-81)。肌强直性营养不良(肌营养不良性肌强直症(dm))对于遗传学家而言是特别受关注的,因为它产生了极其广泛范围的临床特征。dm的经典形式(现称为dm 1型(dm1))是由编码细胞溶质蛋白激酶的基因dmpk的3'-非翻译区(utr)中ctg重复的扩增引起的。如本文所述的crispr系统可以靶向过表达的rna或毒性rna(例如,dmpk基因)或者dm1骨骼肌、心脏或脑中的任何错误调控的选择性剪接。
[0258]
本文所述的crispr系统还可以靶向影响引起各种疾病的rna依赖性功能的反式作用突变,这些各种疾病例如像普拉德威利综合征(prader willi syndrome)、脊髓性肌萎缩症(sma)和先天性角化不良。可以使用本文所述的crispr系统治疗的疾病列表汇总于cooper等人,“rna and disease[rna和疾病],”cell[细胞],136.4(2009):777-793和wo 2016205764中,将这些文献中的每一个通过援引以其全文并入本文。
[0259]
本文所述的crispr系统也可以用于治疗各种tau蛋白病,包括例如原发性和继发性tau蛋白病,诸如原发性年龄相关tau蛋白病(part)/神经元纤维缠结(nft)突出的老年性痴呆(其中nft与阿尔茨海默病(ad)中看到的那些相似,但没有斑块)、拳击员痴呆(慢性创伤性脑病)和进展性核上性麻痹。tau蛋白病的可用列表和治疗这些疾病的方法描述于例如wo 2016205764中,将该文献通过援引以其全文并入本文。
[0260]
本文所述的crispr系统也可以用于靶向破坏可能引起剪接缺陷和疾病的顺式作用剪接密码的突变。这些疾病包括例如由缺失smn1基因引起的运动神经元退行性疾病(例如,脊髓性肌萎缩症)、杜氏(duchenne)肌营养不良(dmd)、与染色体17有关联的额颞叶痴呆和帕金森综合征(ftdp-17)以及囊性纤维化。
[0261]
本文所述的crispr系统可以进一步用于抗病毒活性、特别是抗rna病毒。效应子蛋白可以使用经选择以靶向病毒rna序列的合适rna指导物靶向病毒rna。
[0262]
此外,可以使用体外rna感测测定来检测特定rna底物。rna靶向效应子蛋白可以用于活细胞中的基于rna的感测。应用的实例是通过感测例如疾病特异性rna进行的诊断。
[0263]
本文所述的crispr系统的治疗性应用的详细描述可见于例如us 8795965、ep 3009511、wo 2016205764和wo 2017070605中,将这些文献中的每一个通过援引以其全文并入本文。
[0264]
在植物中的应用
[0265]
本文所述的crispr系统在植物中具有广泛多种的效用。在一些实施例中,crispr系统可以用于工程化植物基因组(例如,改善产量、制备具有所期望翻译后修饰的产物、或引入用于生产工业产物的基因)。在一些实施例中,crispr系统可以用于将所期望的性状引入至植物(例如,对基因组进行或不进行可遗传修饰),或调控植物细胞或整株植物中内源基因的表达。
[0266]
在一些实施例中,crispr系统可以用于鉴定、编辑和/或沉默编码特定蛋白质(例如,过敏性蛋白质(例如,花生、大豆、扁豆、豌豆、四季豆和绿豆中的过敏性蛋白质))的基因。关于如何鉴定、编辑和/或沉默编码蛋白质的基因的详细描述是描述于例如nicolaou等人,“molecular diagnosis of peanut and legume allergy[花生和豆类过敏的分子诊断],”curr.opin.allergy clin.immunol.[过敏和临床免疫学新见],11(3):222-8(2011)和wo 2016205764中,将这些文献中的每一个通过援引以其全文并入本文。
[0267]
crispr系统的递送
[0268]
通过本披露和本领域中的知识,本文所述的crispr系统、其组分、其核酸分子或者编码或提供其组分的核酸分子可以通过各种递送系统诸如载体(例如,质粒或病毒递送载体)来递送。本文所披露的crispr效应子和/或任何rna(例如,rna指导物)可以使用合适的载体,例如质粒或病毒载体来递送,这些病毒载体诸如腺相关病毒(aav)、慢病毒、腺病毒和其他病毒载体或其组合。可以将效应子和一种或多种rna指导物包装至一种或多种载体,例如质粒或病毒载体中。
[0269]
在一些实施例中,通过例如以下方式将载体(例如,质粒或病毒载体)递送至目的组织:肌肉内注射、静脉内施用、经皮施用、鼻内施用、口服施用或粘膜施用。这种递送可以是经由单剂量或者多剂量进行的。本领域技术人员应理解的是,本文有待递送的实际剂量可以在很大程度上根据多种因素而变化,该多种因素包括但不限于载体选择、靶细胞、生物体、组织、有待治疗的受试者的一般状况、所寻求的转化/修饰的程度、施用途径、施用方式和所寻求的转化/修饰的类型。
[0270]
在某些实施例中,递送是经由腺病毒进行的,这些腺病毒可以是含有至少1x105个腺病毒颗粒(也称为颗粒单位,pu)的一个剂量。在一些实施例中,剂量优选地是至少约1x106个颗粒、至少约1x107个颗粒、至少约1x108个颗粒和至少约1x109个颗粒的腺病毒。递送方法和剂量描述于例如wo 2016205764和us 8454972中,将这些文献中的每一个通过援引以其全文并入本文。
[0271]
在一些实施例中,递送是经由质粒进行的。剂量可以是足以引发应答的质粒数量。在一些情况下,质粒组合物中质粒dna的合适量可以是从约0.1至约2mg。质粒通常包括(i)启动子;(ii)可操作地连接至启动子、编码核酸靶向crispr效应子的序列;(iii)选择性标记物;(iv)复制起点;以及(v)在(ii)的下游并可操作地连接至(ii)的转录终止子。质粒还可以编码crispr复合物的rna组分,但是这些组分中的一种或多种可以代替地编码在不同的载体上。施用频率在医学或兽医学从业者(例如,医师、兽医师)或本领域技术人员的范围
之内。
[0272]
在另一个实施例中,递送是经由脂质体或脂质转染配制品等进行的,并且可以通过本领域技术人员已知的方法制备。此类方法描述于例如wo 2016205764、us 5593972、us 5589466和us 5580859中,将这些文献中的每一个通过援引以其全文并入本文。
[0273]
在一些实施例中,递送是经由纳米颗粒或外来体进行的。例如,外来体已显示在递送rna方面是特别有用的。
[0274]
将本文所述的crispr系统的一种或多种组分引入至细胞的另外手段是通过使用细胞穿透肽(cpp)。在一些实施例中,将细胞穿透肽连接至crispr效应子。在一些实施例中,将crispr效应子和/或rna指导物偶联至一种或多种cpp以便转运至细胞(例如,植物原生质体)中。在一些实施例中,将crispr效应子和/或一种或多种rna指导物由偶联至用于细胞递送的一种或多种cpp的一个或多个环状或非环状dna分子编码。
[0275]
cpp是来源于蛋白质或嵌合序列的少于35个氨基酸的短肽,这些短肽能够以非受体依赖性方式将生物分子跨细胞膜转运。cpp可以是阳离子肽、具有疏水性序列的肽、两亲性肽、具有富含脯氨酸且抗微生物的序列的肽和嵌合肽或二分肽。cpp的实例包括例如tat(其是l型hiv病毒复制所需的核转录激活蛋白)、穿膜肽、卡波西成纤维细胞生长因子(fgf)信号肽序列、整合素β3信号肽序列、多精氨酸肽args序列、富含鸟嘌呤的分子转运蛋白和甜箭肽。cpp和使用它们的方法描述于例如等人,“prediction of cell-penetrating peptides[细胞穿透肽的预测],”methods mol.biol.[分子生物学方法],2015;1324:39-58;ramakrishna等人,“gene disruption by cell-penetrating peptide-mediated delivery of cas9 protein and guide rna[通过细胞穿透肽介导的cas9蛋白和指导rna递送进行的基因破坏],”genome res.[基因组研究],2014年6月;24(6):1020-7;以及wo 2016205764中,将这些文献中的每一个通过援引以其全文并入本文。
[0276]
用于本文所述的crispr系统的各种递送方法还描述于例如us 8795965、ep 3009511、wo 2016205764和wo 2017070605中,将这些文献中的每一个通过援引以其全文并入本文。
[0277]
实例
[0278]
本发明在以下实例中进一步描述,这些实例不限制权利要求中所述的本发明的范围。
[0279]
实例1-对clust.091979crispr-cas系统的组分的鉴定
[0280]
使用上文所述的计算方法鉴定此蛋白质家族。clust.091979系统包括与从环境收集的未培养宏基因组序列中发现的crispr系统相关联的单效应子,这些环境不限于肠道、牛肠道、人肠道、羊肠道、陆生、粪便和哺乳动物消化系统环境(表5)。示例性clust.091979效应子包括下表5和表6中所示的那些。将seq id no:1-4、14、15、17-19、21-25、27-33、35-49、51-56中列出的效应子序列比对以鉴定序列相似性的区域,如图1a-1l中所示。条形图描绘了序列相似性,其中最高条指示具有最高序列相似性的残基。序列相似性的非限制性区域示出于表7中。序列相似性的区域表明,本文所披露的效应子是具有代表核酸酶的保守c末端ruvc结构域的家族。
[0281]
表51.代表性clust.091979效应子蛋白
[0282]
[0283]
[0284][0285]
表62.代表性clust.091979效应子蛋白的氨基酸序列
[0286]
[0287]
[0288]
[0289]
[0290]
[0291]
[0292]
[0293]
[0294]
[0295]
[0296][0297]
表73.clust.091979效应子的保守序列。
[0298]
[0299][0300]
这些系统的直接重复序列和间隔子长度的实例示出于表8中。
[0301]
表84.代表性clust.091979直接重复的核苷酸序列和间隔子长度
[0302]
[0303]
[0304]
[0305]
[0306]
[0307][0308]
实例2-对反式激活rna元件的鉴定
[0309]
除了效应子蛋白和crrna之外,本文所述的一些crispr系统还可以包括激活稳健性酶活性的另外小型rna,将其称为反式激活rna(tracrrna)。此类tracrrna典型地包括与crrna杂交的互补区。crrna-tracrrna杂交体形成具有效应子的复合物,该复合物导致可编程酶活性的激活。
[0310]
·
通过针对与crrna的直接重复部分同源的短序列基序搜索侧接crispr阵列的基因组序列来鉴定tracrrna序列。搜索方法包括对完整直接重复(dr)或dr子序列的精确或简并匹配。例如,可以将长度n个核苷酸的dr分解成一组重叠6-10nt k聚体。可以将这些k聚体与侧接crispr基因座的序列比对,并且可以将具有1或更多个k聚体对齐的同源性的区域鉴
定为dr同源区以用于作为tracrrna的实验验证。替代性地,可以针对完整dr或dr子序列以及来自侧接crispr系统的元件的基因组序列的短k聚体序列计算rna共折叠自由能。可以将具有低最小自由能结构的侧接序列元件鉴定为dr同源区以用于作为tracrrna的实验验证。
[0311]
·
tracrrna元件常常很靠近crispr相关基因或crispr阵列出现。作为搜索dr同源区以鉴定tracrrna元件的替代方案,可以通过克隆或基因合成来分离侧接crispr效应子或crispr阵列的非编码序列以用于tracrrna的直接实验验证。
[0312]
·
可以使用crispr系统的宿主生物的小型rna测序或在非天然物种中异源性表达的合成序列进行tracrrna元件的实验验证。可以使用小型rna序列自起源基因组基因座的比对来鉴定含有dr同源区和完整tracrrna元件的典型模式化加工的表达rna产物。
[0313]
·
通过rna测序鉴定的完整tracrrna候选物可以通过以下方式来进行体外或体内验证:在与tracrrna候选物组合或不组合的情况下表达crrna和效应子,并且监测效应子酶活性的激活。
[0314]
·
在工程化构建体中,tracrrna的表达可以由启动子驱动,这些启动子包括但不限于用于在哺乳动物细胞中表达的u6、u1和h1启动子或用于在细菌中表达的j23119启动子。
[0315]
·
在一些情况下,可以将tracrrna与crrna融合并且表达为单rna指导物。
[0316]
·
该系统可以包括包含在表9中列出的非编码序列内的tracrrna。例如,在一些实施例中,该系统包括seq id no:152-204中任一个中列出的tracrrna。
[0317]
表95.代表性clust.091979系统的非编码序列
[0318]
[0319]
[0320]
[0321]
[0322]
[0323]
[0324][0325]
实例3-对酶活性的新型rna调节子的鉴定
[0326]
除了效应子蛋白和crrna之外,本文所述的一些crispr系统还可以包括激活或调节效应子活性的另外小型rna,将其在本文中称为rna调节子。
[0327]
·
预期rna调节子很靠近crispr相关基因或crispr阵列出现。为了鉴定和验证rna调节子,可以通过克隆或基因合成来分离侧接crispr效应子或crispr阵列的非编码序列以用于直接实验验证。
[0328]
·
可以使用crispr系统的宿主生物的小型rna测序或在非天然物种中异源性表达的合成序列进行rna调节子的实验验证。可以使用小型rna序列与起源基因组基因座的比对来鉴定含有dr同源区和模式化加工的表达rna产物。
[0329]
·
通过rna测序鉴定的候选rna调节子可以通过以下方式来进行体外或体内验证:在与候选rna调节子组合或不组合的情况下表达crrna和效应子,并且监测效应子酶活性的
改变。
[0330]
·
在工程化构建体中,rna调节子可以由启动子驱动,这些启动子包括用于在哺乳动物细胞中表达的u6、u1和h1启动子或用于在细菌中表达的j23119启动子。
[0331]
·
在一些情况下,可以将rna调节子与crrna、tracrrna或两者人工融合并且表达为单rna元件。
[0332]
实例4-工程化clust.091979crispr-cas系统的功能验证
[0333]
鉴定了clust.091979crispr-cas系统的组分,选择来自命名为auxo013988882(seq id no:1)的宏基因组源和来自命名为srr3181151(seq id no:4)的宏基因组源的基因座用于功能验证。
[0334]
dna合成和效应子文库克隆
[0335]
为了测试示例性clust.091979 crispr-cas系统的活性,使用pet28a( )载体设计和合成系统。简而言之,合成编码clust.091979 auxo013988882效应子(表6中所示的seq id no:1)的大肠杆菌密码子优化的核酸序列和编码clust.091979srr3181151效应子(表6中所示的seq id no:4)的大肠杆菌密码子优化的核酸序列(金斯瑞公司(genscript))并且将其单独地克隆至来源于pet-28a( )(emd-密理博公司(emd-millipore))的定制表达系统中。载体包含在lac启动子控制下编码clust.091979效应子的核酸和大肠杆菌核糖体结合序列。载体还包含在clust.091979效应子的开放阅读框后、用于由j23119启动子驱动的crispr阵列文库的接受位点。用于clust.091979 auxo013988882效应子(seq id no:1)的非编码序列列出于seq id no:98中,并且用于clust.091979srr3181151效应子(seq id no:4)的非编码序列列出于seq id no:99中,如表9中所示。测试另外的条件,其中将clust.091979效应子在没有非编码序列的情况下单独地克隆至pet28a( )中。参见图4a。
[0336]
计算设计含有“重复-间隔子-重复”序列的寡核苷酸文库合成(ols)池,其中“重复”表示crispr阵列中发现的与效应子相关联的共有直接重复序列,并且“间隔子”表示拼接pacyc184质粒或大肠杆菌必需基因的序列。特别地,用于clust.091979auxo013988882效应子(seq id no:1)的重复序列列出于seq id no:57中,并且用于clust.091979srr3181151效应子(seq id no:4)的重复序列列出于seq id no:60中,如表8中所示。间隔子长度由内源crispr阵列中发现的间隔子长度的模式决定。将重复-间隔子-重复序列附加有限制性位点,从而使得能够将片段双向克隆至前面提及的crispr阵列文库受体位点中;和独特的pcr引发位点,以使得能够从更大的池中特异性扩增特定重复-间隔子-重复文库。
[0337]
接下来,使用金门组装(golden gate assembly)方法将重复-间隔子-重复文库克隆至质粒中。简而言之,首先使用独特的pcr引物将每种重复-间隔子-重复从ols池(安捷伦基因组学(agilent genomics))中扩增出并且对其使用bsai预线性化质粒骨架以减少潜在背景。将两种dna片段用ampure xp(贝克曼库尔特公司(beckman coulter))纯化,之后添加至金门组装主混合物(新英格兰实验室(new england biolabs))并且根据制造商的说明书孵育。将金门反应物进一步纯化并且浓缩以使得在细菌筛选的随后步骤中实现最大转化效率。
[0338]
按照卢西根公司(lucigen)推荐的方案使用基因pulser (伯乐公司(bio-rad))将含有不同的重复-间隔子-重复元件和crispr效应子的质粒文库电穿孔至e.cloni
srr3181151效应子的强耗减靶标的位置。分析耗减靶标的侧接序列以确定clust.091979 auxo013988882和clust.091979 srr3181151的pam序列。clust.091979 auxo013988882和clust.091979 srr3181151的pam序列的weblogo表示(crooks等人,genome research[基因组研究]14:1188-90,2004)示出于图7和图10中,其中“20”位置对应于相邻于靶标的5’端的核苷酸。
[0344]
因此,clust.091979 crispr-cas的多种效应子显示出体内活性。
[0345]
实例5-通过clust.091979靶向哺乳动物基因
[0346]
此实例描述了使用来自clust.091979的核酸酶进行的对多种靶标的插入缺失评估,这些核酸酶通过瞬时转染引入至哺乳动物细胞中。
[0347]
将seq id no:4和seq id no:10的效应子克隆至pcda3.1骨架(英杰公司(invitrogen))中。然后将质粒大量制备并且稀释至1μg/μl。对于rna指导物制备,通过含有靶序列支架的超聚体(ultramer)和u6启动子衍生编码crrna的dsdna片段。将超聚体重悬于ph 7.5的10mm tris
·
hcl中至最终原液浓度为100μm。随后再次使用10mm tris
·
hcl将工作原液稀释至10μm以充当用于pcr反应的模板。将crrna的扩增在具有以下组分的50μl反应物中进行:0.02μl前面提及的模板、2.5μl正向引物、2.5μl反向引物、25μl新英格兰生物学实验室公司hifi聚合酶和20μl水。循环条件是:1x(98℃下30s)、30x(98℃下10s,67℃下15s)、1x(72℃下2min)。将pcr产物用1.8x spri处理净化并且归一化至25ng/μl。所制备的crrna序列及其相应靶序列示出于表10中。seq id no:205、seq id no:207、seq id no:252、seq id no:254、seq id no:256、seq id no:258、seq id no:260、seq id no:262、seq id no:264、seq id no:266、seq id no:268、seq id no:270、seq id no:272、seq id no:274和seq id no:276的成熟crrna的直接重复序列列出于seq id no:60中。seq id no:209和seq id no:214的成熟crrna的直接重复列出于seq id no:62中。seq id no:211、seq id no:278、seq id no:280、seq id no:282、seq id no:284、seq id no:286和seq id no:288的成熟crrna的直接重复列出于seq id no:213中。
[0348]
表10.用于瞬时转染测定的rna指导物和靶序列。
[0349]
[0350][0351]
在转染前大约16小时,将100μl在dmem/10%fbs 青霉素/链霉素中的25,000个hek293t细胞铺板至96孔板中的每个孔中。在转染的当天,细胞是70%-90%汇合的。对于待转染的每个孔,制备0.5μl lipofectamine 2000和9.5μl opti-mem的混合物,并且然后在室温下孵育5-20分钟(溶液1)。在孵育之后,将lipofectamine:optimem混合物添加至最多至10μl的含有182ng效应子质粒和14ng crrna以及水的单独混合物(溶液2)。在阴性对照的情况下,在溶液2中不包含crrna。将溶液1和溶液2通过向上和向下吸移进行混合,且然后在
室温下孵育25分钟。在孵育后,将20μl溶液1和溶液2混合物逐滴添加至含有细胞的96孔板的每个孔。在转染后72小时,通过以下方式使细胞胰蛋白酶化:向每个孔的中心添加10μl tryple并且孵育大约5分钟。然后将100μl d10培养基添加至每个孔并且混合以重悬细胞。然后将细胞以500g旋转沉降10分钟,并且弃去上清液。将quickextract缓冲液添加至原始细胞悬浮液体积的量的1/5。将细胞在65℃下孵育15分钟,在68℃下孵育15分钟,并且在98℃下孵育10分钟。
[0352]
通过两轮pcr制备用于下一代测序的样品。使用第一轮(pcr1)来扩增根据靶标的特定基因组区域。通过柱纯化对pcr1产物进行纯化。进行第2轮pcr(pcr2)以添加依诺米那衔接子和索引。然后将反应物池化并且通过柱纯化进行纯化。用150次循环nextseq v2.5中等或高输出试剂盒进行测序运行。
[0353]
图11a、图11b、图11c和图11d示出了在分别用seq id no:4或seq id no:10的效应子转染后,hek293t细胞中aavs1、vegfa和emx1靶基因座的插入缺失百分比。条反映了在两个生物平行测定中测量的平均插入缺失百分比。对于seq id no:4和seq id no:10的效应子,插入缺失百分比在每种靶标处高于阴性对照的插入缺失百分比。
[0354]
如图11a中所示,由seq id no:4的效应子和seq id no:205的crrna形成的复合物在seq id no:206的aavs1靶标处具有活性,并且由seq id no:4的效应子和seq id no:207的crrna形成的复合物在seq id no:208的vegfa靶标处具有活性。如图11b中所示,由seq id no:4的效应子和seq id no:252的crrna形成的复合物在seq id no:253的aavs1靶标处具有活性,由seq id no:4的效应子和seq id no:254的crrna形成的复合物在seq id no:255的aavs1靶标处具有活性,由seq id no:4的效应子和seq id no:256的crrna形成的复合物在seq id no:257的aavs1靶标处具有活性,由seq id no:4的效应子和seq id no:258的crrna形成的复合物在seq id no:259的aavs1靶标处具有活性,并且由seq id no:4的效应子和seq id no:274的crrna形成的复合物在seq id no:275的aavs1靶标处具有活性。还如图11b中所示,由seq id no:4的效应子和seq id no:260的crrna形成的复合物在seq id no:261的emx1靶标处具有活性。还如图11b中所示,由seq id no:4的效应子和seq id no:262的crrna形成的复合物在seq id no:263的vegfa1靶标处具有活性,由seq id no:4的效应子和seq id no:264的crrna形成的复合物在seq id no:265的vegfa1靶标处具有活性,由seq id no:4的效应子和seq id no:266的crrna形成的复合物在seq id no:267的vegfa1靶标处具有活性,由seq id no:4的效应子和seq id no:268的crrna形成的复合物在seq id no:269的vegfa1靶标处具有活性,由seq id no:4的效应子和seq id no:270的crrna形成的复合物在seq id no:271的vegfa1靶标处具有活性,由seq id no:4的效应子和seq id no:272的crrna形成的复合物在seq id no:273的vegfa1靶标处具有活性,并且由seq id no:4的效应子和seq id no:274的crrna形成的复合物在seq id no:275的vegfa1靶标处具有活性。seq id no:4的效应子对于图11a和图11b中的每种靶标利用5
’‑
tttg-3’pam。
[0355]
如图11c中所示,由seq id no:10的效应子和seq id no:209的crrna形成的复合物在seq id no:210的aavs1靶标处具有活性,由seq id no:10的效应子和seq id no:211的crrna形成的复合物在seq id no:212的aavs1靶标处具有活性,并且由seq id no:10的效应子和seq id no:214的crrna形成的复合物在seq id no:215的vegfa靶标处具有活性。
如图11d中所示,由seq id no:10的效应子和seq id no:278的crrna形成的复合物在seq id no:279的aavs1靶标处具有活性,由seq id no:10的效应子和seq id no:280的crrna形成的复合物在seq id no:281的aavs1靶标处具有活性,由seq id no:10的效应子和seq id no:284的crrna形成的复合物在seq id no:285的aavs1靶标处具有活性,并且由seq id no:10的效应子和seq id no:286的crrna形成的复合物在seq id no:287的aavs1靶标处具有活性。还如图11d中所示,由seq id no:10的效应子和seq id no:288的crrna形成的复合物在seq id no:289的emx1靶标处具有活性,并且由seq id no:10的效应子和seq id no:282的crrna形成的复合物在seq id no:283的vegfa靶标处具有活性。seq id no:10的效应子对于图11c和图11d中的靶标利用5
’‑
attg-3’pam和5
’‑
gtta-3’pam。
[0356]
此实例表明clust.091979家族中的核酸酶在哺乳动物细胞中具有活性。
[0357]
其他实施例
[0358]
应理解,虽然已经结合其具体实施方式对本发明进行了描述,但前面的描述旨在说明而非限制本发明的范围,本发明的范围由所附权利要求的范围限定。其他方面、优点和修改在以下权利要求的范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。