1.本发明涉及染色体核型分析技术领域,更具体地,涉及一种基于深度学习的染色体核型分析方法及系统。
背景技术:
2.染色体核型分析是遗传科学研究和辅助临床诊断的重要手段之一。在诊断染色体异常的常规临床工作流程中,遗传学家需要通过光学显微镜根据每一条明暗相间的带纹目视检查该标本的染色体数目和形态,因此存在专业要求高、耗时长的问题。
3.目前有半自动商业染色体核型分析系统投入使用,用于辅助遗传学家进行光学显微镜下的分析工作,比如cytovision,ikaros,asi hiband等。上述系统通过光学显微镜拍摄标本玻片上的染色体,然后基于图像方法进行染色体分割、分类等操作。然而上述自动化操作大部分都是集中于核型分析中的某一步任务,如单条染色体类别预测、中期图中的染色体检测、特定染色体的分割等,在实际操作中,需要用户手动调整图像中染色体的清晰度、分割染色体,或将染色体拖到其对应类别的位置上,缺乏从拍摄图到核型图的全流程全自动解决方案,导致目前的染色体核型分析工作存在耗时长、效率低、准确率无法保证。
技术实现要素:
4.本发明为克服上述现有技术所述的缺乏从拍摄图到核型图的全自动分析操作,导致染色体核型分析工作存在耗时长、效率低、准确率无法保证的缺陷,提供一种基于深度学习的染色体核型分析方法,以及一种基于深度学习的染色体核型分析系统。
5.为解决上述技术问题,本发明的技术方案如下:
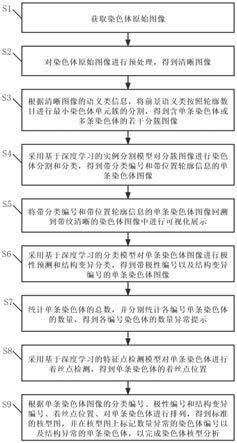
6.一种基于深度学习的染色体核型分析方法,包括以下步骤:
7.s1、获取染色体原始图像;
8.s2、对染色体原始图像进行预处理,得到清晰图像;
9.s3、根据清晰图像的语义类信息,将前景语义类按照轮廓数目进行最小染色体单元簇的分割,得到含单条染色体或多条染色体的若干分簇图像;
10.s4、采用基于深度学习的实例分割模型对分簇图像进行染色体分割和分类,得到带分类编号和带轮廓位置信息的单条染色体图像;
11.s5、将带分类编号和带轮廓位置信息的单条染色体图像回溯到带纹清晰的染色体图像中进行可视化展示;
12.s6、采用基于深度学习的分类模型对单条染色体图像进行极性预测和结构变异分类,得到带极性编号以及结构变异编号的单条染色体图像;
13.s7、统计单条染色体的总数,并分别统计各编号单条染色体的数量,得到各编号染色体的数量异常提示;
14.s8、采用基于深度学习的特征点检测模型对单条染色体进行着丝点检测,得到单条染色体的着丝点位置;
15.s9、根据单条染色体图像的分类编号、极性编号和结构变异编号、着丝点位置、对单条染色体进行排列,得到标准的核型图,并在核型图上标记数量异常的染色体编号以及结构异常的单条染色体,以完成染色体核型分析。
16.进一步的,本发明还提出了一种基于深度学习的染色体核型分析系统,包括:
17.图像采集模块,用于获取染色体原始图像;
18.图像预处理模块,用于对染色体原始图像进行预处理,得到清晰图像;
19.分簇模块,用于根据清晰图像的语义类信息,将前景语义类按照轮廓数目进行最小染色体单元簇的分割,得到含单条染色体或多条染色体的若干分簇图像;
20.分割及分类模块,用于基于深度学习的染色体实例分割模型对分簇图像进行染色体分割和分类,得到带分类编号的单条染色体图像;以及用于采用基于深度学习的分类模型对单条染色体图像进行极性预测和结构变异分类,得到带极性编号以及结构变异编号的单条染色体图像;
21.着丝点检测模块,用于采用基于深度学习的特征点检测模型对单条染色体进行着丝点检测,得到单条染色体的着丝点位置;
22.可视化模块,用于将带分类编号的单条染色体图像回溯到带纹清晰的染色体图像中,根据单条染色体图像的分类编号、极性编号和结构变异编号、着丝点位置、对单条染色体进行排列,得到标准的核型图,并在核型图上标记数量异常的染色体编号以及结构异常的单条染色体,将核型图进行可视化展示。
23.与现有技术相比,本发明技术方案的有益效果是:本发明适用于染色体分割、染色体分类、染色体排列、染色体计数等任务,通过对图像进行预处理操作,得到去噪的带纹清晰的染色体图像,便于后续的染色体分簇、染色体分割、染色体分类等作业任务的操作;本发明还实现了从拍摄端到分析端的全流程自动化操作,能够有效提高用户对染色体核型分析的工作效率。
附图说明
24.图1为实施例1的基于深度学习的染色体核型分析方法的流程图。
25.图2为染色体原始图像。
26.图3为带纹清晰的染色体图像。
27.图4为清晰图像。
28.图5为分簇图像。
29.图6为单条染色体图像。
30.图7为可视化展示的染色体图像。
31.图8为以着丝点排列的染色体核型图像。
32.图9为以底部对齐排列的染色体核型图像。
33.图10为实施例2的基于深度学习的染色体核型分析系统的架构图。
具体实施方式
34.附图仅用于示例性说明,不能理解为对本专利的限制;
35.对于本领域技术人员来说,附图中某些公知结构及其说明可能省略是可以理解
的。
36.下面结合附图和实施例对本发明的技术方案做进一步的说明。
37.实施例1
38.本实施例提出一种基于深度学习的染色体核型分析方法,如图1所示,为本实施例的基于深度学习的染色体核型分析方法的流程图。
39.本实施例提出的基于深度学习的染色体核型分析方法中,包括以下步骤:
40.步骤1:获取染色体原始图像。
41.本实施例通过设备拍摄采集染色体图像,得到染色体原始图像。如图2所示,为本实施例的染色体原始图像。
42.步骤2:对染色体原始图像进行预处理,得到清晰图像。
43.本实施例中,对染色体图像进行预处理的步骤包括:采用基于对抗生成网络的图像转换模型对采集的染色体原始图像进行风格转换,得到如图3所示的带纹清晰的染色体图像;采用基于神经网络的语义分割模型对带纹清晰的染色体图像进行背景消除,得到如图4所示的干净清晰图像。
44.其中,图像转换模型是一种设计为端到端的模型,用于将源域图像转换到目标域图像,其中源域提供图像内容,目标域提供图像的属性或风格,在源域提供的图像内容下实现目标域的风格化,从而实现源域图像到目标域图像的转换。
45.本实施例通过将不同设备或拍摄装置采集的染色体原始图像转换为带纹清晰的染色体图像,便于后续染色体分割和分类操作,缩小图像的差异,减小对不同拍摄风格的染色体图像所带来的分割和分类误差。
46.在一实施例中,图像转换模型包括第一生成器、第二生成器、第一判别器和第二判别器;将所述训练集输入染色体图像转换模型中进行训练时,第一生成器根据输入的染色体原始图像x生成相应的带纹清晰的图像g;第一判别器将第一生成器生成的图像g根据目标域图像集y进行判别,得到源域图像集x在目标域图像集y中的映射,且存在一个最佳的映射使图像g与目标域图像集y具有相同的分布;从目标域图像集y中选择一张带纹清晰的染色体图像输入第二生成器,第二生成器生成图像g
′
;第二判别器将第二生成器生成的图像g
′
根据源域图像集x进行判别,得到标域图像集y在源域图像集x中的映射,且存在一个最佳的映射使图像g
′
与源域图像集x具有相同的分布。
47.在另一实施例中,采用pix2pix、cyclegan、duaigan、discogan、unit、munit、stargan、apade等模型作为图像转换模型,且不限于所述图像转换模型。
48.在一实施例中,采用基于神经网络的语义分割模型对带纹清晰的染色体图像进行背景消除的步骤包括:所述语义分割模型将图像中的染色体区域作为前景语义类,将图像中的背景区域作为背景语义类,预测出输入图像的每一个像素点所属语义类,再根据每一个像素点的语义类生成蒙版,得到前景语义蒙版和背景语义蒙版,最后将背景语义蒙版删除,保留前景语义蒙版,从而实现染色体和背景内容的分割,得到消除背景噪音的清晰图像。
49.其中,语义分割模型采用u-net、fcn、segnet、pspnet、deeplab中的一种或多种,且不限于所述语义分割模型。
50.步骤3:根据清晰图像的语义类信息,将前景语义类按照轮廓数目进行最小染色体
单元簇的分割,得到含单条染色体或多条染色体的若干分簇图像。
51.其中,将前景语义类按照轮廓数目进行最小染色体单元簇的分割的步骤包括:
52.s3.1、对所述清晰图像中的前景语义蒙版进行轮廓提取,对提取的n个轮廓分别计算其最小外围矩形框,并分别保存n个最小外围矩形框的左上角坐标[pointx,pointy]、矩形框的长度值length和宽度值width;
[0053]
进一步的,将n个最小外围矩形框的左上角坐标[pointx,pointy]、矩形框的长度值length和宽度值width以列表的形式存储;
[0054]
s3.2、以所述最小外围矩形框的大小创建n个纯色背景图片,然后将提取的n个轮廓逐一绘制在相应的纯色背景图片中,得到n个轮廓蒙版;
[0055]
s3.3、根据所述轮廓蒙版在所述清晰图像中进行roi区域图像显示,得到轮廓蒙版对应的染色体簇,将该roi区域保存为分簇图像,并对所述分簇图像进行图像尺寸归一化,得到如图5所示的分簇图像。
[0056]
进一步的,本实施例中对图像尺寸归一化的步骤包括:
[0057]
若分簇图像尺寸长度length值大于预设长度l或者分簇图像的宽度width大于预设宽度w,即如果长度值length大于预设长度l,则以分簇图的长边为填充后图片的长度,将宽边按l/w的比例进行两边等距填充,使得填充后图像的长宽比等于l/w,将图片填充为长方形图片后将其尺寸重新设置为l*w;如果宽度值width大于w,则以分簇图的宽边为填充后图片的长度,将长边按l/w的比例进行两边等距填充,使得填充后图像的长宽比等于l/w,将图片填充为长方形图片后将其尺寸重新设置为l*w;
[0058]
若分簇图像的长度值length小于预设长度l和宽度值width小于预设宽度w,则对分簇图像进行边缘填充白色至图像尺寸为l*w,填充时将染色体图像置于图像中部,即对分簇图像进行上下两边和左右两边的等距填充。本实施例中的预设长度l的取值范围为100~1000,预设宽度w的取值范围为100~1000。
[0059]
步骤4:采用基于深度学习的染色体实例分割模型对分簇图像进行染色体分割和分类,得到带分类编号的单条染色体图像。
[0060]
本实施例中对分簇图像进行染色体分割和分类的步骤包括:构建基于深度学习的染色体实例分割模型,将所述分簇图像输入染色体实例分割模型,得到分簇图像中染色体编号及其对应的染色体蒙版;根据所述染色体蒙版在对应的分簇图像上进行裁剪,得到如图6所示的单条染色体图像,然后将所述染色体编号标注在裁剪图像上,得到一系列的带分类编号的单条染色体图像。
[0061]
其中,在对分簇图像进行分类过程中,通过对分簇图像中每一个像素点的前景语义和背景语义,区分出前景语义中的实例类别,即预测分簇图像中每一条染色体的编号。经过染色体分割和分类后,得到分簇图像中每一条染色体编号及其对应的染色体蒙版。
[0062]
在一具体实施例中,染色体实例分割模型采用mask rcnn、blendmask、solo、solov2等。
[0063]
进一步的,构建用于训练所述染色体实例分割模型的训练数据集的步骤如下:
[0064]
(4-a)采集临床现存的核型图,对核型图按区域裁剪,得到各编号染色体图像及对应的标签。
[0065]
(4-b)利用图像配准的方式将各编号染色体图像在预存储的清晰图像上进行特征
匹配,得到清晰图像中与之匹配的染色体蒙版,并将染色体图像的编号赋值给清晰图像中匹配的染色体。
[0066]
(4-c)以清晰图像大小为目标尺寸,为每一张完成特征匹配的染色体蒙版创建背景语义类蒙版的背景图片。
[0067]
(4-d)将每一张完成特征匹配的染色体蒙版以填充内轮廓的形式绘制在所述背景图片上。
[0068]
(4-e)判断清晰图像上的完成特征匹配的染色体蒙版是否存在重合区域,若是,则对有重合区域的染色体蒙版进行合并生成含多条染色体的分簇图蒙版,例如新的交叉蒙版、粘连蒙版、重叠蒙版、交叉重叠蒙版或交叉粘连蒙版等,同时保存所述分簇图蒙版上的染色体蒙版及其编号。
[0069]
(4-f)将所述染色体蒙版和分簇图蒙版作为感兴趣区域,将完成特征匹配的染色体蒙版进行roi区域图像显示。
[0070]
(4-g)根据所述染色体蒙版和分簇图蒙版提取最小矩形区域,将所述染色体蒙版和分簇图蒙版及其对应的roi区域显示后的图像进行最小矩形区域裁剪,再对裁剪后的图像进行图像尺寸归一化处理,得到单条染色体图像和含多条染色体的分簇图。
[0071]
本步骤的图像尺寸归一化处理步骤与上述s3.3步骤对分簇图像进行图像尺寸归一化处理相同。
[0072]
(4-h)对单条染色体图像和含多条染色体的分簇图,将其对应的染色体标签及染色体蒙版轮廓信息写入标注文件,制作实例分割标注信息,得到大量单条及多条染色体图像及其对应的标注文件,组成用于训练所述染色体实例分割模型的训练数据集。
[0073]
经过上述步骤,能够自动制作大量单条及多条染色体图像及其对应的以json文件形式存储的标注文件。
[0074]
步骤5:将带分类编号和带轮廓位置信息的单条染色体图像回溯到带纹清晰的染色体图像中进行可视化展示。
[0075]
其中,将带分类编号的单条染色体图像回溯到带纹清晰的染色体图像中进行可视化展示的步骤包括:
[0076]
(5-a)对于所述带分类编号的单条染色体图像的染色体蒙版,计算其最小外围矩形,得到最小外围矩形列表,以及单条染色体所在的分簇图进行分簇时图像的长度length和宽度width;
[0077]
(5-b)根据每个单条染色体图像分簇的长度length和宽度width对单条染色体图像进行图像尺寸归一化处理;其具体步骤如下:
[0078]
若单条染色体图像分簇的长度length和宽度width进行判断,并对图像尺寸进行调整:当长度length大于预设长度l,或宽度width大于预设宽度w时,则对该单条染色体图像的染色体蒙版重新设置尺寸:当长度值length大于预设长度l,则以单条染色体图像的染色体蒙版的长边为填充后图片的长度,将宽边按l/w的比例进行两边等距填充,使得填充后图像的长宽比等于l/w,将图片填充为长方形图片后将其尺寸重新设置为l*w;当宽度值width大于预设宽度w,则以单条染色体图像的宽边为填充后图片的长度,将长边按l/w的比例进行两边等距填充,使得填充后图像的长宽比等于l/w,将图片填充为长方形图片后将其尺寸重新设置为l*w;
[0079]
若单条染色体图像的长度值length小于预设长度l和宽度值width小于预设宽度w,则对单条染色体图像的染色体蒙版进行边缘填充至图像尺寸为l*w,填充时将染色体图像置于图像中部,即对单条染色体图像进行上下两边和左右两边的等距填充。本实施例中的l的取值范围为100~1000,w的取值范围为100~1000。
[0080]
(5-c)对完成图像尺寸调整的单条染色体图像中的染色体蒙版进行轮廓提取,然后将分簇图最小外围矩阵起始点作为该轮廓映射至清晰图像的起始点,将轮廓映射至清晰图像;
[0081]
(5-d)遍历完成基本单位的所有单条染色体图像中的染色体蒙版后,将完成分割和分类的带分类编号的单条染色体图像回溯到所述清晰图像中进行可视化显示。
[0082]
在实际可视化展示中,染色体图像中所有单条染色体以彩色轮廓分割,且单条染色体图像的分类编号以不同染色体之间的轮廓色彩区分,得到如图7所示的可视化展示的染色体图像。
[0083]
步骤6:采用基于深度学习的分类模型对单条染色体图像进行极性预测和结构变异分类,得到带极性编号以及结构变异编号的单条染色体图像。
[0084]
本步骤中,构建基于深度学习的染色体分类模型,将单条染色体图像输入染色体分类模型,得到单条染色体图像中染色体极性编号及异常编号。其中,构建用于训练所述染色体分类模型的具体步骤如下:
[0085]
(6-a)采集临床现存的核型图,对核型图按区域裁剪,得到各编号染色体图像及对应的标签;
[0086]
(6-b)为各编号染色体图像制作极性标签,将随机选择一定量的染色体图像垂直翻转操作,将该部分图像标注为方向向下(用编号a表示),剩余各编号图像标注为方向向上(用编号b表示);
[0087]
(6-c)为上述已标注极性标签的图像制作结构异常标签,将有结构异常的染色体图像标记为异常(用编号an表示),将没有结构异常的染色体图像标记为正常(用编号n表示);
[0088]
(6-d)将各染色体图像对应的染色体编号、极性标签、结构异常标签写入标注文件,制作单条染色体图像标注信息,得到大量单条染色体图像及其对应的标注文件,组成用于训练所述染色体分类模型的训练数据集;
[0089]
(6-e)将(6-d)得到的训练数据集输入染色体分类模型中进行训练,根据模型输出的分类结果对染色体分类模型的参数进行调整,完成基于深度学习的染色体分类模型的构建。
[0090]
本实施例中采用的基于深度学习的染色体分类模型包括alexnet、zfnet、vgg、inception、resnet、wideresnet(inception-resnet_v1/v2)、densenet、resnext、dpn、senet等中的一种或多种。
[0091]
步骤7:统计单条染色体的总数,并分别统计各编号单条染色体的数量,得到各编号染色体的数量异常提示。
[0092]
步骤8:采用基于深度学习的特征点检测模型对单条染色体进行着丝点检测,得到单条染色体的着丝点位置。
[0093]
本步骤中,通过构建基于深度学习的特征点检测模型,将单条染色体图像输入染
色体特征点检测模型,得到单条染色体图像中着丝点位置(x,y)。其中,构建用于训练所述染色体特征点检测模型的步骤如下:
[0094]
(8-a)采集临床现存的核型图,对核型图按区域进行裁剪,得到各编号染色体图像;
[0095]
(8-b)为各编号染色体图像制作着丝点位置标签,标注图像中染色体着丝点位置的坐标(x,y);
[0096]
(8-c)将其对应的染色体编号、着丝点位置写入标注文件,制作单条染色体图像标注信息,得到大量单条染色体图像及其对应的标注文件,组成用于训练所述染色体特征点检测模型的训练数据集;
[0097]
(8-d)将(8-c)得到的训练数据集输入染色体特征点检测模型中,根据检测结果对染色体特征点检测模型中的参数进行调整,完成染色体特征点检测模型的构建。
[0098]
本实施例中的染色体特征点检测模型包括mtcnn、tcdcn等中的一种或多种。
[0099]
步骤9:根据单条染色体图像的分类编号、极性标签和结构变异标签、着丝点位置,对单条染色体进行排列,得到标准的核型图,并在核型图上标记数量异常的染色体编号以及结构异常的单条染色体,以完成染色体核型分析。
[0100]
在另一实施例中,为更直观地观察染色体核型图,还设置有以下步骤:对所述带分类编号、极性标签、结构变异标签、着丝点位置的单条染色体图像按染色体编号顺序进行排列,生成染色体核型图。其具体步骤如下:
[0101]
1)以像素值255,创建染色体排列图像的白色背景图像;
[0102]
2)对每一张分簇图像,以染色体实例分割输出的单条染色体蒙版作为roi,在分簇图像上进行感兴趣区域显示,得到分割后的单条染色体图像;
[0103]
3)将上述得到的染色体图像进行最小矩形区域裁剪,并进行相应角度的旋转,将染色体摆正;
[0104]
4)将旋转摆正后的单条染色体图像按染色体编号顺序,依次放置在排列图中的对应位置,并在该染色体下方合适位置打印对应的染色体编号,以及分类编号、极性编号和结构变异编号、着丝点位置,并在核型图上标记数量异常的染色体编号以及结构异常的单条染色体,得到染色体核型排列图。
[0105]
其中,所述染色体核型排列图中单条染色体图像的排列方式包括以着丝粒排列和/或底部对齐排列,具体根据用户的使用需求选择,分别得到如图8、9所示的染色体核型排列图。本实施例提出的染色体核型分析方法可应用于染色体分割、染色体分类、染色体排列、染色体计数、核型图输出等任务,实现染色体核型分析从拍摄至分析全流程的自动化操作,即能够有效实现对光学显微镜拍摄的染色体原始图像进行全自动进行核型分析。
[0106]
实施例2
[0107]
本实施例提出一种基于深度学习的染色体核型分析系统,应用于实施例1提出的一种基于深度学习的染色体核型分析方法。如图10所示,为本实施例的基于深度学习的染色体核型分析系统的架构图。
[0108]
本实施例提出的基于深度学习的染色体核型分析系统中,包括:
[0109]
图像采集模块1,用于获取染色体原始图像;
[0110]
图像预处理模块2,用于对染色体原始图像进行预处理,得到清晰图像;
[0111]
分簇模块3,用于根据清晰图像的语义类信息,将前景语义类按照轮廓数目进行最小染色体单元簇的分割,得到含单条染色体或多条染色体的若干分簇图像;
[0112]
分割及分类模块4,用于基于深度学习的染色体实例分割模型对分簇图像进行染色体分割和分类,得到带分类编号的单条染色体图像;以及用于采用基于深度学习的分类模型对单条染色体图像进行极性预测和结构变异分类,得到带极性编号以及结构变异编号的单条染色体图像;
[0113]
着丝点检测模块5,用于采用基于深度学习的特征点检测模型对单条染色体进行着丝点检测,得到单条染色体的着丝点位置;
[0114]
可视化模块6,用于将带分类编号的单条染色体图像回溯到带纹清晰的染色体图像中,根据单条染色体图像的分类编号、极性编号和结构变异编号、着丝点位置、对单条染色体进行排列,得到标准的核型图,并在核型图上标记数量异常的染色体编号以及结构异常的单条染色体,将核型图进行可视化展示。
[0115]
在具体实施过程中,图像采集模块1与外接设备连接,获取染色体原始图像,并将获取的染色体原始图像传输至图像预处理模块2中。
[0116]
图像预处理模块2采用基于对抗生成网络的图像转换模型对输入的染色体原始图像进行风格转换,得到带纹清晰的染色体图像,以及采用基于神经网络的语义分割模型对带纹清晰的染色体图像进行背景消除,得到清晰图像。图像预处理模块2将其完成预处理得到的清晰图像传输至分簇模块3。
[0117]
分簇模块3根据清晰图像的语义类信息,将前景语义类按照轮廓数目进行最小染色体单元簇的分割,得到含单条染色体或多条染色体的若干分簇图像,再将分簇图像传输至分割及分类模块4中。
[0118]
分割及分类模块4搭载有完成训练的基于深度学习的染色体实例分割模型,分割及分类模块4将所述分簇图像输入染色体实例分割模型,得到分簇图像中染色体编号及其对应的染色体蒙版;根据所述染色体蒙版在对应的分簇图像上进行裁剪,并将所述染色体编号标注在裁剪图像上,得到一系列的带分类编号的单条染色体图像,再将一系列的带分类编号的单条染色体图像传输至可视化模块6中。
[0119]
同时,分割及分类模块4采用预设的基于深度学习的分类模型对单条染色体图像进行极性预测和结构变异分类,得到带极性编号以及结构变异编号的单条染色体图像,再将带极性编号以及结构变异编号的单条染色体图像传输至可视化模块5中。
[0120]
着丝点检测模块5采用预设的基于深度学习的特征点检测模型对单条染色体进行着丝点检测,得到单条染色体的着丝点位置,再将单条染色体的着丝点位置传输至可视化模块5中。
[0121]
可视化模块6以清晰图像为单位,将带分类编号的单条染色体图像回溯到带纹清晰的染色体图像中根据单条染色体图像的分类编号、极性编号和结构变异编号、着丝点位置、对单条染色体进行排列,得到标准的核型图,并在核型图上标记数量异常的染色体编号以及结构异常的单条染色体,以完成染色体核型分析。
[0122]
本实施例构建一个能够实现从拍摄图到核型图的全流程全自动的染色体核型分析系统,通过图像预处理模块2去除拍摄图中大量的背景噪声,配合分簇模块3、分割及分类模块4、着丝点检测模块5等功能模块实现染色体分割、染色体分类、染色体排列、染色体计
数等任务的连通,从而减少遗传学家的工作量,并提高染色体核型分析工作效率。
[0123]
实施例3
[0124]
本实施例提出一种基于深度学习的染色体核型分析系统,应用于实施例1提出的基于深度学习的染色体核型分析方法。
[0125]
本实施例提出的基于深度学习的染色体核型分析系统中,包括处理器和存储器,其中存储器上存储有计算机程序,所述处理器执行存储器中的计算机程序时实现实施例1所述的基于深度学习的染色体核型分析方法的步骤。
[0126]
相同或相似的标号对应相同或相似的部件;
[0127]
显然,本发明的上述实施例仅仅是为清楚地说明本发明所作的举例,而并非是对本发明的实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以穷举。凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明权利要求的保护范围之内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。