1.本发明涉及计算机辅助药物研发技术领域,具体涉及一种高效分子三维构象的表达方法。
背景技术:
2.药物和受体分子的作用是一种构象动态匹配过程,药物和受体分子都进行了一系列重要构象变化。能被受体识别并与受体结构互补的构象,才产生特定的药理效应,即药效构象。所以,药物和受体的相互作用是在三维空间实现的,分子的三维构象分析是药物分子设计的关键。随着计算机化学和分子图形学的发展,药物和受体的三维结构模拟已成为可能,计算机辅助三维药物设计大大提高了药物开发的效率,已成为药物研发领域的研究热点。
3.传统的分子构象信息存储使用三维坐标的显示表达,例如mdl、sdf、pdb和 xyz格式等。然而,由于分子含有多个柔性可旋转键的三维结构,为了表征其构象空间,需要使用大量的构象(100-1000)做为其构象空间的代表,因此,以上使用三维坐标的显示表达会占据很大的文件存储空间,这对于千万级以上的大型分子库的构象空间表达会面临很大的技术挑战,不仅要占据巨大的文件存储空间,也给数据的读写构成巨大的io瓶颈。
技术实现要素:
4.本发明的目的在于针对现有技术中的不足,而提供一种高效分子三维构象的表达方法,该方法高效地还原出分子构象中各原子的坐标,突破了千万级以上的超大规模化学分子数据库的分子三维构象的存储限制,为药物分子的三维筛选提供了有效解决手段。
5.本发明的目的通过以下技术方案实现:
6.提供一种高效分子三维构象的表达方法,包括以下步骤:
7.步骤a、建立基础片段库辅助文件,以获得所有三维构象元数据文件中所需参考的基础片段的三维结构数据;
8.步骤b、基于分子片段组装技术构建分子三维构象,
9.步骤c、记录生成分子三维构象的元数据,所述元数据包含以下信息:
10.(1)构成分子的基础片段信息,包括基础片段的哈希代码、基础片段的构象数;
11.(2)基础片段的连接关系,包括每个子片段连接涉及的基础片段、连接的原子指标和连接键的旋转刻度;
12.(3)基础片段间合理构象组合的标示:构成子片段的两个基础片段各自的三维构象以怎样的旋转角度组合是合理的;
13.(4)每个基础片段当前步骤中在后续连接步骤中还可用的连接键;
14.(5)每个基础片段的连接键对应的可用于确定该基础片段沿该旋转键旋转角度的锚定原子;
15.(6)由基础片段组合成子片段中原子指标的映射关系;
16.(7)整体分子的构象数量以及各个构象能量;
17.(8)整体分子的原子顺序以及原子键连信息和立体信息;
18.(9)分子的药效团信息。
19.上述技术方案中,步骤a中,所述基础片段库包含以下信息:
20.(1)基础片段的唯一表征代码即哈希代码;
21.所述基础片段为组合成分子的最小片段单元,所述基础片段的唯一表征代码为哈希代码,其完全由该基础片段分子的拓扑图以及立体信息唯一地决定;
22.(2)基础片段是否在公用库或者自定义;
23.(3)如果基础片段是自定义,则该基础片段有多少个三维构象以及对应的三维坐标;
24.上述技术方案中,哈希代码的计算方式为:
25.1)对药效团长度和类型进行编码,随机整数,一旦选定就固定了;
26.2)以每个原子为中心,将该原子的编码分别与该原子各个连接键的距离编码的乘积进行加和,作为该原子的新编码;如此迭代2~3次;
27.3)计算所有原子中心的新编码的加和即得到哈希代码。
28.上述技术方案中,步骤b包括以下步骤:
29.b1、从基础片段的三维构象组合成较大的子片段
30.(1)组合成子片段ab的两个基础片段分别定义为基础片段a和基础片段b,基础片段a由基础片段库中选取na个合理三维结构,基础片段b由基础片段库中选取 nb个合理三维结构;
31.(2)基础片段a与基础片段b以一个可旋转的单键连接,并通过nr个旋转刻度,nr取值10~30,每个旋转刻度为360/nr度,得到na*nb*nr个可能的构象组合;
32.(3)从na*nb*nr个构象组合中过滤掉高能量的不合理构象,保留nab个三维结构作为组合而成的子片段ab的合理三维构象;
33.b2、按照步骤b1获得所有组合而成的子片段的合理三维构象;
34.b3、对所有组合而成的子片段的合理三维构象,按照步骤b1的方法,直到完成整个分子的三维构象。
35.上述技术方案中,步骤b1中,所述合理三维结构是指通过常规能量计算方法计算三维结构的能量,并选取低能量且没有范德华距离冲突的三维结构。
36.上述技术方案中,还包括步骤d、元数据的压缩存储:将步骤c记录的元数据与二进制格式或者gz压缩格式组合输出到文件中,重建三维构象时该文件可以直接读人内存。
37.本发明的有益效果:
38.本发明的一种高效分子三维构象的表达方法,包括步骤a、建立基础片段库辅助文件,以获得所有三维构象元数据文件中所需参考的基础片段的三维结构数据;步骤b、基于分子片段组装技术构建分子三维构象;步骤c、记录生成分子三维构象的元数据以及步骤d、元数据的压缩存储。步骤c中只记录了分子组装步骤中合理的组合必要且完整的信息,并以此在构象还原时可以极高的效率重构三维构象,这是因为生成构象时需对大量可能组合的能量计算以便从中挑选出极少一部分可以保留的组合,而依据这些保留的合理组合重新组装三维结构时跳过了生成构象时的大量计算。因此,与现有技术相比,本发明避免了显式地
记录每个分子构象中各原子的三维坐标,而是记录了高效还原每个构象三维坐标的meta信息(包括构成分子的子片段及其连接的拓扑关系、每个可旋转键的扭转角度、每个构象中各个子片段的构象指标等),利用这些meta信息并结合基础片段库构象,可以高效地还原出分子构象中各原子的坐标。由于meta信息的存储量要比三维构象的显式表达要小100-1000倍,而meta 数据还原三维坐标的速度要比最快的三维构象生成的算法还要快一个数量级以上。由此,本发明的表达方法突破了超大规模化学分子数据库的分子三维构象的存储限制,为药物分子的三维筛选提供了有效解决手段。
附图说明
39.利用附图对本发明作进一步说明,但附图中的实施例不构成对本发明的任何限制,对于本领域的普通技术人员,在不付出创造性劳动的前提下,还可以根据以下附图获得其它的附图。
40.图1为实施例2的基础片段库辅助文件的界面演示图。
41.图2为实施例2的元数据文件的界面演示图。
具体实施方式
42.下面结合具体实施例对本发明作进一步的详细说明,但并不构成对本发明的任和限制。
43.实施例1。
44.本实施例的一种高效分子三维构象的表达方法,包括以下步骤:
45.步骤a、建立基础片段库辅助文件,以获得所有三维构象元数据文件中所需参考的基础片段的三维结构数据;所述基础片段库包含以下信息:
46.(1)基础片段的唯一表征代码即哈希代码;
47.所述基础片段为组合成分子的最小片段单元,所述基础片段的唯一表征代码为哈希代码,其完全由该基础片段分子的拓扑图以及立体信息(例如分子手性、立体关系) 唯一地决定;
48.(2)基础片段是否在公用库或者自定义;
49.(3)如果基础片段是自定义,则该基础片段有多少个三维构象以及对应的三维坐标。
50.步骤b、基于分子片段组装技术构建分子三维构象,包括以下步骤:
51.b1、从基础片段的三维构象组合成较大的子片段
52.(1)组合成子片段ab的两个基础片段分别定义为基础片段a和基础片段b,基础片段a由基础片段库中选取na个合理三维结构,基础片段b由基础片段库中选取 nb个合理三维结构;
53.(2)基础片段a与基础片段b以一个可旋转的单键连接,并通过nr个旋转刻度,nr取值10~30,每个旋转刻度为360/nr度,得到na*nb*nr个可能的构象组合;
54.(3)从na*nb*nr个构象组合中过滤掉高能量的不合理构象,保留nab个三维结构作为组合而成的子片段ab的合理三维构象;
55.b2、按照步骤b1获得所有组合而成的子片段的合理三维构象;
56.b3、对所有组合而成的子片段的合理三维构象,按照步骤b1的方法,直到完成整个分子的三维构象。
57.步骤c、记录生成分子三维构象的元数据,所述元数据包含以下信息:
58.(1)构成分子的基础片段信息,包括基础片段的哈希代码、基础片段的构象数和每个构象的三维坐标;
59.(2)基础片段的连接关系,包括每个子片段连接涉及的基础片段、连接的原子指标和连接键的旋转刻度;
60.(3)基础片段间合理构象组合的标示:构成子片段的两个基础片段各自的三维构象以怎样的旋转角度组合是合理的;
61.(4)每个基础片段当前步骤中在后续连接步骤中还可用的连接键;
62.(5)每个基础片段的连接键对应的可用于确定该基础片段沿该旋转键旋转角度的锚定原子;
63.(6)由基础片段组合成子片段中原子指标的映射关系;
64.(7)整体分子的构象数量以及各个构象能量;
65.(8)整体分子的原子顺序以及原子键连信息和立体信息。
66.(9)分子的药效团信息。
67.步骤d、元数据的压缩存储
68.将步骤c记录的元数据与二进制格式或者gz压缩格式组合输出到文件中,重建三维构象时该文件可以直接读人内存。
69.本实施例中,步骤a中的哈希代码的计算方式为:
70.1)对药效团长度和类型进行编码,随机整数,一旦选定就固定了;
71.2)以每个原子为中心,将该原子的编码分别与该原子各个连接键的距离编码的乘积进行加和,作为该原子的新编码;如此迭代2~3次;
72.3)计算所有原子中心的新编码的加和即得到哈希代码。
73.本实施例中,步骤b1中的合理三维结构是指通过常规能量计算方法计算三维结构的能量,并选取低能量且没有范德华距离冲突的三维结构。
74.实施例2。
75.本实施例的具体实施方式以ch
3-sih
2-geh
2-oh分子为例,该分子的三维构象的表达方法如下:
76.一、建立基础片段库辅助文件,以获得所有三维构象元数据文件中所需参考的基础片段的三维结构数据:
77.该基础片段库辅助文件如图1所示:
78.第一行:基础片段库的文件名。这一行为空,表明全部基础片段是自定义的。
79.第二行:从第二行开始记录每个基础片段的表达。每个记录的第一行表明该记录是自定义(0)还是公用库(1)以及该记录的基础片段的哈希代码。
80.如果基础片段为自定义(0),那么紧接着上面一行的是该基础片段有多少个原子以及有多少三维构象;然后,紧接着是每个三维构象中各原子的x、y、z坐标。至此,这个基础片段库的表达结束。
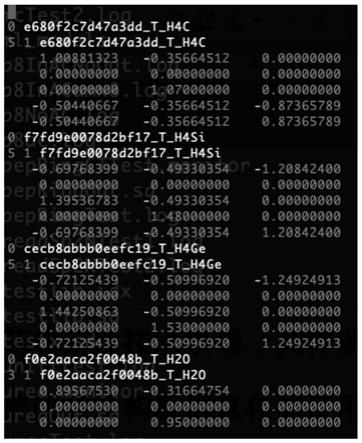
81.然后,按照以上表达方式,重复其它基础片段库的信息。
82.由于该基础片段库辅助文件只记录涉及的非重复的基础片段,而且许多常用基础片段可以指向一个公用库,因此这个文件通常很小。在构象重构时,一般是从公用库和这个辅助文件自定义的基础片段库先将构象元数据文件里涉及的所有基础片段的三维结构一次性调入内存里,从而可以随机地将元数据文件里任何一个分子的构象重建。
83.二、构建分子三维构象:
84.该分子ch
3-sih
2-geh
2-oh包含四个片段单元:ch3、sih2、geh2和oh,分别对应四个基础片段分子ch4、sih4、geh4及h2o。根据基础片段库辅助文件的信息,自下而上地从基础片段组合出整体分子的过程如下:
85.(1)ch4 sih4→
ch3sih386.(2)geh4 h2o
→
geh3oh
87.(3)ch3sih3 geh3oh
→
ch3sih2geh2oh
88.三、记录生成该分子三维构象的元数据:
89.如图2所示,元数据文件包含以下信息:
90.第一行:该分子的化学名称(csigeoh8)。
91.第二行:该分子的哈希代码。
92.第三行至“m end”一行,为整体分子的原子顺序以及原子键连信息和立体信息。
[0093]“m end”一行之后的内容包含:
[0094]
(1)构成分子的基础片段信息,包括基础片段的哈希代码、基础片段的构象数;
[0095]
(2)基础片段的连接关系,包括每个子片段连接涉及的基础片段、连接的原子指标和连接键的旋转刻度;
[0096]
(3)片段间合理构象组合的标示:构成子片段的两个基础片段各自的三维构象以怎样的旋转角度组合是合理的;
[0097]
(4)每个片段当前步骤中在后续连接步骤中还可用的连接键;
[0098]
(5)每个基础片段的连接键对应的可用于确定该基础片段沿该旋转键旋转角度的锚定原子;
[0099]
(6)由基础片段组合成子片段中原子指标的映射关系。
[0100]
(7)分子的药效团信息。
[0101]
补充说明:
[0102]
图2中“30001-120-1#ia,ib,ja,jb,nedges of fraga and fragb indexes”一行中记录的前四个数“ia”、“ib”、“ja”、“jb”分别表征对于基础片段a和基础片段b中的连接原子(ja,jb)对应到组合而成的子片段ab中的原子指标(ia,ib);
[0103]
该行中记录的其余的数表征基础片段a和基础片段b中可用连接键对应到子片段 ab中可用的连接键。如果是数字
“‑
1”,则表明该连接键连接完毕,不再可用。这里表明,基础片段a无可用连接键,而基础片段b原来还有两个可连接键,现在只剩一个了。
[0104]
四、元数据的压缩存储:
[0105]
将上述记录的元数据与二进制格式或者gz压缩格式组合输出到文件中,重建三维构象时该文件可以直接读人内存。
[0106]
以下分别对vi文件压缩比例和构象重构速度进行测试:
[0107]
1、ac格式与传统sd文本格式构象存储效率的对照结果如表1所示:
[0108]
表1.ac格式与传统sd文本格式构象存储效率的对照结果
[0109]
最大构象数量1002505001000ac7.2k11k17k24ksdf2.7m6.6m14m22mratio375600820920
[0110]
由表1的数据可知,本发明的构象存储文件的压缩效率是传统构象存储文件的 300~1000倍,本发明大量节省了构象存储需要的存储空间,从而为超大规模的分子构象库存储提供了高效的解决方案。
[0111]
2、从ac格式读入分子三维坐标的速率与线程数的关系如表2所示:
[0112]
表2.从ac格式读入分子三维坐标的速率与线程数的关系
[0113][0114][0115]
由表2的数据可以得出,本发明的构象生成速度为每秒进行68万~272.2万构象的生成(4线程并行)或81.6万~408.4万构象的生成(8线程并行)。
[0116]
以上所举实施例为本发明的较佳实施方式,仅用来方便说明本发明,并非对本发明作任何形式上的限制,任何所属技术领域中具有通常知识者,若在不脱离本发明所提技术特征的范围内,利用本发明所揭示技术内容所作出局部更动或修饰的等效实施例,并且未脱离本发明的技术特征内容,均仍属于本发明技术特征的范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。