1.本发明涉及一种染料的吸收光谱研究领域,特别涉及一种基于xgboost算法快速预测染料的最大吸收波长的方法及其系统。
背景技术:
2.染料作为一种着色剂,大量用于纺织品的染色和印花上,油漆、塑料、纸张、皮革、光电通讯、食品、涂料、化妆品、摄影材料中也有广泛的使用。1856年,英国科学家伯琴合成了苯胺紫,开启了合成染料的时代。1862年p.griess发现偶氮染料——卑斯麦棕(bismarck brown),合成染料发展至今已有一百五十多年历史,偶氮染料已经成为品种最多的一类,占全世界所有染料的70%左右。
3.近年来,随着人们对于美好生活的向往和对时尚与美的追求,纺织行业中出现了许多新颖的染料制品,颜色种类繁多。为了满足人们对于色彩提出的更高要求,研究者们期望寻找一种快速得到与染料颜色直接相关的参数的方法。最大吸收波长是反映染料颜色的最重要参数之一。目前,得到染料的最大吸收波长的方法主要是先将合成后的染料配成一定浓度的溶液,再使用紫外
‑
可见分光光度仪进行检测,虽然这种方法能够较为准确的检测出染料的最大吸收波长,但其耗费的时间较长,实验成本较高,在开发新型符合特定颜色要求的染料工作上会有一定的困难。
4.随着人工智能和大数据的发展,计算机技术用于化学领域的研发日益广泛,这使得将算法运用于染料的最大吸收波长进行预测,进一步地开发新型染料成为可能。extreme gradient boosting(可译为极限梯度提升算法),简称xbgoost算法,是由华盛顿大学陈天奇博士于2014年提出,该算法是基于累加迭代的梯度提升决策树,为梯度增强回归树(gbrt)的改进与具体实现。xbgoost算法致力于让提升树突破自身的计算极限,以实现运算快速、性能优异的工程目标。它同时也支持并行计算,适用于小样本集以及大规模样本集,可训练百万级的样本。但如何将xbgoost算法应用于快速预测染料的参数还需要进一步地探索和研究。
技术实现要素:
5.为了解决现有技术染料最大吸收波长的检测成本高、耗时长的问题,本发明的目的在于克服已有技术存在的不足,提供一种实现快速预测染料的最大吸收波长的方法及其系统,通过计算染料的二维描述符,利用最大相关最小冗余(mrmr)的变量筛选方法,同时借助xgboost算法建立模型,快速预测染料的最大吸收波长值。通过以上方法,可以在数分钟内快速得到结果,大大降低时间成本及检测成本,整个过程无需实验和繁杂的计算。
6.为达到上述发明创造目的,本发明采用如下技术方案:
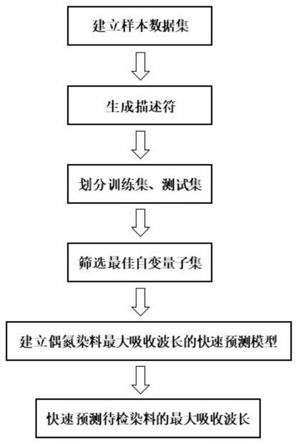
7.一种基于xgboost算法快速预测染料的最大吸收波长的方法,包括如下步骤:
8.1)利用计算机系统,从文献中查找偶氮型染料分子结构以及对应的最大吸收波长实验值,进行数据预处理,整理样本的分子结构式、获取样本的最大吸收波长实验值,将预
处理后的数据集样本作为后续建模的数据集样本;所述的步骤1)中的对数据样本进行预处理包括整理样本的分子结构式、获取样本的最大吸收波长实验值;
9.2)对所述数据集的样本使用化学绘图程序,绘得所收集的染料分子结构,利用描述符生成程序生成相应的描述符;
10.3)以所述数据集样本的最大吸收波长实验值为目标变量,使用产生的描述符为自变量,通过变量筛选方法删除冗余自变量,保留最佳自变量子集;
11.4)对所述数据集进行随机划分为训练集和测试集;
12.5)对最佳自变量子集进行自变量进行二次筛选,对筛选出的最佳自变量进行转换,获取新的自变量子集;
13.6)目标变量为以所述步骤1)的数据集为样本,自变量为所述步骤6)的自变量子集,以所述目标变量与自变量,使用xgboost算法构建训练模型,建立偶氮型染料最大吸收波长的快速预测模型;
14.7)根据所建立的偶氮型染料最大吸收波长的快速预测模型和待测的偶氮型染料分子结构,快速预测待检测的偶氮型染料的最大吸收波长。
15.优选地,在所述步骤5)中,获取的新的自变量有22个,分别为:
16.p(1)=
‑
0.1468x1 0.07953x2 0.005442x3‑
0.1144x4‑
0.03946x5‑
0.7645x6‑
0.1187x7
‑
0.02542x8 0.2509x9 0.2106x10 1.518x11
‑
1.490x12 0.3446x13 1.073x14 0.1408x15 0.008882x16 3.373x17
‑
0.08313x18 0.08137x19
‑
0.1829x20 0.1180x21
‑
0.1042x22
‑
2.007;
17.p(2)=
‑
0.3452x1‑
0.001333x2‑
0.1305x3 0.03473x4 0.06318x5 0.5968x6‑
0.08493x7‑
0.05796x8‑
0.4417x9‑
0.0002596x
10
0.8958x
11
0.6900x
12
‑
0.1266x
13
0.3495x
14
‑
0.002020x
15
‑
0.03937x
16
4.958x
17
‑
0.2235x
18
0.06097x
19
‑
0.8796x
20
‑
0.04725x
21
0.007784x
22
‑
3.015;
18.p(3)=
‑
0.04478x1 0.03094x2 0.1164x3 0.4955x4 0.1280x5 0.5930x6‑
0.2875x7 0.1254x8 0.4020x9 0.08689x
10
‑
0.6100x
11
1.735x
12
‑
0.01940x
13
0.004682x
14
0.05438x
15
0.2704x
16
6.064x
17
‑
0.1437x
18
0.04459x
19
0.1100x
20
‑
0.09971x
21
0.5054x
22
‑
3.849;
19.p(4)=
‑
0.009321x1‑
0.06079x2 0.2526x3‑
0.2809x4 0.1216x5 0.06794x6‑
0.4561x7 0.05122x8 1.249x9‑
0.07666x
10
‑
0.7976x
11
2.530x
12
‑
0.03591x
13
1.490x
14
0.1424x
15
0.0004158x
16
7.116x
17
‑
0.1939x
18
‑
0.004350x
19
‑
0.03845x
20
‑
0.1580x
21
‑
0.1890x
22
‑
3.298;
20.p(5)= 0.1486x1‑
0.04740x2 0.06389x3‑
0.4567x4‑
0.08375x5‑
1.068x6‑
0.1309x7 0.08396x8‑
0.2237x9‑
0.02917x
10
‑
1.069x
11
0.6590x
12
0.09570x
13
0.5122x
14
0.1219x
15
0.1383x
16
1.394x
17
‑
0.3684x
18
‑
0.005981x
19
0.4988x
20
‑
0.2064x
21
‑
0.1966x
22
0.1455;
21.p(6)=
‑
0.06321x1‑
0.06233x2 0.09175x3‑
0.4032x4‑
0.03185x5‑
0.8999x6‑
0.6305x7 0.006485x8‑
0.08835x9‑
0.004323x
10
0.4255x
11
‑
0.3270x
12
‑
0.04173x
13
‑
1.365x
14
0.1730x
15
0.2213x
16
‑
5.996x
17
‑
0.2242x
18
0.01693x
19
0.2991x
20
‑
0.3007x
21
0.04562x
22
3.766;
22.p(7)= 0.01173x1‑
0.05926x2 0.04441x3‑
0.4263x4‑
0.2526x5 0.6295x6‑
0.2542x7‑
0.02312x8‑
1.104x9 0.06854x
10
0.1134x
11
1.756x
12
0.5732x
13
‑
0.4101x
14
0.2815x
15
0.1898x
16
‑
4.937x
17
‑
0.07380x
18
‑
0.01904x
19
‑
0.2896x
20
‑
0.1891x
21
0.3324x
22
‑
0.1618;
23.p(8)= 0.1105x1‑
0.02617x2 0.02652x3‑
0.8271x4‑
0.05887x5 0.5631x6 0.02257x7‑
0.1536x8‑
1.503x9 0.3102x
10
‑
0.9672x
11
0.7700x
12
0.3457x
13
‑
0.06278x
14
0.2976x
15
0.1385x
16
‑
0.9929x
17
0.06927x
18
0.03850x
19
‑
0.1615x
20
‑
0.3619x
21
0.3208x
22
0.2358;
24.p(9)= 0.02127x1‑
0.1786x2‑
0.06685x3‑
0.4015x4 0.3178x5‑
0.1032x6 0.2255x7‑
0.1113x8‑
0.9447x9 0.08232x
10
‑
1.404x
11
‑
0.8030x
12
0.6998x
13
0.1931x14 0.3646x
15
0.2034x
16
‑
0.09102x
17
0.08943x
18
0.02135x
19
‑
0.2601x
20
‑
0.3098x
21
0.7253x
22
0.9554;
25.p(10)=
‑
0.1770x1‑
0.1774x2 0.006306x3‑
0.2404x4‑
0.04884x5‑
0.2527x6 0.02039x7 0.03926x8‑
1.702x9 0.07748x
10
0.5582x
11
‑
2.127x
12
0.2517x
13
0.8850x
14
0.1794x
15
0.04091x
16
5.594x
17
0.1376x
18
‑
0.05464x
19
0.2750x
20
‑
0.2324x
21
0.6710x
22
2.790;
26.p(11)=
‑
0.05616x1‑
0.06217x2‑
0.02304x3‑
0.1938x4 0.2139x5 0.1186x6 0.2535x7 0.08223x8‑
0.5295x9 0.3386x
10
2.094x
11
‑
1.292x
12
‑
0.1709x
13
1.838x
14
0.1669x
15
‑
0.1044x
16
0.6906x
17
0.01153x
18
‑
0.1321x
19
0.02713x
20
‑
0.1830x
21
0.8217x
22
0.6820;
27.p(12)=
‑
0.3046x1‑
0.01073x2‑
0.02445x3‑
0.6758x4 0.2464x5‑
0.03164x6 0.01964x7 0.1548x8‑
0.7268x9 0.3027x
10
‑
1.149x
11
0.1688x
12
‑
0.1605x
13
‑
1.350x
14
0.1300x
15
‑
0.3169x
16
‑
0.3204x
17
0.007568x
18
‑
0.06604x
19
0.3098x
20
0.02328x
21
0.2713x
22
0.8352;
28.p(13)=
‑
0.04337x1‑
0.08791x2 0.07980x3‑
0.6369x4 0.1290x5‑
0.7322x6 0.3031x7 0.1606x8‑
0.8637x9 0.2216x
10
0.1595x
11
0.7907x
12
‑
0.6508x
13
‑
3.742x
14
0.1753x
15
‑
0.3929x
16
1.428x
17
‑
0.004318x
18
‑
0.009281x
19
‑
0.3420x
20
0.07558x
21
0.6384x
22
1.523;
29.p(14)= 0.02989x1‑
0.1218x2 0.05419x3‑
0.6072x4 0.1960x5‑
1.762x6 0.02483x7 0.2813x8‑
1.613x9 0.2832x
10
‑
0.1367x
11
2.201x
12
‑
0.6199x
13
‑
0.6117x
14
0.08369x
15
‑
0.4650x
16
‑
2.183x
17
0.06825x
18
0.05490x
19
‑
0.8920x
20
0.06140x
21
0.7572x
22
‑
0.7424;
30.p(15)= 0.1169x1‑
0.1483x2 0.01328x3‑
0.3053x4 0.1536x5‑
0.6943x6‑
0.1216x7 0.06248x8‑
0.6836x9 0.2833x
10
0.9688x
11
2.265x
12
0.1987x
13
‑
0.9578x
14
‑
0.05165x
15
‑
0.6580x
16
‑
0.9617x
17
‑
0.02333x
18
0.06494x
19
0.3898x
20
0.03080x
21
0.9222x
22
‑
0.9825;
31.p(16)=
‑
0.1550x1‑
0.1281x2 0.1309x3‑
0.05866x4‑
0.1344x5‑
1.094x6 0.2870x7 0.06733x8 0.5596x9 0.3920x
10
‑
0.3636x
11
1.244x
12
0.4942x
13
‑
1.000x
14
‑
0.1326x
15
‑
0.6449x
16
‑
2.707x
17
‑
0.04000x
18
0.0535x
19
‑
0.2715x
20
‑
0.06113x
21
1.050x
22
0.9641;
32.p(17)= 0.08381x1‑
0.1054x2‑
0.1433x3‑
0.4815x4‑
0.1768x5‑
0.4814x6‑
0.08084x7 0.1586x8 1.579x9 0.3521x
10
0.2724x
11
0.6562x
12
0.4360x
13
‑
1.893x
14
‑
0.09452x
15
‑
0.1887x
16
1.826x
17
0.05249x
18
0.01277x
19
‑
0.5550x
20
‑
0.07673x
21
0.1618x
22
0.06534;
33.p(18)= 0.07249x1‑
0.1885x2‑
0.1806x3‑
0.09214x4‑
0.3839x5 0.5123x6‑
0.1603x7 0.07763x8 1.838x9 0.3805x
10
‑
1.214x
11
‑
0.5214x
12
‑
0.3239x
13
0.2161x
14
0.01676x
15
‑
0.1699x
16
‑
0.4673x
17
0.004565x
18
0.04535x
19
‑
0.03797x
20
0.01406x
21
0.3348x
22
‑
1.027;
34.p(19)=
‑
0.1175x1‑
0.1880x2‑
0.01379x3‑
0.3925x4‑
0.07044x5‑
0.2917x6 0.09341x7‑
0.06911x8 0.5469x9 0.3977x
10
0.06104x
11
1.581x
12
‑
0.2019x
13
0.1832x
14
‑
0.07002x
15
0.4297x
16
‑
0.4373x
17
‑
0.003169x
18
0.0001358x
19
0.2545x
20
0.08886x
21
‑
0.06477x
22
‑
1.118;
35.p(20)= 0.08123x1‑
0.2253x2 0.08497x3‑
0.5679x4 0.03546x5 0.2170x6‑
0.2738x7‑
0.1333x8‑
0.1056x9 0.5705x
10
‑
1.070x
11
‑
0.9166x
12
‑
0.05864x
13
‑
0.2105x
14
‑
0.1546x
15
0.4839x
16
0.2875x
17
‑
0.09273x
18
‑
0.04427x
19
‑
0.5460x
20
0.1642x
21
0.1383x
22
0.05128;
36.p(21)=
‑
0.02327x1 0.08022x2‑
0.08145x3‑
0.5957x4‑
0.1105x5‑
1.042x6‑
0.2198x7‑
0.1200x8 0.4716x9‑
0.1289x
10
‑
0.7229x
11
0.7876x
12
‑
0.1554x
13
0.3089x
14
‑
0.02132x
15
0.09991x
16
1.897x
17
0.01432x
18
‑
0.04045x
19
‑
0.04416x
20
0.03642x
21
1.123x
22
‑
0.4297;
37.p(22)=
‑
0.04836x1 0.06341x2‑
0.005342x3‑
1.683x4‑
0.05461x5 0.3883x6 0.01348x7 0.002202x8 0.7124x9‑
0.2961x
10
0.02117x
11
‑
0.5521x
12
‑
0.09357x
13
1.039x
14
‑
0.07988x
15
0.2085x
16
‑
0.01309x
17
‑
0.003115x
18
0.03063x
19
0.1140x
20
0.04054x
21
1.328x
22
‑
0.5431;
38.其中,x1:b_sm05_ea(dm),由偶极矩加权的边缘邻接矩阵的5阶矩阵;x2:a_f03[c
‑
o],拓扑距离为3处的c
‑
o键频率;x3:b_sm06_aea(ri),共振积分加权的增强边缘邻接的6阶矩阵;x4:a_gats7i,由电离势加权的滞后为7的geary自相关;x5:b_c
‑
001,甲基数;x6:a_gats2e,由sanderson电负性加权的滞后为2的geary自相关;x7:b_hy,亲水因素;x8:a_pipc09,9阶分子多径数;x9:b_spmin8_bh(e),由sanderson电负性加权的burden矩阵的最小特征值8;x
10
:a_no,氧原子数;x
11
:b_mats3v,由范德瓦尔斯体积加权的滞后3的莫兰自相关;x
12
:a_gats2m,质量加权的滞后2的geary自相关;x
13
:b_naasc,aasc类型的原子数;x
14
:a_eta_betap_a,etaπ和孤对平均vem计数;x
15
:b_pipc10,10阶分子多径数;x
16
:a_ats7m,按质量加权的滞后7(对数函数)的broto
‑
moreau自相关;x
17
:b_spmaxa_ea(dm),由偶极矩加权的边缘邻接矩阵的归一化前导特征值;x
18
:b_sm07_aea(ri),共振积分加权的增强边缘邻接的7阶矩阵;x
19
:a_spad_ea(dm),由偶极矩加权的边缘邻接矩阵的谱绝对偏差;x
20
:a_hy,亲水因素;x
21
:b_nbm,多重键的数量;x
22
:a_gats7v,由范德瓦尔斯体积加权的滞后为7的geary自相关。
[0039]
优选地,在所述的步骤3)中,变量筛选方法如下:
[0040]
首先计算各个特征之间以及与目标值之间的相关系数,根据相关系数大小排序,选取特征或直接删除特征之间相关系数大于阈值的冗余特征,然后在特征集合中找到与类别相关性最大,但特征之间冗余性最小的一组特征,选为最佳自变量子集。
[0041]
优选地,在所述的步骤2)中,使用chemdraw软件,绘得所收集的染料分子结构,将
其分子结构导出smiles字符串形式,再用dragon软件生成相应的描述符。
[0042]
进一步优选地,在所述的步骤3)中,以实验的最大吸收波长值为目标变量,利用dragon软件所生成的描述符作为自变量,删除变量间相关系数为0.99的变量对中的一个变量,保留其中与目标变量相关系数较大的那个自变量,获得初步筛选的自变量数据集和目标变量数据集。
[0043]
优选地,在所述的步骤4)中,对所述数据集进行随机划分为训练集和测试集,按照数据量计算,测试集所占比例不超过整体数据集的20%。
[0044]
优选地,在所述的步骤5)中,对最佳自变量子集进行自变量进行二次筛选,利用最大相关最小冗余(mrmr)方法结合xgboost回归留一法,选出建模的最佳自变量子集。
[0045]
一种实现快速预测染料的最大吸收波长的系统,执行本发明基于xgboost算法快速预测染料的最大吸收波长的方法,其特征在于,包括:
[0046]
输入模块:利用计算机系统,从文献中采集偶氮型染料的分子结构及其对应的最大吸收波长实验值并作为输入数据;
[0047]
数据分析模块:利用输入模块获得的数据,执行基于xgboost算法快速预测染料的最大吸收波长的方法,快速预测待检测的偶氮型染料的最大吸收波长;
[0048]
输出模块:将快速预测待检测的偶氮型染料的最大吸收波长数据输出。
[0049]
本发明与现有技术相比较,具有如下显而易见的突出实质性特点和显著优点:
[0050]
1.绿色环保:本发明避免了繁杂的实验合成过程,无需合成染料分子,无需采购药品试剂,无需购买实验仪器,符合绿色环保理念;
[0051]
2.时效高:本发明使用的自变量均为分子二维结构得到的描述符,通过dragon软件在一分钟内可生成数百个分子的描述符,时效性高,使用便捷,只需一人操作即可完成;
[0052]
3.周期短:本发明经过变量筛选以及xgboost的建模,可提前预测染料分子的最大吸收波长,为染料研发人员提供有利的参考,大大降低研发成本,有效地缩短研发周期,避免了盲目的“试错”实验;
[0053]
4.成本低:本发明在dragon软件生成的自变量基础上进行了一定的筛选和对变量进行转换,再以xgboost建模,操作过程简单,仅仅利用计算机即可完成全部过程。
附图说明
[0054]
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,可以根据本附图说明获得其它实施例的附图。
[0055]
图1为本发明方法的主要流程图。
[0056]
图2为本发明的偶氮染料的最大吸收波长的xgboost回归建模结果图。
[0057]
图3为本发明的偶氮染料的最大吸收波长的xgboost回归留一法交叉验证结果图。
[0058]
图4为本发明的偶氮染料的最大吸收波长的xgboost回归独立测试集结果图。
具体实施方式
[0059]
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完
整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
[0060]
需要说明的是,机器学习或深度学习方法是目前使用较为广泛的数据挖掘方法,在多种算法中,xgboost(extreme gradient boosting)是一种既适用于小样本集,又适用于大规模样本集的并行计算方法,是基于累加迭代的梯度提升决策树,为梯度增强回归树(gbrt)的改进与具体实现。传统的gbrt算法只是利用了泰勒展开式的一阶导数,然而xgboost算法对目标函数进行了改进,引入目标误差函数进行二阶导数的展开,从而提高了模型的预测精度,因此,该方法适用于对染料的最大吸收波长进行快速预测。
[0061]
本发明提出一种基于xgboost算法快速预测染料的最大吸收波长的方法。首先,立足于偶氮染料分子结构的本身特点,利用dragon软件生成描述符,并探讨进一步筛选自变量的方式;其次,基于xgboost的基本原理,研究基于xgboost算法的快速预测染料最大吸收波长的具体框架流程。
[0062]
以下结合具体的实施例子对上述方案做进一步说明,本发明的优选实施例详述如下:
[0063]
实施例一:
[0064]
在本实施例中,请参见图1和图2,一种基于xgboost算法快速预测染料的最大吸收波长的方法,包括步骤:
[0065]
1)利用计算机系统,从文献中查找偶氮型染料分子结构以及对应的最大吸收波长实验值,进行数据预处理,整理样本的分子结构式、获取样本的最大吸收波长实验值,将预处理后的数据集样本作为后续建模的数据集样本;
[0066]
2)对所述数据集的样本使用化学绘图程序,绘得所收集的染料分子结构,利用描述符生成程序生成相应的描述符;
[0067]
3)以所述数据集样本的最大吸收波长实验值为目标变量,使用产生的描述符为自变量,通过变量筛选方法删除冗余自变量,保留最佳自变量子集;
[0068]
4)对所述数据集进行随机划分为训练集和测试集;
[0069]
5)对最佳自变量子集进行自变量进行二次筛选,对筛选出的最佳自变量进行转换,获取新的自变量子集;
[0070]
6)目标变量为以所述步骤1)的数据集为样本,自变量为所述步骤6)的自变量子集,以所述目标变量与自变量,使用xgboost算法构建训练模型,建立偶氮型染料最大吸收波长的快速预测模型;
[0071]
7)根据所建立的偶氮型染料最大吸收波长的快速预测模型和待测的偶氮型染料分子结构,快速预测待检测的偶氮型染料的最大吸收波长。
[0072]
本实施例方法的优势在于无需合成样本、无需送样检测、成本低廉且可同时快速计算大量样本。对于预测染料的最大吸收波长,并为后续筛选符合特定颜色要求的染料提供有利参考,从而提高研发效率,缩短研发周期。
[0073]
实施例二
[0074]
本实施例与实施例一基本相同,特别之处在于:
[0075]
在本实施例中,在所述的步骤2)中,使用chemdraw软件,绘得所收集的染料分子结
构,将其分子结构导出smiles字符串形式,再用dragon软件生成相应的描述符。
[0076]
在本实施例中,在所述的步骤3)中,以实验的最大吸收波长值为目标变量,利用dragon软件所生成的描述符作为自变量,删除变量间相关系数为0.99的变量对中的一个变量,保留其中与目标变量相关系数较大的那个自变量,获得初步筛选的自变量数据集和目标变量数据集。本实施例计算各个特征之间以及与目标值之间的相关系数,根据相关系数大小排序,选取特征或直接删除特征之间相关系数大于阈值的冗余特征,然后在特征集合中找到与类别相关性最大,但特征之间冗余性最小的一组特征,选为最佳自变量子集。
[0077]
在本实施例中,在所述的步骤4)中,对所述数据集进行随机划分为训练集和测试集,按照数据量计算,测试集所占比例不超过整体数据集的20%。
[0078]
在本实施例中,在所述的步骤5)中,对最佳自变量子集进行自变量进行二次筛选,利用最大相关最小冗余(mrmr)方法结合xgboost回归留一法,选出建模的最佳自变量子集。
[0079]
本实施例基于xgboost算法快速预测染料的最大吸收波长的方法,建立数据集样本;生成描述符;随机划分训练集和测试集;自变量筛选;建立快速预测模型。本实施例基于可靠的文献数据和建模方法,所建染料最大吸收波长预测模型具有高效便捷、成本低、绿色环保等优点。
[0080]
实施例三
[0081]
本实施例与上述实施例基本相同,特别之处在于:
[0082]
在本实施例中,在所述步骤5)中,获取的新的自变量为:
[0083]
p(1)=
‑
0.1468x1 0.07953x2 0.005442x3‑
0.1144x4‑
0.03946x5‑
0.7645x6‑
0.1187x7
‑
0.02542x8 0.2509x9 0.2106x10 1.518x11
‑
1.490x12 0.3446x13 1.073x14 0.1408x15 0.008882x16 3.373x17
‑
0.08313x18 0.08137x19
‑
0.1829x20 0.1180x21
‑
0.1042x22
‑
2.007;
[0084]
p(2)=
‑
0.3452x1‑
0.001333x2‑
0.1305x3 0.03473x4 0.06318x5 0.5968x6‑
0.08493x7‑
0.05796x8‑
0.4417x9‑
0.0002596x
10
0.8958x
11
0.6900x
12
‑
0.1266x
13
0.3495x
14
‑
0.002020x
15
‑
0.03937x
16
4.958x
17
‑
0.2235x
18
0.06097x
19
‑
0.8796x
20
‑
0.04725x
21
0.007784x
22
‑
3.015;
[0085]
p(3)=
‑
0.04478x1 0.03094x2 0.1164x3 0.4955x4 0.1280x5 0.5930x6‑
0.2875x7 0.1254x8 0.4020x9 0.08689x
10
‑
0.6100x
11
1.735x
12
‑
0.01940x
13
0.004682x
14
0.05438x
15
0.2704x
16
6.064x
17
‑
0.1437x
18
0.04459x
19
0.1100x
20
‑
0.09971x
21
0.5054x
22
‑
3.849;
[0086]
p(4)=
‑
0.009321x1‑
0.06079x2 0.2526x3‑
0.2809x4 0.1216x5 0.06794x6‑
0.4561x7 0.05122x8 1.249x9‑
0.07666x
10
‑
0.7976x
11
2.530x
12
‑
0.03591x
13
1.490x
14
0.1424x
15
0.0004158x
16
7.116x
17
‑
0.1939x
18
‑
0.004350x
19
‑
0.03845x
20
‑
0.1580x
21
‑
0.1890x
22
‑
3.298;
[0087]
p(5)= 0.1486x1‑
0.04740x2 0.06389x3‑
0.4567x4‑
0.08375x5‑
1.068x6‑
0.1309x7 0.08396x8‑
0.2237x9‑
0.02917x
10
‑
1.069x
11
0.6590x
12
0.09570x
13
0.5122x
14
0.1219x
15
0.1383x
16
1.394x
17
‑
0.3684x
18
‑
0.005981x
19
0.4988x
20
‑
0.2064x
21
‑
0.1966x
22
0.1455;
[0088]
p(6)=
‑
0.06321x1‑
0.06233x2 0.09175x3‑
0.4032x4‑
0.03185x5‑
0.8999x6‑
0.6305x7 0.006485x8‑
0.08835x9‑
0.004323x
10
0.4255x
11
‑
0.3270x
12
‑
0.04173x
13
‑
1.365x
14
0.1730x
15
0.2213x
16
‑
5.996x
17
‑
0.2242x
18
0.01693x
19
0.2991x
20
‑
0.3007x
21
0.04562x
22
3.766;
[0089]
p(7)= 0.01173x1‑
0.05926x2 0.04441x3‑
0.4263x4‑
0.2526x5 0.6295x6‑
0.2542x7‑
0.02312x8‑
1.104x9 0.06854x
10
0.1134x
11
1.756x
12
0.5732x
13
‑
0.4101x
14
0.2815x
15
0.1898x
16
‑
4.937x
17
‑
0.07380x
18
‑
0.01904x
19
‑
0.2896x
20
‑
0.1891x
21
0.3324x
22
‑
0.1618;
[0090]
p(8)= 0.1105x1‑
0.02617x2 0.02652x3‑
0.8271x4‑
0.05887x5 0.5631x6 0.02257x7‑
0.1536x8‑
1.503x9 0.3102x
10
‑
0.9672x
11
0.7700x
12
0.3457x
13
‑
0.06278x
14
0.2976x
15
0.1385x
16
‑
0.9929x
17
0.06927x
18
0.03850x
19
‑
0.1615x
20
‑
0.3619x
21
0.3208x
22
0.2358;
[0091]
p(9)= 0.02127x1‑
0.1786x2‑
0.06685x3‑
0.4015x4 0.3178x5‑
0.1032x6 0.2255x7‑
0.1113x8‑
0.9447x9 0.08232x
10
‑
1.404x
11
‑
0.8030x
12
0.6998x
13
0.1931x14 0.3646x
15
0.2034x
16
‑
0.09102x
17
0.08943x
18
0.02135x
19
‑
0.2601x
20
‑
0.3098x
21
0.7253x
22
0.9554;
[0092]
p(10)=
‑
0.1770x1‑
0.1774x2 0.006306x3‑
0.2404x4‑
0.04884x5‑
0.2527x6 0.02039x7 0.03926x8‑
1.702x9 0.07748x
10
0.5582x
11
‑
2.127x
12
0.2517x
13
0.8850x
14
0.1794x
15
0.04091x
16
5.594x
17
0.1376x
18
‑
0.05464x
19
0.2750x
20
‑
0.2324x
21
0.6710x
22
2.790;
[0093]
p(11)=
‑
0.05616x1‑
0.06217x2‑
0.02304x3‑
0.1938x4 0.2139x5 0.1186x6 0.2535x7 0.08223x8‑
0.5295x9 0.3386x
10
2.094x
11
‑
1.292x
12
‑
0.1709x
13
1.838x
14
0.1669x
15
‑
0.1044x
16
0.6906x
17
0.01153x
18
‑
0.1321x
19
0.02713x
20
‑
0.1830x
21
0.8217x
22
0.6820;
[0094]
p(12)=
‑
0.3046x1‑
0.01073x2‑
0.02445x3‑
0.6758x4 0.2464x5‑
0.03164x6 0.01964x7 0.1548x8‑
0.7268x9 0.3027x
10
‑
1.149x
11
0.1688x
12
‑
0.1605x
13
‑
1.350x
14
0.1300x
15
‑
0.3169x
16
‑
0.3204x
17
0.007568x
18
‑
0.06604x
19
0.3098x
20
0.02328x
21
0.2713x
22
0.8352;
[0095]
p(13)=
‑
0.04337x1‑
0.08791x2 0.07980x3‑
0.6369x4 0.1290x5‑
0.7322x6 0.3031x7 0.1606x8‑
0.8637x9 0.2216x
10
0.1595x
11
0.7907x
12
‑
0.6508x
13
‑
3.742x
14
0.1753x
15
‑
0.3929x
16
1.428x
17
‑
0.004318x
18
‑
0.009281x
19
‑
0.3420x
20
0.07558x
21
0.6384x
22
1.523;
[0096]
p(14)= 0.02989x1‑
0.1218x2 0.05419x3‑
0.6072x4 0.1960x5‑
1.762x6 0.02483x7 0.2813x8‑
1.613x9 0.2832x
10
‑
0.1367x
11
2.201x
12
‑
0.6199x
13
‑
0.6117x
14
0.08369x
15
‑
0.4650x
16
‑
2.183x
17
0.06825x
18
0.05490x
19
‑
0.8920x
20
0.06140x
21
0.7572x
22
‑
0.7424;
[0097]
p(15)= 0.1169x1‑
0.1483x2 0.01328x3‑
0.3053x4 0.1536x5‑
0.6943x6‑
0.1216x7 0.06248x8‑
0.6836x9 0.2833x
10
0.9688x
11
2.265x
12
0.1987x
13
‑
0.9578x
14
‑
0.05165x
15
‑
0.6580x
16
‑
0.9617x
17
‑
0.02333x
18
0.06494x
19
0.3898x
20
0.03080x
21
0.9222x
22
‑
0.9825;
[0098]
p(16)=
‑
0.1550x1‑
0.1281x2 0.1309x3‑
0.05866x4‑
0.1344x5‑
1.094x6 0.2870x7 0.06733x8 0.5596x9 0.3920x
10
‑
0.3636x
11
1.244x
12
0.4942x
13
‑
1.000x
14
‑
0.1326x
15
‑
0.6449x
16
‑
2.707x
17
‑
0.04000x
18
0.0535x
19
‑
0.2715x
20
‑
0.06113x
21
1.050x
22
0.9641;
[0099]
p(17)= 0.08381x1‑
0.1054x2‑
0.1433x3‑
0.4815x4‑
0.1768x5‑
0.4814x6‑
0.08084x7 0.1586x8 1.579x9 0.3521x
10
0.2724x
11
0.6562x
12
0.4360x
13
‑
1.893x
14
‑
0.09452x
15
‑
0.1887x
16
1.826x
17
0.05249x
18
0.01277x
19
‑
0.5550x
20
‑
0.07673x
21
0.1618x
22
0.06534;
[0100]
p(18)= 0.07249x1‑
0.1885x2‑
0.1806x3‑
0.09214x4‑
0.3839x5 0.5123x6‑
0.1603x7 0.07763x8 1.838x9 0.3805x
10
‑
1.214x
11
‑
0.5214x
12
‑
0.3239x
13
0.2161x
14
0.01676x
15
‑
0.1699x
16
‑
0.4673x
17
0.004565x
18
0.04535x
19
‑
0.03797x
20
0.01406x
21
0.3348x
22
‑
1.027;
[0101]
p(19)=
‑
0.1175x1‑
0.1880x2‑
0.01379x3‑
0.3925x4‑
0.07044x5‑
0.2917x6 0.09341x7‑
0.06911x8 0.5469x9 0.3977x
10
0.06104x
11
1.581x
12
‑
0.2019x
13
0.1832x
14
‑
0.07002x
15
0.4297x
16
‑
0.4373x
17
‑
0.003169x
18
0.0001358x
19
0.2545x
20
0.08886x
21
‑
0.06477x
22
‑
1.118;
[0102]
p(20)= 0.08123x1‑
0.2253x2 0.08497x3‑
0.5679x4 0.03546x5 0.2170x6‑
0.2738x7‑
0.1333x8‑
0.1056x9 0.5705x
10
‑
1.070x
11
‑
0.9166x
12
‑
0.05864x
13
‑
0.2105x
14
‑
0.1546x
15
0.4839x
16
0.2875x
17
‑
0.09273x
18
‑
0.04427x
19
‑
0.5460x
20
0.1642x
21
0.1383x
22
0.05128;
[0103]
p(21)=
‑
0.02327x1 0.08022x2‑
0.08145x3‑
0.5957x4‑
0.1105x5‑
1.042x6‑
0.2198x7‑
0.1200x8 0.4716x9‑
0.1289x
10
‑
0.7229x
11
0.7876x
12
‑
0.1554x
13
0.3089x
14
‑
0.02132x
15
0.09991x
16
1.897x
17
0.01432x
18
‑
0.04045x
19
‑
0.04416x
20
0.03642x
21
1.123x
22
‑
0.4297;
[0104]
p(22)=
‑
0.04836x1 0.06341x2‑
0.005342x3‑
1.683x4‑
0.05461x5 0.3883x6 0.01348x7 0.002202x8 0.7124x9‑
0.2961x
10
0.02117x
11
‑
0.5521x
12
‑
0.09357x
13
1.039x
14
‑
0.07988x
15
0.2085x
16
‑
0.01309x
17
‑
0.003115x
18
0.03063x
19
0.1140x
20
0.04054x
21
1.328x
22
‑
0.5431
[0105]
其中,x1:b_sm05_ea(dm),由偶极矩加权的边缘邻接矩阵的5阶矩阵;x2:a_f03[c
‑
o],拓扑距离为3处的c
‑
o键频率;x3:b_sm06_aea(ri),共振积分加权的增强边缘邻接的6阶矩阵;x4:a_gats7i,由电离势加权的滞后为7的geary自相关;x5:b_c
‑
001,甲基数;x6:a_gats2e,由sanderson电负性加权的滞后为2的geary自相关;x7:b_hy,亲水因素;x8:a_pipc09,9阶分子多径数;x9:b_spmin8_bh(e),由sanderson电负性加权的burden矩阵的最小特征值8;x
10
:a_no,氧原子数;x
11
:b_mats3v,由范德瓦尔斯体积加权的滞后3的莫兰自相关;x
12
:a_gats2m,质量加权的滞后2的geary自相关;x
13
:b_naasc,aasc类型的原子数;x
14
:a_eta_betap_a,etaπ和孤对平均vem计数;x
15
:b_pipc10,10阶分子多径数;x
16
:a_ats7m,按质量加权的滞后7(对数函数)的broto
‑
moreau自相关;x
17
:b_spmaxa_ea(dm),由偶极矩加权的边缘邻接矩阵的归一化前导特征值;x
18
:b_sm07_aea(ri),共振积分加权的增强边缘邻接的7阶矩阵;x
19
:a_spad_ea(dm),由偶极矩加权的边缘邻接矩阵的谱绝对偏差;x
20
:a_hy,亲水因素;x
21
:b_nbm,多重键的数量;x
22
:a_gats7v,由范德瓦尔斯体积加权的滞后为7的geary自相关。
[0106]
本实施例对筛选出的最佳自变量进行转换,获得新的自变量,提供丰富的变量条件和变量资源。
[0107]
实施例四
[0108]
本实施例与上述实施例基本相同,特别之处在于:
[0109]
在本实施例中,一种基于xgboost算法快速预测染料的最大吸收波长的方法,包括以下步骤:
[0110]
(1)利用计算机系统,在文献中查找单偶氮型染料分子结构以及对应的最大吸收波长实验值,共找到符合要求的单偶氮型染料分子212个,部分结构以及最大吸收波长值如表1所示:
[0111]
表1.部分文献中单偶氮型染料分子结构及其最大吸收波长实验值表
[0112][0113]
(2)使用dragon软件对chemdraw软件绘出的偶氮染料二维结构生成的描述符共10540个,部分描述符如表2所示:
[0114]
表2.dragon生成的部分偶氮染料描述符表
[0115]
a_mwa_amwa_sva_sea_mvb_nskb_ntab_nbmb_scbob_rbn121.176.73211.08517.8040.6161573182200.268.70714.72423.6360.64933120152.148.94911.48317.7930.675933120242.357.57319.30432.2870.603157318298.169.8166.99910.0040.71143141205.2310.80214.4519.760.761114314185.1214.1874.9666.3380.8281143141
99.1511.0176.4939.2220.7211143141242.357.57319.30432.2870.6031354181152.148.94911.48317.7930.6751573182
[0116]
(3)以最大吸收波长值为目标变量,dragon生成的描述符为自变量,以变量自相关为0.99初步筛选自变量,获得2173个描述符;
[0117]
(4)随机划分训练集和测试集,比例为4:1,训练集与测试集的样本量分别为169和43;
[0118]
(5)以最大相关最小冗余(mrmr)结合xgboost回归算法筛选描述符,选出了22个最佳描述符分别为x1:b_sm05_ea(dm),由偶极矩加权的边缘邻接矩阵的5阶矩阵;x2:a_f03[c
‑
o],拓扑距离为3处的c
‑
o键频率;x3:b_sm06_aea(ri),共振积分加权的增强边缘邻接的6阶矩阵;x4:a_gats7i,由电离势加权的滞后为7的geary自相关;x5:b_c
‑
001,甲基数;x6:a_gats2e,由sanderson电负性加权的滞后为2的geary自相关;x7:b_hy,亲水因素;x8:a_pipc09,9阶分子多径数;x9:b_spmin8_bh(e),由sanderson电负性加权的burden矩阵的最小特征值8;x
10
:a_no,氧原子数;x
11
:b_mats3v,由范德瓦尔斯体积加权的滞后3的莫兰自相关;x
12
:a_gats2m,质量加权的滞后2的geary自相关;x
13
:b_naasc,aasc类型的原子数;x
14
:a_eta_betap_a,etaπ和孤对平均vem计数;x
15
:b_pipc10,10阶分子多径数;x
16
:a_ats7m,按质量加权的滞后7(对数函数)的broto
‑
moreau自相关;x
17
:b_spmaxa_ea(dm),由偶极矩加权的边缘邻接矩阵的归一化前导特征值;x
18
:b_sm07_aea(ri),共振积分加权的增强边缘邻接的7阶矩阵;x
19
:a_spad_ea(dm),由偶极矩加权的边缘邻接矩阵的谱绝对偏差;x
20
:a_hy,亲水因素;x
21
:b_nbm,多重键的数量;x
22
:a_gats7v,由范德瓦尔斯体积加权的滞后为7的geary自相关,部分样本的22个最佳描述符的数据如表3所示:
[0119]
表3.22个最佳描述符的部分数据表
[0120]
x1x2x3x4x5x6x7x8x9x
10
x
11
02
‑
31.90961.494
‑
0.39500.7241
‑
0.4074.118600.50100.8062.0423.7103
‑
0.1854.11860000.8812.042003
‑
0.18509
‑
31.46760.998
‑
0.3954.080.7243
‑
0.40700
‑
6.87030.638
‑
0.294000
‑
0.43804
‑
6.871.53230.44
‑
0.2945.602
‑
0.43800
‑
6.87031.13
‑
0.294000
‑
0.43800
‑
6.87030.532
‑
0.294000
‑
0.4382.7749
‑
5.281.46720.998
‑
0.1274.08030.16306
‑
3060.881
‑
0.39500.7243
‑
0.407x
12
x
13
x
14
x
15
x
16
x
17
x
18
x
19
x
20
x
21
x
22
0.87200.44400.4090.101
‑
3.3034.485
‑
0.84831.9980.88700.53800.4720.26304.4850.67930.6430.73200.63600.5270.26309.71
‑
0.61430.6150.73800.2502.7450.101
‑
3.30313.96
‑
0.67331.4990.77800.417000.13808.241
‑
0.61430
0.83300.85702.4120.138013.037
‑
0.62630.4341.27400.6000.13808.791
‑
0.359300.57500.5000.138010.498
‑
0.46300.73800.2502.7450.157
‑
5.4113.96
‑
0.67341.4990.73200.63600.5270.101
‑
3.3039.71
‑
0.61430.615
[0121]
(6)基于上述步骤(5)筛选出的描述符,根据以下公式进行转换,生成新的描述符,详细参见表4所示;
[0122]
表4.转换后生成的部分新的描述符表
[0123]
p(1)p(2)p(3)p(4)p(5)p(6)p(7)p(8)p(9)p(10)p(11)
‑
3.17820.93262.73420.8387
‑
2.0609
‑
0.5855
‑
1.0507
‑
1.14401.6655
‑
1.27250.8755
‑
2.1190
‑
2.87660.6954
‑
0.33380.3965
‑
2.2410
‑
1.10340.7956
‑
0.83930.74530.9077
‑
1.0234
‑
1.2532
‑
0.1511
‑
0.5920
‑
0.4816
‑
2.5011
‑
0.79282.10550.34960.45970.3345
‑
1.07100.54263.7107
‑
0.0215
‑
1.11290.6361
‑
1.6558
‑
0.83770.6952
‑
2.5634
‑
0.6051
‑
2.38970.3521
‑
1.3444
‑
1.0557
‑
0.7755
‑
0.1320
‑
0.83070.41981.70000.3583
‑
1.2323
‑
1.05800.35971.4789
‑
0.9158
‑
0.5661
‑
0.7803
‑
1.4076
‑
0.62870.88400.1023
‑
0.4554
‑
3.31060.8612
‑
0.13870.4932
‑
0.7565
‑
0.90100.19041.04721.2316
‑
0.6188
‑
1.5444
‑
1.76160.1799
‑
1.6415
‑
1.4686
‑
0.69030.0008
‑
1.37540.26061.89810.8092
‑
1.1241
‑
0.19921.13882.6359
‑
1.9418
‑
0.34580.3525
‑
0.2280
‑
0.3770
‑
0.9234
‑
1.5266
‑
0.0878
‑
0.80970.56241.07201.0985
‑
0.40260.3831
‑
1.65180.22171.1666
‑
1.89010.3023p(12)p(13)p(14)p(15)p(16)p(17)p(18)p(19)p(20)p(21)p(22)0.6781
‑
0.2404
‑
0.89220.58031.2131
‑
0.71190.1493
‑
0.80470.50990.4298
‑
0.26510.23570.99950.03690.72230.82110.0873
‑
0.2796
‑
0.0590
‑
0.8305
‑
0.2994
‑
0.3939
‑
0.93200.53230.1565
‑
0.08060.79330.1857
‑
0.1140
‑
0.15760.58440.09470.64440.94600.1229
‑
0.1649
‑
0.58850.40050.1342
‑
0.4190
‑
0.45440.38360.14070.15401.07890.34720.74190.1733
‑
0.36050.30580.0274
‑
0.1176
‑
0.03680.3212
‑
0.1299
‑
0.0725
‑
1.80641.1486
‑
1.0105
‑
0.7966
‑
0.27950.24660.1686
‑
0.5786
‑
0.2804
‑
1.45960.9426
‑
0.39790.65730.9147
‑
0.5049
‑
0.08660.07570.6217
‑
0.58680.22220.02340.8346
‑
0.11990.4177
‑
0.0857
‑
0.50110.00980.1934
‑
0.3530
‑
0.07520.19930.1140
‑
0.96280.2668
‑
0.09750.1320
‑
0.45440.3061
‑
0.3581
‑
0.73230.04250.0039
‑
0.20331.5820
‑
0.3709
‑
0.02300.13860.58970.0203
‑
0.12240.18121.29560.45481.0225
[0124]
(7)在上述步骤(6)生成的新的描述符基础上,使用xgboost回归建立偶氮染料的最大吸收波长的快速预测模型;
[0125]
(8)根据建立的偶氮染料的最大吸收波长快速预测模型与待测的偶氮染料分子,快速预测待测的偶氮染料最大吸收波长值;
[0126]
在本实施例中,基于169个xgboost回归算法建立的偶氮染料的最大吸收波长预测模型的建模效果,如图2所示。利用xgboost回归算法对169个偶氮染料的数据进行回归建模,建立单偶氮染料的最大吸收波长的xgboost回归定量预测模型,模型预测值与文献报道的实验值的相关系数为0.99,平均绝对误差为0.04,均方根误差为0.23。
[0127]
在本实施例中,基于169个xgboost回归算法建立的偶氮染料的最大吸收波长预测模型的独立测试集预测结果,如图4所示。通过建立的偶氮染料的xgboost回归算法预测模型对独立测试集的43个样本进行预测,预测结果良好,偶氮染料的预测值与文献报道的实验值的平均绝对误差为28.32。
[0128]
实施例五
[0129]
本实施例与上述实施例基本相同,特别之处在于:
[0130]
在本实施例中,一种实现快速预测染料的最大吸收波长的系统,执行上述实施例所述基于xgboost算法快速预测染料的最大吸收波长的方法,包括:
[0131]
输入模块:利用计算机系统,从文献中采集偶氮型染料的分子结构及其对应的最大吸收波长实验值并作为输入数据;
[0132]
数据分析模块:利用输入模块获得的数据,执行基于xgboost算法快速预测染料的最大吸收波长的方法,快速预测待检测的偶氮型染料的最大吸收波长;
[0133]
输出模块:将快速预测待检测的偶氮型染料的最大吸收波长数据输出。
[0134]
本实施例系统的优势在于,运行程序时无需合成样本、无需送样检测、成本低廉且可同时快速计算大量样本。对于预测染料的最大吸收波长,并为后续筛选符合特定颜色要求的染料提供有利参考,从而提高研发效率,缩短研发周期。
[0135]
上面对本发明实施例结合附图进行了说明,但本发明不限于上述实施例,还可以根据本发明的发明创造的目的做出多种变化,凡依据本发明技术方案的精神实质和原理下做的改变、修饰、替代、组合或简化,均应为等效的置换方式,只要符合本发明的发明目的,只要不背离本发明的技术原理和发明构思,都属于本发明的保护范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。