1.本发明涉及生物信息学和大数据技术领域,尤其涉及基于深度学习框架的基于图卷积网络的微生物与疾病关联关系预测方法及系统。
背景技术:
2.研究表明,人体内的微生物群不但在人类的某些生理过程中起着重要作用,例如可为人体采集或存储能量、促进人体对碳水化合物的吸收、以及保护人体免受外来微生物和病原体的侵害等,而且微生物群的组成变化还将对人体健康有重要影响。
3.微生物和疾病之间密不可分的关系吸引了科学家们开展许多项目来调查研究微生物和疾病之间的关联,例如人类微生物组工程和地球微生物群工程,旨在深入了解微生物组成及其在人类疾病中的重要作用和建立一个地球微生物多样性的全球目录。另外,为了进一步研究微生物和疾病之间的关联关系,人们先后建立一系列的微生物和疾病关联数据库,基于这些数据库,研究人员提出了大量预测模型和方法来预测微生物和疾病之间的潜在关联。
4.深入了解微生物和疾病之间的关系,不但可以揭露更多人类疾病的发病机制,而且能对疾病的预防、治疗和诊断提供帮助。然而传统的实验方法检测微生物和疾病之间的关联大多为湿实验,例如:独立培养法和定量法。这需要很长且密集的时间来观察,另外,由于疾病种类的多样性,难以验证涉及不同疾病的某些宿主和微生物之间的相互作用是偶然的还是必然的。这就导致了用生物技术上确认微生物和疾病之间的关联耗时长、成本高和具有盲目性等缺点,因此,通过计算方法来预测发掘潜在的微生物和疾病之间关联变得越来越重要。
技术实现要素:
5.本发明目的在于提供微生物与疾病关联关系预测方法,以解决生物实验方法预测微生物与疾病关联关系实验耗时长、成本高和具有盲目性的技术问题。
6.为实现上述目的本发明提供基于图卷积网络的微生物与疾病关联关系预测方法,包括以下步骤:
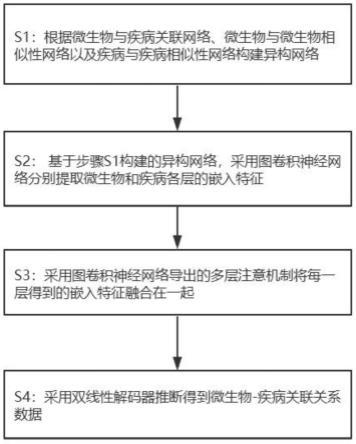
7.s1:根据微生物与疾病关联网络、微生物与微生物相似性网络以及疾病与疾病相似性网络构建异构网络,包括,s101:从hmdad数据库中下载微生物-疾病关联关系数据,其中包括39种疾病和292种微生物之间的483种实验关联通过重复数据消除操作,从hmdad导出了450个关联,涉及39种疾病和292种微生物;令表示下载得到的微生物种类集合,微生物种类集合,表示下载得到的疾病种类集合,得到邻接矩阵对于mi(1≤i≤nm)和dj(1≤j≤nd),若它们之间存在关联则令a
i,j
=1,否则a
i,j
=0;,nm表示微生物种类的个数,nd表示疾病种类的个数。
8.s102:根据微生物-疾病关联网络计算微生物-微生物之间的第一相似性和第二相
似性并构建微生物相似性网络;
9.s103:根据微生物-疾病关联网络来计算疾病-疾病之间的第一相似性和第二相似性来构建疾病相似性网络;
10.s104:基于上述得到的微生物相似性网络、疾病相似性网络和微生物-疾病关联网络,构建一个异构网络;
11.s2:基于步骤s1构建的异构网络,采用图卷积神经网络分别提取微生物和疾病各层的嵌入特征;
12.s3:采用图卷积神经网络导出的多层注意机制将每一层得到的嵌入特征融合在一起;
13.s4:采用双线性解码器推断得到微生物-疾病关联关系数据。
14.进一步的,步骤s1中异构网络的构建,采用如下公式进行计算:
15.整合第一相似性和第二相似性来构建微生物相似性网络,两个微生物{mi,mj}∈m之间的相似性sm定义如下:
[0016][0017]
其中,cm(mi,mj)为微生物mi和微生物mj之间的第二相似性,其计算公式如下:
[0018][0019]
其中,a(i,:)和a(j,:)分别代表邻接矩阵a的第i和第j行;km(mi,mj)为微生物mi和微生物mj之间的第一相似性,其计算公式如下:
[0020]
km(mi,mj)=exp(-γm||a(i,:)-a(j,:)||2)
[0021]
其中||
·
||2表示l2范数,参数γm可计算如下:
[0022][0023]
疾病相似性网络通过疾病第二相似性和第一相似性构建,两个疾病{di,dj}∈d之间的相似性sd定义如下:
[0024][0025]
其中,cd(di,dj)为疾病di和疾病dj之间的第二相似性,其计算公式如下:
[0026][0027]
其中,a(:,i)代表邻接矩阵a的第i列。
[0028]
kd(di,dj)是疾病di和疾病dj之间的第一相似性,其计算公式如下:
[0029]
kd(di,dj)=exp(-γd||a(:,i)-a(:,j)||2)
[0030]
其中||
·
||2表示l2范数,参数γd可计算如下:
[0031][0032]
基于上述得到的微生物相似性矩阵疾病相似性矩阵和微生物-疾病关联关系邻接矩阵a,构建邻接矩阵如下:
[0033][0034]
进一步的,其特征在于步骤s2包括,
[0035]
将步骤s1得到的邻接矩阵作为模型的输入,定义的gcn公式如下:
[0036][0037]
设l表示gcn的节点层数,l∈{1,...,l},h
l
代表第1层节点的嵌入特征,是第1层的权重矩阵,k是第1嵌入特征的维数,σ(
·
)代表非线性激活函数,采用以下整流线性单元relu作为激活函数。
[0038][0039]
进一步的,所述步骤s3包括,
[0040]
h0代表y中节点的初始嵌入特征,初始化h0公式如下:
[0041][0042]
得出微生物和疾病的最终嵌入特征,如下所示:
[0043][0044]
代表微生物的最终的嵌入特征矩阵,代表疾病最终的嵌入特征矩阵,wi代表自学习注意力分数定义如下:
[0045][0046]
进一步的,所述步骤s4包括,
[0047]
采用如下定义的双线性解码器重建潜在的微生物疾病关联的邻接矩阵:
[0048]a′
=f(hm,hd):
[0049][0050]
是训练矩阵。
[0051]
本发明还提供一种基于图卷积网络的微生物与疾病关联关系预测系统,包括存储器、处理器以及存储在存储器上并可在处理器上运行的计算机程序,处理器执行计算机程序时实现上述任一方法的步骤。
[0052]
本发明具有以下有益效果:
[0053]
本发明的图卷积神经网络的微生物与疾病关联关系预测,充分利用异构网络中邻居节点信息和图注意力网络自动提取特征的特点,从而提高微生物与疾病关联关系的预测效果。可为研究人员提供一定的参考信息,大大减少了实验的工作量。
[0054]
除了上面所描述的目的、特征和优点之外,本发明还有其它的目的、特征和优点。下面将参照附图,对本发明作进一步详细的说明。
附图说明
[0055]
构成本技术的一部分的附图用来提供对本发明的进一步理解,本发明的示意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。在附图中:
[0056]
图1是本发明基于多层注意力机制图卷积神经网络的基于图卷积网络的微生物与疾病关联关系预测方法的框架图。
[0057]
图2为本发明流程图。
具体实施方式
[0058]
本发明的发明构思是根据已知的微生物-疾病关联网络、微生物-微生物相似性网络以及疾病-疾病相似性网络构建异构网络;然后,利用图卷积神经网络分别提取微生物和疾病各层的嵌入特征;每一层的这些嵌入特征将通过采用多层注意机制融合在一起;最后,进一步推断微生物与疾病之间的可能关联。本发明充分利用多种相似性度量和端到端的深度学习模型,从而在构建的复杂网络中给待预测的微生物节点与疾病节点建立了更多的连接,从而提高微生物与疾病关联关系的预测效果。
[0059]
本发明提供基于图卷积网络的微生物与疾病关联关系预测方法,包括以下步骤:
[0060]
s1:根据微生物与疾病关联网络、微生物与微生物相似性网络以及疾病与疾病相似性网络构建异构网络,包括,s101:从hmdad数据库中下载微生物-疾病关联关系数据,其中包括39种疾病和292种微生物之间的483种实验关联通过重复数据消除操作,从hmdad导出了450个关联,涉及39种疾病和292种微生物;令表示下载得到的微生物种类集合,微生物种类集合,表示下载得到的疾病种类集合,得到邻接矩阵对于mi(1≤i≤nm)和dj(1≤j≤nd),若它们之间存在关联则令a
i,j
=1,否则a
i,j
=0;,nm表示微生物种类的个数,nd表示疾病种类的个数。
[0061]
s102:根据微生物-疾病关联网络计算微生物-微生物之间的第一相似性和第二相似性并构建微生物相似性网络;
[0062]
s103:根据微生物-疾病关联网络来计算疾病-疾病之间的第一相似性和第二相似性来构建疾病相似性网络;
[0063]
s104:基于上述得到的微生物相似性网络、疾病相似性网络和微生物-疾病关联网络,构建一个异构网络;
[0064]
s2:基于步骤s1构建的异构网络,采用图卷积神经网络分别提取微生物和疾病各层的嵌入特征;
[0065]
s3:采用图卷积神经网络导出的多层注意机制将每一层得到的嵌入特征融合在一
起;
[0066]
s4:采用双线性解码器推断得到微生物-疾病关联关系数据。
[0067]
进一步的,步骤s1中异构网络的构建,采用如下公式进行计算:
[0068]
整合第一相似性和第二相似性来构建微生物相似性网络,两个微生物{mi,mj}∈m之间的相似性sm定义如下:
[0069][0070]
其中,cm(mi,mj)为微生物mi和微生物mj之间的第二相似性,其计算公式如下:
[0071][0072]
其中,a(i,:)和a(j,:)分别代表邻接矩阵a的第i和第j行;km(mi,mj)为微生物mi和微生物mj之间的第一相似性,其计算公式如下:
[0073]
km(mi,mj)=exp(-γm||a(i,:)-a(j,:)||2)
[0074]
其中||
·
||2表示l2范数,参数γm可计算如下:
[0075][0076]
疾病相似性网络通过疾病第二相似性和第一相似性构建,两个疾病{di,dj}∈d之间的相似性sd定义如下:
[0077][0078]
其中,cd(di,dj)为疾病di和疾病dj之间的第二相似性,其计算公式如下:
[0079][0080]
其中,a(:,i)代表邻接矩阵a的第i列。
[0081]
kd(di,dj)是疾病di和疾病dj之间的第一相似性,其计算公式如下:
[0082]
kd(di,dj)=exp(-γd||a(:,i)-a(:,j)||2)
[0083]
其中||
·
||2表示l2范数,参数γd可计算如下:
[0084][0085]
基于上述得到的微生物相似性矩阵疾病相似性矩阵和微生物-疾病关联关系邻接矩阵a,构建邻接矩阵如下:
[0086][0087]
进一步的,其特征在于步骤s2包括,
[0088]
将步骤s1得到的邻接矩阵作为模型的输入,定义的gcn公式如下:
[0089][0090]
设l表示gcn的节点层数,l∈{1,...,l},h
l
代表第1层节点的嵌入特征,是第1层的权重矩阵,k是第1嵌入特征的维数,σ(
·
)代表非线性激活函数,采用以下整流线性单元relu作为激活函数。
[0091][0092]
进一步的,所述步骤s3包括,
[0093]
h0代表y中节点的初始嵌入特征,初始化h0公式如下:
[0094][0095]
得出微生物和疾病的最终嵌入特征,如下所示:
[0096][0097]
代表微生物的最终的嵌入特征矩阵,代表疾病最终的嵌入特征矩阵,wi代表自学习注意力分数定义如下:
[0098][0099]
进一步的,所述步骤s4包括,
[0100]
采用如下定义的双线性解码器重建潜在的微生物疾病关联的邻接矩阵:
[0101]a′
=f(hm,hd):
[0102][0103]
是训练矩阵。
[0104]
本发明还提供一种基于图卷积网络的微生物与疾病关联关系预测系统,包括存储器、处理器以及存储在存储器上并可在处理器上运行的计算机程序,处理器执行计算机程序时实现上述任一方法的步骤。
[0105]
本发明具有以下有益效果:
[0106]
本发明的图卷积神经网络的微生物与疾病关联关系预测,充分利用异构网络中邻居节点信息和图注意力网络自动提取特征的特点,从而提高微生物与疾病关联关系的预测效果。可为研究人员提供一定的参考信息,大大减少了实验的工作量。
[0107]
以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。