基于变构概率对近视/高度近视相关snp风险的排序方法和系统
技术领域
1.本发明涉及生信领域,更具体地,涉及一种基于变构概率对近视/高度近视相关snp风险的排序方法、系统、诊断设备和计算机可读存储介质。
背景技术:
2.近视除了影响视力质量外,近视还会增加永久性视力障碍和潜在致盲疾病的风险,包括近视黄斑病变、视网膜脱离和青光眼,人们普遍认为,近视的发病是环境因素和遗传因素相互作用的结果。随着新一代测序(ngs)的兴起和全基因组关联研究(gwas)的普及,大量近视相关的单核苷酸多态性(snp)位点已被确定,然而,这些snps如何诱导近视的发展。
3.rna剪接是真核细胞基因表达中非常重要的一个生物过程,通过rna剪接,可以产生许多具有功能的、带有编码信息的mrna(信使rna),对生物发育及进化至关重要,与此同时,人类细胞中大量的转录变异源于rna剪接中的遗传干扰,研究表明部分snp可能引起剪接变异,而异常剪接通过调节表达显著影响疾病的发生和发展,新一代测序(ngs)已经发现前所未有数量的剪接异构体,其中许多与各种生理和病理条件有关。另一方面,越来越多的证据表明rna二级结构(rss)广泛参与了大量生物功能过程,包括近视的发生和发展。
技术实现要素:
4.本发明发现snp诱导的rna变构会影响前mrna剪接过程,是导致近视发生和发展的重要途径,故而,基于近视相关snp的研究从rna二级结构的角度计算snp诱导的rna变构对剪接相关元件的影响,从而对snp的近视风险进行排序。
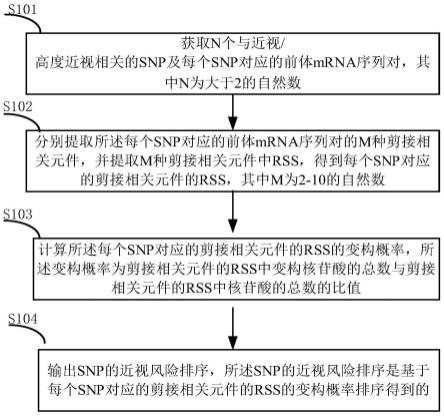
5.本发明提供了一种基于变构概率对近视/高度近视相关snp风险的排序方法,包括:
6.获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对,其中n为大于2的自然数;
7.分别提取所述每个snp对应的前体mrna序列对的m种剪接相关元件,并提取m种剪接相关元件中rss,得到每个snp对应的剪接相关元件的rss,其中m为2-10的自然数;
8.计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;
9.输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率排序得到的。
10.可选的,所述前体mrna序列对为具有和不具有snp的一对或多对前体mrna序列对;rna剪接会产生1个或数个可变剪接,当只存在一个可变剪接时,所述前体mrna序列对为具有和不具有snp的一对前体mrna序列对,当存在多个可变剪接时,所述前体mrna序列对为具有和不具有snp的多对前体mrna序列对;
11.进一步,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:
12.计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;
13.评估每个snp对应的前体mrna序列对的结构异质性,得到每个snp对应的结构异质性得分;
14.输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率及每个snp对应的结构异质性得分的集合;
15.可选的,所述每个snp对应的结构异质性得分为评估snp对前体mrna序列的局部结构异质影响程度,得到每个snp对应的局部结构异质性得分,优选的,通过rnasnp来评估snp对前体mrna序列的局部结构异质影响程度;
16.可选的,所述每个snp对应的结构异质性得分为评估snp对前体mrna序列的全局结构异质影响程度,得到每个snp对应的全局结构异质性得分;优选的,通过rnasmc来评估snp对前体mrna序列的全局结构异质影响程度。
17.进一步,所述每个snp对应的结构异质性得分包括局部结构异质性得分和/或全局结构异质性得分,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分的集合;
18.优选的,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分,分别选择剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分排序靠前的snp组合,得到三组snp组合,并取三组snp组合的交集;
19.优选的,所述snp的近视风险排序是基于每个snp对应的每种剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分,分别基于每种剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分选择排序靠前的snp组合,得到7组snp组合,并取7组snp组合的交集。
20.进一步,所述获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对:
21.获取snp侧翼序列,并截取所述snp上下游各n个碱基作为种子序列,其中n为15-50的自然数;
22.在数据库下载转录本信息作为参考序列;
23.基于短序列比对工具,分别获得snp种子序列对应的参考序列位置;
24.根据snp匹配的正负链及上下游片段对应的起始碱基位置信息,筛选snp定位于参考序列的片段,得到snp及其对应的前体mrna序列对;
25.优选的,根据以下原则筛选snp定位于参考序列的片段pm:
[0026][0027]
ini
up
,ini
down
分别代表snp上游,下游序列匹配到参考序列的碱基起始位置,seed
up
,seed
down
分别对应snp上下游侧翼序列,ref
forward
,ref
reverse
分别表示种子序列位于参考序列的正负链情况。
[0028]
进一步,所述m种剪接相关元件包括下列剪切相关元件中的一种或几种:5'剪接位
点、3'剪接位点、分支点、多嘧啶束mrna剪接过程、剪接调节元件;优选的,所述剪接调节元件包括外显子剪接增强子(ese)、内含子剪接增强剂(ise)、外显子拼接消音器(ess)、内含子拼接消音器;
[0029]
可选的,采用下列方法提取所述前体mrna序列的m种剪接相关元件:基于基因组注释信息提取前体mrna序列的5'剪接位点、3'剪接位点;优选的,采用下列方法中的一种或几种提取前体mrna序列的5'剪接位点、3'剪接位点:deep splicer、splice finder、splice2deep、splicerover、deepss、spliceai;采用下列方法中的一种或几种提取前体mrna序列的分支点、多嘧啶束mrna剪接区域:svm-bpfinder、bpp、branchpointer、labranchor、rnabps;采用下列方法中的一种或几种提取前体mrna序列的剪接调节元件:hsf、protein-specific prediction of rna-binding sites based on information entropy、rbpmmap、graphprot、rna-binding protein targets、ionmf、ideep、circrna-rbp web server;
[0030]
可选的,所述rss包括茎(s)、发夹环(h)、内环(i)、外环(e)、凸起环(b)、多支环(m);可选的,所述提取m种剪接相关元件中rss是采用rna结构预测算法预测剪接相关元件rna的二级结构,进而采用rna motif预测算法提取二级结构中的rss;
[0031]
优选的,所述rna结构预测算法包括下列算法中的一种或几种:rna structure,rnafold,mfold,sfold,maxexpect;优选的,所述rna motif预测算法包括下列算法中的一种或几种:bprna,dotaligner,cmfinder,rnaz,qrna。
[0032]
进一步,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:基于snp的近视风险排序得到x个候选的snp,其中x为自然数整数,对候选的snp进行分子对接能力影响分析,所述分子对接能力影响分析是计算候选的snp对应的不含有snp的前体mrna序列分子对接评分减去含有snp的前体mrna序列分子对接评分差值比不含有snp的前体mrna序列分子对接评分的比值;
[0033]
可选的,所述mrna序列分子对接评分采用hdock server评分。
[0034]
进一步,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:基于snp的近视风险排序得到x个候选的snp,其中x为自然数整数,将候选的snp映射到基因组的基因功能元件中,所述基因功能元件包括外显子、内含子、5’utr和3’utr。
[0035]
本技术的目的在于提供一种基于变构概率对近视/高度近视相关snp风险的排序设备,所述设备包括:存储器和处理器;
[0036]
所述存储器用于存储程序指令;
[0037]
所述处理器用于调用程序指令,当程序指令被执行时,用于执行上述的基于变构概率对近视/高度近视相关snp风险的排序方法。
[0038]
本技术的目的在于提供一种基于变构概率对近视/高度近视相关snp风险的排序系统,包括:
[0039]
获取单元,用于获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对,其中n为大于2的自然数;
[0040]
提取单元,用于分别提取所述每个snp对应的前体mrna序列对的m种剪接相关元件,并提取m种剪接相关元件中rss,得到每个snp对应的剪接相关元件的rss,其中m为2-10的自然数;
[0041]
计算单元,用于计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;
[0042]
排序单元,用于输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率排序得到的。
[0043]
本技术的目的在于提供一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现上述的基于变构概率对近视/高度近视相关snp风险的排序方法。
[0044]
本技术的优点:
[0045]
1.提供了基于变构概率对近视/高度近视相关snp风险的排序方法,从含有或不含有该snp的前体mrna序列对中提取剪接相关元件的rss,并计算snp引起的剪接相关元件的rss的变构概率,量化了snp对rna剪接的影响,进而对多个snp进行风险排序,为发现近视相关snp提供了新的途径;
[0046]
2.本技术还进一步结合了结构异质性评分,包括基于rnasnp的局部结构异质性评分和/或基于rnasmc的全局结构异质性得分,从更多评分维度上进行更为精准的评分;
[0047]
3.本技术还进一步对候选的snp进行分子对接能力影响分析和/或基因组的基因元件位置分析,从候选的snp中筛选有潜在功能的snp,节约后续实验验证成本;
[0048]
4.本技术提出snp在全基因组范围内的编码,非编码基因,及外显子,内含子等功能元件的精确定位算法,由此实现snp在参考序列上端对端的精准、无错配比对。
附图说明
[0049]
为了更清楚地说明本发明实施例中的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获取其他的附图。
[0050]
图1是本发明实施例提供的一种基于变构概率对近视/高度近视相关snp风险的排序方法示意流程图;
[0051]
图2是本发明实施例提供的一种基于变构概率对近视/高度近视相关snp风险的排序的系统示意图;
[0052]
图3是本发明实施例提供的一种基于变构概率对近视/高度近视相关snp风险的排序的设备示意图;
[0053]
图4是本发明具体实施例提供的近视相关snp数据收集图;(a)数据源的venn图;(b)snp的基因定位;(c)基因调控元件的snp定位;(d)眼组织中的基因座分布;(e)角膜中的基因座分布;(f)虹膜中的基因位点分布;(g)视网膜中的基因位点分布;(h)巩膜和脉络膜的基因座分布。;
[0054]
图5是本发明具体实施例提供的近视相关的snp在前体mrna的剪接相关元件中引起大量的结构异质性;(a)snp诱导的前mrna rna二级结构变异的一个例子。红框表示局部结构异质性存在的位置。蓝色方框表示全球结构异质性存在的位置。黑色三角形分别表示snp、起始位点和终止位点的位置;(b)rnasnp中p值的分布。rnasnp网站建议p值》0.2作为结
构变化不太明显;(c)rnasmc得分分布(得分范围为0~10)。rnasmc分数反映了整体结构异质性。得分越低,全球范围内的结构异质性越高。小于9的分数被认为是显著的结构异质性;(d、e)表和venn图显示了特定区域中发生的变构rss与前mrna配对的数量;
[0055]
图6是本发明具体实施例提供的剪接相关元件的结构背景特征参与解释其变构概率;a、b:剪接相关元件的rna变构图谱。蓝色方框表示外显子。浅蓝色框表示内含子。虚线分别表示5'ss、bp位点和3'ss。mann-whitney u非参数检验的统计检验;c剪接相关元件的rna结构分析。mann-whitney u非参数检验的统计检验;d描述变构概率和结构基序比线性关系的散点图。r:皮尔逊相关系数。e、使用皮尔逊相关检验的变构概率和结构基序的相关矩阵。*:p值≤0.05,**:p值≤0.01,***:p值≤0.001,****:p值≤0.0001.;
[0056]
图7是本发明具体实施例提供的剪接相关元件的变构图谱;a-e rss的整体变构特征谱;图中的纵轴表示变构概率(%),其上限为2%,横轴表示每种变构类型;f-h 3种变构模式中每种变构类型的排名分数,通过wilcoxon秩和检验进行统计检验。*:p值《0.05,**:p值《0.01,***:p值《0.001和****:p值《0.0001;
[0057]
图8是本发明具体实施例提供的距离和基因组位置因素参与调节snp变构效应;a可能参与调节变构概率(ap)的因素示意图;b直方图显示了不同距离区间下的变构概率。横轴表示核苷酸和snp之间的绝对距离(nt)。纵轴表示变构概率的百分比。横向虚线表示5%变构概率。垂直的粉红色虚线将距离效应分为远端和近端占优势;c显示近端和远端变构特征的优势比占优势。黑框表示无显著优势比。蓝色框表示比值比(p《0.05)小于1。黄色框表示比值比(p《0.01)大于1;d violin图显示了不同基因组片段(外显子与内含子)下的变构概率。进行配对t检验以分析每个变构结构基序中的ap差异。**:p值≤0.01,***:p值≤0.001,****:p值≤0.0001
[0058]
图9是本发明具体实施例提供的具有高结构异质性风险的snp广泛影响rna和剪接相关蛋白之间的分子相互作用图;a维恩图表示rnasnp、rnasmc和5种剪接相关元件筛选的交集;b前10名高风险snp;c显示rbp-rna分子对接状态变化的对接信息散点图,绿点表示hdock分数偏移,紫色和粉色点分别表示rna结合残基的平均值和中值偏移;
[0059]
图10是本发明具体实施例提供的lim2第一内含子5
′
s处的rna结构特征可调节剪接;a、b实验设计示意图,以验证5'ss的二级结构在拼接过程中的作用,为了与整个5'ss完全形成牢固的发夹结构,在原始5'ss序列(粉色方框和圆圈)的上游立即插入一个短序列(蓝色方框和圆圈);c、d通过hek293t中的小基因剪接分析确定剪接事件,使用同一对引物通过rt-pcr鉴定未剪接和剪接产物,并以不同长度的条带显示,通过image j/fiji测量未切片、拼接产品和背景的平均灰度值,以计算拼接效率,e显示lim2 rna和u1snrnp之间对接状态变化幅度的雷达图;
[0060]
图11是本发明具体实施例提供的近视的具体剪接相关元件的各核酸位点的rss的变构概率a。
具体实施方式
[0061]
为了使本技术领域的人员更好地理解本发明方案,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述。
[0062]
在本发明的说明书和权利要求书及上述附图中的描述的一些流程中,包含了按照
特定顺序出现的多个操作,但是应该清楚了解,这些操作可以不按照其在本文中出现的顺序来执行或并行执行,操作的序号如s101、s102等,仅仅是用于区分开各个不同的操作,序号本身不代表任何的执行顺序。另外,这些流程可以包括更多或更少的操作,并且这些操作可以按顺序执行或并行执行。需要说明的是,本文中的“第一”、“第二”等描述,是用于区分不同的消息、设备、模块等,不代表先后顺序,也不限定“第一”和“第二”是不同的类型。
[0063]
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域技术人员在没有作出创造性劳动前提下所获取的所有其他实施例,都属于本发明保护的范围。
[0064]
图1是本发明实施例提供的一种基于变构概率对近视/高度近视相关snp风险的排序方法示意流程图,具体地,所述方法包括如下步骤:
[0065]
s101:获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对,其中n为大于2的自然数;
[0066]
在一个实施例中,所述前体mrna序列对为具有和不具有snp的一对或多对前体mrna序列对;rna剪接会产生1个或数个可变剪接,当只存在一个可变剪接时,所述前体mrna序列对为具有和不具有snp的一对前体mrna序列对,当存在多个可变剪接时,所述前体mrna序列对为具有和不具有snp的多对前体mrna序列对。
[0067]
在一个实施例中,所述获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对:
[0068]
获取snp侧翼序列,并截取所述snp上下游各n个碱基作为种子序列,其中n为15-50的自然数;
[0069]
在数据库下载转录本信息作为参考序列;
[0070]
基于短序列比对工具,分别获得snp种子序列对应的参考序列位置;
[0071]
根据snp匹配的正负链及上下游片段对应的起始碱基位置信息,筛选snp定位于参考序列的片段,得到snp及其对应的前体mrna序列对。
[0072]
在一个具体实施例中,所述获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对的方法为:首先,我们在dbsnp数据库中获取snp侧翼序列,为保证比对结果的精确性,分别截取snp上下游各30个碱基,作为种子序列;其次,在ensmbl,gencode等数据库下载基因或功能元件对应的转录本信息,作为参考序列。最后,基于短序列比对工具-bowtie 2,设置严格比对参数,如
“‑‑
n-ceil c,3
‑‑
np 0
‑‑
end-to-end-a
‑‑
score-min c,0”,分别获得snp上下游的种子对应的参考序列位置。根据snp匹配的正负链及上下游片段对应的起始碱基位置信息,根据以下原则筛选snp精确定位于参考序列的片段pm:
[0073][0074]
ini
up
,ini
down
分别代表snp上游,下游序列匹配到参考序列的碱基起始位置,seed
up
,seed
down
分别对应snp上下游侧翼序列,ref
forward
,ref
reverse
分别表示种子序列位于参考序列的正负链情况。pm为snp精确比对到基因等参考序列上,由此实现snp在参考序列上端对端的精准、无错配比对。
[0075]
在一个实施例中,获取n个与近视/高度近视相关的snp的方法为:基于数据库检索
方式获取近视/高度近视相关snp集合,将待测snp与snp集合匹配,当待测snp在snp集合范围内,得到n个与近视/高度近视相关的snp,进行后续方法步骤;
[0076]
s102:分别提取所述每个snp对应的前体mrna序列对的m种剪接相关元件,并提取m种剪接相关元件中rss,得到每个snp对应的剪接相关元件的rss,其中m为2-10的自然数;
[0077]
在一个实施例中,所述m种剪接相关元件包括下列剪切相关元件中的一种或几种:5'剪接位点、3'剪接位点、分支点、多嘧啶束mrna剪接区域、剪接调节元件;优选的,所述剪接调节元件包括外显子剪接增强子(ese)、内含子剪接增强剂(ise)、外显子拼接消音器(ess)、内含子拼接消音器;
[0078]
在一个具体实施例中,采用下列方法提取所述前体mrna序列的m种剪接相关元件:基于基因组注释信息提取前体mrna序列的5'剪接位点、3'剪接位点;优选的,采用下列方法中的一种或几种提取前体mrna序列的5'剪接位点、3'剪接位点:deep splicer、splice finder、splice2deep、splicerover、deepss、spliceai;采用下列方法中的一种或几种提取前体mrna序列的分支点、多嘧啶束mrna剪接区域:svm-bpfinder、bpp、branchpointer、labranchor、rnabps;采用下列方法中的一种或几种提取前体mrna序列的剪接调节元件:hsf、protein-specific prediction of rna-binding sites based on information entropy、rbpmmap、graphprot、rna-binding protein targets、ionmf、ideep、circrna-rbp web server。
[0079]
在一个实施例中,所述rss包括茎(s)、发夹环(h)、内环(i)、外环(e)、凸起环(b)、多支环(m);可选的,所述提取m种剪接相关元件中rss是采用rnafold算法预测剪接相关元件rna的二级结构,进而采用bprna提取二级结构中的rss。
[0080]
s103:计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;
[0081]
在一个具体实施例中,计算所述每个snp对应的剪接相关元件的rss的变构概率ap,对于前体mrna序列区域,整体变构概率ap的计算公式为:
[0082][0083]
其中,apr表示序列区域内的变构概率,n1表示序列区域中变构核苷酸的总数,n2表示序列区域中核苷酸的总数。
[0084]
在一个实施例中,所述s103的另一种方法为:计算所述每个snp对应的每种剪接相关元件中各核酸位点的rss的变构概率,所述变构概率为每种剪接相关元件中各核酸位点的rss变构核苷酸的总数与每种剪接相关元件中各核酸位点的rss核苷酸的总数的比值。
[0085]
在一个具体实施例中,计算述每个snp对应的每种剪接相关元件中各核酸位点的rss的变构概率ap,对于前体mrna序列区域,各核酸位点的rss的变构概率ap的计算公式为:
[0086][0087]
其中,apn表示序列区域内各核酸位点的rss的变构概率,n1表示序列区域中特定位点中变构核苷酸的总数,n2表示序列区域中特定位点中核苷酸的总数。
[0088]
在一个具体实施例中,计算每种剪接相关元件中各核酸位点的rss的变构概率ap,结果如图11所示。
[0089]
所述变构核苷酸是指相对不具有snp的前体mrna序列,具有snp的前体mrna序列rss发生改变的核苷酸。
[0090]
s104:输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率排序得到的。
[0091]
在一个实施例中,输出snp的近视风险排序,所述snp的近视风险排序为每种剪接相关元件的rss的变构概率的集合;具体如下:
[0092]
snp
risk
∈{ap5′
ss
,ap3′
ss
,ap
bp
,ap
ppt
,ap
sre
}
[0093]
其中,ap5′
ss
为5'剪接位点的变构概率,ap3′
ss
为3'剪接位点的变构概率,ap
bp
为分支点的变构概率,ap
ppt
为多嘧啶束mrna剪接区域的变构概率,ap
sre
为剪接调节元件的变构概率。
[0094]
在一个实施例中,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括计算所述snp与发生变构的碱基的距离,当距离低于阈值,输出snp的近视风险排序中加入距离风险提示项;可选的,所述snp与发生变构的碱基的距离为snp在基因组上的绝对位置与发生变构的碱基在基因组上的绝对位置的距离;可选的,所述阈值为350bp。
[0095]
在一个实施例中,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;评估每个snp对应的前体mrna序列对的结构异质性,得到每个snp对应的结构异质性得分;输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率及每个snp对应的结构异质性得分的集合;可选的,所述每个snp对应的结构异质性得分为评估snp对前体mrna序列的局部结构异质影响程度,得到每个snp对应的局部结构异质性得分,优选的,通过rnasnp来评估snp对前体mrna序列的局部结构异质影响程度;可选的,所述每个snp对应的结构异质性得分为评估snp对前体mrna序列的全局结构异质影响程度,得到每个snp对应的全局结构异质性得分;优选的,通过rnasmc来评估snp对前体mrna序列的全局结构异质影响程度。
[0096]
在一个实施例中,所述通过rnasmc来评估snp对前体mrna序列的全局结构异质影响程度为:获得前体mrna序列对预测的二级结构,提取所述前体mrna序列对的rss,通过序列比对,对前体mrna序列对的序列进行比较、匹配、评分和重构,找到最优匹配区域,选取最优匹配区域序列中rss,比较所述rss,获得前体mrna序列对在所述最优匹配区域内rss的相似度评分;
[0097]
在一个具体实施例中,所述相似度评分计算公式如下:
[0098][0099]
其中,b、e、h、i、m、s分别表示前体mrna序列中的凸环、外环、发夹环、内环、多分支环、茎;up1和up2表示每种rss的两个rna的空间排列位置集;un1和un2表示这两个rna中rss的个数;相似度评分的取值范围是0~10。
[0100]
在一个实施例中,所述每个snp对应的结构异质性得分包括局部结构异质性得分和/或全局结构异质性得分,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分的集合;优选的,所述
snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分,分别选择剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分排序靠前的snp组合,得到三组snp组合,并取三组snp组合的交集;优选的,所述snp的近视风险排序是基于每个snp对应的每种剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分,分别基于每种剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分选择排序靠前的snp组合,得到7组snp组合,并取7组snp组合的交集;
[0101]
优选的,所述近视风险排序为7组snp组合的交集:
[0102]
snp
risk
∈{rnasnp
score
,rnasmc
score
,ap5′
ss
,ap3′
ss
,ap
bp
,ap
ppt
,ap
sre
}
[0103]
其中,rnasnp
score
为局部结构异质性得分,rnasmc
score
为全局结构异质性得分,ap5′
ss
为5'剪接位点的变构概率,ap3′
ss
,为3'剪接位点的变构概率,ap
bp
为分支点的变构概率,ap
ppt
为多嘧啶束mrna剪接区域的变构概率,ap
sre
为剪接调节元件的变构概率。
[0104]
在一个实施例中,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:基于snp的近视风险排序得到x个候选的snp,其中x为自然数整数,对候选的snp进行分子对接能力影响分析,所述分子对接能力影响分析是计算候选的snp对应的不含有snp的前体mrna序列分子对接评分减去含有snp的前体mrna序列分子对接评分差值比不含有snp的前体mrna序列分子对接评分的比值;所述mrna序列分子对接评分采用hdock server评分。
[0105]
在一个具体实施例中,获得含有与不含有snp前体mrna序列对的三维结构的pdb格式文件,利用hdock软件,我们获得了rna结合残基的对接能量评分和位置信息,以量化前体mrna序列对之间结合亲和力和对接位点残基的差异:
[0106][0107]
δd作为对接能量分数的相对差,d1表示不含有snp的前体mrna序列对接评分,d2表示含有snp的前体mrna序列对接得分;
[0108][0109]
l
m1
表示不含有snp的前体mrna序列中rna结合残基的平均位置,表示含有snp的前体mrna序列中rna结合残基的平均位置;
[0110][0111]
l
med1
表示不含有snp的前体mrna序列中rna结合残基的中值位置,l
med2
表示含有snp的前体mrna序列中rna结合残基的中值位置。
[0112]
在一个实施例中,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:基于snp的近视风险排序得到x个候选的snp,其中x为自然数整数,将候选的snp映射到基因组的基因功能元件中,所述基因功能元件包括外显子、内含子、5’utr和3’utr。
[0113]
在一个具体实施例中,发明人以近视为例进行了深入研究snp诱导的mrna变构风险得分。从基因型和表型数据库(dbgap)收集人类近视相关的snp,nhgri-ebi已发表的全基因组关联研究(gwas)和人工文献挖掘。最终集合中总共包含1145个snp。ensembl变体效应
预测器(vep)用于注释基因组中的snp。从单核苷酸多态性数据库(dbsnp)检索等位基因信息,为了质量控制,snps严格筛选,步骤如下:去除无参考的snps,去除多位点变异的snps,去除位于基因间区域的snp。总共1541对野生型(wt)和突变型(mt)pre-mrnas(前体mrna序列对)涉及806个近视相关的snp,作为来自ensemble(grch38)的fasta序列(见图4)。剪接相关元件由5'剪接位点(5'ss)、3'剪接位点(3'ss)、分支点(bp)、多嘧啶束(py-tract)和剪接调控元件(sre)组成(图5)。其中,5'ss和3'ss的基因组信息来自ensemble(grch38)的基因组注释。bp和py-tract由branchpointer识别,从uniprot的功能筛选确定的sre,通过rbpmap检测得到。我们将rna变构的核苷酸位点映射到每个与剪接相关元件,并计算了变构区域中涉及的mrna前体序列对。在1541对pre-mrna中,121涉及5'ss变构,102涉及3'ss变构,78涉及bp变构,118涉及py-tract变构,979涉及sre变构(图5e)。我们的研究结果表明,剪接相关元件的rna结构受到广泛干扰。rna二级结构由viennarna包中的rnafold预测(版本2.4.18)。通过bprna挖掘rna二级结构基序。这些rna亚结构可以分为两类:(1)配对状态(pair):stem(s);(2)非配对状态(unpair):发夹环(h)、内环(i)、外环(e)、凸环(b)和多分支环(m)。我们计算了每个剪接相关元件的变构概率(ap),包括5'ss、3'ss、分支点位点、py-tract和59种sre结合区域(图6)。对于剪接位点,5'ss-1(5'ss区域上游-1位置核苷酸)、5'ss 1和3'ss-1比周围的核苷酸具有更好的抗变构性能。bp在5个区域中对rna变构效应最敏感,ap最高可达4.2%。对于py-tract,ap在(3%~3.5%)上下波动,没有明显的峰谷。同时,还绘制了wt型转录本前体上这些剪接相关基序的rna结构谱(图6)。不难发现,变构概率与rss之间存在一定的关系(图6)。pearson相关性检验(图6)显示motifs与变构效应高度负相关(r=-0.812;r=pearson相关系数),motifh(r=0.790)和m(r=0.753)高度正相关,基序i(r=0.669)和e(r=0.447)呈中度正相关,基序b呈低正相关。相关分析结果表明,motif s有利于rna结构的稳定,有助于剪接相关区域抵抗snp诱导的变构。ap与结构特征谱之间的显著相关性表明,不同rss状态下的核苷酸可能具有不同的变构概率,因此我们进一步研究了每个剪接相关元件的变构特征谱。为了便于观察变构特征,我们进一步将20种特定变构类型分为3种变构模式。pair》unpair模式包括s》b、s》h、s》i、s》m;unpair》pair模式包括b》s、h》s、i》s、m》s;unpair》unpair模式包含所有剩余的基序变构类型。无论在任何区域变构中,unpair》pair的ap最大,pair》unpair的ap最低,这意味着unpair状态的核苷酸更容易受到snp和转换引发的二次结构冲击。到配对状态。此外,3'ss、bp和py-tract中的最大ap除5ss外均为i》s。具体而言,5'ss的前三个ap分别为b》s(4.6%)、m》s(3.7%)和i》s(3.5%)(图7)。值得注意的是,5
′
ss(5
′
ss 1)下游的 1位点在5
′
ss序列中尤为特殊,b》s的ap比其他5
′
ss位点高出2倍以上。3'ss的前三个ap分别为i》s(4.6%)、b》s(3.5%)、m》s(3%)(图7)。我们研究了ap与变构核苷酸到snp的距离之间的关系。统计结果表明,随着核苷酸与snp距离的增加,rna变构效应逐渐减弱。以350nt为界(图8b),我们将snp的变构效应分为近端变构效应和远端变构效应。通过风险评估,我们发现两种变构效应对rna结构的调节作用不同。与远端变构效应相比,近端变构效应对源自s(s》h除外)和m》s的变构具有更强的变构风险。然而,远端变构起源于h(h》b除外)、b(b》s除外)和s》h的变构风险高于近端变构效应。其次,鉴于分布在内含子或外显子上的sre结合区域在ap上表现出较大差异,我们进行了配对t检验以确定差异是否具有统计学意义。结果表明,某些基序衍生的变构概率在内含子和外显子之间存在显着差异,包括“s》b”、“s》h”、“s》i”、“s》m”、“b》i”。、“b》s”、“h》i”、“h》s”、“i》
b”、“i》h”、“i》s”、“m》h”、“m》s”。我们将两种结构异质性评分(rnasnp、rnasmc)和五种剪接相关元件相结合,以获得剪接相关基序的结构异质性风险高的snp。为了进一步探索snps对剪接相关rbps与pre-mrna对接能力的影响,我们通过hdockserver模拟了pre-mrna与top10风险得分snps(见图9)和剪接相关蛋白的对接过程。hdock评估结果发现,top10风险得分的snp广泛干扰前mrna和剪接相关蛋白之间的对接分数和对接位点。为了形象地呈现剪接相关元件中的结构干扰与剪接效率之间的二元相互作用,我们在近视相关基因lim2的第一个外显子-内含子-外显子区域的5'ss中使用rna结构干扰进行了功能验证。以enst00000596399.2)为例。据报道lim2基因与轴向延长和白内障有关。为此设计了1个对照组和3个实验组。为避免剪接过程中u1snrna和5'ss之间的碱基配对无序,每个实验组都保留了5'ss序列内容。为了形成整个5's完全碱基配对的牢固发夹结构,在5's序列的上游立即插入一个短序列。5'ss上游的-1和-2个核苷酸被认为足以调节拟南芥中的剪接,并且它已被评估为在lim2的天然序列中处于基序b(未配对状态)。然后,我们在插入的序列中创建了两种与5'ss序列碱基配对的突变,以破坏5'ss上游-1和-2核苷酸的碱基配对状态(图10)。我们通过hek293t细胞中的小基因剪接测定评估了这些设计构建体上的剪接事件(图10)。首先,我们验证了天然序列构建体在hek293t细胞中完全剪接(图10,enst00000596399.2的泳道:(1)当整个5的序列与上游插入的序列完全碱基配对时,剪接被显着抑制(图10,enst00000596399.2的第2道),通过引入突变“aa”或“gc”以强制在5'ss上游的-1和-2位置处于未配对状态,我们发现剪接事件被部分挽救(图10,enst00000596399的泳道3和泳道4;(2)与茎设计组相比,剪接效率分别从8%提高到30.83%或50.08%(图10)。
[0114]
图2是本发明实施例提供的一种基于变构概率对近视/高度近视相关snp风险的排序系统,包括:
[0115]
获取单元,用于获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对,其中n为大于2的自然数;
[0116]
提取单元,用于分别提取所述每个snp对应的前体mrna序列对的m种剪接相关元件,并提取m种剪接相关元件中rss,得到每个snp对应的剪接相关元件的rss,其中m为2-10的自然数;
[0117]
计算单元,用于计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;
[0118]
排序单元,用于输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率排序得到的。
[0119]
图3是本发明实施例提供的一种基于变构概率对近视/高度近视相关snp风险的排序设备示意图,所述设备包括:存储器和处理器;
[0120]
所述存储器用于存储程序指令;
[0121]
所述处理器用于调用程序指令,当程序指令被执行时,用于执行上述的一种基于变构概率对近视/高度近视相关snp风险的排序方法。
[0122]
本发明实施例提供的一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现上述的一种基于变构概率对近视/高度近视相关snp风险的
排序方法。
[0123]
本验证实施例的验证结果表明,为适应症分配固有权重相对于默认设置来说可以适度改善本方法的性能。
[0124]
所属领域的技术人员可以清楚地了解到,为描述的方便和简洁,上述描述的系统,装置和单元的具体工作过程,可以参考前述方法实施例中的对应过程,在此不再赘述。
[0125]
在本技术所提供的几个实施例中,应该理解到,所揭露的系统,装置和方法,可以通过其它的方式实现。例如,以上所描述的装置实施例仅仅是示意性的,例如,所述单元的划分,仅仅为一种逻辑功能划分,实际实现时可以有另外的划分方式,例如多个单元或组件可以结合或者可以集成到另一个系统,或一些特征可以忽略,或不执行。另一点,所显示或讨论的相互之间的耦合或直接耦合或通信连接可以是通过一些接口,装置或单元的间接耦合或通信连接,可以是电性,机械或其它的形式。
[0126]
所述作为分离部件说明的单元可以是或者也可以不是物理上分开的,作为单元显示的部件可以是或者也可以不是物理单元,即可以位于一个地方,或者也可以分布到多个网络单元上。可以根据实际的需要选择其中的部分或者全部单元来实现本实施例方案的目的。
[0127]
另外,在本发明各个实施例中的各功能单元可以集成在一个处理单元中,也可以是各个单元单独物理存在,也可以两个或两个以上单元集成在一个单元中。上述集成的单元既可以采用硬件的形式实现,也可以采用软件功能单元的形式实现。
[0128]
本领域普通技术人员可以理解上述实施例的各种方法中的全部或部分步骤是可以通过程序来指令相关的硬件来完成,该程序可以存储于一计算机可读存储介质中,存储介质可以包括:只读存储器(rom,read only memory)、随机存取存储器(ram,random access memory)、磁盘或光盘等。
[0129]
本领域普通技术人员可以理解实现上述实施例方法中的全部或部分步骤是可以通过程序来指令相关的硬件完成,所述的程序可以存储于一种计算机可读存储介质中,上述提到的存储介质可以是只读存储器,磁盘或光盘等。
[0130]
以上对本发明所提供的一种计算机设备进行了详细介绍,对于本领域的一般技术人员,依据本发明实施例的思想,在具体实施方式及应用范围上均会有改变之处,综上所述,本说明书内容不应理解为对本发明的限制。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。