一种胶质瘤pten基因突变的检测方法
1.技术领域
2.本发明涉及医学影像处理技术领域,更具体地,涉及基因突变的检测方法。
背景技术:
3.胶质瘤是最常见的原发性脑肿瘤。自2016年起《中枢神经系统肿瘤who分类》将包括异柠檬酸脱氢酶-1基因突变、o6-甲基鸟嘌呤dna甲基转移酶启动子甲基化在内的一系列预后、治疗相关标志物纳入胶质瘤的诊断,建立了整合诊断的模式。而2021版《中枢神经系统肿瘤who分类》进一步规定了没有分子病理结果作为依据不能将胶质瘤分级。
4.目前,胶质瘤标志物检测依赖于手术切除或活检。然而这类方法存在有创性的缺陷,导致难以在手术前获取检测结果,无法在术前制定最佳手术方案时发挥作用。磁共振成像等医学影像可无创地获取肿瘤的信息。近年来影像组学的提出为磁共振影像高通量定量分析提供了技术支撑。影像组学在脑胶质瘤标志物和个体预后评估等方面有了一定的应用。然而由于影像组学只能提取有限的特征,因而能自动学习相关特征的深度学习也被引入医学影像的研究。
5.但由于深度学习需要大量样本学习充足的特征的特征,因而影像组学特征可作为深度学习特征的补充。目前研究仍采用基于自然图像预训练的二维深度学习模型及传统的算法整合。
6.此外,目前影像组学及深度学习均是基于人工绘制肿瘤区域或基于人工绘制训练的自动绘制肿瘤区域的深度学习模型。存在主观性且耗时长。
7.因此,希望提供一种基于目标训练的三维深度学习特征提取器及目标热点区域影像组学特征整合方法,其能够获得准确且高效的脑胶质瘤标志物预测结果。
技术实现要素:
8.本发明的目的在于克服上述现有技术的特点,提供一种胶质瘤pten基因突变的检测方法。本发明通过自多模态脑磁共振影像中提取深度学习及影像组学特征,并使用深度学习模型进行最终预测,来提高预测的准确性。
9.本发明通过以下技术方案实现的:一种胶质瘤pten基因突变的检测方法,包括以下步骤:获取胶质瘤患者t1加权,造影剂强化t1加权,t2加权,液体衰减反转恢复序列多模态磁共振图像;将获取的多模态图像进行标准化;将获取标准化的多模态图像训练成深度残差网络模型以提取深度学习特征并获取类激活图;将获取的类激活图使用最大类间方差法进行二值化以获得感兴趣区域;
将获取的标准化的感兴趣区域多模态图像提取出降维的影像组学特征;整合提取的深度学习特征及提取的降维的影像组学特征进行训练从而整合模型用于预测pten突变状态。
10.进一步地,对多模态磁共振图像获取的图像颅骨剥离,n4偏置场校正,直方图匹配,重采样,刚性配准,变形的标准化处理。
11.进一步地,深度残差网络是三维resnet-18。所述深度学习特征是指全肿瘤区域经过深度残差网络池化层输出的特征。所述类激活图是指最后一层特征图经过双线性插值上采样与原图大小一致的图。
12.进一步地,影像组学特征包括实性肿瘤区域及全肿瘤区域的几何特征,强度特征,纹理特征,及经过三层小波分解的八个子带的几何特征,强度特征,纹理特征。
13.进一步地,整合网络为以降维的影像组学特征与深度学习特征为输入的四层全连接网络。
14.更进一步的,纹理特征包括灰度共生矩阵,灰度行程矩阵,灰度区域大小矩阵,领域灰度差矩阵提取的特征。
15.更进一步的,降维的影像组学特征为经过影像组学特征降维模型输出的特征。
16.更进一步的,步骤影像组学特征降维模型为四层全连接网络。
附图说明
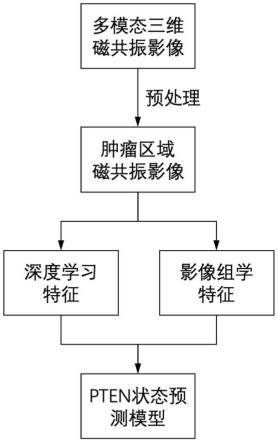
17.图1为本发明所述一种胶质瘤pten基因突变的检测方法的一具体实施例的流程示意图;图2为图1所述具体实施例中pten状态预测模型结构示意图;图3为图2所述深度残差网络模型结构及类激活图示意图;图4为图2所述感兴趣区域多模态图像及影像组学特征降维模型结构示意图;图5为图2所述整合模型结构示意图;附图标记说明:1-深度残差网络模型;2-影像组学特征降维模型;3-整合模型;4-类激活图;5-感兴趣区域;11-标准化的多模态图像;12-池化层;21-感兴趣区域多模态图像;22-最大信息系数前30%的影像组学特征;23-全连接层;31-降维的影像组学特征;32-深度学习特征。
具体实施方式
18.下面结合实施例对本发明作进一步详细的说明。
19.如图1和图2所示,本实施例提供 一种胶质瘤pten基因突变的检测方法,包括以下步骤:(1)获取胶质瘤患者t1加权,造影剂强化t1加权,t2加权,液体衰减反转恢复序列磁共振图像。
20.(2)对步骤(1)获取的多模态图像进行标准化。
21.(3)根据步骤(2)获取的标准化的多模态图像11训练深度残差网络模型以提取深度学习特征并获取类激活图4。
22.(4)根据步骤(3)获取的类激活图使用最大类间方差法进行二值化以获得感兴趣
区域5。
23.(5)根据步骤(4)获取的标准化的感兴趣区域多模态图像21提取降维的影像组学特征31。
24.(6)根据步骤(3)提取的深度学习特征及步骤(5)提取降维的影像组学特征训练整合模型3用于预测pten突变状态。
25.在本实施例中,所述图像标准化的具体流程如下:采用advanced normalization tools开源软件包中的antsbrainextraction.sh脚本进行颅骨剥离,得到脑图像。采用simpleitk开源软件包中n4biasfieldcorrectionimagefilter函数进行n4偏置场强校正。然后,随机选取一位患者的磁共振图像作为模板,采用simpleitk开源软件包中histogrammatchingimagefilter函数对其他图像与模板进行直方图匹配。采用advanced normalization tools开源软件包中的resample_image函数对图像重采样至体素大小1mm
×
1mm
×
1mm。采用advanced normalization tools开源软件包中的registration函数将非t1序列图像与t1序列图像进行刚性配准。采用pil软件包最近邻差值法将图像调整大小至240
×
240
×
155(长
×
宽
×
高)。
26.如图1、图2和图3所示,在本实施例中,所述深度学习特征的提取具体流程如下:首先,采用pil软件包最近邻差值法将全肿瘤区域图像调整大小至240
×
240
×
32(长
×
宽
×
高)。并将t1加权,造影剂强化t1加权,t2加权,液体衰减反转恢复序列图像进行合并为240
×
240
×
32
×
4(长
×
宽
×
高
×
通道)多模态图像。其次,采用numpy软件包对pten基因突变的多模态图像进行2次-15度至15度的水平旋转以平衡样本量。最后,采用深度残差网络模型1三维resnet-18为基础网络,采用标准化的多模态图像11为输入,采用全外显子测序所获得的pten基因突变状态为标注进行训练,并以池化层12的输出作为深度学习特征。
27.在本实施中,所述感兴趣区域多模态图像提取具体流程如下:首先,使用pytorch软件包的grid_sample函数将深度残差网络模型最后一层特征图以双线性插值法上采样至240
×
240
×
32(长
×
宽
×
高)获得类激活图4。其次,使用最大类间方差法确定二值化分割阈值,以大于阈值区域为感兴趣区域。
28.作为一可选实施例,深度残差网络模型1的权重采用glort算法进行初始化,损失函数采用二值交叉熵函数,优化器采用均方根梯度下降算法,学习率采用0.001,正则权重采用0.001,批次大小采用16。
29.在本实施例中,所述影像组学特征的提取采用pyradiomics软件包提取实性肿瘤区域及全肿瘤区域的几何特征,强度特征,纹理特征,及经过三层小波分解的八个子带的几何特征,强度特征,纹理特征。纹理特征包括灰度共生矩阵,灰度行程矩阵,灰度区域大小矩阵,领域灰度差矩阵提取的特征。
30.如图4所示,作为一可选实施例,所述影像组学特征的降维具体流程如下:构建影像组学特征降维模型2进行特征降维。
31.作为一可选实施例,所述影像组学特征降维模型2采用四层全连接神经网络作为基础,前三层采用线性整流函数作为激活函数,第四层采用sigmoid函数作为激活函数。在训练过程中第一层随机失活20%神经元,第二层随机失活50%神经元。以第三层输出作为降维的影像组学特征31。
32.如图5所示,在本实施例中,所述的整合模型3以四层全连接神经网络为基础网络。
采用降维的影像组学特征31与深度学习特征32的拼接为输入,采用全外显子测序所获得的pten基因突变状态为标注进行训练。
33.作为以可选实施例,整合模型3的损失函数采用二值交叉熵函数,优化器采用均方根梯度下降算法,学习率采用0. 1,正则权重采用0.005,批次大小采用16。
34.以上所述的仅是本发明的一些实施方式。对于本领域的普通技术人员来说,在不脱离本发明创造构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。
