1.本发明涉及信息抽取技术领域,特别涉及一种基于多任务学习的生物医学实体抽取方法。
背景技术:
2.当前生物医学领域的相关研究资料在不断增加,pubmed已有3400多万的医学文献引用。这些医学文献包含着海量的知识,记录着最新的医学研究进展,但是医学文献以文本的形式存储知识,研究人员在查阅资料时会花费较多的时间。当前开始逐渐使用医学知识图谱来组织和管理知识,研究人员可以方便地检索内容,也可以基于医学知识图谱开发药物发现、知识问答及数据分析平台等应用。但是当前构建医学知识图谱由医学相关的专家手动地从文本资料中抽取知识,可以保证数据内容的准确性,但是会花费较高的人工成本和时间成本,并且需要后期维护和更新数据内容。因此自动地从文本资料中抽取结构化知识构建医学知识图谱是当前的重要研究方向。
3.实体抽取(entity extraction,ee)的目标是从医学文本中正确地抽取出医学实体。早期方法主要是基于词典和规则,根据具体任务要求来人工地构建,此类方法泛化性比较差。近年来深度学习方法被广泛应用,kocaman等人基于双向长短期记忆网络-卷积神经网络(bilstm-cnn)构建混合模型用于医学实体抽取任务。该方法使用预训练的词嵌入来获取词表征,但是词表征不包含上下文信息。xu等人使用预训练语言模型bert从文本中提取上下文语义特征,然后基于bert和bilstm-crf构建混合模型完成生物医学实体抽取任务。上述基于深度学习的实体抽取方法采用单任务学习方式训练模型,并没有关注各个任务之间的关联性。
4.在生物医学领域中,实体名称普遍存在同义词的情况,例如cancer的同义词有tumor、neoplasm、malignancy等等。实体标准化目标是将表示同一实体的不同实体名称建立起映射关系,此任务也被称为实体链接、实体消岐等。在构建医学知识图谱时一般会从多个知识来源中获取知识,可能会使用不同的名称来表示同一个实体。因此实体标准化是一个重要的任务,这可以有效地减少医学知识图谱的冗余性和歧义性。早期主要是基于规则和词典的方法,根据词形变换规则、词典映射完成任务,此类方法的精确率较高但是召回率较低。当前逐渐使用深度学习方法,使用预训练语言模型得到实体名称的实体表征,然后通过计算实体表征之间的相似度来完成任务。sung等人提出了biosyn模型,使用biobert对实体名称进行编码得到表征,称为密集表征。biosyn除了使用密集表征,额外引入了实体的字形特征,使用词频-逆向文件频率(term frequency
–
inverse document frequency,tf-idf)计算得到稀疏表征。将密集表征与稀疏表征进行加权求和得到实体表征。liu等人构建了专用于实体标准化的预训练语言模型sapbert,使得实体表征额外包含同义词信息。上述基于深度学习的实体标准化模型同样采用单任务学习方式训练模型,忽略子任务之间的关联性。因此,存在准确率低的问题。
技术实现要素:
5.本发明要解决的技术问题是提供一种准确率高的基于多任务学习的生物医学实体抽取方法。
6.为了解决上述问题,本发明提供了一种基于多任务学习的生物医学实体抽取方法,所述基于多任务学习的生物医学实体抽取方法包括以下步骤:
7.s1、获取一段医学文本;
8.s2、将医学文本输入多任务实体抽取模型,利用多任务实体抽取模型从医学文本中抽取实体;
9.其中,所述多任务实体抽取模型包括第一预训练语言模型和多个交互式指针网络解码层,每个交互式指针网络解码层对应一个子任务,每个子任务对应目标实体类别的实体抽取,所述交互式指针网络解码层包括中间层、起始层、线性交互层和结束层;医学文本输入所述第一预训练语言模型得到文本表征,所述中间层对文本表征进行特征提取和降维后输入所述起始层,所述起始层得到实体开始表征,所述实体开始表征输入所述线性交互层得到交互表征,交互表征与文本表征进行求和并输入所述结束层得到实体结束表征,将实体开始表征和实体结束表征进行解码得到实体的开始边界和结束边界,完成从文本中抽取实体。
10.作为本发明的进一步改进,所述多任务实体抽取模型包括八个交互式指针网络解码层,分别对应基因/蛋白质、化合物/药物、疾病、dna、rna、细胞类型、细胞系、物种共八种实体类别的实体抽取。
11.作为本发明的进一步改进,所述第一预训练语言模型采用biobert。
12.作为本发明的进一步改进,还包括以下步骤:
13.s3、为不同的实体类别分别构建对应的医学词典,每个实体由id标识;
14.s4、构建多任务实体标准化模型,将抽取的实体输入所述多任务实体标准化模型,并输出得到实体密集表征;
15.s5、计算出实体稀疏表征,将实体密集表征与实体稀疏表征进行加权求和得到实体表征,分别计算出抽取的实体和医学词典中所有实体的实体表征,通过内积计算它们之间的相似度,从而得到抽取的实体在医学词典中的id。
16.作为本发明的进一步改进,所述多任务实体标准化模型包括第二预训练语言模型和多个bert层,每个bert层对应一个子任务,每个子任务对应目标类别的实体标准化;将抽取的实体输入所述第二预训练语言模型得到实体上下文表征,并根据实体类别将实体上下文表征输入到对应的bert层,bert层输出得到实体密集表征。
17.作为本发明的进一步改进,所述第二预训练语言模型采用sapbert。
18.作为本发明的进一步改进,步骤s5中,利用tf-idf算法计算出实体稀疏表征。
19.本发明还提供了一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现上述任意一项所述方法的步骤。
20.本发明还提供了一种计算机可读存储介质,其上存储有计算机程序,其特征在于,该程序被处理器执行时实现上述任意一项所述方法的步骤。
21.本发明的有益效果:
22.本发明基于多任务学习的生物医学实体抽取方法通过多任务实体抽取模型进行实体抽取,将第一预训练语言模型作为共享的编码层,来学习各个子任务的共同语义特征,并将学习的语义特征共享给各个子任务,以加强模型之间的联系并减少对训练数据量的依赖,并通过各个交互式指针网络解码层学习各个子任务特有的特征信息,多任务实体抽取模型可以并行地从文本中抽取多种类别的实体,从而可以更快地、准确地完成任务。
23.上述说明仅是本发明技术方案的概述,为了能够更清楚了解本发明的技术手段,而可依照说明书的内容予以实施,并且为了让本发明的上述和其他目的、特征和优点能够更明显易懂,以下特举较佳实施例,并配合附图,详细说明如下。
附图说明
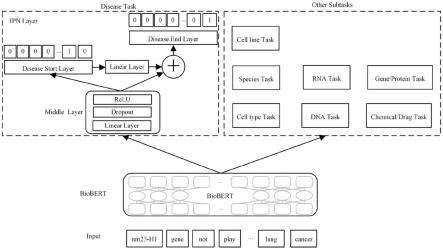
24.图1是本发明优选实施例中多任务实体抽取模型的结构示意图;
25.图2是本发明优选实施例中多任务实体标准化模型的结构示意图。
具体实施方式
26.下面结合附图和具体实施例对本发明作进一步说明,以使本领域的技术人员可以更好地理解本发明并能予以实施,但所举实施例不作为对本发明的限定。
27.实施例一
28.本实施例公开了一种基于多任务学习的生物医学实体抽取方法,包括以下步骤:
29.s1、获取一段医学文本;
30.s2、将医学文本输入多任务实体抽取模型,利用多任务实体抽取模型从医学文本中抽取实体;
31.通常的,实体抽取模型由编码层和解码层组成。编码层从文本中提取语义特征,解码层根据语义特征来抽取出存在的实体。实体抽取方法常用的解码层有mlp,crf以及pn。mlp使用全局特征信息预测实体标签。crf通过局部特征信息预测实体的标签。例如,在bio标注模式下,如果预测当前单词的标签为b,则crf预测后面相邻单词的标签为i的概率更大。由于crf的解码速度较慢,训练会花费较长的时间。pn由两个独立的模块组成,分别用于预测实体的开始边界和结束边界。特别地,在抽取医学实体时,实体的开始边界和结束边界之间存在着一定的关联,因此本发明基于指针网络提出交互式指针网络(interactive pointer network,ipn),将预测实体开始边界的特征信息用于预测实体结束边界。
32.具体地,本发明中多任务实体抽取模型包括第一预训练语言模型和多个交互式指针网络(interactive pointer network,ipn)解码层,每个交互式指针网络解码层对应一个子任务,每个子任务对应目标实体类别的实体抽取,所述交互式指针网络解码层包括中间层(middle layer)、起始层(start layer)、线性交互层(interactive linear layer)和结束层(end layer),参照图1。具体地,中间层包括线性层、dropout层及relu激活函数。
33.医学文本输入所述第一预训练语言模型得到文本表征,所述中间层对文本表征进行特征提取和降维后输入所述起始层,所述起始层得到实体开始表征,所述实体开始表征输入所述线性交互层得到交互表征,交互表征与文本表征进行求和并输入所述结束层得到实体结束表征,将实体开始表征和实体结束表征进行解码得到实体的开始边界和结束边界,完成从文本中抽取实体。
34.在本实施例中,所述多任务实体抽取模型包括八个交互式指针网络解码层,分别对应基因/蛋白质(gene/protein)、化合物/药物(chemical/drug)、疾病(disease)、dna、rna、细胞类型(cell type)、细胞系(cell line)、物种(species)共八种实体类别的实体抽取。
35.当前预训练语言模型已经得到广泛的关注,自bert(bidirectional encoder representation from transformers)模型的提出,预训练语言模型在各个领域得到应用。bert基于transformer模型架构,通过深层网络和自注意力机制,使得模型可以从文本中提取深层次的语义特征。bert通过自监督任务完成模型参数的更新和优化。相比于预训练的词嵌入,预训练语言模型得到的文本表征包含上下文信息,可以提高模型的性能。biobert与bert模型架构相同,使用来自pubmed和pmc的医学文献作为语料集。相比于bert,在生物医学领域的多个任务中取得更优实验结果。本发明中在实体抽取模型中使用biobert作为预训练语言模型。用于学习各个子任务的共同语义特征,并将这些特征共享给每个子任务,这可以加强模型之间的联系并减少对训练数据量的依赖。在实体抽取任务中,识别目标类别的实体是一个子任务,每种实体类别分别对应一个ipn解码层,将编码层输出的共享特征输入到每个解码层,使得交互式指针网络解码层学习到每个子任务特有的特征信息。
36.在一些实施例中,本发明一种基于多任务学习的生物医学实体抽取方法还包括以下步骤:
37.s3、为不同的实体类别分别构建对应的医学词典,每个实体由id标识;
38.可选地,本发明中从5个数据库收集相关内容,共构建了6个医学词典,词典内容统计如表1所示。例如对于疾病实体和化合物实体,从医学主题词表(medical subject headings,mesh)中收集医学实体的同义词,然后构建得到词典。
[0039][0040]
表1生物医学词典内容统计
[0041]
s4、构建多任务实体标准化模型,将抽取的实体输入所述多任务实体标准化模型,
并输出得到实体密集表征;
[0042]
所述多任务实体标准化模型包括第二预训练语言模型和多个bert层,每个bert层对应一个子任务,每个子任务对应目标类别的实体标准化;将抽取的实体输入所述第二预训练语言模型得到实体上下文表征,并根据实体类别将实体上下文表征输入到对应的bert层,bert层输出得到实体密集表征。参照图2。其中,模型利用实体的语义特征和字形特征完成实体之间相似度的计算,相比于利用单一特征,可以取得更好的实验结果。
[0043]
通常地,biobert、bert等模型适用于自然语言处理领域的绝大多数任务,训练模型所使用的语料集为文本。此外,通常也会为特定任务构建预训练语言模型,liu等人基于度量学习构建了专用于实体标准化的预训练模型sapbert。sapbert的语料集来自统一医学语言系统(unified medical language system,umls),将umls id与实体名称组合为(name,umls id),然后基于度量学习完成模型的自监督训练。本发明在实体标准化模型中使用sapbert作为预训练语言模型。sapbert作为共享模型,每个bertlayer对应特定子任务,共计5个子任务。
[0044]
s5、计算出实体稀疏表征,将实体密集表征与实体稀疏表征进行加权求和得到实体表征,分别计算出抽取的实体和医学词典中所有实体的实体表征,通过内积计算它们之间的相似度,从而得到抽取的实体在医学词典中的id。可选地,利用tf-idf算法计算出实体稀疏表征。
[0045]
为了验证本发明的有效性,在生物医学领域7个公共数据集及5个私有数据集上训练和评估模型,公开数据集的统计如表2所示。
[0046]
由于生物医学领域实体标准化数据集较少,在已得到医学词典的基础上构建私有实体标准化数据集。首先从词典中随机选择实体名称,然后按照一定的规则进行转变,主要是大小写的转变、单词的随机替换或删除。这些规则可以让模型使用这些数据集训练之后更具有鲁棒性和泛化性。最终共得到6个私有数据集,训练集共6000个实体,测试集共4000个实体。
[0047]
[0048][0049]
表2公开数据集统计
[0050]
深度学习框架pytorch1.7.0搭建实验模型。预训练语言模型使用biobert-base v1.1与sapbert,隐藏层维度设为768,输入序列长度为512。训练集的批次大小为32,学习率为1e-5,优化器为adam,训练轮数为15。middle layer和interactive layer的维数设为128,dropout率设为0.2。
[0051]
实体抽取任务使用f1值作为评测指标。实体标准化任务使用hits@k来评估模型性能,hits@k表示在预测的k个结果中存在正确结果的概率,k为1时等价于准确率。
[0052]
解码层ipn、pn、crf及mlp的实验比较如表3所示,从中可以看出ipn的实验结果优于其他三种解码层,在8个数据集上的平均f1值分别比pn、crf、mlp高0.3%、0.57%、1.04%。这证明了ipn解码层的有效性,通过将实体开始边界的特征信息用于预测实体的结束边界,促进了特征之间的交互,从而提高了模型的实验效果。
[0053][0054]
表3不同解码层的性能对比
[0055]
为了验证多任务实体抽取模型(multi-task entity extraction model,mt-eem)的有效性,使用每个数据集训练对应的单任务实体抽取模型(single-task entity extraction model,st-eem)。st-eem与mt-eem的实验比较结果如表4所示,从表中看出mt-eem的实验结果明显优于st-eem。mt-eem在五个数据集上的性能表现更优,平均f1值比st-eem高0.59%。这证明了多任务学习方式在实体抽取任务中的有效性。
[0056][0057]
表4 st-eem与mt-eem的性能比较
[0058]
bern2是常用的生物医学实体抽取工具,其共有两个版本,这里对比的版本是使用biobert作为预训练语言模型。mtm-cw是基于bilstm-crf的多任务实体抽取模型,由于数据集的不统一,这里仅作参考作用。mt-eem与bern2及mtm-cw的实验对比结果如表5所示,从中可以看出mt-eem的实验结果优于bern2,平均f1值比bern2高0.7%。bern2同样采用多任务学习方式,但是解码层使用mlp,因此这也证明ipn解码层的有效性。此外,mtm-cw使用预训练的词嵌入获取词表征,这证明biobert获取的词表征可以使得模型取得更好的性能。
[0059][0060]
表5与其他实体抽取模型的性能比较
[0061]
多任务实体标准化模型(multi-task entity normalization model,mt-enm)首先在3个公开的数据集上完成训练与评估。单任务实体标准化模型(single-task entity normalization model,st-enm)的实验结果来自sapbert论文,sapbert在多个数据集上取得了目前最高的性能表现。mt-enm和st-enm在公共数据集的实验比较结果如表6所示,从中看出mt-enm的实验结果优于st-enm,在两个任务上hits@1值高于st-enm。但是在ncbi-disease数据集上mt-enm的性能表现比较低,原因可能是子任务的数量较少,并没有充分地利用子任务之间的特征信息。
[0062][0063]
表6st-enm与mt-enm在公开数据集的性能比较
[0064]
多任务实体抽取模型可识别8种实体类别,因此多任务实体标准化模型在已构建的5个私有数据集进行训练与评估。mt-enm与st-enm的实验比较如表7所示,mt-enm在三个任务上取得更佳的性能表现,平均hits@1值为94.52%,比st-enm高0.47%。mt-enm共有5个子任务,使得共享模型学习到更多促进子任务的特征信息。这证明当子任务数量越多的时候,多任务实体标准化模型可以取得更佳的实验结果。
[0065]
由于species实体对应的ncbi-taxonomy词典数据内容过多,暂时无法完成模型的训练与评估,因此mt-enm只在5个私有数据集上完成训练和评估。
[0066]
[0067][0068]
表7 st-enm与mt-enm在私有数据集的性能比较
[0069]
本发明提出了基于多任务学习的生物医学实体抽取模型和实体标准化模型。相比于单任务学习方式,多任务学习方式可以提高模型的实验结果以及减少模型的训练时间。特别地,当子任务数量较多时,多任务模型的参数量会远少于单任务模型从而可以快速地准确地从医学文献中抽取结构化知识,完成自动构建医学知识图谱。在实体抽取任务中提出的交互式指针网络相比于其他的解码层取得了更优的实验结果。
[0070]
实施例二
[0071]
本实施例公开了一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现上述实施例一中所述基于多任务学习的生物医学实体抽取方法的步骤。
[0072]
实施例三
[0073]
本实施例公开了一种计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行时实现上述实施例一中所述基于多任务学习的生物医学实体抽取方法的步骤。
[0074]
以上实施例仅是为充分说明本发明而所举的较佳的实施例,本发明的保护范围不限于此。本技术领域的技术人员在本发明基础上所作的等同替代或变换,均在本发明的保护范围之内。本发明的保护范围以权利要求书为准。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。