1.本发明涉及生物信息学领域,涉及融合蛋白质的多个生物特征,被用于识别关键蛋白质,具体是一种基于弱一致性模型识别关键蛋白质的方法。
背景技术:
2.自然界中有成千上万种蛋白质,每一种蛋白质的功能都不一样,其中有些蛋白质至关重要,是细胞生存或者繁衍的必不可少的物质基础,杀死它们会导致细胞死亡或者停止繁衍,这些蛋白质被称为关键蛋白质或致死蛋白质。由于关键蛋白质的重要作用,研究关键蛋白质已经成为一个热点。研究表明,关键蛋白质也与致病基因有关,识别关键蛋白质也有助于疾病研究。并且,关键蛋白质的识别也有助于新疗法和抗生素的开发和推动合成生物学的发展。
3.目前识别关键蛋白质的方法主要分为生物实验方法和计算方法。基因敲除、rna干扰、有条件基因敲除及基因编辑技术等生物实验方法虽然已识别了多个物种的关键蛋白质的金标准集,但是它们不仅周期长、效率低、费用高,而且在人类或者其它灵长类动物身上实验还受到严格的道德伦理的约束。因此,急需开发新的识别关键蛋白质的方法。
4.随着高通量技术的出现和蛋白质相互作用(protein-protein interaction,简称ppi)网络数据的积累,用计算方法来识别关键关键蛋白质已经成为趋势。近年来,一些基于ppi网络拓扑特征的方法被提出来了,如度中心性(degree centrality,简称dc),介数中心性(betweenness centrality,简称bc),接近性中心性(closeness centrality,简称cc),子图中心性(subgraph centrality,简称sc),特征向量中心(eigenvector centrality,简称ec),信息中心性(information centrality,简称ic)和邻居中心性(neighbor centrality,简称nc)等。
5.这类中心性方法的预测准确度依赖于ppi网络数据的可靠。然而,ppi数据中包含了很多假阳性和假阴性,这都影响了关键蛋白质识别的准确度。于是,研究人员逐渐开发出融合多个蛋白质特征的方法。例如,li等人将ppi网络的拓扑特征和基因表达数据结合在一起,从而提出了pec方法。peng等人使用直系同源信息来衡量蛋白质的保守特征,用边聚集系数(ecc)来反映ppi网络的拓扑特征,然后使用随机游走模型将它们结合起来设计出了ion方法。li等人根据ppi网络的拓扑特征提出了邻居接近中心性,然后用expoc模型将直系同源数据和ncc结合起来设计出ncco方法;li等人通过使用改进的的poc模型结合ecc系数、直系同源数据和皮尔逊相关系数(pcc)设计出e_poc方法。zhong等人提出了一种新的名为jdc的度量方法,该方法提供了一种动态阈值方法来将基因表达数据进行二值化,并结合jaccard相似性指数和ecc来预测必需蛋白质。
6.上述方法都是基于多特征融合来识别关键蛋白质的方法,在识别准确度上都有一定的提高,其中,ncco和e_poc两个方法都发现两类特征之间存在一致性关系。在通过大量观察和统计分析之后,本发明发现,两类特征之间更倾向于满足弱一致性关系,并在此基础上提出了弱一致性模型来识别关键蛋白质。
技术实现要素:
7.本发明针对现有代表性方法的结合模型中存在的不足,提出了一种弱一致性模型来识别关键蛋白质。这种方法的识别效率和实用价值高,为关键蛋白质的识别提供了新思路。
8.实现本发明目的的技术方案是:
9.基于弱一致性模型识别关键蛋白质的方法,包括如下步骤:
10.1)设置弱一致性的条件:设蛋白质u和蛋白质v满足弱一致性,即u<<v,表示u弱压制v,弱一致性的条件如下:
11.a(u)》a(v)-ξand b(u)》b(v)-ξ,
12.其中u,v代表蛋白质;a和b表示两种特征得分;ξ是容错因子,为正数;
13.使用图g(v,e)表示ppi网络,v是蛋白质节点的集合,e是边的集合,在确定了蛋白质间的弱压制关系后,结合模型被定义为:
[0014][0015]
其中dm
u,v
表示蛋白质u和蛋白质v是否满足弱压制关系,其进一步定义如下:
[0016][0017]
其中α是介于[0,1]之间的可调节参数;
[0018]
2)弱一致性模型评估:
[0019]
为了检验弱一致性模型识别关键蛋白质的能力,用弱一致性模型替换了如下几个代表性方法中的结合模型,来观察是否比原方法有更高的关键蛋白质识别率;2-1)pec方法使用边聚集系数用来衡量两个蛋白质连接的紧密程度,使用基于基因表达谱的皮尔逊相关系数来衡量蛋白质间连接的可靠程度,然后使用乘法将边聚集系数和皮尔逊相关系数结合起来,pec的计算公式为:
[0020][0021]
其中ecc(u,v)分别表示蛋白质u和蛋白质v之间的边聚集系数;nv代表蛋白质v的邻域;pcc(u,v)表示蛋白质u和蛋白质v之间的皮尔逊相关系数,ecc(u,v)和pcc(u,v)的公式分别如下:
[0022][0023][0024]
其中nu和nv分别是蛋白质u和蛋白质v的邻域;du和dv分别表示蛋白质u和蛋白质v的度;s表示基因表达数据的样本数;g(u,i)和g(v,i)分别代表蛋白质u和蛋白质v在样本i中的基因表达水平;g(u)和g(v)分别是蛋白质u和蛋白质v的基因表达水平的均值;σ(u)和σ(v)分别表示蛋白质u和蛋白质v的基因表达水平的标准差,使用公式(1)的弱一致性模型替
换公式(3),然后将替换结合模型后的方法命名为fpec,fpec满足条件为:
[0025]
ecc(u)》ecc(v)-ξand pcc(u)》pcc(v)-ξ,
[0026]
其中ecc(u)和ecc(v)分别表示蛋白质u、蛋白质v的边聚集系数;pcc(u)和pcc(v)分别代表蛋白质u和蛋白质v的皮尔逊相关系数;ξ是容错因子,为正数,采用结合模型给出最终得分,计算公式如下:
[0027][0028]
其中α是介于[0,1]之间的可调节参数,nu代表蛋白质u的邻域;
[0029]
2-2)ion方法使用直系同源信息来衡量蛋白质的保守特征,用边聚集系数来反映ppi网络的拓扑特征,然后使用随机游走模型将边聚集系数和直系同源信息结合起来,ion的计算公式为:
[0030]
pr
t 1
=(1-α)d α*h*pr
t
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(7),其中pr表示排序得分,t是迭代的次数,d代表直系同源矩阵,h为归一化的ecc矩阵,d的公式如下所示:
[0031][0032]
其中o(u)为蛋白质u在参考生物体中存在直系同源蛋白质的参考物种数目,h的公式如下所示:
[0033][0034]
其中nu为蛋白质u的邻域,ecc(u,v)的公式如下:
[0035][0036]
其中,z
u,v
是包含边(u,v)的三角形的数量,ku和kv分别是蛋白质u和蛋白质v的度;min()是取最小值函数,使用公式(1)的弱一致性模型替换公式(7),然后将替换结合模型后的方法命名为fion,fion满足的条件为:
[0037]
d(u)》d(v)-ξandhr(u)》hr(v)-ξ,
[0038]
其中d(u)表示蛋白质u的直系同源得分;hr表示h*pr
t
;ξ是容错因子,为正数,采用结合模型给出最终得分,计算公式如下:
[0039][0040]
其中α是介于[0,1]之间的可调节参数;nu代表蛋白质u的邻域;
[0041]
2-3)ncco方法根据ppi网络的拓扑特征提出了邻居接近中心性即ncc,然后用expoc模型将直系同源数据和ncc结合起来,ncco的计算公式为:
[0042]
fs(u)=∑
u<v
(ncc(u)-ncc(v)) (os(u)-os(v))
ꢀꢀꢀ
(12),其中uξv表示u压制v;ncc(u)表示蛋白质u的邻居接近中心性得分;os表示直系同源得分,os的公式与公式(8)一致,ncc的计算公式为:
[0043]
cc(u)=(n-1)/∑vp(u,v)
[0044][0045]
其中n为ppi网络中蛋白质的总数,p(u,v)表示蛋白质u、v之间的最短路径;du和dv表示蛋白质u和蛋白质v的度;min()为取最小值函数;nu表示蛋白质u的邻域;t
u,v
是包含蛋白质u和蛋白质v的所有三角形的点的集合,使用公式(1)的弱一致性模型替换公式(12),然后将替换结合模型后的方法命名为fncco,fncco满足的条件为:
[0046]
ncc(u)》ncc(v)-ξand os(u)》os(v)-ξ,
[0047]
其中ξ是容错因子,为正数,采用结合模型给出最终得分,计算公式如下:
[0048][0049]
其中α是介于[0,1]之间的可调节参数;nu代表蛋白质u的邻域;
[0050]
2-4)e_poc方法采用边聚集系数来衡量蛋白质聚集在一起的程度;使用皮尔逊相关系数表示两个蛋白质的基因表达水平,来衡量边的可靠性;采用直系同源信息来衡量蛋白质本身的重要性;最后使用拓展的poc模型将皮尔逊相关系数、边聚集系数、直系同源信息融合在一起,e_poc的计算公式为:
[0051][0052][0053]
其中ps代表直系同源得分,ps的公式与公式(8)相同;α是介于[0,1]之间的可调参数;pn代表边聚集系数和皮尔逊相关系数的相加值,其公式如下:
[0054]
pn(u)=ecc(u) pcc(u)
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(16),
[0055]
其中ecc表示边聚集系数,ecc的公式与公式(10)相同,pcc表示皮尔逊相关系数,pcc的公式和公式(5)一致,使用公式(1)的弱一致性模型替换公式(15),然后将替换结合模型后的方法命名为fe_poc,fe_poc满足条件为:
[0056]
pn(u)》pn(v)-ξand ps(u)》ps(v)-ξ,
[0057]
其中ξ是容错因子,为正数,采用结合模型给出最终得分,计算公式如下:
[0058][0059]
其中α是介于[0,1]之间的可调节参数;nu代表蛋白质u的邻域;
[0060]
2-5)jdc方法采用一种动态阈值方法来将基因表达数据进行二值化,并使用乘法将jaccard相似性指数和边聚集系数结合起来预测关键蛋白,jdc的计算公式为:
[0061][0062]
其中nu代表蛋白质u的邻域;jaccard(u,v)代表边(u,v)的jaccard相似度;ecc(u,v)代表边(u,v)的边聚集系数,ecc与公式(10)一样,jaccard相似度的计算公式如下:
[0063][0064]
其中,su和sv表示蛋白质u和蛋白质v的基因表达数据的布尔值,jaccard相似度系数在0到1之间,使用公式(1)的弱一致性模型替换公式(18),然后将替换结合模型后的方法命名为fjdc,fjdc满足条件为:
[0065]
jaccard(u)》jaccard(v)-ξand ecc(u)》ecc(v)-ξ,
[0066]
其中jaccard(u)和jaccard(v)分别表示蛋白质u和蛋白质v的jaccard相似度系数;ecc(u)和ecc(v)分别表示蛋白质u和蛋白质v的边聚集系数,ξ是容错因子,为较小的正数,采用结合模型给出最终得分,计算公式如下:
[0067][0068]
其中α是介于[0,1]之间的可调节参数;nu代表蛋白质u的邻域。
[0069]
本技术方案的优点:
[0070]
(1)本技术方案首次提出了不同特征得分之间存在一种弱压制关系,满足这种关系的蛋白质更倾向于是关键蛋白质;
[0071]
(2)根据特征之间的弱压制关系,本技术方案设计了弱一致性模型来整合不同的特征得分;
[0072]
(3)将弱一致性模型应用在五种经典算法上,结果表明弱一致性模型拥有更高的识别率与实用价值。
[0073]
这种方法识别率和实用价值高,为关键蛋白质的识别提供了新思路。
附图说明
[0074]
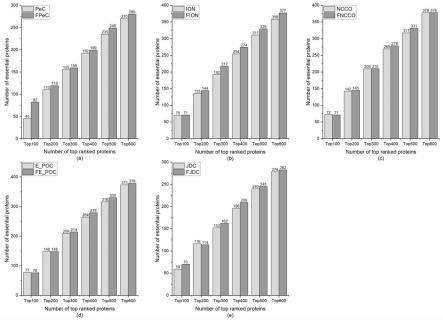
图1为实施例中基于柱状图比较实验结果示意图;
[0075]
图2为实施例中基于precision-recall(准确率-召回率)曲线比较实验结果示意图;
[0076]
图3为实施例中基于jackknife曲线比较实验结果示意图。
具体实施方式
[0077]
下面结合附图及具体实施例对发明作进一步的详细描述,但不是对本发明的限定。
[0078]
实施例:
[0079]
本例实验在一台电脑(intel(r)core(tm)i5-6500 3.20ghz cpu,内存为16g)上进行,操作系统为windows 10,编译运行工具为matlab2021。
[0080]
本例使用s.cerevisiae物种的数据集作为测试数据。ppi数据集来自于三个数据库:biogrid,intact,mint和下载于2014年1月的consolidated ppi数据集,其中有6099个蛋白质,560702个相互作用。关键蛋白质数据集整合了mips,sgd,deg和sgdp四个数据库中的数据,包含了1285个关键蛋白质。直系同源蛋白质数据集从inparanoid的第7版中提取,该数据集由inparaniod程序生成,包含100个参考物种的全基因组之间的蛋白质对比。基因表达数据由tu等人构建,共有6777个基因产物和36个样本。
[0081]
为了验证弱一致性模型的性能,本例将pec、ion、ncco、e_poc和jdc这五种代表性方法作为测试方法,并在s.cerevisiae数据集上进行了实验。在这五种代表性方法中,ion和e_poc都包含参数α,它们在s.cerevisiae数据集上的α值如表1所示。
[0082]
本例在使用弱一致性模型替换pec,ion,ncco,e_poc和jdc的结合模型后,引入了参数ξ和α,fpec,fion,fncco,fe_poc和fjdc在s.cerevisiae数据集上的最佳参数值如表2所示。
[0083]
表1 ion和e_poc在s.cerevisiae上的参数
[0084][0085]
基于弱一致性模型识别关键蛋白质的方法,包括如下步骤:
[0086]
1)设置弱一致性的条件:设蛋白质u和蛋白质v满足弱一致性,即u<<v,表示u弱压制v,弱一致性的条件如下:
[0087]
a(u)》a(v)-ξand b(u)》b(v)-ξ,
[0088]
其中u,v代表蛋白质;a和b表示两种特征得分;ξ是容错因子,为较小的正数;
[0089]
使用图g(v,e)表示ppi网络,v是蛋白质节点的集合,e是边的集合,在确定了蛋白质间的弱压制关系后,结合模型被定义为:
[0090]
[0091]
其中dm
u,v
表示蛋白质u和蛋白质v是否满足弱压制关系,其进一步定义如下:
[0092][0093]
其中α是介于[0,1]之间的可调节参数;
[0094]
2)弱一致性模型评估:
[0095]
为了检验弱一致性模型识别关键蛋白质的能力,本例用弱一致性模型替换了如下几个代表性方法中的结合模型,来观察是否比原方法有更高的关键蛋白质识别率;
[0096]
2-1)pec方法使用边聚集系数用来衡量两个蛋白质连接的紧密程度,使用基于基因表达谱的皮尔逊相关系数来衡量蛋白质间连接的可靠程度,然后使用乘法将边聚集系数和皮尔逊相关系数结合起来,pec的计算公式为:
[0097][0098]
其中ecc(u,v)分别表示蛋白质u和蛋白质v之间的边聚集系数;nv代表蛋白质v的邻域;pcc(u,v)表示蛋白质u和蛋白质v之间的皮尔逊相关系数,ecc(u,v)和pcc(u,v)的公式分别如下:
[0099][0100][0101]
其中nu和nv分别是蛋白质u和蛋白质v的邻域;du和dv分别表示蛋白质u和蛋白质v的度;s表示基因表达数据的样本数;g(u,i)和g(v,i)分别代表蛋白质u和蛋白质v在样本i中的基因表达水平;g(u)和g(v)分别是蛋白质u和蛋白质v的基因表达水平的均值;σ(u)和σ(v)分别表示蛋白质u和蛋白质v的基因表达水平的标准差,本例使用公式(1)的弱一致性模型替换公式(3),然后将替换结合模型后的方法命名为fpec,fpec满足条件为:
[0102]
ecc(u)》ecc(v)-ξandpcc(u)》pcc(v)-ξ,
[0103]
其中ecc(u)和ecc(v)分别表示蛋白质u、蛋白质v的边聚集系数;pcc(u)和pcc(v)分别代表蛋白质u和蛋白质v的皮尔逊相关系数;ξ是容错因子,为较小的正数,采用结合模型给出最终得分,计算公式如下:
[0104][0105]
其中α是介于[0,1]之间的可调节参数,nu代表蛋白质u的邻域,具体流程如下:
[0106]
[0107][0108]
2-2)ion方法使用直系同源信息来衡量蛋白质的保守特征,用边聚集系数来反映ppi网络的拓扑特征,然后使用随机游走模型将边聚集系数和直系同源信息结合起来,ion的计算公式为:
[0109]
pr
t 1
=(1-α)d α*h*pr
t
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(7),
[0110]
其中pr表示排序得分,t是迭代的次数,d代表直系同源矩阵,h为归一化的ecc矩阵,d的公式如下所示:
[0111][0112][0113]
其中o(u)为蛋白质u在参考生物体中存在直系同源蛋白质的参考物种数目,h的公式如下所示:
[0114][0115]
其中nu为蛋白质u的邻域,ecc(u,v)的公式如下:
[0116][0117]
其中,z
u,v
是包含边(u,v)的三角形的数量,ku和kv分别是蛋白质u和蛋白质v的度;min()是取最小值函数,本例使用公式(1)的弱一致性模型替换公式(7),然后将替换结合模型后的方法命名为fion,fion满足的条件为:
[0118]
d(u)》d(v)-ξandhr(u)》hr(v)-ξ,
[0119]
其中d(u)表示蛋白质u的直系同源得分;hr表示h*pr
t
;ξ是容错因子,为较小的正数,采用结合模型给出最终得分,计算公式如下:
[0120][0121]
其中α是介于[0,1]之间的可调节参数;nu代表蛋白质u的邻域,具体流程如下:
[0122][0123]
2-3)ncco方法根据ppi网络的拓扑特征提出了邻居接近中心性即ncc,然后用expoc模型将直系同源数据和ncc结合起来,ncco的计算公式为:
[0124]
fs(u)=∑
u<v
(ncc(u)-ncc(v)) (os(u)-os(v))
ꢀꢀꢀꢀ
(12),
[0125]
其中u<v表示u压制v;ncc(u)表示蛋白质u的邻居接近中心性得分;os表示直系同源得分,os的公式与公式(8)一致,ncc的计算公式为:
[0126]
cc(u)=(n-1)/∑vp(u,v)
[0127][0128]
其中n为ppi网络中蛋白质的总数,p(u,v)表示蛋白质u、v之间的最短路径;du和dv表示蛋白质u和蛋白质v的度;min()为取最小值函数;nu表示蛋白质u的邻域;t
u,v
是包含蛋白质u和蛋白质v的所有三角形的点的集合,本例使用公式(1)的弱一致性模型替换公式(12),然后将替换结合模型后的方法命名为fncco,fncco满足的条件为:
[0129]
ncc(u)》ncc(v)-ξand os(u)》os(v)-ξ,
[0130]
其中ξ是容错因子,为较小的正数,采用结合模型给出最终得分,计算公式如下:
[0131][0132]
其中α是介于[0,1]之间的可调节参数;nu代表蛋白质u的邻域,具体流程如下:
[0133][0134]
2-4)e_poc方法采用边聚集系数来衡量蛋白质聚集在一起的程度;使用皮尔逊相关系数表示两个蛋白质的基因表达水平,来衡量边的可靠性;采用直系同源信息来衡量蛋白质本身的重要性;最后使用拓展的poc模型将皮尔逊相关系数、边聚集系数、直系同源信息融合在一起,e_poc的计算公式为:
[0135][0136][0137]
其中ps代表直系同源得分,ps的公式与公式(8)相同;α是介于[0,1]之间的可调参数;pn代表边聚集系数和皮尔逊相关系数的相加值,其公式如下:
[0138]
pn(u)=ecc(u) pcc(u)
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(16),
[0139]
其中ecc表示边聚集系数,ecc的公式与公式(10)相同,pcc表示皮尔逊相关系数,pcc的公式和公式(5)一致,本例使用公式(1)的弱一致性模型替换公式(15),然后将替换结合模型后的方法命名为fe_poc,fe_poc满足条件为:
[0140]
pn(u)》pn(v)-ξand ps(u)》ps(v)-ξ,
[0141]
其中ξ是容错因子,为较小的正数,采用结合模型给出最终得分,计算公式如下:
[0142][0143]
其中α是介于[0,1]之间的可调节参数;nu代表蛋白质u的邻域,具体流程如下:
[0144][0145][0146]
2-5)jdc方法采用一种动态阈值方法来将基因表达数据进行二值化,并使用乘法将jaccard相似性指数和边聚集系数结合起来预测关键蛋白,jdc的计算公式为:
[0147][0148]
其中nu代表蛋白质u的邻域;jaccard(u,v)代表边(u,v)的jaccard相似度;ecc(u,v)代表边(u,v)的边聚集系数,ecc与公式(10)一样,jaccard相似度的计算公式如下:
[0149][0150]
其中,su和sv表示蛋白质u和蛋白质v的基因表达数据的布尔值,jaccard相似度系数在0到1之间,本例使用公式(1)的弱一致性模型替换公式(18),然后将替换结合模型后的方法命名为fjdc,fjdc满足条件为:
[0151]
jaccard(u)》jaccard(v)-ξand ecc(u)》ecc(v)-ξ,
[0152]
其中jaccard(u)和jaccard(v)分别表示蛋白质u和蛋白质v的jaccard相似度系数;ecc(u)和ecc(v)分别表示蛋白质u和蛋白质v的边聚集系数,ξ是容错因子,为较小的正
数,采用结合模型给出最终得分,计算公式如下:
[0153][0154]
其中α是介于[0,1]之间的可调节参数;nu代表蛋白质u的邻域,具体流程如下:
[0155][0156][0157]
本例与现有方法对比结果如图1-图3所示,采用本例方法替换了结合模型后,识别关键蛋白质的效率明显高于现有方法。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。