1.本发明涉及生物信息领域,具体地,涉及一种可重构基于硬件的核苷酸比对工具。
背景技术:
2.自2001年人类基因组初稿完成以来,基因组数据一直远远超过摩尔定律,预计今年下半年会达到千兆字节的规模,在未来其也不可估计。基因组数据能够在医疗、检测疾病和在人的生命形成过程之中都有贡献。
3.数据的增长主要由于dna测序技术的发展。生物信息学是一跨学科的领域,改进了使用各种算法存储、检索、组织和分析生物数据的方法。在分子生物学中,测序使研究人员能够获得关于基因突变、疾病和表型关联等信息。在进化生物学中,使用测序获得的信息可以提供关于不同生物如何相关以及如何演变。
技术实现要素:
4.针对现有技术中的缺陷,本发明的目的是提供一种可重构基于硬件的核苷酸比对工具。
5.根据本发明提供的可重构基于硬件的核苷酸比对工具,包括计算机、fpga模块以及通用异步收发器桥;
6.所述计算机,用于运行blastn算法以根据目标序列查询相似的核苷酸序列;
7.所述fpga模块,用于加速blastn算法的史密斯
‑
沃特曼部分;
8.所述通用异步收发器桥,用于计算机和fpga模块之间的串行通信。
9.优选地,所述史密斯
‑
沃特曼部分时包括如下步骤:
10.读取目标序列,根据所述目标序列查询到所有可能的待匹配序列;
11.对每段待匹配序列进行评分生成匹配分数,去除匹配分数低于预设置分数阈值的待匹配序列;
12.将匹配分数最高的若干个核苷酸序列确定为相似序列。
13.优选地,所述史密斯
‑
沃特曼部分时还包括如下步骤:
14.根据所述目标序列对应的相似序列进行对所述目标序列进行扩展查询;
15.在扩展时根据目标序列的每段对应的主题序列进行评分生成匹配分数,根据最高匹配分数筛选出每段的相似序列段;
16.当匹配分数低于预设置的分数阈值时,扩展查询停止,并丢弃分数低于分数阈值的相关序列;
17.对于所有相似序列段进行合并,并按e值递增排序,并返回e值和s值,s值表示两序列的同源性,分值越高表明它们之间相似程度越大;e值就是s值的可靠性的评价,表明在随机的情况下,其他序列与目标序列相似度要大于该条相似序列的可能性,所以分值越低越好。
18.优选地,根据目标序列查询相似的核苷酸序列包括如下步骤:
19.将目标序列读入关联数组,其中每个序列被分成长度为n的字,且为了减少内存占用,避免为每个序列存储重复的字;
20.将每个序列都与其前一序列的索引相关联;
21.将匹配得到的序列用灰尘过滤算法再次进行处理,得到的结果如果在相似度指定范围内,那么认为是一对相邻序列;
22.对相邻序列的进行扩展查询,接而打分,并将低分的序列去除,最高分序列配对的写入指定的用户文件。
23.本发明提供的可重构基于硬件的核苷酸比对工具,包括计算机、fpga模块以及通用异步收发器桥;
24.所述计算机,用于运行hardware
‑
blastn算法以根据目标序列查询相似的核苷酸序列;
25.所述fpga模块,用于加速hardware
‑
blastn算法的部分模块进行加速;
26.所述通用异步收发器桥,用于计算机和fpga模块之间的串行通信。
27.优选地,所述根据目标序列查询相似的核苷酸序列时包括如下步骤:
28.读取目标序列,根据所述目标序列查询到所有可能的待匹配序列;
29.对每段待匹配序列进行评分生成匹配分数,去除匹配分数低于预设置分数阈值的待匹配序列;
30.将匹配分数最高的若干个核苷酸序列确定为相似序列。
31.优选地,所述根据目标序列查询相似的核苷酸序列时还包括如下步骤:
32.根据所述目标序列对应的相似序列进行对所述目标序列进行扩展查询;
33.在扩展时根据目标序列的每段对应的主题序列进行评分生成匹配分数,根据最高匹配分数筛选出每段的相似序列段;
34.当匹配分数低于预设置的分数阈值时,扩展查询停止,并丢弃分数低于分数阈值的相关序列;
35.对于所有相似序列段进行合并,并按e值递增排序,并返回e值。
36.优选地,根据目标序列查询相似的核苷酸序列包括如下步骤:
37.将目标序列读入关联数组,其中每个序列被分成长度为n的字,且为了减少内存占用,避免为每个序列存储重复的字;
38.将每个序列都与其前一序列的索引相关联;
39.将匹配得到的序列用灰尘过滤算法再次进行处理,得到的结果如果在相似度指定范围内,那么认为是一对相邻序列;
40.对相邻序列的进行扩展查询,接而打分,并将低分的序列去除,最高分序列配对的写入指定的用户文件。
41.与现有技术相比,本发明具有如下的有益效果:
42.本发明设置有计算机和fpga模块,通过计算机运行blastn算法以根据目标序列查询相似的核苷酸序列,通过所述fpga模块加速blastn算法的部分模块,利用硬件并行处理的优势去执行测序,提高了测序效率。
附图说明
43.通过阅读参照以下附图对非限制性实施例所作的详细描述,本发明的其它特征、目的和优点将会变得更明显:
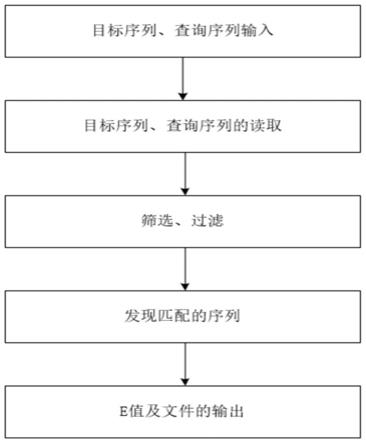
44.图1为本发明实施例中可重构基于硬件的核苷酸比对工具的使用流程框图;
45.图2为本发明实施例中算法结构在逻辑门阵列的框架图。
具体实施方式
46.下面结合具体实施例对本发明进行详细说明。以下实施例将有助于本领域的技术人员进一步理解本发明,但不以任何形式限制本发明。应当指出的是,对本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进。这些都属于本发明的保护范围。
47.图1为本发明实施例中可重构基于硬件的核苷酸比对工具的使用流程框图,图2为本发明实施例中算法结构在逻辑门阵列的框架图,如图1、图2所示,本发明提供的可重构基于硬件的核苷酸比对工具,包括计算机、fpga模块以及通用异步收发器桥;
48.所述计算机,用于运行blastn算法以根据目标序列查询相似的核苷酸序列;
49.所述fpga模块,用于加速blastn算法的史密斯
‑
沃特曼部分;
50.所述通用异步收发器桥,用于计算机和fpga模块之间的串行通信。
51.在本发明实施例中,所述史密斯
‑
沃特曼部分时包括如下步骤:
52.读取目标序列,根据所述目标序列查询到所有可能的待匹配序列;
53.对每段待匹配序列进行打分,去除低分序列,对剩余待匹配序列的进行匹配;
54.将匹配分数极高者确定为相似序列。
55.根据所述目标序列对应的相似序列进行对所述目标序列进行扩展查询;
56.在扩展时根据目标序列的每段对应的主题序列进行评分,根据分数筛选相关的序列;
57.当分数低于预设置的分数阈值,扩展查询停止,并丢弃分数低于阈值的相关序列;
58.对于所有的高分序列进行合并,并按e值递增排序,并返回e值。
59.在本发明实施例中,根据目标序列查询相似的核苷酸序列包括如下步骤:
60.将目标序列读入关联数组,其中每个序列被分成长度为n的字,且为了减少内存占用,避免为每个序列存储重复的字;
61.将每个序列都与其前一序列中出现的索引相关联;
62.将匹配得到的序列用灰尘过滤算法再次进行处理,得到的结果如果在相似度指定范围内,那么认为是一对相邻序列;
63.对相邻序列的进行扩展查询,接而打分,并将低分的序列去除,最后高分序列配对的写入指定的用户文件。
64.在本发明变形例中,本发明提供的可重构基于硬件的核苷酸比对工具,包括计算机、fpga模块以及通用异步收发器桥;
65.所述计算机,用于运行hardware
‑
blastn算法以根据目标序列查询相似的核苷酸序列;
66.所述fpga模块,用于加速blastn算法的部分模块进行加速;
67.所述通用异步收发器桥,用于计算机和fpga模块之间的串行通信。
68.在本发明实施例中,所述根据目标序列查询相似的核苷酸序列时包括如下步骤:
69.读取目标序列,根据所述目标序列查询到所有可能的待匹配序列;
70.对每段待匹配序列进行打分,去除低分序列,对剩余待匹配序列的进行匹配;
71.将匹配分数极高者确定为相似序列。
72.所述根据目标序列查询相似的核苷酸序列时还包括如下步骤:
73.根据所述目标序列对应的相似序列进行对所述目标序列进行扩展查询;
74.在扩展时根据目标序列的每段对应的主题序列进行评分,根据分数筛选相关的序列;
75.当分数低于预设置的分数阈值,扩展查询停止,并丢弃分数低于阈值的相关序列;
76.对于所有相似序列段进行合并,并按e值递增排序,并返回e值和s值,s值表示两序列的同源性,分值越高表明它们之间相似程度越大;e值就是s值的可靠性的评价,表明在随机的情况下,其他序列与目标序列相似度要大于该条相似序列的可能性,所以分值越低越好。
77.更为具体地,根据目标序列查询相似的核苷酸序列包括如下步骤:
78.将目标序列读入关联数组,其中每个序列被分成长度为n的字,且为了减少内存占用,避免为每个序列存储重复的字;
79.将每个序列都与其前一序列中出现的索引相关联;
80.将匹配得到的序列用灰尘过滤算法再次进行处理,得到的结果如果在相似度指定范围内,那么认为是一对相邻序列;
81.对相邻序列的进行扩展查询,接而打分,并将低分的序列去除,最后高分序列配对的写入指定的用户文件。
82.当对两个输入序列执行比对时。为此,设置一个2d矩阵,其中每个轴的长度加1。这个矩阵用零填充,它将被移动,从索引(1,1)开始,使行和列零只包含零。遍历开始,每个单元格的分数由以下值中最大一个决定:零,左单元格之和与匹配分数之和。单元格上一格匹配分数之和,或者对角线左匹配分数之和。当这个过程发生时,最高得分值的位置和值被记录。当矩阵被填满时,发生回溯,其中遍历从最大值,以及相应的向上、向左或对角移动被执行,直到达到零,其中向上或向左对应于间隙,对角对应于匹配。此时,对齐已经完成,并且可以获得相应的相似性百分比和分数。
83.对于硬件设计,需要两个主要组件:一个带有hardware
‑
blastn硬件算法的计算机,以及一个用于加速hardware
‑
blastn算法部分的硬件阵列。使用带有一个通用串行总线通用异步收发器桥开发板,用于pc机和现场可编程门阵列之间的串行通信。硬件实现被设计成让现场可编程门阵列根据其速度限制计算hardware
‑
blastn的部分。这些速度限制受到现场可编程门阵列速度或现场可编程门阵列与个人电脑之间串行通信速度的制约。因此,如果给定更强的现场可编程门阵列或现场可编程门阵列,算法进一步加速。
84.当使用本发明提供的基于硬件的核苷酸比对工具时,可以在蛋白序列库、核酸基因序列库中进行高效率比对,首先实验者先在c 程序中输入两条dna序列,生成.exe文件。而后在电脑的cmd界进行操作与现场可编程门阵列板卡的连接。本发明利用硬件并行处理的优势去执行,以此来提高测序效率。而后比对结果返回。
85.本发明不仅在个性化医疗有价值,如检测人类易患某些疾病(包括癌症、自闭症和衰老等)的基因组突变。也有助于理解导致表型多样性的分子因素(如头发和皮肤颜色等)。不单一在人类,也可使用在其他生命物种中。本发明不仅于对测序算法在硬件上的进行了研究,利用硬件结构对其运算算法进行加速,在可编程逻辑门阵列上代码进行算法编程。
86.以上对本发明的具体实施例进行了描述。需要理解的是,本发明并不局限于上述特定实施方式,本领域技术人员可以在权利要求的范围内做出各种变形或修改,这并不影响本发明的实质内容。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。