用于有效地确定组织的区域中的多种细胞类型的密度和空间关系特征的方法
1.相关申请的交叉引用
2.本技术要求于2020年4月27日提交的美国申请第63/016,004号的优先权的权益,该美国申请的全部内容出于所有目的通过引用并入本文。
背景技术:
3.免疫组织化学(ihc)载玻片图像分析可涉及计算分析。例如,组织切片的多重免疫荧光(mif)染色允许同时检测多个生物标志物以及它们在单细胞水平下的共表达。mif使得能够表征肿瘤微环境中的免疫环境,这对于对免疫疗法的应答具有显著影响。初级分析可对所有不同类型的标志物/细胞表型进行检测和分类。在初始分析后,第二次分析是用于生成不同的特征(诸如密度或空间关系)以关联临床研究或试验来预测预后或药物性能的非常重要的步骤。需要有效地生成二级分析的有用结果的方法和系统。这些改进和其他改进通过本文所述的实施例得以实现。
技术实现要素:
4.本发明的实施例允许有效地确定组织的区域中的多种细胞类型的密度和空间关系特征。效率可能是一个或多个方面的结果。用于图像的相同高倍率被用于分析目标区域(例如,肿瘤)中的特征(例如,细胞、表型、上皮肿瘤(epitumor)),避免了针对不同的倍率进行调整的计算。特征形状可能是复杂的,并可通过像素坐标(例如,对应于沿特征周边的每个点或特征中心处的点)的大的集合来描述。使用像素坐标的集合执行空间评定(例如,以确定特征是否在给定的目标区域如肿瘤区内)则可能涉及大量的计算。为了提高计算效率,具有较简单形状的边界区(例如,框)可被限定为在特征周围的区域。边界区加快了对特征是否在肿瘤或另一特定目标区域内的分析。例如,如果边界框的四个顶点都在目标区域内,则可确定特征充分地在该区域内。哈希表可用于识别代表目标区域、组织和肿瘤中的特征的多边形之间的关系。此外,用于二级分析的参数被识别为生物标志物,这可能有助于表征肿瘤或其他目标区域。
5.实施例可包括一种使用生物学样品的图像来识别针对肿瘤的生物标志物的方法。所述方法可包括识别所述图像内的肿瘤区。所述肿瘤区可描绘所述肿瘤的边界。所述方法可进一步包括识别所述图像的多个区域。所述多个区域中的每个区域对应于组织块或生物学对象(例如,癌周、基质、细胞表型等)。所述方法还可包括:针对所述多个区域中的每个区域,限定针对所述区域的包围所述区域的边界区(例如,边界框或边界椭圆形),其中所述边界区包括多边形或椭圆形。所述方法可包括:针对所述多个区域的第一子集中的每个区域,确定所述区域将归属于肿瘤,其中针对所述第一子集中的每个区域的所述边界区完全在所述肿瘤区内。另外,所述方法可包括:针对所述多个区域的第二子集中的每个区域,确定针对所述区域的所述边界区部分地在所述肿瘤区内。所述方法可进一步包括:针对所述多个区域的所述第二子集中的每个区域,基于所述区域与所述肿瘤区的交集确定是否将所述区
域归属于所述肿瘤。所述方法还可包括:针对归属于所述肿瘤的每个区域,访问表征生物学观察的度量。所述方法可包括基于所访问的度量来生成结果的值。所述方法还可包括:将所述结果的所述值与参考值进行比较,所述参考值通过使用归属于另一肿瘤的区域来确定,或者通过使用归属于所述肿瘤区的完全外部的区域来确定。另外,所述方法可包括基于所述比较来确定所述结果是否为生物标志物
6.实施例可包括一种分析生物学样品的图像的方法。所述方法可包括确定第一目标区域(例如,肿瘤)内的细胞表型的第一计数或第一密度。所述方法还可包括确定第二目标区域(例如,基质)内的细胞表型的第二计数或第二密度。第二目标区域可在第一目标区域内。所述方法可进一步包括生成输出。所述输出可包括所述第一计数或所述第一密度以及所述第二计数或所述第二密度。
7.实施例可包括一种分析生物学样品的图像的方法。所述方法可包括识别肿瘤中的多个第一细胞。该多个第一细胞中的每个第一细胞可具有第一表型。所述方法还可包括识别所述肿瘤中的多个第二细胞。该多个第二细胞中的每个第二细胞可具有第二表型。所述方法可进一步包括:针对所述多个第一细胞中的每个第一细胞,使用最近邻搜索计算到所述多个第二细胞中的第二细胞的最短距离。另外,所述方法可包括基于计算出的最短距离生成结果。
8.实施例可包括一种分析生物学样品的图像的方法。所述方法可包括识别肿瘤中的多个第一细胞。该多个第一细胞中的每个第一细胞可具有第一表型。所述方法还可包括:识别所述肿瘤中的一个或多个区域,其中所述一个或多个区域中的每个区域对应于组织块或生物学对象(例如,癌周、基质等)。所述方法可进一步包括:针对所述多个第一细胞中的每个第一细胞,使用最近邻搜索计算所述第一细胞与所述一个或多个区域中的每个区域之间的最短距离。另外,所述方法可包括基于计算出的最短距离生成结果。
9.实施例可包括一种分析生物学样品的图像的方法。所述方法可包括:识别肿瘤中的多个区域,其中所述多个区域中的每个区域对应于组织块或生物学对象(例如,癌周、基质等)。所述方法还可包括:针对所述多个区域中的每个区域,确定压缩级别。所述方法可进一步包括基于所述压缩级别生成结果。
10.参考以下具体实施方式和附图,可以更好地理解本发明的实施例的性质和优点。
附图说明
11.图1a和图1b示出了对组织的mif图像的分析。
12.图1c示出了根据本发明的实施例的使用高放大倍率与使用低放大倍率之间的计算时间的比较。
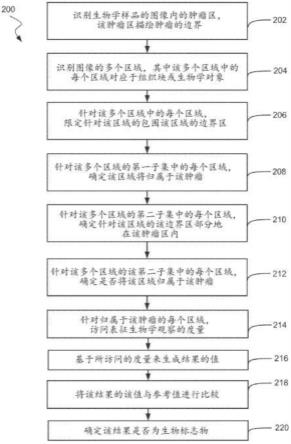
13.图2示出了根据本发明的实施例的分析生物学样品的图像的方法。
14.图3a示出了根据本发明的实施例的用于分析生物学样品的图像的流程图。
15.图4示出了根据本发明的实施例的图像的被分析区域。
16.图5a示出了根据本发明的实施例的肿瘤组织的示例性图像。
17.图5b以图表示出了根据本发明的实施例的肿瘤组织中的细胞类型的分析结果。
18.图6以图表示出了根据本发明的实施例的肿瘤组织中的细胞类型的分析结果。
19.图7示出了根据本发明的实施例的针对细胞表型的密度或计数分析生物学样品的
图像的方法。
20.图8a示出了根据本发明的实施例的针对单个细胞表型的计数或密度的分析。
21.图8b示出了根据本发明的实施例的计数细胞表型的结果。
22.图9示出了根据本发明的实施例的针对表型距离分析生物学样品的图像的方法。
23.图10示出了根据本发明的实施例的对距离的计算。
24.图11示出了根据本发明的实施例的肿瘤中的多种表型。
25.图12示出了根据本发明的实施例的不同表型之间的距离。
26.图13示出了根据本发明的实施例的针对表型之间的距离的结果的表格。
27.图14示出了根据本发明的实施例的上皮肿瘤和细胞的轮廓。
28.图15a示出了根据本发明的实施例的生物学样品的图像。
29.图15b示出了根据本发明的实施例的带有注释肿瘤区的红圈的放大图像。
30.图15c示出了根据本发明的实施例的包括表示上皮肿瘤的蓝色多边形和表示表型的细胞的黑圈的图像。
31.图16示出了根据本发明的实施例的对肿瘤内的单个上皮肿瘤多边形的选择。
32.图17示出了根据本发明的实施例的被五条轮廓线包围的单个上皮肿瘤。
33.图18示出了根据本发明的实施例的带有从细胞到上皮肿瘤的距离的结果的表格。
34.图19示出了根据本发明的实施例的针对从细胞到区域的距离分析生物学样品的图像的方法。
35.图20示出了根据本发明的实施例的针对压缩级别分析生物学样品的图像的方法。
36.图21示出了根据本发明的实施例的具有不同压缩级别的区域的肿瘤。
37.图22示出了根据本发明的实施例的以多边形突出显示的不同压缩级别。
38.图23示出了根据本发明的实施例的针对压缩级别的结果的列表。
39.图24示出了根据本发明的实施例的计算机系统。
具体实施方式
40.组织切片的图像的某些特征,诸如密度和空间关系,可提供有用和重要的临床信息。这些特征可用于了解受试者的临床状态,包括用于诊断以及用于监测治疗。
41.基于低放大倍率掩码的二级分析可用于对图像进行二级分析,以确定诸如密度和空间关系等特征。然而,使用低放大倍率掩码可能是计算上密集的。
42.图1a和图1b示出了对组织的mif图像的分析。两个图都示出了在蓝色矩形区域中显示的放大区段。使用计算出的多边形生成了低放大倍率掩码。
43.图1c示出了一个表,其比较了使用高放大倍率多边形逼近方法和基于低放大倍率掩码的方法的计算时间。根据本发明的实施例,与基于低放大倍率掩码的方法相比,基于高放大倍率多边形的方法的计算时间要快得多。通过使用基于低放大倍率掩码的方法已经实现了两个下采样因子,并且性能示出在图1c中。一个具有以4倍下采样原始mif图像的低放大倍率。另一个具有27倍下采样的低放大倍率。例如,在特定面板中,使用基于高放大倍率多边形的方法的计算时间仅以大约3分钟完成;尽管是最慢的,以4的下采样进行的低放大倍率掩码方法以4.32小时(260分钟)完成,历经88倍长。此外,基于高放大倍率多边形的分析生成准度高。
44.i.边界区
45.在本发明的实施例中可实现准确和计算有效的结果。使用边界区(诸如盒)来包围多边形有助于加快分析。
46.a.识别生物标志物的方法
47.图2示出了分析生物学样品的图像以识别针对肿瘤的生物标志物的方法200。在一些实施例中,方法200可包括捕获生物学样品的图像。图像可能来自各种免疫组织化学(ihc)载玻片图像,其可包括单个载玻片或者两个或更多个相邻载玻片,每个载玻片具有不同的生物标志物或细胞表型。图像可以是组织切片的多重免疫荧光(mif)染色图像。生物学样品可以是单个组织。在一些情况下,可对多个生物学样品进行成像。例如,来自多个活体组织切片、多个组织块或组织微阵列的图像可用于分析。
48.在框202处,可识别图像中的肿瘤区。肿瘤区可描绘肿瘤的边界。在一些实施例中,可使用与除肿瘤之外的生物学结构相对应的区域。其他生物学结构可包括肿瘤、基质、上皮肿瘤或癌周。肿瘤区可通过人工或通过计算机程序来识别。
49.在框204处,可识别图像的多个区域。该多个区域中的每个区域可对应于组织块或生物学对象。组织块或生物学对象可以是基质、上皮肿瘤、癌周、血管(例如,血液)、细胞表型、肿瘤、坏死、组织褶皱、染色伪影或红细胞。组织块或生物学对象可通过人工或通过计算机程序来识别。该多个区域可覆盖整个肿瘤区和/或整个图像。
50.在框206处,可针对多个区域中的每个区域,限定针对该区域的边界区。边界区可包围该区域。在一些实施例中,边界区可外切该区域,使得该区域的至少一个点与边界区的边缘接触。边界区可具有在比该区域大10%、20%、30%、40%、50%、60%、70%、80%、90%或100%的范围内的面积。边界区可包括多边形或椭圆形。例如,边界区可以是边界盒或边界圆。边界区可包括三角形、矩形、五边形、六边形、七边形、八边形、九边形、十边形或者具有11条边或更多条边的多边形。在一些实施例中,多边形可以是规则的。
51.在框208处,该多个区域的第一子集中的每个区域可被确定为归属于肿瘤,其中针对第一子集中的每个区域的边界区完全在肿瘤区内。当多边形或椭圆形的点的子集被确定为在肿瘤区内时,边界区可被确定为完全在肿瘤区内。例如,在2d坐标系中,表示沿两条轴线的极值的点可与肿瘤区进行比较。例如,最上点、最下点、最左点和/或最右点可被确定为在肿瘤区中。在一些实施例中,多边形的顶点可被确定为在肿瘤区中。
52.在一些实施例中,该多个区域的第一子集也可被确定为在被排除的区域之外。被排除的区域可包括生物学结构(例如,肿瘤、癌周、基质、坏死、红细胞、染色伪影或组织褶皱)。对区域在被排除的区域之外的确定将类似于对区域在肿瘤区内的确定。当顶点或极值点被确定为在被排除的区域之外时,边界区可被确定为在被排除的区域之外。
53.在框210处,针对该多个区域的第二子集中的每个区域的边界区可被确定为部分地在肿瘤区内。当表示极值的点中的至少一个点(但少于所有表示极值的点)中的每个点被确定为在肿瘤区之外时,边界区可被确定为部分地在肿瘤区内。在一些实施例中,边界多边形的一个或多个顶点(但不是所有顶点)可被确定为在肿瘤区之外。
54.在一些实施例中,针对该多个区域的第三子集中的每个区域的边界区可被确定为肿瘤区的完全外部。当所有表示极值的点或边界多边形的所有顶点都被确定为在肿瘤区之外时,边界区可被确定为肿瘤区的完全外部。
55.在框212处,是否将区域归属于肿瘤可针对该多个区域的第二子集中的每个区域来确定。该确定基于该区域和肿瘤区的交集/排除。在一些实施例中,交集被计算为与肿瘤区重叠的区域的面积。仅表示肿瘤区内的区域的部分的新子区域可用于分析。
56.在其他实施例中,交集可由肿瘤内的区域的面积或百分比来表征。可将交集与阈值(例如,最小面积或最小百分比)进行比较。当交集超过阈值时,则该区域归属于肿瘤。如果交集未超过阈值,则该区域不归属于肿瘤。交集可使用该区域和肿瘤区的几何结构来计算。
57.在一些实施例中,区域的一定比例可基于交集百分比而归属于肿瘤。例如,与交集百分比相同的百分比可归属于肿瘤。从该区域确定的任何度量都可按交集百分比进行缩放。
58.两个哈希表可用于跟踪多边形/区域与肿瘤/组织区域之间的关系。肿瘤哈希表可用关于区域/多边形是否归属于肿瘤的信息进行更新。此外,肿瘤哈希表还可跟踪关于区域是部分地/完全在肿瘤区之内还是在肿瘤区之外的信息;其还可更新肿瘤区(肿瘤的数量指标)所属的区域/多边形。类似地,组织哈希表可用关于区域/多边形是否归属于组织的信息进行更新。此外,区域是部分地/完全在组织区域之内还是在组织区域之外,以及组织区域(组织的数量指标)所属的区域。
59.在框214处,可针对归属于肿瘤的每个区域,访问表征生物学观察的度量。该度量可以是本文所公开的任何度量。例如,该度量可以是该区域内的特定类型的细胞或表型的计数或密度。特定类型细胞的计数或密度可以是在肿瘤区内的或在肿瘤的完全外部的区域的计数或密度。表征度量可包括:引用哈希表以确定该区域是否归属于肿瘤/组织。
60.例如,该度量可包括不同类型区域的位置。该多个区域可包括与第一组织块或第一生物学对象相对应的第一区域。该多个区域可进一步包括与第二组织块或第二生物学对象相对应的第二区域。第一区域和第二区域中的每一者被确定为归属于肿瘤。表征生物学观察的度量可识别与该区域相关联的位置。
61.在一些实施例中,第一组织块或第一生物学对象可以是第一表型的细胞,该第一表型可以是单个生物标志物或不同生物标志物的共定位。第二组织块或第二生物学对象可以是第二表型的细胞。该度量可包括细胞/表型的位置。
62.在一些实施例中,第一区域可对应于第一表型的细胞,并且第二区域可对应于第二组织块。该度量可包括细胞的位置以及组织或其他区域的位置。
63.该度量可包括区域或表型的不同特征,诸如绝对强度值、强度百分比和分布、形状、面积、压缩级别等。该多个区域中的每个区域可对应于组织块或生物学对象。压缩级别可以是与组织块或生物学对象相对应的区域的压缩级别。压缩级别作为本文件稍后示出的特征示例进行描述。
64.在框216处,可基于所访问的度量来生成结果的值。该结果可能针对归属于肿瘤的区域而生成。例如,对于当该度量包括区域或细胞的位置时而言,该结果可识别由该度量针对特定区域(例如,第一区域)识别的第一位置与由该度量针对另一区域(例如,第二区域)或细胞识别的第二位置之间的距离。该结果可包括所访问的度量中的每个度量。在一些实施例中,该结果可包括所访问的度量的统计值。例如,该结果可包括所访问的度量的平均值、中值、众数、百分位数或标准偏差。当一个或多个所访问的度量超过阈值时,该结果可包
括统计值。
65.在一些实施例中,可将特定组织块或生物学对象中的特定细胞的计数或密度用作度量。该多个区域的子集可对应于组织块或生物学对象。该方法可包括:针对该多个区域的该子集中的每个区域,访问表征生物学观察的度量。生成结果的值可包括:确定包括针对归属于肿瘤的每个区域以及针对归属于该多个区域的该子集的每个区域的度量的函数的值。例如,该结果可以是针对组织块的特定类型细胞的计数或密度与针对肿瘤区的特定类型细胞的计数或密度的比率。该结果可以是利用图7、图8a和图8b所描述的输出中的任何输出。
66.在一些实施例中,各类型细胞之间的距离可用于生成结果的值。该多个区域可包括带有多个第一细胞的第一区域,该多个第一细胞具有第一表型。该多个区域可包括带有多个第二细胞的第二区域,该多个第二细胞具有第二表型。第一区域和第二区域可被确定为归属于肿瘤。表征生物学观察的度量可识别区域内的多个细胞中的每个细胞的位置。该结果可通过以下方式来生成:针对该多个第一细胞中的每个第一细胞,使用最近邻搜索计算到该多个第二细胞中的第二细胞的最短距离。最近邻搜索可以是k最近邻(knn)搜索。可计算出该最短距离的统计值。该结果可包括该统计值。在一些实施例中,可确定具有距具有一定范围的第二细胞的最短距离的第一细胞的计数。该结果可以是该计数。可在识别图像内的肿瘤区之前预先确定该范围。该结果可以是利用图9至图13所描述的结果。
67.在一些实施例中,细胞与组织块或生物学对象之间的距离可用于生成结果的值。该多个区域可包括带有多个第一细胞的第一区域,该多个第一细胞具有第一表型。该多个区域可包括一个或多个第二区域。每个第二区域可对应于组织块或生物学对象。第一区域和该一个或多个第二区域中的每一者可被确定为归属于肿瘤。表征生物学观察的度量可识别每个区域的至少一个位置。该至少一个位置可以是第一区域中该多个第一细胞中的每个细胞的位置。该至少一个位置可以是该一个或多个第二区域中的每个第二区域的位置。每个第二区域的位置可以是该区域的中心或该第二区域的边缘上的点。第二区域的边缘上的点可以是最接近第一细胞的点。结果的值可通过以下方式来生成:针对该多个第一细胞中的每个细胞,使用最近邻搜索计算到该一个或多个区域中的第二区域的最短距离。最近邻搜索可以是k最近邻搜索。该结果可包括计算该最短距离的统计值,并且该结果可以是该统计值。在一些实施例中,该方法可包括:确定在一定范围内的具有该最短距离的第一细胞的计数。可在识别图像内的肿瘤区之前预先确定该范围。该结果可以是该计数。该结果可以是利用图15a至图19所描述的结果。
68.第一细胞与第二区域之间的距离可以是最近垂直距离或最近水平距离。knn搜索可确定该多个第一细胞中具有到给定第二区域的最近垂直距离的第一细胞。该方法可包括:计算具有该最近垂直距离的细胞与给定第二区域之间的距离。也可使用具有该最近水平距离的细胞。
69.可针对每个图像或每个生物学样品报告结果。在其他实施例中,可针对多个图像或多个生物学样品将结果聚合在一起。该聚合可以是针对该多个图像或生物学样品一起报告结果或提供度量的统计值。本文所述的任何结果或度量都可如上所述进行使用。
70.在框218处,可将结果的值与参考值进行比较,该参考值通过使用归属于另一肿瘤的区域来确定,或者通过使用归属于肿瘤区的完全外部的区域来确定。
71.参考值可通过使用肿瘤区的完全外部的区域来确定。例如,针对该多个区域的第
三子集中的每个区域,边界区可被确定为肿瘤区的完全外部。可针对归属于肿瘤的完全外部的每个区域,访问表征生物学观察的度量。以与结果的值被生成的方式相同的方式,基于针对归属于肿瘤的完全外部的每个区域所访问的度量可生成参考值。
72.一个或多个参考受试者中的一个或多个肿瘤可用于确定参考值。该一个或多个参考受试者可能已知患有肿瘤或已知患有一定程度的癌症。
73.在框220处,基于比较可确定结果为它是针对肿瘤的生物标志物。当参考值从非肿瘤区获得时,当结果的值在统计学上不同于该参考值时,该度量可被确定为生物标志物。非肿瘤区可以是来自相同受试者或来自参考受试者的区域。参考受试者可能已知没有癌症。在其中参考值从归属于肿瘤的区域被确定的实施例中,如果结果并没有在统计学上不同于参考值,则该结果可被确定为生物标志物。
74.b.确定肿瘤分类的方法
75.方法可包括确定肿瘤的分类。该方法可包括确定具有肿瘤区的图像中的生物标志物的值。生物标志物可通过方法200来确定。生物标志物的值可与阈值进行比较。阈值可从具有相同肿瘤分类的参考受试者的图像来确定。肿瘤的分类可基于该比较。例如,当生物标志物的值超过阈值时,肿瘤可被归类为较严重的类别(例如,恶性或正在扩大)。在一些实施例中,肿瘤分类可以是癌症的存在情况或严重程度。较严重的肿瘤分类可导致癌症存在或癌症为严重的分类。癌症的分类的实例包括进行性疾病和稳定性疾病。
76.与肿瘤区相对应的肿瘤的分类可使用所访问的度量和/或结果来确定。该分类可以是与肿瘤区或其他目标区域的特定性质相关联的任何数字或其他特征。例如,“ ”号(或文字“阳性”)可表示肿瘤为恶性或正在扩大。该分类可以是二元的(例如,阳性或阴性),或者可具有更多的分类级别(例如,从1到10或从0到1的标度)。该分类可以是肿瘤为良性、稳定的或正在缩小。
77.可基于确定的分类对从其获得了生物学样品的受试者进行处理。例如,可对具有恶性或正在扩大的分类的肿瘤开始或增加肿瘤治疗(例如,药物、放射、疗法或手术)。可对具有良性或正在缩小的分类的肿瘤结束、减少或不开始肿瘤治疗。在对治疗的应答方面的分类可包括完全应答、部分应答和无应答。对于这些分类而言,可在治疗前以及在治疗后一次或多次获取生物标志物的值。生物标志物的值中的变化(增加或减少)可指示对治疗的应答。
78.基于一个或多个生物标志物以及任选地其他临床信息,可确定进一步的行动。该进一步的行动可包括:开始或结束对肿瘤的治疗,在临床试验中登记或取消登记患有肿瘤的受试者,或者对受试者执行附加的诊断测试。生物标志物的值可与阈值进行比较。阈值可从参考受试者来确定。参考受试者可包括已知的健康个体。在一些实施例中,参考受试者可包括患有已知分期的肿瘤的个体。阈值可以是数字,以建立与参考受试者的统计学差异。例如,阈值可以是远离从健康参考受试者确定的结果的值的一个、两个或三个标准偏差。
79.c.示例性边界盒
80.图3示出了方法200的具体实施的实例。在框302处,手动肿瘤/排除注释被加载。肿瘤注释可以是框202中所识别的肿瘤区。排除注释是为了排除与其他生物学结构相关联的某些区域。这些排除区域可包括折叠组织、伪影、异型增生、坏死等。这些被排除的区域是不能用于分析的区域。
81.在框304处,重叠的肿瘤和排除区域被计算出。病理学家/用户可手动生成/绘制肿瘤或被排除的区域,其可包括重叠的肿瘤或重叠的排除区域。在计算任何进一步的步骤之前,可执行完整性检查,以避免重叠的肿瘤或被排除的区域的错误。
82.在框306出,自动检测的多边形被加载。这些多边形可表示上皮肿瘤或血管(例如,组织中为肿瘤或免疫细胞提供营养的血管)。该多边形可以是方法200中的该多个区域。
83.在框308处,构建了两个哈希表来跟踪多边形与肿瘤或组织之间的空间关系和信息。组织是最大的区域,其中肿瘤和多边形是组织块的子集。
84.在框310处,可生成边界盒。边界盒可以是方法200的框206中的边界区。多边形可使用边界盒被识别为完全在肿瘤之内或与肿瘤相交,类似于方法200的框208和框210。
85.在框312处,多边形的交集和排除可被计算出。多边形本身而不是边界盒可用于确定多边形与肿瘤或与被排除的区域的交集。哈希表可针对将多边形识别为完全在肿瘤之内、与肿瘤相交或在肿瘤之外而被更新。
86.在框314、框316、框318和框320处,可报告不同的结果,包括:目标区域(例如,肿瘤)内的表型的计数/密度;不同表型之间的距离;多边形的计数/密度,以及表型与多边形之间的距离。这些结果可关于它们作为针对肿瘤或癌症的生物标志物的用途进行评估,其可包括方法200的框218和框220。
87.图4示出了图像的被分析区域。红线450表示肿瘤区。蓝线452表示一种类型的多边形。绿线454表示被排除的区域。有待考虑的多边形为肿瘤内但在被排除的区域之外的多边形。阴影部分456示出了肿瘤内且在被排除的区域之外的多边形。针对阴影部分456的度量可被确定为二级分析的一部分。
88.d.示例性生物标志物识别
89.癌症可能通过上调肿瘤细胞上以及肿瘤微环境中的程序性死亡1(pd-1)及其配体、程序性死亡配体1(pd-l1)通路而逃避免疫监视和根除。用针对pd-1或pd-l1的抗体阻断这一通路已经在一些癌症患者中引起了显著的临床应答。
90.对卵巢癌组织的图像中可见的t细胞的数量进行分析。研究的t细胞包括cd3、cd8、gzmb和gzmk。在肿瘤区和基质区域中对这些细胞进行了分析。细胞被确定为在肿瘤区中或在基质区域中(在肿瘤区之外)。用于确定在肿瘤区之内或之外的方法包括图2中所描述的方法(例如,框202至框212)。总共研究了17例卵巢癌组织。在这17例组织中,九例组织包括浸润有免疫细胞的肿瘤,并且八例组织包括排斥免疫细胞的肿瘤。
91.图5a示出了肿瘤组织的示例性多重荧光测定图像。顶部图像示出了其中肿瘤组织排斥免疫细胞的组织。顶部图像示出了指向cd3和gzmb的共定位的箭头。底部图像示出了其中免疫细胞已经浸润肿瘤组织的组织。底部图像示出了指向肿瘤内的某些细胞的箭头。绿色箭头指向cd3细胞、gzmb细胞和pd-1细胞的共定位。黄色箭头指向cd3和gzmb的位置。图5a显示,在排斥性肿瘤中,没有发现pd-1细胞,而发现了cd3和gzmb。图5a还显示,在浸润性组织中,发现了pd-1细胞以及cd3和gzmb。
92.图5b以图表示出了对所有17例组织中的pd-1的分析的结果。x轴示出了肿瘤组织和基质组织,其各自在浸润性肿瘤组织和排斥性肿瘤组织之间进行拆分。使用类似于方法200的方法将细胞分配到这些肿瘤区和基质区域。y轴示出了pd1细胞、gzmb细胞和cd3细胞与gzmb和cd3的比率。对于肿瘤区而言,与对于肿瘤区而言的排斥性肿瘤(p值=0.05)相比,
针对pd-1呈阳性的gzmb t细胞的分数在浸润性肿瘤中显著较高。对于基质区域而言,对于浸润性肿瘤而言的针对pd-1呈阳性的gzmb t细胞的分数略微较高(p值=0.3)。结果证实了浸润性肿瘤中gzmb t细胞的较高的活化状态或增加的数量。图5b显示,基于肿瘤区中pd1细胞、gzmb细胞、cd3细胞相对于gzmb细胞、cd3细胞的数量的生物标志物可用于识别浸润性肿瘤组织或排斥性肿瘤组织。浸润性肿瘤组织可用于评估药物性能。在一些实施例中,浸润性肿瘤组织可能意味着免疫细胞正在试图杀死肿瘤细胞并且药物可能是有效的。图5b还显示,用于识别浸润性肿瘤组织或排斥性肿瘤组织的基质中细胞的相对数量将不如使用肿瘤组织中的细胞那样有效。参考图2中的方法200,细胞的数量可以作为度量,并且结果可以是该细胞与其他细胞的数量的比率。参考值可以是针对排斥性肿瘤组织的比率或针对基质的比率。
93.图6以图表示出了涉及所有17例组织中的gzmb和gzmk的分析的结果。x轴示出了排斥性肿瘤组织和浸润性肿瘤组织。y轴示出了cd8细胞和gzmk细胞或gzmb细胞相对于cd8细胞的数量。左图表示出了gzmb结果。右图表示出了gzmk结果。右图表显示,与浸润性样品相比,cd8细胞、gzmk细胞相对于cd8细胞在排斥性样品中较高。左图表显示,对于cd8细胞、gzmb细胞相对于cd8细胞而言,浸润性样品与排斥性样品之间无显著差异。cd8细胞、gzmk细胞相对于cd8细胞的数量可用作针对浸润性肿瘤组织的生物标志物。
94.ii.细胞表型的密度或计数
95.图7示出了分析生物学样品的图像的方法700。该图像可以是本文所述的任何图像。
96.在框702处,可确定具有第一目标区域的细胞表型的第一计数或第一密度。第一目标区域可以是肿瘤或本文所述的任何目标区域。细胞表型可通过本文所述的任何方法(包括方法200)被确定为在第一目标区域内。
97.在框704处,可确定第二目标区域内的细胞表型的第二计数或第二密度。第二目标区域可在第一目标区域内。第二目标区域可包括基质、上皮肿瘤或癌周。细胞表型可通过本文所述的任何方法(包括方法200)被确定为在第二目标区域内。
98.可确定第三目标区域内的细胞表型的第三计数或第三密度。第三目标区域可在第一目标区域内。第三目标区域可以是基质、上皮肿瘤或癌周,并且可不同于第二目标区域。
99.在框706处,可生成输出。该输出可包括第一计数或第一密度。该输出可进一步包括第二计数或第二密度。该输出还可包括第三计数或第三密度。
100.方法700可进一步包括将第二计数或第二密度与阈值进行比较。基于该比较可确定第一目标区域的分类。例如,如果第二计数或第二密度超过阈值,则第一目标区域的分类可以是存在疾患。例如,该分类可以是肿瘤为恶性并正在生长。
101.方法700可包括将第一计数与第一阈值进行比较或将第二计数与第二阈值进行比较。当第一计数超过第一阈值或第二计数超过第二阈值时,生成的输出可包括第一密度和/或第二密度,而不是计数或作为对计数的补充。
102.图8a示出了针对不同细胞表型的计数或密度的分析。肿瘤802用红线勾勒出轮廓。上皮肿瘤包含区域(804)用蓝线勾勒出轮廓,并且上皮肿瘤排除区域(806)用绿线勾勒出轮廓。基质区域也是肿瘤区内被排除在上皮肿瘤区之外的区域。某一表型(表型a)的细胞以品红色勾勒出轮廓示出(例如,细胞808)。
103.图8b示出了对细胞表型进行计数的结果。图8b显示,肿瘤中存在具有表型a的五个细胞。在这五个细胞中,一个细胞在上皮肿瘤内,并且四个细胞在基质中。计算出了肿瘤、上皮肿瘤和基质的面积。上皮肿瘤和基质的面积为肿瘤内的面积。然后通过以下方式来确定表型a的密度:获取细胞的数量并除以对应的面积。在一些实施例中,可使用被细胞覆盖的面积而不是细胞的计数来计算密度。
104.该结果可如用方法200所公开的那样进行使用。
105.iii.表型距离
106.图9示出了分析生物学样品的图像的方法900。该图像可以是本文所述的任何图像。
107.在框902处,可识别肿瘤中的多个第一细胞。该多个第一细胞中的每个第一细胞可具有第一表型。该多个第一细胞可通过本文所述的任何方法(包括方法200)被确定为在肿瘤中。
108.在框904处,可识别肿瘤中的多个第二细胞。该多个第二细胞中的每个第二细胞可具有第二表型。该多个第二细胞可通过本文所述的任何方法(包括方法200)被确定为在肿瘤中。
109.在框906处,针对该多个细胞中的每个第一细胞,可计算出到该多个第二细胞中的第二细胞的最短距离。该最短距离为该第一细胞与该多个第二细胞中的任意第二细胞之间的距离。该计算可使用最近邻搜索,包括例如k最近邻(knn)、k-d树、制高点树(vantage point trees)和球树。与knn搜索和其他最近邻搜索相比,通过使用欧氏距离来计算多个细胞/表型的两个集合的一种常用距离度量会导致非常复杂的计算。针对距离的强力法将需要大量的计算。该方法可用于验证knn搜索或其他最近邻搜索是否在小子集实例中获得准确的结果。
110.在框908处,可基于计算出的最短距离生成结果。该结果可以是该最短距离的列表。在一些实施例中,可计算出统计值的统计值(例如,平均值、中值、众数、百分位数、标准偏差)。该结果可包括该统计值。该结果可如用方法200所公开的那样进行使用。
111.在一些实施例中,可确定在一定范围具有该最短距离的第一细胞的计数。该结果可包括第一细胞的计数。在实施例中,可确定在若干个不同范围内具有最短距离的第一细胞的计数。该结果可包括这些计数。这些计数可被呈现为直方图。
112.图10示出了对从表型a到表型b的距离的计算。图8示出了单个表型a,a1。存在四个表型b:b1、b2、b3和b4。最短距离为a1与b3之间的距离(以虚线表示)。
113.图11示出了多个表型a和表型b。以红圈表示的三个表型a在肿瘤内。以蓝色x表示的两个表型b在肿瘤内。
114.图12示出了表型a与表型b之间的距离。最短距离以虚线示出。发现最短距离为66.0、173.4和201.4。然后可基于该距离计算出统计值。例如,平均最短距离为147.0,并且标准偏差为71.5。
115.表型a和表型b的排列可能比图11中所示的更为复杂。计算每一个单个距离可能是计算上密集且低效的。k最近邻(knn)搜索用于搜索两个不同表型之间的最近邻。利用图11的配置进行的knn搜索得到与计算该两种表型之间的所有距离相同的距离、平均值和标准偏差。与强力法(例如,计算多个细胞/表型的两个集合之间的欧氏距离)相比,knn搜索在计
算上更有效。
116.图13示出了来自该分析的可能的结果表。图13示出了最短距离的平均值和标准偏差。图13还列出了肿瘤中的具有表型b的一定范围的表型a的数量。在该实例中,一个表型a距一个表型b 66.0微米。这一个表型a与针对在表型b的70微米范围内、80微米范围内、90微米范围内和100微米范围内的表型a的数量的值1相对应。
117.iv.细胞到区域的距离
118.从表型的细胞到区域的距离在临床分析中可能是有用的。图14示出了上皮肿瘤和以蓝色x表示的表型的轮廓。最里面的圆圈1402表示上皮肿瘤。表型具有距上皮肿瘤的不同距离。上皮肿瘤内的表型被认为具有为零的距离。到上皮肿瘤的表型距离可在具有到上皮肿瘤的不同距离分布范围的直方图上进行报告。
119.图15a示出了生物学样品的图像。图15b示出了带有注释肿瘤区的红圈的放大图像。图15c示出了包括表示上皮肿瘤的蓝色多边形和表示表型的细胞的黑圈的图像。红色轮廓表示肿瘤区。
120.图16示出了对肿瘤内的单个上皮肿瘤多边形的选择。少数表型a的细胞以黑圈表示。此处上皮肿瘤被选择为多边形的实例。(类似地,血管和/或成纤维细胞活化蛋白基质也可被选择为多边形。)
121.图17示出了被五条轮廓线包围的单个上皮肿瘤。基于距上皮肿瘤多边形的距离来模拟这五条轮廓线。这些轮廓线与上皮肿瘤多边形间隔开10微米、20微米、30微米、40微米和50微米。特定表型的细胞被示出为红点。图17从轮廓线表明,三个细胞在上皮肿瘤的10微米范围内。所有四个细胞都在上皮肿瘤的40微米范围内。
122.图18示出了带有从表型a的细胞到上皮肿瘤的距离的结果的表格。在计算出从细胞到上皮肿瘤的每段距离后,可确定这些距离的平均距离和标准偏差。
123.图19示出了分析生物学样品的图像的方法1900。该图像可以是本文所述的任何图像。
124.在框1902处,可识别肿瘤中的多个第一细胞。该多个第一细胞中的每个第一细胞可具有第一表型。该多个第一细胞可通过本文所述的任何方法被确定为在肿瘤中。
125.在框1904处,可识别肿瘤中的一个或多个区域。该一个或多个区域中的每个区域可对应于组织块或生物学对象。该一个或多个区域可通过本文所述的任何方法被确定为在肿瘤中。
126.在框1906处,可使用最近邻搜索来计算第一细胞与该一个或多个区域中的每个区域之间的最短距离。最近邻搜索的实例包括k最近邻(knn)、k-d树、制高点树和球树knn搜索可确定具有到区域的最近垂直距离的细胞/表型。在一些实施例中,该距离可以是最近水平距离或跨另一维度的距离。可计算出细胞/表型与区域之间的距离。可对计算出的距离进行分类,以分析从细胞/表型到区域的距离分布。可以类似的方式使用其他最近邻搜索。
127.在框1908处,可基于计算出的最短距离来生成结果。该结果可类似于图16。该结果可包括在一定范围内的细胞/表型的计数。该结果可如用方法200所公开的那样进行使用。
128.knn搜索生成了图16中的结果。knn搜索避免了对模拟研究中所使用的周围距离轮廓掩码的计算。与轮廓掩码的使用相比,knn搜索在获得相同结果方面更快且更有效。
129.v.压缩级别
130.图20示出了分析生物学样品的图像的方法2000。该图像可以是本文所公开的任何图像。
131.在框2002处,可识别肿瘤中的多个区域。该多个区域中的每个区域可对应于组织块或生物学对象。框2002可类似于框1304。
132.在框2004处,可确定针对该多个区域中的每个区域的压缩级别。可从面积与一个或多个特征维度的比率来计算出压缩级别。特征维度可以是区域的宽度、长度或区域长轴的长度或区域短轴的长度。压缩级别还可包括血管腔游离出cd31细胞的程度,该细胞是针对血管壁的生物标志物。
133.在框2006处,可基于压缩级别来生成结果。该结果可包括该压缩级别的列表。在一些实施例中,该结果可包括压缩级别的类别中的区域的计数。压缩级别的不同类别可基于压缩的大小(例如,低、中、高)。该结果可包括区域的面积。该结果可进一步包括压缩级别和/或面积的统计值。该结果可如用方法200所公开的那样进行使用。
134.图21示出了带有不同压缩级别的区域的肿瘤。该肿瘤用红线勾勒出轮廓。区域示出为以绿色填充的多边形。具有低压缩级别的多边形在形状上为圆形。具有高压缩级别的多边形长而细,其具有高的高与宽的纵横比。具有中等压缩级别的多边形不像具有高压缩级别的多边形那样长而细,也不像具有低压缩级别的多边形那样圆。
135.图22示出了具有低压缩级别、中等压缩级别和高压缩级别的三个多边形,每个多边形都用粉色轮廓线标记。多边形2202具有低压缩级别。多边形2204具有中等压缩级别。多边形2206具有高压缩级别。
136.图23示出了针对压缩级别的可能的结果列表。列出了低类别、中等类别和高类别中的每种类别中的多边形数量。也列出了平均压缩级别。也报告了多边形的面积和多边形的总面积。
137.vi.计算机系统
138.上述图像分析方法可由计算机系统执行,其可包括图24中的计算机系统10。在一些实施例中,计算机系统包括单个计算机设备,其中子系统可以是计算机设备的部件。在其他实施例中,计算机系统可以包括多个计算机设备,每一个均是带有内部组件的子系统。计算机系统可以包括台式计算机和膝上型计算机、平板电脑、移动电话、其他移动设备和基于云的系统。
139.图24所示的子系统经由系统总线75互连。示出了附加子系统,诸如打印机74、键盘78、存储设备79、与显示适配器82耦合的监视器76(例如,显示屏,诸如lcd或led)等。耦合至i/o控制器71的外围装置和输入/输出(i/o)设备可通过本领域已知的任何数量的装置,诸如输入/输出(i/o)端口77(例如,usb)连接至计算机系统。例如,i/o端口77或外部接口81(例如,以太网、wi-fi等)可用于将计算机系统10连接至广域网,诸如互联网、鼠标输入器件或扫描仪。通过系统总线75的互连允许中央处理器73与每一个子系统通信并控制对来自系统存储器72或存储器件79(例如,固定磁盘,诸如硬盘驱动器,或光盘)的多个指令的执行,以及子系统之间的信息交换。所述系统存储器72和/或存储设备79可以包含计算机可读介质。另一子系统是数据收集设备85,诸如相机、显微镜、麦克风、加速度计等。本文所述的ihc数据可通过数据收集设备85来获取。在一些实施例中,可将图像数据传递到存储设备79上或存储在系统存储器72中。本文提到的任何数据均可以从一个部件输出至另一部件,并可
以输出给用户。
140.计算机系统可包括多个相同的部件或子系统,它们例如通过外部接口81、通过内部接口或经由可移动存储设备连接在一起,该可移动存储设备可从一个部件连接和移动至另一个部件。在某些实施例中,计算机系统、子系统或设备可以通过网络来通信。在这种情况下,一台计算机可以视为客户端,另一台计算机可以视为服务器,其中每一台计算机均可以视为同一计算机系统的一部分。客户端和服务器可以各自包括多个系统、子系统或组件。
141.实施例的各方面可使用硬件电路(例如,专用集成电路或现场可编程门阵列)和/或使用具有一般可编程处理器的计算机软件,以控制逻辑的形式,以模块化或集成方式来实施。如本文所用,处理器可包括单核处理器、在同一集成芯片上的多核处理器、或在单电路板上或联网的多个处理单元,以及专用硬件。基于本文提供的公开内容和启示,本领域普通技术人员将知道并理解使用硬件以及硬件和软件的组合实现本发明实施例的其他方式和/或方法。
142.可使用任何合适的计算机语言,诸如,例如,java、c、c 、c#、objective-c、swift,或脚本语言,诸如perl或python,使用例如传统技术或面向对象技术,将本技术中描述的任何软件组件或功能实现为由处理器执行的软件代码。软件代码可以作为一系列指令或命令存储在计算机可读介质上,以进行存储和/或传输。合适的非暂时性计算机可读介质可包括随机存取存储器(ram)、只读存储器(rom)、磁性介质诸如硬盘驱动器或软盘、或者光学介质诸如光盘(cd)或dvd(数字通用光盘)或蓝光光盘、闪存等。所述计算机可读介质可以是这种存储或传输设备的任何组合。
143.也可使用载波信号对此类程序进行编码和传输,该载波信号适合于经由符合包括互联网在内的各种协议的有线网络、光学网络和/或无线网络进行传输。如此,计算机可读介质可以使用经这种程序编码的数据信号来创建。以程序代码编码的计算机可读介质可与兼容设备一起打包,或者与其他设备分开提供(例如通过互联网下载)。任何此类计算机可读介质可以驻留在单个计算机产品(例如,硬盘驱动器、cd或整个计算机系统)上或内部,并且可以存在于系统或网络内的不同计算机产品上或内部。计算机系统可以包括监测器、打印机或其他合适的显示器,用于向用户提供本文提到的任何结果。
144.本文所述的任何方法都可用包括一个或多个处理器的计算机系统完全或部分地执行,该计算机系统可被配置成执行步骤。因此,实施方案可以针对被配置成执行本文描述的任何方法的步骤的计算机系统,可能具有执行相应步骤或相应步骤组的不同组件。尽管以编号的步骤呈现,但是可以同时或在不同时间或以不同顺序执行本文所述方法的步骤。此外,部分步骤可以与其他方法中的部分步骤一起使用。另外,全部或部分步骤可以任选。另外,任何方法的任何步骤都可以用模块、单元、电路或用于执行这些步骤的系统的其他装置来执行。这些步骤可作为图像分析代码存储在系统存储器72中或存储在存储设备79上。
145.在不脱离本发明实施例的精神和范围的情况下,可以以任何合适的方式组合特定实施例的具体细节。然而,本发明的其他实施例可以针对与每一个单独方面有关的特定实施例,或者这些单独方面的特定组合。
146.为了说明和描述的目的,已经呈现了对本公开的示例性实施例的以上描述。并不旨在穷举本公开或将本公开限制为所描述的精确形式,并且根据以上教导,许多修改和变化是可能的。
147.除非特别指出是相反情况,否则对“一个”、“一种”或“该”的陈述旨在表示“一个或多个”。除非特别指出是相反情况,否则“或”的使用旨在表示“包含或”,而不是“排除或”。提及“第一”部件并不一定要求提供第二部件。此外,除非明确说明,否则对“第一”或“第二”组件的引用并不是将所引用的组件限于特定位置。术语“基于”旨在表示“至少部分基于”。
148.本文提及的所有专利、专利申请、出版物和说明书全文出于所有目的以引用方式并入本文。没有一项被认为是现有技术。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。