1.本发明涉及染色体区域分割技术领域,尤其涉及一种基于深度学习卷积神经网络的人类染色体图像摆正方法。
背景技术:
2.由于染色体是基因的载体,是人类遗传信息的携带者,因此,染色体中数量或结构异常,都可能带来流产、死胎、畸形、智力障碍、生长发育异常、肿瘤等疾病。因此,染色体的异常检测对于临床疾病诊断、预防染色体缺陷造成生理缺陷的患儿出生等具有临床意义。
3.染色体着丝粒是指中期染色体的两条姐妹染色单体的连接处,位于染色体的主缢痕处,着丝粒将两条染色单体分为短臂(p)和长臂(q),染色体分割分类后,需要对染色体进行摆正和同源染色体着丝粒对齐操作,按照国际标准要求,一个分裂相中的所有染色体不仅要按编号排列,且必须按短臂在上,长臂在下的方向放置,同时医生们更希望两条同源同类的染色体按照着丝粒靠近的原则摆放。因此,定位姐妹染色单体长短臂及着丝粒所在位置对于摆正染色体和计算长短臂比值至关重要,此外,着丝粒位置还可以为某些染色体异常诊断提供辅助信息。但由于染色体普遍存在弯曲、重叠、交叉粘连、边缘不平滑等现象,尤其是姐妹染色单体分叉,进一步使得着丝粒识别困难,也为染色体摆正带来很高的技术难度。
4.传统根据特征提取算法识别染色体着丝粒位置,多受先验知识和染色体灰度值、阈值等信息影响,对于复杂情况如背景噪声干扰造成着丝粒位置不是最细处、染色体着丝粒位置被遮挡、姐妹染色单体分叉难以提取骨架线、染色体弯曲法线方向波动大等不确定因素,都会影响最终的着丝粒定位效果,因此传统算法难以做到复杂情况下的适应性。
技术实现要素:
5.本发明的目的在于提供一种基于深度学习卷积神经网络的人类染色体图像摆正方法,以解决传统复杂染色体区域分割方法对着丝粒定位效果有待提升,难以做到复杂情况下的适应性的问题。
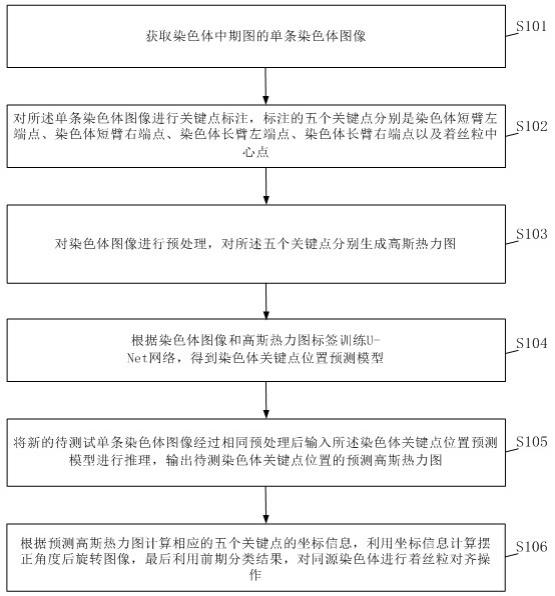
6.本发明提供一种基于深度学习卷积神经网络的人类染色体图像摆正方法,包括:获取染色体中期图的单条染色体图像;对所述单条染色体图像进行关键点标注,标注的五个关键点分别是染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点以及着丝粒中心点;对染色体图像进行预处理,对所述五个关键点分别生成高斯热力图;根据染色体图像和高斯热力图标签训练u-net网络,得到染色体关键点位置预测模型;将新的待测试单条染色体图像经过相同预处理后输入所述染色体关键点位置预测模型进行推理,输出待测染色体关键点位置的预测高斯热力图;根据预测高斯热力图计算相应的五个关键点的坐标信息,利用坐标信息计算摆正
角度后旋转图像,最后利用前期分类结果,对同源染色体进行着丝粒对齐操作。
7.进一步地,对所述单条染色体图像进行关键点标注,标注的五个关键点分别是染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点以及着丝粒中心点之后,还包括:对标注后的数据进行审核,排除脏数据和错误标注。
8.进一步地,对染色体图像进行预处理,对所述五个关键点分别生成高斯热力图包括:对标注的五个关键点生成高斯热力图,每个关键点一张高斯热力图,高斯热力图半径为6,中心点像素为1,周围像素值中心向外逐渐变小至0;将染色体图像由输入的gray格式或者bgr格式转为rgb格式,根据染色体中期图的分割结果,去除图像周边与其交叉粘连的其他染色体和背景杂质干扰;将高斯热力图尺寸和rgb格式图像统一到固定尺寸128*128,对统一尺寸后的rgb格式图像进行归一化,归一化使数据服从高斯分布,归一化采用图像减去其像素均值除以方差。
9.进一步地,根据染色体图像和高斯热力图标签训练u-net网络,得到染色体关键点位置预测模型,包括:网络预测输出五张预测的高斯热力图,计算五张高斯热力图的最大激活值位置,分别得到染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点以及着丝粒中心点的具体坐标;计算五个关键点的预测位置相对于真实标注位置的距离误差,距离误差转换成预测准确率;计算预测的高斯热力图与真实的高斯热力图的损失,利用损失进行梯度反向传播,更新u-net网络权重,u-net网络再次前向推理,计算新的损失,依次反复,直到在验证集中五个关键点的预测准确率达到最高值不再上升时,保存u-net网络此刻的权重,得到最佳权重的染色体关键点位置预测模型。
10.进一步地,计算预测的高斯热力图与真实的高斯热力图的损失,包括:使用损失函数jointsmseloss计算预测高斯热力图与真实高斯热力图的损失,按通道计算均方误差损失函数mseloss,再求加权平均值;oss,再求加权平均值;式中,代表第i张预测输出的高斯热力图的坐标,代表第i张真实的高斯热力图的坐标,n为正整数,代表每张染色体的像素个数,mselossi是单张高斯热力图的均方误差,是第i张高斯热力图的分配权重,在该网络中设置五个关键点的权重相等,都为1,m为正整数,代表每条染色体的高斯热力图张数,每条染色体都有5张高斯热力图,因此m
等于5。
11.进一步地,计算五个关键点的预测位置相对于真实标注位置的预测准确率,包括:使用拉格朗日距离公式计算预测的高斯热力图与真实的高斯热力图之间的距离;式中,下标p_i代表第i个预测点,t_i代表第i个真实标签点,表示第i个预测点的x坐标,第i个预测点的y坐标,表示第i个z真实点的x坐标,表示第i个真实点的y坐标;w代表图像宽度,h代表图像高度,m为正整数,代表预测点的个数;根据五个关键点的预测位置与真实标注位置的拉格朗日距离,确定预测准确率。
12.进一步地,根据五个关键点的预测位置与真实标注位置的拉格朗日距离,确定预测准确率,包括:判断五个关键点的预测位置与真实标注位置的拉格朗日距离是否小于0.5;如果某个关键点的预测位置与真实标注位置的拉格朗日距离小于0.5,将所述关键点记为正确预测的关键点,反之记为预测失败;根据正确的关键点的数量与关键点的总数量的比值,得到预测准确率。
13.进一步地,根据预测高斯热力图计算相应的五个关键点的坐标信息,利用坐标信息计算摆正角度后旋转图像,最后利用前期分类结果,对同源染色体进行着丝粒对齐操作,包括:计算五张预测高斯热力图的最大激活值位置,分别得到染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点以及着丝粒的预测位置坐标;利用染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点的预测位置坐标,计算预测染色体摆正所需的旋转角度,计算旋转矩阵;根据所述旋转矩阵旋转所述单条染色体图像,计算旋转后的关键点坐标,实现染色体长臂在上短臂在下;根据旋转后的着丝粒的位置,和前期分类结果,实现对同源染色体的着丝粒对齐操作。
14.本发明的有益效果如下:本发明提供的一种基于深度学习卷积神经网络的人类染色体图像摆正方法,通过获取染色体中期图的单条染色体图像,对单条染色体图像进行关键点标注,标注的五个关键点分别是染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点以及着丝粒中心点,对染色体图像进行预处理,对五个关键点分别生成高斯热力图,根据染色体图像和高斯热力图标签训练u-net网络,得到染色体关键点位置预测模型,将新的待测试单条染色体图像经过相同预处理后输入染色体关键点位置预测模型进行推理,输出待测染色体关键点位置的预测高斯热力图,根据预测高斯热力图计算相应的五个关键点的坐标信息,利用坐标信息计算摆正角度后旋转图像,最后利用前期分类结果,对同源染色体进行着丝粒对齐操作,实现染色体长臂在上短臂在下的看图习惯和
报告要求,并通过着丝粒位置信息,实现同源染色体的着丝粒对齐操作,应用于分割后的单条染色体摆正和着丝粒对齐,解决了传统复杂染色体区域分割方法对着丝粒定位效果有待提升,难以做到复杂情况下的适应性的问题。
附图说明
15.为了更清楚地说明本发明的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,对于本领域普通技术人员而言,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。
16.图1是本发明一种基于深度学习卷积神经网络的人类染色体图像摆正方法的流程图;图2是着丝粒的相对位置发生变化示意图;图3是单条染色体关键点标注示意图。
具体实施方式
17.为使本发明的目的、技术方案和优点更加清楚,下面将结合本发明具体实施例及相应的附图对本发明技术方案进行清楚、完整地描述。显然,所描述的实施例仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。以下结合附图,详细说明本发明各实施例提供的技术方案。
18.请参阅图1,本发明提供一种基于深度学习卷积神经网络的人类染色体图像摆正方法,包括:s101,获取染色体中期图的单条染色体图像。
19.人类正常染色体核型由23组染色体组成,其中包含22组常染色体和一组性染色体,每组染色体可根据着丝粒在染色体中的不同位置将23组中期染色体的为中央着丝粒染色体、亚中央着丝粒染色体、近端着丝粒染色体。染色体数量和结构的改变,都可能带来不同的影响,而染色体结构异常最常见的表现特征就是着丝粒的相对位置发生变化,如图2。例如罗伯逊易位就是最常见的着丝粒异常表现之一,由于染色体发生罗伯逊易位,导致染色体着丝粒融合,着丝粒位置由近端变成远端。
20.传统着丝粒定位算法受限约束性条件较多,造成传统算法有一定的缺陷,难以兼容复杂情况。因此,本发明提出一种基于深度学习卷积神经网络的人类染色体图像摆正方法,用来解决各种简单或复杂情况下,染色体着丝粒、长短臂定位困难问题,从而实现染色体着丝粒位置对齐,染色体极性摆正,还可对部分染色体提供辅助信息,满足医生对核型分析习惯、和报告显示的需求。
21.s102,对所述单条染色体图像进行关键点标注,标注的五个关键点分别是染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点以及着丝粒中心点。
22.具体地,单条染色体关键点标注示意图如图3所示,此外,对标注后的数据进行审核,排除脏数据和错误标注,保证图像的质量和标注的正确性。
23.s103,对染色体图像进行预处理,对所述五个关键点分别生成高斯热力图。
24.具体地,对标注的五个关键点生成高斯热力图,每个关键点一张高斯热力图,高斯热力图半径为6,中心点像素为1,周围像素值中心向外逐渐变小至0;将染色体图像由输入的gray格式或者bgr格式转为rgb格式,根据染色体中期图的分割结果,去除图像周边与其交叉粘连的其他染色体和背景杂质干扰;将高斯热力图尺寸和rgb格式图像统一到固定尺寸128*128,对统一尺寸后的rgb格式图像进行归一化,归一化使数据服从高斯分布,归一化采用图像减去其像素均值除以方差,归一化使数据服从高斯分布,以降低不同医院、不同来源、不同条带水平的染色体图像带来的差距。
25.s104,根据染色体图像和高斯热力图标签训练u-net网络,得到染色体关键点位置预测模型。
26.具体地,网络预测输出五张预测的高斯热力图,计算五张高斯热力图的最大激活值位置,分别得到染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点以及着丝粒中心点的具体坐标;计算五个关键点的预测位置相对于真实标注位置的距离误差,距离误差转换成预测准确率;计算预测的高斯热力图与真实的高斯热力图的损失,利用损失进行梯度反向传播,更新u-net网络权重,u-net网络再次前向推理,计算新的损失,依次反复,直到在验证集中五个关键点的预测准确率达到最高值不再上升时,保存u-net网络此刻的权重,得到最佳权重的染色体关键点位置预测模型。u-net网络是一种深度学习卷积神经网络。
27.具体地,计算预测的高斯热力图与真实的高斯热力图的损失,包括:使用损失函数jointsmseloss计算预测高斯热力图与真实高斯热力图的损失,按通道计算均方误差损失函数mseloss,再求加权平均值;函数mseloss,再求加权平均值;式中,代表第i张预测输出的高斯热力图的坐标,代表第i张真实的高斯热力图的坐标,n为正整数,代表每张染色体的像素个数,mselossi是单张高斯热力图的均方误差,是第i张高斯热力图的分配权重,在该网络中设置五个关键点的权重相等,都为1,m为正整数,代表每条染色体的高斯热力图张数,每条染色体都有5张高斯热力图,因此m等于5。
28.具体地,计算五个关键点的预测位置相对于真实标注位置的预测准确率,包括:使用拉格朗日距离公式计算预测的高斯热力图与真实的高斯热力图之间的距离;式中,下标p_i代表第i个预测点,t_i代表第i个真实标签点,表示第i个预测点的x
坐标,第i个预测点的y坐标,表示第i个z真实点的x坐标,表示第i个真实点的y坐标;w代表图像宽度,h代表图像高度,m为正整数,代表预测点的个数。
29.根据五个关键点的预测位置与真实标注位置的拉格朗日距离,确定预测准确率。判断五个关键点的预测位置与真实标注位置的拉格朗日距离是否小于0.5;如果某个关键点的预测位置与真实标注位置的拉格朗日距离小于0.5,将所述关键点记为正确预测的关键点,反之记为预测失败;根据正确的关键点的数量与关键点的总数量的比值,得到预测准确率例如,五个预测的高斯热力图中的关键点,其中有四个关键点与真实标注位置之间的拉格朗日距离小于0.5,则该染色体点高斯热力图准确率为0.8。
30.s105,将新的待测试单条染色体图像经过相同预处理后输入所述染色体关键点位置预测模型进行推理,输出待测染色体关键点位置的预测高斯热力图。
31.具体地,只有在u-net网络训练阶段,需要标注五个关键点、生成关键点的高斯热力图,测试阶段只需要对图像进行处理,不需要五个关键点、生成关键点的高斯热力图。
32.s106,根据预测高斯热力图计算相应的五个关键点的坐标信息,利用坐标信息计算摆正角度后旋转图像,最后利用前期分类结果,对同源染色体进行着丝粒对齐操作。
33.具体地,计算五张预测高斯热力图的最大激活值位置,分别得到染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点以及着丝粒的预测位置坐标;利用染色体短臂左端点、染色体短臂右端点、染色体长臂左端点、染色体长臂右端点的预测位置坐标,计算预测染色体摆正所需的旋转角度,计算旋转矩阵;根据所述旋转矩阵旋转所述单条染色体图像,计算旋转后的关键点坐标,实现染色体长臂在上短臂在下;根据旋转后的着丝粒的位置,和前期分类结果,实现对同源染色体的着丝粒对齐操作。此外,还可以通过计算着丝粒在染色体的位置,与同源染色体着丝粒位置进行比较, 如果差距较大,则考虑是否有染色体异常。由以上实施例可知,本发明提供的一种基于深度学习卷积神经网络的人类染色体图像摆正方法,可实现染色体长臂在上短臂在下的看图习惯和报告要求,并通过着丝粒位置信息,实现同源染色体的着丝粒对齐操作,应用于分割后的单条染色体摆正和着丝粒对齐,解决了传统复杂染色体区域分割方法对着丝粒定位效果有待提升,难以做到复杂情况下的适应性的问题。
34.本发明实施例还提供一种存储介质,本发明实施例还提供一种存储介质,所述存储介质中存储有计算机程序,所述计算机程序被处理器执行时实现本发明提供的基于深度学习卷积神经网络的人类染色体图像摆正方法各实施例中的部分或全部步骤。所述的存储介质可为磁碟、光盘、只读存储记忆体(英文:read-onlymemory,简称:rom)或随机存储记忆体(英文:randomaccessmemory,简称:ram)等。
35.本领域的技术人员可以清楚地了解到本发明实施例中的技术可借助软件加必需的通用硬件平台的方式来实现。基于这样的理解,本发明实施例中的技术方案本质上或者说对现有技术做出贡献的部分可以以软件产品的形式体现出来,该计算机软件产品可以存储在存储介质中,如rom/ram、磁碟、光盘等,包括若干指令用以使得一台计算机设备(可以是个人计算机,服务器,或者网络设备等)执行本发明各个实施例或者实施例的某些部分所
述的方法。
36.以上所述的本发明实施方式并不构成对本发明保护范围的限定。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。