单细胞rna测序数据降维方法、设备及可读存储介质
技术领域
1.本发明涉及深度学习技术领域,尤其涉及一种单细胞rna测序数据降维方法、设备及可读存储介质。
背景技术:
2.复杂的生物组织和生命体是由形态各异、功能各异的细胞群组成。单细胞rna测序(scrna-seq)技术是对每一个细胞的rna进行测序,得到所有基因在该细胞的表达量。与传统的批量测序不同,它具备分析单个细胞的生物学状态的能力,被广泛应用于肿瘤生物学、胚胎发育学、器官形成等诸多生物学领域。在scrna-seq数据中,细胞的每一个基因的表达量都可视为该细胞的一个特征,从中挖掘生物信息的关键步骤是将高维且复杂的scrna-seq数据降维到二维以达到可视化的效果,直观地观察到细胞群体的分布。
3.一个scrna-seq数据一般含有几万甚至几十万个细胞,每个细胞含有几万个基因表达特征,目前的降维方法不能从如此庞大且高维的数据中充分挖掘出关键信息。传统降维方法都有各自的局限性,比如pca等线性方法不能在二维或三维空间准确有效捕捉到原始高维数据的特征。近年随着深度学习的发展,一些基于深度学习的聚类方法被提出来,但是面对大量、高维且携带大量噪声的scrna-seq数据,降维效果仍然不佳。同时目前的降维方法缺少对细胞之间结构信息的挖掘,而对于scrna-seq数据来说,细胞之间的结构信息是不能忽视的,生物状态相似的细胞中包含的结构信息蕴藏着大量的生物信息。
4.因此需要一种能够在高维、大量且携带大量噪声的的scrna-seq数据中挖掘细胞之间结构信息的方法,去胜任降维任务。
技术实现要素:
5.为解决上述技术问题,本发明提供了一种单细胞rna测序数据降维方法、设备及可读存储介质。
6.第一方面,本发明提供一种单细胞rna测序数据降维方法,所述单细胞rna测序数据降维方法包括:
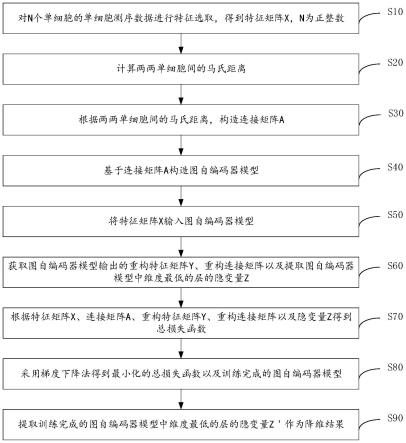
7.对n个单细胞的单细胞测序数据进行特征选取,得到特征矩阵x,n为正整数;
8.计算两两单细胞间的马氏距离;
9.根据两两单细胞间的马氏距离,构造连接矩阵a;
10.基于连接矩阵a构造图自编码器模型;
11.将特征矩阵x输入图自编码器模型;
12.获取图自编码器模型输出的重构特征矩阵y、重构连接矩阵以及提取图自编码器模型中维度最低的层的隐变量z;
13.根据特征矩阵x、连接矩阵a、重构特征矩阵y、重构连接矩阵以及隐变量z得到总损失函数;
14.采用梯度下降法得到最小化的总损失函数以及训练完成的图自编码器模型;
15.提取训练完成的图自编码器模型中维度最低的层的隐变量z'作为降维结果。
16.可选的,所述对n个单细胞的单细胞测序数据进行特征选取,得到特征矩阵x的步骤包括:
17.从n个单细胞的单细胞测序数据中选取满足预设条件的m个基因为特征,得到特征矩阵x,n为正整数,其中,预设条件为:
18.dg>exp[-(m
g-b)] 0.02
[0019]
其中,mg={log2x
ig
|x
ig
>0},i是单位1,x
ig
是细胞i的第g个基因表达量,b为通过二分法得到的与m对应的超参数。
[0020]
可选的,所述根据两两单细胞间的马氏距离,构造连接矩阵a的步骤包括:
[0021]
以每个单细胞为节点,对于细胞i,选取与细胞i的马氏距离最小的前t个细胞作为细胞i的近邻点进行构图,得到连接图;
[0022]
基于连接图,构造连接矩阵a,其中,与细胞j若为细胞i的近邻点,则连接矩阵a中第i行第j列的元素a
ij
为1,否则为0。
[0023]
可选的,图自编码器模型的传播公式为:
[0024][0025]
其中,in是n阶单位矩阵,w
(h-1)为第(h-1)层可训练参数,relu(
·
)为非线性激活函数。
[0026]
可选的,所述根据特征矩阵x、连接矩阵a、重构特征矩阵y、重构连接矩阵以及隐变量z得到总损失函数的步骤包括:
[0027]
根据特征矩阵x以及重构特征矩阵y得到内容重构损失函数l1;
[0028]
根据连接矩阵a以及重构连接矩阵得到图结构重构损失函数l2;
[0029]
根据连接矩阵a以及隐变量z得到低维图结构重构损失函数l3;
[0030]
根据总损失函数计算公式,得到总损失函数loss,总损失函数计算公式为:
[0031]
loss=l1 l2 λ
·
l3[0032]
其中,λ为预设值。
[0033]
可选的,内容重构损失函数l1为:
[0034][0035]
其中,x
ij
为特征矩阵x中第i行第j列的元素,y
ij
为重构特征矩阵y中第i行第j列的元素。
[0036]
可选的,图结构重构损失函数l2为:
[0037][0038]
其中,sigmoid(
·
)为非线性激活函数,t为矩阵的转置操作,a
ij
、分别是a、中第i行第j列的元素。
[0039]
可选的,低维图结构重构损失函数l3为:
[0040][0041]
其中,h=sigmoid(z
t
z),sigmoid(
·
)为非线性激活函数,t为矩阵的转置操作,a
ij
是a中第i行第j列的元素,h
ij
是h中第i行第j列的元素。
[0042]
第二方面,本发明还提供一种单细胞rna测序数据降维设备,所述单细胞rna测序数据降维设备包括处理器、存储器、以及存储在所述存储器上并可被所述处理器执行的单细胞rna测序数据降维程序,其中所述单细胞rna测序数据降维程序被所述处理器执行时,实现如上所述的单细胞rna测序数据降维方法的步骤。
[0043]
第三方面,本发明还提供一种可读存储介质,所述可读存储介质上存储有单细胞rna测序数据降维程序,其中所述单细胞rna测序数据降维程序被处理器执行时,实现如上所述的单细胞rna测序数据降维方法的步骤。
[0044]
本发明中,对n个单细胞的单细胞测序数据进行特征选取,得到特征矩阵x,n为正整数;计算两两单细胞间的马氏距离;根据两两单细胞间的马氏距离,构造连接矩阵a;基于连接矩阵a构造图自编码器模型;将特征矩阵x输入图自编码器模型;获取图自编码器模型输出的重构特征矩阵y、重构连接矩阵以及提取图自编码器模型中维度最低的层的隐变量z;根据特征矩阵x、连接矩阵a、重构特征矩阵y、重构连接矩阵以及隐变量z得到总损失函数;采用梯度下降法得到最小化的总损失函数以及训练完成的图自编码器模型;提取训练完成的图自编码器模型中维度最低的层的隐变量z'作为降维结果。通过本发明,通过马氏距离衡量细胞间的相似性,去除了不同基因表达水平的量纲的影响,排除了基因之间的相互性干扰;图自编码器模型利用到邻近单细胞的信息,使得在庞大、复杂且高维的数据上展开降维任务时保持了细胞之间的结构信息。
附图说明
[0045]
图1为本发明实施例方案中涉及的单细胞rna测序数据降维设备的硬件结构示意图;
[0046]
图2为本发明单细胞rna测序数据降维方法一实施例的流程示意图。
[0047]
本发明目的的实现、功能特点及优点将结合实施例,参照附图做进一步说明。
具体实施方式
[0048]
应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
[0049]
第一方面,本发明实施例提供一种单细胞rna测序数据降维设备,该单细胞rna测序数据降维设备可以是个人计算机(personal computer,pc)、笔记本电脑、服务器等具有数据处理功能的设备。
[0050]
参照图1,图1为本发明实施例方案中涉及的单细胞rna测序数据降维设备的硬件结构示意图。本发明实施例中,单细胞rna测序数据降维设备可以包括处理器1001(例如中央处理器central processing unit,cpu),通信总线1002,用户接口1003,网络接口1004,存储器1005。其中,通信总线1002用于实现这些组件之间的连接通信;用户接口1003可以包括显示屏(display)、输入单元比如键盘(keyboard);网络接口1004可选的可以包括标准的
有线接口、无线接口(如无线保真wireless-fidelity,wi-fi接口);存储器1005可以是高速随机存取存储器(random access memory,ram),也可以是稳定的存储器(non-volatile memory),例如磁盘存储器,存储器1005可选的还可以是独立于前述处理器1001的存储装置。本领域技术人员可以理解,图1中示出的硬件结构并不构成对本发明的限定,可以包括比图示更多或更少的部件,或者组合某些部件,或者不同的部件布置。
[0051]
继续参照图1,图1中作为一种计算机存储介质的存储器1005中可以包括操作系统、网络通信模块、用户接口模块以及单细胞rna测序数据降维程序。其中,处理器1001可以调用存储器1005中存储的单细胞rna测序数据降维程序,并执行本发明实施例提供的单细胞rna测序数据降维方法。
[0052]
第二方面,本发明实施例提供了一种单细胞rna测序数据降维方法。
[0053]
一实施例中,参照图2,图2为本发明单细胞rna测序数据降维方法一实施例的流程示意图。如图2所示,单细胞rna测序数据降维方法包括:
[0054]
步骤s10,对n个单细胞的单细胞测序数据进行特征选取,得到特征矩阵x,n为正整数;
[0055]
本实施例中,从单细胞rna测序数据集中选择n个单细胞的单细胞测序数据,然后对n个单细胞的单细胞测序数据进行特征选取,得到特征矩阵x,n为正整数。
[0056]
进一步地,一实施例中,步骤s10包括:
[0057]
从n个单细胞的单细胞测序数据中选取满足预设条件的m个基因为特征,得到特征矩阵x,n为正整数,其中,预设条件为:
[0058]dg
>exp[-(m
g-b)] 0.02
[0059]
其中,mg={log2x
ig
|x
ig
>0},i是单位1,x
ig
是细胞i的第g个基因表达量,b为通过二分法得到的与m对应的超参数。
[0060]
本实施例中,从单细胞rna测序数据集中选择n个单细胞测序数据。预定取m=2000个基因作为细胞的特征,二分法查找出与m=2000对应的超参数b,使得dg满足:
[0061]dg
>exp[-(m
g-b)] 0.02
[0062]
其中,mg={log2x
ig
|x
ig
>0},i是单位1,x
ig
是细胞i的第g个基因表达量,b为通过二分法得到的与m对应的超参数。
[0063]
步骤s20,计算两两单细胞间的马氏距离;
[0064]
本实施例中,为消除基因之间表达值的差异性,通过马氏距离计算公式,计算两两单细胞间的马氏距离。
[0065]
步骤s30,根据两两单细胞间的马氏距离,构造连接矩阵a;
[0066]
本实施例中,根据两两单细胞间的马氏距离即可确定两两单细胞间相似度,从而基于两两单细胞间的相似度构造连接矩阵a。
[0067]
进一步地,一实施例中,步骤s30包括:
[0068]
以每个单细胞为节点,对于细胞i,选取与细胞i的马氏距离最小的前t个细胞作为细胞i的近邻点进行构图,得到连接图;基于连接图,构造连接矩阵a,其中,与细胞j若为细胞i的近邻点,则连接矩阵a中第i行第j列的元素a
ij
为1,否则为0。
[0069]
本实施例中,t选为(0.01
×
n)和20中的最大值。
[0070]
步骤s40,基于连接矩阵a构造图自编码器模型;
[0071]
本实施例中,基于连接矩阵a构造图自编码器模型,具体的,图自编码器模型的传播公式为:
[0072][0073]
其中,in是n阶单位矩阵,w
(h-1)
为第(h-1)层可训练参数,relu(
·
)为非线性激活函数。作为示意,图自编码器各层的维度为2000-512-256-128-68-10-2-10-68-128-512-2000,维度为2的层的隐变量z将在图自编码器模型训练完毕后提取出来作为降维的结果,最后一层作为重构矩阵y。
[0074]
步骤s50,将特征矩阵x输入图自编码器模型;
[0075]
步骤s60,获取图自编码器模型输出的重构特征矩阵y、重构连接矩阵以及提取图自编码器模型中维度最低的层的隐变量z;
[0076]
步骤s70,根据特征矩阵x、连接矩阵a、重构特征矩阵y、重构连接矩阵以及隐变量z得到总损失函数;
[0077]
本实施例中,根据特征矩阵x以及重构特征矩阵y得到内容重构损失函数l1;
[0078]
根据连接矩阵a以及重构连接矩阵得到图结构重构损失函数l2;
[0079]
根据连接矩阵a以及隐变量z得到低维图结构重构损失函数l3;
[0080]
根据总损失函数计算公式,得到总损失函数loss,总损失函数计算公式为:
[0081]
loss=l1 l2 λ
·
l3[0082]
其中,λ为预设值。
[0083]
进一步地,一实施例中,内容重构损失函数l1为:
[0084][0085]
其中,x
ij
为特征矩阵x中第i行第j列的元素,y
ij
为重构特征矩阵y中第i行第j列的元素。
[0086]
进一步地,一实施例中,图结构重构损失函数l2为:
[0087][0088]
其中,sigmoid(
·
)为非线性激活函数,t为矩阵的转置操作,a
ij
、分别是a、中第i行第j列的元素。
[0089]
进一步地,一实施例中,低维图结构重构损失函数l3为:
[0090][0091]
其中,h=sigmoid(z
t
z),sigmoid(
·
)为非线性激活函数,t为矩阵的转置操作,a
ij
是a中第i行第j列的元素,h
ij
是h中第i行第j列的元素。
[0092]
步骤s80,采用梯度下降法得到最小化的总损失函数以及训练完成的图自编码器
模型;
[0093]
本实施例中,采用梯度下降法调整图自编码器模型的参数,从而得到最小化的总损失函数以及最小化的总损失函数对应的图自编码器模型参数。
[0094]
步骤s90,提取训练完成的图自编码器模型中维度最低的层的隐变量z'作为降维结果。
[0095]
本实施例中,根据步骤s80得到训练完成的图自编码器模型后,即可提取训练完成的图自编码器模型中维度最低的层的隐变量z'作为降维结果。
[0096]
本实施例中,对n个单细胞的单细胞测序数据进行特征选取,得到特征矩阵x,n为正整数;计算两两单细胞间的马氏距离;根据两两单细胞间的马氏距离,构造连接矩阵a;基于连接矩阵a构造图自编码器模型;将特征矩阵x输入图自编码器模型;获取图自编码器模型输出的重构特征矩阵y、重构连接矩阵以及提取图自编码器模型中维度最低的层的隐变量z;根据特征矩阵x、连接矩阵a、重构特征矩阵y、重构连接矩阵以及隐变量z得到总损失函数;采用梯度下降法得到最小化的总损失函数以及训练完成的图自编码器模型;提取训练完成的图自编码器模型中维度最低的层的隐变量z'作为降维结果。通过本实施例,通过马氏距离衡量细胞间的相似性,去除了不同基因表达水平的量纲的影响,排除了基因之间的相互性干扰;图自编码器模型利用到邻近单细胞的信息,使得在庞大、复杂且高维的数据上展开降维任务时保持了细胞之间的结构信息。
[0097]
第三方面,本发明实施例还提供一种可读存储介质。
[0098]
本发明可读存储介质上存储有单细胞rna测序数据降维程序,其中所述单细胞rna测序数据降维程序被处理器执行时,实现如上述的单细胞rna测序数据降维方法的步骤。
[0099]
其中,单细胞rna测序数据降维程序被执行时所实现的方法可参照本发明单细胞rna测序数据降维方法的各个实施例,此处不再赘述。
[0100]
需要说明的是,在本文中,术语“包括”、“包含”或者其任何其他变体意在涵盖非排他性的包含,从而使得包括一系列要素的过程、方法、物品或者系统不仅包括那些要素,而且还包括没有明确列出的其他要素,或者是还包括为这种过程、方法、物品或者系统所固有的要素。在没有更多限制的情况下,由语句“包括一个
……”
限定的要素,并不排除在包括该要素的过程、方法、物品或者系统中还存在另外的相同要素。
[0101]
上述本发明实施例序号仅仅为了描述,不代表实施例的优劣。
[0102]
通过以上的实施方式的描述,本领域的技术人员可以清楚地了解到上述实施例方法可借助软件加必需的通用硬件平台的方式来实现,当然也可以通过硬件,但很多情况下前者是更佳的实施方式。基于这样的理解,本发明的技术方案本质上或者说对现有技术做出贡献的部分可以以软件产品的形式体现出来,该计算机软件产品存储在如上所述的一个存储介质(如rom/ram、磁碟、光盘)中,包括若干指令用以使得一台终端设备执行本发明各个实施例所述的方法。
[0103]
以上仅为本发明的优选实施例,并非因此限制本发明的专利范围,凡是利用本发明说明书及附图内容所作的等效结构或等效流程变换,或直接或间接运用在其他相关的技术领域,均同理包括在本发明的专利保护范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。