1.本发明涉及生物信息学领域,特别涉及利用计算机技术预测大分子-小分子间相互作用的领域。
背景技术:
2.人体是一个复杂系统,由基因、蛋白质、代谢物等多类型、多层次的要素及各类要素间相互作用构成,并通过复杂的相互作用、代偿机制来维持系统的平衡和稳态。对人体内不同类型、不同层面复杂相互作用的深入探索有利于系统性刻画生命体系,促进对癌症等复杂疾病作用机制的理解与研究。
3.长久以来人们更加关注于dna、蛋白质等生物大分子的功能,而代谢物却被认为是一种被动受到酶催化作用的终端产物,很少有研究关注到代谢物在其他生物学过程例如免疫、信号传导方面的调控功能。最近,越来越多的研究发现,代谢物通过与人体内关键蛋白质间的相互作用,同样也参与调控和影响除了代谢以外的众多生物学过程。因此,深入理解代谢物与蛋白质间的相互作用具有重要意义。然而,目前已知的代谢物-蛋白质相互作用主要局限于代谢酶及其反应物和产物间的相互作用,仍然有大量的代谢物-蛋白质相互作用没有得到揭示。
4.尽管高通量的质谱技术已经用于对特定代谢物相关作用蛋白质的鉴定,然而此类方法的实验成本高,并且通常受限于共价结合的相互作用方式。为了更加系统性、全面性地对代谢物-蛋白质相互作用预测,本发明提出了一种基于二维异质网络的代谢物-蛋白质相互作用预测方法,旨在通过计算方法对各类代谢物-蛋白质相互作用进行高效的预测。
技术实现要素:
5.本发明的目的在于提出一种基于二维异质网络的代谢物-蛋白质相互作用预测方法,构建代谢物与蛋白质二维异质生物网络,基于此网络计算任意代谢物和蛋白质间的多维度相关性,并构建随机森林分类模型,对潜在的代谢物与蛋白质相互作用进行预测。
6.本发明解决其问题所采用的技术方案如下:
7.一种基于二维异质网络的代谢物-蛋白质相互作用预测方法,该方法包括:
8.建模过程的步骤:基于二维异质网络构建用于代谢物-蛋白质相互作用预测的随机森林模型;
9.步骤1:构建代谢物-蛋白质二维异质网络;
10.步骤2:针对代谢物-蛋白质相互作用,收集阴性、阳性样本集和;
11.步骤3:对于阴性、阳性样本集和中的任意一对样本,计算每对代谢物-蛋白质间的多维度相关性;
12.步骤4:结合多维度相关性计算结果,训练基于随机森林算法的代谢物-蛋白质相互作用预测模型;
13.实际预测的步骤:基于上述构建好的随机森林模型预测任意一对代谢物与蛋白质
间是否具有相互作用;
14.针对代谢物-蛋白质二维异质网络中任意一对没有直接相连的代谢物和蛋白质,基于步骤3相同方法计算二者之间多维度相关性;将获得的多维相关性带入步骤4内的预测模型,获得这个代谢物与蛋白质间具有相互作用的概率值,当概率值大于预设阈值,判断该代谢物与蛋白质具有相互作用。
15.所述构建代谢物-蛋白质二维异质网络包括如下三个步骤:
16.步骤11:构建蛋白质-蛋白质相互作用网络;
17.从biogrid数据库中读取人类的蛋白质-蛋白质物理相互作用数据,以基因名标记每一个蛋白质,并构建蛋白质-蛋白质相互作用网络;
18.步骤12:构建代谢物-蛋白质相互作用网络;
19.通过从kegg数据库读取人类代谢通路的kgml文件,获取反应式信息;获取每个反应式中的酶、反应物、产物,并且以基因名标记酶,以kegg的化合物id标记反应物、产物;
20.标记相互作用关系用于表示处于作用关系两端的参与到同一个反应中的任意一个酶与一个反应物,或者任意一个酶与任意一个产物间存在相互作用,整合所有反应中非重复的相互作用,构建代谢物-蛋白质相互作用网络;
21.步骤13:构建代谢物-代谢物相互作用网络;
22.从pubchem数据库读取kegg代谢通路中每个代谢物的sdf二维结构文件;分别计算每个代谢物的分子描述符;计算任意两个代谢物的分子描述符间的tanimoto相关系数;
23.利用所有相关性大于0的代谢物-代谢物关联构建代谢物-代谢物相互作用网络;
24.所述步骤2中样本收集包括以下步骤:
25.步骤21:以所述步骤12中代谢物-蛋白质相互作用网络中删除邻居节点个数排名前10的代谢物后,剩余所有具有相互作用的成对的代谢物及蛋白质作为阳性样本集合;
26.步骤22:对阳性集合中所有的代谢物和蛋白质进行随机配对,并保证不与阳性集合重复,随机地生成与阳性集合数量一致的阴性集合;
27.所述步骤3多维度相关性计算包括以下步骤:
28.步骤31:根据蛋白质-蛋白质相互作用构建蛋白质邻接矩阵p;
29.步骤32:根据代谢物-蛋白质相互作用构建代谢物-蛋白质邻接矩阵i;
30.步骤33:根据代谢物-蛋白质相互作用构建代谢物-代谢物邻接矩阵m;
31.步骤34:基于邻接矩阵p、i、m计算4维度的代谢物-蛋白质相关性。
32.所述步骤31中蛋白质邻接矩阵p满足如下条件:
[0033][0034]
其中,pi,j表示矩阵p中的第i行、第j列内数值。
[0035]
所述步骤32中代谢物-蛋白质邻接矩阵i满足如下条件:
[0036][0037]
其中,ii,j表示矩阵i中的第i行、第j列内数值。
[0038]
所述步骤33中代谢物邻接矩阵m满足如下条件:
[0039]mi,j
=代谢物i与代谢物j分子描述符间tanimoto相关系数
[0040]
其中,mi,j表示矩阵m中的第i行、第j列内数值。
[0041]
所述步骤34包括以下步骤:
[0042]
步骤341:针对阳性及阴性样本集合中的任意一个样本,即一个代谢物m,和一个蛋白质p,计算第一维度相关性ns1,
[0043]
ns1(m,p)=ip
m,p
[0044]
步骤342:计算第二维度相关性ns2;
[0045]
ns2(m,p)=ip
2m,p
/σki
m,k
[0046]
步骤343:计算第三维相关性ns3:
[0047]
ns3(m,p)=mi m,p
/σki
m,k
[0048]
步骤344:计算第四维相关性ns4:
[0049]
ns4(m,p)=edgeconnectivity(m,p,g.h),
[0050]
其中,g.h是由同时整合了蛋白质-蛋白质相互作用,代谢物-代谢物相互作用、蛋白质-代谢物相互作用的二维异质网络,edgeconnectivity(m,p,g.h)计算在网络g.h中需要最少删除多少条网络边才可以断开节点m与p所有链接路径。
[0051]
所述步骤4包括以下步骤:
[0052]
步骤41:从阴性集合和阳性集合中随机比例选取样本作为训练集合、测试集合;
[0053]
步骤42:针对训练集合,以所述步骤3中计算获得的每个样本中代谢物与蛋白质间的4个维度的相关性作为样本的特征描述,以样本来自阳性或阴性集合作为类别标签,训练随机森林分类模型;
[0054]
步骤43:针对测试集,同样以所述步骤3中计算获得的每个样本中代谢物与蛋白质间的4个维度的相关性作为样本的特征描述,并输入步骤42训练好的分类模型,从而获得每个测试样本属于阳性、阴性集合的概率值,并以阳性概率值》预设阈值的预测结果判别为阳性,否则为阴性,从而判别预测结果的准确性。
[0055]
所述阳性表示当前测试样本判别为具有相互作用,所述阴性表示当前测试样本判别为不具有相互作用。
[0056]
本发明具有以下有益效果及意义:
[0057]
本发明提出的一种基于二维异质网络的代谢物-蛋白质相互作用预测方法,通过整合已知的蛋白质-蛋白质相互作用、代谢物-代谢物结构相关性、代谢物-蛋白质相互作用构建代谢物-蛋白质二维异质网络,基于该网络对任意代谢物和蛋白质间复杂的网络关联特征进行多维描述,并在此基础上利用随机森林算法构建代谢物-蛋白质相互作用预测,该模型能够可靠预测出潜在的代谢物-蛋白质相互作用。
附图说明
[0058]
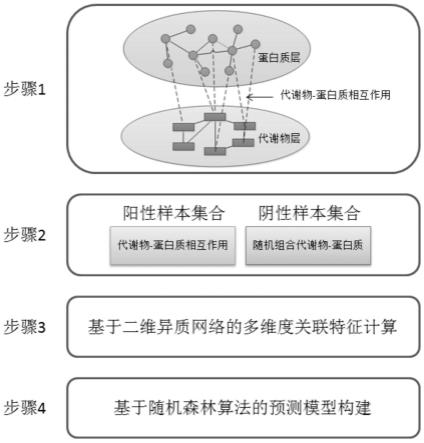
图1是本发明的一种基于二维异质网络的代谢物-蛋白质相互作用预测方法的示意图。
具体实施方式
[0059]
为使本发明的上述目的、特征和优点能够更加明显易懂,下面结合附图对本发明的具体实施方法做详细的说明。在下面的描述中阐述了很多具体细节以便于充分理解本发明。但本发明能够以很多不同于在此描述的其他方式来实施,本领域技术人员可以在不违背发明内涵的情况下做类似改进,因此本发明不受下面公开的具体实施的限制。
[0060]
除非另有定义,本文所使用的所有技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。本文中在发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不是旨在于限制本发明。
[0061]
一、基于二维异质网络构建用于代谢物-蛋白质相互作用预测的随机森林模型;
[0062]
图1是本发明所提供的一种基于二维异质网络的代谢物-蛋白质相互作用预测方法的示意图。如图1所示,本发明提供一种对代谢物-蛋白质间是否具有相互作用进行预测的方法。图1中从上到下依次给出了四个步骤:具体内容包括:
[0063]
步骤1:构建代谢物-蛋白质二维异质网络;
[0064]
所述代谢物-蛋白质二维异质网络包括了代谢物层、蛋白质层两个层面网络并基于已知的代谢层与蛋白质层相互作用连接两层网络,构建一个二维异质网络体系,主要是为后续对代谢物-蛋白质间的关联性描述提供基础。
[0065]
步骤2:针对代谢物-蛋白质相互作用,收集阴性、阳性样本集;
[0066]
所述阳性样本集合是指已知具有相互作用的代谢物-蛋白质对,阴性集合则是非相互作用的代谢物与蛋白质对,收集相同数量的阴性、阳性样本为后续训练随机森林预测模型数据基础。
[0067]
步骤3:计算每对代谢物-蛋白质间的多维度相关性;
[0068]
所述步骤整合利用代谢物-蛋白质二维异质网络中多种类型的拓扑相关性,从多个角度对任意一对代谢物-蛋白质间的网络关联性进行计算,从而获得多维度的相关性特征。
[0069]
步骤4:基于随机森林算法训练代谢物-蛋白质相互作用预测模型;
[0070]
所述步骤在步骤3获得的多维相关性特征的基础上,利用随机森林算法训练代谢物-蛋白质相互作用预测模型,该模型可以根据任意一对代谢物-蛋白质的多维度相关性特征判别二者间具有相互作用的概率。
[0071]
二、基于上述构建好的随机森林模型预测任意一对代谢物与蛋白质间是否具有相互作用;
[0072]
针对步骤1二维异质网络中任意一种代谢物及任意一种蛋白质,基于步骤3相同方法计算二者之间多维度相关性;基于步骤4已经获得的随机森林模型,获得这一对代谢物-蛋白质属于阳性或者阴性集合的概率值,其中阴性、阳性概率值的和为1,且当阳性概率值》0.5时,视作预测结果为阳性,即上述代谢物与蛋白质间具有潜在相互作用,当阳性概率值》0.9时,认为预测结果具有高可信度。
[0073]
以下对上述所涉及的各个步骤进行详细阐述。
[0074]
步骤1:构建代谢物-蛋白质二维异质网络;
[0075]
所述步骤1包括如下步骤:
[0076]
步骤11:构建蛋白质-蛋白质相互作用网络;
[0077]
从biogrid数据库中下载人类的蛋白质-蛋白质物理相互作用数据,以基因名(gene symbol)标记每一个蛋白质,并构建蛋白质-蛋白质相互作用网络;
[0078]
步骤12:构建代谢物-蛋白质相互作用网络;
[0079]
所述步骤12具体包括以下步骤:
[0080]
步骤121:从kegg数据库下载所有人类代谢通路的kgml文件;
[0081]
步骤122:获取每个kgml文件中的反应式信息;
[0082]
步骤123:获取每个反应式中的酶、反应物、产物,并且以基因名标记酶,以kegg的化合物id标记反应物、产物;
[0083]
步骤124:参与到同一个反应中的任意一个酶与一个反应物,或者任意一个酶与任意一个产物间添加相互作用;
[0084]
步骤125:整合所有反应中非重复的相互作用,构建代谢物-蛋白质相互作用网络
[0085]
步骤13:构建代谢物-代谢物相互作用网络;
[0086]
所述步骤13具体包括以下步骤:
[0087]
步骤131:从pubchem数据库下载kegg代谢通路中每个代谢物的sdf二维结构文件;
[0088]
步骤132:基于每个代谢物的二维结构sdf文件,利用r语言chemminer包中sdf2ap函数计算每个代谢物的分子描述符;
[0089]
步骤133:利用r语言chemminer包中cmp.similarity函数计算任意两个代谢物的分子描述符间tanimoto相关系数;
[0090]
步骤134:利用所有相关性大于0的代谢物-代谢物关联构建代谢物-代谢物相互作用网络;
[0091]
所述步骤2包括如下步骤:
[0092]
步骤21:以所述步骤12中代谢物-蛋白质相互作用网络中删除邻居节点个数排名前10的代谢物后剩余所有具有相互作用的成对的代谢物及蛋白质作为阳性样本集合;
[0093]
步骤22:利用r语言sample函数对阳性集合中所有的代谢物和蛋白质进行随机配对,并保证不与阳性集合重复,随机地生成与阳性集合数量一致的阴性集合;
[0094]
所述步骤3包括以下步骤;
[0095]
步骤31:根据蛋白质-蛋白质相互作用构建蛋白质邻接矩阵p,满足如下条件:
[0096][0097]
其中,pi,j表示矩阵p中的第i行、第j列内数值;
[0098]
步骤32:根据代谢物-蛋白质相互作用构建代谢物-蛋白质邻接矩阵i,满足如下条件:
[0099][0100]
其中,ii,j表示矩阵i中的第i行、第j列内数值;
[0101]
步骤33:根据代谢物-蛋白质相互作用构建代谢物-代谢物邻接矩阵m,满足如下条件:
[0102]mi,j
=代谢物i与代谢物j分子描述符间tanimoto相关系数
[0103]
其中,mi,j表示矩阵m中的第i行、第j列内数值;
[0104]
步骤34:基于邻接矩阵p、i、m计算4维度的代谢物-蛋白质相关性;
[0105]
所述步骤34具体包括以下步骤:
[0106]
步骤341:针对阳性及阴性样本集合中的任意一个样本,即一个代谢物m,和一个蛋白质p,计算第一维度相关性ns1,
[0107]
ns1(m,p)=ip
m,p
[0108]
其中ip
m,p
表示矩阵i及矩阵p乘积后所得矩阵中代谢物m对应行和蛋白质p对应列中的结果;
[0109]
步骤342:计算第二维度相关性ns2;
[0110]
ns2(m,p)=ip
2m,p
/σki
m,k
[0111]
其中ip
2m,p
表示矩阵i及矩阵p2乘积后所得矩阵中代谢物m对应行和蛋白质p对应列中的结果,σki
m,k
表示矩阵i中代谢物m对应行内的数值之和;
[0112]
步骤343:计算第三维相关性ns3:
[0113]
ns3(m,p)=mi m,p
/σki
m,k
[0114]
其中mi m,p
表示矩阵m及矩阵i乘积后所得矩阵中代谢物m对应行和蛋白质p对应列中的结果;
[0115]
步骤344:计算第四维相关性ns4:
[0116]
ns4(m,p)=edgeconnectivity(m,p,g.h),
[0117]
其中,g.h是由同时整合了蛋白质-蛋白质相互作用,代谢物-代谢物相互作用、蛋白质-代谢物相互作用的二维异质网络,edgeconnectivity(m,p,g.h)计算在网络g.h中需要最少删除多少条网络边才可以断开节点m与p所有链接路径。
[0118]
所述步骤4具体包括以下步骤:
[0119]
步骤41:从阴性集合和阳性集合中随机选取90%的样本作为训练集合,剩余10%作为测试集;
[0120]
步骤42:针对训练集合,以所述步骤3中计算获得的每个样本中代谢物与蛋白质间的4个维度的相关性作为样本的特征描述,以样本来自阳性或阴性集合作为类别标签,训练随机森林分类模型;
[0121]
步骤43:针对测试集,同样以所述步骤3中计算获得的每个样本中代谢物与蛋白质间的4个维度的相关性作为样本的特征描述,并输入步骤42训练好的分类模型,从而获得每个测试样本属于阳性、阴性集合的概率值,并以阳性概率值》0.5的预测结果为阳性,否则为阴性判别预测结果的准确性。
[0122]
实施例
[0123]
本文中将一种基于二维异质网络的代谢物-蛋白质相互作用预测方法用于对不饱和脂肪酸与免疫系统相关蛋白质间的相互作用分析中,候选脂肪酸4种(包括亚油酸、花生四烯酸、棕榈酸、二十二烷酸),免疫系统相关蛋白质42种(包括pdcd1、ctla4、src、tgfb1、mmp1、mmp9等),获得候选的代谢物-蛋白质对一共4*42=168种,实施效果如下:
[0124]
利用本发明构建的基于二维异质网络的代谢物-蛋白质相互作用预测模型预测上述4种脂肪酸与42种免疫相关蛋白质间具有相互作用的概率。结果显示,具有高可信度的7
组预测结果中,有4组(花生四烯酸-tgfb1,亚油酸-tgfb1,花生四烯酸-src,亚油酸-src)是文献中已经证实具有相互作用的(表1)。该实施例表明了本发明提出的一种基于二维异质网络的代谢物-蛋白质相互作用预测方法的有效性。
[0125]
表1.4种脂肪酸与42种免疫相关蛋白质间相互作用预测结果(阳性概率值》0.9的高度可信结果)
[0126][0127][0128]
以上所述的具体实施例,对本发明的目的、技术方案和有益效果进行了进一步详细说明,应理解的是,以上所述仅为本发明的具体实施例而已,并不用于限制本发明,凡在本发明的精神和原则之内,所做的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。