1.本发明涉及微生物技术领域,尤其是涉及一种预测胃癌淋巴结转移的菌群标志物及其应用。
背景技术:
2.胃癌(gastric cancer)是最常见的恶性肿瘤之一,近年来,胃癌诊疗技术发展迅速,但早期诊断和治疗策略仍然不足。癌症转移是癌症患者预后较差的主要原因之一,而胃癌最常见的转移方式是淋巴结转移。胃癌一旦发生淋巴结转移,预示癌症进一步恶性进展,并且治疗效果往往不甚理想,预后较差。此外,促使胃癌淋巴结转移的具体分子特征尚不明确,而缺乏有效的预测胃癌淋巴结转移的诊断方法和可靠的生物标志物可能是胃癌存活率低的重要原因之一。因此,有必要深入探讨胃癌恶性进展的分子机制,明确胃癌淋巴结转移的潜在分子网络,筛选合适的生物标志物,从而制定有效的诊断和治疗措施。
3.越来越多研究证实人体微生态与机体疾病的发生、发展密切相关,且研究也发现消化道内的微生物对胃肠道癌症的恶性进展有促进作用,因此消化道微生态检测具有预测胃癌淋巴结转移的应用前景,而其中又以胃内原位微生物的诊断价值最大。但目前由于胃内微生物丰度小、胃酸环境不宜微生物长期生存等生理特征,导致胃内微生物的研究较少。
技术实现要素:
4.本发明的目的在于克服上述现有技术的不足之处而提供一种预测胃癌淋巴结转移的菌群标志物及其应用,本发明的菌群标志物为肠杆菌目(enterobacteriales)耶尔森氏菌属(yersinia),以解决目前胃癌淋巴结转移患者缺乏进行早期预警、难以制定个性化精准随诊方案的临床困境,能够协助疾病诊断以及指导药物研究、精准用药。
5.为实现上述目的,本发明采取的技术方案为:
6.第一目的,本发明提供了一种预测胃癌淋巴结转移的菌群标志物,所述菌群标志物为耶尔森氏菌属(yersinia)。本发明的耶尔森氏菌属(yersinia)属于肠杆菌目(enterobacteriales)。
7.本发明发明人经过大量研究及试验发现,无淋巴结转移胃癌患者原发灶内耶尔森氏菌属(yersinia)的相对丰度显著高于有淋巴结转移胃癌患者(2.251%vs0.080%,p=0.005)。经受试者工作曲线分析(receiver operating characteristic curve,roc)发现,采用耶尔森氏菌属(yersinia)的相对丰度作为检验变量,roc曲线的曲线下面积(area under curve,auc)为1.000,最佳的诊断临界相对丰度为0.210%,诊断的特异性为100%,灵敏度为100%。上述结果提示,采用耶尔森氏菌属(yersinia)作为预测胃癌淋巴结转移的菌群标志物,具有较高的诊断效能。
8.第二目的,本发明提供了所述的菌群标志物在制备预测胃癌淋巴结转移的制剂中的应用。
9.第三目的,本发明提供了检测耶尔森氏菌属(yersinia)丰度的试剂在制备预测胃
癌淋巴结转移的制剂中的应用。
10.作为本发明所述应用的优选实施方式,所述试剂是检测胃癌组织中的耶尔森氏菌属(yersinia)丰度的试剂。
11.作为本发明所述应用的优选实施方式,所述试剂包括宏基因组测序试剂、16s rrna扩增子测序试剂、微滴式数字pcr检测试剂、qpcr定量检测试剂中的至少一种。
12.本发明检测肠杆菌目(enterobacteriales)耶尔森氏菌属(yersinia)丰度是通过宏基因组测序试剂、16s rrna扩增子测序试剂、微滴式数字pcr检测试剂、qpcr定量检测试剂中的至少一种进行测定。本发明将胃癌组织菌群(yersinia)的丰度或含量作为预测指标,因此,采用上述的分子生物学方式均可对其进行检测,这种多样化的定量手段可打破特定实验条件和实验技能的限制,使某些不具备特定实验设备的实验室也可实现本发明的数据测量和预测。
13.作为本发明所述应用的优选实施方式,所述试剂还包括聚合酶链式反应、逆转录-聚合酶链式反应、巢式pcr或核酸杂交用试剂。
14.第四目的,本发明提供了所述的菌群标志物在制备预防胃癌患者淋巴结转移药物或功能性食品中的应用。
15.第五目的,本发明提供了检测所述的菌群标志物的试剂在制备预测胃癌淋巴结转移的产品中的应用。
16.作为本发明所述应用的优选实施方式,所述产品为试剂盒或芯片。
17.本发明还开发了一套胃内胃癌组织微生态的检测方法,应用这套方案检测胃癌组织微生物表型与胃癌淋巴结转移的关系,并建立应用胃癌组织微生态筛选胃癌淋巴结转移高危患者的检测模型,为胃癌患者手术治疗后的精准诊疗提供新的手段。
18.与现有技术相比,本发明具有以下有益效果:
19.1)本发明提供了一种预测胃癌淋巴结转移的菌群标志物,可用于预测胃癌淋巴结转移风险的辅助诊断,检测特异性好(100%),灵敏度高(100%),具有指示胃癌组织微生物菌群的状况,指导胃癌组织微生态的调整以降低胃癌患者出现淋巴结转移的可能性;
20.2)本发明使用特定的肠杆菌目(enterobacteriales)耶尔森氏菌属(yersinia)丰度作为输入指标,不局限于测量手段,只要能获取微生物丰度值的实验手段即能够预测胃癌淋巴结转移。
附图说明
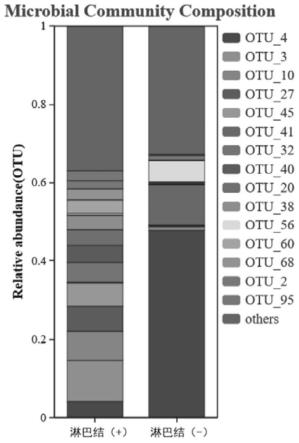
21.图1为胃癌患者胃癌原发灶微生物物种组成柱状图;
22.图2为胃癌患者有无淋巴结转移特征微生物线性判别分析图;
23.图3为各菌群进化关系及不同样本间的差异菌群的物种进化树;
24.图4为应用特征微生物序列检测胃癌患者淋巴结转移的roc曲线分析图。
具体实施方式
25.为更好的说明本发明的目的、技术方案和优点,下面将结合附图和具体实施例对本发明作进一步说明。
26.在以下实施例中,所使用的实验方法如无特殊说明,均为常规方法,所用的材料、
试剂等,如无特殊说明,均可从商业途径得到。
27.本发明的菌群标志物通过新一代测序技术高通量检测胃癌患者的胃癌原发灶组织微生物组成,挖掘胃癌淋巴结转移患者胃内微生态特征,从而确定胃癌淋巴结转移患者胃癌组织菌群的特异性核苷酸序列。
28.实施例1、胃癌组织微生态分析
29.(1)胃癌组织微生物dna提取:
30.在胃癌患者进行手术过程中,待手术医师将胃大体标本分离切除,对胃大体标本进行适当清洗,并使用无菌器械解剖,将部分胃癌病灶切割取下并使用1%pbs溶液和安多福0.1%乙烯吡咯烷酮碘消毒液进行表面清洗和消毒,然后装入冻存管,并立即放入液氮中转运;所获得样品存贮于-80℃低温冰箱,待微生物dna提取前再进行解冻。
31.使用研磨珠均质器(biospec)将解冻的胃癌组织样品充分研磨,所得组织沉淀采用qiaamp powerfecal dna kit(qiagen,germany)进行微生物dna提取。
32.(2)胃癌组织微生物16s rrna扩增子测序:
33.提取后的胃癌组织微生物dna使用341f 5
′‑
cctacgggnggcwgcag-3
′
(seq id no:1)及806r 5
′‑
ggactachvgggtwtctaat-3
′
(seq id no:2)作为扩增引物,使用ucp multiplex pcr kit(qiagen)进行16s rrna基因v3-v4区扩增,扩增后采用qiaseq ultralow input library kit进行文库构建。
34.文库采用high sensitivity dna kit(agilent,usa)及qubit 1
×
dsdna hs assay kit(thermo-fisher,usa)进行定量及混合,混合文库采用1.0nmol/l的naoh进行变性,后以14pm作为上机目标浓度,用ht1进行文库稀释。采用miseq reagent kit v3(illumina,usa)测序试剂盒在miseq平台(illumina)上进行测序。下机数据采用miseq reporter(illumina)进行数据拆分。
35.(3)胃癌组织微生态扩增子数据的生物信息学分析:
36.将miseq reporter拆分后的fastq数据导入clc workbench(qiagen)分析平台,采用microbial genomics module进行扩增子数据分析。具体分析流程如下:对低质量及嵌合体数据进行过滤后,测序获得的reads比对silva数据库,获得胃癌组织微生物的组成及相对丰度图(参考图1,展示丰度前十五的菌群在两组样本中的相对丰度)。
37.实施例2、筛选胃癌淋巴结转移患者的特征胃内菌群
38.(1)研究队列的建立
39.本研究收集了9例胃癌患者原发灶组织样品,术后对所有患者进行病理确诊以确定是否有淋巴结转移,其中4例患者无淋巴结转移,5例患者有淋巴结转移。
40.(2)胃癌淋巴结转移患者的特征胃癌组织菌群
41.胃癌组织菌群微生态分析根据实施例1所述方法执行。根据胃癌患者有无淋巴结转移进行分组,采用线性判别分析效应大小(linear discriminant analysis effect size,lefse)方法分析胃癌淋巴结转移的特征菌群。
42.首先,采用非参数检验kruskal-wallis秩和检验来识别胃癌组织微生物在不同分类学水平的存在的丰度差异,然后使用线性判别分析(linear discriminant analysis,lda)来估计每个差异丰度特征的大小。本研究采用p《0.05作为kruskal-wallis秩和检验中判断差异存在统计学意义的标准,采用lda》2.0作为判断将菌群纳入胃癌淋巴结转移特征
菌株的标准。lefse分析的结果通过柱状图和进化树分支图进行可视化。参考图2,图2展示胃癌无淋巴结转移组(左侧)及胃癌有淋巴结转移组(右侧)密切关联的主要菌种;图3为物种进化树图展示各菌群进化关系及不同样本间的差异菌群。
43.4例无淋巴结转移胃癌患者原发灶内耶尔森氏菌属(yersinia)的相对丰度显著高于5例有淋巴结转移胃癌患者(2.251%vs 0.080%,p=0.005)。经受试者工作曲线分析(receiver operating characteristic curve,roc)发现,采用耶尔森氏菌属(yersinia)的相对丰度作为检验变量,roc曲线的曲线下面积(area under curve,auc)为1.000,最佳的诊断临界相对丰度为0.210%,诊断的特异性为100%,灵敏度为100%。
44.实施例3、采用胃癌组织微生物标记物预测胃癌淋巴结转移患者的诊断效能评估实验
45.(1)胃癌患者验证队列的收集与检测
46.按照实施例2的方案收集8例胃癌患者原发灶组织样品,其中4例有淋巴结转移,4例无淋巴结转移。胃癌组织微生态检测方案按实施例1。
47.(2)采用胃癌组织微生物标记物预测胃癌淋巴结转移患者的诊断效能评估。
48.检测结果提示,4例无淋巴结转移胃癌患者原发癌灶肠杆菌目(enterobacteriales)耶尔森氏菌属(yersinia)的相对丰度明显高于有淋巴结转移的胃癌患者(2.250%vs 0.133%,p=0.021)。经受试者工作曲线分析(receiver operating characteristic curve,roc)发现,采用耶尔森氏菌属(yersinia)的相对丰度作为检验变量,roc曲线的曲线下面积(area under curve,auc)为1.000,最佳的诊断临界相对丰度为0.024%,诊断的特异性为100%,灵敏度为100%(参考图4)。
49.上述结果提示,采用耶尔森氏菌属(yersinia)作为预测胃癌患者淋巴结转移,具有较高的诊断效能。
50.本发明提供了一种预测胃癌早期淋巴结转移的菌群标志物耶尔森氏菌属(yersinia),可用于预测胃癌淋巴结转移风险的辅助诊断,其检测特异性好(100%),灵敏度高(100%),具有指示胃癌组织微生物菌群的状况,指导胃癌组织微生态的调整以降低胃癌患者出现淋巴结转移的可能性;并且只用特定的肠杆菌目(enterobacteriales)耶尔森氏菌属(yersinia)丰度作为输入指标,不局限于测量手段,只要能获取微生物丰度值的实验手段,即能够辅助预测胃癌淋巴结转移风险。
51.最后所应当说明的是,以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,尽管参照较佳实施例对本发明作了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的实质和范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。