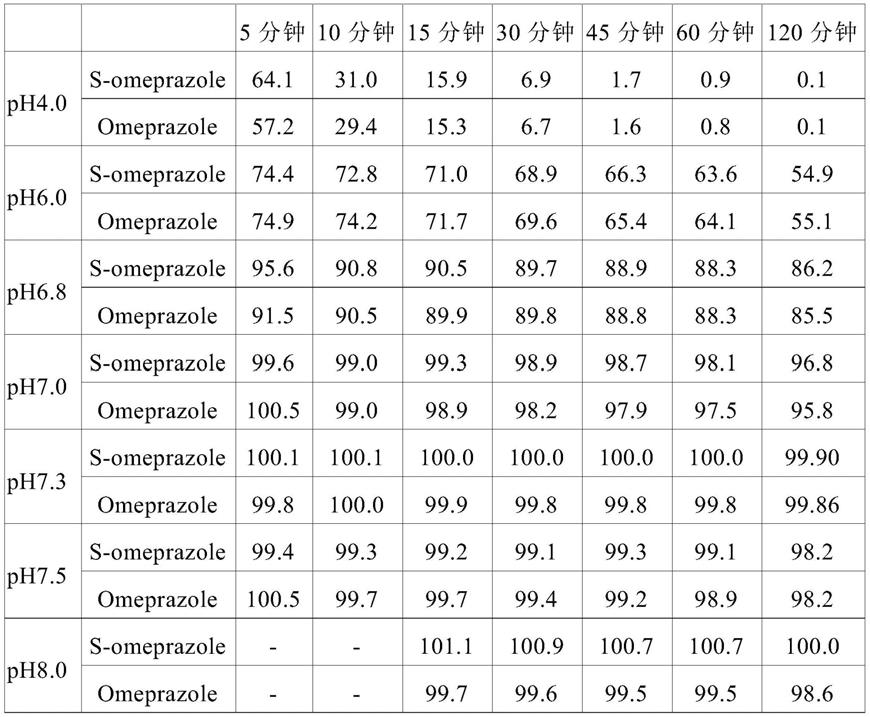
1.本说明书公开了用于预测测试物质在人类中的适应症的方法、用于预测测试物质在人类中的适应症的装置、用于预测测试物质在人类中的适应症的程序、以及用于训练用于预测测试物质在人类中的适应症的人工智能模型的方法以及经训练的人工智能模型。
背景技术:
2.新药的开发从用于寻找新药的候选物质的药物发现研究(发现阶段)开始,然后是使用动物或培养细胞的临床前试验(阶段0)以及人类的阶段i至iii临床试验。只有通过了试验的物质才能申请获得厚生劳动省批准作为医药品制造和销售的许可。然后,即使在经过被批准作为医药品所需的审查并被投入市场之后,也提供用于监测在开发阶段和批准审查阶段期间未预测到的不良事件或效果的时间段。如上所述,一种新药投入市场需要大量的时间和金钱。另一方面,据说物质从发现阶段进入制造和销售批准的概率约为1.6%。还据说,在通过临床前试验的物质中,仅13.8%在从临床前试验通过至阶段iii的临床试验期间展现出无不良事件的效果并获得批准申请。换句话说,超过80%的候选物质在阶段i至iii临床试验期间退出。据说由于退出引起的损失高达每种物质1.5至2亿美元。
3.作为用于在新药的开发中辅助新药的候选物质的探索的方法,专利文献1公开了一种方法,该方法包括:将从源自已经给予测试物质的个体的一个或多于一个器官的细胞或组织所获得的各器官中的器官相关指标因子的测试数据与初步确定的器官相关指标因子的相应标准数据进行比较,以获得用于计算器官相关指标因子的模式的相似度的模式相似度;以及使用器官相关指标因子的模式相似度作为指标来预测测试物质在一个或多于一个器官中和/或在除该一个或多于一个器官以外的器官中的功效或副作用。
4.另外,作为用于预测候选物质在新药的开发中的功效或副作用的方法,专利文献2公开了一种人工智能模型,该人工智能模型用于根据与从已经给予测试物质以准备训练数据的非人类动物收集到的多个不同器官相同的多个不同器官中的转录组(transcriptome)的动态来预测测试物质对人类的一个或多于一个作用。该方法包括将指示从已经针对非人类动物中的各非人类动物分别给予对人类具有已知作用的多个现有物质的非人类动物收集到的多个不同器官中的转录组的动态的数据集以及指示各现有物质对人类的已知作用的数据作为训练数据输入到人工智能模型中,以训练人工智能模型。
5.引用列表
6.专利文献
7.专利文献1:wo2016/208776
8.专利文献2:日本特开6559850
技术实现要素:
9.发明要解决的问题
10.本发明的一个目的是根据响应于向除人类以外的动物给予测试物质的生物标志
物的动态来有效地预测该测试物质在人类中的适应症。
11.通过专利文献2中所公开的方法,仅可以预测关于在获取多个器官的转录组数据作为训练数据时使用的现有物质已经已知的功效。
12.本发明的目的是即使在测试物质具有关于在获取训练数据时使用的现有物质尚未已知的功效的情况下,也预测出该测试物质的功效。
13.用于解决问题的方案
14.本发明也可以包括以下方面作为实施例。
15.项1.本发明的特定实施例涉及一种用于人工智能模型的训练方法。所述训练方法包括:将第一训练数据集、第二训练数据集和第三训练数据集彼此相关联地输入到人工智能模型中,以训练所述人工智能模型;所述第一训练数据集是如下的数据集:指示从已经分别给予在人类中具有已知适应症的多个预定现有物质的相应非人类动物收集到的一个器官或多个不同器官中的各器官中的生物标志物的动态的数据集与指示所给予的预定现有物质的相应名称的标签相关联;所述第二训练数据集是如下的数据集:指示所述多个预定现有物质的相应名称的标签与指示针对所述多个预定现有物质中的各预定现有物质所报告的适应症的标签相关联;所述第三训练数据集是如下的数据集:指示针对所述多个预定现有物质中的各预定现有物质所报告的适应症的标签与关于对应于这些适应症中的各适应症而报告的不良事件的信息相关联;其中,所述人工智能模型用于预测测试物质在人类中的适应症。
16.项2.在根据项1的训练中,通过所述第二训练数据集使所述第一训练数据集和所述第三训练数据集相关联以生成第四训练数据集,并且将所述第四训练数据集输入到所述人工智能模型中。
17.项3.在根据项1或2的训练方法中,关于不良事件的所述信息包括指示所述不良事件的标签、以及所述适应症中的不良事件的有无或发生频率。
18.项4.在根据项1至3中任一项的训练方法中,所述生物标志物是转录组。
19.项5.在根据项1至4中任一项的训练方法中,所述人工智能模型是单类支持向量机即one-class svm。
20.项6.本发明的特定实施例涉及一种用于人工智能模型的训练装置。所述训练装置包括处理部,其中,所述处理部将第一训练数据集、第二训练数据集和第三训练数据集彼此相关联地输入到人工智能模型中,以训练所述人工智能模型,所述第一训练数据集是如下的数据集:指示从已经分别给予在人类中具有已知适应症的多个预定现有物质的相应非人类动物收集到的一个器官或多个不同器官中的各器官中的生物标志物的动态的数据集与指示所给予的预定现有物质的相应名称的标签相关联;所述第二训练数据集是如下的数据集:指示所述多个预定现有物质的相应名称的标签与指示针对所述多个预定现有物质中的各预定现有物质所报告的适应症的标签相关联;所述第三训练数据集是如下的数据集:指示针对所述多个预定现有物质中的各预定现有物质所报告的适应症的标签与关于对应于这些适应症中的各适应症而报告的不良事件的信息相关联;以及其中,所述人工智能模型用于预测测试物质在人类中的适应症。
21.项7.本发明的特定实施例涉及一种用于训练人工智能模型的程序,所述程序在由计算机执行时使所述计算机执行如下的步骤:将第一训练数据集、第二训练数据集和第三
训练数据集彼此相关联地输入到人工智能模型中,以训练所述人工智能模型。在所述程序中,所述第一训练数据集是如下的数据集:指示从已经分别给予在人类中具有已知适应症的多个预定现有物质的相应非人类动物收集到的一个器官或多个不同器官中的各器官中的生物标志物的动态的数据集与指示所给予的预定现有物质的相应名称的标签相关联;所述第二训练数据集是如下的数据集:指示所述多个预定现有物质的相应名称的标签与指示针对所述多个预定现有物质中的各预定现有物质所报告的适应症的标签相关联;所述第三训练数据集是如下的数据集:指示针对所述多个预定现有物质中的各预定现有物质所报告的适应症的标签与关于对应于这些适应症中的各适应症而报告的不良事件的信息相关联;以及其中,所述人工智能模型用于预测测试物质在人类中的适应症。
22.项8.本发明的特定实施例涉及一种用于预测测试物质在人类中的适应症的方法。所述方法包括如下的步骤:获取第一测试数据集,所述第一测试数据集是指示从已经给予测试物质的非人类动物收集到的一个或多于一个器官中的生物标志物的动态的数据集,以及将所述第一测试数据集和第二测试数据集输入到通过根据项1至5中任一项所述的方法训练的人工智能模型中,以使用经训练的人工智能模型基于输入到其中的所述第一测试数据集和所述第二测试数据集来预测所述测试物质在人类中的适应症,所述第二测试数据集是多个已知适应症的标签与关于对应于所述多个已知适应症中的各适应症而报告的不良事件的信息相关联的数据集。
23.项9.在根据实施例8的预测方法中,所述测试物质不包括现有物质或现有物质的等效物质。
24.项10.在根据实施例8或9的预测方法中,所述测试物质是选自现有物质和现有物质的等效物质中的一种。
25.项11.本发明的特定实施例涉及一种用于预测测试物质在人类中的适应症的预测装置。所述预测装置包括处理部,其中,所述处理部将第一测试数据集和第二测试数据集输入到通过根据项1至5中任一项所述的方法训练的人工智能模型中,以使用经训练的人工智能模型基于输入到其中的所述第一测试数据集和所述第二测试数据集来预测所述测试物质在人类中的适应症,所述第一测试数据集是指示与从为了生成第一训练数据集而已经给予所述测试物质的非人类动物收集到的一个或多于一个器官相对应的一个或多于一个器官中的生物标志物的动态的数据集,所述第二测试数据集是多个已知适应症的标签与为了生成第三训练数据集而获取的关于与所述多个已知适应症中的各适应症相对应地报告的不良事件的信息相关联的数据集。
26.项12.本发明的特定实施例涉及一种用于预测测试物质在人类中的适应症的计算机程序,所述计算机程序在由计算机执行时使所述计算机执行如下的步骤:将第一测试数据集和第二测试数据集输入到通过根据项1至5中任一项所述的方法训练的人工智能模型中,以使用经训练的人工智能模型基于输入到其中的所述第一测试数据集和所述第二测试数据集来预测所述测试物质在人类中的适应症,所述第一测试数据集是指示与从为了生成第一训练数据集而已经给予所述测试物质的非人类动物收集到的一个或多于一个器官相对应的一个或多于一个器官中的生物标志物的动态的数据集,所述第二测试数据集是多个已知适应症的标签与关于与所述多个已知适应症中的各适应症相对应地报告的不良事件的信息相关联的数据集。
27.项13.本发明的特定实施例涉及一种用于预测测试物质在人类中的适应症的预测系统。所述系统包括服务器装置,用于发送第一测试数据集,所述第一测试数据集是指示从已经给予所述测试物质的非人类动物收集到的一个或多于一个器官中的生物标志物的动态的数据集,以及预测装置,用于预测所述测试物质对人类的作用,所述预测装置经由网络连接到所述服务器装置。所述服务器装置包括用于发送所述第一测试数据集的通信部,所述预测装置包括处理部和通信部,其中,所述处理部经由所述预测装置的通信部来获取经由所述服务器装置的通信部发送的所述第一测试数据集,以及将所获取的第一测试数据集和第二测试数据集输入到通过根据项1至5中任一项所述的方法训练的人工智能模型中,以使用经训练的人工智能模型基于输入到其中的所述第一测试数据集和所述第二测试数据集来预测所述测试物质在人类中的适应症,所述第一测试数据集是指示与从为了生成第一训练数据集而已经给予所述测试物质的非人类动物收集到的一个或多于一个器官相对应的一个或多于一个器官中的生物标志物的动态的数据集,所述第二测试数据集是如下的数据集:多个已知适应症的标签与为了生成第三训练数据集而获取的关于对应于所述多个已知适应症中的各适应症而报告的不良事件的信息相关联。
28.项14.本发明的特定实施例涉及一种用于使用第一训练数据集、第二训练数据集和第三训练数据集来训练人工智能模型的方法,所述人工智能模型用于预测测试物质在人类中的适应症,所述第一训练数据集是如下的数据集:指示从已分别给予在人类中具有已知适应症的多个预定现有物质的相应非人类动物收集到的一个器官或多个不同器官中的各器官中的生物标志物的动态的数据集与指示为了获取指示所述生物标志物的动态的数据集而给予的现有物质的名称的标签相关联,所述第二训练数据集是如下的数据集:指示所述多个预定现有物质的相应名称的标签与指示针对所述多个预定现有物质中的各预定现有物质所报告的适应症的标签相关联,所述第三训练数据集是如下的数据集:指示所述适应症的标签与关于对应于所述适应症中的各适应症所报告的不良事件的信息相关联。
29.项15.本实施例涉及一种用于使用第一测试数据集和第二测试数据集作为用于预测测试物质在人类中的适应症的测试数据的方法。在所述方法中,所述第一测试数据集是指示与从为了生成第一训练数据集而已给予所述测试物质的非人类动物收集到的一个或多于一个器官相对应的一个或多于一个器官中的生物标志物的动态的数据集,以及所述第二测试数据集是多个已知适应症的标签与关于与所述多个已知适应症中的各适应症相对应地报告的不良事件的信息相关联的数据集。
30.发明的效果
31.即使在测试物质关于在获取训练数据时使用的现有物质具有尚未已知的功效的情况下,也可以预测该测试物质的功效。
附图说明
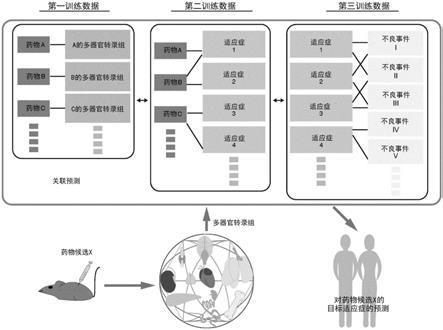
32.图1示出本发明的概要。
33.图2示出专利文献2(现有技术)中所描述的发明的概要。
34.图3示出训练数据的示例。图3的(a)示出第一训练数据的示例。图3的(b)示出第二训练数据的示例。图3的(c)示出第三训练数据的示例。图3的(d)示出第四训练数据的示例。
35.图4的(a)示出训练系统的硬件结构。图4的(b)示出预测系统的硬件结构。
36.图5示出训练装置的硬件结构。
37.图6是示出训练程序的处理的流程的流程图。
38.图7示出预测装置的硬件结构。
39.图8是示出预测程序的处理的流程的流程图。
40.图9示出服务器装置的硬件结构。
41.图10是示出预测系统中的处理的流程的流程图。
42.图11示出来自在不使用测试药物的转录组数据的情况下训练的人工智能的预测结果。
43.图12示出来自使用测试药物的转录组数据进行训练的人工智能的预测结果。
44.图13示出阿仑膦酸钠(alendronate)的一些决策函数值。
具体实施方式
45.1.训练方法和预测方法的概要以及术语的说明
46.首先,概述了如本发明的某些实施例的用于训练人工智能的方法以及预测方法。另外,说明了传统方法与本发明中所包括的训练方法和预测方法之间的差异。
47.该预测方法预测测试物质在人类中的适应症。优选地,预测方法基于与已经给予对人类具有已知作用的现有物质的非人类动物中的生物标志物的动态、已知适应症以及与该已知适应症相对应地报告的不良事件有关的信息,来预测测试物质在人类中的适应症。使用人工智能模型进行预测。
48.(1)训练阶段
49.如图1所示,用于预测的人工智能模型优选地通过包括彼此相关联的三种类型的训练数据集(即,第一训练数据集、第二训练数据集和第三训练数据集)的数据集来训练。
50.如图1所示,第一训练数据集是如下的数据集,在该数据集中,将指示在向非人类动物分别给予了在人类中具有已知适应症的多个预定现有物质之后从相应的非人类动物收集到的一个器官或者多个不同器官中的各器官中的生物标志物的动态的数据集与指示所给予的预定现有物质的相应名称的标签相关联。如图1所示,生成第一训练数据集。例如,向诸如小鼠等的非人类动物分别给予作为预定现有物质的药物a、b和c,并且从非人类动物分别收集器官或作为器官的一部分的组织。接着,分析收集到的器官或组织中的生物标志物的动态,并根据[指示相应器官名和生物标志物的动态的数据]以及[所给予的药物的相应名称]生成第一训练数据集。图3(a)示出第一训练数据集的更具体示例。在图3(a)所示的第一训练数据集的示例中,最左边的列被称为“第一列”。在图3(a)所示的第一列中,作为示例示出药物名“阿立哌唑(aripiprazole)”和药物名“empa”。在第二列和后续列中,示出各器官中的rna的表达水平。“心脏(heart)”和“皮肤(skin)”是器官名的标签,并且“alas2”和“apod”是表达被分析的基因名的标签。在第二列和后续列以及第二行和后续行中,指示相应基因的表达水平的值已作为元素输入。在第一训练数据集中,[指示器官名的标签和指示基因名的标签]以及[指示相应基因的表达水平的值]与指示药物名的标签相对应。
[0051]
如图1所示,第二训练数据集是如下的数据集,在该数据集中,将指示为了获得第一训练数据集而给予的多个预定现有物质的相应名称的标签(图3的(a)的第一列)与指示针对该多个预定现有物质中的各预定现有物质所报告的适应症的标签相关联。图3(b)示出
第二训练数据集的具体示例。在图3(b)所示的第二训练数据集的示例中,最左边的列被称为“第一列”。在图3(b)所示的第一列中,作为示例示出药物名“阿立哌唑”和药物名“empa”。第二列和后续列示出针对第一列中列出的各药物所报告的适应症。这里,“神经损伤”被示出为指示药物名“阿立哌唑”的适应症的名称标签,并且“2型糖尿病”被示出为指示药物名“empa”的适应症的名称标签。
[0052]
如图1所示,第三训练数据是如下的数据集,在该数据集中,将指示如图3(b)所示的针对为了获得第一训练数据集而给予的多个预定现有物质中的各预定现有物质所报告的适应症的标签与关于对应于这些适应症中的各适应症所报告的不良事件的信息相关联。这里,关于不良事件的信息可以包括指示不良事件的名称的标签以及不良事件的有无或发生频率。图3(c)示出第三训练数据集的更具体示例。在图3(c)所示的第三训练数据集的示例中,最左边的列被称为“第一列”。在图3(c)的第一列中示出“神经损伤”,它是图3(b)的“适应症1”中所示的药物名“阿立哌唑”的适应症。另外,作为图3(b)的“适应症1”中所示的药物名“empa”的适应症,“2型糖尿病”示出在图3(c)的第一列中。图3(c)的第二列和后续列的最上列示出指示不良事件的名称的标签,并且这里示出“睡眠障碍”和“血糖下降”。图3(c)的第二列的第二行和后续行中的数值示出相应不良事件的发生频率。
[0053]
在本实施例的训练方法中,输入到人工智能中的是第四训练数据集,该第四训练数据集是通过第二训练数据集将第一训练数据集与第三训练数据集相关联而生成的。
[0054]
图3(d)示出第四训练数据集的示例。在图3(d)所示的第四训练数据集的示例中,最左边的列被称为“第一列”。在第一列和第二列中,示出指示图3(c)中所示的不良事件的名称的标签以及相应不良事件的发生频率。另外,在第四列和后续列中,示出图3(a)中所示的指示器官名的标签和基因名的标签以及基因的表达水平。换句话说,图3(d)示出包括图3(c)的第二列和后续列中的不良事件的发生频率的数据集,其中不良事件的发生频率代替图3(a)中示出药物名的第一列中的标签。
[0055]
(2)预测阶段
[0056]
如上述第1.(1)节中所述地训练的人工智能模型用于预测测试物质在人类中的适应症。输入到经训练的人工智能模型中以预测适应症的测试数据集是第一测试数据集和第二测试数据集。第一测试数据集与第二测试数据集一起输入到经训练的人工智能模型中。
[0057]
第一测试数据集是指示从已经给予测试物质的非人类动物收集到的一个或多于一个器官中的生物标志物的动态的数据集。另外,多个器官与为了生成第一训练数据集而收集到的器官相对应。优选地,第一测试数据是[指示器官名的标签和指示基因名的标签]与[指示相应基因的表达水平的值]相关联的数据,该数据是通过向非人类动物给予一个测试物质并分析从中收集到的一个或多于一个器官中的转录组而获得的。
[0058]
第二测试数据集是多个已知适应症的标签与为了生成第三训练数据集而获取的、关于与多个已知适应症中的各适应症相对应地报告的不良事件的信息相关联的数据集。这里,多个已知适应症不仅可以包括用作第二训练数据的适应症,还可以包括登记在外部数据库中的已知适应症。在已知适应症中,术语“多个”可以意指例如100、500、1000或2000或更多。
[0059]
这里,在预测方法中,测试物质不必一定是现有物质或现有物质的等效物质。在测试物质不是现有物质或现有物质的等效物质的情况下,预测方法用作用于预测新物质的适
应症的方法。
[0060]
另外,在预测方法中,可以包括现有物质或现有物质的等效物质作为测试物质。在这种情况下,预测方法用作用于探索现有物质或现有物质的等效物质的新适应症的药物重新定位方法。在使用本说明书中所描述的预测方法作为药物重新定位方法的情况下,优选将测试物质包括在为了获取第一训练数据集而给予的现有物质中。以这种方式,可以提高预测精度。
[0061]
(3)与传统方法的比较
[0062]
图2中所示的传统方法是专利文献2中所描述的方法,其中该方法例如向诸如小鼠等的非人类动物分别给予作为现有物质的药物a、b和c,并且从相应的非人类动物收集器官或作为器官的一部分的组织。接着,分析所收集到的器官或组织中的生物标志物的动态以生成第一训练数据集。另外,根据现有物质的例如不良事件、适应症、药物动力学和适应症等的人类临床数据库生成第二训练数据。然后,通过使用第一训练数据集和第二训练数据进行训练来生成图2所示的人工智能模型。换句话说,在传统方法中,通过将生物标志物的动态与现有物质的不良事件、适应症、药物动力学或者和适应症中的每一个相关联来构建人工智能模型。另外,在传统方法中使用的测试数据是如下的数据,该数据指示与从为了生成第一训练数据集而已经给予测试物质的非人类动物收集到的一个器官或多个器官相对应的一个器官或多个不同器官中的生物标志物的动态。
[0063]
本实施例与传统方法的不同之处在于,作为训练数据,不仅使用生物标志物的动态,而且还使用代替适应症名的与不良事件有关的信息。另外,作为测试数据,也不仅使用生物标志物的动态,而且还使用与多个已知适应症和不良事件有关的信息。
[0064]
因此,即使在测试物质具有关于获取训练数据时使用的现有物质尚未已知的适应症的情况下,也可以预测测试物质的适应症。
[0065]
(4)术语的说明
[0066]
在本发明中,非人类动物不受限制。示例包括诸如小鼠、大鼠、狗、猫、兔、牛、马、山羊、绵羊和猪等的哺乳动物以及诸如鸡等的鸟类。优选地,非人类动物是诸如小鼠、大鼠、狗、猫、牛、马和猪等的哺乳动物,更优选地是小鼠或大鼠等,并且更优选地是小鼠。非人类动物还包括这些动物的胎儿和雏等。
[0067]
在本发明中,术语“物质”可以包括例如化合物;核酸;糖类;脂类;糖蛋白;糖脂;脂蛋白;氨基酸;多肽;蛋白质;多酚类;趋化因子(chemokines);选自包含上述物质的最终代谢物、中间代谢物和合成原料物质的组的至少一种代谢物质;金属离子;或微生物。另外,物质可以是单质,或者可以是多种物质的混合物。优选地,术语“物质”包括例如医药品、准医药品、药用化妆品、食品、特定保健用食品、具有功能声称食品及其候选。另外,术语“物质”还可以包括在医药批准的临床前试验或临床试验期间中止或中断测试的物质。
[0068]“现有物质”不受限制,只要它是现有物质即可。优选地,它是一种对人类具有已知作用的物质。另外,术语“现有物质的等效物质”可以包括与现有物质在结构上类似并具有类似作用的物质。这里,术语“类似作用”意指具有与现有物质相同的作用,但是作用的强度可以是相同的或不同的。
[0069]“不良事件”不受限制,只要它是被判断为对人类有害的作用即可。优选示例包括诸如faers(https://www.fda.gov/drugs/guidancecomplianceregulatoryinformation/
surveillance/adversedrugeffects/ucm082193.htm)或clinicaltrials.gov(https://clinicaltrials.gov/)等的外部数据库中所列出的不良事件。
[0070]“适应症”不受限制,只要它是应被减轻、治疗、停止进行或预防的人类的疾患或症状即可。疾患或症状的示例包括诸如上述faers、dailymed的所有药物标签(https://dailymed.nlm.nih.gov/dailymed/spl-resources-all-drug-labels.cfm)、医学主题词表(medical subject headings)(https://www.nlm.nih.gov/mesh/meshhome.html)、drugs@fda(https://www.accessdata.fda.gov/scripts/cder/daf/)或国际疾病分类(https://www.who.int/health-topics/international-classification-of-diseases)等的外部数据库中所列出的疾患或症状。更具体地,适应症的示例包括诸如血栓症、栓塞症和狭窄症(stenosis)等的缺血性疾病(特别是心脏、脑、肺、大肠等);诸如动脉瘤、静脉瘤、充血和出血等的循环障碍(主动脉、静脉、肺、肝脏、脾脏、视网膜等);诸如过敏性支气管炎和肾小球肾炎等的过敏性疾病;诸如阿尔茨海默型痴呆症等的痴呆症;诸如帕金森病、肌萎缩性侧索硬化症和重症肌无力症等的变性疾患(神经、骨骼肌等);肿瘤(良性上皮性肿瘤、良性上皮性肿瘤、恶性上皮性肿瘤、恶性非上皮性肿瘤);代谢性疾病(糖类代谢异常、脂类代谢异常、电解质失衡);传染病(细菌、病毒、立克次体(rickettsia)、衣原体、真菌、原虫、寄生虫等);以及诸如肾脏疾病、系统性红斑和多发性硬化等的与自身免疫性疾病等相关联的症状或疾病。
[0071]
不良事件的发生率可通过以下方法获得。通过例如从数据库(诸如如上所述的clinicaltrials.gov、faers、或dailymed的所有药物标签等)中进行文本提取来提取指示不良事件的名称的单词。所提取的一个单词可以算作报告的一个不良事件。对于一种现有物质,可以根据下式获得发生率:发生率=(针对一个不良事件所报告的案例数)/(针对现有物质所报告的不良事件的总案例数)。在数据库中以文本形式登记了与作用相关的说明的情况下,可以在提取与作用相对应的文本之前通过自然语言处理对登记的文本进行语法分析、单词分割或语义分析等。
[0072]“器官”不受限制,只要它是存在于如上所述的哺乳动物或鸟类的身体中的器官即可。例如,在哺乳动物的情况下,器官是选自如下项中的至少一种:循环系统器官(心脏、动脉、静脉、淋巴管等)、呼吸系统器官(鼻腔、副鼻腔、喉头、气管、支气管、肺等)、消化系统器官(唇、颊部、腭、齿、牙龈、舌、唾液腺、咽、食道、胃、十二指肠、空肠、回肠、盲肠、阑尾、上行结肠、横结肠、s状结肠、直肠、肛门、肝脏、胆囊、胆管、胆道、胰腺、胰管等)、泌尿系统器官(尿道、膀胱、输尿管、肾脏)、神经系统器官(大脑、小脑、中脑、脑干、脊髓、末梢神经、自主神经等)、女性生殖系统器官(卵巢、输卵管、子宫、阴道等)、乳房、男性生殖系统器官(阴茎、前列腺、睾丸、附睾(epididymis)、输精管)、内分泌系统器官(下丘脑、脑下垂体、松果体、甲状腺、副甲状腺、肾上腺等)、外皮系统器官(皮肤、毛发、指甲等)、造血系统器官(血液、骨髓、脾脏等)、免疫系统器官(淋巴结、扁桃体、胸腺等)、骨和软组织器官(骨、软骨、骨骼肌、结缔组织、韧带、腱、隔膜、腹膜、胸膜、脂肪组织(棕色脂肪、白色脂肪)等和感觉系统器官(眼球、眼睑、泪腺、外耳、中耳、内耳、耳蜗等)。优选地,“器官”可以是选自如下项中的至少一种:骨髓、胰腺、头盖骨、肝脏、皮肤、脑、脑下垂体、肾上腺、甲状腺、脾脏、胸腺、心脏、肺、主动脉、骨骼肌、睾丸、附睾脂肪、眼球、回肠、胃、空肠、大肠、肾脏和腮腺。优选地,所有骨髓、胰腺、头盖骨、肝脏、皮肤、脑、脑下垂体、肾上腺、甲状腺、脾脏、胸腺、心脏、肺、主动脉、骨骼肌、睾
丸、附睾脂肪、眼球、回肠、胃、空肠、大肠、肾脏和腮腺在根据本发明的预测中使用。术语“多个器官”不受限制,只要器官的数量是两个或多于两个即可。例如,多个器官可以选自2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23或24种类型的器官。
[0073]
术语“器官由来(organ-derived)”意指例如从器官收集,或从所收集到的器官的细胞、组织或体液培养。
[0074]
术语“体液”包括例如血清、血浆、尿、脊髓液、腹水、胸腔积液、唾液、胃液、胰液、胆汁、乳汁、淋巴液和细胞间质液。
[0075]
术语“生物标志物”是指可根据物质的给予、在各器官的细胞或组织中和/或在体液中变化的生物物质。可用作“生物标志物”的生物物质的示例是选自以下项中的至少一种:核酸;糖类;脂类;糖蛋白;糖脂;脂蛋白;氨基酸、多肽;蛋白质;多酚类;趋化因子;选自包含上述物质的最终代谢物、中间代谢物和合成原料物质的组的至少一种代谢物质;以及金属离子等。更优选的是核酸。生物标志物优选是根据物质的给予在各器官的细胞或组织中和/或在体液中变化的生物物质的组。生物物质组的示例可以是选自以下项中的至少一种的组:核酸;糖类;脂类;糖蛋白;糖脂;脂蛋白;氨基酸、多肽;蛋白质;多酚类;趋化因子;选自包含上述物质的最终代谢物、中间代谢物和合成原料物质的组的至少一种代谢物质;以及金属离子等。
[0076]
术语“核酸”优选是指包含在转录组中的rna的组,诸如mrna、非编码rna和microrna,更优选是指mrna的组。rna优选是可在上述器官的细胞或组织中或在体液中的细胞中表达的mrna、非编码rna和/或microrna,更优选是可通过rna-seq等检测到的mrna、非编码rna和/或microrna(https://www.ncbi.nlm.nih.gov/gene?linkname=genome_gene&from_uid=52、http://jp.support.illumina.com/sequencing/sequencing_software/igenome.html)。优选地,可通过rna-seq分析的所有rna用于根据本发明的预测。
[0077]
术语“指示生物标志物的动态的数据集”意指指示生物标志物已经或尚未响应于现有物质的给予而变化的数据集。优选地,生物标志物的动态指示生物标志物已经响应于现有物质的给予而变化。例如,可以通过以下方法获取数据。针对从已经给予现有物质的非人类动物收集到的某些器官由来的组织、细胞或体液,测量各生物标志物的丰度或浓度,以获取已经给予现有物质的个体的各器官的测量值。另外,从尚未给予现有物质的非人类动物,针对与获取已经给予现有物质的个体的测量值的器官相对应的器官由来的组织、细胞或体液,以相同的方式测量各生物标志物的丰度或浓度,以获取非给予个体的测量值。将已经给予现有物质的个体的各器官由来的各生物标志物的测量值同非给予个体中与已经给予现有物质的个体中的生物标志物相对应的各器官的生物标志物的测量值进行比较,以获取指示其间的差的值作为数据。这里,术语“与
…
相对应”是指器官和生物标志物相同或是相同类型。优选地,所述差可以表示为从已经给予现有物质的个体由来的相应生物标志物的测量值与非给予个体中与上述生物标志物相对应的生物标志物的测量值的比(诸如商等)。例如,数据包括通过将从已经给予现有物质的个体由来的器官a中的生物标志物a的测量值除以从非给予个体由来的器官a中的生物标志物a的测量值而获得的商。
[0078]
在生物标志物是转录组的情况下,可以使用可通过rna-seq分析的所有rna。可替代地,可以分析rna的表达,并使用例如wgcna(https://labs.genetics.ucla.edu/horvath/coexpressionnetwork/rpackages/wgcn a/)将rna分割为指示与器官名和基因名
相关联的各rna的动态的数据子集(模块)。对于通过wgcna分割的各模块,可以针对各现有物质计算与1-of-k表示的皮尔逊相关系数,以选择针对各现有物质具有最高相关系数绝对值的模块,并且可以使用包括在所选择的模块中的各器官中的rna作为生物标志物。
[0079]
此外,在响应于现有物质的给予的生物标志物是转录组的情况下,可以使用deseq2分析来获得与尚未给予现有物质的动物的各器官中的转录组相比已经给予现有物质的动物的各器官中的转录组的变化。例如,通过htseq-count对从已经给予现有物质的动物收集到的各器官中的rna的表达水平以及从尚未给予现有物质的动物收集到的各相应器官中的基因的表达水平进行量化,以获得相应的计数数据。然后,比较相应器官和相应器官中的相应基因的表达水平。作为比较的结果,对于各器官中的各基因,输出已经给予现有物质的动物中的基因表达的变化的log2(倍数(fold))值和用作各变化的概率的指标的p值。基于log2(倍数)值,可以判断是否存在诸如转录组等的生物标志物的动态。
[0080]
生物标志物的测量值可以通过已知方法获取。在生物标志物是核酸的情况下,可以通过诸如rna-seq等的测序或定量pcr等来获取测量值。在生物标志物是糖类、脂类、糖脂、氨基酸、多酚类;趋化因子;或选自包含上述物质的最终代谢物、中间代谢物和合成原料物质的组的至少一种代谢物质等的情况下,可以通过例如质谱分析来获取测量值。在生物标志物是糖蛋白、脂蛋白、多肽或蛋白质等的情况下,可以通过例如elisa(酶联免疫吸附测定(enzyme-linked immuno sorbent assay))方法来获取测量值。用于收集从用于测量的器官由来的组织、细胞或体液的方法以及用于测量生物标志物的预处理方法也是已知的。
[0081]“测试物质”是要评价其作用的物质。测试物质可以是现有物质、现有物质的等效物、或新物质。在预测方法中,即使在尚未发现测试物质的作用与现有物质或现有物质的等效物质的作用之间的关系的情况下,也可以预测测试物质对人类的作用。另一方面,在测试物质是选自现有物质或现有物质的等效物中的一种的情况下,可以发现现有物质或现有物质的等效物的未知作用。未知作用可以是一种作用或多种作用。未知作用优选是新适应症。通过预测测试物质在人类中的新适应症,也可以实现药物重新定位。已知向非人类动物给予测试物质。另外,可以以与指示从已经给予现有物质的非人类动物收集到的一个或多于一个器官中的生物标志物的动态的数据相同的方式,获取指示从已经给予测试物质的非人类动物收集到的一个或多于一个器官中的生物标志物的动态的数据。
[0082]
2.人工智能模型的构建
[0083]
2-1.训练数据的生成
[0084]
(1)第一训练数据集的生成
[0085]
第一训练数据集由指示一个器官或多个不同器官中的各器官中的生物标志物的动态的数据集、以及指示现有物质的名称的标签构成。一个器官或多个不同器官可以从已经分别给予对人类具有已知作用的多个现有物质的相应非人类动物收集。第一训练数据集可以存储在图5中示出为数据库tr1的训练装置10的辅存储部104中。
[0086]
指示一个器官或多个不同器官中的各器官中的生物标志物的动态的数据集可以通过上述第1.(4)节中所描述的方法获取。
[0087]
指示相应器官中的生物标志物的动态的各数据项可以与关于所给予的现有物质的名称的信息、关于所收集到的器官的名称的信息、以及关于生物标志物的名称的信息等相关联。“关于名称的信息”可以是名称本身或缩写名称等的标签,或者可以是与相应名称
相对应的标签值。
[0088]
指示生物标志物的动态的数据集中所包括的各数据项用作构成后述的人工智能模型的第一训练数据集中的矩阵的元素。在生物标志物是转录组的情况下,各rna的表达水平与指示生物标志物的动态的数据集中所包括的数据相对应,并且用作构成第一训练数据集的矩阵的元素。例如,在生物标志物是转录组的情况下,可以使用通过deseq2分析所获得的各现有物质的log2(倍数)值作为第一训练数据集的各元素。
[0089]
第一训练数据集的示例如上述第1.(1)节和图3(a)中所示。
[0090]
作为指示生物标志物的动态的数据集,生物标志物的测量值可以直接用作第一训练数据集的元素,或者可以在用作第一训练数据集的元素之前经过标准化或降维等。标准化方法的示例例如是对指示表达差的数据进行变换,使得平均值为0且方差为1。标准化中的平均值可以是各器官中的平均值、各基因中的平均值或所有数据的平均值。另外,可以通过诸如主成分分析等的统计处理来实现降维。进行统计处理情况下的母群体可以针对各器官、各基因或所有数据来设置。例如,在生物标志物是转录组的情况下,仅有具有相对于通过deseq2分析所获得的各现有物质的log2(倍数)值不大于预定值的p值的基因才可以用作第一训练数据集的元素。例如,预定值可以是10-3
或10-4
。优选为10-4
。
[0091]
第一训练数据集中所包括的指示所给予的预定现有物质的相应名称的标签可以是物质本身的名称或者可以被编码。
[0092]
可以响应于现有物质的更新或指示新生物标志物的动态的数据的添加,更新第一训练数据集。
[0093]
(2)第二训练数据集的生成
[0094]
如上述第1.(1)节和图3(b)中所示,通过将指示为了生成第一训练数据集而向非人类动物给予的多个预定现有物质的相应名称的标签与指示针对多个预定现有物质中的各预定现有物质所报告的适应症的标签相关联,来生成第二训练数据集。对于现有物质的适应症,通过针对各现有物质对指示例如现有物质的名称的单词进行搜索,可以从外部数据库(诸如上述第1.(4)节中所述的faers、dailymed的所有药物标签、医学主题词表、drugs@fda或国际疾病分类等)获取与现有物质相对应的适应症的名称的标签。每个现有物质可能存在一种适应症或两种或多于两种适应症。在每个现有物质存在两种或多于两种适应症的情况下,两种或多于两种适应症构成第二训练数据集。可以通过对存储在数据库中的数据集进行文本提取、自然语言处理、数字化处理或图像分析处理等来获取指示针对多个预定现有物质中的各预定现有物质所报告的适应症的标签。例如,在将外部数据库中所存储的、指示与为了生成第一训练数据集而向非人类动物给予的相应现有物质相对应的相应适应症的名称的标签作为文本中的插入进行登记的情况下,可以在提取与作用相对应的文本之前通过自然语言处理对所登记的文本进行语法分析、单词分割或语义分析等。
[0095]
(3)第三训练数据集的生成
[0096]
如上述第1.(1)节和图3(c)中所述,第三训练数据是如下的数据集,在该数据集中,指示如图3(b)所示的针对为获取第一训练数据集而给予的多个预定现有物质中的各预定现有物质所报告的适应症的标签与关于对应于这些适应症中的各适应症所报告的不良事件的信息相关联。对于针对多个预定现有物质中的各预定现有物质所报告的适应症,通过针对各现有物质对例如现有物质的名称的单词进行搜索,可以从外部数据库(诸如
faers、dailymed的所有药物标签、医学主题词表、drugs@fda或国际疾病分类等)获取与现有物质相对应的适应症的名称的标签。通过对指示适应症名的标签进行搜索,可以从外部数据库(诸如faers或clinicaltrials.gov等)获取指示与这些适应症中的各适应症相对应地报告的不良事件的标签。另外,在将指示适应症或不良事件的名称的标签作为文本中的插入进行登记的情况下,可以在提取与作用相对应的文本之前通过自然语言处理对所登记的文本进行语法分析、单词分割或语义分析等。
[0097]
不良事件的发生频率可以通过上述第1.(4)节中所描述的方法计算。
[0098]
(4)第四训练数据集的生成
[0099]
如上述第1.(1)节和图3(d)中所述,通过将针对与指示为了获取第一训练数据而给予的现有物质的名称的标签相对应的适应症所报告的不良事件的发生频率(图3(c)所示的第二列和后续列中的不良事件的发生频率)代入到指示第一训练数据集中所包括的药物名的标签的部分(指示药物名的图3(a)的第一列)中,来生成第四训练数据集。
[0100]
2-2.训练数据向人工智能模型的输入
[0101]
人工智能模型不受限制,只要能够解决与本发明相关联的问题即可。在本实施例中,优选使用可以进行关联预测(link prediction)的人工智能模型。这种人工智能模型的示例包括one-class svm(单类支持向量机)。
[0102]
使用利用one-class svm进行关联预测的情况作为示例来说明输入第四训练数据的示例。要输入到one-class svm中的数据作为第四训练数据集被输入到one-class svm中,该第四训练数据集是通过利用下式的核函数将第一训练数据集与第三训练数据集相关联而获得的:
[0103]
k(gad1,gbd2)=《ga,gb》《d1,d2》
[0104]
这里,《
·
,
·
》表示对各向量进行缩放使得12范数等于1、并且取这两个经缩放向量之间的内积的运算符。
[0105]
作为one-class svm,例如可以使用来自python的“scikit-learn”包,其中参数nu=0.1。
[0106]
2-3.用于训练人工智能模型的系统
[0107]
图4(a)示出训练系统50的硬件结构。训练系统50包括测量部30和训练装置10,其中测量部30是用于获取生物标志物的测量数据的下一代测序器等。训练装置10可以通过无线或有线网络可通信地连接到测量部30,或者可以经由诸如cd-r等的存储介质获取测量部30所获取的数据。
[0108]
(1)用于训练人工智能模型的装置
[0109]
例如,可以使用训练装置10(以下也可以称为“装置10”)来执行人工智能模型的训练。
[0110]
在装置10及装置10中的处理的说明中,对于与上述第1.节和第2-1.节中描述的术语共通的术语,这里并入上述说明。
[0111]
装置10至少包括处理部101和存储部。存储部由主存储部102和/或辅存储部104构成。
[0112]
图5示出装置10的硬件结构。装置10可以连接到输入部111、输出部112和存储介质113。装置10还可以连接到测量部30,其中测量部30是下一代测序器或质谱仪等。另外,装置
10可以可通信地连接到外部数据库60,诸如faers、dailymed的所有药物标签、医学主题词表、drugs@fda、国际疾病分类或clinicaltrials.gov等。
[0113]
在装置10中,处理部101、主存储部102、rom(只读存储器)103、辅存储部104、通信接口(i/f)105、输入接口(i/f)106、输出接口(i/f)107和媒介接口(i/f)108通过总线109连接以进行互相数据通信。
[0114]
处理部101由cpu或mpu等构成。处理部101中的处理可以由gpu辅助。处理部101执行辅存储部104或rom 103中所存储的计算机程序,并处理所获取的数据,由此装置10起作用。处理部101获取如上述第1.节中所述的指示从已经给予现有物质的非人类动物收集到的多个不同器官中的生物标志物的动态的数据集、以及该现有物质对人类的已知作用作为训练数据。另外,处理部101使用这两种类型的训练数据来训练人工智能模型。
[0115]
rom 103由掩模rom、prom、eprom或eeprom等构成,并存储由处理部101执行的计算机程序以及由其使用的数据。rom 103存储当装置10启动时由处理部101执行的引导程序以及与装置10的硬件的操作相关的程序和设置等。
[0116]
主存储部102由诸如sram或dram等的ram(随机存取存储器)构成。主存储部102用于读出存储在rom 103和辅存储部104中的计算机程序。当处理部101执行这些计算机程序时,主存储部102也用作工作空间。主存储部102暂时存储经由网络获取的训练数据等以及由辅存储部104读出的人工智能模型的功能等。
[0117]
辅存储部104由硬盘、诸如闪速存储器等的半导体存储元件、或光盘等构成。在辅存储部104中,存储处理部101所要执行的各种计算机程序以及用于执行计算机程序的各种设置数据。具体地,辅存储部104以非易失性方式存储操作软件(os)1041、训练程序tp、人工智能模型数据库ai1、用于存储第一训练数据集的数据库tr1、用于存储第二训练数据集的数据库tr2、用于存储第三训练数据集的数据库tr3。训练程序tp与操作软件(os)1041协作进行如后所述的用于训练人工智能的处理。
[0118]
通信i/f 105由诸如usb、ieee1394或rs-232c等的串行接口、诸如scsi、ide或ieee1284等的并行接口、以及由d/a转换器或a/d转换器等构成的模拟接口、以及网络接口控制器(nic)等构成。通信i/f 105用作通信部105,并且在处理部101的控制下从测量部30或其他外部装置接收数据,并且在必要时将存储在装置10中或由装置10生成的信息发送到测量部30或外部,或者显示该信息。通信i/f 105可以经由网络与测量部30或其他外部装置(未示出,例如其他计算机或云系统)通信。
[0119]
输入i/f 106由诸如usb、ieee1394或rs-232c等的串行接口、诸如scsi、ide或ieee1284等的并行接口、以及由d/a转换器或a/d转换器等构成的模拟接口等构成。输入i/f 106接受来自输入部111的字符输入、点击或声音输入等。所接受的输入存储在主存储部102或辅存储部104中。
[0120]
输入部111由触摸面板、键盘、鼠标、手写板或麦克风等构成,并向装置10进行字符输入或声音输入。输入部111可以在外部连接到装置10,或者可以与装置10集成。
[0121]
输出i/f 107例如由与输入i/f 106相同的接口构成。输出i/f 107将处理部101所生成的信息输出到输出部112。输出i/f 107将由处理部101生成并存储在辅存储部104中的信息输出到输出部112。
[0122]
输出部112例如由显示器或打印机等构成,并显示从测量部30发送的测量结果、装
置10中的各种操作窗口、各项训练数据和人工智能模型等。
[0123]
媒介i/f 108读出例如存储在存储介质113中的应用软件等。所读出的应用软件等存储在主存储部102或辅存储部104中。另外,媒介i/f 108将处理部101所生成的信息写入存储介质113。媒介i/f 108将由处理部101生成并存储在辅存储部104中的信息写入存储介质113。
[0124]
存储介质113由软盘、cd-rom或dvd-rom等构成。存储介质113通过软盘驱动器、cd-rom驱动器或dvd-rom驱动器等连接到媒介i/f 108。用于使计算机执行操作的应用程序等可以存储在存储介质113中。
[0125]
处理部101可以经由网络获取装置10的控制所需的应用软件和各种设置,而不是从rom 103或辅存储部104中读取它们。还可以将应用程序存储在网络上的服务器计算机的辅存储部中,并且装置10访问该服务器计算机以下载计算机程序并将其存储在rom 103或辅存储部104中。
[0126]
另外,在rom 103或辅存储部104中,例如安装了提供图形用户界面环境的操作系统,诸如由美国微软公司制造和销售的windows(商标)。根据第二实施例的应用程序将在操作系统上操作。换句话说,装置10可以是个人计算机等。
[0127]
(2)用于训练人工智能模型的处理
[0128]
参考图6,说明用于通过训练程序tp来训练人工智能模型的处理的流程。
[0129]
处理部101接受操作者通过输入部111输入的用于开始处理的命令,并且在步骤s1中从存储在辅存储部104中的第一训练数据集数据库tr1、第二训练数据集数据库tr2、第三训练数据集数据库tr3分别获取第一训练数据集、第二训练数据集和第三训练数据集。
[0130]
接着,处理部101接受操作者通过输入部111输入的用于开始第四训练数据集的生成的命令,并且在步骤s2中生成第四训练数据集。
[0131]
接着,处理部101接受操作者通过输入部111输入的用于输入第四训练数据集的命令,并且在步骤s3中将第四训练数据集输入到人工智能模型ai1中,以训练人工智能模型。
[0132]
处理部101将经训练的人工智能模型存储在辅存储部104中。
[0133]
从一个步骤到另一步骤的转变可以根据操作者所输入的命令来进行,或者可以通过前一步骤的完成来触发,使得处理部101可以自动进行转变。
[0134]
在训练处理中,对于与上述第1.节和第2-1.节中描述的术语和说明共通的术语和说明,这里并入上述说明。
[0135]
3.人工智能模型对适应症的预测
[0136]
3-1.测试数据的生成
[0137]
(1)第一测试数据集的生成
[0138]
第一测试数据集是指示一个器官或多个不同器官中的各器官中的生物标志物的动态的数据集,并且可以从与已经获取第一训练数据的一个器官或多个不同器官相对应的一个或多于一个器官获取。指示相应器官中的生物标志物的动态的数据集可以通过如上述第1.(4)节中所述的方法以与指示用作第一训练数据的生物标志物的动态的数据集相同的方式获取。
[0139]
(2)第二测试数据集的生成
[0140]
如上述第1.(2)节所述,第二测试数据是多个已知适应症的标签与关于与该多个
已知适应症中的各适应症相对应地报告的不良事件的信息相关联的数据集。通过对指示适应症名的标签进行搜索,可以从外部数据库(诸如faers或clinicaltrials.gov等)获取多个已知适应症的标签以及指示与这些适应症中的各适应症相对应地报告的不良事件的标签。另外,在将指示适应症或不良事件的名称的标签作为文本中的插入进行登记在的情况下,可以在提取与作用相对应的文本之前通过自然语言处理对所登记的文本进行语法分析、单词分割或语义分析等。
[0141]
不良事件的发生频率可以通过上述第1.(4)节中所描述的方法计算。
[0142]
3-2.预测系统1
[0143]
图4(a)示出预测系统51的硬件结构。预测系统51包括测量部30和预测装置20,其中测量部30是用于获取生物标志物的测量数据的下一代测序器等。预测装置20可以经由无线或有线网络连接到测量部30,或者可以经由诸如cd-r等的存储介质获取测量部30所获取的数据。
[0144]
(1)适应症预测装置
[0145]
例如,可以使用预测装置20(以下可以简称为“装置20”)来实现适应症的预测。
[0146]
在装置20及装置20中的处理的说明中,对于与上述第1.节和第2-1.节中描述的术语共通的术语,这里并入上述说明。
[0147]
图7示出预测装置20(以下也可以称为“装置20”)的硬件结构。装置20至少包括处理部201和存储部。存储部由主存储部202和/或辅存储部204构成。装置20可以连接到输入部211、输出部212和存储介质213。另外,装置20可以连接到测量部30,其中测量部30是下一代测序器或质谱仪等。
[0148]
在装置20中,处理部201、主存储部202、rom(只读存储器)203、辅存储部204、通信接口(i/f)205、输入接口(i/f)206、输出接口(i/f)207和媒介接口(i/f)208通过总线209连接以进行互相数据通信。
[0149]
由于装置20的基本硬件结构与装置10的基本硬件结构相同,因此这里并入上述第2-3.(1)节中的说明。通信接口205用作通信部205。
[0150]
然而,在装置20的辅存储部204中,以非易失性方式存储操作软件(os)2041、预测程序pp、经训练的人工智能模型ai2、用于存储第一测试数据集的数据库ts1和用于存储第二测试数据集的数据库ts2,来代替操作软件(os)1041、训练程序tp、人工智能模型ai1,用于存储第一训练数据集的数据库tr1、用于存储第二训练数据集的数据库tr2和用于存储第三训练数据集的数据库tr3。预测程序pp与操作软件(os)2041协作进行如后所述的适应症预测处理。
[0151]
(2)用于预测适应症的处理
[0152]
参考图8,说明用于通过预测程序pp来预测适应症的处理的流程。
[0153]
处理部201接受操作者通过输入部211输入的用于开始处理的命令,并且在步骤s51中获取存储在辅存储部204中的第一测试数据集和第二测试数据集。
[0154]
接着,处理部201接受操作者通过输入部211输入的用于开始预测的命令,并且在步骤s52中将第一测试数据集数据库ts1、第二测试数据集数据库ts2、第一测试数据集和第二测试数据集输入到经训练的人工智能模型ai2中,以预测测试物质的适应症。
[0155]
此时,经训练的人工智能模型ai2逐一判断所关注的测试物质是否对作为第二测
试数据单独输入的所有适应症有效。具体地,经训练的人工智能模型ai2在lp问题中判断所关注的药物与单个适应症之间是否存在关联。
[0156]
接着,处理部201将结果存储在存储部中。如果测试物质对特定适应症有效,则处理部201根据经训练的人工智能模型ai2导出的结果是标签“1”,并且如果测试物质对特定适应症无效,则该导出的结果是标签
“‑
1”。
[0157]
换句话说,标有“1”的适应症是针对测试物质预测的适应症。
[0158]
此外,在人工智能模型是one-class svm的情况下,计算指示预测的可靠性的决策函数值。在输出许多适应症作为预测结果的情况下,可以预测出较高值指示更有可能的适应症。另外,在输出许多适应症作为预测结果的情况下,可以以相同的方式使用指示在给予具有与作为测试物质的所关注测试物质类似的作用机理的药物之后收集到的一个或多于一个器官中的转录组的动态的数据进行预测。然后,通过对所关注的测试物质的预测结果和具有类似作用机理的其他测试物质的预测结果之间的比较而发现的这两者共通的适应症可以用作预测结果。
[0159]
3-3.预测系统2
[0160]
图4(b)示出预测系统400的结构。
[0161]
预测系统400可通信地连接到测量部30、训练装置10、预测装置20以及用于发送指示生物标志物的动态的数据集的服务器装置40。训练装置10和预测装置20经由服务器装置40获取由测量部30获取的数据。
[0162]
(1)服务器装置
[0163]
关于服务器装置40(在下文中可以简称为“装置40”),对于与上文第1.节和第2-1.节中描述的术语共通的术语,这里并入上述说明。
[0164]
图9示出服务器装置40(也可以称为“装置40”)的硬件结构。装置40至少包括处理部401和存储部。存储部由主存储部402和/或辅存储部404构成。装置40可以连接到输入部411、输出部412和存储介质413。另外,装置40可以通过有线或无线网络可通信地连接到测量部30,其中测量部30是下一代测序器或质谱仪等。
[0165]
在装置40中,处理部401、主存储部402、rom(只读存储器)403、辅存储部404、通信接口(i/f)405、输入接口(i/f)406、输出接口(i/f)407和媒介接口(i/f)408通过总线409连接以进行互相数据通信。
[0166]
由于装置40的基本硬件结构与装置10的基本硬件结构相同,因此这里并入上述第2-3.(1)节中的说明。通信接口405用作通信部405。
[0167]
然而,在装置40的辅存储部404中,以非易失性方式存储操作软件(os)4041、用于存储第一测试数据集的数据库ts1,来代替操作软件(os)1041、训练程序tp、人工智能模型ai1、用于存储第一训练数据集的数据库tr1、用于存储第二训练数据集的数据库tr2和用于存储第三训练数据集的数据库tr3。
[0168]
(2)预测系统2的操作
[0169]
参考图10,说明预测系统的操作。
[0170]
这里,说明从测量部30对生物标志物的测量值的获取到预测结果的输出的顺序流程。
[0171]
在步骤s81中,测量部30获取已经给予现有物质的非人类动物的各器官中的生物
标志物的测量值。测量部30中的测量值的获取可以响应于操作者对用于开始测量的命令的输入而进行。在步骤s82中,测量部30将获取的测量值发送至服务器装置40。发送处理可以响应于操作者对用于开始发送的命令的输入而进行。
[0172]
在步骤s83中,服务器装置40的处理部401经由通信i/f 405获取测量值。此时,通信i/f 405用作通信部。
[0173]
在步骤s84中,响应于操作者通过训练装置10的输入部111输入的用于开始获取测量值的命令,训练装置10的处理部101将用于开始发送测量值的信号从通信i/f 105发送到服务器装置40。服务器装置40的处理部401经由通信i/f 405接受用于开始发送测量值的输入,并且开始从通信i/f 405发送测量值。此时,通信i/f 105和通信i/f 405分别用作通信部105和通信部405。
[0174]
在步骤s85中,训练装置10的处理部101经由通信i/f 105从外部数据库60获取与向非人类动物给予的现有物质的适应症以及与该适应症相对应的不良事件有关的信息。
[0175]
另外,在步骤s84中,训练装置10的处理部101经由通信i/f 105获取从服务器装置40发送的测量值(步骤s86),并将测量值存储在训练装置10的存储部中。步骤s86可以在步骤s85之前进行。
[0176]
接着,在图10的步骤s87中,训练装置10的处理部101根据图6的步骤s1中所示的处理来生成第一训练数据集、第二训练数据集和第三训练数据集。这里并入图6中的步骤s1的说明。
[0177]
接着,在图10的步骤s88中,训练装置10的处理部101根据图6的步骤s2中所示的处理来从第一训练数据集、第二训练数据集和第三训练数据集生成第四训练数据集。这里并入图6中的步骤s2的说明。
[0178]
接着,在图10的步骤s89中,训练装置10的处理部101根据图6的步骤s3至s4中所示的处理将第四训练数据集输入到人工智能模型中,以训练该人工智能模型,并将经训练的人工智能模型存储在存储部中。这里并入图6中的步骤s3至s4的说明。
[0179]
在图10的步骤s90中,在接受来自预测装置20的用于开始发送人工智能模型的命令之后,训练装置10的处理部101经由通信i/f 105向预测装置20发送经训练并存储的人工智能模型。此时,通信i/f 105用作通信部105。
[0180]
接着,在步骤s91中,测量部30获取已经给予测试物质的非人类动物的各器官中的生物标志物的测量值。测量部30中的测量值的获取可以响应于操作者对用于开始测量的命令的输入而进行。在步骤s92中,测量部30将获取的测量值发送至服务器装置40。发送处理可以响应于操作者对用于开始发送的命令的输入而进行。
[0181]
在步骤s93中,服务器装置40的处理部401经由通信i/f 405获取测量值。此时,通信i/f 405用作通信部405。
[0182]
在步骤s94中,响应于操作者通过预测装置20的输入部211输入的用于开始获取测量值的命令,预测装置20的处理部201将用于开始发送测量值的信号从通信i/f 205发送到服务器装置40。服务器装置40的处理部401经由通信i/f 405接受用于开始发送测量值的输入,并且开始从通信i/f 405发送测量值。此时,通信i/f 205和通信i/f 405用作通信部。预测装置20的处理部201经由通信i/f 205获取测量值,并将测量值存储在预测装置20的存储部中。随后,预测装置20的处理部201生成第一测试数据集。如上述第2-4.(1)节所述地生成
第一测试数据集。
[0183]
接着,在步骤s95中,预测装置20的处理部201经由通信i/f 205向训练装置10发送用于开始发送人工智能模型的命令。当接受来自预测装置20的用于开始发送人工智能模型的命令时,训练装置10的处理部101经由训练装置10的通信i/f 105向预测装置20发送经训练的人工智能模型。预测装置20经由通信i/f 205获取经训练的人工智能模型。步骤s95可以在步骤s94之前进行。
[0184]
接着,在步骤s96中,预测装置20的处理部201将在步骤s94中生成的第一测试数据和存储在存储部中的第二测试数据输入到在步骤s95中获取的经训练的人工智能模型ai2中,并且根据图8的步骤s52预测测试物质对人类的作用。在步骤s97中,预测装置20的处理部201输出结果。可替代地,在图10的步骤s94至s97中,预测装置20的处理部201可以预测与现有物质的新适应症相关的预测结果。
[0185]
(3)用于构建预测系统的方法
[0186]
用于构建预测系统的方法包括准备训练装置10和预测装置20的步骤。构建方法还可以包括准备已经给予现有物质的非人类动物的一个或多于一个器官中的生物标志物的测量值、或已经给予测试物质的非人类动物的一个或多于一个器官中的生物标志物的测量值的步骤。
[0187]
4.计算机程序
[0188]
4-1.训练程序
[0189]
训练程序tp是使计算机执行包括上文结合人工智能模型的训练所述的图6的步骤s1至s4的处理以使该计算机用作训练装置10的计算机程序。
[0190]
4-2.预测程序
[0191]
预测程序pp是使计算机执行包括上文结合测试物质的作用的预测所述的步骤s51至s53的处理以使该计算机用作预测装置20的计算机程序。
[0192]
5.存储有计算机程序的存储介质
[0193]
本节涉及存储有上述计算机程序的存储介质。计算机程序存储在诸如硬盘、诸如闪速存储器等的半导体存储元件、或光盘等的存储介质中。另外,计算机程序可以存储在可经由诸如云服务器等的网络连接的存储介质中。计算机程序可以是可下载形式的或存储在存储介质中的程序产品。
[0194]
存储介质中的程序的存储格式不受限制,只要如上所述的装置能够读取该程序即可。在存储介质中的存储优选以非易失性方式。
[0195]
6.变形例
[0196]
在上述第2.节中,示出训练装置10和预测装置20是不同计算机的实施例。然而,一个计算机可以进行人工智能模型的训练和预测。
[0197]
在本说明书中,附至硬件的相同附图标记表示相同的部分或相同的功能。
[0198]
示例
[0199]
以下示出实施例以更详细地说明本发明。然而,本发明不应被解释为限于以下实施例。
[0200]
以下动物实验经karydo therapeutix,inc.的伦理委员会批准而进行。
[0201]
实验示例i.给予药物的小鼠中的基因表达分析
[0202]
i-1.给予药物的小鼠的准备及基因表达分析
[0203]
1.药物的给予
[0204]
(1)阿仑膦酸钠
[0205]
将阿仑膦酸钠三水合物(alendronate sodium salt trihydrate)(wako)在pbs(nacalai tesque,inc.)中的溶解以1.0mg/kg的剂量经皮下注射给11周龄雄性c57bl/6n小鼠,每三天或四天一次,持续八天。药物针对每次给予来新准备。在药物给予后的第8天下午收集各器官。
[0206]
(2)对乙酰氨基酚(acetaminophen)
[0207]
10周龄雄性c57bl/6n小鼠禁食12小时,期间允许小鼠自由饮水。紧接在禁食期后,将溶解在生理盐水(otsuka pharmaceutical co.,ltd.)中的对乙酰氨基酚(wako)以单次300mg/kg的剂量经腹腔内给予至小鼠。在给予后,允许小鼠自由正常饮食。在中午之前进行给予,并且在给予后2小时收集器官。
[0208]
(3)阿立哌唑(aripiprazole)
[0209]
将阿立哌唑(sigma-aldrich)在0.5%(w/v)羧甲基纤维素(carboxymethyl cellulose)400溶液(wako)中的溶解以单次0.3mg/kg的剂量经腹腔内给予至11周龄雄性c57bl/6n小鼠。在下午给予药物,并且在两小时后收集器官。
[0210]
(4)阿赛那平(asenapine)
[0211]
将马来酸阿赛那平(asenapine maleate)(chemscene)在生理盐水中的溶解以单次0.3mg/kg的剂量经皮下给予至11周龄雄性c57bl/6n小鼠。在下午给予药物,并且在两小时后收集器官。
[0212]
(5)顺铂(cisplatin)
[0213]
将顺铂(bristol-myers squibb)以单次20mg/kg的剂量经腹腔内给予至11周龄雄性c57bl/6n小鼠。在药物给予后第三天下午收集器官。
[0214]
(6)氯氮平(clozapine)
[0215]
将氯氮平(sigma-aldrich)以单次0.3mg/kg的剂量经皮下给予至11周龄雄性c57bl/6n小鼠。首先将氯氮平溶解在乙酸中,然后用生理盐水稀释,并用1m naoh调节至ph 6。在药物给予后两小时的下午收集器官。
[0216]
(7)多西环素(doxycycline)
[0217]
9周龄雄性c57bl/6n小鼠用含5%蔗糖(nacalai tesque,inc.)和2mg/ml的多西环素盐酸盐n-水合物(wako)的ro水喂养两周。含有药物的ro水每周用新的ro水更换。在药物给予后第13天的下午收集器官。阴性对照组用含5%蔗糖(nacalai tesque,inc.)的ro水喂养。
[0218]
(8)恩格列净(empagliflozin)
[0219]
将溶解在0.5%羧甲基纤维素中的恩格列净(toronto research chemical)以每日10mg/kg的剂量强制地经口给予至10周龄雄性c57bl/6n小鼠,持续两周。针对每次给予新准备药物。在药物给予开始后的第14天下午收集器官。
[0220]
(9)来那度胺(lenalidomide)
[0221]
将来那度胺(wako)溶解在含有0.5%羧甲基纤维素和0.25%tween-80(nacalai tesque,inc.)的溶液中,并将该溶液以每日50mg/kg的剂量强制地经口给予至8周龄雄性
reads/run,nextseq 500/550high output kit v2.5,cat#20024906)来进行。
[0242]
使用从已经给予各药物的小鼠收集到的各器官的差异基因表达数据作为机器学习框架的各药物的特征。根据专利文献1中所描述的方法进行rna-seq数据处理(转录产物的定位和计数)。
[0243]
使用tophat2对mm10进行小鼠基因组的定位。通过deseq2(1.22.1)来识别药物给予组和阴性对照组(多西环素和来那度胺给予组)或wt小鼠组(已经给予除多西环素和来那度胺以外的药物的对照组)的各器官中的差异基因表达。药物给予组、阴性对照组和wt小鼠组各自分别用n=2进行分析。
[0244]
3.示例
[0245]
利用使用one-class svm的关联预测(lp)来构建人工智能模型,以预测药物的适应症。
[0246]
3-1.训练
[0247]
(1)第一训练数据
[0248]
作为各药物的特征,选择示出各器官中的表达变化p《0.0001的基因。使器官、从所有器官(24器官框架)或单个器官(单器官框架)中选择的所有基因的log2倍数值与器官名的组合、以及为了获取基因表达数据而给予的药物的名称的标签成为集合,并用作第一训练数据。
[0249]
(2)第二训练数据
[0250]
使上述第1.节中的给予小鼠的药物名的标签和各药物的适应症的标签成为集合,并用作第二次训练数据。与药物名相对应的适应症名与fda不良事件报告系统(faers:https://open.fda.gov/data/faers/)一致。
[0251]
(3)第三训练数据
[0252]
从faers(https://www.fda.gov/drugs/guidancecomplianceregulatoryinformation/surveil lance/adversedrugeffects/ucm082193.htm)下载从2014q2至2018q1的不良事件报告数据。从报告数据中提取指示与上述第1.节中的给予小鼠的各药物的适应症名相对应的不良事件的单词。将提取的一个单词视为报告的一个不良事件,并且各不良事件的发生频率(%)分别通过下式计算:(针对一种药物的适应症名报告一个不良事件的案例数)/(针对一种药物的适应症名所报告的所有不良事件的数量)。
[0253]
(4)第四训练数据
[0254]
当药物名定义为例如a和b时,ga和gb分别指示在给予药物a和b时的24个器官中观察到的转录组的模式(第一训练数据集)。另外,当药物a的适应症和药物b的适应症分别由“1”和“2”表示、并且针对适应症1所报告的不良事件(ae)的元素由i、ii
…
n表示时,适应症1的向量被表示为d1=(d
1i
,d
1ii
,
…
,d
1n
)和d2=(d
2i
,d
2ii
,
…
,d
2n
)(第三训练数据集)。另外,由于第二训练数据集包括指示药物a的名称的标签和指示适应症1的名称的标签的集合、以及指示药物b的名称的标签和指示适应症2的名称的标签的集合,因此这些集合可以分别表示为gad1和gbd2(第二训练数据集)。这里,在faers中,当具有适应症1的患者服用的药物a的记录数量超过10时,将适应症视为阳性(有适应)。
[0255]
(5)one-class svm
[0256]
作为要输入到one-class svm中的数据,通过利用下面的核函数将第一训练数据
集与第三训练数据集相关联而获得的第四训练数据集被输入到one-class svm中。
[0257]
k(gad1,gbd2)=《ga,gb》《d1,d2》
[0258]
这里,《
·
,
·
》表示对各向量进行缩放使得12范数等于1、并且取这两个经缩放向量之间的内积的运算符。
[0259]
作为one-class svm,使用来自python的p
‘
scikit-learn’包,其中参数nu=0.1。
[0260]
3-2.预测
[0261]
在经训练的one-class svm中输入响应于所关注药物的给予的24个器官中的转录组的模式(第一测试数据)、以及登记在faers中的[指示所有适应症的名称的标签]和[与适应症相对应的不良事件的名称和不良事件的发生频率的组合(gd)],以使经训练的one-class svm判断所关注的药物是否对所有适应症单独有效。具体地,在lp问题中,使经训练的one-class svm判断所关注的药物与单个适应症之间是否存在关联。当所关注的药物对特定适应症有效时,svm返回标签“1”;并且当所关注的药物对特定适应症无效时,svm返回标签
“‑
1”。
[0262]
3-3.示例1
[0263]
在示例1中,在假设上述第1.节中给予的药物之一的适应症未知的情况下进行预测。换句话说,首先使用与除上述第1.节中给予的药物之一以外的14种药物有关的数据作为训练数据来训练one-class svm。此后,使用所排除的药物作为所关注的药物,并且将响应于给予所关注的药物的转录组的模式作为第一测试数据连同第二测试数据一起输入在经训练的one-class svm中以预测适应症。结果在图11中示出。在图11中,tn表示真阴性,tp表示真阳性,fn表示假阴性,以及fp表示真阳性。真阴性指示“非适应症”能够被预测为“非适应症”的项目数,并且真阳性指示“是适应症”能够被预测为“是适应症”的项目数。假阴性指示“是适应症”被预测为“非适应症”的项目数,并且假阳性指示“非适应症”被预测为“是适应症”的项目数。准确率评分是指示预测的准确率的评分。召回率评分是在预测为“是适应症”的情况下的覆盖率。精确率评分指示在预测为“是适应症”的情况下的可靠性。
[0264]
所有15种药物的准确率评分较高(》0.78)。这些结果指示78%以上的预测适应症或非适应症已被实际报告或尚未报告。另外,召回率评分示出阿仑膦酸钠、阿立哌唑、阿塞那平、氯氮平、恩格列净、鲁拉西酮、奥氮平、依洛尤单抗、利塞膦酸、索非布韦和特立帕肽的较高值(》0.8)。召回率评分指示,可以预测针对这些药物已报告的80%以上的适应症。多西环素的召回率评分为0.527,这指示针对该药物预测了所报告的适应症中的约50%。只有对乙酰氨基酚(apap)示出高精确率评分(1.000),其他均示出低精确率评分(《0.35)。顺铂和来那度胺的精确率评分和f主评分无法计算,因为这两者均示为0tp和0fn。许多药物的精确率评分如此低的原因被认为主要是因为与tp相比,存在更多的fp。
[0265]
这些结果指示,本发明的预测方法可用于预测没有已知适应症的新物质的适应症。
[0266]
3-4.示例2
[0267]
评价本发明是否可用于探索已知物质的新适应症,即所谓的药物重新定位。使用上述第1.节中列出的所有15种药物的数据来训练人工智能,以预测单个药物的适应症。结果在图12中示出。图中的符号与图11中的符号相同。
[0268]
作为结果,对于所有药物,tp数增加,fn数减少。召回率评分也提高。此外,所有药
物的准确率评分和召回率评分均提高,范围在0.770-1.000之间。这些结果指示,报告的适应症和未报告的适应症这两者均可以77%以上的概率捕获。由于fn的数量较大,因此所有药物的精确率评分都很低。在图12中,fp指示先前尚未报告的潜在新适应症。由于fp的数量相对大,因此当需要缩小候选时,可以通过计算fp中的各适应症的决策函数值并对各药物的各适应症进行排序来缩小候选。图13示出阿仑膦酸钠的决策函数值的示例。另外,对于已知具有类似作用机理的药物(例如,阿仑膦酸钠和利塞膦酸,或阿立哌唑和氯氮平)共通并且被预测为fp的适应症被认为很有可能作为重新定位出的适应症。
[0269]
这些结果表明本发明的预测方法对于药物重新定位也是有用的。
[0270]
附图标记列表
[0271]
10/训练装置
[0272]
20/预测装置
[0273]
40/服务器装置
[0274]
101/处理部
[0275]
201/处理部
[0276]
401/处理部
[0277]
400/预测系统
[0278]
105/通信部
[0279]
405/通信部。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。