技术特征:
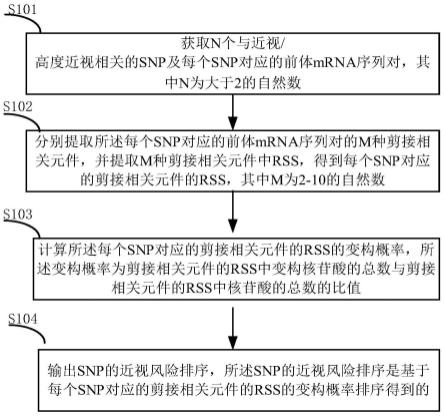
1.一种基于变构概率对近视/高度近视相关snp风险的排序方法,包括:获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对,其中n为大于2的自然数;分别提取所述每个snp对应的前体mrna序列对的m种剪接相关元件,并提取m种剪接相关元件中rss,得到每个snp对应的剪接相关元件的rss,其中m为2-10的自然数;计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率排序得到的。2.根据权利要求1所述的基于变构概率对近视/高度近视相关snp风险的排序方法,其特征在于,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;评估每个snp对应的前体mrna序列对的结构异质性,得到每个snp对应的结构异质性得分;输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率及每个snp对应的结构异质性得分的集合;可选的,所述每个snp对应的结构异质性得分为评估snp对前体mrna序列的局部结构异质影响程度,得到每个snp对应的局部结构异质性得分,优选的,通过rnasnp来评估snp对前体mrna序列的局部结构异质影响程度;可选的,所述每个snp对应的结构异质性得分为评估snp对前体mrna序列的全局结构异质影响程度,得到每个snp对应的全局结构异质性得分;优选的,通过rnasmc来评估snp对前体mrna序列的全局结构异质影响程度。3.根据权利要求2所述的基于变构概率对近视/高度近视相关snp风险的排序方法,其特征在于,所述每个snp对应的结构异质性得分包括局部结构异质性得分和/或全局结构异质性得分,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分的集合;优选的,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分,分别选择剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分排序靠前的snp组合,得到三组snp组合,并取三组snp组合的交集;优选的,所述snp的近视风险排序是基于每个snp对应的每种剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分,分别基于每种剪接相关元件的rss的变构概率、局部结构异质性得分和/或全局结构异质性得分选择排序靠前的snp组合,得到7组snp组合,并取7组snp组合的交集。4.根据权利要求1所述的基于变构概率对近视/高度近视相关snp风险的排序方法,其特征在于,所述获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对:获取snp侧翼序列,并截取所述snp上下游各n个碱基作为种子序列,其中n为15-50的自然数;
在数据库下载转录本信息作为参考序列;基于短序列比对工具,分别获得snp种子序列对应的参考序列位置;根据snp匹配的正负链及上下游片段对应的起始碱基位置信息,筛选snp定位于参考序列的片段,得到snp及其对应的前体mrna序列对;优选的,根据以下原则筛选snp定位于参考序列的片段pm:ini
up
,ini
down
分别代表snp上游,下游序列匹配到参考序列的碱基起始位置,seed
up
,seed
down
分别对应snp上下游侧翼序列,ref
forward
,ref
reverse
分别表示种子序列位于参考序列的正负链情况。5.根据权利要求1-4任意一项所述的基于变构概率对近视/高度近视相关snp风险的排序方法,其特征在于,所述m种剪接相关元件包括下列剪切相关元件中的一种或几种:5'剪接位点、3'剪接位点、分支点、多嘧啶束mrna剪接过程、剪接调节元件;优选的,所述剪接调节元件包括外显子剪接增强子(ese)、内含子剪接增强剂(ise)、外显子拼接消音器(ess)、内含子拼接消音器;可选的,采用下列方法提取所述前体mrna序列的m种剪接相关元件:基于基因组注释信息提取前体mrna序列的5'剪接位点、3'剪接位点;优选的,采用下列方法中的一种或几种提取前体mrna序列的5'剪接位点、3'剪接位点:deep splicer、splice finder、splice2deep、splicerover、deepss、spliceai;采用下列方法中的一种或几种提取前体mrna序列的分支点、多嘧啶束mrna剪接区域:svm-bpfinder、bpp、branchpointer、labranchor、rnabps;采用下列方法中的一种或几种提取前体mrna序列的剪接调节元件:hsf、protein-specific prediction of rna-binding sites based on information entropy、rbpmmap、graphprot、rna-binding protein targets、ionmf、ideep、circrna-rbp web server;可选的,所述rss包括茎(s)、发夹环(h)、内环(i)、外环(e)、凸起环(b)、多支环(m);可选的,所述提取m种剪接相关元件中rss是采用rna结构预测算法预测剪接相关元件rna的二级结构,进而采用rna motif预测算法提取二级结构中的rss;优选的,所述rna结构预测算法包括下列算法中的一种或几种:rna structure,rnafold,mfold,sfold,maxexpect;优选的,所述rna motif预测算法包括下列算法中的一种或几种:bprna,dotaligner,cmfinder,rnaz,qrna。6.根据权利要求1-4任意一项所述的基于变构概率对近视/高度近视相关snp风险的排序方法,其特征在于,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:基于snp的近视风险排序得到x个候选的snp,其中x为自然数整数,对候选的snp进行分子对接能力影响分析,所述分子对接能力影响分析是计算候选的snp对应的不含有snp的前体mrna序列分子对接评分减去含有snp的前体mrna序列分子对接评分差值比不含有snp的前体mrna序列分子对接评分的比值;可选的,所述mrna序列分子对接评分采用hdock server评分。7.根据权利要求1-4任意一项所述的基于变构概率对近视/高度近视相关snp风险的排序方法,其特征在于,所述基于变构概率对近视/高度近视相关snp风险的排序方法还包括:基于snp的近视风险排序得到x个候选的snp,其中x为自然数整数,将候选的snp映射到基因
组的基因功能元件中,所述基因功能元件包括外显子、内含子、5’utr和3’utr。8.一种基于变构概率对近视/高度近视相关snp风险的排序设备,所述设备包括:存储器和处理器;所述存储器用于存储程序指令;所述处理器用于调用程序指令,当程序指令被执行时,用于执行权利要求1-7所述的基于变构概率对近视/高度近视相关snp风险的排序方法。9.一种基于变构概率对近视/高度近视相关snp风险的排序系统,包括:获取单元,用于获取n个与近视/高度近视相关的snp及每个snp对应的前体mrna序列对,其中n为大于2的自然数;提取单元,用于分别提取所述每个snp对应的前体mrna序列对的m种剪接相关元件,并提取m种剪接相关元件中rss,得到每个snp对应的剪接相关元件的rss,其中m为2-10的自然数;计算单元,用于计算所述每个snp对应的剪接相关元件的rss的变构概率,所述变构概率为剪接相关元件的rss中变构核苷酸的总数与剪接相关元件的rss中核苷酸的总数的比值;排序单元,用于输出snp的近视风险排序,所述snp的近视风险排序是基于每个snp对应的剪接相关元件的rss的变构概率排序得到的。10.一种计算机可读存储介质,其上存储有计算机程序,其特征在于,所述计算机程序被处理器执行时实现如权利要求1-7任意一项所述的基于变构概率对近视/高度近视相关snp风险的排序方法。
技术总结
本发明涉及基于变构概率对近视/高度近视相关SNP风险的排序方法和系统。所述方法包括:获取N个与近视/高度近视相关的SNP及每个SNP对应的前体mRNA序列对;分别提取所述每个SNP对应的前体mRNA序列对的M种剪接相关元件,并提取M种剪接相关元件中RSS,得到每个SNP对应的剪接相关元件的RSS;计算所述每个SNP对应的剪接相关元件的RSS的变构概率,所述变构概率为剪接相关元件的RSS中变构核苷酸的总数与剪接相关元件的RSS中核苷酸的总数的比值;输出SNP的近视风险排序。本申请在发现近视相关SNP上具有重要应用价值。上具有重要应用价值。上具有重要应用价值。
技术研发人员:徐良德 王宏 于晓光 闻旭冉
受保护的技术使用者:温州谱希医学检验实验室有限公司
技术研发日:2022.11.08
技术公布日:2023/2/3
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。